一种基于机器学习的三维肺切片细胞分类和蛋白定量方法与流程
本发明涉及生物,尤其是指一种基于机器学习的三维肺切片细胞分类和蛋白定量方法。
背景技术:
1、以肺纤维化和肺部肿瘤为代表的肺部疾病已经成为人类健康的严重威胁。肺纤维化是以成纤维细胞增殖及大量细胞外基质聚集并伴随炎症损伤、组织结构破坏为特征的一大类肺疾病的终末期改变,是正常的肺泡组织被损坏后经过异常修复而导致的结构异常(疤痕形成)。肺纤维化是一种与衰老密切相关的疾病,在疾病的发展过程中,肺内微组织结构和细胞形态及特定蛋白表达出现明显异常。
2、对肺部组织细胞及其空间结构进行分类分析,有助于准确识别和定位细胞,分析细胞基因表达状况以及胞内基因组折叠和表达调控机制;并对染色质、蛋白相互作用进行分析;进而对疾病发病机理和药物疗效机制进行分析,为研究细胞生物学、生物医学和药物研发提供重要的支持。
3、高通量测序技术使得研究者可以在单细胞精度上解析细胞病变的发生和发展,但多组学研究的高成本让众多研究者望而却步。因此,现有组织细胞研究方法仍然集中在传统的western blot(wb)、免疫荧光(immunofluorescence,if)、免疫组化(immunohistochemistry, ihc)等低成本手段。
4、然而,基于wb、if及ihc等方法分析肺组织细胞图像信息时,一方面,由于传统图像识别和细胞分类依赖于临床病理医生的经验判断,不易标准化和广泛应用;另一方面,传统方法可能会造成肺组织空间微结构的丢失,并且其细胞分类鉴定能力受限于该目标细胞具有相应的细胞标志物。具体表现在:wb分析会造成肺内单细胞信息及空间信息的丢失,不能反应肺组织的单细胞和空间结构信息;if分析则受限于荧光通道且依赖于特定细胞标志物的存在;在蛋白定量方面,虽然流式细胞术与wb的结合可以对特定细胞中的蛋白进行定量,但在组织消化过程中仍然无法避免空间信息的丢失;而采用if分析定量胞内蛋白则会受到细胞标志物的限制,无法实现多种类型细胞的并行识别。
5、近年来,基于机器学习的图像识别也逐步被应用于疾病诊断中。然而,该类方法通常需要较大数据量和强大计算能力支持,对信息分析技术要求高,使用便捷性差,不易操作。
6、因此,临床上迫切需要成本更低、操作更简便、准确性更高的肺组织细胞分类方法和模型。
技术实现思路
1、本发明要解决的技术问题是提供一种基于机器学习的三维肺切片细胞分类和蛋白定量方法,该方法基于训练后的人工智能,可以不依赖传统的特异性细胞标志物,即可在肺切片的免疫荧光图片中,准确实现肺部包括干细胞在内的多种细胞类型的自动识别、分类及其细胞核中目标蛋白的定量。
2、为了解决上述技术问题,本发明采用如下技术方案:
3、一种基于机器学习的三维肺切片细胞分类和蛋白定量方法,包括以下步骤:
4、s1,构建肺组织标准化成像数据集;
5、s2,基于ilastik构建机器学习模型;
6、s3,模型验证,并对细胞中的目标蛋白进行定量。
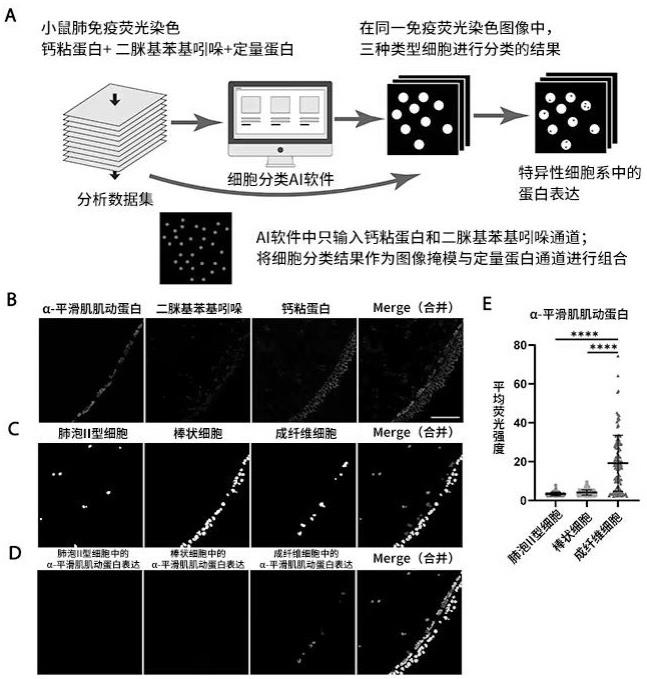
7、优选地,步骤s1中构建肺组织数据集,具体操作如下:
8、s11,对肺部的石蜡包埋切片进行免疫荧光染色:三个通道分别为e-cadherin,dapi,细胞标志物;
9、s12,使用共聚焦显微镜进行层切拍摄。
10、优选地,步骤s12显微镜拍摄的具体操作方法包括以下步骤:
11、s121,共聚焦显微镜z轴层切拍摄,保证xyz步长一致;
12、s122,保证各图片曝光程度一致;
13、s123,只保留e-cadherin和dapi通道,删除其他各细胞标记物通道;
14、s124,利用leica las x软件的auto scale给出的参照,用fiji分别对e-cadherin和dapi通道进行归一化。
15、优选地,步骤s2中构建机器学习模型,具体操作如下:
16、s21,单独提取出只包含e-cadherin和dapi通道的图片输入程序,建立细胞识别模型;
17、s22,按步骤s21操作方法分别建立at2细胞、club细胞、肌成纤维细胞的细胞识别模型;
18、s23,建立模型后,根据细胞标志物的染色结果计算识别准确率。
19、优选地,步骤s21中建立细胞识别模型的具体操作包括如下步骤:
20、s211,利用pixel classification分割细胞核与背景;
21、s212,输入只包含e-cadherin和dapi通道的图片以及第一步的分割结果,利用未输入的细胞标志物通道作为参照,标记目标细胞与其他细胞,训练模型并批量处理。
22、优选地,步骤s3中对模型进行验证:在模型建立并验证准确率后则不再需要细胞标志物通道,可以替换为其他目标蛋白;
23、对细胞中的目标蛋白进行定量,具体操作如下:
24、s31,将只包含e-cadherin和dapi通道的图片输入各个模型,分别得到同一张图片上的不同的细胞识别结果;
25、s32,将识别结果与需要定量的蛋白通道融合,使其只显示特定类型细胞中的蛋白表达;
26、s33,对目标细胞中的蛋白进行定量。
27、优选地,步骤s33中对目标细胞进行定量的具体操作包括以下步骤:
28、s331,对肺切片进行免疫荧光染色,染色要求和参数与构建模型时相同;
29、s332,去除目标蛋白通道,提取e-cadherin和dapi两个通道,按照构建模型时的方法进行标准化后,将图片输入已建立好的三个细胞分类模型,分别输出三类细胞的识别结果;
30、s333,将识别结果中的目标细胞进行提取;
31、s334,对提取的目标细胞中的目的蛋白进行提取;
32、s335,对目标细胞内的蛋白进行定量:分别使用不同细胞分选模型输出的结果与目的蛋白通道合成可以对不同细胞进行定量,从而实现同一张免疫荧光图片上不同细胞的识别和不同细胞中的蛋白定量。
33、本发明的有益效果:
34、本发明采用3d免疫荧光组织切片完整保留组织的结构分层特征和细胞组成,从而构建肺组织标准化成像数据集;并利用生物图像分析软件ilastik结合ai模型运算,构建机器学习模型——ai细胞分类器;本发明提供的方法训练后的人工智能,可以不依赖传统的特异性细胞标志物,即可在肺切片的免疫荧光图片中,准确实现肺部细胞包括干细胞在内的多种细胞类型的自动识别、分类及其细胞核中目标蛋白的定量。
35、相较于现有的基于细胞标志物法识别细胞的方法,本专利建立的ai细胞分类器不依赖于特定细胞标志物,可以在一张if图像中一次性区分多种类型的细胞,自动化程度及准确率高,可识别的肺部细胞种类更多,适合不同细胞的批量化识别、分类以及细胞信息的提取,并定量分析核内因子和表观遗传修饰。
36、即:
37、(1)3d空间信息识别能力:解决了荧光通道不足的问题,同时能够保留切片上的细胞组织空间信息。该方法能够在维持肺组织3d结构基础上,对肺部组织细胞及其空间结构进行分析,不会造成空间信息的丢失;
38、(2)自动化非标志物依赖性批量细胞分类能力:可在同一切片上对不同细胞进行分类鉴定,可以一次性分辨三种以上细胞类型;并且,基于机器学习构建的ai细胞分类器,只需要少量的图像和若干标志物,即可实现较高的分类精度;随着机器学习输入的图像更多,细胞识别的精度会更高;
39、(3)定量分析:对所分类的细胞中的蛋白、核酸,均可同步进行定量分析。
- 还没有人留言评论。精彩留言会获得点赞!