新型粉尘螨蛋白质的制作方法
本发明涉及源自粉尘螨的新型蛋白质以及该蛋白质的利用。
背景技术:
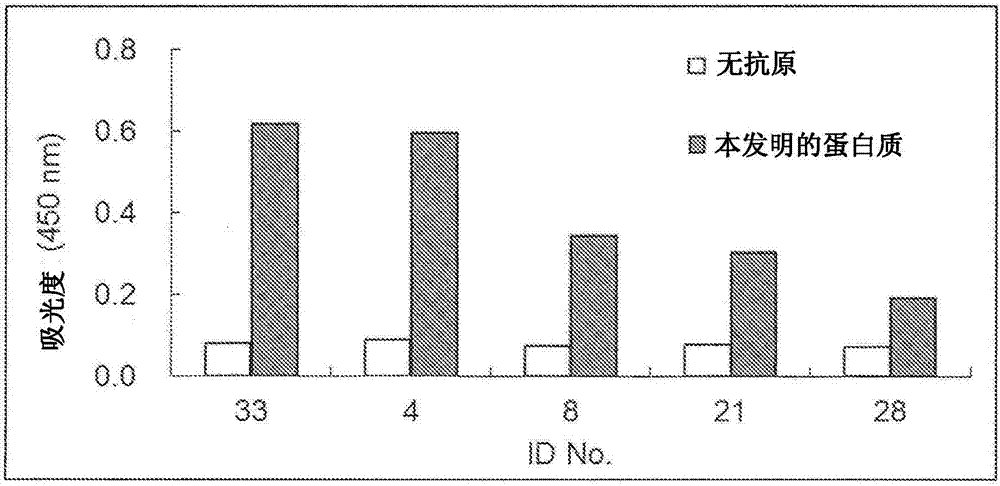
:作为过敏性疾病的代表性疾病,可以列举花粉症等的过敏性鼻炎、过敏性结膜炎、特应性支气管哮喘、特应性皮肤炎等,但近年来,日本的这些过敏性疾病的患病率日趋增加。已知特应性支气管哮喘、特应性皮肤炎的发病原因的大部分是由包括螨虫的虫体、螨虫的排泄物、霉、宠物等动物的毛等在内的室内灰尘造成的,但是已知特别是在源自室内生存的尘螨的过敏原发挥了重要的作用。(非专利文献1)。在室内生存的代表性的尘螨绝大多数由粉尘螨(dermatophagoidesfarinae,以下简称为“derf”))和屋尘螨(dermatophagoidespteronyssinus,以下简称为“derp”))构成。已知这些尘螨在温暖且湿润的条件下繁殖得最好,而在高气密化、高隔热化的建筑物增加的现代,一年中室内被保持在适于尘螨的条件的机会增大,其生存数量也增大。可以认为这是过敏性疾病的增加的原因的一个因素。在这些过敏性疾病的治疗中,使用抗组胺药、类固醇系抗炎症药、抗白三烯药、脱颗粒抑制药、th2细胞因子抑制药等,但所有都限于对症疗法。然而,近年来,以皮下过敏原免疫疗法(scit)、舌下过敏原免疫疗法(slit)为代表的过敏原免疫疗法相继开发。过敏原免疫疗法是通过在体内逐次少量投与过敏原来控制对于过敏原的免疫应答的治疗法,被认为这是目前能够根治过敏性疾病的唯一的方法。在过敏原免疫疗法的开发中,关于作为原因的过敏原的研究和理解是必不可少的。例如,在日本被认为生存数量多的粉尘螨虫体和排泄物中被报告有被称为derf1、derf2、derf3的抗原性高的过敏原,而且已经被克隆(非专利文献2、3)。另外,根据过去的报告,已知螨虫过敏的患者80%以上具有对这些抗原任意1种以上特异性的ige(非专利文献4)。此外,也积极进行着粉尘螨过敏原的探索研究,对于以螨虫为原因的过敏性疾病的预防和治疗要求更加有用的新型过敏原蛋白质(非专利文献5)。现有技术文献非专利文献非专利文献1:korsgaardj,etal.,allergy,53:77-83,1998非专利文献2:dilworthrj,etal.,clinicalandexperimentalallergy,21:25-32,1991非专利文献3:yuukit.etal.,jpn.j.allegol.,39(6):557-561,1990非专利文献4:heymannpw,etal.,j.allergyclin.immunol.,83:1055-1067,1989非专利文献5:thomaswr,etal.,int.arch.allergyimmunol.,129:1-18,2002技术实现要素:发明所要解决的课题本发明涉及提供新型的粉尘螨蛋白质、以及使用其的以粉尘螨为原因的过敏性疾病的诊断药、预防药和治疗药等。用于解决课题的方法本发明的发明人为了解决上述课题而进行研究的结果,从粉尘螨排泄物中,成功获得了与源自以粉尘螨为原因的过敏性疾病患者的血清中的ige显示高的反应性、分子量110kda附近的新的蛋白质,而且发现其作为以粉尘螨为原因的过敏性疾病的诊断药、预防药、治疗药有用。即,本发明涉及以下的1)~14)。1)选自以下的(a)~(c)中的粉尘螨蛋白质或其片段肽。(a)由序列号2所示的氨基酸序列构成的蛋白质。(b)由在序列号2所示的氨基酸序列中取代、缺失或添加1个或多个氨基酸的氨基酸序列构成、且具有粉尘螨过敏原活性的蛋白质。(c)由与序列号2所示的氨基酸序列具有90%以上的同一性的氨基酸序列构成、且具有粉尘螨过敏原活性的蛋白质。2)编码选自以下的(a)~(c)中的粉尘螨蛋白质或其片段肽的聚核苷酸。(a)由序列号2所示的氨基酸序列构成的蛋白质。(b)由在序列号2所示的氨基酸序列中取代、缺失或添加1个或多个氨基酸的氨基酸序列构成、且具有粉尘螨过敏原活性的蛋白质。(c)由与序列号2所示的氨基酸序列具有90%以上的同一性的氨基酸序列构成、且具有粉尘螨过敏原活性的蛋白质。3)选自以下的(d)~(f)中的聚核苷酸。(d)由序列号1所示的碱基序列构成的聚核苷酸。(e)与由与序列号1所示的碱基序列互补的碱基序列构成的聚核苷酸在严格条件下杂交、编码具有粉尘螨过敏原活性的蛋白质的聚核苷酸。(f)由与序列号1所示的碱基序列具有90%以上的同一性的碱基序列构成、且编码具有粉尘螨过敏原活性的蛋白质的聚核苷酸。4)以上述1)的粉尘螨蛋白质或其片段肽为有效成分的以粉尘螨为原因的过敏性疾病的预防或治疗剂。5)以上述1)的粉尘螨蛋白质或其片段肽为有效成分的以粉尘螨为原因的过敏性疾病的诊断药。6)与上述1)的粉尘螨蛋白质或其片段肽结合的结合蛋白质。7)如上述6)所述的结合蛋白质,其为抗体分子。8)如上述6)所述的结合蛋白质,其为单克隆抗体。9)上述1)的粉尘螨蛋白质或其片段肽用于制造以粉尘螨为原因的过敏性疾病的预防或治疗剂的使用。10)上述1)的粉尘螨蛋白质或其片段肽用于制造以粉尘螨为原因的过敏性疾病的诊断药的使用。11)如上述1)所述的粉尘螨蛋白质或其片段,其用于以肽粉尘螨为原因的过敏性疾病的预防或治疗。12)如上述1)所述的粉尘螨蛋白质或其片段,其用于以粉尘螨为原因的过敏性疾病的诊断。13)一种以粉尘螨为原因的过敏性疾病的预防或治疗方法,其中,对患者投与上述1)的粉尘螨蛋白质或其片段肽。14)一种以粉尘螨为原因的过敏性疾病的诊断方法,其中,对被测试者投与上述1)的粉尘螨蛋白质或其片段肽。发明的效果本发明的粉尘螨蛋白质由于与源自以粉尘螨为原因的过敏性疾病患者的血清中的ige显示高的反应性,所以能够在以粉尘螨为原因的过敏性疾病的诊断、预防和治疗中利用。该粉尘螨蛋白质是与公知的粉尘螨过敏原、特别是derf1(分子量约27kda)、derf2(分子量约15kda)、derf3(分子量约29kda)不同的蛋白质,因此通过将它们组合,能够成为更有用的过敏原免疫疗法。附图说明图1是用sds-聚丙烯酰胺凝胶电泳(sds-page)解析粉尘螨粗提取液和粗纯化物(用利用fplc的阴离子交换色谱分级)得到的图。图2是表示使用以粉尘螨为原因的过敏性疾病患者(rastscore≥2)5名的血清的纯化过敏原特异性的ige抗体效价的分析结果的图。图3是表示使用以粉尘螨为原因的过敏患者疾病(2名)的末梢血的过敏原活性评价(嗜碱性粒细胞上的cd203c表达增强)的结果的图。图中百分数表示解析对象细胞中的p4门(gate)内分布(population)。具体实施方式粉尘螨蛋白质本发明的粉尘螨蛋白质是选自以下的(a)~(c)中的蛋白质、或其片段肽。(a)由序列号2所示的氨基酸序列构成的蛋白质。(b)由在序列号2所示的氨基酸序列中取代、缺失或添加1个或多个氨基酸的氨基酸序列构成、且具有粉尘螨过敏原活性的蛋白质。(c)由与序列号2所示的氨基酸序列具有90%以上的同一性的氨基酸序列构成、且具有粉尘螨过敏原活性的蛋白质。(a)的由序列号2所示的氨基酸序列构成的蛋白质为由粉尘螨,通过利用fplc的阴离子交换色谱分级(fraction)后,通过超滤法除去100kda以下的蛋白而进行分离、纯化的蛋白质,具有以下的性质。i)与以粉尘螨为原因的过敏性疾病患者的血清中ige显示结合反应。ii)将以粉尘螨为原因的过敏性疾病患者的嗜碱性粒细胞活化。iii)由sds-聚丙烯酰胺凝胶电泳(sds-page)测定的分子量为约110kda。因此,该蛋白质是与以derf1(分子量约27kda)、derf2(分子量约15kda)、derf3(分子量约29kda)为代表的公知的粉尘螨过敏原不同的(非专利文献5)、新型的粉尘螨蛋白质。在本发明的粉尘螨蛋白质中,只要具有粉尘螨过敏原活性,则包括由在该序列号2所示的氨基酸序列中取代、缺失或添加1个或多个氨基酸的氨基酸序列构成的蛋白质(上述(b))。作为包含这样的氨基酸序列的粉尘螨蛋白质,可以列举例如具有序列号2所示的氨基酸序列的粉尘螨蛋白质的同种型等。其中,被缺失、取代或添加的多个氨基酸是指例如1~10个、更优选1~5个氨基酸。另外,上述的添加包括在两末端的1~数个氨基酸的添加。本说明书中,“过敏原”是指引起过敏性疾病的原因物质。另外,所谓“粉尘螨过敏原活性”不仅包括与肥大细胞上的ige结合、对特应性的人引起速发型过敏反应的活性(deweck,al.etal.,int.arch.allergyimmunol.,146:177-189,2008),还包括仅与血清中的ige结合的活性(参照实施例2(i))。另外,本发明的粉尘螨蛋白质中,只要具有粉尘螨过敏原活性,则还包括将相当于序列号2所示的氨基酸序列的序列适当比对时,由与序列号2所示的氨基酸序列具有90%以上的同一性的蛋白质构成的蛋白质((上述(c))。其中,与序列号2所示的氨基酸序列的同一性优选为95%以上,更优选为98%以上。该氨基酸序列的同一性例如能够适用使用blast(国家生物信息中心基本局部比对搜索工具,basiclocalalignmentsearchtoolatthenationalcenterforbiologicalinformation),以可选参数为初期设定值进行计算的方法。另外,本发明的粉尘螨蛋白质可以为融合蛋白质质这样的较大蛋白质的一部分。其中,作为融合蛋白质中所添加的序列,可以列举例如多重组氨酸残基那样有助于纯化的序列、确保重组生产时的稳定性的添加序列等。本发明的粉尘螨蛋白质可以仅是过敏原活性所必须的区域的片段肽。例如,优选列举包含特别是由以粉尘螨为原因的过敏患者疾病的t细胞所特异性识别的表位(t细胞表位)的部分片段。与hlaii类分子结合并抗原提呈的肽的长度,从肽解析的结果(chicz,r.m.etal.,j.exp.med.,178:27-47,1993)来看,可以认为由约10~34的氨基酸残基构成,因此至少包含1个t细胞表位的片段也包括这样长度的肽。t细胞表位能够通过利用10mer~20mer左右的重叠肽的研究、例如jahn-schmidb.j.etal.,allergyclinimmunol.126(5):1068-71,2010、sone,t,etal.,j.immunol.,161:448-457,1998等中记载的方法来鉴定。编码粉尘螨蛋白质的聚核苷酸本发明的聚核苷酸编码上述粉尘螨蛋白质或其片段肽,优选列举(d)由序列号1所示的碱基序列构成的聚核苷酸、(e)与由与序列号1所示的碱基序列互补的碱基序列构成的聚核苷酸在严格条件下杂交、且编码具有粉尘螨过敏原活性的蛋白质的聚核苷酸、(f)由与序列号1所示的碱基序列具有90%以上的同一性的碱基序列构成、且编码具有粉尘螨过敏原活性的蛋白质的聚核苷酸。(e)和(f)的聚核苷酸包括(d)的聚核苷酸的变异体。该变异体包括天然的等位基因变异体、使用该领域中公知的变异诱发技术能够生成的天然不存在的变异体。本发明的聚核苷酸不仅包括双链dna,还包括构成其的正义链和反义链等各种单链dna、rna。反义链能够作为探针等利用。dna包括例如由克隆、化学合成技术或它们的组合所得到的cdna、基因组dna等。此外,本发明的聚核苷酸中,除了本发明的编码多肽的碱基序列以外,还可以包括非翻译区(utr)的序列、载体序列(包含表达载体序列)等的碱基序列。这里,所谓严格条件,可以列举例如molecularcloning:alaboratorymanual(secondedition,j.sambrooket.al,1989)中记载的条件等。即,可以列举在含有6×ssc(1×ssc的组成:0.15m氯化钠、0.015m柠檬酸钠、ph7.0)、0.5%sds、5×denhardt和100mg/ml鲱鱼精子dna的溶液中,与探针一起,在65℃恒温8~16小时,使其杂交的条件等。另外,与序列号1所示的碱基序列的同一性优选为95%以上,更优选为98%以上。该碱基序列的同一性能够适用例如使用blast、将可选参数作为初期设定值进行计算的方法。编码粉尘螨蛋白质的聚核苷酸的取得编码本发明的粉尘螨蛋白质的聚核苷酸能够由粉尘螨克隆,作为克隆方法,可以列举已知的方法,例如使用鸟枪法、pcr法进行的方法。例如,制备与本发明的聚核苷酸的碱基序列的一部分特异性杂交的探针,使用该探针对于基因组dna文库或cdna文库进行筛选即可。作为这样的探针,只要为与本发明的聚核苷酸或其互补链的至少一部分特异性杂交的探针,则可以使用任何序列和长度的探针。另外,本发明的聚核苷酸也能够通过用适当的原理取得与包含本发明的聚核苷酸的一部分或全部的聚核苷酸杂交的序列来获得。例如,可以列举使用包含上述的本发明的聚核苷酸的一部分的聚核苷酸作为引物进行的pcr法、使用包含上述的本发明的聚核苷酸的一部分的聚核苷酸作为探针的方法。例如,使用pcr等扩增手段的方法,能够通过从本发明的聚核苷酸的5'侧和3'侧的序列(或其互补序列)中分别制备引物,使用这些引物以基因组dna(或cdna)等为模板进行pcr等扩增反应,将两引物之间所夹的dna区域扩增,由此大量取得包含该聚核苷酸的dna片段。另外,本发明中提供的聚核苷酸例如也能够通过利用定向或随机的变异导入法等方法,改变由序列号1所示的碱基序列构成的聚核苷酸来制作。这里,在定向地导入变异时的变异计划例如能够通过参考聚核苷酸序列上的特征性序列来进行。另外,作为随机地导入变异的方法,可以列举例如pcr法、利用突变原处理的方法。作为定向地导入变异的方法,可以列举部位特异性的突变诱导法,更具体而言,能够使用例如site-directedmutagenesissystemmutan-superexpresskm试剂盒(takarabio)等进行。另外,也能够使用重组pcr法(pcrprotocols,academicpress,newyork,1990)。粉尘螨蛋白质的制造本发明的粉尘螨蛋白质能够从粉尘螨分离纯化而得到。分离纯化方法没有特别限定,例如将粉尘螨提取液使用凝胶过滤、离子交换色谱法、亲和色谱等现有公知的方法分离、纯化即可。另外,也能够将通过在适当的载体中插入本发明的聚核苷酸所制作的重组载体导入宿主细胞,使粉尘螨蛋白质在细胞内或细胞外表达,来采取。用于插入本发明的聚核苷酸的载体只要能够在大肠杆菌、枯草杆菌等细菌、酵母或动物细胞等宿主中复制,就没有特别限定,可以列举例如质粒dna、噬菌体dna等。表达载体的构建所使用的载体dna可以使用广泛普及的容易获得的载体。例如可以列举puc19(takarabio)、ptv118n(takarabio)、pmamneo(clontech)、pgex(gehealthcare)、pet160(invitrogen)、pdest(invitrogen)等。在使用该重组载体转化宿主时,能够使用原生质体法、感受态细胞法、电穿孔法等进行。作为宿主,没有特别限制,能够使用所有宿主,例如能够使用动物、源自动物的细胞、植物、源自植物的细胞、微生物等。所得到的转化体使用包含能够利用的碳源、氮源、金属盐、维生素等的培养基在适当的条件下培养即可。能够从这样得到的培养液中,通过一般的方法进行蛋白质的收集、纯化,得到本发明的粉尘螨蛋白质。以粉尘螨为原因的过敏性疾病的预防或治疗本发明的粉尘螨蛋白质或其片段肽与源自以粉尘螨为原因的过敏性疾病患者的血清中ige显示高的反应性,因此能够作为以粉尘螨为原因的过敏性疾病的预防或治疗剂,能够向患者给药,并用于以粉尘螨为原因的过敏性疾病的预防或治疗。这里,以粉尘螨为原因的过敏性疾病的预防或治疗,具体是指对粉尘螨的特异性抗原为原因的所有过敏性疾病的预防或治疗,作为该过敏性疾病,可以列举例如特应性支气管哮喘、过敏性鼻炎、过敏性结膜炎、特应性皮肤炎等。该以粉尘螨为原因的过敏性疾病的预防或治疗可以列举例如对以粉尘螨为原因的过敏性疾病的过敏原免疫疗法。另外,本发明的粉尘螨蛋白质为与以现有公知的derf1、derf2为代表的过敏原不同的蛋白质,因此通过与它们组合,能够成为更加有用的过敏原免疫疗法。使用本发明的以粉尘螨为原因的过敏性疾病的预防或治疗剂作为过敏原免疫疗法剂的情况下,优选将粉尘螨蛋白质、或其片段肽直接或者干燥后制成粉末状,或者根据需要通过常规方法添加一般使用的各种添加剂、例如稳定剂、赋形剂、助溶剂、乳浊剂、缓冲剂、止痛剂、保存剂、着色剂等,调制成复方制剂。例如,能够将粉末状的被纯化的粉尘螨蛋白质溶解到添加了苯酚的生理盐水中,用作过敏原免疫疗法用抗原的原液。在使用本发明的以粉尘螨为原因的过敏性疾病的预防或治疗剂作为过敏原免疫疗法剂的情况下,在投与时,能够包含具有免疫赋活作用的佐剂。作为佐剂,能够列举例如完全弗氏佐剂(completefreund'sadjuvant(cfa))、不完全弗氏佐剂(incompletefreund'sadjuvant(ifa))、明矾、脂质a、单磷酰脂质a、卡介苗bcg(bacillus-calmette-guerrin)等的细菌制剂、cpg-dna、dsrna等的核酸、结核菌素等的细菌成分制剂、钥孔血蓝蛋白或酵母甘露聚糖等的天然高分子物质、胞壁酰三肽和胞壁酰二肽或它们的衍生物、明矾(alum)、非离子性嵌段共聚物、白细胞介素2(il-2)、粒细胞巨噬细胞集落刺激因子(granulocytemacrophagecolonystimulatingfactor)(gm-csf)、干扰素-α(ifn-α)、干扰素-β(ifn-β)等的细胞因子等,能够使用它们中的1种或组合2种以上使用。佐剂也可以与本发明的粉尘螨蛋白质、或其片段肽以混合状态、或作为乳液同时投与使用。本发明的以粉尘螨为原因的过敏性疾病的预防或治疗剂能够通过通常的给药途径例如经皮、经粘膜、经口、皮内、皮下、肌肉内、腹腔内、滴鼻等的给药方法进行。配合给药形态,可以制备成片剂、胶囊剂、颗粒剂、散剂、含片、舌下片、糖浆剂、注射剂、栓剂、吸入剂、经皮吸收剂、滴眼剂、滴鼻剂、鼻腔内喷雾剂、湿布剂、湿敷剂、软膏、乳剂、霜剂等的各种剂型的制剂。本发明的以粉尘螨为原因的过敏性疾病的预防或治疗剂的给药量和给药次数根据给药途径、症状等的不同而不同,例如能够以每1次给药量为约0.1μg~100mg的范围的方式适当选择,以每周1次到数次左右给药。以粉尘螨为原因的过敏性疾病的诊断本发明的粉尘螨蛋白质或其片段肽能够成为以粉尘螨为原因的过敏性疾病诊断药、具体而言对于以粉尘螨为原因的过敏性疾病的皮内反应诊断药,通过将其少量注射到被测试者的皮内,能够掌握生物体对于粉尘螨的过敏状态、免疫状态,用于抗原的确定、疾病的诊断。在作为皮内反应诊断试剂使用的情况下,将通过上述方法取得的本发明的粉尘螨蛋白质或其片段肽例如进行干燥后制成粉末状,将其溶解于含有苯酚的生理盐水中,稀释使用。与粉尘螨蛋白质或其片段肽结合的结合蛋白质与本发明的粉尘螨蛋白质或其片段肽结合的结合蛋白质是能够与本发明的粉尘螨蛋白质或其片段肽特异性结合的蛋白质。作为结合蛋白质,可以列举例如免疫球蛋白(iga、igd、igg、igm、igy等)、fab段、f(ab')2段、单链抗体段(scfv)、单域抗体等(carter,pj.,nat.rev.immunol.,6:343-357,2006)的抗体分子、affibody、darpins、avimer等的抗体样分子,作为抗体分子,可以列举多克隆抗体、单克隆抗体(小鼠抗体、嵌合抗体、人源化抗体和人抗体等),但不限定于这些。上述结合蛋白质能够使用各种公知的方法制作,制作方法没有特别限定。上述结合蛋白质能够在表达本发明的粉尘螨蛋白质的生物体或其组织或者细胞的鉴定等中利用。例如,能够用于测定大气中或室内的空间或人的粘膜中的粉尘螨蛋白质的有无等。该测定能够通过公知的免疫学方法进行,例如能够通过elisa法进行。另外,也能够用于以粉尘螨为原因的过敏性疾病的治疗。以下,利用实施例对本发明进行具体说明,但本发明不受这些实施例限定。实施例实施例1源自粉尘螨的蛋白质的纯化将长时间饲养螨虫后的培养基加湿干燥,通过180μm的筛后,分散在phosphatebufferedsaline(磷酸盐缓冲溶液,pbs)中,立即通过53μm的筛,去除不溶物。将分散有排泄物的pbs在37℃静置2小时,提取可溶性成分。在4℃以5,000rpm离心30分钟,将上清分离,用分级分子量12,000-14,000的透析膜透析2天,去除低分子成分。将其使用离心浓缩器(vivaspin20、mwco10kda)浓缩,得到粗提取物。在粗提取物中加入10倍量的20mmtris-hcl(ph9.0)后,上样于阳离子柱(vivapuresmaxim),离心(500g、5分)后,回收洗脱组分。将洗脱组分上样于阴离子色谱柱(unoq1ml、bio-rad),以0~1m的nacl梯度使其洗脱。回收含有目标蛋白质的组分,使用离心浓缩器(vivaspin6、mwco100kda)去除低分子组分,得到纯化蛋白质。将纯化蛋白质用sds-聚丙烯酰胺凝胶电泳(sds-page)解析的结果表示在图1中。实施例2纯化抗原的过敏原活性确认(i)与以粉尘螨为原因的过敏性疾病患者血清ige的反应性的确认制备本发明蛋白质以及已知的螨虫抗原(derf1)20μg/ml溶液,以成为1μg/50μl/孔的方式添加在maxisorp96孔板(nunc)中后,在4℃静置一晩,由此使抗原固相化。第二天将板清洗后,添加含有1%牛血清白蛋白的pbs,在室温静置1小时,由此实施封闭。在板清洗后,添加5倍稀释的患者血清,在室温静置2小时。作为阴性对照,使用市售的人ab型血清(cellect(r)humanabserum;mpbiomedicals)。板清洗后,添加以含有1%牛血清白蛋白的pbs稀释7000倍的2次抗体(hrp标记山羊抗人ige抗体、novus),室温静置40分钟。清洗板后,用tmbsubstratereagentset(tmb底物试剂盒)(bdbiosciences)通过室温静置以遮光状态实施20分钟tmb显色。结束后,通过添加1n硫酸,使显色停止,微孔板检测器测定波长450nm的吸光度。将其结果表示在图2中。(ii)以粉尘螨为原因的过敏性疾病患者嗜碱性粒细胞的活化已知在被过敏原刺激的嗜碱性粒细胞中,cd203c的表达增强(deweck,al.etal.,int.arch.allergyimmunol.,146:177-189,2008)。根据该原理,使用allergenicity试剂盒(beckmancoulter),分析患者血液中的嗜碱性粒细胞活化。在肝素采血的全血100μl中加入本发明蛋白质过敏原溶液(最终浓度1~100ng/ml)20μl和包含cd3-pc7、crth2-fitc和cd203c-pe的抗体混合物20μl,在37℃孵育15分钟。加入反应停止液100μl和溶血固定液2ml,在室温反应10分钟。离心(200xg、5分钟)之后,去除上清,加入磷酸缓冲液(pbs)3ml,再离心。去除上清后,将细胞悬浮于加有0.1%甲醛的pbs,利用流式细胞仪(facscanto、bdbiosciences)测定。对于所得到的数据,以cd3-pc7阴性且crth2-fitc阳性为指标,选择血球中的嗜碱性粒细胞,分析cd203c的表达强度。将其结果表示于图3。与仅添加pbs时相比,确认到添加过敏原时的cd203c的表达增强。实施例3碱基序列和氨基酸序列的确定<部分内部氨基酸序列的确定>为了明确本发明蛋白质内部的部分氨基酸序列,对利用蛋白酶有限酶解得到的片段进行解析。将纯化蛋白50μg进行sds-page后,将分子量110kda条带附近的凝胶切碎,在1ml的纯化水中进行搅拌、清洗(20分钟×2次)。添加300μl的7mol/l胍/0.5mol/ltris-hcl/0.01mol/ledta(ph8.5)和10μl的2-巯基乙醇,进行氮置换和超声波处理后,通过在室温静置2小时进行还原。接着,添加10μl的4-乙烯基吡啶,在进行氮置换和超声波处理(1分钟)之后,在室温静置30分钟,由此进行烷基化。将反应液废弃后,新加入1ml的0.1vol%三氟乙酸,确认为酸性后,搅拌1小时。将溶液废弃,在1ml的0.025mol/l碳酸氢铵/50vol%乙腈中搅拌(20分钟×2次)。将溶液废弃,在1ml的乙腈中搅拌5分钟后,将溶液废弃,用离心蒸发器将凝胶减压干燥固化。将溶解于0.025mol/l碳酸氢铵的赖氨酰内肽酶以酶/底物比1/20添加,在冰上静置,使凝胶膨润1小时。追加0.025mol/l碳酸氢铵,使溶液完全浸透凝胶,之后再在37℃反应17小时,回收反应液([1]液)。为了提取肽,在剩余的凝胶中添加100μl的0.1vol%三氟乙酸/50vol%乙腈,搅拌20分钟后,回收提取液([2]液)。进而在100μl的乙腈中搅拌20分钟后,回收提取液([3]液)。将[1]~[3]液混合并减压浓缩后,加入反相高效液相层析(xbridgec18),以乙腈0~80重量%的梯度进行洗脱、分级。收集主要的2个肽,利用蛋白测序(procise492clc;appliedbiosystems)进行n末端氨基酸序列解析的结果,得到了thr-asp-asp-phe-phe-pro-tyr-ala-ser-asp-glu-his-ala-tyr-trp-thr-gly-tyr-phe-thr〔部分氨基酸序列1(序列号3)〕和gln-gly-asp-tyr-val-glu-phe-asp-phe-val-val-gly-pro-ile-x-val〔部分氨基酸序列2(序列号4)〕。<粉尘螨rna的提取>从在液体氮中冻结的粉尘螨中,使用rneasyminikit(qiagen)按照以下的操作提取rna。对粉尘螨10mg,加入600μlbufferrlt,通过18g注射针进行破碎。将粉尘螨悬浊液加入到qiashredder旋转柱(qiagen)进行离心(15000rpm2分钟),回收穿柱液。在回收的rna溶液中添加70%乙醇水溶液600μl后,添加于rneasymini柱中,进行离心(10000rpm15秒)。进而将柱用bufferrw1350μl清洗后,用rnase-freednaseset(qiagen)进行dnase处理。将柱用bufferrw1350μl清洗后,按照rneasyminikit的操作说明进行rna的纯化。纯化后,添加rnasefreeh2o30μl,回收总rna(totalrna)。<粉尘螨cdna的制作>从所得到的总rna中,使用superscriptiiifirst-strandsynthesissystemforrt-pcr(invitrogen)合成cdna。将总rna3μg悬浮于8μl的纯化水中,加入oligo(dt)201μl、10mmdntp1μl后,在65℃孵育5分钟。在冰上冷却2分钟后,加入10xrtbuffer2μl、25mmmgcl21μl、0.1mdtt2μl、rnaseout(40u/μl)1μl、superscriptiiireversetranscriptase1μl并混合。在50℃孵育50分钟后,在85℃进行5分钟处理,停止反应。<cdna序列的确定>由所得到的部分氨基酸序列3、4设计简并引物。作为相当于部分氨基酸序列3的正义引物,设计primer1及其下游的primer2。同样作为相当于部分氨基酸序列4的反义引物,设计primer3及其上游的primer4。使用这些引物,以螨虫cdna为模板,进行利用kodfxneo(toyobo)的巢式pcr。将pcr产物在2%琼脂糖凝胶(nacalaitesque)上电泳后,将包含扩增的条带的凝胶片段用wizardsvgelandpcrclean-upsystem(promega)纯化。纯化的pcr产物使用primer2、primer4的引物进行利用kodfxneo的pcr,将pcr产物用wizardsvgelandpcrclean-upsystem(promega)纯化。纯化后,使用bigdyexterminatorkit(appliedbiosystems)和dna测序仪(3130xl;appliedbiosystems)分析碱基序列,取得中间序列。接着,为了分析3'侧序列,使用5'/3'racekit2ndgeneration(roche),制作在3'末端添加了锚序列的粉尘螨cdna。使用根据由中间序列得到的序列设计的primer5、primer6的2阶段的引物,进行利用kodfxneo的巢式pcr。分析所得到的pcr产物的碱基序列,取得3'末端序列。通过中间序列和3'末端序列的解析,取得1850bp的部分序列。对与该部分序列具有相同性的蛋白质利用ncbi(http://www.ncbi.nlm.nih.gov/)数据库进行检索,结果得知,与capitellateleta(海蠕虫)的假定蛋白captedraft_151096(genbank:elt89240.1)的相同性为37%,与lottiagigantean(霸王莲花青螺)的假定蛋白lotgidraft_207233(genbank:eso84761.1)的相同性为40%,与amphimedonqueenslandica(大堡礁海绵)的predicted:lysosomalalpha-mannosidase-like(溶酶体α-甘露糖苷酶样)(ncbireferencesequence:xp_003383866.1)的相同性为47%。因此,分析elt89240.1、eso84761.1、xp_003383866.1之间的相同序列,设计对于在n末端附近氨基酸序列一致的区域的简并引物primer7、primer8。利用由中间序列设计的primer9、primer10和primer7、primer8,从粉尘螨cdna使用kodfxneo实施巢式pcr。分析所得到的pcr产物的碱基序列,取得5'侧的部分序列。进而为了分析5'侧序列,使用smarterracecdna扩增试剂盒(clontech公司)实施5'race法。将粉尘螨总rna使用nucleotrapmrnamini(macherey-nagel)纯化粉尘螨polyamrna。使用纯化的polyamrna用smarterracecdna扩增试剂盒取得在5'末端包含接头序列的cdna。对该cdna用由5′侧部分序列设计的primer11用primestarhsdna聚合酶(takarabio)进行降落pcr。关于反应条件,按照smarterracecdna扩增试剂盒的操作说明。分析所得到的pcr产物的碱基序列,由此取得直到5′侧末端的序列。[表1]使用引物primer15’-cngaygayttyttyccntayg-3’序列号5primer25’-gaygarcaygcntaytgga-3′序列号6primer35’-aartcraaytcnacrtartcnc-3’序列号7primer45’-raaytcnacrtartcnccytg-3’序列号8primer55’-gttggtcataaaggacaacg-3’序列号9primer65’-tcggtatcgttcaagaagta-3’序列号10primer75’-gtnccncayacncaygayg-3’序列号11primer85’-ncaygaygaygtnggntgg-3’序列号12primer95’-ttgtggccacacttttacgtccag-3′序列号13primer105’-ccattgcttcacgtaatctt-3’序列号14primer115’-agccatttcccgggaatgaccaaatg-3’序列号15表中r=a+g、y=c+t、n=a+t+g+c的混合碱基编码本发明的粉尘螨蛋白质的cdna由3006bp的orf构成。由碱基序列推定的氨基酸数为1002aa。根据信号序列预测软件(signalp4.1、http://www.cbs.dtu.dk/services/signalp/)的解析,最初的27个氨基酸为信号肽。因此,作为天然物的本发明的粉尘螨蛋白质的氨基酸数为975aa,由该氨基酸序列预测的本发明的粉尘螨蛋白质的分子量为113.5kda。该预测分子量与sds-page的分子量约110kda一致。使用ncbi的blast检索进行由所确定的碱基序列推定的氨基酸序列的相同性检索,结果显示与以derf1、derf2为代表的已知的derf由来过敏原没有相同性,为新型的蛋白质。序列表<110>大鹏药品工业株式会社<120>新型粉尘螨蛋白质<130>th0094<150>jp2014-244038<151>2014-12-02<160>15<170>patentinversion3.5<210>1<211>3006<212>dna<213>粉尘螨<220><221>cds<222>(1)..(3006)<220><221>sig_peptide<222>(1)..(81)<220><221>mat_peptide<222>(82)..(3006)<400>1atggattatcataatctatcattattggtggtggctctattgtctgtg48metasptyrhisasnleuserleuleuvalvalalaleuleuserval-25-20-15accatattcacaacaattcaacaatctgaatcggttgttattaaagtg96thrilephethrthrileglnglnsergluservalvalilelysval-10-5-115gaaaatctgccagaacgttgtgattattcacaatgtccaaaatgggat144gluasnleuprogluargcysasptyrserglncysprolystrpasp101520cctaatgatattaatgtacatttggtggcccatacacatgatgatgtt192proasnaspileasnvalhisleuvalalahisthrhisaspaspval253035ggttggttaaaaacggtggaacaatattattatggcctaaaaaatgat240glytrpleulysthrvalgluglntyrtyrtyrglyleulysasnasp404550attcaacgtgccggagtgcaatatattttggatacagtaattgaagaa288ileglnargalaglyvalglntyrileleuaspthrvalilegluglu556065ttaatacgtaataaacaacgacgttttatttatgttgagattgcattc336leuileargasnlysglnargargpheiletyrvalgluilealaphe70758085ttttggaaatggtggcaagaacaagatgaagatcaacgtatgatcgta384phetrplystrptrpglngluglnaspgluaspglnargmetileval9095100cgagaattagtacgtactggacaattggaatttattaatggtggctgg432arggluleuvalargthrglyglnleuglupheileasnglyglytrp105110115tcaatgccagatgaagcagcaacacattataattcacttattgatcaa480sermetproaspglualaalathrhistyrasnserleuileaspgln120125130tctacatggggtctaagacaattgaatgatacattcggtaaatgtggt528serthrtrpglyleuargglnleuasnaspthrpheglylyscysgly135140145catccaaaagtaacatggcaaattgatccatttggtcattcccgggaa576hisprolysvalthrtrpglnileasppropheglyhisserargglu150155160165atggctaatctttatgcacagatgggttatgatgcattattttttgca624metalaasnleutyralaglnmetglytyraspalaleuphepheala170175180cgtcaagattatcaagatcgtgaaaatcgtatgacaaatcgtaaattg672argglnasptyrglnasparggluasnargmetthrasnarglysleu185190195gaacatgtatggcaaggatcagatgatcttggtactgctggtgatata720gluhisvaltrpglnglyseraspaspleuglythralaglyaspile200205210ttcactggtatgatgtttagtggttatggacctattgaattcaattgg768phethrglymetmetpheserglytyrglyproileglupheasntrp215220225gatataacaaatggccccgaagatgctgttgttgataatcctgagtca816aspilethrasnglyprogluaspalavalvalaspasnprogluser230235240245gaagaatataatgttccggataaaatacgacgttttgtggaaaaagca864gluglutyrasnvalproasplysileargargphevalglulysala250255260aaatattttgcacagtattatgcaacaaatcattttatgtttccaatg912lystyrphealaglntyrtyralathrasnhisphemetphepromet265270275ggtacggattttcaatatggtgatgcacatacatggtttaaaaatctg960glythrasppheglntyrglyaspalahisthrtrpphelysasnleu280285290gataaattgatcaaagctgtaaataatgctggaaaaggtgttcgggca1008asplysleuilelysalavalasnasnalaglylysglyvalargala295300305ttctattcaacaccatcatgctatgcacgtgcattatatgaaacgaat1056phetyrserthrprosercystyralaargalaleutyrgluthrasn310315320325cgtacatggaccacgaaaacagatgatttttttccctatgcatcagat1104argthrtrpthrthrlysthraspaspphepheprotyralaserasp330335340gaacatgcatattggactggatatttcaccagtagaccggctttaaaa1152gluhisalatyrtrpthrglytyrphethrserargproalaleulys345350355cgtatggaacgtatgggcaataatctattacaagcatgtaaacaattg1200argmetgluargmetglyasnasnleuleuglnalacyslysglnleu360365370gatattttggccggaaatgatggacgttttgaaatgaatataacaaga1248aspileleualaglyasnaspglyargpheglumetasnilethrarg375380385ttacgtgaagcaatgggtgtcatgcaacatcatgatgctgtaaccgga1296leuargglualametglyvalmetglnhishisaspalavalthrgly390395400405acggaaaaacaacatgttgcatttaattatgcaaaaatgttagattca1344thrglulysglnhisvalalapheasntyralalysmetleuaspser410415420gcaatgctacaatgtcgtcatgtaattagtgaatcatatcgaaagtta1392alametleuglncysarghisvalileserglusertyrarglysleu425430435tttccaacacaaacaaaagaacagcatgaattttgtccatatttaaat1440pheprothrglnthrlysgluglnhisgluphecysprotyrleuasn440445450ataagttcatgtccttcaacagaaatgggtgaatcacgtacgatacat1488ilesersercysproserthrglumetglygluserargthrilehis455460465ctttataatccacttggtcatcgtttagtgaatcgaacaatacgtgta1536leutyrasnproleuglyhisargleuvalasnargthrileargval470475480485cctgtaaaggatggttattattatcaagttcgtgaccaaaatgatcat1584provallysaspglytyrtyrtyrglnvalargaspglnasnasphis490495500tcaatacctgctgttttgatatcaataccagaatttgttcgtaaaatt1632serileproalavalleuileserileprogluphevalarglysile505510515cctggacgtaaaagtgtggccacaaaagaattagtattccgtgtacct1680proglyarglysservalalathrlysgluleuvalpheargvalpro520525530attattgaatcacttggtatacgtagatttcatatgattgccactaaa1728ileilegluserleuglyileargargphehismetilealathrlys535540545gaaaaacaacaggattcagctgttgaaatccaaggagaaaaatttgtt1776glulysglnglnaspseralavalgluileglnglyglulyspheval550555560565ggtcataaaggacaacgatttcaacttaaagatggtttgattattgaa1824glyhislysglyglnargpheglnleulysaspglyleuileileglu570575580tttgattctaatggaaaaattgcaacaatgactcgaaataatcaatcg1872pheaspserasnglylysilealathrmetthrargasnasnglnser585590595atatcgatatcgaatgaattccgtttgtttcatggtgctgatattggt1920ileserileserasnglupheargleuphehisglyalaaspilegly600605610cgtcattcaggtgcctatattttccgtccaagtgaacagaaaactttt1968arghisserglyalatyrilepheargprosergluglnlysthrphe615620625cctgtcacagaaaaaatggaagcaacattgtatgtagatcaaaaattc2016provalthrglulysmetglualathrleutyrvalaspglnlysphe630635640645ggtatcgttcaagaagtacatcaacaatttgattcatttgttggtcaa2064glyilevalglngluvalhisglnglnpheaspserphevalglygln650655660atcatacgattggataaacaaggtgattatgtggaatttgattttgtt2112ileileargleuasplysglnglyasptyrvalglupheasppheval665670675gttggaccaattccagtggatgatctaattggtaaagaaatcattaca2160valglyproileprovalaspaspleuileglylysgluileilethr680685690cgatatagtacgaatcttgcaaatgatgaaacattctttaccgattca2208argtyrserthrasnleualaasnaspgluthrphephethraspser695700705aatggtcgacaaatgttacgaagacgttggaattatcgtccatcatgg2256asnglyargglnmetleuargargargtrpasntyrargprosertrp710715720725aaatatgaaatcgaagaaccagtatcgggtaattattatccggttaat2304lystyrgluileglugluprovalserglyasntyrtyrprovalasn730735740tcacgtattgctattcgtgatgatagaaaatcattgcagatgacaatt2352serargilealaileargaspasparglysserleuglnmetthrile745750755atgactgatcgatcacaaggtggttcattatcacctgaacaaattaat2400metthraspargserglnglyglyserleuserprogluglnileasn760765770ggcagcgttgatctaatggtacatcgtcgtttattacatgatgattat2448glyservalaspleumetvalhisargargleuleuhisaspasptyr775780785tttggtgtcgatgaaccattaaatgaaccaggtgttgatggtcatggc2496pheglyvalaspgluproleuasngluproglyvalaspglyhisgly790795800805atagttatacgtggcagacatttattactattagatacattggaaaaa2544ilevalileargglyarghisleuleuleuleuaspthrleuglulys810815820gctgccgaaaaacatcgtccacttgcacaggaaatgtttatggagcca2592alaalaglulyshisargproleualaglnglumetphemetglupro825830835attattagttttacatcatcgatggagaaaaatcagccaatatataag2640ileileserphethrsersermetglulysasnglnproiletyrlys840845850ggattaacgaaagatctgcctggaaatgttcatcttcttaccctggaa2688glyleuthrlysaspleuproglyasnvalhisleuleuthrleuglu855860865caatggcattcaaaacgttatctattacgtcttgaacatttttatcaa2736glntrphisserlysargtyrleuleuargleugluhisphetyrgln870875880885cgttttgaagatccatcattgagtaatccagccactgtatcgttacgc2784argphegluaspproserleuserasnproalathrvalserleuarg890895900catttatttcaatcatttgaaataactgccgttgaagaattaacgctt2832hisleupheglnserphegluilethralavalglugluleuthrleu905910915ggtgcaaatcaaccgatatcagcgttgaaaaatcgtttacaatatcgt2880glyalaasnglnproileseralaleulysasnargleuglntyrarg920925930tatattagaccattaaatgagcaacaatcatcgatcataacggatcca2928tyrileargproleuasngluglnglnserserileilethrasppro935940945ataattgaaggtgaaaatttcgatattcatcttgaaccgatgcagata2976ileilegluglygluasnpheaspilehisleugluprometglnile950955960965cgtacatttttaatcgatattaaacgaaat3006argthrpheleuileaspilelysargasn970975<210>2<211>1002<212>prt<213>粉尘螨<400>2metasptyrhisasnleuserleuleuvalvalalaleuleuserval-25-20-15thrilephethrthrileglnglnsergluservalvalilelysval-10-5-115gluasnleuprogluargcysasptyrserglncysprolystrpasp101520proasnaspileasnvalhisleuvalalahisthrhisaspaspval253035glytrpleulysthrvalgluglntyrtyrtyrglyleulysasnasp404550ileglnargalaglyvalglntyrileleuaspthrvalilegluglu556065leuileargasnlysglnargargpheiletyrvalgluilealaphe70758085phetrplystrptrpglngluglnaspgluaspglnargmetileval9095100arggluleuvalargthrglyglnleuglupheileasnglyglytrp105110115sermetproaspglualaalathrhistyrasnserleuileaspgln120125130serthrtrpglyleuargglnleuasnaspthrpheglylyscysgly135140145hisprolysvalthrtrpglnileasppropheglyhisserargglu150155160165metalaasnleutyralaglnmetglytyraspalaleuphepheala170175180argglnasptyrglnasparggluasnargmetthrasnarglysleu185190195gluhisvaltrpglnglyseraspaspleuglythralaglyaspile200205210phethrglymetmetpheserglytyrglyproileglupheasntrp215220225aspilethrasnglyprogluaspalavalvalaspasnprogluser230235240245gluglutyrasnvalproasplysileargargphevalglulysala250255260lystyrphealaglntyrtyralathrasnhisphemetphepromet265270275glythrasppheglntyrglyaspalahisthrtrpphelysasnleu280285290asplysleuilelysalavalasnasnalaglylysglyvalargala295300305phetyrserthrprosercystyralaargalaleutyrgluthrasn310315320325argthrtrpthrthrlysthraspaspphepheprotyralaserasp330335340gluhisalatyrtrpthrglytyrphethrserargproalaleulys345350355argmetgluargmetglyasnasnleuleuglnalacyslysglnleu360365370aspileleualaglyasnaspglyargpheglumetasnilethrarg375380385leuargglualametglyvalmetglnhishisaspalavalthrgly390395400405thrglulysglnhisvalalapheasntyralalysmetleuaspser410415420alametleuglncysarghisvalileserglusertyrarglysleu425430435pheprothrglnthrlysgluglnhisgluphecysprotyrleuasn440445450ilesersercysproserthrglumetglygluserargthrilehis455460465leutyrasnproleuglyhisargleuvalasnargthrileargval470475480485provallysaspglytyrtyrtyrglnvalargaspglnasnasphis490495500serileproalavalleuileserileprogluphevalarglysile505510515proglyarglysservalalathrlysgluleuvalpheargvalpro520525530ileilegluserleuglyileargargphehismetilealathrlys535540545glulysglnglnaspseralavalgluileglnglyglulyspheval550555560565glyhislysglyglnargpheglnleulysaspglyleuileileglu570575580pheaspserasnglylysilealathrmetthrargasnasnglnser585590595ileserileserasnglupheargleuphehisglyalaaspilegly600605610arghisserglyalatyrilepheargprosergluglnlysthrphe615620625provalthrglulysmetglualathrleutyrvalaspglnlysphe630635640645glyilevalglngluvalhisglnglnpheaspserphevalglygln650655660ileileargleuasplysglnglyasptyrvalglupheasppheval665670675valglyproileprovalaspaspleuileglylysgluileilethr680685690argtyrserthrasnleualaasnaspgluthrphephethraspser695700705asnglyargglnmetleuargargargtrpasntyrargprosertrp710715720725lystyrgluileglugluprovalserglyasntyrtyrprovalasn730735740serargilealaileargaspasparglysserleuglnmetthrile745750755metthraspargserglnglyglyserleuserprogluglnileasn760765770glyservalaspleumetvalhisargargleuleuhisaspasptyr775780785pheglyvalaspgluproleuasngluproglyvalaspglyhisgly790795800805ilevalileargglyarghisleuleuleuleuaspthrleuglulys810815820alaalaglulyshisargproleualaglnglumetphemetglupro825830835ileileserphethrsersermetglulysasnglnproiletyrlys840845850glyleuthrlysaspleuproglyasnvalhisleuleuthrleuglu855860865glntrphisserlysargtyrleuleuargleugluhisphetyrgln870875880885argphegluaspproserleuserasnproalathrvalserleuarg890895900hisleupheglnserphegluilethralavalglugluleuthrleu905910915glyalaasnglnproileseralaleulysasnargleuglntyrarg920925930tyrileargproleuasngluglnglnserserileilethrasppro935940945ileilegluglygluasnpheaspilehisleugluprometglnile950955960965argthrpheleuileaspilelysargasn970975<210>3<211>20<212>prt<213>粉尘螨<400>3thraspaspphepheprotyralaseraspgluhisalatyrtrpthr151015glytyrphethr20<210>4<211>16<212>prt<213>粉尘螨<220><221>misc_feature<222>(15)..(15)<223>xaa可以是任何自然产生的氨基酸<400>4glnglyasptyrvalglupheaspphevalvalglyproilexaaval151015<210>5<211>21<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(2)..(2)<223>n=a+t+g+c<220><221>misc_feature<222>(5)..(5)<223>y=c+t<220><221>misc_feature<222>(8)..(8)<223>y=c+t<220><221>misc_feature<222>(11)..(11)<223>y=c+t<220><221>misc_feature<222>(14)..(14)<223>y=c+t<220><221>misc_feature<222>(17)..(17)<223>n=a+t+g+c<220><221>misc_feature<222>(20)..(20)<223>y=c+t<400>5cngaygayttyttyccntayg21<210>6<211>19<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(3)..(3)<223>y=c+t<220><221>misc_feature<222>(6)..(6)<223>r=a+g<220><221>misc_feature<222>(9)..(9)<223>y=c+t<220><221>misc_feature<222>(12)..(12)<223>n=a+t+g+c<220><221>misc_feature<222>(15)..(15)<223>y=c+t<400>6gaygarcaygcntaytgga19<210>7<211>22<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(3)..(3)<223>r=a+g<220><221>misc_feature<222>(6)..(6)<223>r=a+g<220><221>misc_feature<222>(9)..(9)<223>y=c+t<220><221>misc_feature<222>(12)..(12)<223>n=a+t+g+c<220><221>misc_feature<222>(15)..(15)<223>r=a+g<220><221>misc_feature<222>(18)..(18)<223>r=a+g<220><221>misc_feature<222>(21)..(21)<223>n=a+t+g+c<400>7aartcraaytcnacrtartcnc22<210>8<211>21<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(1)..(1)<223>r=a+g<220><221>misc_feature<222>(4)..(4)<223>y=c+t<220><221>misc_feature<222>(7)..(7)<223>n=a+t+g+c<220><221>misc_feature<222>(10)..(10)<223>r=a+g<220><221>misc_feature<222>(13)..(13)<223>r=a+g<220><221>misc_feature<222>(16)..(16)<223>n=a+t+g+c<220><221>misc_feature<222>(19)..(19)<223>y=c+t<400>8raaytcnacrtartcnccytg21<210>9<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>9gttggtcataaaggacaacg20<210>10<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>10tcggtatcgttcaagaagta20<210>11<211>19<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(3)..(3)<223>n=a+t+g+c<220><221>misc_feature<222>(6)..(6)<223>n=a+t+g+c<220><221>misc_feature<222>(9)..(9)<223>y=c+t<220><221>misc_feature<222>(12)..(12)<223>n=a+t+g+c<220><221>misc_feature<222>(15)..(15)<223>y=c+t<220><221>misc_feature<222>(18)..(18)<223>y=c+t<400>11gtnccncayacncaygayg19<210>12<211>19<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(1)..(1)<223>n=a+t+g+c<220><221>misc_feature<222>(4)..(4)<223>y=c+t<220><221>misc_feature<222>(7)..(7)<223>y=c+t<220><221>misc_feature<222>(10)..(10)<223>y=c+t<220><221>misc_feature<222>(13)..(13)<223>n=a+t+g+c<220><221>misc_feature<222>(16)..(16)<223>n=a+t+g+c<400>12ncaygaygaygtnggntgg19<210>13<211>24<212>dna<213>人工序列<220><223>合成寡核苷酸<400>13ttgtggccacacttttacgtccag24<210>14<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>14ccattgcttcacgtaatctt20<210>15<211>26<212>dna<213>人工序列<220><223>合成寡核苷酸<400>15agccatttcccgggaatgaccaaatg26当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1