乳酸乳球菌乳亚种YF11的非编码小RNAs144的制作方法
本发明涉及细菌非编码小RNA(small non-coding RNA,sRNA),特别是涉及一个乳酸乳球菌乳亚种YF11的非编码小RNA s144、含有乳酸乳球菌乳亚种YF11的非编码小RNAs144的载体、含该载体的乳酸乳球菌及应用。
背景技术:
细菌非编码小RNA(small non-coding RNA,sRNA)是近年来在细菌等原核生物中发现的一类RNA调控子,它们不编码蛋白质,通常位于基因间区,长度在50-500nt之间,广泛参与体内多种生命活动的调控。细菌sRNA是细菌代谢、毒力和适应环境压力的重要调节因子,在应对环境变化的基因表达调控中发挥重要作用,sRNA的功能涉及从结构调节到催化作用,影响生物体内各种加工过程,一个单独的sRNA就能调控大量的基因并对细胞生理产生深远影响。sRNA主要通过碱基配对与靶标mRNA结合导致mRNA翻译和稳定性的变化来发挥生物调控作用。迄今为止,大部分细菌sRNA的研究集中在大肠杆菌等模式生物中,在各种细菌中共发现超过150种sRNA,在大肠杆菌中发现了约80种sRNA。但随着研究的不断深入,已有越来越多的研究转向其他细菌。
Nisin(乳链菌肽)是由乳酸乳球菌(Lactococcus lactis)产生的一种具有抑菌作用的天然小分子多肽,对于大多数革兰氏阳性菌具有抑菌效果,并且通过与其他的抑菌剂联合还能够抑制某些革兰氏阴性菌的生长。Nisin是用于食品的生物防腐剂,已广泛应用于乳制品、肉制品和酒精饮料等防腐保鲜中,并且极有潜力用于药物,兽药及保健品中。但目前nisin生产成本较高,阻碍了其在生产应用领域的发展。
乳酸乳球菌在发酵过程中能够产生乳酸,乳酸的积累导致发酵液的pH下降,进而导致乳酸乳球菌生长停止,甚至死亡。
技术实现要素:
本发明的目的是克服现有技术的不足,提供一种乳酸乳球菌乳亚种YF11的非编码小RNA s144。
本发明的第二个目的是提供一种含有乳酸乳球菌乳亚种YF11的非编码小RNAs144的载体。
本发明的第三个目的是提供一种含有上述载体的重组乳酸乳球菌。
本发明的第四个目的是提供一种含有上述载体的重组乳酸乳球菌在发酵生产乳链菌肽的应用。
本发明的技术方案概述如下:
乳酸乳球菌乳亚种YF11的非编码小RNAs144,该核苷酸序列如序列表中SEQ ID NO.1所示;或与序列表SEQ ID NO.1所示的核苷酸序列具有90%以上同源性并与耐酸相关的核苷酸序列。
含有乳酸乳球菌乳亚种YF11的非编码小RNA s144的载体,其核苷酸序列如序列表中SEQ ID NO.2所示。
含有上述载体的重组乳酸乳球菌。
上述重组乳酸乳球菌在发酵生产乳链菌肽的应用。
本发明优点:
实验结果表明:乳酸乳球菌乳亚种YF11的非编码小RNA s144过表达后,含有s144过表达的乳酸乳球菌在酸性环境下的生存能力明显增强,提高在酸性发酵液的存活率,增加nisin的稳定性,从而提高nisin产量。
附图说明
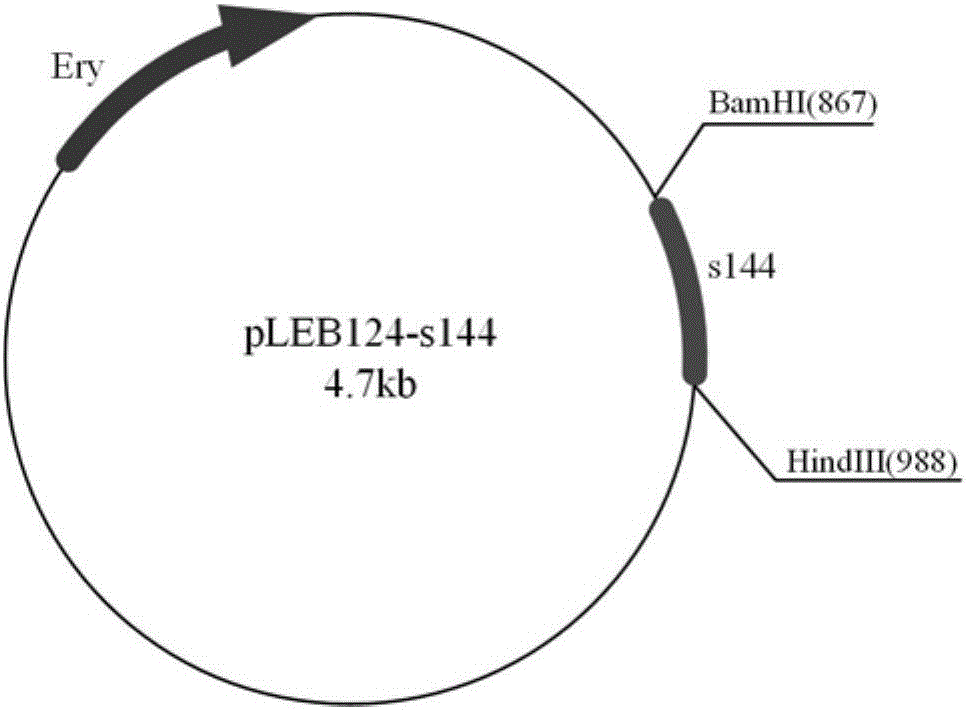
图1为乳酸乳球菌乳亚种YF11的非编码小RNA s144过表达载体pLEB124-s144的物理图谱。
图2为菌株的在不同pH胰蛋白胨水溶液应激3h后存活率。
图3为菌株生物量随时间变化图。
图4为菌株的nisin效价随时间变化图。
图2-图4中:
含有空质粒pLEB124的乳酸乳球菌乳亚种YF11对照菌株图示中记为YF11+菌;
含有载有乳酸乳球菌乳亚种YF11的非编码小RNA s144的pLEB124载体的重组乳酸乳球菌(本发明的菌株)菌株命名为乳酸乳球菌乳亚种YF11-s144,图示中记为s144菌;
具体实施方式
下面结合具体实施例对本发明作进一步的说明,本发明的实施例是为了使本领域的技术人员能够更好地理解本发明,但并不对本发明作任何限制。
各实施例所用的乳酸乳球菌乳亚种(Lactococcus lactis subsp.lactis)YF11菌株保藏于中国微生物菌种保藏管理委员会普通微生物中心,保藏号为CGMCC NO.12429,保藏时间为2015年5月10日。地址为北京市朝阳区北辰西路1号院3号中国科学院微生物研究所,邮编100101。
本发明中所用的实验方法如未作特别说明均是采用了常规方法。
所用质粒pLEB124购买于苏州金唯智生物科技有限公司。
实施例1
乳酸乳球菌乳亚种YF11的非编码小RNA s144(简称s144)序列的获得
1、文库的构建及测序
Small RNA文库:提取样品总RNA后用切胶回收片段(18-150nt),按照Illumina TruSeqTM SmallRNA Sample Preparation protocol构建small文库,最后建好的文库用Illumina HiSeq2000进行测序。
链特异性文库:将提取样品总RNA去除rRNA后打断成短片段,以mRNA为模板先后合成两条cDNA链,适当修饰后将第二条cDNA链,PCR扩增后将建好的测序文库用Illumina HiSeq2000进行测序。
2、数据预处理与生物信息学分析
测序所得的原始数据需要去除杂质数据:含adaptor的reads、N的比例大于10%的reads、低质量reads,最后获得clean reads。然后对clean reads进行拼接处理,获得Full-length Tags,将tags比对到基因组、基因、rRNA和tRNA上,得到比对统计信息。
基因表达量的计算使用RPKM法(Reads Per Kb per Million reads),其计算公式为:
其中,RPKM为某基因的表达量,C为唯一比对到该基因的reads数,N为唯一比对到参考基因的总reads数,L为该基因编码区的碱基数。
3、样品的制备与总RNA的提取
取出-80℃保藏的乳酸乳球菌乳亚种YF11菌株,接种到种子培养基中,30℃活化培养三代,然后以10%的接种量分别转接到pH 7.0和pH5.0的发酵培养基中,继续应激培养1h,4℃,8000rpm离心5min去上清收集菌体,无菌生理盐水洗涤菌体三次,用液氮迅速冷冻。在冰上使用细胞破碎仪对菌体进行机械破壁(超声5s,暂停5s,持续10min)后用trizol试剂重悬并混匀。使用ZR RNA MiniPrepTM提取菌体总RNA,RNA样品-80℃保存在加有RNA酶抑制剂的RNase free水中。按照试剂盒说明书进行操作。检测提取的总RNA的浓度和纯度,使用合格的样品进行后续cDNA的合成。
使用RevertAid First Strand cDNA Synthesis Kit试剂盒,按照cDNA的合成体系将各反应物加入PCR管中,3000r/min离心30s。cDNA合成程序:25℃温育5min,42℃水浴60min进行合成cDNA反应,70℃加热5min终止反应。测定cDNA样品的浓度和纯度,-20℃保存备用。
表1:cDNA的合成体系组成:
乳酸乳球菌种子培养基配方:酵母粉15g,蛋白胨15g,KH2PO420g,蔗糖15g,NaCl 1.5g,MgSO4·7H2O 0.15g,加水至1L,调节pH至7.2,琼脂粉15g(固体培养基),121℃高温蒸气灭菌20min;
乳酸乳球菌发酵培养基配方酵母粉15g,蛋白胨15g,KH2PO42%,蔗糖2%,玉米浆3g,半胱氨酸2.6g,NaCl 1.5g,MgSO4·7H2O 0.15g,加水至1L,调节pH至7.2,121℃高温蒸气灭菌20min。
4、实时荧光定量PCR验证
将所得的cDNA按试剂说明书(480 SYBR Green I Master)稀释至一定浓度用作qRT-PCR反应的模板,按照RT-PCR反应体系将模板加入离心管中,将样品转移至96孔板,设置3个平行,再加入其他反应物,以上操作步骤均在冰上进行,验证引物设计如表2所示。3000r/min简短离心后,进行RT-PCR反应。RT-PCR反应程序为:95℃预变性10min;95℃变性10s,50℃复性10s,72℃延伸1min,共55个循环;72℃延伸5min;熔解曲线65℃起始1min,95℃终止。
RT-PCR反应体系:
表2
引物序列如下:
说明:用SEQ ID NO.3、SEQ ID NO.4,结合表2其它组分,扩增得到16S RNA目标片段;
用SEQ ID NO.5、SEQ ID NO.6,结合表2其它组分,扩增得到s144目标片段。
5、小RNA的预测与确定
对于small RNA文库,根据比对信息,过滤后得到那些比对到基因间区IGR(Intergenic region)和AM(Antisense to mRNA)类型的All-unitags且总表达量大于20的All-unitags列为候选的sRNA将其列为候选sRNA,该sRNA为乳酸乳球菌乳亚种YF11的非编码小RNA s144,其核苷酸序列如SEQ ID NO.1所示。
对于链特异性文库,从中挑选出长度大于等于100bp且平均覆盖深度大于等于2的gene model(TAR),再从中找出位于基因间区(一个基因3’端下游100bp到下一个基因5’端上游100bp之间)的作为新转录本,这些转录本与蛋白库(NR)比对不上的作为候选sRNA。对候选的小RNA,为了验证所预测的候选sRNA在发酵培养基中酸应激1h后表达量的变化情况,采用检测目的sRNA相对于内参基因16S rRNA的相对定量方法,即2-ΔΔCp法。具体计算方法如下:
ΔCp=Cp(目的基因)-Cp(内参基因)
ΔΔCp=ΔCp(实验组)-ΔCp(对照组)
相对倍数(实验组/对照组)=2-ΔΔCp,以pH=7.0条件下的为对照组,相对转录水平设为1,其他条件为实验组。重复3次实验,计算平均值。
为了验证深度测序所得候选sRNA在酸胁迫下相对表达差异,通过qRT-PCR的方法检测sRNA基因经过酸应激处理后表达量的变化,使用2-ΔΔCt法分析耐酸相关sRNA在pH 5.0(实验组)和pH 7.0(对照组)应激下的差异表达相对倍数。从荧光定量RCR结果中筛选出了在酸性胁迫下表达量明显上调的sRNA。
荧光定量PCR验证sRNA上调情况见表3:
表3
实施例2、s144的功能分析
为了分析s144在乳酸乳球菌的生存和耐受性方面发挥的作用,构建了s144过表达菌株
和缺失菌株,并进行了相关的检测实验。
1、s144过表达菌株的构建及验证
以乳酸乳球菌乳亚种YF11的基因组为模版,根据s144的核苷酸序列设计引物sRNA-F与sRNA-R进行PCR1扩增。扩增产物经琼脂糖凝胶电泳纯化后用DNA纯化回收试剂盒回收后与质粒pLEB124用BamHI和HindIII双酶切,然后用T4DNA连接酶连接。连接产物转化TG1大肠杆菌感受态细胞,转化产物涂布具有红霉素(150ug/ml)抗性的LB平板。对平板上的菌落用引物General-sRNA-F、General-sRNA-R进行PCR2反应验证,扩增出315bp的片段认为是阳性克隆,将阳性菌落于LB液体培养基中过夜培养,提取质粒送测序。结果正确的质粒即为过表达载体(含有乳酸乳球菌乳亚种YF11的非编码小RNA s144的载体),将其命名为pLEB124-s144,其核苷酸序列如SEQ ID NO.2所示。将过表达载体pLEB124-s144电转到乳酸乳球菌乳亚种YF11感受态细胞中,涂布含有红霉素(20ug/ml)抗性的种子培养基平板,30℃培养36小时,用引物General-sRNA-F、General-sRNA-R进行PCR3筛选阳性克隆转化子即为乳酸乳球菌过表达株,即为本发明的含载有乳酸乳球菌乳亚种YF11的非编码小RNA s144的pLEB124载体的重组乳酸乳球菌菌株命名为乳酸乳球菌乳亚种YF11-s144,简称YF11-s144,图示中记为s144菌;
(1)引物序列
sRNA‐F:CGCGGATCCTCCTCCCCTTTCATATTTAGAA(SEQ ID NO.7)
sRNA‐R:CCCAAGCTTCAAATTACTTGAGGGCATCC(SEQ ID NO.8)
General‐sRNA‐F:ATTACTGAAGGGAACCTAGAATAGT(SEQ ID NO.9)
General‐sRNA‐R:CGACCTGAGGTAATTATAACCC(SEQ ID NO.10)
(2)PCR反应体系
PCR1反应体系:
PCR1反应条件:95℃预变性2min;95℃变性20s,Tm‐5℃复性20s,72℃延伸20s,共35个循环;72℃延伸5min。重叠延伸PCR反应程序:95℃预变性2min;95℃变性20s,Tm+5℃复性20s(每个循环温度下降0.5℃,直至Tm‐5℃),72℃延伸20s,进行20个循环;95℃预变性2min;95℃变性20s,Tm‐5℃复性20s,72℃延伸20s,再进行15个循环;72℃延伸5min。PCR2反应体系:
PCR2反应条件:97℃预变性7min;94℃变性30s,55℃复性30s,72℃延伸1min,共30个循环;72℃延伸5min。
2、不同的乳酸乳球菌在不同pH的胰蛋白胨水溶液中应激后生存能力分析。
乳酸乳球菌由于发酵后期发酵液的pH值降低会严重影响菌体的生长。为研究小RNA分子在酸性应激下的生存能力,用胰蛋白胨水溶液作应激培养基,应激3小时后稀释涂布计算存活率。
使用的菌株:YF11+菌和YF11-s144菌
具体方法包括以下步骤:
将YF11菌和YF11-s144菌分别接入种子培养基中,30℃静置培养。活化三代后,取6mL对数生长后期(约8h),8000r/min,4℃离心10min,弃上清收集菌体。用等体积生理盐水悬浮菌体,8000r/min,4℃离心10min,弃上清收集菌体,再重复一次该洗涤步骤。用3mL生理盐水悬浮菌体,各取500μL菌悬液接入8mL不同pH梯度的胰蛋白胨水培养基(胰蛋白胨水培养基:胰蛋白胨2%,氯化钠(NaCl)0.5%,分别调节至pH 7.0,5.0,4.0,3.0,2.0,121℃高温蒸气灭菌20min)中,于30℃静置应激3h。将pH 7.0、pH 5.0、pH 4.0条件下的菌体培养液用生理盐水稀释至10-7,将pH3.0和pH2.0条件下的菌体培养液用生理盐水稀释至10-6。每个样品取100μL稀释液涂布于pH 7.2的种子培养基平板上,30℃倒置培养。统计各个平板上菌落数,三个平行样本取平均值;其pH 5.0、pH 4.0、pH3.0和pH2.0条件下的菌落数分别除以pH 7.0条件下的菌落数计算系列pH梯度胰蛋白胨水培养基胁迫3h后的存活率;之后将实验组和对照组菌株在相同pH条件下的存活率作比值以此衡量乳酸乳球菌乳亚种YF11-s144耐酸性变化情况。检测结果如图2所示,可以看出,在酸应激后,过表达菌株的生存率高于对照组,说明过表达的sRNA基因有助于菌株在低pH下生存。
3、s144过表达菌株生长状况的测定
为检测小RNA s144对菌体生存能力的影响,进行了发酵实验:取出-20℃保存的YF11菌、YF11+菌、YF11-s144菌和YF11-Δs144菌活化3代后按5%的接种量接入到发酵培养基(乳酸乳球菌发酵培养基(g/L):酵母膏15,蛋白胨15,磷酸二氢钾20,蔗糖20,玉米浆3,半胱氨酸2.6,无碘精盐1.5,MgSO4·7H2O 0.15,pH 7.2)中,30℃恒温静置培养,每隔2h取发酵液样品,8000r/min离心5min分离菌体。用等体积的生理盐水重新悬浮菌体,然后对菌悬液进行稀释测定OD600值,以生理盐水为对照,保证OD600测量值在0.3-0.7之间线性范围内,绘制发酵液生物量随时间变化曲线如图3。可以看出,过表达菌株乳酸乳球菌乳亚种YF11-s144菌体量同期对比较空质粒对照组高,生长情况比空质粒对照组的好。推测该小RNA分子与菌体的生存能力相关。
4.nisin效价测定试验
取出-20℃保存的YF11+菌和YF11-s144菌活化3代后按5%的接种量接入到发酵培养基中,30℃恒温静置培养,每隔2h取分别取500μL这四种菌的发酵液样品与500μL无菌HCl溶液(0.02mol/L)混匀,然后煮沸5min,8000r/min再离心5min。取上清液用无菌HCl溶液(0.02mol/L)稀释分别制成这四种菌在不同时间点的样品。
将4℃保存的藤黄八叠球菌(Micrococcus flavus ATCC 10240,购买于2012年6月17日,购于上海研生实业有限公司)斜面取出,用2mL无菌生理盐水洗下菌苔,将得到的菌悬液收集到无菌离心管中,取100μL菌悬液混匀于6mL无菌生理盐水。nisin效价培养基(26mL)加热融化后冷却至70℃左右,加入1.5%的吐温-20(390μL),再冷却至50℃左右,加入上述菌悬液迅速摇匀,倒入9mm无菌培养皿内。待其凝固后用灭菌直径7mm的打孔器在平板上均匀打出8个孔(其中6个孔加入样品,2个孔加入nisin标准液)。称取8mg nisin标准品溶于8mL无菌HCl溶液(0.02mol/L)制成10000IU/mL的标准溶液,然后以此稀释制成2000IU/mL、1000IU/mL、200IU/mL、100IU/mL、50IU/mL和25IU/mL系列nisin标准溶液。每个样品设置三个平行,每个孔内加入100μL的样品或标准溶液,加样时尽量让四株菌在同一时间点取得的样品分布在同一平板上,37℃恒温培养24h。用游标卡尺测量抑菌圈直径,并以直径平均值为横坐标,nisin效价的对数值为纵坐标,绘制标准曲线。然后将样品的抑菌圈直径值代入标准方程计算得到样品的nisin效价。
由图4可看出s144相比于对照菌株nisin产量有所增长,说明s144与nisin产量有关。
SEQUENCE LISTING
<110> 天津大学
<120> 乳酸乳球菌乳亚种YF11的非编码小RNA s144
<130>
<160> 10
<170> PatentIn version 3.3
<210> 1
<211> 115
<212> RNA
<213> Lactococcus lactis subsp.lactis
<400> 1
auuucagaua uuaauuguua auucuagaac ucaucgguug aacaaucgga aaucaacaau 60
uaauaucuga uucaaauagg gaaaagagaa uucuccuucg ggaggauuuu uuaau 115
<210> 2
<211> 4697
<212> DNA
<213> 人工合成
<400> 2
gaccggaaat atttacaaaa atcaatttaa caattcctta aaacatgcag gaattgacga 60
tttaaacaat attagctttg aacaattctt atctcttttc aatagctata aattatttaa 120
taagtaagtt aagggatgca taaactgcat cccttaactt gtttttcgtg tgcctatttt 180
ttgtgaatcg gattcacaga tgtctgcctg ttcatccgcg tccagctcgt tgagtttctc 240
cagaagcgtt aatgtctggc ttctgataaa gcgggccatg ttaagggcgg ttttttcctg 300
tttggtcact gatgcctccg tgtaaggggg atttctgttc atgggggtaa tgataccgat 360
gaaacgagag aggatgctca cgatacgggt tactgatgat gaacatgccc ggttactgga 420
acgttgtgag ggtaaacaac tggcggtatg gatgcggcgg gaccagagaa aaatcactca 480
gggtcaatgc cagcgcttcg ttaatacaga tgtaggtgtt ccacagggta gccagcagca 540
tcctgcgatg cagatccgga acataatggt gcagggcgct gacttccgcg tttccagact 600
ttacgaaaca cggaaaccga agaccattca tgttgttgct caggtcgcag acgttttgca 660
gcagcagtcg cttcacgttc gctcgcgtat cggtgattca ttctgctaac cagtaaggca 720
accccgccag cctagccggg tcctcaacga caggagcacg atcatgcgca cccgtggcca 780
ggacccaacg ctgcccgaaa ttctgcagga attcgaatgg ccatgggacg tcgacctgag 840
gtaattataa cccgggccct atatatggat ccatttcaga tattaattgt taattctaga 900
actcatcggt tgaacaatcg gaaatcaaca attaatatct gattcaaata gggaaaagag 960
aattctcctt cgggaggatt ttttaataag cttacttaag ttaatttttt tcactattct 1020
aggttccctt cagtaatacg tttcaaaccc atctctttta ggttcgaaag ccatgtgatt 1080
tcacatcgct ttttgcccaa tgcaaaaata cctcttataa catctctctt cttgaattat 1140
attttttcaa tatgatcatt gtataagttc ttcttactcg taccttattt tatcaaattt 1200
gacagaattt tgatagaata gtagtaataa atttttgtaa gcggttttta cataatgaaa 1260
tatttaactt ttatcatgta aatcaatttt gggggaaaat ttttatgaat catattccag 1320
tttttggcga aaagaatgaa ggaaaagact atcaagcaag atatggcgtc tactcaattg 1380
tagcaagaga gaacaaagaa atttgccttg ttcaagcccc taacggaatt ccaaatcttt 1440
aaaatgcccc ttaaaattca aaataaaggc atttaaaatt taaatatttc ttgtgataaa 1500
gtttgttaaa aaggagtggt tttatgactg ttatgtggtt atcgatagat ctgcgcgcga 1560
tcgatatcag cgctttaaat ttgcgcatgc tagctatagt tctagaggta ccggttgtta 1620
acgttagccg gctacgtata ctccggaata ttaataggcc taggatgcat atggcggccg 1680
cctgcagcaa ctcttcgtaa gtcatcttgc ccaacgtgaa tttttctagt gctatatatg 1740
ttttctccgc tgaggtattc attctattat gacattcatt cactaactca ttttcatttg 1800
ttattggctc atgtatatgt tctttttttc gaaatgcttc ataatatctt ataccaaata 1860
ttctctggga catcgcattg atgtgaatta attcgatcat tttgtttatt gcaatcgtat 1920
tgctattaat cgcaacatca aaccaaaata aaagccccct tcgactttcg tcaggggggc 1980
ttttatttat tcaataatcc ctcctctcaa taaatctatt gttgtactta attcaacttc 2040
catttctctg tatctttcaa tacgctcttt taagtcctta atttcttttt ttaattcctc 2100
attttcagca aataactctt tttctttgtt tgtcatttta tttcccccgt ttcagcatca 2160
agaacctttg cataacttgc tctatatcca cactgataat tgccctcaaa ccataatcta 2220
aaggcgctag agtttgttga aacaatatct tttacatcat tcgtatttaa aattccaaac 2280
tccgctcccc taaggcgaat aaaagccatt aaatcttttg tatttaccaa attatagtca 2340
tccactatat ctaaaagtaa attcttcaat tctctttttt ggctttcatc aagtgttata 2400
tagcggtcaa tatcaaaatc attaatgttc aaaatatctt ttttgtcgta tatatgttta 2460
ttcttagcaa tagcgtcctt tgattcatga gtcaaatatt catatgaacc tttgatataa 2520
tcaagtatct caacatgagc aactgaacta ttccccaatt ttcgcttaat cttgttccta 2580
acgctttcta ttgttacagg atttcgtgca atatatataa cgtgatagtg tggtttttta 2640
tagtgctttc catttcgtat aacatcacta ctattccatg tatctttatc ttttttttcg 2700
tccatatcgt gtaaaggact gacagccata gatacgccca aactctctaa tttttctttc 2760
caatcattag gaattgagtc aggatataat aaaaatccaa aatttctagc tttagtattt 2820
ttaatagcca tgatataatt accttgtcaa aaacaagtag cgaaaactcg tatccttcta 2880
aaaacgcgag ctttcgctta ttttttttgt tctgattcct ttcttgcata ttcttctata 2940
gctaacgccg caaccgcaga ttttgaaaaa cctttttgtt tcgccatatc tgttaatttt 3000
ttatcttgct cttttgtcag agaaatcata actctttttt tcgattctga aatcaccatt 3060
taaaaaactc caatcaaata attttataaa attagtgtat cactttgtaa tcataaaaac 3120
aacaataaag ctacttaaat atagatttat aaaaaacgtt ggcgaaaacg ttggcgattc 3180
gttggcgatt gaaaaacccc tcaaaccctt gagccagttg ggatagagcg tttttggcac 3240
aaaaattggc actcggcact taatgggggg tcgtagtacg gaagcaaaat tcgcttcctt 3300
tcccccccat ttttttccaa attccaaatt tttttcaaaa attttccagc gctaccgctc 3360
ggcaaaattg caagcaattt ttaaaatcaa acccatgagg gaatttcatt ccctcaaact 3420
cccttgagcc tcctccaacc gaaatagaag gacgctgcgc ttattatttc attcagtcat 3480
cggctttcat aatctaacag acaacatctt cgctgcaaag ccacgctacg ctcaagggct 3540
tttacgctac gataacgcct gttttaacga ttatgccgat aactaaacga aataaacgct 3600
aaaacgtctc agaaacgatt ttgagacgtt ttaataaaaa atcgtgcggt aaagctcatc 3660
agcgtggtcg tgaagcagct tatcggataa taaatatata taaacgtata tagatttcat 3720
aaagtctaac acactagact tatttacttc gtaattaagt cgttaaaccg tgtgctctac 3780
gaccaaaact ataaaacctt taagaacttt ctttttttac aagaaaaaag aaattagata 3840
aatctctcat atcttttatt caataatcgc atccgattgc agtataaatt taacgatcac 3900
tcatcatgtt catatttatc agagctcgtg ctataattat actaatttta taaggaggaa 3960
aaaatatggg catttttagt atttttgtaa tcagcacagt tcattatcaa ccaaacaaaa 4020
aataagtggt tataatgaat cgttaataag caaaattcat ataaccaaat taaagagggt 4080
tataatgaac gagaaaaata taaaacacag tcaaaacttt attacttcaa aacataatat 4140
agataaaata atgacaaata taagattaaa tgaacatgat aatatctttg aaatcggctc 4200
aggaaaaggc cattttaccc ttgaattagt aaagaggtgt aatttcgtaa ctgccattga 4260
aatagaccat aaattatgca aaactacaga aaataaactt gttgatcacg ataatttcca 4320
agttttaaac aaggatatat tgcagtttaa atttcctaaa aaccaatcct ataaaatata 4380
tggtaatata ccttataaca taagtacgga tataatacgc aaaattgttt ttgatagtat 4440
agctaatgag atttatttaa tcgtggaata cgggtttgct aaaagattat taaatacaaa 4500
acgctcattg gcattacttt taatggcaga agttgatatt tctatattaa gtatggttcc 4560
aagagaatat tttcatccta aacctaaagt gaatagctca cttatcagat taagtagaaa 4620
aaaatcaaga atatcacaca aagataaaca aaagtataat tatttcgtta tgaaatgggt 4680
taacaaagaa tacaaga 4697
<210> 3
<211> 21
<212> DNA
<213> 人工合成
<400> 3
gatgatacat agccgacctg a 21
<210> 4
<211> 18
<212> DNA
<213> 人工合成
<400> 4
ttccctactg ctgcctcc 18
<210> 5
<211> 22
<212> DNA
<213> 人工合成
<400> 5
tctagaactc atcggttgaa ca 22
<210> 6
<211> 19
<212> DNA
<213> 人工合成
<400> 6
taaaaaatcc tcccgaagg 19
<210> 7
<211> 31
<212> DNA
<213> 人工合成
<400> 7
cgcggatcct cctccccttt catatttaga a 31
<210> 8
<211> 29
<212> DNA
<213> 人工合成
<400> 8
cccaagcttc aaattacttg agggcatcc 29
<210> 9
<211> 25
<212> DNA
<213> 人工合成
<400> 9
attactgaag ggaacctaga atagt 25
<210> 10
<211> 22
<212> DNA
<213> 人工合成
<400> 10
cgacctgagg taattataac cc 22
- 还没有人留言评论。精彩留言会获得点赞!