一种利用羰基还原酶制备克唑替尼中间体的方法与流程
本发明涉及生物制药技术领域,特别涉及一种利用羰基还原酶制备克唑替尼中间体的方法。
背景技术:
赛可瑞(Xalkori,克唑替尼)是由辉瑞公司研制的抑制Met/ALK/ROS的ATP竞争性的多靶点蛋白激酶抑制剂。分别在ALK、ROS和MET激酶活性异常的肿瘤患者中证实克唑替尼对人体有显著临床疗效。Xalkori是肿瘤药物研发史上最快速的药物之一,2011年在美国上市后引起轰动。
克唑替尼的化学结构式如下:
(S)-2’,6’-二氯-3’-氟苯乙醇是克唑替尼化学合成过程中一个重要的中间体。但目前主要以化学合成为主,但该方法步骤繁琐,产品收率低,污染大,不适于规模化生产(US7858643B2)。目前(S)-2’,6’-二氯-3’-氟苯乙醇的生物制备方法,在催化过程中需要使用昂贵的辅酶NADP和使用葡萄糖脱氢酶进行辅酶再生,不利于该中间体的廉价制备。
技术实现要素:
本发明的目的在于解决现有技术存在的上述不足,提供一种利用羰基还原酶制备克唑替尼中间体的方法,方法简单易行,产品收率高,生产成本低、环保,从而满足克唑替尼的大规模工业化生产要求。
本发明解决其技术问题所采用的技术方案是:
一种利用羰基还原酶制备克唑替尼中间体的方法,以2’,6’-二氯-3’-氟苯乙酮为底物,磷酸缓冲液为反应介质,加入羰基还原酶、辅因子及氢供体形成生物催化反应体系,发生生物催化反应生成(S)-2’,6’-二氯-3’-氟苯乙醇。
作为优选,所述羰基还原酶的氨基酸序列如SEQ ID NO.1所示,所述羰基还原酶的编码基因如SEQ ID NO.2所示。
作为优选,所述辅因子为NAD+。
作为优选,所述氢供体为异丙醇。
作为优选,每升生物催化反应体系中2’,6’-二氯-3’-氟苯乙酮的用量为50-100g。
作为优选,每升生物催化反应体系中羰基还原酶的用量为3-10g。
作为优选,每升生物催化反应体系中辅因子的用量为0.05-0.1g。
作为优选,每升生物催化反应体系中氢供体的用量为50-100mL。
作为优选,所述磷酸缓冲液的pH为6.5-7.5。
作为优选,所述生物催化反应的温度为25-35℃,反应时间8-12h。
本发明的有益效果是:本发明采用新型的羰基还原酶催化还原反应,具有生物催化剂使用量小、反应条件温和、反应时间短、收率高、光学纯度好等优势。与现有技术的化学法相比,经济性高、污染小,具有重要的工业应用价值。
附图说明
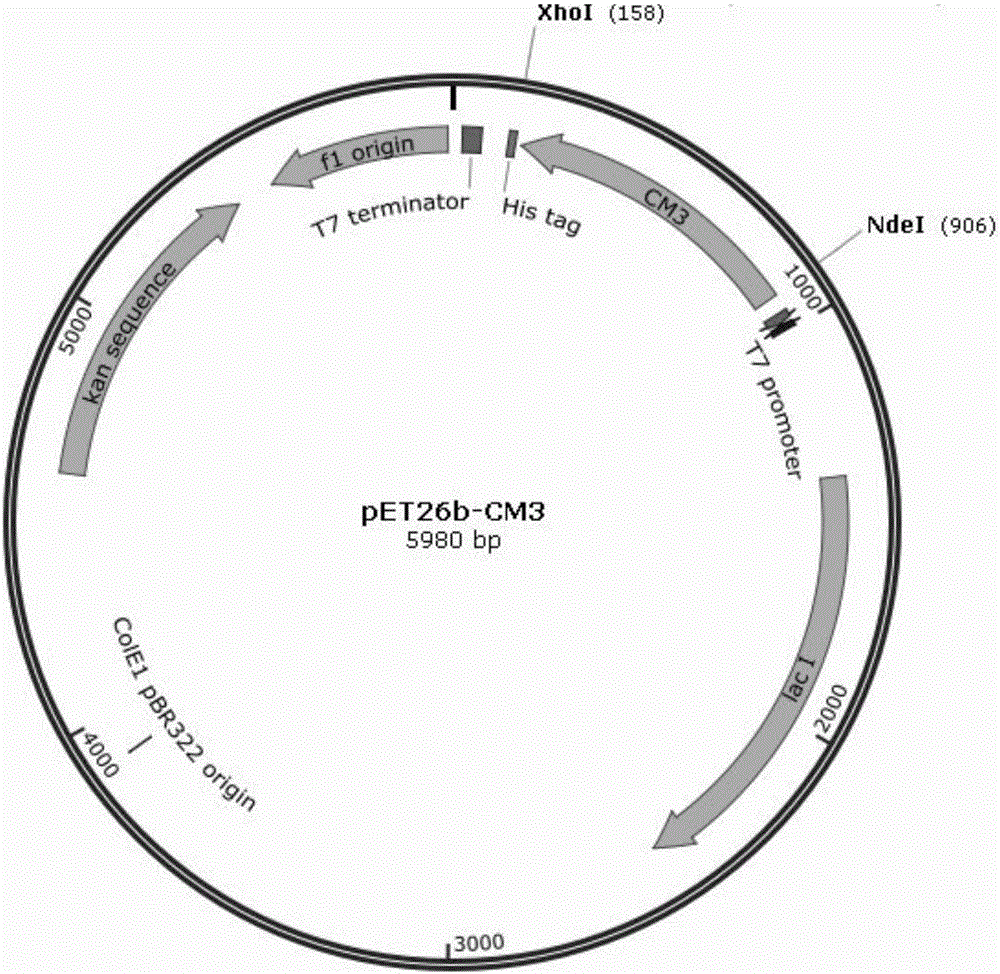
图1是本发明构建的重组羰基还原酶的表达质粒pET26-CM3;
图2是大肠杆菌BL21(DE3)/pET26-CM3诱导表达后重组羰基还原酶的SDS-PAGE电泳图;其中M是蛋白分子量标准品,1号是破碎后上清液,2号是纯化后的重组羰基还原酶。
图3是反应过程中底物和产物HPLC液相图。(A)反应0h的液相图;(B)反应6h后的液相图。
具体实施方式
下面通过具体实施例,对本发明的技术方案作进一步的具体说明。
本发明中,若非特指,所采用的原料和设备等均可从市场购得或是本领域常用的。下述实施例中的方法,如无特别说明,均为本领域的常规方法。
本发明的工艺路线如下:
实施例1:
(1)羰基还原酶编码基因的合成:根据羰基还原酶CM3的原始基因序列(genebank Sequence ID:ABB91667.1),按照大肠杆菌密码子分析用表对原始基因序列进行密码子优化并进行目的基因的全合成,基因合成委托上海捷瑞生物科技有限公司进行,获得的编码基因序列如SEQ ID NO.2所示,翻译后的羰基还原酶氨基酸序列如SEQ ID NO.1所示。合成后的编码基因两端分别带用NdeI和XhoI酶切位点并连接到pET26b(+)上,获得重组表达载体pET26b-CM3(见图1)。
(2)羰基还原酶的表达:
制备大肠杆菌BL21(DE3)感受态细胞,将重组表达载体pET26b-CM3通过热激转化法转到表达菌株BL21(DE3)中并涂布到含有50μg/mL的LB平板上(10g/L蛋白胨,0.5g/L酵母粉,10g/L氯化钠,20g/L琼脂,pH7.0),37℃过夜培养。次日,挑取单菌落,将单菌落接入到5mL含卡那霉素的LB培养液中,37℃,振荡培养过夜,次日取1mL菌液加入到含卡那霉素的100mL TB培养基中(12g/L蛋白胨,24g/L酵母粉,4mL/L甘油,0.1M磷酸缓冲液,pH 7.0),37℃下振荡培养至OD600至3.0,然后添加IPTG至终浓度为0.1mM,25℃下诱导培养过夜。图2说明重组羰基还原酶在大肠杆菌中获得了正确表达。
(3)细胞破碎:5000g离心10min后收集菌体,用0.1M磷酸缓冲液(pH7.0)缓冲液重悬菌体,超声破碎后所得液体即为粗酶液。
(4)生物催化反应:将5g底物2’,6’-二氯-3’-氟苯乙酮加入到含有85mL磷酸缓冲液(0.1M,pH7.0)的250mL反应器中,随后,将15mL含有5mL异丙醇,10mg NAD+,500mg羰基还原酶的0.1M磷酸缓冲液(pH7.0)加入该反应器中。在30℃下磁力搅拌反应,并用碱调节反应的pH值,使其维持在7.0左右。利用薄层层析法定性检测底物的消耗及产物的生成情况。HPLC检测底物完全消耗(约8-10小时),加NaCl至饱和,用乙酸乙酯萃取3次,合并有机相萃取。无水硫酸钠除水,抽滤,减压浓缩,得油状液体,手性ee值大于99,收率为90.5%。图3说明利用该重组羰基还原酶可以催化底物生成产物,6h后产物峰明显增大,底物峰减小。
(5)产物鉴定:利用核磁共振1H NMR对目标产物进行鉴定,(S)-2’,6’-二氯-3’-氟苯乙醇核磁数据1H NMR(400MHz,chloroform-D)δppm 1.65(d,J=6.8Hz,3H)5.58(q,J=6.9Hz,1H)6.96-7.10(m,1H)7.22-7.36(m,1H)。
实施例2:
本实施例与实施例不同之处在于步骤(4):
将100g底物2’,6’-二氯-3’-氟苯乙酮加入到含有850mL磷酸缓冲液(0.1M,pH7.0)的2.5L反应器中,随后,将150mL含有100mL异丙醇,50mg NAD+,10g羰基还原酶的0.1M磷酸缓冲液(pH7.0)加入该反应器中。在30℃下磁力搅拌反应,并用碱调节反应的pH值,使其维持在7.0左右。利用薄层层析法定性检测底物的消耗及产物的生成情况。HPLC检测底物完全消耗,加NaCl至饱和,用乙酸乙酯萃取3次,合并有机相萃取。无水硫酸钠除水,抽滤,减压浓缩,得油状液体,手性ee值大于99,产品收率为89.5%。
实施例3:
本实施例与实施例不同之处在于步骤(4):
将80g底物2’,6’-二氯-3’-氟苯乙酮加入到含有850mL磷酸缓冲液(0.1M,pH7.0)的2.5L反应器中,随后,将150mL含有70mL异丙醇,80mg NAD+,3g羰基还原酶的0.1M磷酸缓冲液(pH7.0)加入该反应器中。在30℃下磁力搅拌反应,并用碱调节反应的pH值,使其维持在7.0左右。利用薄层层析法定性检测底物的消耗及产物的生成情况。HPLC检测底物完全消耗,加NaCl至饱和,用乙酸乙酯萃取3次,合并有机相萃取。无水硫酸钠除水,抽滤,减压浓缩,得油状液体,手性ee值大于99,产品收率为88.6%。
以上所述的实施例只是本发明的一种较佳的方案,并非对本发明作任何形式上的限制,在不超出权利要求所记载的技术方案的前提下还有其它的变体及改型。
SEQUENCE LISTING
<110> 浙江海洋大学
<120> 一种利用羰基还原酶制备克唑替尼中间体的方法
<130> 2016.12
<160> 2
<170> PatentIn version 3.3
<210> 1
<211> 241
<212> PRT
<213> 人工序列
<400> 1
Met Thr Thr Thr Ser Asn Ala Leu Val Thr Gly Gly Ser Arg Gly Ile
1 5 10 15
Gly Ala Ala Ser Ala Ile Lys Leu Ala Gln Glu Gly Tyr Ser Val Thr
20 25 30
Leu Ala Ser Arg Ser Val Asp Lys Leu Asn Glu Val Lys Ala Lys Leu
35 40 45
Pro Ile Val Gln Asp Gly Gln Lys His Tyr Ile Trp Glu Leu Asp Leu
50 55 60
Ala Asp Val Glu Ala Ala Ser Ser Phe Lys Gly Ala Pro Leu Pro Ala
65 70 75 80
Ser Ser Tyr Asp Val Phe Val Ser Asn Ala Gly Val Ala Ala Phe Ser
85 90 95
Pro Thr Ala Asp His Asp Asp Lys Glu Trp Gln Asn Leu Leu Ala Val
100 105 110
Asn Leu Ser Ser Pro Ile Ala Leu Thr Lys Ala Leu Leu Lys Asp Val
115 120 125
Ser Glu Arg Pro Ala Asp Asn Pro Leu Gln Ile Ile Tyr Ile Ser Ser
130 135 140
Val Ala Gly Leu His Gly Ala Ala Gln Val Ala Val Tyr Ser Ala Ser
145 150 155 160
Lys Ala Gly Leu Asp Gly Phe Met Arg Ser Val Ala Arg Glu Val Gly
165 170 175
Pro Lys Gly Ile His Val Asn Ser Ile Asn Pro Gly Tyr Thr Lys Thr
180 185 190
Glu Met Thr Ala Gly Ile Glu Ala Leu Pro Asp Leu Pro Ile Lys Gly
195 200 205
Trp Ile Glu Pro Glu Ala Ile Ala Asp Ala Val Leu Phe Leu Ala Lys
210 215 220
Ser Lys Asn Ile Thr Gly Thr Asn Ile Val Val Asp Asn Gly Leu Ile
225 230 235 240
Ala
<210> 2
<211> 726
<212> DNA
<213> 人工序列
<400> 2
atgacgacta cttcaaatgc gctcgtcact ggaggcagcc gcggcattgg cgctgcttcc 60
gccattaagc tggctcagga gggctacagt gttacgctgg cctctcgcag tgttgataaa 120
ctgaatgaag taaaggcgaa actcccaatt gtacaggacg ggcagaagca ctacatttgg 180
gaactcgatc tggctgatgt ggaagctgct tcgtcgttca agggtgctcc tttgcctgct 240
agcagctacg acgtcttcgt ttcgaacgcg ggcgtcgctg cgttctcgcc cacagccgac 300
cacgatgata aggagtggca gaacttgctc gccgtgaact tgtcgtcgcc cattgccctc 360
acgaaggccc tcttgaagga cgtctccgaa aggcctgcgg acaatccgtt gcagattatc 420
tacatttcgt cggtggccgg cttgcatggc gccgcgcagg tcgccgtgta cagtgcatct 480
aaggccggtc ttgatggttt tatgcgctcc gtcgcccgtg aggtgggccc gaagggcatc 540
catgtgaact ccatcaaccc cggatacacc aagactgaaa tgaccgcggg cattgaagcc 600
ctgcctgatt tgcctatcaa ggggtggatc gagcccgagg caattgctga cgcggttctg 660
tttctggcaa agtccaagaa tatcaccggc acaaacattg tggtcgacaa tggcttgatt 720
gcttag 726
- 还没有人留言评论。精彩留言会获得点赞!