一种在生理pH条件下有活力的草酸氧化酶及其应用的制作方法
本发明涉及一种在生理ph条件下有活力的草酸氧化酶基因及其编码的蛋白与应用,尤其涉及该草酸氧化酶在用于制备降低草酸过多症疾病病人血液或尿液中草酸浓度的药物中的应用,属基因工程技术领域。
背景技术:
泌尿系结石疾病是一种全球范围内的重大健康疾病,绝大多数泌尿系结石由草酸钙或草酸钙与磷酸钙成分组成。许多疾病均与人体内的草酸量过高有关,比如原发性高草酸尿症、第二高草酸尿症、孤独症、外阴痛、草酸过多引起的晚期肾结石疾病、心脏传导障碍、克罗恩氏症、炎症性肠病、结肠炎、泌尿性结石、哮喘、慢性阻塞性肺病、纤维肌痛、zellweger综合征等,以及其他慢性疾病。原发性高草酸尿症(primaryhyperoxaluria,ph)包括i型,ii型和iii型,都是极为罕见的基因突变导致的乙醛酸盐代谢异常遗传疾病,该疾病因肝脏中特定代谢酶的缺失导致草酸在体内的过度积累:i型疾病因乙醛酸盐氨基转移酶缺失导致,ii型疾病因乙醛酸/羟基丙酮酸还原酶缺失导致,iii型疾病因肝脏特有的线粒体酶4-羟基-2-氧-戊二醛缩酶缺失导致,还有一些非以上三种类型的原发性高草酸尿疾病,原因正在进一步的研究中。原发性高草酸尿症疾病是一种非常严重的遗传病,晚期患者体内的血液中草酸浓度可达100μmol/l,远高于正常人草酸水平(<5μmol/l),伴随着患者肾功能的衰竭,过饱和的草酸钙会在肾脏、甲状腺、心肌层、骨骼、皮肤、血管及眼中沉积形成结石,损坏人体重要器官,危及生命。第二高草酸尿症(secondaryhyperoxaluria,sh)是一种食源性草酸吸收过度或食入大量草酸前体导致的疾病,原因包括胃肠道疾病引起的,如肠炎、囊胞性纤维症、短肠综合征、或富含草酸食物或草酸前体饮食偏好。第二高草酸尿症是一种非常常见的疾病,其危害没有原发性高草酸尿症严重,但是高血草酸及尿草酸浓度仍然会让患者患上草酸过多引起的慢性疾病或晚期尿路系统衰竭。
人体通过食源性吸收的草酸占泌尿草酸总量的10~70%不等,食物中的草酸可以通过整个胃肠道消化系统吸收,包括胃,小肠和大肠,因此在消化吸收系统器官中去除食物中的草酸可以有效的防止草酸被胃肠道吸收。业界公认,草酸钙浓度的过高会导致结石的形成,因此,降低草酸的浓度有助于降低结石形成的风险。
需要长期进行透析的慢性肾功能衰竭(crf)和肾功能衰竭晚期(esrf)病人也很容易导致草酸过多症。该类病人的肾功能缺失导致草酸无法被有效清除,同时透析过程中需要注射维生素c作为抗氧化药物,而其在人体内会被代谢转化为草酸。此类病人体内血液中草酸一般在30-90μmol/l。2006年数据显示,美国每年有34万人需要透析,该人数还在一直上升,因此,该类病人也处于患草酸过多疾病的风险之中。
zellweger综合征(zsd)病人也与草酸过多有很高的关联性(83%),zsd是一种过氧化物酶缺失导致的疾病,虽然zsd病人草酸代谢异常的机理尚不清楚,但是zsd病人体内的血草酸浓度与原发性高草酸尿症病人是差不多的。
迄今为止几乎没有有效的治疗措施可以显著的降低草酸过多症患者形成结石的风险,其中最主要被使用的一些方法仅仅是通过口服草酸降解酶或草酸降解细菌来限制食物中草酸的吸收。然而,这些方法都会遇到一个共同的挑战,那就是胃环境中的强酸性加上高活力的胃蛋白酶会降解草酸降解酶蛋白,肠道中的胰蛋白酶和糜蛋白酶也会降解草酸降解酶。而且,大多数已知的草酸降解酶的最适ph值均为弱酸性,所以在中性偏碱的肠道中的无活力或活力很低。口服草酸降解菌同样遇到胃的强酸环境和肠道的蛋白酶解的挑战,无法形成优势菌群发挥降草酸作用。
自然界中主要有两种草酸降解酶:草酸脱羧酶和草酸氧化酶(www.brenda-enzymes.org/),然而并没有发现一种草酸脱羧酶在生理环境(ph7.4)有降解草酸活力。第一次被明确验证有草酸氧化酶活力的蛋白来自于大麦,其在大麦萌发过程中起重要作用,故又被称为萌发蛋白(germinprotein)。所有文献中曾报道的萌发蛋白类似的(germ-like)草酸氧化酶,其均在酸性条件下具有降解草酸的活力。有文章(biochem.j.,1962,85,33)报道三角梅叶片在ph7.4条件下有草酸氧化酶活力,但是其提取溶液没有草酸氧化酶活力。报道的在ph>7下具有活力的草酸氧化酶,其氨基酸序列未知,wo2011/066282专利描述了一种草酸降解酶来自于真菌,其专利实施例5中的酶并没有被纯化出来,也没有任何序列信息,本专利发明人也曾尝试纯化该酶,但没有成功。makotok等人也曾发表过一篇文章(journaloftheinstituteofbrewing,vol115,2009,232-237),文章中说其来自于一种大麦种子和大麦芽的草酸氧化酶在中性ph下有活力。但是本专利发明人提取过相同种属的大麦中的草酸氧化酶,发现其在ph7.4并无草酸氧化酶活力。因此,寻找在生理ph条件下有活力的草酸氧化酶,并获得其氨基酸序列,实现基因工程重组表达,工业化生产,利用其降低人体中的草酸含量的治疗措施仍然是非常必要的。
技术实现要素:
为了克服现有技术中的不足,本申请筛选了上千种植物样本,测试其在生理ph条件(ph7.4)下是否有草酸氧化酶活力,筛选到甜菜茎,香蕉皮,三角梅,夜来香,紫茉莉,守宫木,麻风树等候选物种,然后通过基因克隆手段克隆到编码草酸氧化酶的基因序列,通过基因工程重组表达系统(大肠杆菌,毕赤酵母,烟草及豌豆)重组表达,验证基因序列的正确性以及其草酸氧化酶的活力性质。
本发明提供了一组在ph7.4条件下有活力的草酸氧化酶,可以降解草酸或草酸盐,其基因序列与seqidno.1-4中任意一项至少有50%的一致性。
本发明提供了一组在ph7.4条件下有活力的草酸氧化酶,可以降解草酸或草酸盐,其基因序列与seqidno.1-4中任意一项至少有55%,60%,65%,70%,75%,80%,85%,90%,95%,98%,99%,100%的一致性。
本发明的另一个目的是提供一组在ph7.4条件下有活力的草酸氧化酶,可以降解草酸或草酸盐,其氨基酸序列与seqidno.5-8中任意一项至少有50%的一致性。
本发明还提供一组在ph7.4条件下有活力的草酸氧化酶,可以降解草酸或草酸盐,其氨基酸序列与seqidno.5-8中任意一项有至少55%,60%,65%,70%,75%,80%,85%,90%,95%,98%,99%,100%的一致性。
以上草酸氧化酶可以从甜菜茎,香蕉皮,三角梅,夜来香,紫茉莉,守宫木,麻风树等植物物种中提取得到。
本发明还提供一组在ph7.4条件下有活力的草酸氧化酶,可以降解草酸或草酸盐,是用不同分子量的聚乙二醇修饰的草酸氧化酶。
所述的聚乙二醇的分子量是2kd,5kd,10kd,20kd,4臂-20kd,30kd,40kd或4臂-40kd中的任意一种。
本发明提供一种包含草酸氧化酶基因的表达载体,其中草酸氧化酶基因序列与seqidno.1-4中任意一项至少有50%的一致性。
本发明提供一种包含草酸氧化酶基因的表达载体,其中草酸氧化酶基因序列与seqidno.1-4中任意一项至少有55%,60%,65%,70%,75%,80%,85%,90%,95%,98%,99%,100%的一致性。
本发明提供一种重组表达菌种,包含以上表达载体,且能产生草酸氧化酶。
本发明提供一种转基因植物株,部分植物(包括叶片,根,花等)或植物细胞,其转化有含以上草酸氧化酶基因序列的表达载体。
本发明还提供了该草酸氧化酶在制备降低草酸过多症疾病病人草酸浓度的药物中的应用。所述的草酸过多症疾病包括原发性高草酸尿症,二型高草酸尿症,zellweger症,或慢性泌尿系统损坏性疾病等。
由于该草酸氧化酶在ph7.4条件下有酶活力,因此,含有草酸氧化酶的药物可以制成针剂或经肠溶材料包埋。
本发明提供的一组草酸氧化酶,它在ph7.4条件下的比活力不低于其最大比活力的20%,而且其是至今为止发现的唯一被公开的且被验证了的在ph7.4或ph7.0~8.0有活力的草酸氧化酶。该草酸氧化酶的发现为研发一种适用于治疗草酸过多疾病的重组草酸氧化酶药物提供了可能性。本发明也可以使用草酸氧化酶制备预防草酸过多疾病的药物。
附图说明
图1为毕赤酵母表达载体zα-5102图谱。
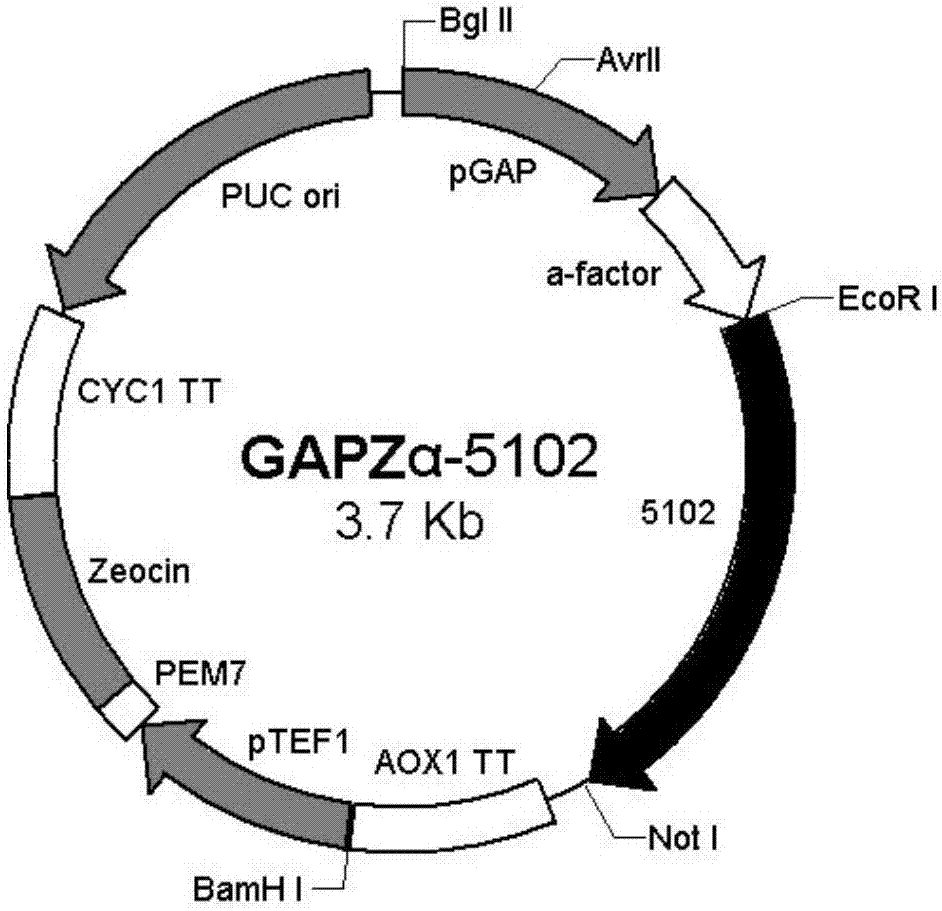
图2为毕赤酵母表达载体gapzα-5102图谱。
图3为毕赤酵母表达载体zα-5601图谱。
图4为毕赤酵母表达载体gapzα-5601图谱。
图5为毕赤酵母表达载体zα-30640图谱。
图6为sds-page分析毕赤酵母x33中草酸氧化酶zα-5102的表达。
图7为sds-page分析毕赤酵母x33中草酸氧化酶zα-5601的表达。
图8为sds-page分析毕赤酵母x33中草酸氧化酶zα-30640的表达。
图9为sds-page分析毕赤酵母x33中草酸氧化酶gapzα-5601的表达。
图10为植物表达载体图谱。
图11为烟草叶片中草酸氧化酶活力显色。
图12为草酸氧化酶活力检测,ph7.4。
左侧试管中含有2mm草酸,右侧试管中不含草酸。
图13为不同分子量peg修饰的草酸氧化酶在大鼠模型中对血草酸的影响结果图。
图14为不同分子量peg修饰的草酸氧化酶在大鼠模型中对尿草酸的影响结果图。
图15为草酸氧化酶b5102和不同分子量peg修饰的b5102样品免疫sd大鼠,其抗体效价结果图。
具体实施方式
下面通过实施例的方式进一步说明本发明,但并不因此将本发明限制在所述的实施例范围之中。下列实施例中未注明具体条件的实验方法,按照常规方法和条件,或按照商品说明书选择。
【实施例1】产草酸氧化酶植物的筛选与活力测试
本专利发明人从广西药用植物园,昆明植物园,武汉植物园及海南亚热带植物园采集了千余种植物样品,进行草酸氧化酶的筛选实验。
产草酸氧化酶植物筛选时,样品先经过处理,处理过程如下:将一定量的植物样品(叶,根,茎或花)用剪刀剪碎,放入定量的盛有纯水的烧杯中,用高速均质机匀浆为悬浊液(颗粒<100μm),匀浆液经离心后分为可溶部分(上清液)与不可溶部分(沉淀),不可溶部分用纯水重悬,两者分别用来测降解草酸的活力。
草酸氧化酶活力测试方法有以下两种:
(1)hplc方法:取40μl样品与360μl的12mm的草酸溶液(用naoh调节至ph7.4)混匀后,37℃反应48小时,反应液加终止液(100μl1.5m硫酸溶液)终止反应。反应液经高速离心去除沉淀,上清液过0.4μm膜后用hplc测定草酸含量。一个酶活单位定义为在上述体系中每分钟降低1微摩尔草酸所需要的酶量。
(2)显色法测草酸氧化酶活力:
20μl样品溶液与580μl活力显色液(含有10mm草酸;4mm3,5-二氯-2-羟基苯磺酸钠(dhbs);2u的辣根过氧化物酶;1mm的4-氨基安替比林(4-aa);50mm的磷酸缓冲液,ph6.0或7.4),37℃反应30min,用酶标仪监测od492的吸光度。一个酶活单位定义为在上述反应体系中每分钟降低1微摩尔草酸所需要的酶量。
经过筛选,我们发现香蕉皮(bananapeel),甜菜茎(beetstem),三角梅叶片(bougainvilleaspectabilisleaves),紫茉莉嫩芽(mirabilisjalapayoungleaf),夜来香叶片(telosmacordatum(brum.f.)),麻风树叶片(jatrophacarcasl.leaf),守宫木(sauropusandrogynus(l.)leaf)等植物样品在ph7.4的环境下有明确的草酸氧化酶活力。
【实施例2】香蕉和甜菜草酸氧化酶基因的克隆
(1)在甜菜茎及香蕉皮样品中提取草酸氧化酶粗品
称取一定量的甜菜茎及香蕉皮样品,切碎后放入盛有纯水的烧杯中,经高速匀浆机匀浆为小于100μm的颗粒,离心去除上清液(无活力或极低的草酸氧化酶活力),沉淀用含表面活性剂(1%硫磺酸脱氧胆酸钠)及蔗糖(50%w/v)的缓冲溶液(2mmpbs,ph7.0)重悬后,4℃抽提2~5天,草酸氧化酶的粗提物经q-sepharose色谱柱纯化,收集洗脱样品峰,后经透析除去样品中的表面活性剂及盐成分。草酸氧化酶样品经native-page分离后仍具有草酸氧化酶活性,通过native-page胶的草酸氧化酶活力显色后鉴定样品洗脱峰及草酸氧化酶条带,经sds-page胶分离后,草酸氧化酶条带被切取后委托中国农业大学分析测试中心做氮端(n-terminal)氨基酸测序及ms-ms质谱序列分析。
(2)基因组序列检索
根据n-terminal测序结果,将氮端的蛋白序列在已公布的香蕉及甜菜的转录基因组草图中进行t-blastn检索,得到候选的基因序列,然后与质谱的序列分析结果进行比对,确定目标草酸氧化酶基因序列。
(3)基因克隆
根据已确定的草酸氧化酶基因dna序列设计特异引物(表1),在香蕉及甜菜的cdna文库中进行pcr扩增,将pcr产物进行纯化回收后测序鉴定是否克隆到草酸氧化酶基因。本实验一共克隆到来自甜菜的b5100,b5102,b5601,来自香蕉的m30640等草酸氧化酶候选基因(如seqidno.1-4所示)。同时也克隆到了多条germin-like基因序列:108,122,303,1013(如seqidno.9-12所示)。
表1草酸氧化酶克隆引物
pcr体系(50μl):
pcr程序:
【实施例3】大肠杆菌系统表达重组草酸氧化酶
(1)质粒的构建
将草酸氧化酶(oxox)b5100,b5102,b5601,m30640基因克隆构建入pat载体(该载体是来源于pet-32a载体),然后将重组质粒转入e.coliorigamib感受态细胞。
(2)摇瓶小量表达
将b5100,b5102,b5601,m30640的甘油菌种涂布于lb琼脂糖平板(含100μg/ml氨苄抗生素),37℃倒置培养过夜至有单克隆菌斑出现。挑取单菌落接种于200ml摇瓶培养基中(含50mllb培养基,100μg/ml氨苄抗生素),220rpm,37℃培养,od600至0.6~0.8,加入mncl2终浓度为1mm,iptg终浓度为0.6mm进行诱导表达,诱导5h后,9500g离心15min收集菌体,菌体重悬于50mm精氨酸溶液中,冰浴超声破碎30min,并用50mm精氨酸重悬洗涤2次,包涵体中有80%为草酸氧化酶蛋白。菌体产量在该发酵条件下可达0.2~0.6g/l。
(3)7l发酵罐大量表达
将b5100,b5102,b5601,m30640基因单克隆菌斑接入500ml摇瓶(含200ml的lb培养基及100μg/ml的氨苄抗生素),过夜培养,作为种子液。将种子液按0.5%的接种量接入7l的发酵罐(含3.5l的基础培养基:甘油12g/l;酵母粉12g/l),200rpm,37℃培养,10%的氨水调节ph稳定在6.85,约8h后,开始匀速补加补料培养基(30g/l甘油,60g/l酵母粉),当od600达到20时,添加iptg至终浓度为0.8mm,继续诱导20h,放罐收集菌体。菌体产量在该发酵条件下可达1~5g/l。
(4)包涵体蛋白洗涤与复性
包涵体洗涤:
大肠杆菌菌体细胞用washingbuffer(50mmtris,50mmnacl,5mmedta,5mmdtt,1%tritonx-100,2m尿素,用盐酸调ph8.0)重悬,然后按照1:1000加入核酸酶(benzonase,含终浓度为0.5mm的mgcl2),搅拌15min,菌液变稀后进行高压均质,高压均质的条件为1100bar,反复破碎4次。将细胞破碎液进行8000g离心10min,弃上清,收集包涵体沉淀。包涵体用适量的washbuffer充分重悬,搅拌约30min,8000g离心10min,弃上清收集沉淀,反复洗涤三次后用纯水重悬包涵体,充分重悬后8000g离心10min,得到包涵体。sds-page电泳分析蛋白纯度,是否能够到达复性要求(纯度>80%)。
包涵体复性:
草酸氧化酶包涵体采用快速稀释方法复性:洗涤合格的包涵体溶解于8m尿素溶液中,测定蛋白浓度后,1ml/min匀速滴加到预冷的复性溶液中快速稀释复性,蛋白终浓度为100μg/ml,磁力搅拌混匀后置于4℃复性24h。复性溶液配方是以下所列任意一种:
1.20mmtris,300mmnacl,1mmmncl2,1mmgsh/0.2mmgssg,ph8.0
2.20mmtris,300mmnacl,1mmmncl2,1mmgsh/0.2mmgssg,400mmarginine,ph8.0
3.20mmtris,300mmnacl,1mmmncl2,1mmgsh/0.2mmgssg,100mmα-cyclodextrin,ph8.0
4.20mmtris,300mmnacl,1mmmncl2,1mmgsh/0.2mmgssg,2%β-cyclodextrin,ph8.0
5.20mmtris,300mmnacl,1mmmncl2,1mmgsh/0.2mmgssg,40%sucrose,ph8.0
6.20mmtris,300mmnacl,1mmmncl2,1mmgsh/0.2mmgssg,40%glucose,ph8.0
7.20mmtris,1mmmncl2,1mmgsh/0.2mmgssg,ph8.0
8.20mmtris,1mmmncl2,1mmgsh/0.2mmgssg,400mmarginine,ph8.0
9.20mmtris,1mmmncl2,1mmgsh/0.2mmgssg,50mmbetaine,ph8.0
10.20mmtris,1mmmncl2,50mmbetaine,ph8.0
11.20mmtris,0.5-5mmmncl2,1mmgsh/0.2mmgssg,50mmbetaine,ph8.0
12.20mmtris,1mmmncl2,1mmgsh/0.2mmgssg,50mmbetaine,ph4.0-10.0
13.20mmtris,1mmmncl2,50mmbetaine,0.05mm1-hexanol,50mmacetamide,300mmkcl,ph8.0
复性后蛋白经phenylsepharose树脂纯化,活力可达0.1~15u/mg蛋白。
缩略词:
1、gsh谷胱甘肽6、sucrose蔗糖
2、gssg氧化型谷胱甘肽7、glucose葡萄糖
3、arginine精氨酸8、betaine甜菜碱
4、α-cyclodextrinα-环糊精9、hexanol己醇
5、β-cyclodextrinβ-环糊精10、acetamide乙酰胺
【实施例4】毕赤酵母系统表达草酸氧化酶
(1)载体构建
将b5100,b5102,b5601,m30640基因通过xhoi和noti酶切位点克隆进ppiczαbandpgapzαa载体,构建的重组质粒为zα-5102,gapzα-5102,zα-5601,gapzα-5601,zα-30640(图1~5)。重组质粒zα-5102,zα-30640,zα-5601用pmei(mssi)内切酶进行线性化,gapza-b5102,gapzα-5601,gapzα-30640用blni(avrii)进行线性化。线性化后的载体通过电转方式转化毕赤酵母x33菌种进行表达。
表2.构建ppiczαbandpgapzαa表达载体引物名称及序列
pcr体系(50ul):
pcr程序:
(2)蛋白表达
用ypds培养基(10g/l酵母粉,20g/l蛋白胨,20g/l葡萄糖,1m山梨醇,20g/l琼脂,含100μg/lzeocin抗生素)平板筛选的阳性克隆,再用高浓度抗生素的ypds平板(1mg/mlzeocin)进行拷贝数的筛选,筛选到的高拷贝数的克隆子用摇瓶表达验证。
筛选到的高拷贝的zα-5102,zα-30640,zα-5601的克隆子接入4ml的ypd培养基,28℃培养18~20h,按4%的接种量转接500ml摇瓶(含50ml培养基)表达培养基bmgy(酵母粉1%(w/v),蛋白胨2%(w/v),100mm磷酸钾缓冲液ph6.0,无氨基酵母氮源1.34%(w/v),甘油1%(w/v),生物素0.04%(w/v)),28℃,220rpm生长18~20h后,od600到达3.0~6.0,离心收集菌体后重悬于bmmy培养基(酵母粉1%(w/v),蛋白胨2%(w/v),100mm磷酸钾缓冲液atph6.0,无氨基酵母氮源1.34%(w/v),1.0%(v/v)甲醇,生物素0.04%(w/v),5mmmncl2)进行诱导表达。诱导表达温度为28℃,每天补加甲醇至终浓度为1.0%,连续诱导90~120h。培养物经9500g离心后取上清液用于显色法测定草酸氧化酶活力及sds-page鉴定分析。
筛选到的高拷贝的gapza-b5102,gapzα-5601,gapzα-30640菌株,转接于bypd培养基(10g/l酵母粉,20g/l蛋白胨,20g/l葡萄糖,生物素400μg/l,mncl25mmand100mm磷酸钾缓冲液,ph6.0),28℃,220rpm震荡培养,48h后补加2%的葡萄糖,培养72h后,收集培养物离心,上清用于草酸氧化酶活力测定与sds-page分析。
结果显示,四种草酸氧化酶(b5100,b5102,b5601,m30640)均可以在酵母系统中成功表达,产量为0.01~0.1mg/ml培养基,sds-page分析显示目的条带为24kd(图6~9),显色法活力测试显示其在ph7.4下均有草酸降解活力。
【实施例5】植物系统表达草酸氧化酶
(1)载体构建
将b5102,b5601,m30640基因通过pcr扩增引入xbai及kpni酶切位点,然后经此两个位点插入phte载体,得到phte-5601,phte-5102andphte-30640(图10)。阳性重组质粒经菌液pcr及内切酶酶切验证后用冻融复苏法转入根癌农杆菌gv3101中。
表3构建植物表达载体引物名称及序列
pcr体系(50μl):
pcr程序:
(2)转化烟草叶片进行瞬时表达
野生型本生烟草(n.benthamiana)于温室中(16/8h光照/黑暗,25℃)培养5~8周。将转化有b5102,b5601,m30640基因的农杆菌菌种接种于3mllb培养基中(含25mg/l利福平及50mg/l的卡那霉素),28℃,220rpm过夜培养至od600>1.2作为种子液。种子液按1:100比例接入2llb培养基(含25mg/l利福平,50mg/l卡那霉素and20μm乙酰丁香酮),220rpm,28℃培养至od600为0.8~1.2(约18h)。5000rpm离心5min,去除上清,菌体沉淀用5倍体积的细菌重悬液(10mm2-(n-吗啡啉)乙烷磺酸钠(mes)ph5.5,10mmmgso4,和100mm乙酰丁香酮),室温培养4h。
将农杆菌菌液通过1ml或3ml注射器注入烟草叶片中进行侵染,每个注射点一般约有7平方厘米的范围会被侵染到,每个叶片需侵染几个点。侵染后的植物置于温室中生长7天左右,每天给予正常浇水和施肥。
(3)叶片草酸氧化酶活力测试实验
侵染后培养7天的烟草叶片被收获用于草酸氧化酶活力测试。测试方法为:侵染点周围叶片(含阴性对照组)被切为直径约1cm的圆盘状,叶片经高速匀浆后离心,上清和沉淀置于草酸氧化酶活力测定缓冲液中(40mm丁二酸,2.5mm草酸,5u/ml辣根过氧化物酶,0.6mg/ml4-氯-1-萘酚,60%(v/v)乙醇,ph5.0),室温孵育过夜显色,上清中未现酶活力,沉淀中呈现草酸氧化酶活力。
同样的侵染方法用于豌豆苗的侵染表达实验。
烟草和豌豆两种表达系统的结果显示,用于表达的3个草酸氧化酶基因均具有草酸氧化酶活力(图11,图12),其表达量约为0.01~5mg/g新鲜叶片,但是表达的绝大部分草酸氧化酶均存在于叶片的固体材料中,不可溶或不能有效溶出。
【实施例6】草酸氧化酶(大肠杆菌表达)在不同ph值条件下的活力测试
测试方法:
10μl草酸氧化酶溶液加入到190μl的活力测试溶液中(800mg/l4-氨基安替比林,4.8mm3,5-二氯-2-羟基苯磺酸钠,10u/ml辣根过氧化物酶,5mm草酸,不同ph缓冲液),混匀后置于37℃孵育10~60min,酶标仪读取od492吸光值。
不同ph缓冲液组成:ph4.5~6.0缓冲液选用50mm的柠檬酸缓冲液,ph6.0~8.0选用50mm的磷酸盐缓冲液。
测试结果显示,草酸氧化酶5100最大活力为18.2u/mg,5102最大活力为19.5u/mg,5601最大活力为18.7u/mg,30640最大活力为14u/mg。为了方便比较,我们将四种酶在不同ph值下的活力换算为相对活力整理到表3中:
表3四种草酸氧化酶的在不同ph值下的相对比活力
【实施例7】草酸氧化酶的peg化修饰
草酸氧化酶包涵体洗涤:
大肠杆菌细胞用washbuffer(50mmtris,50mmnacl,5mmedta,5mmdtt,1%tritonx-100,2m尿素,用盐酸调ph8.0)充分重悬(浓度约30g/l),然后按照1:1000加入核酸酶(benzonase,终浓度为0.5mm的mgcl2),搅拌15min,菌液变稀后进行高压均质。高压均质的条件为1100bar,反复破碎4次。细胞破碎液离心,8000g,10min,弃上清,收集沉淀。
包涵体清洗:包涵体用适量的washbuffer充分重悬,搅拌约30min,8000g离心10min,弃上清收集沉淀,反复洗涤三次后用纯水重悬包涵体,充分重悬后8000g离心10min,得到的包涵体称重,sds-page分析包涵体蛋白纯度,是否能够到达复性要求(纯度>80%)。
q柱变性纯化草酸氧化酶包涵体:
洗涤合格的包涵体(纯度>80%)溶解于8m尿素溶液(20mmtris-hcl,8murea,ph8.0),13000g,离心10min,弃去沉淀,上清上样与平衡好的q柱(平衡液平衡5柱体积),用洗脱液b(8murea,30g/lnacl,tris-hcl,ph8.0)洗脱6柱体积(cv)洗脱杂质蛋白,换用洗脱液c(8murea,160g/lnacl,tris-hcl,ph8.0)洗脱,收集目标样品峰,经q柱纯化,草酸氧化酶包涵体蛋白纯度可达95%,纯化后的样品用超滤管浓缩至5mg/ml用于下一步的复性实验。
草酸氧化酶包涵体蛋白复性
按实施例3中叙述进行操作。
phenylsepharose树脂纯化
上样前,phenylsepharose柱用平衡液(10mmtris-hcl,500mmnacl,ph8.0)平衡5柱体积(cv)。在复性后的草酸氧化酶b5102中加入nacl至终浓度为0.5m,混匀后过0.45μm的滤膜澄清。澄清液上样与平衡好的phenylsepharose柱子,用washbuffer(10mmtris-hcl,250mmnacl,ph8.0)洗涤1.5柱体积后换用洗脱液(10mmtris-hcl,20%isopropanol,ph7.0)洗脱。收集目标蛋白样品峰,并加nacl至50mm进行盐析沉淀,盐析后样品经12000g离心10min,弃去上清,沉淀重新溶解于硼酸缓冲液(10mmna2b4o7~h3bo3,ph9.0)中,用于后期的peg化修饰。纯化得到的样品进行sds-page分析蛋白纯度,草酸氧化酶活力测试评定复性效果。
草酸氧化酶的peg化修饰
溶解于硼酸盐溶液的草酸氧化酶b5102蛋白用硼酸缓冲液调整浓度至2mg/ml。按b5102蛋白上的赖氨酸的摩尔数的10倍比列投入mpeg-sc(methoxypegsuccinimidylcarboxymethylester)修饰物到温和磁力搅拌的b5102蛋白溶液中进行peg化修饰反应,混匀后28℃反应6h,加入与mpeg摩尔量相等的甘氨酸终止反应。我们试验了2kd,5kd,10kd,20kd,4臂-20kd,30kd,40kdand4臂-40kd的peg修饰物。
peg修饰后的b5102样品用分子排阻色谱(gehiloadsuperdex16/600gl,usa)进行分离纯化。分子排阻色谱在上样前用磷酸缓冲液(10mmk2hpo4~kh2po4,ph7.4)进行平衡5柱体积,按柱体积的5%进行上样,然后再用磷酸缓冲液进行洗脱,流速1ml/min,收集样品峰。
【实施例8】不同分子量peg修饰的草酸氧化酶在大鼠模型中降低血草酸及尿草酸实验
30只5周龄sd雄性大鼠,体重约为130~150g,购买自湖北省动物实验中心。大鼠饲养与独立供气笼(ivc)系统(温度18~26℃,湿度40~70%)中适应一周后,随机分入大鼠代谢笼中,分为5组,其中一组为对照组,四组为实验组,每组6只大鼠。经过适应期后,对照组和实验组大鼠的血草酸和尿草酸均稳定后,3种不同分子量peg(20k,30k,40k)修饰的草酸氧化酶b5102通过尾静脉注射方式给药到不同的实验组大鼠,其中一组实验组大鼠注射相同剂量体积的生理盐水作为安慰剂组,药物给药剂量为0.2mg/天/次,连续给药3天,对照组大鼠没有做任何治疗处理。对照组大鼠喂常规鼠粮和饮水,实验组大鼠喂常规鼠粮和1%乙二醇饮水。给药后,所有大鼠的血草酸和尿草酸每天进行检测监控。每天通过尾静脉采集0.3ml血液用于测定血草酸浓度,新采集的血液经5000g离心5min后收集血清,加入盐酸调节ph至2.0~4.0,防止血清中维生素c(vc)转化为草酸。尿液每12h(8:30am和8:30pm)收集一次,并用盐酸调节ph至1.5~2.0,防止vc转化为草酸。所有大鼠的血草酸和尿草酸均被检测,稳定后可以进行药效实验。
血草酸测定采用amplexred(10-acetyl-3,7-dihydroxyphenoxazine)荧光酶标法检测,检测流程如下:
(1)盐酸酸化后的血清样品用10k超滤膜离心管5000g离心30min超滤去除血清蛋白,温度4℃。
(2)将超滤液加入96孔荧光酶标板(全黑色不透明)中,每个样品点6个平行孔,前3个孔加入草酸反应试剂(100mm柠檬酸缓冲液,ph5.4,10μmamplexred,1u/mlhrp,0.1u/ml草酸氧化酶),后3个孔加入背景校准试剂(100mm柠檬酸缓冲液,ph5.4,10μmamplexred,1u/mlhrp),震荡混匀后25℃孵育30min。
(3)用荧光酶标仪(激发光波长:538nm,发射光波长:590nm)检测荧光值。
(4)根据样品净荧光值(草酸测试组平均荧光值-背景校准组平均荧光值)与草酸标准曲线荧光值进行对照,计算样品中的草酸浓度。
尿草酸浓度测定采用美国trinity公司的草酸测定试剂盒,具体操作步骤严格按照操作说明书执行。
结果显示,与安慰剂组相比,给药组(20k,30k,40kpeg-oxox-b5102)可以降40%~50%血草酸浓度,同时降20%~40%的尿草酸浓度。(图13和图14)
【实施例9】peg-oxox免疫原性的测定
草酸氧化酶b5102和不同分子量peg(2k,5k,10k,20k,30k,40k)修饰的b5102样品通过尾静脉注射sd大鼠进行免疫原性评估。注射剂量为0.2mg/次,每周一次,持续免疫四周,第一次免疫前及免疫后不同时间点取血清样品测定抗体效价,评估免疫原性(免疫前0天,第7天,第14天,第21天,第28天,第45天,第60天)。
测定流程如下:
(1)用包被液(50mmcarbonate/bicarbonatebuffer,ph9.6)稀释peg修饰的b5102蛋白样品至10μg/ml,点100μl至高吸附96孔酶标板孔中,用锡箔纸包裹置于4℃过夜包被。
(2)将包被板置于室温,倒空液体并拍干残留液体,用300μl洗涤液(1.5mmkh2po4;8.1mmna2hpo4·12h2o;136mmnacl;2.7mmkcl;0.05%tween-20)清洗两次。
(3)每个孔中加300μl封闭液(1.5mmkh2po4;8.1mmna2hpo4·12h2o;136mmnacl;2.7mmkcl;5%脱脂奶粉),孵育1h。
(4)倒空液体并拍干残留液体,用300μl洗脱液清洗两次。
(5)将血清样品用洗涤液做梯度稀释(50倍,100倍,200倍,400倍,800倍,……)混匀后加100μl至每酶标孔中,37℃孵育1h或室温孵育3h。
(6)倒空液体并拍干残留液体,每个孔中加满洗涤液,倒空液体并拍干残留液体,重复3次。
(7)用洗涤液将hrp偶联的羊抗大鼠抗体(二抗)稀释到最优浓度(一般为1/5000~1/20000),并向每个测试孔中加入100μl二抗,室温孵育1h。
(8)倒空液体并拍干残留液体,每个孔中加满洗脱液,倒空液体并拍干残留液体,重复3~5次。
(9)将6mltmb试剂a(0.5mmedta-na;5mm柠檬酸;10%甘油;0.04%tetramethylbenzidine)和6ml试剂b(165mm乙酸钠;8.3mm柠檬酸;0.06%30%-h2o2)混匀。
(10)每个孔中加100μltmb显色液,室温孵育30min后,每个孔中加100μl终止反应液(2mh2so4)。
(11)酶标仪测定od450吸光值。
结果显示,草酸氧化酶的抗体效价在第28天时达到最高值。第28天的结果(图15)显示草酸氧化酶经peg修饰后,草酸氧化酶的抗体效价显著下降,且随着修饰peg分子量的增大,下降幅度更大,20k,30k,40kpeg修饰的草酸氧化酶几乎检测不到抗体。
sequencelisting
<110>武汉康复得生物科技股份有限公司
<120>一种在生理ph条件下有活力的草酸氧化酶及其应用
<160>34
<170>patentinversion3.3
<210>1
<211>651
<212>dna
<213>beetstem
<400>1
tctgatcctggtctcctacaggatttttgtgtgggtgtaaatgaccctgattcagcagtg60
tttgtaaatggaaaattctgcaagaacccaaaagacgtgacaatcgacgatttcttatac120
aaagggtttaatattccctcagacacaaacaacactcaaagagcagaagccacactagta180
gatgtcaatcgatttccagcacttaacacattaggtgtagccatggctcgtgtagacttt240
gcgtcctttggcctaaacacacctcatttgcaccctcgtggttctgagatattcgcggtg300
ctagaggggactttatatgccggcattgtcaccaccgattacaagcttttcgacacggtg360
ttgagaaagggtgacatgattgttttccctcaaggcttaatccacttccagcttaatctt420
ggcaagacagatgctcttgctattgcctcttttgggagccaatttcctggacgagttaat480
gttgctaatggtgtctttggaactacgccacaaattttggatgatgtacttacccaagcg540
tttcaggtagatgagatggtgattcagcaacttcgatctcagttttcaggtcaaaacata600
tcaatcaacactggaagatctattcttaaactcttaactgatgttgcttga651
<210>2
<211>651
<212>dna
<213>beetstem
<400>2
tctgatcctggtctcctacaggatttttgtgtgggtgtaaatgaccctgattcagcagtg60
tttgtaaatggaaaattctgcaagaacccaaaagacgtgacaatcgacgatttcttatac120
aaagggtttaatattccctcagacacaaacaacactcaaagagcagaagccacactagta180
gatgtcaatcgatttccagcacttaacacattaggtgtagccatggctcgtgtagacttt240
gcgtcctttggcctaaacacacctcatttgcaccctcgtggttctgagatattcgcggtc300
ctagaggggactttatatgccggcattgtcaccaccgataataagcttttcgacacggtg360
ttgagaaagggtgacatgattgttttccctcaaggcttaatccacttccagcttaatctt420
ggcaagacagatgctcttgctattgcctcttttgggagccaatttcctggacgagttaat480
gttgctaatggtgtctttggaactacgccacaaattttggatgatgtacttacccaagcg540
tttcaggtagataagatggtgattgagcaacttcgatctcagttttcaggtccaaacaca600
tcaatcaacactggaagatctattcttaaactcttaactgatgttgcttga651
<210>3
<211>651
<212>dna
<213>beetstem
<400>3
tccgatcctgcaccccttcaagatttttgtattgctgtaaatgatcccaattctgcagtg60
cttgtgaatggaaagctttgtaagaacccaaaagaagtgacaatagatgatttcttgtac120
aaagggtttaatatacctgcagacacaaacaacactcaaggagcaagtgccacactagtg180
gacattactctattccctgcagttaacacacaaggagtctccatggctcgtgtggacttt240
gcgccatttggactaaacacccctcatttacatcctcgtggctcagaggttttcgcagtg300
atggaagggattatgtatgctggttttgtgaccactgattataagctctatgatacaatt360
ataaaaaagggtgatattattgtgtttccacaaggtctaattcatttccaacttaatctt420
gggaagacagatgctttagcaattgcctcatttgggagccaaaatccagggagaattaat480
atcgctgacagtgtgtttggtactactccgcgtgttctagatgatgtgcttaccaaagga540
tttcaaatcgatgagttgttggtcaagcaacttcgttctcagttttctactgataatata600
tcaacaagcactggaaggtcatttttgaaattgctatctgaaacttattga651
<210>4
<211>642
<212>dna
<213>bananapeel
<400>4
tttgatccgagtcctctccaagacttttgcgttgctgactacgactccaacgtgtttgtg60
aacggattcgcctgcaagaaagctaaggatgtcacggcagatgacttctacttcaccggc120
ttagacaagcccgcgagcaccgccaacgagcttggcgcaaacatcactctcgtcaacgtg180
gaacgactcccaggcctcaactcccttggcgtcgccatgtctcgcatcgactacgcgccc240
ttcggtctcaaccctcctcactcgcatccacgatcgtcggagatactgcacgtggcggaa300
ggaacgctctacgccggcttcgtcacctccaacacggaaaacggcaaccttctcttcgct360
aagaagctgaagaagggcgacgcgtttgtgttccccaggggcctcatacacttccagttc420
aacatcggggacaccgatgcggtggcgttcgctaccttcggcagccagagcccgggtctc480
gtcaccaccgccaacgcactgttcggatcgaagccgcccatcgctgattacattcttgcc540
caggccgtgcagcttagcaagacgaccgtgggctggcttcagcagcagcagtggttggac600
atcgctcaagaatatggacaacgcttagttcaagctaattga642
<210>5
<211>216
<212>prt
<213>beetstem
<400>5
seraspproglyleuleuglnaspphecysvalglyvalasnasppro
151015
aspseralavalphevalasnglylysphecyslysasnprolysasp
202530
valthrileaspasppheleutyrlysglypheasnileproserasp
354045
thrasnasnthrglnargalaglualathrleuvalaspvalasnarg
505560
pheproalaleuasnthrleuglyvalalametalaargvalaspphe
65707580
alaserpheglyleuasnthrprohisleuhisproargglyserglu
859095
ilephealavalleugluglythrleutyralaglyilevalthrthr
100105110
asptyrlysleupheaspthrvalleuarglysglyaspmetileval
115120125
pheproglnglyleuilehispheglnleuasnleuglylysthrasp
130135140
alaleualailealaserpheglyserglnpheproglyargvalasn
145150155160
valalaasnglyvalpheglythrthrproglnileleuaspaspval
165170175
leuthrglnalapheglnvalaspglumetvalileglnglnleuarg
180185190
serglnpheserglyglnasnileserileasnthrglyargserile
195200205
leulysleuleuthraspvalala
210215
<210>6
<211>216
<212>prt
<213>beetstem
<400>6
seraspproglyleuleuglnaspphecysvalglyvalasnasppro
151015
aspseralavalphevalasnglylysphecyslysasnprolysasp
202530
valthrileaspasppheleutyrlysglypheasnileproserasp
354045
thrasnasnthrglnargalaglualathrleuvalaspvalasnarg
505560
pheproalaleuasnthrleuglyvalalametalaargvalaspphe
65707580
alaserpheglyleuasnthrprohisleuhisproargglyserglu
859095
ilephealavalleugluglythrleutyralaglyilevalthrthr
100105110
aspasnlysleupheaspthrvalleuarglysglyaspmetileval
115120125
pheproglnglyleuilehispheglnleuasnleuglylysthrasp
130135140
alaleualailealaserpheglyserglnpheproglyargvalasn
145150155160
valalaasnglyvalpheglythrthrproglnileleuaspaspval
165170175
leuthrglnalapheglnvalasplysmetvalilegluglnleuarg
180185190
serglnpheserglyproasnthrserileasnthrglyargserile
195200205
leulysleuleuthraspvalala
210215
<210>7
<211>216
<212>prt
<213>beetstem
<400>7
seraspproalaproleuglnaspphecysilealavalasnasppro
151015
asnseralavalleuvalasnglylysleucyslysasnprolysglu
202530
valthrileaspasppheleutyrlysglypheasnileproalaasp
354045
thrasnasnthrglnglyalaseralathrleuvalaspilethrleu
505560
pheproalavalasnthrglnglyvalsermetalaargvalaspphe
65707580
alapropheglyleuasnthrprohisleuhisproargglyserglu
859095
valphealavalmetgluglyilemettyralaglyphevalthrthr
100105110
asptyrlysleutyraspthrileilelyslysglyaspileileval
115120125
pheproglnglyleuilehispheglnleuasnleuglylysthrasp
130135140
alaleualailealaserpheglyserglnasnproglyargileasn
145150155160
ilealaaspservalpheglythrthrproargvalleuaspaspval
165170175
leuthrlysglypheglnileaspgluleuleuvallysglnleuarg
180185190
serglnpheserthraspasnileserthrserthrglyargserphe
195200205
leulysleuleusergluthrtyr
210215
<210>8
<211>213
<212>prt
<213>bananapeel
<400>8
pheaspproserproleuglnaspphecysvalalaasptyraspser
151015
asnvalphevalasnglyphealacyslyslysalalysaspvalthr
202530
alaaspaspphetyrphethrglyleuasplysproalaserthrala
354045
asngluleuglyalaasnilethrleuvalasnvalgluargleupro
505560
glyleuasnserleuglyvalalametserargileasptyralapro
65707580
pheglyleuasnproprohisserhisproargsersergluileleu
859095
hisvalalagluglythrleutyralaglyphevalthrserasnthr
100105110
gluasnglyasnleuleuphealalyslysleulyslysglyaspala
115120125
phevalpheproargglyleuilehispheglnpheasnileglyasp
130135140
thraspalavalalaphealathrpheglyserglnserproglyleu
145150155160
valthrthralaasnalaleupheglyserlysproproilealaasp
165170175
tyrileleualaglnalavalglnleuserlysthrthrvalglytrp
180185190
leuglnglnglnglntrpleuaspilealaglnglutyrglyglnarg
195200205
leuvalglnalaasn
210
<210>9
<211>639
<212>dna
<213>germin-like
<400>9
atggctcccctactctaccttgtagtattcttgcttgctccttttctctcccatgctgcg60
gatcccgatcctttgctagatttttgtgtagcggaccttaatgcctctccctcatttgct120
aatttcccttgcaaacaaacctcaaatgtgacctctgaagatttcttctttgatgggttt180
atgaatgagggaaacacatcaaactcgtttggatcaagggtcacacccggaaacgtcctc240
acatttcctgcccttaatatgctcgggatttcaatgaatcgggttgatcttgctgtggat300
gggatgaacccgccccattcccacccacgagcaagtgagagcggtgtggtgatgaagggg360
agagttctagtagggttcgtaaccacggggaatgtgtactattcaaaggtgttggttcca420
ggacagatgtttgtaatcccaagggggttggttcattttcaaaagaatgttggacaaaat480
aaggcactcatcattacagctttcaatagtcagaatccaggagtagtgttattatcctca540
accctgtttggtacaaacccttcaattccagatgatgttttaagccaaactttcctagtg600
gaccagagcattgtcgaaggaataaaatccaacttttga639
<210>10
<211>718
<212>dna
<213>germin-like
<400>10
atggaagtcgtcgcagctgtatcttttctggccgtgttattggctctggtttcccctgcc60
ctcgccaatgatcctgatatcttttctggccgtgttattggctctggtttcccctgccct120
cgccaatgatcctgatatgctccaagatgtttgtgtcgctgattccacctctggagtgaa180
attgaatggatttgcatgcaaggatgcagcaagcattacaccagaagacttcttctttgc240
tggaatatccaaacccggaatgacaaacaatacaatgaaatccctagtaaccggagctaa300
cgtcgaaaagataccgggtttaaacacactcggagtgtccatgggtcgtatcgacttcgg360
cccaggtggtcttaacccacctcacactcacccacgagccacagaaatggtctttgtgtt420
atatggagaattggacgttggtttcctaactacttctaataagctcatttctaagcatat480
taaaactggtgaaacttttgtttttcctagagggttagtccactttcagaaaaataatgg540
ggataaacctgctgctttagtcactgcttttaatagtcagttgcctggcacccaatcaat600
agctgccacgttgtttacgtcgaccccacctgttccagataatgttttaactatgacttt660
ccaagtcggtactaaacaagtccagaagatcaaggctaggctcgctcctaagaagtaa718
<210>11
<211>669
<212>dna
<213>germin-like
<400>11
atggcggctgtttgggtagtcttggtggtgctagcggcggcttttgctgttggggtcttt60
gccagcgatcctgatatgcttcaagatgtttgtgttgctgatcgtacatctggaatatta120
gtgaatggattcacatgtaaaaatatgaccatgataacccctgaagacttcttcttcacc180
ggaatttcacaaccaggccaaatcacaaataaaatccttggttctcgagtcaccggagcg240
aatgtgcaggacatccctggtctcaacaccttgggagtctcgatggctcgtgtcgacttt300
actccctacggtctaaacccacctcacattcaccctagaatcgtccaccctcgtgccact360
gaaatgatctatgttcttaagggtgaattgtacgttggttttataacgaccgacaataag420
ctcatttccaaggttgttaaagctggagaagtatttgttttccctagaggtttggctcac480
tttcagaaaaacatgttgaaatatccagctgctgcattagctgccttcaacagccaactt540
ccaggcactcaacaatttgcagctgctctctttacttccaatcctcctgtgtctaatgat600
gtgttggctcaggcttttaacattgacgaacacaatgtcaaaaagattagggctggcctt660
actccatag669
<210>12
<211>660
<212>dna
<213>germin-like
<400>12
atggagtcgcactacacgaagagaccattcctcctctttctctccttcaccgtcctcctc60
gtgttgatccgcgctgaccctgatcctctccaggacttctgcgtcgccgacctcggagct120
actgtggtcgtcaatgggttcccgtgcaagcccgcgtccggagtcacgtccgacgacttc180
ttcttcgccggactgtccagggagggcaacaccagcaatatcttcgggtccaacgtgacc240
aacgccaacatgctcagcttcccggggctcaacaccctcggcgtctccatgaaccgcgtc300
gacgtcgcccccggcggcaccaacccgccccacagccacccgagggctaccgagctcatc360
atcctcctcaagggccggctgctggtggggttcatcagcaccagtaaccagttcttctcc420
aaggtcttgaaccccggcgagatgttcgtggtgcccaaggggctcatccacttccagtac480
aacgtcggcaaggagaaggcgctcgccatcaccaccttcgacagccagctccccggagta540
gtgatcgcctccaccaccctgttcgcatcgaatccggcgattcccgacgatgtgctggcc600
aaagcttttcaggtggacgcgaaggtcgtcgctctcatcaagtccaagtttgagagataa660
<210>13
<211>212
<212>prt
<213>germin-like
<400>13
metalaproleuleutyrleuvalvalpheleuleualapropheleu
151015
serhisalaalaaspproaspproleuleuaspphecysvalalaasp
202530
leuasnalaserproserphealaasnpheprocyslysglnthrser
354045
asnvalthrsergluaspphephepheaspglyphemetasnglugly
505560
asnthrserasnserpheglyserargvalthrproglyasnvalleu
65707580
thrpheproalaleuasnmetleuglyilesermetasnargvalasp
859095
leualavalaspglymetasnproprohisserhisproargalaser
100105110
gluserglyvalvalmetlysglyargvalleuvalglyphevalthr
115120125
thrglyasnvaltyrtyrserlysvalleuvalproglyglnmetphe
130135140
valileproargglyleuvalhispheglnlysasnvalglyglnasn
145150155160
lysalaleuileilethralapheasnserglnasnproglyvalval
165170175
leuleuserserthrleupheglythrasnproserileproaspasp
180185190
valleuserglnthrpheleuvalaspglnserilevalgluglyile
195200205
lysserasnphe
210
<210>14
<211>219
<212>prt
<213>germin-like
<400>14
metgluvalvalalaalavalserpheleualavalleuleualaleu
151015
valserproalaleualaasnaspproaspmetleuglnaspvalcys
202530
valalaaspserthrserglyvallysleuasnglyphealacyslys
354045
aspalaalaserilethrprogluaspphephephealaglyileser
505560
lysproglymetthrasnasnthrmetlysserleuvalthrglyala
65707580
asnvalglulysileproglyleuasnthrleuglyvalsermetgly
859095
argileasppheglyproglyglyleuasnproprohisthrhispro
100105110
argalathrglumetvalphevalleutyrglygluleuaspvalgly
115120125
pheleuthrthrserasnlysleuileserlyshisilelysthrgly
130135140
gluthrphevalpheproargglyleuvalhispheglnlysasnasn
145150155160
glyasplysproalaalaleuvalthralapheasnserglnleupro
165170175
glythrglnserilealaalathrleuphethrserthrproproval
180185190
proaspasnvalleuthrmetthrpheglnvalglythrlysglnval
195200205
glnlysilelysalaargleualaprolyslys
210215
<210>15
<211>217
<212>prt
<213>germin-like
<400>15
metalaalavaltrpvalvalleuvalvalleualaalaalapheala
151015
valglyvalphealaseraspproaspmetleuglnaspvalcysval
202530
alaaspargthrserglyileleuvalasnglyphethrcyslysasn
354045
metthrmetilethrprogluaspphephephethrglyilesergln
505560
proglyglnilethrasnlysileleuglyserargvalthrglyala
65707580
asnvalglnaspileproglyleuasnthrleuglyvalsermetala
859095
argvalaspphethrprotyrglyleuasnproprohisilehispro
100105110
argalathrglumetiletyrvalleulysglygluleutyrvalgly
115120125
pheilethrthraspasnlysleuileserlysvalvallysalagly
130135140
gluvalphevalpheproargglyleualahispheglnlysasnmet
145150155160
leulystyrproalaalaalaleualaalapheasnserglnleupro
165170175
glythrglnglnphealaalaalaleuphethrserasnproproval
180185190
serasnaspvalleualaglnalapheasnileaspgluhisasnval
195200205
lyslysileargalaglyleuthrpro
210215
<210>16
<211>219
<212>prt
<213>germin-like
<400>16
metgluserhistyrthrlysargpropheleuleupheleuserphe
151015
thrvalleuleuvalleuileargalaaspproaspproleuglnasp
202530
phecysvalalaaspleuglyalathrvalvalvalasnglyphepro
354045
cyslysproalaserglyvalthrseraspaspphephephealagly
505560
leuserarggluglyasnthrserasnilepheglyserasnvalthr
65707580
asnalaasnmetleuserpheproglyleuasnthrleuglyvalser
859095
metasnargvalaspvalalaproglyglythrasnproprohisser
100105110
hisproargalathrgluleuileileleuleulysglyargleuleu
115120125
valglypheileserthrserasnglnphepheserlysvalleuasn
130135140
proglyglumetphevalvalprolysglyleuilehispheglntyr
145150155160
asnvalglylysglulysalaleualailethrthrpheaspsergln
165170175
leuproglyvalvalilealaserthrthrleuphealaserasnpro
180185190
alaileproaspaspvalleualalysalapheglnvalaspalalys
195200205
valvalalaleuilelysserlysphegluarg
210215
<210>17
<211>25
<212>dna
<213>artificial
<223>b5100/5102f
<400>17
atgtctgatcctggtctcctacagg25
<210>18
<211>24
<212>dna
<213>artificial
<223>b5100/5102r
<400>18
agcaacatcagttaagagtttaag24
<210>19
<211>25
<212>dna
<213>artificial
<223>b5601f
<400>19
atgtccgatcctgcaccccttcaag25
<210>20
<211>20
<212>dna
<213>artificial
<223>b5601r
<400>20
tcaataagtttcagatagca20
<210>21
<211>25
<212>dna
<213>artificial
<223>m30640f
<400>21
atgtttgatccgagtcctctccaag25
<210>22
<211>25
<212>dna
<213>artificial
<223>m30640r
<400>22
tcaattagcttgaactaagcgttgt25
<210>23
<211>36
<212>dna
<213>artificial
<223>毕赤酵母系统b5102f
<400>23
ccgctcgagaaaagatctgatcctggtctcctacag36
<210>24
<211>37
<212>dna
<213>artificial
<223>毕赤酵母系统b5102r
<400>24
aaatatgcggccgctcaagcaacatcagttaagagtt37
<210>25
<211>33
<212>dna
<213>artificial
<223>毕赤酵母系统b5601f
<400>25
ccgctcgagaaaagatccgatcctgcacccctt33
<210>26
<211>39
<212>dna
<213>artificial
<223>毕赤酵母系统b5601r
<400>26
aaatatgcggccgctcaataagtttcagatagcaatttc39
<210>27
<211>35
<212>dna
<213>artificial
<223>毕赤酵母系统m30640f
<400>27
ccgctcgagaaaagatttgatccgagtcctctcca35
<210>28
<211>38
<212>dna
<213>artificial
<223>毕赤酵母系统m30640r
<400>28
aaatatgcggccgctcaattagcttgaactaagcgttg38
<210>29
<211>33
<212>dna
<213>artificial
<223>植物表达系统5102-f
<400>29
gctctagaatgtctgatcctggtctcctacagg33
<210>30
<211>34
<212>dna
<213>artificial
<223>植物表达系统5102-r
<400>30
cggggtacctcaagcaacatcagttaagagttta34
<210>31
<211>34
<212>dna
<213>artificial
<223>植物表达系统5601-r
<400>31
gctctagaatgtccgatcctgcaccccttcaaga34
<210>32
<211>34
<212>dna
<213>artificial
<223>植物表达系统5601-r
<400>32
cggggtacctcaataagtttcagatagcaatttc34
<210>33
<211>33
<212>dna
<213>artificial
<223>植物表达系统30640-f
<400>33
gctctagaatgtttgatccgagtcctctccaag33
<210>34
<211>34
<212>dna
<213>artificial
<223>植物表达系统30640-r
<400>34
cggggtacctcaattagcttgaactaagcgttgt34
- 还没有人留言评论。精彩留言会获得点赞!