靶向表皮生长因子受体变体III的嵌合抗原受体的制作方法
本发明涉及嵌合抗原受体(CAR)。CAR能够利用配体结合结构域特性的重新指导针对所选择靶标的免疫细胞特异性和反应性。特别地,本发明涉及特异性地结合表皮生长因子受体变体III的CAR(EGFRvIII特异性CAR)。本发明还涉及编码EGFRvIII特异性CAR的多核苷酸和在其表面上表达EGFRvIII特异性CAR的分离的细胞。本发明还涉及用于改造在其表面上表达EGFRvIII特异性CAR的免疫细胞的方法。本发明特别地用于治疗实体瘤例如多形性胶质母细胞瘤(GBM)、非小细胞肺癌、头颈癌、乳腺癌、卵巢癌和前列腺癌。本发明还涉及包含EGFRvIII特异性CAR的免疫细胞(EGFRvIII特异性CAR-T细胞)、包含EGFRvIII特异性CAR-T细胞的组合物和使用EGFRvIII特异性CAR-T细胞治疗EGFRvIII-介导的病理的方法。
背景
EGFR变体III(EGFRvIII)即EGFR的肿瘤特异性突变体是通常与野生型EGFR基因扩增相关的基因组重排产物。EGFRvIII由外显子2-7的框内缺失形成,导致267个氨基酸的缺失与在连接处的甘氨酸取代。截短的受体失去其结合配体的能力,但获得组成型激酶活性。有意义的是,EGFRvIII始终与相同肿瘤细胞中的全长野生型EGFR共表达。此外,表达EGFRvIII的细胞表现出增加的增殖、侵袭、血管生成和对凋亡的抗性。
EGFRvIII最常见于多形性胶质母细胞瘤(GBM)中。据估计25-35%的GBM携带这种截短的受体。此外,它的表达通常反映出更具有攻击性的表型和差的预后。除GBM外,EGFRvIII的表达还在其它实体瘤中有报道,例如非小细胞肺癌、头颈癌、乳腺癌、卵巢癌和前列腺癌。相反,EGFRvIII未在健康组织中表达。正常组织中没有表达使得EGFRvIII成为研发肿瘤特异性靶向疗法的理想靶标。
遗传修饰T细胞以识别恶性肿瘤相关抗原的过继转移已经显示作为治疗癌症的新方法的希望(参见,例如Brenner等人,Current Opinion in Immunology,22(2):251-257(2010);Rosenberg等人,Nature Reviews Cancer,8(4):299-308(2008))。T细胞能够被遗传修饰以表达嵌合抗原受体(CAR),其为由抗原识别部分和T细胞活化结构域组成的融合蛋白(参见,例如Eshhar等人,Proc.Natl.Acad.Sci.USA、90(2):720-724(1993)和Sadelain等人,Curr.Opin.Immunol,21(2):215-223(2009))。因此,使用抗-EGFRvIII拮抗剂包括EGFRvIII特异性CAR和EGFRvIII特异性CAR-T细胞治疗实体瘤例如多形性胶质母细胞瘤可以形成富有希望的治疗剂。
概述
提供了结合EGFRvIII的嵌合抗原受体(CAR)。经证实,在T细胞中表达的某些EGFRvIII特异性CAR在接触EGFRvIII后有效激活T细胞。有利地,本文提供的EGFRvIII特异性CAR结合人EGFRvIII。另外有利地,本文提供的EGFRvIII特异性CAR-T细胞在接触表达EGFRvIII的细胞后显示出脱粒活性、增加的干扰素γ产生和/或细胞毒性活性。
在一个方面,本发明提供包含胞外配体结合结构域、第一跨膜结构域和胞内信号传导结构域的EGFRvIII特异性CAR,其中胞外配体结合结构域包含:(a)重链可变(VH)区,其包含:(i)VH互补决定区1(CDR1),其包含SEQ ID NO:62、63、64、74、75、76、80、81、82、88、89、90、109、110、111、115、116、117、121、122、123、137、138或139中所示的序列;(ii)VH CDR2,其包含SEQ ID NO:70、71、77、78、83、84、86、87、91、92、112、113、118、119、124、125、127、128、140或141中所示的序列;和iii)VH CDR3,其包含SEQ ID NO:73、79、85、114、120、126、129或142中所示的序列;和/或(b)轻链可变(VL)区,其包含(i)VL CDR1,其包含SEQ ID NO:149、156、159、162、165、182、185、187或195中所示的序列;(ii)VL CDR2,其包含SEQ ID NO:152、157、160、163、183、186、188或196中所示的序列;和(iii)VL CDR3,其包含SEQ ID NO:153、158、161、164、184、189或197中所示的序列。
在另一个方面,本发明提供包含胞外配体结合结构域、第一跨膜结构域和胞内信号传导结构域的EGFRvIII特异性CAR,其中胞外配体结合结构域包含单链Fv片段(scFv),所述scFv包含重链可变(VH)区,其包含3个来自VH区的CDR,所述VH区包含SEQ ID NO:5、9、11、13、15、37、39、41、43或48中所示的序列;和/或轻链可变(VL)区,其包含3个来自VL区的CDR,所述VL区包含SEQ ID NO:6、10、12、14、16、38、40、42或49中所示的序列。在一些实施方案中,VH区可以包含SEQ ID NO:5、9、11、13、15、37、39、41、43或48中所示的序列或其具有非CDR内的残基中的一个或几个保守氨基酸取代的变体,和/或VL区可以包含SEQ ID NO:6、10、12、14、16、38、40、42或49中所示的氨基酸序列或其具有非CDR内的残基中的一个或几个保守氨基酸取代的变体。例如,在一些实施方案中,scFv的VH或VL区可以包含上述氨基酸序列或其变体,该变体具有非CDR内的残基中的不超过10、9、8、7、6、5、4、3、2或1个保守取代。
在一些实施方案中,本发明提供包含胞外配体结合结构域、第一跨膜结构域和胞内信号传导结构域的EGFRvIII特异性CAR,其中胞外配体结合结构域包含单链Fv片段(scFv),所述scFv包含:重链可变(VH)区,其包含SEQ ID NO:11、15、30、37或41中所示的序列;和/或轻链可变(VL)区,其包含SEQ ID NO:12、16、31、38或42中所示的序列。在一些实施方案中,VH包含SEQ ID NO:11中所示的序列,且VL包含SEQ ID NO:12中所示的序列。在一些实施方案中,VH包含SEQ ID NO:15中所示的序列,且VL包含SEQ ID NO:16中所示的序列。在一些实施方案中,VH包含SEQ ID NO:30中所示的序列,且VL包含SEQ ID NO:31中所示的序列。在一些实施方案中,VH包含SEQ ID NO:37中所示的序列,且VL包含SEQ ID NO:38中所示的序列。在一些实施方案中,VH包含SEQ ID NO:41中所示的序列,且VL包含SEQ ID NO:42中所示的序列。
在一些实施方案中,胞内信号传导结构域包含CD3ζ信号传导结构域。在一些实施方案中,胞内信号传导结构域包含4-1BB信号传导结构域。在一些实施方案中,CAR还可以包含第二胞内信号传导结构域。在一些实施方案中,第二胞内信号传导结构域可以包含4-1BB信号传导结构域。在一些实施方案中,第一胞内信号传导结构域包含CD3ζ信号传导结构域,且第二胞内信号传导结构域包含4-1BB信号传导结构域。
在一些实施方案中,CAR可以包含胞外配体结合结构域与第一跨膜结构域之间的柄结构域。在一些实施方案中,所述柄结构域可以选自:人CD8α铰链、人CD28铰链、IgG1铰链和FcγRIIIα铰链。
在一些实施方案中,第一跨膜结构域可以包含CD8α链跨膜结构域。
在一些实施方案中,CAR可以包含对EGFRvIII无特异性的另一个胞外配体结合结构域。
在CAR的一些实施方案中,胞外配体结合结构域、第一跨膜结构域和胞内信号传导结构域位于单一多肽上。
在一些实施方案中,CAR可以包含第二跨膜结构域,其中第一跨膜结构域和胞外配体结合结构域位于第一多肽上,且其中第二跨膜结构域和胞内信号传导结构域位于第二多肽上,其中第一跨膜结构域包含来自高亲和力IgE受体(FcεRI)的α链的跨膜结构域,且第二跨膜结构域包含来自FcεRI的γ或β链的跨膜结构域。在一些实施方案中,CAR可以包含第三多肽,其包含与来自共刺激分子的胞内信号传导结构域融合的第三跨膜结构域,其中第三跨膜结构域包含来自FcεRI的γ或β链的跨膜结构域。
在另一个方面,本发明提供分离的多核苷酸,其包含编码如本文所述的EGFRvIII特异性CAR的核酸序列。
在另一个方面,本发明提供表达载体,其包含编码如本文所述的EGFRvIII特异性CAR抗体的核酸序列。
在另一个方面,本发明提供改造的免疫细胞,其在其细胞表面膜上表达如本文所述的EGFRvIII特异性CAR。在一些实施方案中,改造的免疫细胞可以包含对EGFRvIII无特异性的另一种CAR。
在一些实施方案中,改造的免疫细胞可以包含编码自杀多肽的多核苷酸。在一些实施方案中,所述自杀多肽是RQR8。在一些实施方案中,编码自杀多肽的多核苷酸位于与包含编码EGFRvIII特异性CAR的核酸序列的多核苷酸不同的核酸分子中。在一些实施方案中,编码自杀多肽的多核苷酸是与包含编码EGFRvIII特异性CAR的核酸序列的多核苷酸相同的核酸分子的组成部分。
在一些实施方案中,含有EGFRvIII-特异性CAR的改造的免疫细胞可以包含在与EGFRvIII-特异性CAR的多肽链分开的多肽中的自杀多肽。
在一些实施方案中,如本文所述的EGFRvIII特异性CAR还包含在与CAR相同的多肽链中的自杀多肽。例如,自杀多肽可以位于CAR的scFv与铰链序列之间。在一些实施方案中,CAR中的自杀多肽可以具有如本文提供的R2形式。在一些实施方案中,自杀多肽包含由利妥昔单抗识别的表位。
本文还提供了编码EGFRvIII特异性CAR的多核苷酸,所述多核苷酸还编码CAR中的自杀多肽。
在一些实施方案中,免疫细胞可以来源于炎症T-淋巴细胞、细胞毒性T-淋巴细胞、调节T-淋巴细胞、记忆T-淋巴细胞、辅助T-淋巴细胞、自然杀伤T-淋巴细胞或自然杀伤细胞。
在一些实施方案中,改造的免疫细胞可以包含一种或多种内源性基因中的破坏,其中内源性基因编码TCRα、TCRβ、CD52、糖皮质激素受体(GR)、脱氧胞苷激酶(dCK)或免疫检查点蛋白,例如程序性死亡-1(PD-1)。
在一些实施方案中,免疫细胞得自健康供体。在一些实施方案中,免疫细胞得自患者。
在另一个方面,本发明提供用作药物的改造的免疫细胞,其在其细胞表面膜上表达如本文所述的EGFRvIII特异性CAR。在一些实施方案中,所述药剂用于治疗EGFRvIII相关癌症(例如具有EGFRvIII表达的任意癌症),其选自多形性胶质母细胞瘤、间变性星形细胞瘤、巨细胞胶质母细胞瘤、胶质肉瘤、间变性少突胶质瘤、间变性室管膜瘤、间变性少突星形细胞瘤、脉络丛癌、间变性神经节胶质瘤、松果体母细胞瘤、松果体细胞瘤、脑膜瘤、髓上皮瘤、室管膜母细胞瘤、髓母细胞瘤、幕上原始神经外胚层瘤、不典型畸胎样/横纹肌样瘤、混合性胶质瘤、头颈癌、非小细胞肺癌、乳腺癌、卵巢癌、前列腺癌、髓母细胞瘤、结直肠癌、肛门癌、宫颈癌、肾癌、皮肤癌、胰腺癌、肝癌、膀胱癌、胃癌、甲状腺癌、间皮瘤、子宫癌、淋巴瘤和白血病。
在另一个方面,本发明提供改造免疫细胞的方法,其包括:提供免疫细胞;和在细胞表面上表达如本文所述的至少一种EGFRvIII特异性CAR。
在一些实施方案中,该方法包括:提供免疫细胞;将向细胞中导入编码所述EGFRvIII特异性CAR的至少一种多核苷酸;和将所述多核苷酸表达入所述细胞。
在一些实施方案中,该方法包括提供免疫细胞;向该细胞中导入编码所述EGFRvIII特异性CAR的至少一种多核苷酸;和导入对EGFRvIII无特异性的至少一种其它CAR。
在另一个方面,本发明提供治疗患有与恶性细胞相关的病症的方法,该方法包括:提供表达在表面上如本文所述的EGFRvIII特异性CAR的免疫细胞;和将所述免疫细胞施用于所述患者。
在另一个方面,本发明提供包含如本文所述的改造的免疫细胞的药物组合物。
在另一个方面,本发明提供治疗与表达受试者中的EGFRvIII的恶性细胞相关的病症的方法,其包括对有此需要的受试者施用有效量的请求保护的药物组合物,其包含如本文所述的改造的免疫细胞。在一些实施方案中,所述病症是癌症。在一些实施方案中,所述癌症是EGFRvIII相关癌症,其选自多形性胶质母细胞瘤、间变性星形细胞瘤、巨细胞胶质母细胞瘤、胶质肉瘤、间变性少突胶质瘤、间变性室管膜瘤、间变性少突星形细胞瘤、脉络丛癌、间变性神经节胶质瘤、松果体母细胞瘤、松果体细胞瘤、脑膜瘤、髓上皮瘤、室管膜母细胞瘤、髓母细胞瘤、幕上原始神经外胚层瘤、不典型畸胎样/横纹肌样瘤、混合性胶质瘤、头颈癌、非小细胞肺癌、乳腺癌、卵巢癌、前列腺癌、髓母细胞瘤、结直肠癌、肛门癌、肾癌、宫颈癌、肝癌、胰腺癌、胃癌、甲状腺癌、间皮瘤、子宫癌和膀胱癌。
在另一个方面,本发明提供抑制具有表达EGFRvIII的恶性细胞的受试者中肿瘤生长或进展的方法,其包括对有此需要的受试者施用有效量的包含如本文所述的改造的免疫细胞的药物组合物。
在另一个方面,本发明提供抑制受试者中表达EGFRvIII的恶性细胞的转移的方法,其包括对有此需要的受试者施用有效量的包含如本文所述的改造的免疫细胞的药物组合物。
在另一个方面,本发明提供诱导具有表达EGFRvIII的恶性细胞的受试者中肿瘤消退的方法,其包括对有此需要的受试者施用有效量的包含如本文所述的改造的免疫细胞的药物组合物。
附图简述
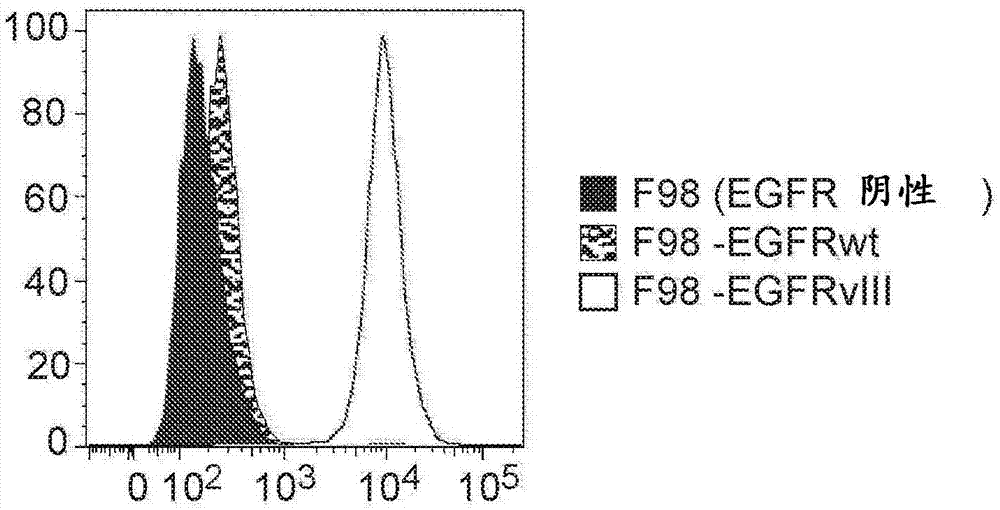
图1A、图1B和图1C显示3种EGFRvIII抗体mAb 42G9(图1A)、32A10(图1B)和32G8(图1C)与3种F98细胞系F98(EGFR阴性)、F98-EGFRwt和F98-EGFRvIII的FACS结合直方图的实例。X-轴是荧光强度;Y-轴是最大值/校准化至模式的百分比。
图2A显示概括含有不同EGFRvIII特异性克隆的CAR的EGFRvIII特异性CAR表达的柱状图。
图2B显示概括结合重组EGFR-mFc和EGFRvIII-mFc的EGFRvIII特异性CAR T细胞百分比的柱状图。
图2C显示概括CAR T细胞中含有10种不同EGFRvIII特异性克隆的CAR的表达的柱状图。
图3显示概括表达不同EGFRvIII特异性克隆的EGFRvIII特异性CAR T细胞单独或与不同细胞系共培养时的脱粒活性的柱状图:不表达任意EGFR蛋白的细胞(NCI-H522和U87-KO),表达高水平野生型EGFR的细胞(U87-KO-EGFRwt)或表达低(NCI-H522-EGFRvIII)和高(U87-KO-EGFRvIII)水平EGFRvIII的细胞。
图4显示概括表达不同EGFRvIII特异性克隆的EGFRvIII特异性CAR T细胞单独或与不同细胞系共培养时的IFNγ分泌的柱状图:不表达任意EGFR蛋白的细胞(NCI-H522和U87-KO),表达高水平野生型EGFR的细胞(U87-KO-EGFRwt)或表达低(NCI-H522-EGFRvIII)和高(U87-KO-EGFRvIII)水平EGFRvIII的细胞。
图5显示比较表达不同EGFRvIII特异性克隆的EGFRvIII特异性CAR T细胞对表达野生型EGFR的细胞系(U87-KO-EGFRwt)vs表达高(U87-KO-EGFRvIII)和低EGFRvIII(NCI-H522-EGFRvIII)的细胞系的细胞毒性的柱状图。
图6显示概括皮下GBM异种移植物模型中表达不同EGFRvIII特异性克隆的EGFRvIII特异性CAR T细胞针对GBM细胞的抗肿瘤活性的示意图。
图7显示概括T细胞转导后第4天、第9/10天和第14/15天时在CAR中表达4种不同EGFRvIII特异性克隆并且在CAR中携带CAR内自杀序列R2的CAR T细胞的CAR表达的柱状图。
图8显示概括T细胞转导后第4天和第14/15天时在CAR中表达4种不同EGFRvIII特异性克隆并且携带CAR内自杀序列R2的CAR T细胞的CAR表达的柱状图。
图9显示概括表达4种不同EGFRvIII特异性克隆并且携带CAR内自杀序列R2的CAR T细胞对表达高水平(U87-KO-EGFRvIII)和低水平EGFRvIII(NCI-H522-EGFRvIII)的细胞系的细胞毒性的柱状图。
详述
本文公开的本发明提供了嵌合抗原受体(CAR)和包含特异性结合EGFRvIII(例如人EGFRvIII)的CAR的免疫细胞(例如CAR-T细胞)。本发明还提供了编码这些CAR的多核苷酸,包含这些CAR-T细胞的组合物和制备和使用这些CAR和CAR-T细胞的方法。本发明还提供了用于治疗受试者中与EGFRvIII-介导的病理相关的病症例如癌症的方法。
一般技术
除非另有指示,否则本发明的实践采用本领域的范围内的分子生物学(包括重组技术)、微生物学、细胞生物学、生物化学、免疫学、病毒学、单克隆抗体生成和改造的常规技术。这类技术在文献中有完整地解释,例如:Molecular Cloning:A Laboratory Manual,第2版(Sambrook等人,1989)Cold Spring Harbor Press;Oligonucleotide Synthesis(M.J.Gait,ed.,1984);Methods in Molecular Biology,Humana Press;Cell Biology:A Laboratory Notebook(J.E.Cellis,ed.,1998)Academic Press;Animal Cell Culture(R.I.Freshney,ed.,1987);Introduction to Cell和Tissue Culture(J.P.Mather和P.E.Roberts,1998)Plenum Press;Cell和Tissue Culture:Laboratory Procedures(A.Doyle,J.B.Griffiths和D.G.Newell,eds.,1993-1998)J.Wiley和Sons;Methods in Enzymology(Academic Press,Inc.);Handbook of Experimental Immunology(D.M.Weir和C.C.Blackwell,eds.);Gene Transfer Vectors for Mammalian Cells(J.M.Miller和M.P.Calos,eds.,1987);Current Protocols in Molecular Biology(F.M.Ausubel等人,eds.,1987);PCR:The Polymerase Chain Reaction,(Mullis等人,eds.,1994);Current Protocols in Immunology(J.E.Coligan等人,eds.,1991);Short Protocols in Molecular Biology(Wiley和Sons,1999);Immunobiology(C.A.Janeway和P.Travers,1997);Antibodies(P.Finch,1997);Antibodies:a practical approach(D.Catty.,ed.,IRL Press,1988-1989);Monoclonal antibodies:a practical approach(P.Shepherd和C.Dean,eds.,Oxford University Press,2000);Using antibodies:a laboratory manual(E.Harlow和D.Lane(Cold Spring Harbor Laboratory Press,1999);The antibodies(M.Zanetti和J.D.Capra,eds.,Harwood Academic Publishers,1995)。
定义
如本文所用,术语“胞外配体结合结构域”是指能够结合配体的寡肽或多肽。优选地,该结构域能够与细胞表面分子发生相互作用。例如,可以选择识别配体的胞外配体结合结构域,所述配体用作与特定疾病状态相关的靶细胞上细胞表面标记。
术语“柄结构域”或“铰链结构域”在本文中可以互换使用,以指起连接跨膜结构域与胞外配体结合结构域作用的寡肽或多肽。特别地,柄结构域用于为胞外配体结合结构域提供更多的柔性和易接近性。
术语“胞内信号传导结构域”是指转导效应子功能信号和指导细胞执行特殊功能的蛋白质部分。
如本文所用,“共刺激分子”是指特异性地结合共刺激配体的T细胞上的同源结合配偶体,由此介导细胞的共刺激应答,例如,但不限于增殖。共刺激分子包括但不限于I类MHC分子、BTLA和Toll配体受体。共刺激分子的实例包括CD27、CD28、CD8、4-1BB(CD137)、OX40、CD30、CD40、PD-1、ICOS、淋巴细胞功能相关抗原-1(LFA-1)、CD2、CD7、LIGHT、NKG2C、B7-H3和特异性地结合CD83的配体等。
“共刺激配体”是指特异性地结合T细胞上同源共刺激信号分子的抗原呈递细胞上的分子,由此提供除通过例如结合TCR/CD3与载有肽的MHC分子的复合物所提供的第一信号之外的信号,其介导T细胞应答,包括但不限于增殖活化、分化、细胞因子产生等。共刺激配体可以包括但不限于CD7、B7-1(CD80)、B7-2(CD86)、PD-L1、PD-L2、4-1BBL、OX40L、诱导型共刺激配体(ICOS-L)、胞间粘附分子(ICAM、CD30L、CD40、CD70、CD83、HLA-G、MICA、M1CB、HVEM、淋巴毒素β受体、3/TR6、ILT3、ILT4、结合Toll配体受体的激动剂或抗体和特异性地结合B7-H3的配体。共刺激配体还特别地涵盖特异性地结合呈递在T细胞上共刺激分子的抗体,例如,但不限于CD27、CD28、4-1BB、OX40、CD30、CD40、PD-1、ICOS、淋巴细胞功能相关抗原-1(LFA-1)、CD2、CD7、LIGHT、NKG2C、B7-H3、特异性地结合CD83的配体。
“抗体”是能够通过位于免疫球蛋白分子的可变区上的至少一种抗原识别位点特异性地结合靶标例如碳水化合物、多核苷酸、脂质等的免疫球蛋白分子。如本文所用,该术语不仅涵盖完整多克隆或单克隆抗体,而且涵盖其抗原结合片段(例如Fab、Fab’、F(ab’)2、Fv)、单链(scFv)和结构域抗体(包括,例如鲨鱼和骆驼抗体)和包含抗体的融合蛋白和任意其它修饰的包含抗原识别位点的免疫球蛋白分子结构。抗体包括任意类别的抗体,例如IgG、IgA或IgM(或其亚类),且抗体不需为任意特定类别。根据其重链恒定区的抗体氨基酸序列的不同,可以将免疫球蛋白指定为不同类别。存在5种主要类别的免疫球蛋白:IgA、IgD、IgE、IgG和IgM,并且它们中的几种可以被进一步分成亚类(同种型),例如IgG1、IgG2、IgG3、IgG4、IgA1和IgA2。对应于免疫球蛋白不同类别的重链恒定区分别称作α、δ、ε、γ和μ。不同类别免疫球蛋白的亚单位结构和三维构型是众所周知的。
如本文所用,术语抗体的“抗原结合片段”或“抗原结合部分”是指保留特异性结合指定抗原(例如EGFRvIII)的能力的完整抗体的一个或多个片段。抗体的抗原结合功能可以由完整抗体的片段执行。术语抗体的“抗原结合片段”内涵盖的结合片段的实例包括Fab;Fab’;F(ab’)2;由VH和CH1结构域组成的Fd片段;由抗体单臂的VL和VH结构域组成的Fv片段;单结构域抗体(dAb)片段(Ward等人,Nature 341:544-546,1989)和分离的互补决定区(CDR)。
“优先结合”或“特异性结合”(本文中互换使用)靶标(例如EGFRvIII蛋白)的抗体、抗体缀合物或多肽是本领域充分理解的术语,并且测定这类特异性或优先结合的方法也是本领域众所周知的。如果分子以比与可选细胞或物质更长的持续时间和/或更大的亲和力与特定细胞或物质更频繁地、更快速地反应或缔合,则认为该分子表现出“特异性结合”或“优先结合”。如果抗体以比结合其它物质更大的亲和力、亲合力、更容易地和/或更长的持续时间与靶标结合,则该抗体“特异性结合”或“优先结合”靶标。例如,特异性结合或优先结合EGFRvIII表位的抗体是以比它结合其它EGFRvIII表位或非-EGFRvIII表位更大的亲和力、亲合力、更容易地和/或更长的持续时间结合该表位的抗体。还应当理解,通过阅读该定义,例如,特异性结合或优先结合第一靶标的抗体(或部分或表位)可特异性结合或优先结合或可不特异性结合或优先结合第二靶标。照此,“特异性结合”或“优先结合”不一定必然要求(尽管可以包括)专属结合。通常,但不一定必然,涉及的结合是指优先结合。
抗体“可变区”是指单独或组合方式的抗体轻链可变区或抗体重链可变区。正如本领域知晓的,重链和轻链可变区各自由通过3个互补决定区(CDR)也称作高变区连接的4个构架区(FR)组成。每条链中的CDR通过FR极为贴近地保持在一起,并且与来自另一链的CDR一起促成抗体的抗原结合位点形成。存在至少两种用于确定CDR的技术:(1)基于交叉物种序列变异性的方法(即Kabat等人Sequences of Proteins of Immunological Interest,(第5版,1991,National Institutes of Health,Bethesda MD));和(2)基于抗原-抗体复合物的结晶学研究的方法(Al-lazikani等人,1997、J.Molec.Biol.273:927-948)。如本文所用,CDR可以指通过任一方法或通过两种方法的组合定义的CDR。
可变结构域的“CDR”是根据Kabat、Chothia、Kabat和Chothia的累积、AbM、接触和/或构象定义的定义或本领域众所周知的任意CDR测定方法鉴定的可变区内的氨基酸残基。可以将抗体CDR鉴定为最初由Kabat等人定义的高变区。参见,例如Kabat等人,1992、Sequences of Proteins of Immunological Interest,第5版,Public Health Service,NIH,Washington D.C。还可以将CDR的位置鉴定为最初由Chothia等描述的结构环结构。参见,例如Chothia等人,Nature 342:877-883,1989。CDR鉴定的其它方法包括“AbM定义”,其是Kabat与Chothia之间的折中,并且使用Oxford Molecular的AbM抗体模型化软件(现在的)衍生或基于MacCallum等人,J.Mol.Biol.,262:732-745,1996中举出的基于观察到的抗原接触的CDR的“接触定义”。在另一个方法中,本文称作CDR的“构象定义”是CDR的位置可以被鉴定为对抗原结合提供热焓贡献的残基。参见,例如Makabe等人,Journal of Biological Chemistry,283:1156-1166,2008。另外的CDR边界定义并不严格遵循上述方法之一,尽管如此,但与Kabat CDR的至少一部分重叠,不过,它们可以根据预测或实验发现缩短或延长,所述预测或实验发现为特定残基或残基组乃至完整CDR并不显著影响抗原结合。如本文所用,CDR可以指本领域已知的任意方法包括方法的组合定义的CDR。本文使用的方法可以应用根据这些方法的任意方法定义的CDR。对于含有一种以上CDR的任意给定的实施方案,可以根据Kabat、Chothia、扩展的、AbM、接触和/或构象定义的任意来定义CDR。
如本文所用,“单克隆抗体”是指得自基本上同源抗体群的抗体,即包含该群的个体抗体是相同的,除了可能天然存在的可以以最少量存在的突变。单克隆抗体是高度特异性针对单一抗原位点。此外,与典型地包括定向于不同决定簇(表位)的不同抗体的多克隆抗体制品相反,每个单克隆抗体针对抗原上的单一决定簇。修饰语“单克隆”表示得自抗体基本上同源群体的抗体特性,并且不是被解释为需要通过任何特定方法产生抗体。例如,根据本发明使用的单克隆抗体可以通过首先由Kohler和Milstein,Nature 256:495,1975描述的杂交瘤方法制备或可以通过重组DNA方法例如美国专利号4,816,567中所述的方法制备。单克隆抗体还可以分离自使用例如McCafferty等人,Nature 348:552-554,1990中所述的技术生成的噬菌体文库。
如本文所用,“人源化”抗体是指非-人(例如鼠)抗体的形式,其为嵌合免疫球蛋白、免疫球蛋白链或其片段(例如Fv、Fab、Fab’、F(ab')2或抗体的其它抗原结合亚序列),它们含有来源于非-人免疫球蛋白的最小序列。优选地,人源化抗体是人免疫球蛋白(接受者(recipient)抗体),其中来自接受者的互补决定区(CDR)的残基被来自具有期望的特异性、亲和力和能力的非-人物种(供体抗体)例如小鼠、大鼠或家兔的CDR的残基替代。在一些情况中,人免疫球蛋白的Fv构架区(FR)残基被相应的非-人残基替代。此外,人源化抗体可以包含既不是接受者抗体中,也不是在导入的CDR或构架序列中发现的残基,但包括它们是为了进一步精制和优化抗体性能。通常,人源化抗体将包含基本上至少一种(和典型地两种)可变结构域的基本上全部,其中全部或基本上全部CDR区相当于非-人免疫球蛋白的那些,且全部或基本上全部FR区是人免疫球蛋白共有序列的那些。人源化抗体还最佳地将包含免疫球蛋白恒定区或结构域(Fc)、典型地人免疫球蛋白的至少一部分。优选具有如WO 99/58572中所述修饰的Fc区的抗体。人源化抗体的其它形式具有一个或多个CDR(CDR L1、CDR L2、CDR L3、CDR H1、CDR H2或CDR H3),它们针对原始抗体发生改变,也称作“来源于”来自原始抗体的一个或多个CDR的一个或多个CDR。
如本文所用,“人抗体”是指具有相当于由人产生和/或已经使用本领域技术人员已知或本文公开的制备人抗体的任意技术制备的抗体的氨基酸序列的抗体。人抗体的该定义包括包含至少一种人重链多肽或至少一种人轻链多肽的抗体。一种这类实例是包含鼠轻链和人重链多肽的抗体。可以使用本领域已知的各种技术生产人抗体。在一个实施方案中,人抗体选自噬菌体文库,其中该噬菌体文库表达人抗体(Vaughan等人,Nature Biotechnology,14:309-314、1996;Sheets等人,Proc.Natl.Acad.Sci.(USA)95:6157-6162,1998;Hoogenboom和Winter,J.Mol.Biol.,227:381,1991;Marks等人,J.Mol.Biol.,222:581,1991)。还可以通过免疫接种已经通过转基因方式导入人免疫球蛋白基因座替代内源性基因座的动物例如小鼠制备人抗体,其中内源性免疫球蛋白基因部分或完全失活。该方法描述在美国专利号5,545,807;5,545,806;5,569,825;5,625,126;5,633,425;和5,661,016中。或者,可以通过使产生针对靶抗原的抗体的人B淋巴细胞无限增殖制备人抗体(这类B淋巴细胞可以从个体或cDNA的单细胞克隆中回收或可以在体外免疫接种)。参见,例如Cole等人Monoclonal Antibody and Cancer Therapy,Alan R.Liss,p.77,1985;Boerner等人,J.Immunol.,147(1):86-95,1991;和美国专利号5,750,373。
术语“嵌合抗体”旨在指其中可变区序列来源于一个物种而恒定区序列来源于另一个物种的抗体,例如其中可变区序列来源于小鼠抗体而恒定区序列来源于人抗体的抗体。
术语“多肽”、“寡肽”、“肽”和“蛋白质”在本文中可以互换使用来指任意长度的氨基酸的链。例如,链可以相对短(例如10-100个氨基酸)或更长。链可以是直链或支链,它可以包含修饰的氨基酸,和/或可以被非氨基酸中断。该术语还涵盖已被天然修饰或通过中断修饰的氨基酸链;例如,二硫键形成、糖基化、脂化、乙酰化、磷酸化或任意其它操作或修饰,例如与标记成分缀合。还包括在该定义范围内是例如含有氨基酸的一个或多个类似物(包括例如非天然氨基酸等)以及本领域已知的其它修饰的多肽。应当理解,多肽可以作为单一的链或缔合的链出现。
“单价抗体”包含一个抗原结合位点/分子(例如IgG或Fab)。在一些情况中,单价抗体可以具有一个以上可以结合位点,但结合位点来自不同抗原。
“二价抗体”包含两个抗原结合位点/分子(例如IgG)。在一些情况中,两个结合位点具有相同抗原特异性。然而,二价抗体可以是双特异性的。
“双特异性”或“双重特异性”是具有两个不同抗原结合位点的杂交抗体。双特异性抗体的两个抗原结合位点结合两个不同表位,其可以位于相同或不同蛋白靶标上。
“双功能”是在两个臂上具有相同抗原结合位点(即相同氨基酸序列)、但每个结合位点可以识别两个不同抗原的抗体。
本发明的抗体可以使用本领域众所周知的技术生产,例如重组技术、噬菌体展示技术、合成技术或这类技术的组合或本领域易于知晓的其它技术(参见,例如Jayasena,S.D.,Clin.Chem.,45:1628-50,1999和Fellouse,F.A.等人,J.Mol.Biol.,373(4):924-40、2007)。
正如本领域已知的,在本文中可以互换使用的“多核苷酸”或“核酸”是指任意长度的核苷酸的链且包括DNA和RNA。核苷酸可以为脱氧核糖核苷酸、核糖核苷酸、修饰的核苷酸或碱基和/或其类似物或可以通过DNA或RNA聚合酶并入链的任意底物。多核苷酸可以包含修饰的核苷酸,例如甲基化核苷酸及其类似物。如果存在,则对核苷酸结构的修饰可以在链的组装前或后受到影响。核苷酸的序列可以被非核苷酸成分中断。多核苷酸可以在聚合后被修饰,例如通过用标记成分缀合。其它类型的修饰包括,例如,“帽”,用类似物取代天然存在核苷酸的一个或多个,核苷酸间修饰,例如具有不带电荷的键(例如膦酸甲酯、磷酸三酯、磷酸酰胺化物、氨基甲酸酯等)和具有带电荷的键(例如硫代磷酸、二硫代磷酸等)的那些,含有侧链部分(pendant moieties)的那些,例如蛋白质(例如核酸酶、毒素、抗体、信号肽、聚-L-赖氨酸等),具有嵌入剂的那些(例如吖啶、补骨脂素等)、含有螯合剂的那些(例如金属、放射性金属、硼、氧化金属等),含有烷基化剂的那些,具有修饰的键的那些(例如α端基核酸等),以及多核苷酸的未修饰形式。此外,糖中通常存在的任意羟基可以例如被膦酸酯基、磷酸酯基替代,被标准保护基保护,或被活化以制备至额外核苷酸的额外键或可以缀合至固相支持物。5’和3’末端OH可以被磷酸化或被1-20个碳原子的胺或有机加帽基团取代。其它羟基也可以被衍生成标准保护基。多核苷酸还可以含有本领域通常已知的核糖或脱氧核糖的类似形式,包括例如,2’-O-甲基-、2’-O-烯丙基、2’-氟-或2’-叠氮基-核糖、碳环糖类似物、α-或β-端基糖、表异构体糖例如阿拉伯糖、木糖或来苏糖、吡喃糖、呋喃糖、景天庚醛糖、无环类似物和无碱基核苷类似物例如甲基核苷。一个或多个磷酸二酯键可以被备选连接基替代。这些备选连接基包括但不限于这样的实施方案,其中磷酸酯被P(O)S(“硫代酸酯”)、P(S)S(“二硫代酸酯”)、(O)NR2(“酰胺化物”)、P(O)R、P(O)OR’、CO或CH2(“甲缩醛”)替代,其中R或R’各自独立地为H或任选含有醚(-O-)键的取代或未取代的烷基(1-20C)、芳基、烯基、环烷基、环烯基或芳烷基(araldyl)。并非多核苷酸中的所有键需要相同。上述描述适用于本文涉及的所有多核苷酸,包括RNA和DNA。
正如本领域已知的,抗体的“恒定区”是指单独或组合形式的抗体轻链的恒定区或抗体重链的恒定区。
如本文所用,“基本上纯”是指这样的材料,其为至少50%纯(即不含污染物),更优选至少90%纯,更优选至少95%纯,又更优选至少98%纯,且最优选至少99%纯。
“宿主细胞”包括个体细胞或细胞培养物,其可以为或已经为并入多核苷酸插入物的载体的接受者。宿主细胞包括单宿主细胞的后代,且后代因天然、偶然或有意的突变而不一定与原始亲代细胞完全相同(在形态或基因组DNA方面(complement))。宿主细胞包括被本发明多核苷酸体内转染的细胞。
如本文所用,“免疫细胞”是指功能牵涉固有和/或适应性免疫应答的起始和/或执行的造血起源的细胞。
正如本领域已知的,术语“Fc区”用于定义免疫球蛋白重链的C-末端区。“Fc区”可以是天然序列Fc区或变体Fc区。尽管免疫球蛋白重链的Fc区的边界可以发生变化,但是人IgG重链Fc区通常定义为从Cys226位或从Pro230位上的氨基酸残基到其羧基末端的一段序列。Fc区中残基的编号是如Kabat中的EU索引的编号。Kabat等人,Sequences of Proteins of Immunological Interest,第5版.Public Health Service,National Institutes of Health,Bethesda,Md.,1991。免疫球蛋白的Fc区通常包含两个恒定区CH2和CH3。
如本领域中所用的,“Fc受体”和“FcR”描述结合抗体Fc区的受体。优选的FcR是天然序列人FcR。此外,优选的FcR结合IgG抗体(γ受体)且包括FcγRI、FcγRII和FcγRIII亚类的受体,包括等位基因变体和这些受体的可变剪接形式。FcγRII受体包括FcγRIIA(“活化受体”)和FcγRIIB(“抑制受体”),其具有主要在其胞质结构域中不同的相似氨基酸序列。FcR综述于Ravetch和Kinet,Ann.Rev.Immunol.,9:457-92,1991;Capel等人,Immunomethods,4:25-34、1994;和de Haas等人,J.Lab.Clin.Med.,126:330-41,1995中。“FcR”还包括新生儿受体FcRn,其负责将母源IgG转移至胎儿(Guyer等人,J.Immunol.,117:587,1976;和Kim等人,J.Immunol.,24:249,1994)。
如本文在抗体方面所用,术语“竞争”是指第一抗体或其抗原结合片段(或部分),它们以与第二抗体或其抗原结合部分充分类似的方式结合表位,使得第一抗体与其同源表位结合的产物在第二抗体存在下与没有第二抗体存在下结合第一抗体相比以可检测到的方式减少。其中第二抗体与其表位结合中第一抗体存在下也以可检测到的方式减少的可选方案能够,但不一定是这种情况。即第一抗体能够抑制第二抗体与其表位结合,而无需第二抗体抑制第一抗体与其相应表位结合。然而,如果每种抗体可检测到地抑制另一抗体与同源表位或配体结合,无论是否达到相同、更大或更低的程度,则认为这些抗体彼此“交叉竞争”结合其相应的表位。竞争和交叉竞争抗体均被本发明所涵盖。与通过这类竞争或交叉竞争发生的机制无关(例如位阻、构象改变或结合其共同表位或部分),本领域技术人员可以理解,基于本文提供的教导,这类竞争和/或交叉竞争抗体被涵盖并且能够用于本文公开的方法。
如本文所用,“自体”是指用于治疗患者的细胞、细胞系或细胞群来源于所述患者或来源于人白细胞抗原(HLA)相容性供体。
如本文所用,“同种异体”是指用于治疗患者的细胞或细胞群并非来源于该患者,而是来源于供体。
如本文所用,“治疗”是获得有益或期望的临床效果的方法。出于本发明的目的,有益或期望的临床效果包括但不限于如下的一种或多种:减少肿瘤或癌细胞的增殖(或破坏肿瘤或癌细胞),抑制肿瘤细胞转移,使表达EGFRvIII的肿瘤收缩或减小其尺寸,使EGFRvIII相关疾病(例如癌症)消退,减轻因EGFRvIII相关疾病(例如癌症)导致的症状,提高患有EGFRvIII相关疾病(例如癌症)的那些患者的生活质量,降低治疗EGFRvIII相关疾病(例如癌症)所需其它药物的剂量,延迟EGFRvIII相关疾病(例如癌症)进展,治愈EGFRvIII相关疾病(例如癌症),和/或延长患有EGFRvIII相关疾病(例如癌症)的患者的存活期。
“改善(ameliorating)”是指与不施用EGFRvIII特异性CAR相比一种或多种症状的减轻或好转(improvement)。“改善”还包括缩短或减少症状的持续时间。
如本文所用,药物、化合物或药物组合物的“有效剂量”或“有效量”是足以实现任意一种或多种有益或期望的效果的量。对于预防用途,有益或期望的效果包括消除或降低风险,减轻疾病的严重程度或延迟其发作,包括疾病的生化、组织学和/或行为症状、其并发症和疾病发展期间呈现的中间病理表型。对于治疗用途,有益或期望的效果包括临床效果,例如减少各种EGFRvIII相关疾病或病症(例如多形性胶质母细胞瘤)的一种或多种症状的发生率或改善它们,降低治疗该病所需的其它药物的剂量,增强另一种药物的效果,和/或延迟患者的EGFRvIII相关疾病进展。有效剂量可以一次或多次施用来施用。出于本发明的目的,药物、化合物或药物组合物的有效剂量为足以直接或间接实现预防或治疗的用量。正如临床环境中可以理解的,药物、化合物或药物组合物的有效剂量可以结合另一种药物、化合物或药物组合物实现,也可以不结合它们实现。因此,在施用一种或多种治疗剂的环境中,可以考虑“有效剂量”,并且如果结合一种或多种另外的活性剂,则可以考虑以有效量给予单一药剂,可以或实现期望的效果。
“个体”或“受试者”是哺乳动物,更优选人。哺乳动物还包括但不限于灵长类、马、狗、猫、小鼠和大鼠。
如本文所用,“载体(vector)”是指能够递送且优选在宿主细胞中表达一种或多种目的基因或序列的构建体。载体的实例包括但不限于病毒载体、裸DNA或RNA表达载体、质粒、粘粒或噬菌体载体、与阳离子缩合剂相关的DNA或RNA表达载体、包封在脂质体中的DNA或RNA表达载体、和某些真核细胞例如生产者细胞(producer cell)。
如本文所用,“表达控制序列”是指指导核酸转录的核酸序列。表达控制序列可以是启动子例如组成型或诱导型启动子,或增强子。表达控制序列可操作地连接至待被转录的核酸序列。
如本文所用,“药学上可接受的载体(carrier)”或“药学上可接受的赋形剂”包括当与活性成分合并时,其允许所述成分保留生物活性并且不与受试者免疫系统反应的任意材料。实例包括但不限于任意标准的药用载体,例如磷酸盐缓冲盐水、水、乳剂例如油/水乳剂和各种类型的湿润剂。用于气雾剂或胃肠外施用的优选稀释剂是磷酸盐缓冲盐水(PBS)或生理(0.9%)盐水。包含这类载体的组合物通过众所周知的常规方法配制(参见,例如Remington's Pharmaceutical Sciences,第18版,A.Gennaro,ed.,Mack Publishing Co.,Easton,PA,1990;和Remington,The Science和Practice of Pharmacy第21版.Mack Publishing,2005)。
如本文所用,术语“kon”或“ka”是指抗体与抗原结合的速率常数。具体而言,可以使用例如全长抗体和/或Fab抗体片段和相应抗原测量该速率常数(kon/ka和koff/kd)和平衡解离常数。
如本文所用,术语“koff”或“kd”是指抗体从抗体/抗原复合物中解离的速率常数。
如本文所用,术语“KD”是指抗体-抗原相互作用的平衡解离常数。
可以使用基于表面等离子体共振的生物传感器分别对结合和解离速率常数kon和koff进行测定,以便表征分析物/配体相互作用,其条件为分析物在结合以低容量通过捕获试剂固定在传感器表面上的配体方面是单价的。使用如Karlsson等人,Anal.Biochem 349、136-147,2006中所述的动力学滴定方法进行分析。根据Myszka,J.Mol.Recognit 12,279-284,1999中的推荐内容,选择用于指定测定的传感器芯片、捕获试剂和测定缓冲液以得到传感器表面上配体的稳定捕获,将分析物与表面的非特异性结合减少到最低限度,并且产生对于动力学分析合适的分析物-结合响应。双重参考分析物-结合响应/分析物/配体相互作用,并且与1:1Langmuir“质量转运限制模型”拟合,其中ka、kd和Rmax为Myszka&Morton等人,Biophys.Chem 64,127-137(1997)中所述的全局参数。平衡解离常数KD从动力学速率常数之比推导,KD=koff/kon。优选在25℃或37℃下进行这类测定。
本文数值或参数中涉及的“约”包括(且描述)涉及数值或参数自身的实施方案。例如,涉及“约X”的描述包括“X”的描述。数值范围包括定义范围的数值。一般而言,术语“约”是指变量的指示数值并且是指在指示数值的实验误差内的变量的所有数值(例如对于平均值在95%置信区间内)或指示数值的10%内,不论哪一个更大。如果术语“约”在时间段(年、月、周、天等)的环境中使用,则术语“约”是指该时间段加上或减去下一个下级时间段的1个量(例如约1年是指11-13个月;约6个月是指6个月加上或减去1周;约1周是指6-8天等)或指示数值的10%内,不论哪一个更大。
应当理解,无论本文使用语言“包含”描述实施方案如何,还提供术语“由……组成”和/或“基本上由……组成”中描述的其它类似实施方案。
如果本发明的方面或实施方案以马库什群组或其它可选群组方式描述,则本发明不仅涵盖作为整体列出的完整群组以及各自群组的每个成员和主要群组的所有可能的子群组,而且涵盖不存在于群组成员中的一个或多个的主要群组。本发明还考虑要求保护发明中的任意群组成员的一个或多个的显而易见的排除。
除非另有定义,否则本文所用的所有技术和科学术语具有与本发明所属技术领域普通技术人员通常所通常理解的相同的含义。在出现矛盾的情况中,以本说明书包括定义为准。在本说明书和权利要求书的全文中,词语“包含(comprise)”或变化形式例如“包含(comprises)”或“包含(comprising)”应当理解为是指包含所述整数或整数组,但不排除任何其它整数或整数组。除非上下文中另有要求,否则单数术语应当包括复数,且复数术语应当包括单数。
示例性方法和材料如本文所述,不过,与本文所述相似或等同的方法和材料也可以用于实施或测试本发明。所述材料、方法和实例仅是示例性的,且不旨在起限定作用。
EGFRvIII特异性CAR及其制备方法
本发明提供结合EGFRvIII(例如人EGFRvIII(例如SEQ ID NO:201,登录号:P00533特征标识符VAR_066493或GenBank登录号AJN69267))的CAR。本文提供的EGFRvIII特异性CAR包括单链CAR和多链CAR。CAR利用单克隆抗体的抗原结合特性而具有以非-MHC-限制性方式重新指导T细胞对EGFRvIII的特异性和反应性的能力。非-MHC-限制性抗原识别使得表达CAR的T细胞具有识别独立于抗原加工的抗原的能力,由此绕过肿瘤逃逸的主要机制。
在一些实施方案中,本文提供的CAR包含胞外配体结合结构域(例如单链可变片段(scFv))、跨膜结构域和胞内信号传导结构域。在一些实施方案中,胞外配体结合结构域、跨膜结构域和胞内信号传导结构域在一个多肽中,即在单链中。本文还提供了多链CAR和多肽。在一些实施方案中,多链CAR包含:包含跨膜结构域和至少一种胞外配体结合结构域的第一多肽和包含跨膜结构域和至少一种胞内信号传导结构域的第二多肽,其中这些多肽组装在一起形成多链CAR。
在一些实施方案中,EGFRvIII特异性多链CAR基于IgE的高亲和力受体(FcεRI)。在肥大细胞和嗜碱细胞上表达的FcεRI触发过敏反应。FcεRI是由单一α亚单位、单一β亚单位和二硫键连接的两个γ亚单位组成的四聚体复合物。α亚单位含有IgE-结合结构域。β和γ亚单位含有介导信号转导的ITAM。在一些实施方案中,FcRα链的胞外结构域缺失并且被EGFRvIII特异性胞外配体结合结构域替代。在一些实施方案中,多链EGFRvIII特异性CAR包含特异性地结合EGFRvIII的scFv、CD8α铰链和FcRβ链的ITAM。在一些实施方案中,CAR可以包含也可以不包含FcRγ链。
在一些实施方案中,胞外配体结合结构域包含scFv,所述scFV包含通过柔性接头连接的靶抗原特异性单克隆抗体的轻链可变(VL)区和重链可变(VH)区。单链可变区片段通过使用短连接肽连接轻链和/或重链可变区而制备(Bird等人,Science 242:423-426,1988)。连接肽的实例是具有氨基酸序列(GGGGS)4(SEQ ID NO:202)的GS接头,其桥接一个可变区羧基末端与另一个可变区氨基末端之间的约3.5nm。已经设计并且使用了其它序列的接头(Bird等人,1988,同上)。通常,接头可以是短的柔性多肽并且优选由约20个或更少的氨基酸残基组成。接头可以进而被修饰用于另外的功能,例如连接药物或连接至固相支持物。单链变体可以通过重组或合成方式产生。为了合成生产scFv,可以使用自动化合成仪。为了重组生产scFv,可以将包含编码scFv的多核苷酸的合适的质粒导入合适的宿主细胞,即真核宿主细胞(例如酵母、植物、昆虫或哺乳动物细胞)或原核宿主细胞(例如大肠杆菌)。可以通过常规操作例如连接多核苷酸制备编码目的scFv的多核苷酸。可以使用本领域已知的标准蛋白质纯化技术分离得到的scFv。
在一些实施方案中,胞外配体结合结构域包含:(a)VH区,其包含(i)VH互补决定区1(CDR1),其包含SEQ ID NO:62、63、64、74、75、76、80、81、82、88、89、90、93、94、95、99、100、101、109、110、111、115、116、117、121、122、123、132、133,134、137、138、139、143、144或145中所示的序列;(ii)VH CDR2,其包含SEQ ID NO:65、66、68、69、70、71、77、78、83、84、86、87、91、92、96、97、98、102、103、105、106、112、113、118、119、124、125、127、128、130、131、135、136、140、141、146或147中所示的序列;和iii)VH CDR3,其包含SEQ ID NO:67、72、73、79、85、104、107、108、114、120、126、129、142或148中所示的序列;和/或VL区,其包含(i)VL CDR1,其包含SEQ ID NO:149、154、156、159、162、165、166、168、169、170、171、173、174、176、178、181、182、185、187、190、192、195或198中所示的序列;(ii)VL CDR2,其包含SEQ ID NO:150、152、155、157、160、163、172、175、179、183、186、188、191、193、196或199中所示的序列;和(iii)VL CDR3,其包含SEQ ID NO:151、153、158、161、164、167、177、180、184、189、194、197或200中所示的序列。在一些实施方案中,VH和VL通过柔性接头连接在一起。在一些实施方案中,柔性接头包含SEQ ID NO:202中所示的氨基酸序列。
在一些实施方案中,胞外配体结合结构域包含:(a)VH区,其包含(i)VH互补决定区1(CDR1),且包含SEQ ID NO:62、63、64、74、75、76、80、81、82、88、89、90、109、110、111、115、116、117、121、122、123、137、138或139中所示的序列;(ii)VH CDR2,其包含SEQ ID NO:70、71、77、78、83、84、86、87、91、92、112、113、118、119、124、125、127、128、140或141中所示的序列;和iii)VH CDR3,其包含SEQ ID NO:73、79、85、114、120、126、129或142中所示的序列,和/或(b)VL区,其包含(i)VL CDR1,且包含SEQ ID NO:149、156、159、162、165、182、185、187或195中所示的序列;(ii)VL CDR2,且包含SEQ ID NO:152、157、160、163、183、186、188或196中所示的序列;和(iii)VL CDR3,其包含SEQ ID NO:153、158、161、164、184、189或197中所示的序列。
在另一个方面,提供了CAR,其特异性地结合EGFRvIII,其中CAR包含胞外配体结合结构域,所述胞外配体结合结构域包含:VH区,其包含SEQ ID NO:1、3、5、7、9、11、13、15、17、19、21、23、25、27、30、32、34、35、37、39、41、43、44、46、48或50中所示VH序列的VH CDR1、VH CDR2和VH CDR3;和/或VL区,其包含SEQ ID NO:2、4、6、8、10、12、14、16、18、20、22、24、26、28、29、31、33、36、38、40、42、45、47、49或51中所示VL序列的VL CDR1、VL CDR2和VL CDR3。在一些实施方案中,VH和VL通过柔性接头连接在一起。在一些实施方案中,柔性接头包含SEQ ID NO:202中所示的氨基酸序列。
在一些实施方案中,CAR包含胞外配体结合结构域,所述胞外配体结合结构域包含:VH区,其包含SEQ ID NO:5、9、11、13、15、37、39、41、43或48中所示VH序列的VH CDR1、VH CDR2和VH CDR3;和/或VL区,其包含SEQ ID NO:6、10、12、14、16、38、40、42或49中所示VL序列的VL CDR1、VL CDR2和VL CDR3。在一些实施方案中,VH和VL通过柔性接头连接在一起。在一些实施方案中,柔性接头包含SEQ ID NO:202中所示的氨基酸序列。
在一些实施方案中,本发明的CAR包含胞外配体结合结构域,所述胞外配体结合结构域具有如表1中列出的部分轻链序列的任意一种和/或如表1中列出的部分重链序列的任意一种。在表1中,加下划线的序列为根据Kabat的CDR序列且加粗显示的序列为根据Chothia的CDR序列。表1中的不同mAbs在本文中也可以称作不同抗-EGFRvIII抗体“克隆”。
表1
本文还提供了结合EGFRvIII的CAR的胞外配体结合结构域的CDR部分(包括Chothia、Kabat CDR和CDR接触区)。CDR区的确定尽在本领域技术水平范围内。应当理解,在一些实施方案中,CDR可以是Kabat和Chothia CDR的组合(也称作“组合的CR”或“扩展的CDR”)。在一些实施方案中,CDR是Kabat CDR。在其他实施方案中,CDR是Chothia CDR。换句话说,在具有一种以上CDR的实施方案中,CDR可以是Kabat、Chothia、组合CDR或其组合的任意种。表2提供了本文提供的CDR序列的实例。
表2
本发明涵盖对表1中所示本发明变体的CAR和多肽的修饰,包括具有不显著影响其特性的修饰的功能等效CAR和已增强或降低活性和/亲和力的变体。例如,可以突变氨基酸序列以得到具有期望的与EGFRvIII的结合亲和力的抗体。多肽的修饰是本领域的常规操作且无需在本文中详细描述。修饰多肽的实例包括具有氨基酸残基的保守取代、不显著有害地改变功能活性或成熟(增强)多肽对其配体的亲和力或化学类似物的应用的氨基酸的一个或多个缺失或添加的多肽。
氨基酸序列插入包括长度范围为一个残基至含有一百个或更多残基的多肽的氨基-末端和/或羧基-末端融合,以及单一或多个氨基酸残基的序列内插入。末端插入的实例包括具有N-末端甲硫氨酰基残基的抗体或与表位标签融合的抗体。抗体分子的另外的插入变体包括与增加抗体在血液循环中的半衰期的酶或多肽的抗体的N-末端或C-末端的融合体。
取代变体具有在抗体分子中被除去的至少一个氨基酸残基和在其位置上插入的不同残基。用于取代诱变最为关注的位点包括高变区,但也考虑FR改变。保守取代如表3中“保守取代”标题下所示。如果这类取代导致生物活性改变,则可以导入更显著的改变,命名为表3中的“示例性取代”或如下参照氨基酸类别中进一步描述,并且筛选产物。在一些实施方案中,本文提供的抗体的取代变体与参比亲代抗体相比,在VH或VL区中具有不超过15、14、13、12、11、10、9、8、7、6、5、4、3、2或1个保守取代。在一些实施方案中,取代不位于VH或VL区的CDR内。
表3:氨基酸取代
在一些实施方案中,本发明提供包含胞外配体结合结构域的CAR,所述胞外配体结合结构域结合EGFRvIII并且与本文所述的抗体或本文所述的CAR(例如表5A)竞争性结合EGFRvIII,包括m62G7、h62G7、h62G7-H14/L1-DV、h62G7-L6/EQ、42G9、32A10、20B9、14C11、21E11、49B11、46E10、12H6、19A9、21E7、11B11、12B2、11F10、17G11、29D5、30D8、20E12、26B9、32G8、34E7、20G5、C6和B5。
在一些实施方案中,本发明提供CAR,其包含抗体与基于CDR接触区的EGFRvIII抗体的CDR部分。CDR接触区是赋予抗体抗原特异性的抗体区。通常,CDR接触区包括CDR中和Vernier区的残基位置,其受到约束以维持结合特异性抗原的抗体环结构。参见,例如Makabe等人,J.Biol.Chem.,283:1156-1166、2007。CDR接触区的确定尽在本领域技术水平范围内。
如本文所述的EGFRvIII特异性CAR对EGFRvIII(例如人EGFRvIII(例如(SEQ ID NO:201))的结合亲和力(KD)可以为约0.001nM至约5000nM。在一些实施方案中,结合亲和力为约5000nM、4500nM、4000nM、3500nM、3000nM、2500nM、2000nM、1789nM、1583nM、1540nM、1500nM、1490nM、1064nM、1000nM、933nM、894nM、750nM、705nM、678nM、532nM、500nM、494nM、400nM、349nM、340nM、353nM、300nM、250nM、244nM、231nM、225nM、207nM、200nM、186nM、172nM、136nM、113nM、104nM、101nM、100nM、90nM、83nM、79nM、74nM、54nM、50nM、45nM、42nM、40nM、35nM、32nM、30nM、25nM、24nM、22nM、20nM、19nM、18nM、17nM、16nM、15nM、12nM、10nM、9nM、8nM、7.5nM、7nM、6.5nM、6nM、5.5nM、5nM、4nM、3nM、2nM、1nM、0.5nM、0.3nM、0.1nM、0.01nM或0.001nM中的任一个。在一些实施方案中,结合亲和力小于约5000nM、4000nM、3000nM、2000nM、1000nM、900nM、800nM、250nM、200nM、100nM、50nM、30nM、20nM、10nM、7.5nM、7nM、6.5nM、6nM、5nM、4.5nM、4nM、3.5nM、3nM、2.5nM、2nM、1.5nM、1nM或0.5nM中的任一个。
本发明CAR的胞内信号传导结构域负责在胞外配体结合结构域与靶标结合后的胞内信号传导,导致免疫细胞活化和免疫应答。胞内信号传导结构域具有激活其中表达CAR的免疫细胞的正常效应子功能的至少一种的能力。例如,T细胞的效应子功能可以是溶细胞活性或辅助细胞活性,包括细胞因子分泌。
在一些实施方案中,用于CAR的胞内信号传导结构域可以是例如但不限于T细胞受体和起协同作用以便在抗原受体衔接后启动信号转导的辅助受体的胞质序列,以及这些序列的任意衍生物或变体和具有相同功能能力的合成序列。胞内信号传导结构域包含两种不同类别的胞质信号传导序列:启动抗原依赖性初级活化的那些和以抗原不依赖性方式起作用以提供二次或共刺激信号的那些。初级胞质信号传导序列可以包含信号传导基序,其称作ITAM的基于免疫受体酪氨酸的活化基序。ITAM是充分定义的信号传导基序,其在用作syk/zap70类酪氨酸激酶结合位点的各种受体的胞质内尾中发现。用于本发明的ITAM的实例可以包括来源于TCRζ、FcRγ、FcRβ、FcRε、CD3γ、CD3δ、CD3ε、CD5、CD22、CD79a、CD79b和CD66d的那些非限制性实例。在一些实施方案中,CAR的胞内信号传导结构域可以包含CD3ζ(zeta)信号传导结构域,其具有与SEQ.ID NO:205中所示氨基酸序列至少约70%,优选至少80%,更优选至少90%、95%、97%或99%的序列同一性。在一些实施方案中,本发明CAR的胞内信号传导结构域包含共刺激分子的结构域。
在一些实施方案中,本发明CAR的胞内信号传导结构域包含共刺激分子的组成部分,共刺激分子选自41BB(GenBank:AAA53133.)和CD28(NP_006130.1)的片段。在一些实施方案中,本发明CAR的胞内信号传导结构域包含氨基酸序列,其包含与SEQ.ID NO:213(CD28信号传导结构域)或SEQ.ID NO:204(4-1BB信号传导结构域)中所示氨基酸序列至少70%,优选至少80%,更优选至少90%、95%、97%或99%的序列同一性。
CAR表达在细胞的表面膜上。因此,CAR能够包含跨膜结构域。用于本文公开的CAR的合适的跨膜结构域具有以下能力:(a)在细胞优选免疫细胞、例如但不限于淋巴细胞或自然杀伤(NK)细胞的表面表达,和(b)与配体结合结构域和胞内信号传导结构域发生相互作用,以指导免疫细胞针对预定的靶细胞的细胞应答。跨膜结构域可以来源于天然或合成来源。跨膜结构域可以来源于任意膜结合蛋白或跨膜蛋白。作为非限制性实例,跨膜多肽可以是T细胞受体的亚单位,例如α、β、γ或δ,构成CD3复合物的多肽,IL-2受体p55(a链),p75(β链)或γ链,Fc受体特别是Fcγ受体III或CD蛋白的亚单位链。或者,跨膜结构域可以是合成的并且可以主要包含疏水性残基,例如亮氨酸和缬氨酸。在一些实施方案中,所述跨膜结构域来源于人CD8α链(例如NP_001139345.1)。CAR还可以包含胞外配体结合结构域与所述跨膜结构域之间的柄结构域。柄结构域可以包含至多300个氨基酸,优选10个至100个氨基酸,且最优选25个至50个氨基酸。柄区可以来源于全部或部分天然存在的分子,例如来自CD8、CD4或CD28胞外区的全部或部分,或来自抗体恒定区的全部或部分。或者,柄结构域可以是对应于天然存在的柄序列的合成序列,或可以是完整的合成柄序列。在一些实施方案中,所述柄结构域是人CD8α链的一部分(例如NP_001139345.1)。在另一个具体的实施方案中,所述跨膜和铰链结构域包含人CD8α链的一部分,优选包含分别与SEQ ID NO:210和SEQ ID NO:208中所示氨基酸序列具有至少70%,优选至少80%,更优选至少90%、95%、97%或99%的序列同一性。在一些实施方案中,本文公开的CAR可以包含特异性地结合EGFRvIII的胞外配体结合结构域、CD8α人铰链和跨膜结构域、CD3ζ信号传导结构域和4-1BB信号传导结构域。
表4提供能够用于本文公开的CAR的结构域的示例性序列。
表4:CAR成分的示例性序列
表5A提供了本发明的示例性EGFRvIII特异性CAR的氨基酸序列。在表5A中,信号/前导肽序列用粗体表示,且GS接头[(GGGGS)4(SEQ ID NO:202)]加下划线。
表5A:示例性EGFRvIII特异性CAR的氨基酸序列
表5B提供了本发明的EGFRvIII特异性CAR的示例性scFv的核酸序列。在表5B中,编码CD8α信号/前导肽的序列加下划线。
表5B:示例性EGFRvIII特异性scFv的核酸序列
表5C提供了本发明的示例性EGFRvIII特异性CAR的示例性核酸序列。
表5C:示例性EGFRvIII特异性CAR的核酸序列
在癌细胞中常观察到靶抗原的下调或突变,产生抗原丢失逃逸变体。因此,为了抵消肿瘤逃逸并使免疫细胞对靶标更具特异性,EGFRvIII特异性CAR能够包含一个或多个额外的胞外配体结合结构域,以同时结合靶标中的不同元件,从而增强免疫细胞活化和功能。在一个实施方案中,胞外配体结合结构域可以串联放置在相同的跨膜多肽上,并且任选地可以通过接头分开。在一些实施方案中,所述不同的胞外配体结合结构域可以放置在组成CAR的不同跨膜多肽上。在一些实施方案中,本发明涉及CAR群,每个CAR包含不同的胞外配体结合结构域。具体而言,本发明涉及一种改造免疫细胞的方法,包括提供免疫细胞并在细胞表面表达CAR群,每个CAR包含不同的胞外配体结合结构域。在另一个具体实施方案中,本发明涉及一种改造免疫细胞的方法,包括提供免疫细胞和向细胞中导入编码多肽的多核苷酸,所述多肽组成CAR群,每个CAR包含不同的胞外配体结合结构域。CAR群意指至少两个、三个、四个、五个、六个或更多个CAR,每个CAR包含不同的胞外配体结合结构域。根据本发明的不同胞外配体结合结构域可以优选地同时结合靶标中的不同元件,从而增强免疫细胞活化和功能。本发明还涉及分离的免疫细胞,其包含CAR群,每个CAR包含不同的胞外配体结合结构域。
在另一个方面,本发明提供编码本文所述的任意CAR和多肽的多核苷酸。可以通过本领域公知的方法制备和表达多核苷酸。
在另一个方面,本发明提供包含本发明任意细胞的组合物(例如药物组合物)。在一些实施方案中,该组合物包含含有编码本文所述任意CAR的多核苷酸的细胞。
本文进一步描述了多核苷酸组合物的表达载体和施用。
在另一个方面,本发明提供制备本文所述任意多核苷酸的方法。
与任意这类序列互补的多核苷酸也被本发明涵盖。多核苷酸可以是单链(编码或反义)或双链的,并且可以是DNA(基因组、cDNA或合成的)或RNA分子。RNA分子包括HnRNA分子,其含有内含子且以一对一方式对应于DNA分子;和不含内含子的mRNA分子。另外的编码或非编码序列可以、但不一定存在于本发明的多核苷酸中,且多核苷酸可以、但不一定连接至其它分子和/或支持材料。
多核苷酸可包含天然序列(即,编码抗体或其部分的内源序列)或可包含这种序列的变体。多核苷酸变体含有一个或多个取代、添加、缺失和/或插入,使得相对于天然免疫反应性分子,编码的多肽的免疫反应性没有变小。通常可如本文所述评估对编码的多肽的免疫反应性的影响。变体优选表现出与编码天然抗体或其部分的多核苷酸序列至少约70%同一性,更优选至少约80%同一性,更优选至少约90%同一性,最优选至少约95%同一性。
如果两个序列中的核苷酸或氨基酸序列在如下所述进行最大对应性比对时是相同的,则称两个多核苷酸或多肽序列是“相同的”。通常通过在比较窗口上比较序列来鉴定和比较序列相似性的局部区域来执行两个序列之间的比较。如本文所用,“比较窗口”是指至少约20个、通常为30-约75个或40-约50个连续位置的段,其中可以两个序列最佳比对后将序列与相同数量的连续位置的参比序列进行比较。
用于比较的序列的最佳比对可以使用Lasergene生物信息学软件套件(DNASTAR,Inc.,Madison,WI)中的Megalign程序,使用默认参数进行。该程序体现了以下参考文献中描述的几种序列对比方案:Dayhoff,M.O.,1978,A model of evolutionary change in proteins-Matrices for detecting distant relationships.In Dayhoff,M.O.(ed.)Atlas of Protein Sequence and Structure,National Biomedical Research Foundation,Washington DC第5卷,增刊3,pp.345-358;Hein J.,1990,Unified Approach to Alignment and Phylogenes pp.626-645Methods in Enzymology第183卷,Academic Press,Inc.,San Diego,CA;Higgins,D.G.和Sharp,P.M.,1989,CABIOS 5:151-153;Myers,E.W.和Muller W.,1988、CABIOS 4:11-17;Robinson,E.D.,1971,Comb.Theor.11:105;Santou,N.,Nes,M.,1987、Mol.Biol.Evol.4:406-425;Sneath,P.H.A.和Sokal,R.R.,1973,Numerical Taxonomy the Principles and Practice of Numerical Taxonomy,Freeman Press,San Francisco,CA;Wilbur,W.J.和Lipman,D.J.,1983、Proc.Natl.Acad.Sci.USA 80:726-730。
优选地,“序列同一性百分比”通过在至少20个位置的比较窗口上比较两个最佳比对的序列来确定,其中比较窗口中的多核苷酸或多肽序列的部分可以包含对于两个序列的最佳比对,与参比序列(其不包含添加或缺失)相比,20%或更少,通常5%至15%或10%至12%的添加或缺失(即,缺口)。通过确定两个序列中相同核酸碱基或氨基酸残基出现的位置数来计算百分比,以产生匹配位置的数目,将匹配位置的数量除以参比序列中的位置总数(即窗口大小)和将结果乘以100以产生序列同一性百分比。
变体也可以或可选地与天然基因或其部分或互补基本上同源。此类多核苷酸变体能够在中等严紧条件下与编码天然抗体的天然存在的DNA序列(或互补序列)杂交。
合适的“中等严紧条件”包括在5×SSC、0.5%SDS、1.0mM EDTA(pH8.0)的溶液中预洗涤;在50℃-65℃、5×SSC下杂交过夜;然后在65℃下用含有0.1%SDS的2X、0.5X和0.2X SSC各自洗涤两次保持20分钟。
如本文所用,“高严紧条件”或“高严格条件”是指:(1)采用低离子强度和高温洗涤,例如在50℃下用0.015M氯化钠/0.0015M柠檬酸钠/0.1%十二烷基硫酸钠;(2)在杂交过程中使用变性剂,例如甲酰胺,例如,在42℃下50%(v/v)甲酰胺与0.1%牛血清白蛋白/0.1%Ficoll/0.1%聚乙烯吡咯烷酮/50mM磷酸钠缓冲液,pH6.5,含750mM氯化钠,75mM柠檬酸钠;或(3)42℃使用50%甲酰胺,5×SSC(0.75M NaCl,0.075M柠檬酸钠),50mM磷酸钠(pH6.8),0.1%焦磷酸钠,5×Denhardt's溶液,超声波鲑鱼精子DNA(50μg/ml),0.1%SDS和10%硫酸葡聚糖,42℃,0.2x SSC(氯化钠/柠檬酸钠)和50%甲酰胺,55℃洗涤,接着是在55℃下由含0.1%SSC的EDTA组成的高严格洗涤液。本领域技术人员将认识到如何根据需要调节温度、离子强度等以适应探针长度等因素。
本领域普通技术人员将理解,由于遗传密码的简并性,存在编码如本文所述的多肽的许多核苷酸序列。这些多核苷酸中的一些与任何天然基因的核苷酸序列具有最小的同源性。尽管如此,本发明特别考虑了由于密码子使用的差异而变化的多核苷酸。此外,包含本文提供的多核苷酸序列的基因的等位基因也在本发明的范围内。等位基因是内源基因,其由于一个或多个突变例如核苷酸的缺失、添加和/或取代而改变。得到的mRNA和蛋白质可以但不必具有改变的结构或功能。可以使用标准技术鉴定等位基因(例如杂交、扩增和/或数据库序列比较)。
可以使用化学合成、重组方法或PCR获得本发明的多核苷酸。化学多核苷酸合成的方法在本领域中是众所周知的,并且在本文中不需要详细描述。本领域技术人员能够使用本文提供的序列和商业DNA合成仪来产生所需的DNA序列。
为了使用重组方法制备多核苷酸,可以将包含所需序列的多核苷酸插入合适的载体中,并且可以进而将载体导入合适的宿主细胞中用于复制和扩增,如本文进一步讨论的。可以通过本领域已知的任何工具将多核苷酸插入宿主细胞中。通过直接摄取、内吞作用、转染、F-交配或电穿孔导入外源多核苷酸来转化细胞。一旦导入,外源多核苷酸可以作为非整合载体(例如质粒)或整合到宿主细胞基因组中维持在细胞内。如此扩增的多核苷酸可以通过本领域众所周知的方法从宿主细胞中分离。参见,例如Sambrook等人,1989。
或者,PCR允许复制DNA序列。PCR技术是本领域众所周知的且描述在美国专利号4,683,195、4,800,159、4,754,065和4,683,202以及PCR:The Polymerase Chain Reaction,Mullis等人编辑,Birkauswer Press,Boston,1994中。
可以通过使用在合适载体中分离的DNA并且将其插入合适的宿主细胞来得到RNA。当细胞复制且DNA被转录成RNA时,然后可以使用本领域技术人员众所周知的方法分离RNA,如例如上文Sambrook等人,1989中所举出的。
合适的克隆载体可以根据标准技术进行构建,或者可以选自本领域可获得的大量克隆载体。虽然选择的克隆载体可以根据预期使用的宿主细胞而变化,但是有用的克隆载体通常具有自我复制的能力,可以具有针对特定限制性内切核酸酶的单个靶标,和/或可以携带可用于选择包含载体的克隆的用于标记的基因。合适的实例包括质粒和细菌病毒,例如pUC18、pUC19、Bluescript(例如pBS SK+)及其衍生物、mp18、mp19、pBR322、pMB9、ColE1、pCR1、RP4、噬菌体DNA和穿梭载体,例如pSA3和pAT28。这些和许多其它克隆载体购自商品供应商,例如BioRad、Strategene和Invitrogen。
表达载体通常是可复制的多核苷酸构建体,其含有根据本发明的多核苷酸。暗示表达载体必须可以作为附加体或作为染色体DNA的整合部分在宿主细胞中复制。合适的表达载体包括但不限于PCT公开号WO 87/04462中公开的质粒、病毒载体,包括腺病毒、腺相关病毒、逆转录病毒、粘粒和表达载体。载体成分通常可包括但不限于以下一种或多种:信号序列;复制起点;一种或多种标记基因;合适的转录控制元件(例如启动子、增强子和终止子)。为了表达(即翻译),通常还需要一个或多个翻译控制元件,例如核糖体结合位点、翻译起始位点和终止密码子。
含有目的多核苷酸的载体可以通过许多适当的工具中的任何一种导入宿主细胞,包括电穿孔、使用氯化钙、氯化铷、磷酸钙、DEAE-葡聚糖或其它物质的转染;微粒轰击;脂质体转染;和感染(例如,其中载体是传染原,例如痘苗病毒)。导入载体或多核苷酸的选择通常取决于宿主细胞的特征。
编码本文公开的EGFRvIII特异性CAR的多核苷酸可存在于表达盒或表达载体中(例如用于导入细菌宿主细胞的质粒,或病毒载体,例如用于转染昆虫宿主细胞的杆状病毒载体,或质粒或病毒载体例如用于转染哺乳动物宿主细胞的慢病毒)。在一些实施方案中,多核苷酸或载体可包括编码核糖体跳跃序列的核酸序列,例如但不限于编码2A肽的序列。在小核糖核酸病毒的口蹄疫病毒(Aphthovirus)亚组中鉴定出的2A肽导致核糖体从一个密码子“跳跃”到下一个密码子而没有在密码子编码的两个氨基酸之间形成肽键(参见(Donnelly和Elliott 2001;Atkins,Wills等人2007;Doronina,Wu等人2008))。“密码子”是指mRNA上(或DNA分子的有义链上)的三个核苷酸,其被核糖体翻译成一个氨基酸残基。因此,当多肽被框内2A寡肽序列分开时,可以从imRNA内的单个连续开放阅读框合成两个多肽。这种核糖体跳跃机制在本领域中是众所周知的,并且已知由用于表达由单一信使RNA编码的几种蛋白质的几种载体使用。
为了将跨膜多肽引导到宿主细胞的分泌途径中,在一些实施方案中,在多核苷酸序列或载体序列中提供分泌信号序列(也称为信号肽、前导序列、前原(prepro)序列或前(pre)序列)。分泌信号序列与跨膜核酸序列可操作地连接,即,两个序列在正确的阅读框架中连接并定位以将新合成的多肽引导至宿主细胞的分泌途径。分泌信号序列通常位于编码目的多肽的核酸序列的5',尽管某些分泌信号序列可位于目的核酸序列的其他位置(参见,例如Welch等人,美国专利号5,037,743;Holland等人,美国专利号5,143,830)。在一些实施方案中,信号肽包含SEQ ID NO:206或214中所示的氨基酸序列。本领域技术人员将认识到,鉴于遗传密码的简并性,这些多核苷酸分子之间可能存在相当大的序列变异。在一些实施方案中,本发明的核酸序列经密码子优化以在哺乳动物细胞中表达,优选用于在人细胞中表达。密码子优化是指在给定物种的高表达的基因中通常很少的密码子中目的序列与在这些物种的高度表达的基因中频繁出现的密码子的交换,编码相同的氨基酸的这种密码子作为被交换的密码子。
改造免疫细胞的方法
本文提供了制备用于免疫疗法的免疫细胞的方法。在一些实施方案中,该方法包括将本发明的CAR导入免疫细胞并且扩增该细胞。在一些实施方案中,本发明涉及改造免疫细胞的方法,其包括:提供细胞并且在该细胞表面上表达如上所述的至少一种CAR。用于改造免疫细胞的方法描述在例如PCT专利申请公开号WO/2014/039523、WO/2014/184741、WO/2014/191128、WO/2014/184744和WO/2014/184143中,每篇文献通过引用整体并入本文。在一些实施方案中,该方法包括:用如上所述编码CAR的至少一种多核苷酸转化所述细胞并且在细胞中表达多核苷酸。
在一些实施方案中,多核苷酸存在于用于细胞中稳定表达的慢病毒载体中。
在一些实施方案中,该方法还可以包括通过使至少一种基因失活进行遗传修饰的步骤,所述基因表达例如但不限于TCR的成分,用于免疫抑制剂的靶标,HLA基因,和/或免疫检查点蛋白,例如,PDCD1或CTLA-4。通过使基因失活,预期目的基因不以功能蛋白形式被表达。在一些实施方案中,待失活的基因选自例如但不限于TCRα、TCRβ、dCK、CD52、GR、PD-1和CTLA-4。在一些实施方案中,该方法包括通过导入细胞稀有切割反应内切核酸酶使一种或多种基因失活,所述稀有切割反应内切核酸酶能够通过选择性DNA切割使基因选择性失活。在一些实施方案中,稀有切割反应内切核酸酶可以是例如转录激活物样效应核酸酶(TALE-核酸酶)或Cas9内切核酸酶。
在一些实施方案中,另外的催化结构域与稀有切割内切核酸酶一起使用以增强其灭活靶向基因的能力。例如,另外的催化结构域可以是DNA末端加工酶。DNA末端加工酶的非限制性实例包括5-3'外切核酸酶,3-5'外切核酸酶,5-3'碱性外切核酸酶,5'瓣状内切核酸酶,解旋酶,磷酸酶,水解酶和模板非依赖性DNA聚合酶。此类催化结构域的非限制性实例由蛋白质结构域或蛋白质结构域的催化活性衍生物组成,其选自hExoI(EXO1_HUMAN),酵母ExoI(EXO1_YEAST),大肠杆菌(E.coli)ExoI,人TREX2,小鼠TREX1,人TREX1,牛TREX1,大鼠TREX1,TdT(末端脱氧核苷酸转移酶),人DNA2,酵母DNA2(DNA2_YEAST)。在一些实施方案中,另外的催化结构域可以具有3′-5′-外切核酸酶活性,并且在一些实施方案中,所述另外的催化结构域是TREX,更优选TREX2催化结构域(WO2012/058458)。在一些实施方案中,所述催化结构域由单链TREX多肽编码。另外的催化结构域可以与核酸酶融合蛋白或嵌合蛋白融合。在一些实施方案中,另外的催化结构域使用例如肽接头融合。
在一些实施方案中,该方法进一步包括向细胞中导入包含与靶核酸序列的一部分同源的至少一个序列的外源核酸的步骤,使得在靶核酸序列和外源核酸之间发生同源重组。在一些实施方案中,所述外源核酸包含分别与靶核酸序列的区域5'和3'同源的第一和第二部分。外源核酸还可以包含位于第一和第二部分之间的第三部分,其与靶核酸序列的区域5'和3'不具有同源性。在切割靶核酸序列后,在靶核酸序列和外源核酸之间刺激同源重组事件。在一些实施方案中,可以在供体基质内使用至少约50bp,大于约100bp或大于约200bp的同源序列。外源核酸可以是例如但不限于约200bp至约6000bp、更优选约1000bp至约2000bp。共有核酸同源物位于侧接断裂位点上游和下游的区域中,并且待导入的核酸序列位于两个臂之间。
在一些实施方案中,核酸连续地包含与所述切割上游序列同源的第一同源区;使靶向基因失活的序列,该基因选自TCRα,TCRβ,CD52,糖皮质激素受体(GR),脱氧胞苷激酶(dCK)和免疫检查点蛋白例如程序性死亡-1(PD-1);和与所述切割下游序列同源的第二同源区。多核苷酸导入步骤可以在导入或表达稀有切割反应内切核酸酶的同时、之前或之后进行。取决于其中发生断裂事件的靶核酸序列的位置,这种外源核酸可用于敲除基因(例如当外源核酸位于基因的开放阅读框内)或用于导入新序列或目的基因。通过使用这种外源核酸的序列插入可以用于修饰靶向的现有基因,通过该基因的校正或替换(作为非限制性实例的等位基因交换),或用于上调或下调靶向基因的表达(作为非限制性实例的启动子交换),靶向基因校正或替换。在一些实施方案中,选自TCRα、TCRβ、CD52、GR、dCK和免疫检查点蛋白的基因的失活可以在由特异性TALE-核酸酶靶向的精确基因组位置完成,其中所述特异性TALE-核酸酶催化切割和其中外源核酸连续包含至少一个同源区和一个序列以灭活选自TCRα、TCRβ、CD52、GR、dCK、免疫检查点蛋白的一种靶基因,其通过同源重组整合。在一些实施方案中,通过分别使用几种TALE-核酸酶并且特异性地靶向一种确定的基因和用于特异性基因失活的几种特异性多核苷酸,可以连续或同时使几种基因失活。
在一些实施方案中,该方法包括灭活一种或多种选自TCRα、TCRβ、CD52、GR、dCK和免疫检查点蛋白的其他基因。在一些实施方案中,基因的失活可以通过向细胞中导入至少一种稀有切割反应内切核酸酶来实现,使得稀有切割反应内切核酸酶特异性地催化细胞基因组的靶向序列中的切割;并且任选地将外源核酸导入到细胞中,所述外源核酸连续包含与切割上游序列同源的第一同源区,待被插入细胞基因组的序列和与切割下游序列同源的第二同源区;其中导入的外源核酸灭活基因和整合至少一种编码至少一种目的重组蛋白的外源多核苷酸序列。在一些实施方案中,外源多核苷酸序列整合在编码选自TCRα、TCRβ、CD52、GR、dCK和免疫检查点蛋白的蛋白质的基因内。
在另一个方面,遗传修饰细胞的步骤可包括:通过灭活表达免疫抑制剂的靶标的至少一种基因来修饰T细胞,和任选地在免疫抑制剂存在下扩增细胞。免疫抑制剂是通过几种作用机制之一抑制免疫功能的药剂。免疫抑制剂能够减少免疫应答的程度和/或贪婪(voracity)。免疫抑制剂的非限制性实例包括钙调磷酸酶抑制剂,雷帕霉素的靶,白细胞介素-2α链阻断剂,肌苷一磷酸脱氢酶抑制剂,二氢叶酸还原酶抑制剂,皮质类固醇和免疫抑制抗代谢物。一些细胞毒性免疫抑制剂通过抑制DNA合成起作用。其他可通过激活T细胞或通过抑制辅助细胞的活化来起作用。根据本发明的方法允许通过使T细胞中的免疫抑制剂的靶失活来赋予对T细胞的免疫抑制抗性以用于免疫疗法。作为非限制性实例,免疫抑制剂的靶标可以是免疫抑制剂的受体,例如但不限于CD52、糖皮质激素受体(GR)、FKBP家族基因成员和亲环蛋白家族基因成员。
在一些实施方案中,该方法的遗传修饰涉及在提供的细胞中表达一种稀有切割反应内切核酸酶,使得稀有切割反应内切核酸酶特异性地催化一种靶向基因的切割,从而使靶向基因失活。在一些实施方案中,改造细胞的方法包括至少一种以下步骤:提供T细胞,例如来自细胞培养物或来自血样;选择表达免疫抑制剂靶标的T细胞中的基因;向T细胞中导入能够通过DNA切割、优选通过双链断裂选择性灭活编码免疫抑制剂靶标的基因的稀有切割反应内切核酸酶,和任选地在免疫抑制剂存在下扩增细胞。
在一些实施方案中,该方法包括:提供T细胞,例如来自细胞培养物或来自血样;选择T细胞中的基因,其中该基因表达免疫抑制剂靶标;用编码稀有切割反应内切核酸酶的核酸转化T细胞,所述稀有切割反应内切核酸酶能够通过DNA切割、优选通过双链断裂选择性灭活编码免疫抑制剂靶标的基因,并将稀有切割反应内切核酸酶表达到T细胞中;和任选地在免疫抑制剂存在下扩增细胞。
在一些实施方案中,稀有切割反应内切核酸酶特异性靶向CD52或GR。在一些实施方案中,选择用于灭活的基因编码CD52,并且免疫抑制治疗包含靶向CD52抗原的人源化抗体。在一些实施方案中,选择用于灭活的基因编码GR,且免疫抑制治疗包含皮质类固醇例如地塞米松。在一些实施方案中,选择用于灭活的基因是FKBP家族基因成员或其变体,并且免疫抑制治疗包括FK506,也称作他克莫司或藤霉素。在一些实施方案中,FKBP家族基因成员是FKBP12或其变体。在一些实施方案中,选择用于灭活的基因是亲环蛋白家族基因成员或其变体,并且免疫抑制治疗包括环孢菌素。
在一些实施方案中,稀有切割反应内切核酸酶可以是例如兆核酸酶、锌指核酸酶或TALE-核酸酶。在一些实施方案中,稀有切割反应内切核酸酶是TALE-核酸酶。
本文还提供了适用于免疫疗法的改造T细胞的方法,其中所述方法包括:通过灭活至少一种免疫检查点蛋白来遗传修饰T细胞。在一些实施方案中,免疫检查点蛋白是例如PD-1和/或CTLA-4。在一些实施方案中,遗传修饰细胞的方法包括:通过灭活至少一种免疫检查点蛋白来修饰T细胞;和扩增细胞。免疫检查点蛋白包括但不限于程序性死亡1(PD-1、也称作PDCD1或CD279,登录号:NM-005018)、细胞毒性T-淋巴细胞抗原4(CTLA-4,也称作CD152,GenBank登录号AF414120.1)、LAG3(也称作CD223,登录号:NM-002286.5)、Tim3(也称作HAVCR2,GenBank登录号:JX049979.1)、BTLA(也称作CD272,登录号:NM-181780.3)、BY55(也称作CD160,GenBank登录号:CR541888.1)、TIGIT(也称作VSTM3,登录号:NM-173799)、B7H5(也称作C10orf54,小鼠vista基因同源物,登录号:NM-022153.1)、LAIR1(也称作CD305,GenBank登录号:CR542051.1)、SIGLEC10(GeneBank登录号:AY358337.1)、2B4(也称作CD244,登录号:NM-001166664.1),它们直接抑制免疫细胞。例如,CTLA-4是在某些CD4和CD8T细胞上表达的细胞表面蛋白;当被其抗原呈递细胞上的配体(B7-1和B7-2)接合时,T细胞活化和效应子功能受到抑制。
在一些实施方案中,所述改造细胞的方法包括至少一个以下步骤:提供T细胞,例如来自细胞培养物或来自血样;将能够通过DNA切割、优选通过双链断裂选择性灭活一个编码免疫检查点蛋白的基因的稀有切割反应内切核酸酶导入T细胞中;并扩增细胞。在一些实施方案中,该方法包括:提供T细胞,例如来自细胞培养物或来自血样;用编码稀有切割反应内切核酸酶的核酸转染所述T细胞,所述稀有切割反应内切核酸酶能够通过DNA切割、优选通过双链断裂选择性灭活编码免疫检查点蛋白的基因;将稀有切割反应内切核酸酶表达到T细胞中;扩增细胞。在一些实施方案中,该稀有切割反应内切核酸酶特异性地靶向选自PD-1、CTLA-4、LAG3、Tim3、BTLA、BY55、TIGIT、B7H5、LAIR1、SIGLEC10、2B4、TCRα和TCRβ的基因。在一些实施方案中,稀有切割反应内切核酸酶可以是兆核酸酶、锌指核酸酶或TALE-核酸酶。在一些实施方案中,稀有切割反应内切核酸酶是TALE-核酸酶。
在一些实施方案中,本发明可特别适用于同种异体免疫疗法。在此类实施方案中,细胞可通过以下方法修饰,该方法包括:使编码T细胞中T细胞受体(TCR)组分的至少一种基因失活;和扩增T细胞。在一些实施方案中,该方法的遗传修饰依赖于在提供的待改造细胞中表达一种稀有切割反应内切核酸酶,使得稀有切割反应内切核酸酶特异性地催化一种靶向基因中的切割,从而使靶向基因失活。在一些实施方案中,所述改造细胞的方法包括至少一种以下步骤:提供T细胞,例如来自细胞培养物或来自血样;向T细胞中导入能够通过DNA切割、优选通过双链断裂选择性灭活至少一种基因编码T细胞受体(TCR)的组分的稀有切割反应内切核酸酶,和扩增细胞。
在一些实施方案中,该方法包括:提供T细胞,例如来自细胞培养物或来自血样;用编码稀有切割反应内切核酸酶的核酸转染所述T细胞,所述稀有切割反应内切核酸酶能够通过DNA切割、优选通过双链断裂选择性灭活至少一种编码T细胞受体(TCR)成分的基因;将稀有切割内切核酸酶表达到T细胞中;分选转染的T细胞,其在细胞表面不表达TCR;和扩增细胞。
在一些实施方案中,稀有切割反应内切核酸酶可以是兆核酸酶、锌指核酸酶或TALE-核酸酶。在一些实施方案中,稀有切割反应内切核酸酶是TALE-核酸酶。在一些实施方案中,TALE-核酸酶识别和切割编码TCRα或TCRβ的序列。在一些实施方案中,TALE-核酸酶包含选自SEQ ID NO:218、219、220、221、222、223、224或225中所示的氨基酸序列的多肽序列。
TALE-核酸酶多肽序列:
重复TRAC_T01-L
LTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGRPALE(SEQ ID NO:218)
重复TRAC_T01-R
LTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGRPALE(SEQ ID NO:219)
重复TRBC_T01-L
LTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNGGGRPALE(SEQ ID NO:220)
重复TRBC_T01-R
NPQRSTVWYLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGRPALE(SEQ ID NO:221)
重复TRBC_T02-L
LTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGRPALE(SEQ ID NO:222)
重复TRBC_T02-R
LTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGRPALE(SEQ ID NO:223)
重复CD52_T02-L
LTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPQQVVAIASNGGGRPALE(SEQ ID NO:224)
重复CD52_T02-R
LTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNNGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGKQALETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQALETVQALLPVLCQAHGLTPEQVVAIASHDGGKQALETVQRLLPVLCQAHGLTPQQVVAIASNGGGRPALE(SEQ ID NO:225)
在另一个方面,遗传修饰细胞的另一步骤可以是扩增TCRα缺陷型T细胞的方法,包括向T细胞中导入pTα(也称作preTCRα)或其功能变体,并任选地通过刺激CD3复合物扩增细胞。在一些实施方案中,该方法包括:a)用编码至少pTα片段的核酸转化细胞以支持CD3表面表达;b)将所述pTα表达到细胞中;和c)任选地通过刺激CD3复合物扩增细胞。
还提供了制备用于免疫疗法的T细胞的方法,包括本文提供的用于扩增T细胞的方法的步骤。在一些实施方案中,pTα多核苷酸序列可以被随机导入或通过同源重组导入。在一些实施方案中,插入可以与TCRα基因的失活相关。
可以使用pTα的不同功能变体。肽的“功能变体”是指与整个肽或其片段基本相似的分子。pTα或其功能变体的“片段”是指比全长pTα更短的肽的分子的任何子集。在一些实施方案中,pTα或功能变体可以是例如全长pTα或C-末端截短的pTα形式。C-末端截短的pTα在C末端缺少一个或多个残基。作为非限制性实例,C-末端截短的pTα形式缺少来自蛋白质C-末端的18、48、62、78、92、110或114个残基。肽的氨基酸序列变体可以通过编码肽的DNA中的突变来制备。这些功能变体包括例如氨基酸序列内残基的缺失或插入或取代。只要最终构建体具有所需活性、特别是功能性CD3复合物的恢复,也可以进行缺失、插入和取代的任何组合以实现最终构建体。在优选的实施方案中,在不同的如上所述pTα形式中导入至少一种突变以影响二聚化。作为非限制性实例,突变残基可以是人pTα蛋白或使用CLUSTALW方法对pTα家族或同源成员的比对位置的至少W46R、D22A、K24A、R102A或R117A。优选地,如上所述的pTα或其变体包含突变的残基W46R或突变的残基D22A、K24A、R102A和R117A。在一些实施方案中,所述pTα或变体也与信号转导结构域例如作为非限制性实例的CD28、OX40、ICOS、CD27、CD137(4-1BB)和CD8融合。如上所述的pTα或变体的胞外结构域可以与TCRα蛋白的片段、特别是TCRα的跨膜和胞内结构域融合。pTα变体也可以与TCRα的胞内结构域融合。
在一些实施方案中,pTα形式可以与胞外配体结合结构域融合。在一些实施方案中,pTα或其功能变体与单链抗体片段(scFv)融合,所述scFv包含通过柔性接头连接的靶抗原特异性单克隆抗体的轻和重的可变片段。
术语“TCRα缺陷型T细胞”是指缺乏功能性TCRα链表达的分离的T细胞。这可以通过不同的方式实现,作为非限制性实例,通过改造T细胞使得其不在其细胞表面上表达任何功能性TCRα或通过改造T细胞使得其在其表面上产生非常少的功能性TCRα链或通过改造T细胞来表达突变或截短形式的TCRα链。TCRα缺陷型T细胞不能再通过CD3复合物扩增。因此,为了克服该问题并允许TCRα缺陷型T细胞的增殖,将pTα或其功能变体导入细胞中,从而恢复功能性CD3复合物。在一些实施方案中,该方法还包括向所述T细胞中导入稀有切割反应内切核酸酶,其能够通过DNA切割选择性灭活一种编码T细胞受体(TCR)的一种成分的基因。在一些实施方案中,稀有切割反应内切核酸酶是TALE-核酸酶。
在另一个方面,通过本文描述的方法获得的改造的T细胞可以与双特异性抗体接触。例如,T细胞可以在施用于患者之前离体与双特异性抗体接触,或者在施用于患者后体内接触。双特异性抗体包含两个可变区,其具有不同的抗原特性,这些特性有助于使改造的细胞接近靶抗原。作为非限制性实例,双特异性抗体可以针对肿瘤标记和淋巴细胞抗原例如但不限于CD3,并且具有重新指导和激活针对肿瘤的任何循环T细胞的潜力。
在一些实施方案中,本发明的编码多肽的多核苷酸可以是例如通过电穿孔直接导入细胞的mRNA。在一些实施方案中,cytoPulse技术可用于瞬时透化活细胞以将材料递送到细胞中。可以修改参数以确定具有最小死亡率的高转染效率的条件。
本文还提供了转化T细胞的方法。在一些实施方案中,该方法包括:使T细胞与RNA接触并向T细胞施加由下列组成的灵敏(agile)脉冲序列:(a)具有电压范围约为2250至3000V/厘米的电脉冲;(b)脉冲宽度为0.1ms;(c)步骤(a)和(b)的电脉冲之间的脉冲间隔为约0.2至10ms;(d)具有电压范围约为2250至3000V、脉冲宽度约为100ms的电脉冲,并且步骤(b)的电脉冲与步骤(c)的首个电脉冲之间的脉冲间隔约为100ms;以及(e)电压约为325V的四个电脉冲,其中4个电脉冲中的每一个之间的脉冲宽度约为0.2ms和脉冲间隔为2ms。在一些实施方案中,转化T细胞的方法,包括使所述T细胞与RNA接触并向T细胞施加灵敏脉冲序列,所述灵敏脉冲序列包括:(a)具有电压为约2250、2300、2350、2400、2450、2500、2550、2400、2450、2500、2600、2700、2800、2900或3000V/厘米的电脉冲;(b)脉冲宽度为0.1ms;(c)和步骤(a)和(b)的电脉冲之间的脉冲间隔约为0.2、0.5、1、2、3、4、5、6、7、8、9或10ms;(d)电压范围从约2250开始,2250、2300、2350、2400、2450、2500、2550、2400、2450、2500、2600、2700、2800、2900或3000V,脉冲宽度为100ms的一个电脉冲,步骤(b)的电脉冲和步骤(c)的首个电脉冲之间的脉冲间隔为100ms;和(e)4个电脉冲,其中电压约为325V,脉冲宽度约为0.2ms,并且在4个电脉冲中的每一个之间具有约2ms的脉冲间隔。包含在上述值范围中的任何值都在本申请中公开。电穿孔介质可以是本领域已知的任何合适的介质。在一些实施方案中,电穿孔介质具有跨越约0.01-约1.0毫西门子范围的电导率。
在一些实施方案中,作为非限制性实例,RNA编码稀有切割反应内切核酸酶,稀有切割反应内切核酸酶的一种单体,例如半TALE核酸酶,CAR,多链嵌合抗原受体的至少一种成分,pTα或其功能性变体,外源核酸,和/或一种另外的催化结构域。
改造的免疫细胞
本发明还提供了包含本文所述的任何CAR多核苷酸的改造的免疫细胞。在一些实施方案中,可以通过质粒载体将CAR作为转基因导入免疫细胞中。在一些实施方案中,质粒载体还可含有例如选择标记,其提供对接收载体的细胞的鉴定和/或选择。
在将编码CAR多肽的多核苷酸导入细胞后,可以在细胞中原位合成CAR多肽。或者,CAR多肽可以在胞外产生,然后导入细胞中。将多核苷酸构建体导入细胞的方法是本领域已知的。在一些实施方案中,稳定转染方法可用于将多核苷酸构建体整合到细胞的基因组中。在其它实施方案中,瞬时转染方法可用于瞬时表达多核苷酸构建体,并且多核苷酸构建体未整合到细胞的基因组中。在其它实施方案中,可以使用病毒介导的方法。可以通过任何合适的工具例如重组病毒载体(例如逆转录病毒、腺病毒)、脂质体等将多核苷酸导入细胞中。瞬时转染方法包括例如但不限于显微注射、电穿孔或粒子轰击。多核苷酸可以包括在载体例如质粒载体或病毒载体中。
本文还提供了通过上述本文提供的改造细胞的方法获得的分离的细胞和细胞系。在一些实施方案中,分离的细胞包含至少一种如上所述的CAR。在一些实施方案中,分离细胞包含CAR群,每个CAR包含不同的胞外配体结合结构域。
本文还提供了根据上述任一方法获得的分离的免疫细胞。能够表达异源DNA的任何免疫细胞可用于表达目的CAR的目的。在一些实施方案中,免疫细胞是T细胞。在一些实施方案中,免疫细胞可以衍生自例如但不限于干细胞。干细胞可以是成体干细胞,非人胚胎干细胞,更特别是非人干细胞,脐带血干细胞,祖细胞,骨髓干细胞,诱导的多能干细胞,全能干细胞或造血干细胞。代表性人细胞是CD34+细胞。分离的细胞也可以是树突细胞,杀伤性树突细胞,肥大细胞,NK细胞,B细胞或T细胞,其选自炎性T淋巴细胞,细胞毒性T淋巴细胞,调节T-淋巴细胞,记忆T-淋巴细胞或辅助T-淋巴细胞。在一些实施方案中,细胞可以衍生自CD4+T-淋巴细胞和CD8+T-淋巴细胞。
在扩增和遗传修饰之前,可以通过各种非限制性方法从受试者获得细胞来源。细胞可以从许多非限制性来源获得,包括外周血单核细胞,骨髓,淋巴结组织,脐带血,胸腺组织,来自感染部位的组织,腹水,胸腔积液,脾组织和肿瘤。在一些实施方案中,可以使用本领域技术人员可获得和已知的任何数量的T细胞系。在一些实施方案中,细胞可以衍生自健康供体,衍生自被诊断患有癌症的患者或衍生自被诊断患有感染的患者。在一些实施方案中,细胞可以是呈现不同表型特征的混合细胞群的一部分。
本文还提供了从根据任何上述方法的转染的T细胞获得的细胞系。本文还提供了对免疫抑制治疗具有抗性的修饰细胞。在一些实施方案中,本发明的分离细胞包含编码CAR的多核苷酸。
本发明的免疫细胞可以在T细胞遗传修饰之前或之后,使用一般描述的方法进行活化和扩增,所述方法例如但不限于美国专利号6,352,694;6,534,055;6,905,680;6,692,964;5,858,358;6,887,466;6,905,681;7,144,575;7,067,318;7,172,869;7,232,566;7,175,843;5,883,223;6,905,874;6,797,514;6,867,041;和美国专利申请公开号20060121005中所述的方法。可以在体外或体内扩增T细胞。通常,可以通过例如与刺激CD3TCR复合物和T细胞表面上的共刺激分子的药剂接触以产生T细胞的激活信号而扩增本发明的T细胞。例如,钙离子载体A23187,佛波醇12-肉豆蔻酸酯13-乙酸酯(PMA)或有丝分裂凝集素如植物血凝素(PHA)可用于产生T细胞的激活信号。
在一些实施方案中,T细胞群可以通过与例如固定在表面上的抗-CD3抗体或其抗原结合片段或抗-CD2抗体接触或通过与结合钙离子载体的蛋白激酶C活化剂(例如苔藓虫素)接触而在体外被刺激。为了共刺激T细胞表面上的辅助分子,使用结合辅助分子的配体。例如,在适于刺激T细胞增殖的条件下,可使T细胞群与抗-CD3抗体和抗-CD28抗体接触。适合T细胞培养的条件包括适当的培养基(例如最小必需培养基或RPMI培养基1640或X-Vivo 15,(Lonza)),其可含有增殖和生存所必需的因子,包括血清(例如胎牛血清或人血清),白细胞介素-2(IL-2),胰岛素,IFN-γ,IL-4,IL-7,GM-CSF,IL-10,IL-2,IL-15,TGFβ和TNF或任何其它本领域技术人员已知的用于细胞生长的添加剂。用于细胞生长的其它添加剂包括但不限于表面活性剂、人血浆蛋白粉(plasmanate)和还原剂,例如N-乙酰-半胱氨酸和2-巯基乙醇。培养基可包括RPMI 1640,AIM-V,DMEM,MEM,a-MEM,F-12,X-Vivo 10和X-Vivo 20,Optimizer,并且添加氨基酸、丙酮酸钠和维生素,无血清或补充有适量的血清(或血浆)或一组确定的激素,和/或一定量的足以使T细胞生长和扩增的细胞因子。抗生素,例如青霉素和链霉素,仅包括在实验培养物中,而不包括在要注入受试者的细胞培养物中。靶细胞维持在支持生长所必需的条件下,例如,适当的温度(例如37℃)和气氛(例如空气加5%CO2)。已暴露于不同刺激时间的T细胞可表现出不同的特征。
在一些实施方案中,通过与组织或细胞共培养,可以扩增本发明的细胞。细胞也可以在体内例如在将细胞施用于受试者后在受试者的血液中扩增。
在一些实施方案中,本发明分离的细胞包含一种失活的基因,其选自CD52、dCK、GR、PD-1、CTLA-4、LAG3、Tim3、BTLA、BY55、TIGIT、B7H5、LAIR1、SIGLEC10、2B4、HLA、TCRα和TCRβ和/或表达CAR、多链CAR和/或pTα转基因。在一些实施方案中,分离的细胞包含编码含有多链CAR的多肽的多核苷酸。在一些实施方案中,本发明分离的细胞包含2种失活的基因,其选自:CD52和GR、CD52和TCRα、CDR52和TCRβ、GR和TCRα、GR和TCRβ、TCRα和TCRβ、PD-1和TCRα、PD-1和TCRβ、CTLA-4和TCRα、CTLA-4和TCRβ、LAG3和TCRα、LAG3和TCRβ、Tim3和TCRα、Tim3和TCRβ、BTLA和TCRα、BTLA和TCRβ、BY55和TCRα、BY55和TCRβ、TIGIT和TCRα、TIGIT和TCRβ、B7H5和TCRα、B7H5和TCRβ、LAIR1和TCRα、LAIR1和TCRβ、SIGLEC10和TCRα、SIGLEC10和TCRβ、2B4和TCRα、2B4和TCRβ和/或表达CAR、多链CAR和pTα转基因。
在一些实施方案中,通过灭活TCRα和/或TCRβ基因,使TCR在本发明的细胞中不起作用。在一些实施方案中,提供了获得源自个体的修饰细胞的方法,其中细胞可以独立于主要组织相容性复合物(MHC)信号传导途径增殖。可以通过该方法获得的可以独立于MHC信号传导途径增殖的修饰细胞包括在本发明的范围内。本文公开的修饰细胞可用于治疗需要其的患者对抗宿主对移植物(HvG)排斥和移植物抗宿主病(GvHD);因此在本发明的范围内是治疗需要其的患者对抗宿主对移植物(HvG)排斥和移植物抗宿主病(GvHD)的方法,包括通过向所述患者施用有效量的包含灭活的TCRα和/或TCRβ基因的修饰细胞来治疗所述患者。
在一些实施方案中,免疫细胞被改造成对一种或多种化疗药物具有抗性。化疗药物可以是例如嘌呤核苷酸类似物(PNA),因此使免疫细胞适合用于组合过继免疫疗法和化疗的癌症治疗。示例性PNA包括例如单独或组合形式的氯法拉滨、氟达拉滨和阿糖胞苷。PNA被脱氧胞苷激酶(dCK)代谢成单磷酸、二磷酸和三磷酸PNA。它们的三磷酸形式与DNA合成竞争ATP,作为促凋亡剂并且是参与三核苷酸生产的核糖核苷酸还原酶(RNR)的有效抑制剂。本文提供了包含失活的dCK基因的EGFRvIII特异性CAR-T细胞。在一些实施方案中,使用编码针对dCK基因的特异性TAL-核酸酶的多核苷酸通过例如mRNA的电穿孔转染T细胞来制备dCK敲除细胞。dCK敲除EGFRvIII特异性CAR-T细胞对PNA包括例如氯法拉滨和/或氟达拉滨具有抗性以及维持对表达EGFRvIII的细胞的T细胞细胞毒性活性。在另一个实例中,化疗药物可以是例如CD52靶向分子,例如单克隆抗-CD52抗体(例如阿仑珠单抗)。CD52是一种存在于淋巴细胞表面的蛋白质,且抗-CD52抗体可通过抗体依赖性和补体依赖性细胞毒性作用诱导免疫细胞凋亡和裂解,导致淋巴细胞清除(lymphodepletion)。本文提供了包含失活的CD52基因的EGFRvIII特异性CAR-T细胞。在一些实施方案中,CD52敲除细胞是通过用编码针对CD52基因的特异性TAL-核酸酶的多核苷酸通过例如mRNA的电穿孔转染T细胞而制备的。CD52敲除EGFRvIII特异性CAR-T细胞对抗-CD52分子具有抗性,包括例如阿仑珠单抗和在抗-CD52分子存在下维持对表达EGFRvIII的细胞的T细胞细胞毒性活性。
在一些实施方案中,本发明分离的细胞或细胞系可以包含pTα或其功能性变体。在一些实施方案中,分离的细胞或细胞系可以通过使TCRα基因失活被进一步遗传修饰。
在一些实施方案中,CAR-T细胞包含编码自杀多肽例如RQR8的多核苷酸。参见,例如WO2013153391A,其通过引用整体并入本文。在包含编码自杀多肽的多核苷酸的CAR-T细胞中,在CAR-T细胞表面上表达自杀多肽。在一些实施方案中,自杀多肽包含SEQ ID NO:226中所示的氨基酸序列。
CPYSNPSLCSGGGGSELPTQGTFSNVSTNVSPAKPTTTACPYSNPSLCSGGGGSPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCNHRNRRRVCKCPRPW(SEQ ID NO.226)。
在一些实施方案中,自杀多肽还可以包含在氨基末端上的信号肽。当在CAR-T细胞表面上表达自杀多肽时,利妥昔单抗与该多肽R表位[即由利妥昔单抗识别的表位–CPYSNPSLC(SEQ ID NO:256)])结合导致细胞溶胞。超过一个利妥昔单抗的分子可以结合细胞表面上表达的每个多肽。该多肽的每个R表位可以结合利妥昔单抗的分开的分子。EGFRvIII特异性CAR-T细胞的缺失可以在体内出现,例如通过将利妥昔单抗施用于患者。决定缺失转移的细胞可以来源于在可归因于转移的细胞的患者中检测到的不期望的作用,例如,当检测到不可接受的水平的毒性时。
在一些实施方案中,自杀多肽可以含有1、2、3个或更多的由抗体(例如利妥昔单抗)识别的表位。
在一些实施方案中,可以在EGFRvIII特异性CAR T细胞中以与含有CAR多肽的多肽分开的多肽的形式提供自杀多肽。在一些实施方案中,可以EGFRvIII特异性CAR T细胞中以与CAR多肽相同的多肽链的形式提供自杀多肽。在含有自杀多肽的CAR中,典型地以CAR的胞外部分的形式提供自杀多肽。
在含有自杀多肽的CAR中,自杀多肽可含有利妥昔单抗[CPYSNPSLC(SEQ ID NO:256)]识别的表位的氨基酸序列的一个或多个拷贝。自杀多肽可以位于CAR中的不同位置。例如,自杀多肽可以是scFv的N-末端,或者它可以是CAR中scFv的C-末端。在一些实施方案中,自杀肽可以在CAR的scFv和铰链区之间。在一些实施方案中,CAR可以含有多于一个自杀多肽。例如,CAR可以含有scFv的N-末端的自杀多肽和scFv的C-末端的自杀多肽。这些多肽中的每一个可含有利妥昔单抗识别的表位的一个或多个拷贝。例如,CAR可以含有在scFv的N-末端的位置的第一自杀多肽,其中第一自杀多肽含有利妥昔单抗识别的表位的一个拷贝,和在scFv的C-末端的位置的第二自杀多肽,其中第二自杀多肽含有利妥昔单抗识别的表位的两个拷贝。
本文还提供了编码EGFRvIII-特异性CAR的核酸,所述EGFRvIII-特异性CAR在CAR中含有自杀多肽。
在实例中,与EGFRvIII特异性CAR在相同的多肽链中的自杀多肽可具有本文所述提供的序列作为“R2自杀序列”。R2自杀序列含有利妥昔单抗识别的表位[CPYSNPSLC(SEQ ID NO:256)]的两个拷贝。此类EGFRvIII-特异性CAR在本文中可称为“EGFRvIII-R2 CAR”。表5D提供了本发明的示例性EGFRvIII-R2 CAR的氨基酸序列。在表5D中,信号/前导肽序列以粗体显示,GS接头[(GGGGS)4(SEQ ID NO:202)]加下划线显示,且R2自杀序列以粗体并加下划线显示。表5E提供了编码本发明的示例性EGFRvIII-R2 CAR的示例性核酸序列。
表5D:具有R2自杀序列的示例性EGFRvIII特异性CAR的氨基酸序列
表5E:具有R2自杀序列的示例性EGFR VIII特异性CAR的核酸序列
治疗应用
通过上述方法得到的分离的细胞或衍生自这类分离细胞的细胞可以用作药物。在一些实施方案中,这类药物可以用于治疗癌症,包括实体瘤和液体瘤。在一些实施方案中,所述癌症是EGFRvIII相关癌症(例如表达任意EGFRvIII的癌症),包括但不限于胶质母细胞瘤(例如多形性胶质母细胞瘤)、间变性星形细胞瘤、巨细胞胶质母细胞瘤、胶质肉瘤、间变性少突胶质瘤、间变性室管膜瘤、间变性少突星形细胞瘤、脉络丛癌、间变性神经节胶质瘤、松果体母细胞瘤、松果体细胞瘤、脑膜瘤、髓上皮瘤、室管膜母细胞瘤、髓母细胞瘤、幕上原始神经外胚层瘤、不典型畸胎样/横纹肌样瘤、混合性胶质瘤、头颈癌、非小细胞肺癌、乳腺癌、卵巢癌、前列腺癌、髓母细胞瘤、结直肠癌、肛门癌、宫颈癌、肾癌、皮肤癌、胰腺癌、肝癌、膀胱癌、胃癌、甲状腺癌、间皮瘤、子宫癌、淋巴瘤或白血病。
在一些实施方案中,如本文所述的药物可以用于:1)抑制具有表达EGFRvIII的恶性细胞的受试者中肿瘤生长或进展;2)抑制受试者中表达EGFRvIII的恶性细胞的转移;和3)诱导具有表达EGFRvIII的恶性细胞的受试者中的肿瘤消退。
在一些实施方案中,本发明的分离细胞或来源于分离细胞的细胞系可以用于制备用于治疗有此需要的患者中的癌症的药物。
本文还提供了用于治疗患者的方法。在一些实施方案中,该方法包括对有此需要的患者提供本发明的免疫细胞。在一些实施方案中,该方法包括对有此需要的患者施用本发明的转化的免疫细胞的步骤。
在一些实施方案中,本发明的T细胞可以在经历稳健的体内T细胞扩增并且可以持续延长的一段时间。
本发明的治疗方法可以是改善、治愈或预防。本发明的方法可以是自体免疫疗法的一部分或同种异体免疫疗法治疗的一部分。本发明特别适用于同种异体免疫疗法。可以使用标准方案将来自供体的T细胞转化为非同种异体反应细胞,并根据需要再生,从而产生CAR-T细胞,其可以施用于一个或几个患者。这种CAR-T细胞疗法可以作为“现成的”治疗产品获得。
可以与所公开的方法一起使用的细胞描述于例如前一部分中。治疗可用于治疗诊断患有例如癌症的患者。可以治疗的癌症包括例如与EGFRvIII相关的癌症,包括任何上面列出的癌症。用本发明的CAR和CAR-T细胞治疗的癌症的类型包括但不限于某些液体和实体瘤,例如多形性胶质母细胞瘤、头颈癌、非小细胞肺癌、乳腺癌、卵巢癌和前列腺癌。还包括成人肿瘤/癌症和儿科肿瘤/癌症。
在一些实施方案中,治疗可以与一种或多种针对癌症的疗法组合进行,其选自抗体疗法、抗体-药物缀合物疗法、化疗、靶向疗法、细胞因子疗法、疫苗疗法、溶瘤病毒疗法、树突细胞疗法、基因疗法、纳米粒疗法、激素疗法、手术切除、激光疗法、肿瘤治疗领域和放射治疗。例如,本发明的CAR和CAR-T细胞可以结合如下手段施用于患者(例如在如下手段之前、与之同时或在其之后)1)标准护理,包括放射、手术切除、化疗(例如替莫唑胺、丙卡巴肼、卡莫司汀、洛莫司汀、长春新碱等),抗体疗法,例如贝伐珠单抗,抗-血管生成疗法,和/或肿瘤治疗领域;2)疫苗,包括EGFRvIII疫苗;3)抗体-药物缀合物疗法,包括但不限于靶向HER受体家族例如EGFR、HER2、HER3和HER4的药物缀合物;4)靶向疗法,例如激酶抑制剂(例如依维莫司);和5)免疫疗法,包括但不限于抗-PD-1、抗-PD-L1、抗-PD-L2、抗-41BB、抗-TIM3、抗-LAG3、抗-TIGIT、抗-OX40、抗-HVEM、抗-BTLA、抗-CD40、抗-CD47、抗-CSF1R、抗-CSF1、抗-MARCO、抗-IL8、抗-CXCR4和抗-CTLA4抗体。
在一些实施方案中,治疗可以施用于经受免疫抑制治疗的患者。实际上,由于编码这种免疫抑制剂的受体的基因的失活,本发明优选依赖于已经对至少一种免疫抑制剂(例如环孢菌素,硫唑嘌呤,甲氨蝶呤,霉酚酸酯和FK506)产生抗性的细胞或细胞群。在这方面,免疫抑制治疗应当有助于在患者体内选择和扩增根据本发明的T细胞。施用根据本发明细胞或细胞群可以以任何方便的方式进行,包括通过气雾剂吸入,注射,摄取,输血,植入或移植。本文所述的组合物可以皮下,皮内,肿瘤内,颅内,结内,髓内,肌内,静脉内或淋巴内注射或腹膜内施用于患者。在一个实施方案中,本发明的细胞组合物优选通过静脉内注射施用。
在一些实施方案中,治疗可以施用于经受淋巴清除方案的患者。用细胞毒性剂或全身照射消耗免疫调节元件可以增强本发明CAR和CAR-T细胞的抗肿瘤活性。例如,淋巴细胞清除方案中的细胞毒性剂包括但不限于氟达拉滨、环磷酰胺和/或阿仑珠单抗。
在一些实施方案中,施用细胞或细胞群可包括施用例如每kg体重约104-约109个细胞,包括在这些范围内的细胞数的所有整数值。在一些实施方案中,施用细胞或细胞群可以包括每kg体重施用约105-106个细胞,包括在这些范围内的细胞数的所有整数值。细胞或细胞群可以一剂或多剂施用。在一些实施方案中,所述有效量的细胞可以作为单剂量施用。在一些实施方案中,所述有效量的细胞可以在一段时间内以超过一剂施用。施用时机在处置医师的判断范围内,并取决于患者的临床状况。细胞或细胞群可以从任何来源获得,例如血库或供体。当个体需求变化时,确定特定疾病或病况的指定细胞类型的有效量的最佳范围在本领域技术范围内。有效量是指提供治疗或预防益处的量。施用剂量取决于接受者的年龄、健康状况和体重,同时治疗的种类(如果有的话),治疗频率和期望效果的性质。在一些实施方案中,通过胃肠外施用有效量的细胞或包含那些细胞的组合物。在一些实施方案中,施用可以是静脉内施用。在一些实施方案中,可以通过在肿瘤内注射直接进行施用。
试剂盒
本发明还提供了用于本发明方法的试剂盒。本发明的试剂盒包括一个或多个容器,其包含编码EGFRvIII特异性CAR的多核苷酸或包含编码如本文所述的EGFRvIII特异性CAR的多核苷酸的改造的免疫细胞和根据本文所述的任何本发明方法使用的说明书。通常,这些说明书包括对用于上述治疗性治疗的改造的免疫细胞的施用的描述。
与本文所述的改造的免疫细胞的使用相关的说明书通常包括关于预期治疗的剂量、施用方案和施用途径的信息。容器可以是单位剂量、散装包装(例如多剂量包装)或亚单位剂量。在本发明的试剂盒中提供的说明书典型地是在标签或包装插页(例如包括在试剂盒中的纸张)上的书面说明书,但机器可读的说明书(例如在磁性或光学存储盘上载有的说明书)也是可接受的。
本发明的试剂盒处于合适的包装中。合适的包装包括但不限于小瓶,瓶子,广口瓶,软包装(例如密封的Mylar或塑料袋)等。还考虑了与特殊性装置结合使用的包装,例如吸入器,鼻腔施用装置(例如雾化器)或输液装置,例如微型泵。试剂盒可以具有无菌进入口(例如,容器可以是静脉内溶液袋或具有可由皮下注射针刺穿的塞子的小瓶)。容器也可以具有无菌进入口(例如,容器可以是静脉内溶液袋或具有可由皮下注射针刺穿的塞子的小瓶)。组合物中的至少一种活性剂是EGFRvIII抗体。容器可进一步包含第二药物活性剂。
试剂盒可以任选地提供额外的成分例如缓冲液和解释性信息。通常,该试剂盒包含容器和在容器上或与容器相关的标签或包装插页。
出于所有目的通过引用并入本文的是美国临时专利申请号62/281,533(2016年1月21日提交)和62/431,758(2016年12月8日提交)的内容。
生物保藏
将本发明有代表性的材料保藏在美国典型培养物保藏中心,10801University Boulevard,Manassas,Va.20110-2209,USA,为________。具有ATCC保藏号______的载体_____为编码______轻链可变区的多肽,且具有ATCC保藏号______的载体______为编码______重链可变区的多肽。保藏根据国际公认用于专利程序和条例目的(布达佩斯条约)下微生物保藏的布达佩斯条约的规定进行。这确保了从保藏之日起维持保藏活培养物30年的可行性。保藏将由ATCC根据布达佩斯条约提供,并受Pf izer,Inc.与ATCC之间的协议约束,该协议确保相关的任何美国或外国专利申请向公众发布或在向公众公开时永久和不受限制地提供培养物的后代,以先到者为准并确保后代可供根据35U.S.C.§122由美国专利和商标专员以及与之相关的专员规则(包括37C.F.R.§1.14,特别是参照886 OG 638)授予使用的权利。
本申请的受让人已经同意,如果在合适的条件下培养时保藏的材料的培养物可能死亡或丢失或被破坏,则该材料将在通知时立即替换为另一同一材料。保藏材料的可用性不应被解释为违反任何政府根据其专利法授予的权利来实施本发明的许可。
提供以下实施例仅用于示例目的,并不意图以任何方式限制本发明的范围。实际上,除了本文所示和所述的那些之外,本发明的各种修改对于本领域技术人员来说将从前面的描述中变得显而易见,并且落入所附权利要求书的范围内。
实施例
实施例1:重组抗-EGFRvIII鼠-人嵌合抗体和人源化抗体的亲和力测定
本实施例测定嵌合和人源化抗-EGFRvIII抗体在25℃和37℃下的亲和力。
将由杂交瘤产生的抗-EGFRvIII小鼠(m)抗体m62G7测序并且亚克隆入用于作为鼠-人嵌合抗体表达的合适的载体。将小鼠抗体m62G7的CDR移植在人框架上并且表达为人IgG1重组抗体h62G7。通过在重链和轻链的CDR中导入突变制备h62G7的亲和变体。在配备研究级抗-人Fc偶联CM4传感器芯片的表面等离子体共振BiacoreTM T200生物传感器(GE Healthcare Inc.,Piscataway,NJ)上测定重组抗-EGFRvIII嵌合抗体m62G7和人源化h62G7抗体的亲和力。然后由抗-人Fc捕获抗-EGFRvIII抗体。然后将单体8-组氨酸标记的人EGFRvIII胞外结构域作为分析物以最高浓度为1000nM的10倍稀释系列进行注射。在25℃和37℃下测定抗-EGFRvIII抗体对人EGFRvIII的亲和力(表6)。这些抗体在相同测定条件下无一表现出可检测到的与1000nM 8-组氨酸标记的重组野生型蛋白EGFRwt的结合。
在表6中,参照重链变化、然后参照轻链变化描述h62G7变体。例如,抗体克隆“h62G7-EQ/L6”是指包含重链中“EQ”变化(本文也称作“h62G7-EQ”)和轻链中“L6”变化(本文也称作“h62G7-L6”)的h62G7克隆。这些重链和轻链氨基酸序列提供于表2中。此外,在本申请中,h62G7变体可以涉及首先书写为重链或轻链变体–由此例如“h62G7-EQ/L6”和“h62G7-L6/EQ”均是指包含h62G7-EQ重链和h62G7-L6轻链的抗体。
表6
实施例2:人抗-EGFRvIII抗体的亲和力测定
本实施例测定各种人抗-EGFRvIII抗体在37℃下的亲和力。
为了生成针对EGFRvIII的人抗体,用低聚甲醛-固定的表达EGFRvIII、F98-npEGFRvIII(美国典型培养物保藏中心,Manassas,VA)和定向于EGFRvIII中的连接区的肽(SEQ ID NO:227:CGSGSGLEEKKGNYVVTDH)的大鼠胶质母细胞瘤细胞系的交替方案免疫接种转基因AlivaMab小鼠(Ablexis LLC,San Francisco,CA)。使用标准技术生成杂交瘤。为了测定这些杂交瘤与EGFRvIII的结合亲和力和特异性,应用配备抗-小鼠Fc偶联CM4传感器芯片的BiacoreTM T200生物传感器(BiacoreTM AB,Uppsala,Sweden–现在的GE Healthcare)通过抗-小鼠Fc捕获培养物上清液中的抗体。然后将单体8-组氨酸标记的人EGFRvIII胞外结构域作为分析物以最高浓度1000nM的10倍稀释系列进行注射。在37℃测定抗-EGFRvIII抗体对人EGFRvIII的亲和力(表7)。这些杂交瘤抗体在相同测定条件下无一表现出可检测到的与1000nM 8-组氨酸标记的重组野生型蛋白EGFRwt的结合。
表7
实施例3:通过流式细胞术测定抗-EGFRvIII抗体与表达EGFRvIII的细胞系的结合特异性
本实施例证实抗-EGFRvIII抗体与表达EGFRvIII的细胞的细胞结合特异性。
为了评价由AlivaMab小鼠生成的抗-EGFRvIII抗体的细胞结合特异性,使用3种同基因大鼠胶质母细胞瘤细胞和人癌细胞系:F98(不表达人EGFR的任何形式)、F98-EGFRwt(表达野生型EGFR)、F98-npEGFRvIII(表达EGFRvIII)和A431(具有野生型EGFR超表达的表皮样癌细胞系),它们均得自美国典型培养物保藏中心(Manassas,VA)。对于细胞染色,将500,000个细胞与50μl杂交瘤上清液一起在4℃孵育45min,用结合缓冲液(PBS(磷酸盐缓冲盐水)+0.5%BSA(牛血清白蛋白))洗涤,然后与来自Jackson ImmunoResearch Laboratories(West Grove,PA)的FITC-缀合的山羊抗-小鼠Fc特异性二抗一起孵育。表8A和8B显示表达EGFRvIII的细胞系上EGFRvIII抗体(克隆20G5除外)的平均荧光强度(MFI)是在非表达细胞系上的平均荧光强度的至少10倍。图1A、图1B和图1C显示已经克隆且表达为重组人IgG1抗体、42G9(图1A)、32A10(图1B)和32G8(图1C)的3种EGFRvIII特异性克隆与3种F98细胞系的FACS结合直方图的实例。
表8A
表8B
这些结果证实抗-EGFRvIII抗体与表达EGFRvIII的细胞的结合特异性。
实施例4:来自噬菌体文库的全人抗-EGFRvIII抗体的亲和力测定
本实施例测定各种人抗-EGFRvIII抗体在25℃下的亲和力。
将人抗-EGFRvIII抗体测序并且亚克隆入用于作为重组人IgG1抗体表达的合适的载体。在25℃下(表9)在配备抗-人Fc偶联CM4传感器芯片的表面等离子体共振BiacoreTMT200生物传感器(GE Healthcare Inc.,Piscataway,NJ)上测定抗体亲和力。用抗-人Fc捕获抗-EGFRvIII抗体。然后将单体8-组氨酸标记的人EGFRvIII胞外结构域作为分析物以从1000nM开始的10倍稀释系列进行注射。在2种抗体中,仅C6在25℃下表现出极弱的、但可检测到的与1000nM 8-组氨酸标记的重组野生型蛋白EGFRwt的结合。
表9
实施例5:重组单链Fv形式的抗-EGFRvIII抗体的亲和力测定
本实施例测定各种重组单链Fv形式的抗-EGFRvIII抗体在37℃的亲和力。
为了将常规抗体转化成单链Fv(scFv)片段,将重链可变结构域和轻链可变结构域通过(GGGGS)4(SEQ ID NO:202)接头连接在一起。然后使scFv片段与人IgG1Fc部分融合,以有利于重组表达和纯化。在37℃在如上所述配备抗-人Fc偶联CM4传感器芯片的表面等离子体共振BiacoreTM T200生物传感器(GE Healthcare Inc.,Piscataway,NJ)上测定scFv-Fc融合蛋白的亲和力。用抗-人Fc捕获scFv-Fc蛋白。然后将单体8-组氨酸标记的人EGFRvIII胞外结构域作为分析物以从1000nM开始的10倍稀释系列进行注射。表10显示scFv重新形成的融合蛋白保持与EGFRvIII结合,且HL(在N-末端具有重链可变结构域)和LH(在N-末端具有轻链可变结构域)中scFv-Fc蛋白的亲和力媲美于表6、7和9中列出的其常规抗体对应物。还测试了这些scFv-Fc蛋白在25℃下与1000nM 8-组氨酸标记的重组野生型蛋白EGFRwt的结合,但其中无一表现出显著结合。
表10
实施例6:EGFRvIII特异性嵌合抗原受体(CAR)T细胞的产生和检测
本实施例测定EGFRvIII特异性CAR T细胞的表达和抗原结合特异性。
根据提供者的说明解冻来自由AllCells(Alameda,CA)提供的健康供体的PBMC,并且在补充5%人血清的X-VivoTM-15培养基(Lonza,Walkersville,MD)中培养过夜。将T细胞在补充了20ng/mL人IL-2、5%人血清和以珠:细胞之比1:1的Dynabead人T激活剂CD3/CD28(Life Technologies,Carlsbad,CA)的X-VivoTM-15培养基(Lonza)中活化3天。然后用在EF1α启动子控制下携带BFP-T2A-EGFRvIII特异性CAR表达盒的双反顺子慢病毒载体以5的感染复数(MOI)转导T细胞。在这种构建体中,将EGFRvIII特异性CAR与BFP(蓝色荧光蛋白)共表达,以有利于检测EGFRvIII特异性CAR。EGFRvIII特异性CAR含有不同抗-EGRRvIII克隆(h62G7-L6/EQ、14C11、20B9、32A10、42G9、C6、20E12、26B9、30D8和32G8;本文另外部分所述)的VH和VL序列。将CAR T细胞维持在培养物中转导后至多14天。通过流式细胞术,使用用于检测的BFP(蓝色荧光蛋白)随时间推移(初级T细胞慢病毒转导后第4、9和14天)监测表达EGFRvIII特异性CAR的细胞百分比(图2A)(根据BFP阳性活细胞百分比确定)。
为了测定CAR的靶标结合特异性,分别由与小鼠IgG1-Fc结构域融合的人EGFRvIII或人野生型EGFR胞外结构域构成的重组蛋白EGFRvIII-mFc和EGFR-mFc在HEK293细胞中产生。通过将不同EGFRvIII特异性CAR T细胞与EGFRvIII-mFc或EGFR-mFc蛋白一起孵育,然后与PE-标记的山羊抗-小鼠Fc二抗(Jackson ImmunoResearch)孵育测定靶标结合特异性,然后在编码包含不同EGFRvIII特异性克隆(h62G7-L6/EQ、14C11、20B9、32A10、42G9、C6、20E12、26B9、30D8和32G8)的CAR的载体转导T细胞后第4天时通过流式细胞术分析。如图2B中所示,包含h62G7-L6/EQ、14C11、20B9、32A10、42G9、20E12、26B9和30D8的VH和VL序列的克隆CAR T细胞结合EGFRvIII-mFc,但不显著结合EGFR-mFc。在转导后第14天时,测定如通过细胞上BFP检测的CAR表达和细胞的重组EGFRvIII-mFc结合的相关性。有代表性的数据集如图2C中所示,其显示转导后第14天时在CAR T细胞中包含10种不同EGFRvIII特异性克隆(h62G7-L6/EQ、14C11、20B9、32A10、42G9、C6、20E12、26B9、30D8和32G8)的CAR的表达,如细胞上BFP检测和细胞的重组EGFRvIII-mFc结合所测定的。
这些结果证实含有包含克隆h62G7-L6/EQ、14C11、20B9、32A10、42G9、20E12、26B9和30D8的VH和VL序列的CAR的CAR T细胞结合EGFRvIII-mFc,但不显著结合EGFR-mFc。
实施例7:EGFRvIII特异性CAR T细胞的靶标依赖性和非依赖性脱粒
本实施例测定EGFRvIII特异性CAR T细胞在靶标表达癌细胞系存在和不存在下的脱粒活性。
5种细胞系用于评价CAR T细胞的脱粒活性。人肺癌细胞系NCI-H522和胶质母细胞瘤细胞系U87MG得自美国典型培养物保藏中心(Manassas,VA)并且在补充了10%热灭活胎牛血清(Mediatech Inc.,Manassas,VA)的RPMI 1640培养基中培养。由于大部分癌细胞系不表达EGFRvIII,所以用编码全长EGFRvIII基因(SEQ ID NO:201)的慢病毒载体转导NCI-H522(NCI-H522不表达可检测到水平的野生型EGFR或EGFRvIII),以生成表达低水平EGFRvIII的“NCI-H522-EGFRvIII”。为了生成表达各种EGFR蛋白的同基因人胶质母细胞瘤细胞系,首先敲除U87MG的内源性野生型EGFR基因,以生成无EGFR的细胞系“U87-KO”。然后用编码全长野生型EGFR基因(登录号NP_005219)的慢病毒载体转导U87-KO细胞,以得到过表达EGFR的细胞系“U87-KO-EGFRwt”。类似地,通过用编码全长EGFRvIII基因(SEQ ID NO:201)的载体的慢病毒转导从U87-KO生成“U87-KO-EGFRvIII”(过表达EGFRvIII)。
对于脱粒测定,将表达各种EGFRvIII特异性CAR(包含克隆h62G7-L6/EQ、14C11、20B9、32A10、42G9、C6、20E12、26B9、30D8和32G8)的100,000个T-细胞在96-孔板中与表达各种水平EGFRvIII蛋白或EGFRwt蛋白的等数量癌细胞一起孵育。在37℃与5%CO2下将共培养物在终体积为100μl的X-VivoTM-15培养基(Lonza,Walkersville,MD)中培养6小时。在细胞刺激期间通过在共培养开始时添加荧光抗-CD107a抗体(Miltenyi Biotec,San Diego,CA)与终浓度为1μg/ml的抗-CD49d(BD Pharmingen,San Diego,CA)、1μg/ml的抗-CD28(Miltenyi Biotec)和1x莫能星(eBioscience,San Diego,CA)溶液进行CD107a染色。6h孵育期后,用可固定活性染料和荧光染料-缀合的抗-CD8抗体(Miltenyi Biotec)染色细胞并且通过流式细胞术分析。将脱粒活性测定为活/CD8+/BFP+/CD107a+细胞%,并且通过测定平均荧光强度信号(MFI)确定CD8+/BFP+细胞中的CD107a染色。在CAR转导后第14天时进行脱粒测定。图3显示来自两个PBMC供体的每种EGFRvIII特异性CAR的结果。包含克隆C6和32G8的CAR T细胞除外,仅当将EGFRvIII特异性CAR T细胞与表达EGFRvIII例如NCI-H522-EGFRvIII和U87-KO-EGFRvIII的细胞系一起共培养时,观察到脱粒活性增加(相对于单独的CAR-T)。
这些结果证明在表达EGFRvIII的肿瘤细胞存在下EGFRvIII特异性CAR T细胞表现出脱粒活性,但在不表达EGFRvIII的肿瘤细胞存在下不表现出脱粒活性。
实施例8:EGFRvIII特异性CART细胞的干扰素γ(IFNγ)分泌
本实施例显示在EGFRvIII特异性CAR T细胞与表达靶蛋白的细胞一起共培养时EGFRvIII特异性CAR T细胞的IFNγ分泌水平。
将表达含有不同EGFRvIII特异性克隆(h62G7-L6/EQ、14C11、20B9、32A10、42G9、C6、20E12、26B9、30D8和32G8)的CAR的EGFRvIII特异性CAR T-细胞在96-孔板(100,000细胞/孔)中与表达各种水平的EGFRvIII或EGFRwt蛋白的等数量的细胞(细胞NCI-H522、NCI-H522-EGFRvIII、U87-KO、U87-KO-EGFRwt和U87-KO-EGFRvIII;各自如上述实施例7中所述)一起孵育。在37℃与5%CO2下将共培养物在终体积为100μl的X-VivoTM-15培养基(Lonza)中培养18小时。然后采集上清液并且冷冻。用人IFN-γQuantikine ELISA试剂盒(R&D system,Minneapolis,MN),根据制造商的说明测定上清液中的IFNγ。如图4中所示,对于大部分CAR T细胞,IFNγ分泌水平与共培养细胞表达的EGFRvIII的量相关:低表达子NCI-H522-EGFRvIII诱导少量的IFNγ,高表达子U87-KO-EGFRvIII诱导大量IFNγ。表达EGFRvIII特异性克隆32G8CAR的T细胞在所有测试条件下都不分泌显著水平的IFNγ。另一方面,EGFRvIII特异性克隆C6CAR T细胞在与U87-KO-EGFRwt或U87-KO-EGFRvIII细胞共培养时分泌高水平的IFNγ。
这些结果证实对于大部分EGFRvIII特异性CAR T细胞,IFNγ分泌水平与共培养细胞表达的EGFRvIII的量相关。
实施例9:EGFRvIII特异性CAR T细胞的细胞毒性
本实施例测定EGFRvIII特异性CAR T细胞在与表达靶蛋白的细胞系一起共培养时EGFRvIII特异性CAR T细胞的细胞毒性。
将表达含有不同EGFRvIII特异性克隆(h62G7-L6/EQ、14C11、20B9、32A10、42G9、C6、20E12、26B9、30D8和32G8)的CAR的EGFRvIII特异性CAR T-细胞与表达各种水平的EGFRvIII蛋白的20,000个靶细胞一起接种在96-孔板上(400,000细胞/孔)。靶细胞为:上述U87-KO-EGFRwt、NCI-H522-EGFRvIII和U87-KO-EGFRvIII。将靶(EGFRvIII阳性:NCI-H522-EGFRvIII和U87-KO-EGFRvIII)和对照(EGFRvIII阴性:U87-KO-EGFRwt)细胞铺板并且用荧光胞内染料CFSE标记,然后将它们与EGFRvIII特异性CAR T-细胞一起共培养。将共培养物在37℃与5%CO2下孵育4小时。该孵育期后,用可固定活性染料标记细胞并且通过流式细胞术分析。测定每种细胞群(EGFRvIII阳性靶细胞或EGFRvIII阴性对照细胞)的活力,并且计算特异性细胞裂解%。T细胞的CAR转导后14天进行细胞毒性测定。全部EGFRvIII特异性CAR T细胞(32G8除外)能够裂解表达低水平靶标的细胞(NCI-H522-EGFRvIII)和表达高水平靶标的细胞(U87-KO-EGFRvIII)至各种程度。此外,EGFRvIII特异性克隆C6CAR T细胞裂解表达野生型EGFR和EGFRvIII的细胞(图5)。
这些结果证实EGFRvIII-特异性CAR T细胞有效地杀伤表达EGFRvIII的细胞。
实施例10:EGFRvIII特异性CART细胞在U87-KO-EGFRvIII模型中的体内研究
本实施例显示EGFRvIII特异性CAR T细胞在皮下U87-KO-EGFRvIII GBM异种移植物(xenograph)模型中的抗肿瘤活性。
将3百万个U87-KO-EGFRvIII GBM肿瘤细胞(上述)皮下植入5-6周龄NSG小鼠(Jackson Laboratory,Sacramento,CA)。通过测径器装置每周1次测量肿瘤体积并且使用如下公式计算:肿瘤体积=(长x宽2)/2。在肿瘤植入后第8天时,将动物根据肿瘤大小随机分成5只动物/组并且通过尾静脉推注施用单剂量的6百万个CAR阳性EGFRvIII特异性CAR T细胞(表达EGFRvIII特异性克隆14C11、32A10或26B9;该克隆如本文另外部分所述)或等同总数的未转导T细胞。图6显示14C11、32A10和26B9EGFRvIII特异性CAR T细胞、但不是未转导的T细胞抑制体内U87-KO-EGFRvIII异种移植物肿瘤生长。
这些结果证实EGFRvIII-特异性CAR T细胞有效地抑制体内表达EGFRvIII的细胞的肿瘤生长。
实施例11:含有R2自杀序列的EGFRvIII特异性CAR的表面表达
本实施例显示含有R2自杀序列的EGFRvIII特异性CAR在T细胞中的表达。
R2自杀序列是基于CD20表位的自杀序列并且含有利妥昔单抗识别表位的两个串联拷贝。R2序列插入在本文另外部分所述各种EGFRvIII特异性CAR(14C11、32A10、30D8和26B9)的scFv与CD8α铰链序列之间以生成不同EGFRvIII-R2 CAR(本文分别称作“14C11-R2”、“32A10-R2”、“30D8-R2”和“26B9-R2”)。T细胞被在EF1α启动子控制下带有不同EGFRvIII-R2 CAR表达盒的慢病毒载体以5或25的感染复数(MOI)转导(对于克隆26B9-R2和30D8-R2)。将CAR T细胞维持在培养物中转导后至多14或15天。采用针对所述细胞的生物素化重组蛋白:EGFRvIII-mFc或利妥昔单抗(缀合有EZ-LinkTM Sulfo-NHS-SS-生物素;ThermoFisher,Waltham,MA)接着PE缀合的链霉抗生物素蛋白(BD Biosciences,San Diego,CA)通过流式细胞术随时间推移(T细胞转导后第4天、第9/10天和第14/15天)监测CAR表达。如使用生物素化EGFRvIII-mFc和生物素化利妥昔单抗测得CAR阳性细胞百分比分别如图7和8中所示。将与未转导T细胞(NTD)的结合提供为对照。
这些结果证实包含R2自杀序列的EGFRvIII-特异性CAR的T细胞中的表达。
实施例12:EGFRvIII-特异性R2 CAR T细胞的细胞毒性
本实施例显示EGFRvIII-特异性R2 CAR T细胞在与表达靶标的细胞一起共培养时的细胞毒性。
细胞毒性测定如实施例9中所述,使用实施例11中所述EGFRvIII-R2 CAR 14C11-R2、32A10-R2、30D8-R2和26B9-R2进行。所测试的全部EGFRvIII特异性R2 CAR T细胞能够裂解表达低水平靶标的细胞(NCI-H522-EGFRvIII)和表达高水平靶标的细胞(U87-KO-EGFRvIII)至各种程度(图9)。
这些结果证实EGFRvIII-特异性R2 CAR T细胞有效地杀伤表达EGFRvIII的细胞。
尽管已经参考各种应用、方法、试剂盒和组合物描述了所公开的教导,但是应当理解,在不脱离本文的教导和所要求保护的发明的情况下,可以进行各种改变和修改。提供前述实施例是为了更好地示例所公开的教导,并且不旨在限制本文给出的教导的范围。虽然已经根据这些示例性实施例描述了本教导,但是本领域技术人员将容易理解,这些示例性实施方案的许多变化和修改是可能的而无需过多的实验。所有这些变化和修改都在当前教导的范围内。
本文引用的所有参考文献,包括专利、专利申请、论文、教科书等,以及其中引用的参考文献,就尚未描述的而言,通过引用整体并入本文。如果所并入的文献和类似材料中的一个或多个与本申请不同或矛盾,包括但不限于定义的术语、术语用法、所描述的技术等,则以本申请为准。
前述说明书和实施例详述了本发明的某些具体实施方案,并描述了发明人考虑的最佳方式。然而,应当理解,无论前述说明书如何详细地出现在文本中,本发明都可以以许多种方式实施,并且本发明应当根据所附权利要求书及其任何等效方案来解释。
序列表
<110> 辉瑞大药厂
<120> 靶向表皮生长因子受体变体III的嵌合抗原受体
<130> PC72270Prov2
<160> 256
<170> PatentIn version 3.5
<210> 1
<211> 115
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 1
Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Thr Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Thr Leu His Trp Val Lys Gln Ser His Val Lys Ser Leu Glu Trp Ile
35 40 45
Gly Gly Ile Asp Pro Ile Asn Gly Gly Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Glu Ala Met Asp Ser Trp Gly Gln Gly Thr Ser Val Thr
100 105 110
Val Ser Ser
115
<210> 2
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 2
Asp Val Val Met Thr Gln Thr Pro Leu Thr Leu Ser Val Thr Ile Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Gly Lys Thr Tyr Leu Asn Trp Leu Leu Gln Arg Pro Gly Gln Ser
35 40 45
Pro Lys Arg Leu Ile Tyr Leu Val Ser Lys Leu Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Phe Tyr Tyr Cys Val Gln Asp
85 90 95
Thr His Phe Pro Leu Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
100 105 110
<210> 3
<211> 115
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 3
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Thr Leu His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Asn Pro Ile Asn Gly Gly Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Glu Ala Met Asp Ser Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 4
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 4
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Gly Lys Thr Tyr Leu Asn Trp Phe Gln Gln Arg Pro Gly Gln Ser
35 40 45
Pro Arg Arg Leu Ile Tyr Leu Val Ser Lys Leu Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Val Gln Asp
85 90 95
Thr His Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 5
<211> 115
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 5
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Thr Leu His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Trp Pro Ile Thr Gly Gly Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Glu Ala Gln Gly Ser Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 6
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 6
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Gly Lys Thr Tyr Leu Asn Trp Phe Gln Gln Arg Pro Gly Gln Ser
35 40 45
Pro Arg Arg Leu Ile Tyr Gln Val Ser Lys Leu Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Gly Gln Asp
85 90 95
Thr His Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 7
<211> 115
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 7
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Thr Leu His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Trp Pro Ile Thr Gly Gly Thr Thr Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Glu Ala Glu Gly Ser Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser
115
<210> 8
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 8
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Asp Lys Thr Tyr Thr Asn Trp Phe Gln Gln Arg Pro Gly Gln Ser
35 40 45
Pro Arg Arg Leu Ile Tyr Glu Val Ser Lys Leu Asp Val Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Gly Gln Asp
85 90 95
Thr His Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
<210> 9
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 9
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Leu Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Pro
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Phe Ala His Ile Phe Ser Thr Asp Glu Lys Ser Leu Lys Leu Ser
50 55 60
Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Ala Pro Val Asp Ser Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Phe Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 10
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 10
Glu Val Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Arg Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Ser Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ser Thr Ile Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Ser Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 11
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 11
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Ala
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Phe Ser Thr Asp Glu Lys Ser Ile Arg Arg Ser
50 55 60
Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Phe
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 12
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 12
Glu Val Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Val Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Asn
20 25 30
Phe Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ala Pro Arg Leu Leu Leu
35 40 45
Tyr Gly Ala Thr Thr Arg Ala Thr Gly Leu Pro Gly Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Asn Ile Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Ile Tyr Phe Cys Gln Gln Tyr Lys Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Ser Lys Val Asp Ile Lys
100 105
<210> 13
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 13
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Ala
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Gly His Ile Phe Ser Thr Asp Glu Lys Ser Tyr Ser Thr Ser
50 55 60
Leu Arg Gly Arg Ile Thr Ile Ser Lys Asp Thr Ser Arg Gly Leu Val
65 70 75 80
Val Leu Thr Leu Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Phe Trp Gly
100 105 110
Pro Gly Phe Leu Val Thr Val Ser Ser
115 120
<210> 14
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 14
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Val Ser Gln Ser Ile Gly Ala Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Phe Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Val Arg Phe Ser Gly
50 55 60
Gly Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Ile Tyr Ser Cys Gln Gln Tyr Ile Tyr Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Thr Val Asp Ile Lys
100 105
<210> 15
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 15
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Asn Asn Ala
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Phe Ala His Ile Phe Ser Thr Asp Glu Lys Ser Phe Arg Thr Ser
50 55 60
Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Ile Leu Val Thr Val Ser Ser
115 120
<210> 16
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 16
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Asn Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly
50 55 60
Ser Asp Ser Gly Thr Glu Phe Ser Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Tyr Lys Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Glu Ile Lys
100 105
<210> 17
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 17
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Val
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Phe Ala His Ile Phe Ser Ser Asp Glu Lys Ser Ile Arg Arg Ser
50 55 60
Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Phe Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Asn
115 120
<210> 18
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 18
Asp Met Val Val Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Gly Ser Asp
20 25 30
Leu Ala Trp Tyr Gln Gln Pro Pro Gly Gln Ser Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Thr Gly Val Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Thr Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Asn Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 19
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 19
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Val
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Phe Ala His Ile Phe Ser Ser Asp Glu Lys Ser Ile Arg Arg Ser
50 55 60
Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 20
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 20
Glu Met Glu Val Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Asn Ile Gly Ser Asp
20 25 30
Leu Ala Trp Tyr Gln Gln Gln Ser Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Ser Gly Ala Ser Thr Arg Ala Thr Gly Val Pro Thr Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Thr Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Asn Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 21
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 21
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Ala
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Phe Ser Thr Asp Glu Lys Ser Ile Arg Arg Ser
50 55 60
Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Ile Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 22
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 22
Glu Val Val Met Thr Gln Ser Pro Pro Asn Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Thr Ser Asn
20 25 30
Phe Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ser Pro Arg Leu Leu Leu
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Thr Gly Val Pro Gly Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Asn Ile Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Tyr Lys Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Ser Lys Val Asp Ile Lys
100 105
<210> 23
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 23
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Ala
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Phe Ser Thr Asp Glu Lys Ser Ile Arg Arg Ser
50 55 60
Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 24
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 24
Glu Val Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Gly Val Ser Ser Asn
20 25 30
Phe Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ser Pro Arg Leu Leu Leu
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Thr Gly Val Pro Gly Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Asn Ile Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Ile Tyr Phe Cys Gln Gln Tyr Lys Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Ser Lys Val Asp Ile Lys
100 105
<210> 25
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 25
Gln Val Thr Leu Glu Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Ala
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Pro Glu
35 40 45
Trp Phe Ala His Ile Phe Ser Thr Asp Glu Lys Ser Leu Arg Leu Ser
50 55 60
Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 26
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 26
Glu Val Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Asn Arg Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Phe Gly Thr Ser Thr Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Asp Ser Leu Gln Ser
65 70 75 80
Glu His Ser Gly Leu Tyr Tyr Cys Gln Gln Tyr Asn Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 27
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 27
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Ala
20 25 30
Lys Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Ala His Ile Phe Ser Thr Asp Glu Lys Ser Ile Arg Arg Ser
50 55 60
Leu Arg Ser Arg Leu Thr Met Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Val Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 28
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 28
Glu Val Leu Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Thr Asn
20 25 30
Phe Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ala Pro Arg Leu Leu Leu
35 40 45
Phe Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Gly Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Asn Ile Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Ile Tyr Phe Cys Gln Gln Tyr Lys Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Ser Lys Val Glu Ile Lys
100 105
<210> 29
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 29
Asp Val Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Asn Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Asn Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Phe Gly Ser Ser Thr Arg Ala Thr Gly Ile Pro Ala Ser Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Asn Ser Leu Gln Ser
65 70 75 80
Glu His Ser Ala Val Tyr Tyr Cys Gln Gln Tyr Asn Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 30
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 30
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Pro
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Gly His Ile Phe Ser Ser Asp Glu Lys Ser Tyr Arg Leu Ser
50 55 60
Leu Arg Ser Arg Leu Ser Ile Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Val Arg Asp Ser Ser Asn Tyr Gly Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 31
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 31
Glu Val Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ile Asn Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Thr Ser Thr Arg Ala Thr Asp Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Asp Tyr Asn Asn Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 32
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 32
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Ile Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Cys Gly Phe Ser Leu Ser Asn Pro
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Leu Gly His Ile Phe Ser Ser Asp Glu Lys Ser Tyr Arg Leu Phe
50 55 60
Leu Arg Ser Arg Leu Ser Ile Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Met Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asp Tyr Glu Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 33
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 33
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Thr Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Gly Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Thr Arg Ala Ser Gly Val Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Ser Cys Gln Glu Tyr Asn Asn Trp Pro Phe
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 34
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<220>
<221> misc_feature
<222> (83)..(83)
<223> Xaa can be any naturally occurring amino acid
<400> 34
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Phe Gly Phe Ser Leu Ser Asn Pro
20 25 30
Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro Gly Lys Ala Pro Glu
35 40 45
Trp Leu Gly His Ile Phe Ser Ser Asp Glu Lys Ser Tyr Arg Leu Ser
50 55 60
Leu Arg Ser Arg Leu Ser Ile Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Phe Xaa Met Thr Asn Met Asp Pro Gly Asp Pro Ala Thr Tyr Tyr
85 90 95
Cys Val Arg Asp Ser Ser Asn Tyr Glu Glu Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 35
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 35
Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu Val Lys Pro Thr Glu
1 5 10 15
Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Asn Pro
20 25 30
Arg Met Gly Val Ser Trp Leu Arg Gln Pro Pro Gly Lys Ala Leu Glu
35 40 45
Trp Phe Ala His Ile Phe Ser Thr Asp Glu Lys Ser Tyr Ser Pro Ser
50 55 60
Leu Arg Gly Arg Leu Thr Val Ser Lys Asp Thr Ser Lys Ser Gln Val
65 70 75 80
Val Leu Thr Leu Thr Asn Met Asp Pro Val Asp Thr Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 36
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 36
Lys Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Asn Gln Ile Val Ser Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Val
35 40 45
Phe Gly Thr Ser Thr Arg Ala Thr Gly Ile Pro Ile Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Val Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Val Cys Gln Gln Tyr Asn Asp Trp Pro Phe
85 90 95
Thr Phe Gly Pro Gly Thr Lys Val Asp Ile Lys
100 105
<210> 37
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 37
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe Thr Phe Ser Asp Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Val Val
50 55 60
Pro Leu Asn Gly Arg Phe Ile Ile Ser Arg Asp Asp Ser Arg Asn Thr
65 70 75 80
Leu Tyr Leu Gln Leu Asn Asn Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Val Pro Gly Ser Tyr Gly Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 38
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 38
Asp Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Asn
20 25 30
Lys Arg Asn Asn Tyr Leu Asp Trp Phe Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Ala Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Gly Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Gln Gln Thr Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 39
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 39
Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe Thr Phe Ser Tyr Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Ile Ala Asp Gly Gly Ala Thr Asp Tyr Ala Ala
50 55 60
Pro Val Arg Asn Arg Phe Thr Ile Ser Arg Asp Asp Ser Arg Asn Thr
65 70 75 80
Leu Tyr Leu Glu Met His Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Ile Pro Gly Asn Asp Ala Phe Asp Met Trp Gly Gln
100 105 110
Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 40
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 40
Asp Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Ser Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Gly Lys Asn Tyr Leu Asp Trp Phe Leu His Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Ile Asp Phe Ile Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Gln Gln Thr Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 41
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 41
Glu Val Gln Leu Val Glu Ser Trp Gly Val Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ile Phe Asn Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Arg Ile Lys Ser Lys Ser Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asp Thr
65 70 75 80
Leu Tyr Leu Gln Met Asn Gly Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Phe Cys Thr Thr Ala Pro Gly Gly Pro Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 42
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 42
Asp Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Arg
20 25 30
Asp Gly Phe Asn Tyr Leu Asp Trp Phe Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Ala Ser Ser Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Asp Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 43
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 43
Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe Thr Phe Ser Tyr Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Ile Thr Asp Gly Gly Val Ile Asp Tyr Ala Ala
50 55 60
Pro Val Arg Asn Arg Cys Thr Ile Ser Arg Asp Asp Ser Arg Asn Thr
65 70 75 80
Leu Tyr Leu Glu Met His Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Ile Pro Gly Asn Asp Asp Phe Asp Met Trp Gly Gln
100 105 110
Gly Arg Met Val Thr Val Ser Ser
115 120
<210> 44
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 44
Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe Thr Phe Ser Tyr Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Ile Asn Asp Gly Gly Ala Thr Asp Tyr Ala Ser
50 55 60
Pro Val Arg Asn Arg Phe Thr Ile Ser Arg Asp Asp Ser Arg Asn Met
65 70 75 80
Leu Tyr Leu Glu Met His Ser Leu Lys Thr Glu Asp Thr Ala Val Tyr
85 90 95
Tyr Cys Thr Thr Ile Pro Gly Asn Asp Ala Phe Asp Met Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 45
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 45
Asp Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Ser Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Thr Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Gly Lys Asn Tyr Leu Asp Trp Phe Leu His Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Phe Leu Gly Ser Ile Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Ile Asp Phe Ile Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Gln Gln Thr Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 46
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 46
Glu Val Gln Leu Val Glu Ser Gly Gly Asp Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Thr Asn Ala
20 25 30
Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Arg Ile Lys Ser Lys Ile Asp Gly Gly Thr Thr Asp Tyr Ala Ala
50 55 60
Pro Val Lys Gly Arg Phe Ile Ile Ser Arg Asp Asp Ser Lys Asn Thr
65 70 75 80
Leu Ser Leu Gln Met Asn Ser Leu Lys Thr Glu Asp Thr Ala Met Tyr
85 90 95
Tyr Cys Thr Thr Ala Pro Gly Gly Pro Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Ser Leu Val Thr Val Ser Ser
115
<210> 47
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 47
Asp Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asp Arg Arg Asn Tyr Leu Asp Trp Phe Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro His Leu Leu Ile Tyr Leu Gly Ser Tyr Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Ile Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
<210> 48
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 48
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Asp Thr Phe Ser Ser Asn
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Ile Pro Ile Phe Gly Thr Ala Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg His Thr Tyr His Glu Tyr Ala Gly Gly Tyr Tyr Gly Gly Ala
100 105 110
Met Asp Pro Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120 125
<210> 49
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 49
Glu Leu Gln Ser Val Leu Thr Gln Pro Pro Ser Ala Ser Gly Thr Pro
1 5 10 15
Gly Gln Arg Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly
20 25 30
Ser Asn Tyr Val Tyr Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys
35 40 45
Ile Leu Ile Tyr Arg Asn Asn Gln Arg Pro Ser Gly Val Pro Asp Arg
50 55 60
Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Ser Gly
65 70 75 80
Leu Arg Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ala Ala Trp Asp Asp
85 90 95
Asn Leu Ser Gly Trp Val Phe Gly Thr Gly Thr Lys Leu Thr Val Leu
100 105 110
<210> 50
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 50
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Asp Ile Ser Gly Gly Gly Gly Arg Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ala Gly Leu Leu Tyr Gly Gly Gly Val Tyr Pro Met Asp Ile
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 51
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 51
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 52
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 52
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu
20 25 30
Pro Val Thr Leu Gly Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln
35 40 45
Ser Leu Leu Tyr Ser Asn Gly Lys Thr Tyr Leu Asn Trp Phe Gln Gln
50 55 60
Arg Pro Gly Gln Ser Pro Arg Arg Leu Ile Tyr Gln Val Ser Lys Leu
65 70 75 80
Asp Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp
85 90 95
Phe Thr Leu Lys Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr
100 105 110
Tyr Cys Gly Gln Asp Thr His Phe Pro Leu Thr Phe Gly Gly Gly Thr
115 120 125
Lys Val Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Gln Ser
145 150 155 160
Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val Ser Cys Lys
165 170 175
Ala Ser Gly Tyr Thr Phe Thr Asp Tyr Thr Leu His Trp Val Arg Gln
180 185 190
Ala Pro Gly Gln Gly Leu Glu Trp Met Gly Gly Ile Trp Pro Ile Thr
195 200 205
Gly Gly Thr Thr Tyr Asn Gln Lys Phe Lys Gly Arg Val Thr Met Thr
210 215 220
Arg Asp Thr Ser Thr Ser Thr Val Tyr Met Glu Leu Ser Ser Leu Arg
225 230 235 240
Ser Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Gly Glu Ala Gln
245 250 255
<210> 53
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 53
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu
20 25 30
Leu Lys Pro Thr Glu Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe
35 40 45
Ser Leu Ser Asn Pro Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro
50 55 60
Gly Lys Ala Leu Glu Trp Phe Ala His Ile Phe Ser Thr Asp Glu Lys
65 70 75 80
Ser Leu Lys Leu Ser Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr
85 90 95
Ser Lys Ser Gln Val Val Leu Thr Met Thr Asn Met Ala Pro Val Asp
100 105 110
Ser Ala Thr Tyr Tyr Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr
115 120 125
Phe Asp Phe Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Glu Val Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Val Ser
165 170 175
Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Arg
180 185 190
Ser Asn Leu Ala Trp Tyr Gln Gln Lys Ser Gly Gln Ala Pro Arg Leu
195 200 205
Leu Ile Tyr Gly Ser Thr Ile Arg Ala Thr Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Ser Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Ser Asp
245 250 255
<210> 54
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 54
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu
20 25 30
Val Lys Pro Thr Glu Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe
35 40 45
Ser Leu Ser Asn Ala Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro
50 55 60
Gly Lys Ala Leu Glu Trp Leu Ala His Ile Phe Ser Thr Asp Glu Lys
65 70 75 80
Ser Ile Arg Arg Ser Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr
85 90 95
Ser Lys Ser Gln Val Val Leu Thr Met Thr Asn Met Asp Pro Val Asp
100 105 110
Thr Ala Thr Tyr Phe Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Glu Val Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser
165 170 175
Pro Gly Glu Arg Val Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser
180 185 190
Ser Asn Phe Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ala Pro Arg Leu
195 200 205
Leu Leu Tyr Gly Ala Thr Thr Arg Ala Thr Gly Leu Pro Gly Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Glu Asn Ile Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Ser Glu Asp Phe Ala Ile Tyr Phe Cys Gln Gln Tyr Lys Asp
245 250 255
<210> 55
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 55
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu
20 25 30
Val Lys Pro Thr Glu Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe
35 40 45
Ser Leu Ser Asn Ala Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro
50 55 60
Gly Lys Ala Leu Glu Trp Leu Gly His Ile Phe Ser Thr Asp Glu Lys
65 70 75 80
Ser Tyr Ser Thr Ser Leu Arg Gly Arg Ile Thr Ile Ser Lys Asp Thr
85 90 95
Ser Arg Gly Leu Val Val Leu Thr Leu Thr Asn Met Asp Pro Val Asp
100 105 110
Thr Ala Thr Tyr Tyr Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr
115 120 125
Phe Asp Phe Trp Gly Pro Gly Phe Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser
165 170 175
Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Val Ser Gln Ser Ile Gly
180 185 190
Ala Asn Leu Ala Trp Tyr Gln Gln Lys Phe Gly Gln Ala Pro Arg Leu
195 200 205
Leu Ile Tyr Gly Ala Ser Thr Arg Ala Thr Gly Ile Pro Val Arg Phe
210 215 220
Ser Gly Gly Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Ser Glu Asp Phe Ala Ile Tyr Ser Cys Gln Gln Tyr Ile Tyr
245 250 255
<210> 56
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 56
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu
20 25 30
Val Lys Pro Thr Glu Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe
35 40 45
Ser Leu Asn Asn Ala Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro
50 55 60
Gly Lys Ala Leu Glu Trp Phe Ala His Ile Phe Ser Thr Asp Glu Lys
65 70 75 80
Ser Phe Arg Thr Ser Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr
85 90 95
Ser Lys Ser Gln Val Val Leu Thr Met Thr Asn Met Asp Pro Val Asp
100 105 110
Thr Ala Thr Tyr Tyr Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Ile Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser
165 170 175
Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser
180 185 190
Asn Asn Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu
195 200 205
Leu Ile Tyr Gly Ala Ser Thr Arg Ala Thr Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Asp Ser Gly Thr Glu Phe Ser Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Ser Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Tyr Lys Asp
245 250 255
<210> 57
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 57
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu
20 25 30
Val Lys Pro Gly Gly Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe
35 40 45
Thr Phe Ser Tyr Ala Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Gly Arg Ile Lys Ser Ile Ala Asp Gly Gly Ala
65 70 75 80
Thr Asp Tyr Ala Ala Pro Val Arg Asn Arg Phe Thr Ile Ser Arg Asp
85 90 95
Asp Ser Arg Asn Thr Leu Tyr Leu Glu Met His Ser Leu Lys Thr Glu
100 105 110
Asp Thr Ala Val Tyr Tyr Cys Thr Thr Ile Pro Gly Asn Asp Ala Phe
115 120 125
Asp Met Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Ser Val Thr Pro
165 170 175
Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr
180 185 190
Ser Asn Gly Lys Asn Tyr Leu Asp Trp Phe Leu His Lys Pro Gly Gln
195 200 205
Ser Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val
210 215 220
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Ile Asp Phe Ile Leu Lys
225 230 235 240
Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met
245 250 255
<210> 58
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 58
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Asn Leu Val Glu Ser Gly Gly Gly Leu
20 25 30
Val Lys Pro Gly Gly Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe
35 40 45
Thr Phe Ser Tyr Ala Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Gly Arg Ile Lys Ser Ile Thr Asp Gly Gly Val
65 70 75 80
Ile Asp Tyr Ala Ala Pro Val Arg Asn Arg Cys Thr Ile Ser Arg Asp
85 90 95
Asp Ser Arg Asn Thr Leu Tyr Leu Glu Met His Ser Leu Lys Thr Glu
100 105 110
Asp Thr Ala Val Tyr Tyr Cys Thr Thr Ile Pro Gly Asn Asp Asp Phe
115 120 125
Asp Met Trp Gly Gln Gly Arg Met Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Ser Val Thr Pro
165 170 175
Gly Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr
180 185 190
Ser Asn Gly Lys Asn Tyr Leu Asp Trp Phe Leu His Lys Pro Gly Gln
195 200 205
Ser Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val
210 215 220
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Ile Asp Phe Ile Leu Lys
225 230 235 240
Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met
245 250 255
<210> 59
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 59
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Val Glu Ser Trp Gly Val Leu
20 25 30
Val Lys Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Ile Phe Asn Asn Ala Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Ile Gly Arg Ile Lys Ser Lys Ser Asp Gly Gly Thr
65 70 75 80
Thr Asp Tyr Ala Ala Pro Val Lys Asp Arg Phe Thr Ile Ser Arg Asp
85 90 95
Asp Ser Lys Asp Thr Leu Tyr Leu Gln Met Asn Gly Leu Lys Thr Glu
100 105 110
Asp Thr Ala Val Tyr Phe Cys Thr Thr Ala Pro Gly Gly Pro Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
145 150 155 160
Asp Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
165 170 175
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Arg
180 185 190
Asp Gly Phe Asn Tyr Leu Asp Trp Phe Leu Gln Lys Pro Gly Gln Ser
195 200 205
Pro Gln Leu Leu Ile Tyr Leu Ala Ser Ser Arg Ala Ser Gly Val Pro
210 215 220
Asp Arg Phe Ser Gly Ser Asp Ser Gly Thr Asp Phe Thr Leu Lys Ile
225 230 235 240
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln
245 250 255
<210> 60
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 60
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
20 25 30
Val Lys Pro Gly Gly Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe
35 40 45
Thr Phe Ser Asp Ala Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr
65 70 75 80
Thr Asp Tyr Val Val Pro Leu Asn Gly Arg Phe Ile Ile Ser Arg Asp
85 90 95
Asp Ser Arg Asn Thr Leu Tyr Leu Gln Leu Asn Asn Leu Lys Thr Glu
100 105 110
Asp Thr Ala Val Tyr Tyr Cys Thr Thr Val Pro Gly Ser Tyr Gly Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp
145 150 155 160
Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly Glu
165 170 175
Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Asn Lys
180 185 190
Arg Asn Asn Tyr Leu Asp Trp Phe Leu Gln Lys Pro Gly Gln Ser Pro
195 200 205
Gln Leu Leu Ile Tyr Leu Ala Ser Asn Arg Ala Ser Gly Val Pro Asp
210 215 220
Arg Phe Ser Gly Gly Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser
225 230 235 240
Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
245 250 255
<210> 61
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 61
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val
20 25 30
Lys Lys Pro Gly Ser Ser Val Lys Val Ser Cys Lys Ala Ser Gly Asp
35 40 45
Thr Phe Ser Ser Asn Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln
50 55 60
Gly Leu Glu Trp Met Gly Val Ile Ile Pro Ile Phe Gly Thr Ala Asp
65 70 75 80
Tyr Ala Gln Lys Phe Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser
85 90 95
Thr Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr
100 105 110
Ala Val Tyr Tyr Cys Ala Arg His Thr Tyr His Glu Tyr Ala Gly Gly
115 120 125
Tyr Tyr Gly Gly Ala Met Asp Pro Trp Gly Gln Gly Thr Leu Val Thr
130 135 140
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Gly Gly Gly Gly Ser Glu Leu Gln Ser Val Leu Thr Gln Pro
165 170 175
Pro Ser Ala Ser Gly Thr Pro Gly Gln Arg Val Thr Ile Ser Cys Ser
180 185 190
Gly Ser Ser Ser Asn Ile Gly Ser Asn Tyr Val Tyr Trp Tyr Gln Gln
195 200 205
Leu Pro Gly Thr Ala Pro Lys Ile Leu Ile Tyr Arg Asn Asn Gln Arg
210 215 220
Pro Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser
225 230 235 240
Ala Ser Leu Ala Ile Ser Gly Leu Arg Ser Glu Asp Glu Ala Asp
245 250 255
<210> 62
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 62
Thr Asp Tyr Thr Leu His
1 5
<210> 63
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 63
Gly Tyr Thr Phe Thr Asp
1 5
<210> 64
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 64
Gly Tyr Thr Phe Thr Asp Tyr Thr Leu His
1 5 10
<210> 65
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 65
Gly Ile Asp Pro Ile Asn Gly Gly Thr Thr Tyr Asn Gln Lys Phe Lys
1 5 10 15
Gly
<210> 66
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 66
Gly Ile Asp Pro Ile Asn Gly Gly Thr Thr Tyr
1 5 10
<210> 67
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 67
Gly Glu Ala Met Asp Ser
1 5
<210> 68
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 68
Gly Ile Asn Pro Ile Asn Gly Gly Thr Thr Tyr Asn Gln Lys Phe Lys
1 5 10 15
Gly
<210> 69
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 69
Gly Ile Asn Pro Ile Asn Gly Gly Thr Thr Tyr
1 5 10
<210> 70
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 70
Gly Ile Trp Pro Ile Thr Gly Gly Thr Thr Tyr Asn Gln Lys Phe Lys
1 5 10 15
Gly
<210> 71
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 71
Gly Ile Trp Pro Ile Thr Gly Gly Thr Thr Tyr
1 5 10
<210> 72
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 72
Gly Glu Ala Glu Gly Ser
1 5
<210> 73
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 73
Gly Glu Ala Gln Gly Ser
1 5
<210> 74
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 74
Ser Asn Pro Arg Met Gly Val Ser
1 5
<210> 75
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 75
Gly Phe Ser Leu Ser Asn Pro Arg
1 5
<210> 76
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 76
Gly Phe Ser Leu Ser Asn Pro Arg Met Gly Val Ser
1 5 10
<210> 77
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 77
His Ile Phe Ser Thr Asp Glu Lys Ser Leu Lys Leu Ser Leu Arg Ser
1 5 10 15
<210> 78
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 78
His Ile Phe Ser Thr Asp Glu Lys Ser Leu
1 5 10
<210> 79
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 79
Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Phe
1 5 10
<210> 80
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 80
Ser Asn Ala Arg Met Gly Val Ser
1 5
<210> 81
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 81
Gly Phe Ser Leu Ser Asn Ala Arg
1 5
<210> 82
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 82
Gly Phe Ser Leu Ser Asn Ala Arg Met Gly Val Ser
1 5 10
<210> 83
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 83
His Ile Phe Ser Thr Asp Glu Lys Ser Ile Arg Arg Ser Leu Arg Ser
1 5 10 15
<210> 84
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 84
His Ile Phe Ser Thr Asp Glu Lys Ser Ile
1 5 10
<210> 85
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 85
Asp Ser Ser Asn Tyr Glu Gly Tyr Phe Asp Tyr
1 5 10
<210> 86
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 86
His Ile Phe Ser Thr Asp Glu Lys Ser Tyr Ser Thr Ser Leu Arg Gly
1 5 10 15
<210> 87
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 87
His Ile Phe Ser Thr Asp Glu Lys Ser Tyr
1 5 10
<210> 88
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 88
Asn Asn Ala Arg Met Gly Val Ser
1 5
<210> 89
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 89
Gly Phe Ser Leu Asn Asn Ala Arg
1 5
<210> 90
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 90
Gly Phe Ser Leu Asn Asn Ala Arg Met Gly Val Ser
1 5 10
<210> 91
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 91
His Ile Phe Ser Thr Asp Glu Lys Ser Phe Arg Thr Ser Leu Arg Ser
1 5 10 15
<210> 92
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 92
His Ile Phe Ser Thr Asp Glu Lys Ser Phe
1 5 10
<210> 93
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 93
Ser Asn Val Arg Met Gly Val Ser
1 5
<210> 94
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 94
Gly Phe Ser Leu Ser Asn Val Arg
1 5
<210> 95
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 95
Gly Phe Ser Leu Ser Asn Val Arg Met Gly Val Ser
1 5 10
<210> 96
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 96
His Ile Phe Ser Ser Asp Glu Lys Ser Ile Arg Arg Ser Leu Arg Ser
1 5 10 15
<210> 97
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 97
His Ile Phe Ser Ser Asp Glu Lys Ser Ile
1 5 10
<210> 98
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 98
His Ile Phe Ser Thr Asp Glu Lys Ser Leu Arg Leu Ser Leu Arg Ser
1 5 10 15
<210> 99
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 99
Ser Asn Ala Lys Met Gly Val Ser
1 5
<210> 100
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 100
Gly Phe Ser Leu Ser Asn Ala Lys
1 5
<210> 101
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 101
Gly Phe Ser Leu Ser Asn Ala Lys Met Gly Val Ser
1 5 10
<210> 102
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 102
His Ile Phe Ser Ser Asp Glu Lys Ser Tyr Arg Leu Ser Leu Arg Ser
1 5 10 15
<210> 103
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 103
His Ile Phe Ser Ser Asp Glu Lys Ser Tyr
1 5 10
<210> 104
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 104
Asp Ser Ser Asn Tyr Gly Gly Tyr Phe Asp Tyr
1 5 10
<210> 105
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 105
His Ile Phe Ser Ser Asp Glu Lys Ser Tyr Arg Leu Phe Leu Arg Ser
1 5 10 15
<210> 106
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 106
His Ile Phe Ser Thr Asp Glu Lys Ser Tyr Ser Pro Ser Leu Arg Gly
1 5 10 15
<210> 107
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 107
Asp Ser Ser Asp Tyr Glu Gly Tyr Phe Asp Tyr
1 5 10
<210> 108
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 108
Asp Ser Ser Asn Tyr Glu Glu Tyr Phe Asp Tyr
1 5 10
<210> 109
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 109
Ser Asp Ala Trp Met Ser
1 5
<210> 110
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 110
Gly Phe Thr Phe Ser Asp
1 5
<210> 111
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 111
Gly Phe Thr Phe Ser Asp Ala Trp Met Ser
1 5 10
<210> 112
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 112
Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr Val Val Pro
1 5 10 15
Leu Asn Gly
<210> 113
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 113
Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr Thr Asp Tyr
1 5 10
<210> 114
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 114
Val Pro Gly Ser Tyr Gly Tyr
1 5
<210> 115
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 115
Ser Tyr Ala Trp Met Ser
1 5
<210> 116
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 116
Gly Phe Thr Phe Ser Tyr
1 5
<210> 117
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 117
Gly Phe Thr Phe Ser Tyr Ala Trp Met
1 5
<210> 118
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 118
Arg Ile Lys Ser Ile Ala Asp Gly Gly Ala Thr Asp Tyr Ala Ala Pro
1 5 10 15
Val Arg Asn
<210> 119
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 119
Arg Ile Lys Ser Ile Ala Asp Gly Gly Ala Thr Asp Tyr
1 5 10
<210> 120
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 120
Ile Pro Gly Asn Asp Ala Phe Asp Met
1 5
<210> 121
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 121
Asn Asn Ala Trp Met Ser
1 5
<210> 122
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 122
Gly Phe Ile Phe Asn Asn
1 5
<210> 123
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 123
Gly Phe Ile Phe Asn Asn Ala Trp Met Ser
1 5 10
<210> 124
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 124
Arg Ile Lys Ser Lys Ser Asp Gly Gly Thr Thr Asp Tyr Ala Ala Pro
1 5 10 15
Val Lys Asp
<210> 125
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 125
Arg Ile Lys Ser Lys Ser Asp Gly Gly Thr Thr Asp Tyr
1 5 10
<210> 126
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 126
Ala Pro Gly Gly Pro Phe Asp Tyr
1 5
<210> 127
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 127
Arg Ile Lys Ser Ile Thr Asp Gly Gly Val Ile Asp Tyr Ala Ala Pro
1 5 10 15
Val Arg Asn
<210> 128
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 128
Arg Ile Lys Ser Ile Thr Asp Gly Gly Val Ile Asp Tyr
1 5 10
<210> 129
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 129
Ile Pro Gly Asn Asp Asp Phe Asp Met
1 5
<210> 130
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 130
Arg Ile Lys Ser Ile Asn Asp Gly Gly Ala Thr Asp Tyr Ala Ser Pro
1 5 10 15
Val Arg Asn
<210> 131
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 131
Arg Ile Lys Ser Ile Asn Asp Gly Gly Ala Thr Asp Tyr
1 5 10
<210> 132
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 132
Thr Asn Ala Trp Met Ser
1 5
<210> 133
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 133
Gly Phe Thr Phe Thr Asn
1 5
<210> 134
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 134
Gly Phe Thr Phe Thr Asn Ala Trp Met Ser
1 5 10
<210> 135
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 135
Arg Ile Lys Ser Lys Ile Asp Gly Gly Thr Thr Asp Tyr Ala Ala Pro
1 5 10 15
Val Lys Gly
<210> 136
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 136
Arg Ile Lys Ser Lys Ile Asp Gly Gly Thr Thr Asp Tyr
1 5 10
<210> 137
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 137
Ser Ser Asn Ala Ile Ser
1 5
<210> 138
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 138
Gly Asp Thr Phe Ser Ser
1 5
<210> 139
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 139
Gly Asp Thr Phe Ser Ser Asn Ala Ile Ser
1 5 10
<210> 140
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 140
Val Ile Ile Pro Ile Phe Gly Thr Ala Asp Tyr Ala Gln Lys Phe Gln
1 5 10 15
Gly
<210> 141
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 141
Val Ile Ile Pro Ile Phe Gly Thr Ala Asp Tyr
1 5 10
<210> 142
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 142
His Thr Tyr His Glu Tyr Ala Gly Gly Tyr Tyr Gly Gly Ala Met Asp
1 5 10 15
Pro
<210> 143
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 143
Ser Asn Tyr Ala Met Ser
1 5
<210> 144
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 144
Gly Phe Thr Phe Ser Asn
1 5
<210> 145
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 145
Gly Phe Thr Phe Ser Asn Tyr Ala Met Ser
1 5 10
<210> 146
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 146
Asp Ile Ser Gly Gly Gly Gly Arg Thr Tyr Tyr Ala Asp Ser Val Lys
1 5 10 15
Gly
<210> 147
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 147
Asp Ile Ser Gly Gly Gly Gly Arg Thr Tyr Tyr
1 5 10
<210> 148
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 148
Ala Gly Leu Leu Tyr Gly Gly Gly Val Tyr Pro Met Asp Ile
1 5 10
<210> 149
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 149
Lys Ser Ser Gln Ser Leu Leu Tyr Ser Asn Gly Lys Thr Tyr Leu Asn
1 5 10 15
<210> 150
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 150
Leu Val Ser Lys Leu Asp Ser
1 5
<210> 151
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 151
Val Gln Asp Thr His Phe Pro Leu Thr
1 5
<210> 152
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 152
Gln Val Ser Lys Leu Asp Ser
1 5
<210> 153
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 153
Gly Gln Asp Thr His Phe Pro Leu Thr
1 5
<210> 154
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 154
Lys Ser Ser Gln Ser Leu Leu Tyr Ser Asn Asp Lys Thr Tyr Thr Asn
1 5 10 15
<210> 155
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 155
Glu Val Ser Lys Leu Asp Val
1 5
<210> 156
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 156
Arg Ala Ser Gln Ser Val Arg Ser Asn Leu Ala
1 5 10
<210> 157
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 157
Gly Ser Thr Ile Arg Ala Thr
1 5
<210> 158
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 158
Gln Gln Tyr Ser Asp Trp Pro Phe Thr
1 5
<210> 159
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 159
Arg Ala Ser Gln Ser Val Ser Ser Asn Phe Ala
1 5 10
<210> 160
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 160
Gly Ala Thr Thr Arg Ala Thr
1 5
<210> 161
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 161
Gln Gln Tyr Lys Asp Trp Pro Phe Thr
1 5
<210> 162
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 162
Arg Val Ser Gln Ser Ile Gly Ala Asn Leu Ala
1 5 10
<210> 163
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 163
Gly Ala Ser Thr Arg Ala Thr
1 5
<210> 164
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 164
Gln Gln Tyr Ile Tyr Trp Pro Phe Thr
1 5
<210> 165
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 165
Arg Ala Ser Gln Ser Val Ser Asn Asn Leu Ala
1 5 10
<210> 166
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 166
Arg Ala Ser Gln Ser Val Gly Ser Asp Leu Ala
1 5 10
<210> 167
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 167
Gln Gln Tyr Asn Asp Trp Pro Phe Thr
1 5
<210> 168
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 168
Arg Ala Ser Gln Asn Ile Gly Ser Asp Leu Ala
1 5 10
<210> 169
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 169
Arg Ala Ser Gln Ser Val Thr Ser Asn Phe Ala
1 5 10
<210> 170
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 170
Arg Ala Ser Gln Gly Val Ser Ser Asn Phe Ala
1 5 10
<210> 171
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 171
Arg Ala Ser Gln Ser Val Asn Arg Asn Leu Ala
1 5 10
<210> 172
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 172
Gly Thr Ser Thr Arg Ala Thr
1 5
<210> 173
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 173
Arg Ala Ser Gln Ser Val Ser Thr Asn Phe Ala
1 5 10
<210> 174
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 174
Arg Ala Ser Gln Ser Val Asn Ser Asn Leu Ala
1 5 10
<210> 175
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 175
Gly Ser Ser Thr Arg Ala Thr
1 5
<210> 176
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 176
Arg Ala Ser Gln Ser Val Ile Asn Asn Leu Ala
1 5 10
<210> 177
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 177
Gln Asp Tyr Asn Asn Trp Pro Phe Thr
1 5
<210> 178
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 178
Arg Ala Ser Gln Ser Val Gly Ser Asn Leu Ala
1 5 10
<210> 179
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 179
Gly Ala Ser Thr Arg Ala Ser Gly
1 5
<210> 180
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 180
Gln Glu Tyr Asn Asn Trp Pro Phe Thr
1 5
<210> 181
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 181
Arg Ala Asn Gln Ile Val Ser Ser Asn Leu Ala
1 5 10
<210> 182
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 182
Arg Ser Ser Gln Ser Leu Leu His Asn Lys Arg Asn Asn Tyr Leu Asp
1 5 10 15
<210> 183
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 183
Leu Ala Ser Asn Arg Ala Ser
1 5
<210> 184
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 184
Met Gln Ala Gln Gln Thr Pro Ile Thr
1 5
<210> 185
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 185
Arg Ser Ser Gln Ser Leu Leu Tyr Ser Asn Gly Lys Asn Tyr Leu Asp
1 5 10 15
<210> 186
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 186
Leu Gly Ser Asn Arg Ala Ser
1 5
<210> 187
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 187
Arg Ser Ser Gln Ser Leu Leu His Arg Asp Gly Phe Asn Tyr Leu Asp
1 5 10 15
<210> 188
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 188
Leu Ala Ser Ser Arg Ala Ser
1 5
<210> 189
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 189
Met Gln Ala Leu Gln Thr Pro Ile Thr
1 5
<210> 190
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 190
Arg Ser Thr Gln Ser Leu Leu Tyr Ser Asn Gly Lys Asn Tyr Leu Asp
1 5 10 15
<210> 191
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 191
Leu Gly Ser Ile Arg Ala Ser
1 5
<210> 192
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 192
Arg Ser Ser Gln Ser Leu Leu Tyr Ser Asp Arg Arg Asn Tyr Leu Asp
1 5 10 15
<210> 193
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 193
Leu Gly Ser Tyr Arg Ala Ser
1 5
<210> 194
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 194
Met Gln Ala Leu Gln Ile Pro Ile Thr
1 5
<210> 195
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 195
Ser Gly Ser Ser Ser Asn Ile Gly Ser Asn Tyr Val Tyr
1 5 10
<210> 196
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 196
Arg Asn Asn Gln Arg Pro Ser
1 5
<210> 197
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 197
Ala Ala Trp Asp Asp Asn Leu Ser Gly Trp Val
1 5 10
<210> 198
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 198
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn
1 5 10
<210> 199
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 199
Ala Ala Ser Ser Leu Gln Ser
1 5
<210> 200
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 200
Gln Gln Ser Tyr Ser Thr Pro Leu Thr
1 5
<210> 201
<211> 943
<212> PRT
<213> Homo sapien
<400> 201
Met Arg Pro Ser Gly Thr Ala Gly Ala Ala Leu Leu Ala Leu Leu Ala
1 5 10 15
Ala Leu Cys Pro Ala Ser Arg Ala Leu Glu Glu Lys Lys Gly Asn Tyr
20 25 30
Val Val Thr Asp His Gly Ser Cys Val Arg Ala Cys Gly Ala Asp Ser
35 40 45
Tyr Glu Met Glu Glu Asp Gly Val Arg Lys Cys Lys Lys Cys Glu Gly
50 55 60
Pro Cys Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe Lys Asp
65 70 75 80
Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn Cys Thr
85 90 95
Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg Gly Asp
100 105 110
Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp Ile Leu
115 120 125
Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala Trp Pro
130 135 140
Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile Ile Arg
145 150 155 160
Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val Ser Leu
165 170 175
Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser Asp Gly
180 185 190
Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn Thr Ile
195 200 205
Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys Ile Ile
210 215 220
Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val Cys His
225 230 235 240
Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg Asp Cys
245 250 255
Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp Lys Cys
260 265 270
Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser Glu Cys
275 280 285
Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile Thr Cys
290 295 300
Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr Ile Asp
305 310 315 320
Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly Glu Asn
325 330 335
Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys His Leu
340 345 350
Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu Glu Gly
355 360 365
Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly Met Val
370 375 380
Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly Leu Phe
385 390 395 400
Met Arg Arg Arg His Ile Val Arg Lys Arg Thr Leu Arg Arg Leu Leu
405 410 415
Gln Glu Arg Glu Leu Val Glu Pro Leu Thr Pro Ser Gly Glu Ala Pro
420 425 430
Asn Gln Ala Leu Leu Arg Ile Leu Lys Glu Thr Glu Phe Lys Lys Ile
435 440 445
Lys Val Leu Gly Ser Gly Ala Phe Gly Thr Val Tyr Lys Gly Leu Trp
450 455 460
Ile Pro Glu Gly Glu Lys Val Lys Ile Pro Val Ala Ile Lys Glu Leu
465 470 475 480
Arg Glu Ala Thr Ser Pro Lys Ala Asn Lys Glu Ile Leu Asp Glu Ala
485 490 495
Tyr Val Met Ala Ser Val Asp Asn Pro His Val Cys Arg Leu Leu Gly
500 505 510
Ile Cys Leu Thr Ser Thr Val Gln Leu Ile Thr Gln Leu Met Pro Phe
515 520 525
Gly Cys Leu Leu Asp Tyr Val Arg Glu His Lys Asp Asn Ile Gly Ser
530 535 540
Gln Tyr Leu Leu Asn Trp Cys Val Gln Ile Ala Lys Gly Met Asn Tyr
545 550 555 560
Leu Glu Asp Arg Arg Leu Val His Arg Asp Leu Ala Ala Arg Asn Val
565 570 575
Leu Val Lys Thr Pro Gln His Val Lys Ile Thr Asp Phe Gly Leu Ala
580 585 590
Lys Leu Leu Gly Ala Glu Glu Lys Glu Tyr His Ala Glu Gly Gly Lys
595 600 605
Val Pro Ile Lys Trp Met Ala Leu Glu Ser Ile Leu His Arg Ile Tyr
610 615 620
Thr His Gln Ser Asp Val Trp Ser Tyr Gly Val Thr Val Trp Glu Leu
625 630 635 640
Met Thr Phe Gly Ser Lys Pro Tyr Asp Gly Ile Pro Ala Ser Glu Ile
645 650 655
Ser Ser Ile Leu Glu Lys Gly Glu Arg Leu Pro Gln Pro Pro Ile Cys
660 665 670
Thr Ile Asp Val Tyr Met Ile Met Val Lys Cys Trp Met Ile Asp Ala
675 680 685
Asp Ser Arg Pro Lys Phe Arg Glu Leu Ile Ile Glu Phe Ser Lys Met
690 695 700
Ala Arg Asp Pro Gln Arg Tyr Leu Val Ile Gln Gly Asp Glu Arg Met
705 710 715 720
His Leu Pro Ser Pro Thr Asp Ser Asn Phe Tyr Arg Ala Leu Met Asp
725 730 735
Glu Glu Asp Met Asp Asp Val Val Asp Ala Asp Glu Tyr Leu Ile Pro
740 745 750
Gln Gln Gly Phe Phe Ser Ser Pro Ser Thr Ser Arg Thr Pro Leu Leu
755 760 765
Ser Ser Leu Ser Ala Thr Ser Asn Asn Ser Thr Val Ala Cys Ile Asp
770 775 780
Arg Asn Gly Leu Gln Ser Cys Pro Ile Lys Glu Asp Ser Phe Leu Gln
785 790 795 800
Arg Tyr Ser Ser Asp Pro Thr Gly Ala Leu Thr Glu Asp Ser Ile Asp
805 810 815
Asp Thr Phe Leu Pro Val Pro Glu Tyr Ile Asn Gln Ser Val Pro Lys
820 825 830
Arg Pro Ala Gly Ser Val Gln Asn Pro Val Tyr His Asn Gln Pro Leu
835 840 845
Asn Pro Ala Pro Ser Arg Asp Pro His Tyr Gln Asp Pro His Ser Thr
850 855 860
Ala Val Gly Asn Pro Glu Tyr Leu Asn Thr Val Gln Pro Thr Cys Val
865 870 875 880
Asn Ser Thr Phe Asp Ser Pro Ala His Trp Ala Gln Lys Gly Ser His
885 890 895
Gln Ile Ser Leu Asp Asn Pro Asp Tyr Gln Gln Asp Phe Phe Pro Lys
900 905 910
Glu Ala Lys Pro Asn Gly Ile Phe Lys Gly Ser Thr Ala Glu Asn Ala
915 920 925
Glu Tyr Leu Arg Val Ala Pro Gln Ser Ser Glu Phe Ile Gly Ala
930 935 940
<210> 202
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 202
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 203
<211> 42
<212> PRT
<213> Homo sapien
<400> 203
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 204
<211> 42
<212> PRT
<213> Homo sapien
<400> 204
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 205
<211> 112
<212> PRT
<213> Homo sapien
<400> 205
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
1 5 10 15
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
20 25 30
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
35 40 45
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
50 55 60
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65 70 75 80
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
85 90 95
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210> 206
<211> 21
<212> PRT
<213> Homo sapien
<400> 206
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro
20
<210> 207
<211> 16
<212> PRT
<213> Homo sapien
<400> 207
Gly Leu Ala Val Ser Thr Ile Ser Ser Phe Phe Pro Pro Gly Tyr Gln
1 5 10 15
<210> 208
<211> 45
<212> PRT
<213> Homo sapien
<400> 208
Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala
1 5 10 15
Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly
20 25 30
Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
35 40 45
<210> 209
<211> 231
<212> PRT
<213> Homo sapien
<400> 209
Glu Pro Lys Ser Pro Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
20 25 30
Asp Thr Leu Met Ile Ala Arg Thr Pro Glu Val Thr Cys Val Val Val
35 40 45
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
50 55 60
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
65 70 75 80
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
85 90 95
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
100 105 110
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
115 120 125
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
130 135 140
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
145 150 155 160
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
165 170 175
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
180 185 190
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
195 200 205
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
210 215 220
Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 210
<211> 24
<212> PRT
<213> Homo sapien
<400> 210
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
1 5 10 15
Ser Leu Val Ile Thr Leu Tyr Cys
20
<210> 211
<211> 52
<212> PRT
<213> Homo sapien
<400> 211
Phe Phe Ile Pro Leu Leu Val Val Ile Leu Phe Ala Val Asp Thr Gly
1 5 10 15
Leu Phe Ile Ser Thr Gln Gln Gln Val Thr Phe Leu Leu Lys Ile Lys
20 25 30
Arg Thr Arg Lys Gly Phe Arg Leu Leu Asn Pro His Pro Lys Pro Asn
35 40 45
Pro Lys Asn Asn
50
<210> 212
<211> 215
<212> PRT
<213> Homo sapien
<400> 212
Met Asp Thr Glu Ser Asn Arg Arg Ala Asn Leu Ala Leu Pro Gln Glu
1 5 10 15
Pro Ser Ser Val Pro Ala Phe Glu Val Leu Glu Ile Ser Pro Gln Glu
20 25 30
Val Ser Ser Gly Arg Leu Leu Lys Ser Ala Ser Ser Pro Pro Leu His
35 40 45
Thr Trp Leu Thr Val Leu Lys Lys Glu Gln Glu Phe Leu Gly Val Thr
50 55 60
Gln Ile Leu Thr Ala Met Ile Cys Leu Cys Phe Gly Thr Val Val Cys
65 70 75 80
Ser Val Leu Asp Ile Ser His Ile Glu Gly Asp Ile Phe Ser Ser Phe
85 90 95
Lys Ala Gly Tyr Pro Phe Trp Gly Ala Ile Phe Phe Ser Ile Ser Gly
100 105 110
Met Leu Ser Ile Ile Ser Glu Arg Arg Asn Ala Thr Tyr Leu Val Arg
115 120 125
Gly Ser Leu Gly Ala Asn Thr Ala Ser Ser Ile Ala Gly Gly Thr Gly
130 135 140
Ile Thr Ile Leu Ile Ile Asn Leu Lys Lys Ser Leu Ala Tyr Ile His
145 150 155 160
Ile His Ser Cys Gln Lys Phe Phe Glu Thr Lys Cys Phe Met Ala Ser
165 170 175
Phe Ser Thr Glu Ile Val Val Met Met Leu Phe Leu Thr Ile Leu Gly
180 185 190
Leu Gly Ser Ala Val Ser Leu Thr Ile Cys Gly Ala Gly Glu Glu Leu
195 200 205
Lys Gly Asn Lys Val Pro Glu
210 215
<210> 213
<211> 41
<212> PRT
<213> Homo sapien
<400> 213
Arg Ser Lys Arg Ser Arg Gly Gly His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 214
<211> 18
<212> PRT
<213> Homo sapien
<400> 214
Met Ile Pro Ala Val Val Leu Leu Leu Leu Leu Leu Val Glu Gln Ala
1 5 10 15
Ala Ala
<210> 215
<211> 43
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 215
Leu Gly Glu Pro Gln Leu Cys Tyr Ile Leu Asp Ala Ile Leu Phe Leu
1 5 10 15
Tyr Gly Ile Val Leu Thr Leu Leu Tyr Cys Arg Leu Lys Ile Gln Val
20 25 30
Arg Lys Ala Ala Ile Thr Ser Tyr Glu Lys Ser
35 40
<210> 216
<211> 22
<212> PRT
<213> Homo sapien
<400> 216
Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val
1 5 10 15
Glu Glu Asn Pro Gly Pro
20
<210> 217
<211> 21
<212> PRT
<213> Homo sapien
<400> 217
Gly Ser Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys Gly Asp Val Glu
1 5 10 15
Glu Asn Pro Gly Pro
20
<210> 218
<211> 255
<212> PRT
<213> Homo sapien
<400> 218
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
35 40 45
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
50 55 60
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
65 70 75 80
Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
85 90 95
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
100 105 110
Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
115 120 125
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
130 135 140
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
145 150 155 160
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
165 170 175
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
180 185 190
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
195 200 205
Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu
210 215 220
Thr Val Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
225 230 235 240
Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln
245 250 255
<210> 219
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 219
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly
35 40 45
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
50 55 60
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His
65 70 75 80
Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
85 90 95
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
100 105 110
Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu
115 120 125
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
130 135 140
Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
145 150 155 160
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
165 170 175
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
180 185 190
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
195 200 205
Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu
210 215 220
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
225 230 235 240
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln
245 250 255
<210> 220
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 220
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly
35 40 45
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
50 55 60
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
65 70 75 80
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
85 90 95
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
100 105 110
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
115 120 125
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
130 135 140
Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
145 150 155 160
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
165 170 175
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
180 185 190
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
195 200 205
Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu
210 215 220
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
225 230 235 240
Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln
245 250 255
<210> 221
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 221
Asn Pro Gln Arg Ser Thr Val Trp Tyr Leu Thr Pro Gln Gln Val Val
1 5 10 15
Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln
20 25 30
Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln
35 40 45
Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr
50 55 60
Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro
65 70 75 80
Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu
85 90 95
Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu
100 105 110
Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln
115 120 125
Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His
130 135 140
Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly
145 150 155 160
Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln
165 170 175
Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp
180 185 190
Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu
195 200 205
Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser
210 215 220
Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro
225 230 235 240
Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
245 250 255
<210> 222
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 222
Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly
35 40 45
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
50 55 60
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His
65 70 75 80
Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
85 90 95
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
100 105 110
Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu
115 120 125
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
130 135 140
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
145 150 155 160
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
165 170 175
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
180 185 190
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu
195 200 205
Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu
210 215 220
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
225 230 235 240
Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys Gln
245 250 255
<210> 223
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 223
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
35 40 45
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
50 55 60
Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn
65 70 75 80
Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
85 90 95
Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala
100 105 110
Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Ala Leu Leu
115 120 125
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala
130 135 140
Ile Ala Ser Asn Asn Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
145 150 155 160
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
165 170 175
Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln Ala Leu Glu Thr Val
180 185 190
Gln Ala Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
195 200 205
Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu
210 215 220
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
225 230 235 240
Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln
245 250 255
<210> 224
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 224
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His Asp Gly
35 40 45
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
50 55 60
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His
65 70 75 80
Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
85 90 95
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
100 105 110
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
115 120 125
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
130 135 140
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
145 150 155 160
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val
165 170 175
Val Ala Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val
180 185 190
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
195 200 205
Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu
210 215 220
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
225 230 235 240
Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln
245 250 255
<210> 225
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 225
Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys
1 5 10 15
Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala
20 25 30
His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala Ser Asn Asn Gly
35 40 45
Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val Leu Cys
50 55 60
Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala Ile Ala Ser His
65 70 75 80
Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu Pro Val
85 90 95
Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val Val Ala Ile Ala
100 105 110
Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg Leu Leu
115 120 125
Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Glu Gln Val Val Ala
130 135 140
Ile Ala Ser His Asp Gly Gly Lys Gln Ala Leu Glu Thr Val Gln Arg
145 150 155 160
Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln Gln Val
165 170 175
Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu Thr Val
180 185 190
Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr Pro Gln
195 200 205
Gln Val Val Ala Ile Ala Ser Asn Gly Gly Gly Lys Gln Ala Leu Glu
210 215 220
Thr Val Gln Arg Leu Leu Pro Val Leu Cys Gln Ala His Gly Leu Thr
225 230 235 240
Pro Glu Gln Val Val Ala Ile Ala Ser Asn Ile Gly Gly Lys Gln
245 250 255
<210> 226
<211> 136
<212> PRT
<213> Homo sapien
<400> 226
Cys Pro Tyr Ser Asn Pro Ser Leu Cys Ser Gly Gly Gly Gly Ser Glu
1 5 10 15
Leu Pro Thr Gln Gly Thr Phe Ser Asn Val Ser Thr Asn Val Ser Pro
20 25 30
Ala Lys Pro Thr Thr Thr Ala Cys Pro Tyr Ser Asn Pro Ser Leu Cys
35 40 45
Ser Gly Gly Gly Gly Ser Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro
50 55 60
Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro
65 70 75 80
Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp
85 90 95
Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu
100 105 110
Ser Leu Val Ile Thr Leu Tyr Cys Asn His Arg Asn Arg Arg Arg Val
115 120 125
Cys Lys Cys Pro Arg Pro Val Val
130 135
<210> 227
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 227
Cys Gly Ser Gly Ser Gly Leu Glu Glu Lys Lys Gly Asn Tyr Val Val
1 5 10 15
Thr Asp His
<210> 228
<211> 804
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 228
atggctctgc ccgtcaccgc tctgctgctg cccctggctc tgctgctgca cgctgctcgc 60
cctgatgtgg tcatgactca gtctcccctg tctctgcccg tcaccctggg acagcccgcc 120
agcatctcct gcaagagctc ccagagcctg ctgtactcca acggcaagac ctatctgaat 180
tggttccagc agagacccgg ccagagccct cggagactga tctaccaggt gtctaagctg 240
gacagcggcg tgcctgatcg cttctctgga agcggatccg gaaccgactt tacactgaag 300
atcagccggg tggaggcaga ggacgtgggc gtgtactatt gcggccagga tacccacttc 360
ccactgacat ttggcggcgg caccaaggtg gagatcaagg gaggaggagg aagcggagga 420
ggaggaagcg gcggcggcgg ctctggcggc ggcggcagcc aggtgcagct ggtgcagagc 480
ggagcagagg tgaagaagcc tggcgcctcc gtgaaggtgt cttgtaaggc cagcggctac 540
acattcaccg attatacact gcactgggtg cggcaggccc ctggccaggg actggagtgg 600
atgggaggaa tctggcctat caccggagga accacataca accagaagtt taagggcaga 660
gtgacaatga ccagggacac atctaccagc acagtgtata tggagctgtc tagcctgcgc 720
tccgaggata cagccgtgta ctattgcgcc agaggcgagg cacagggatc ttggggacag 780
ggcaccctgg tgacagtgtc ctct 804
<210> 229
<211> 807
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 229
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
cctcaggtga ccctgaagga gagcggccct gtgctgctga agccaacaga gaccctgaca 120
ctgacctgca cagtgtctgg cttcagcctg tccaaccccc ggatgggcgt gagctggatc 180
agacagcccc ctggcaaggc cctggagtgg ttcgcccaca tcttttctac cgatgagaag 240
agcctgaagc tgtccctgag atctaggctg accctgagca aggacacatc taagagccag 300
gtggtgctga ccatgacaaa catggcccct gtggactccg ccacatacta ttgcgccaga 360
gacagctcca attacgaggg ctatttcgac ttttggggcc agggcaccct ggtgacagtg 420
tctagcggcg gaggaggatc cggaggagga ggatctggcg gcggcggctc cggcggcggc 480
ggctccgagg tggtgctgac ccagagccct gccacactgt ccgtgtctcc aggcgagaga 540
gccaccctgt cttgtagggc cagccagtcc gtgcgcagca atctggcctg gtaccagcag 600
aagtccggcc aggccccaag actgctgatc tatggctcca ccatcagggc cacaggagtg 660
ccagcacgct tctctggaag cggatccggc acagagttta ccctgacaat ctcctctctg 720
cagtccgagg atttcgccgt gtactattgc cagcagtact ctgactggcc cttcaccttt 780
ggccctggca caaaggtgga tatcaag 807
<210> 230
<211> 807
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 230
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
ccacaggtga ccctgaagga gtccggcccc gtgctggtga agcctacaga gaccctgaca 120
ctgacctgca cagtgtccgg cttctctctg agcaacgccc gcatgggcgt gtcttggatc 180
aggcagcccc ctggcaaggc cctggagtgg ctggcccaca tcttttccac cgacgagaag 240
tctatccgga gaagcctgcg ctccaggctg accctgagca aggatacatc caagtctcag 300
gtggtgctga ccatgacaaa catggacccc gtggataccg ccacatactt ctgcgccaga 360
gacagctcca attacgaggg ctattttgat tactggggcc agggcaccct ggtgacagtg 420
tctagcggag gaggaggaag cggaggagga ggatctggcg gcggcggctc tggcggcggc 480
ggcagcgagg tggtcatgac ccagagccca gccacactga gcgtgtcccc tggcgagagg 540
gtgaccctgt cctgtagggc atctcagagc gtgtcctcta acttcgcctg gtatcagcag 600
agaccaggcc aggcaccaag gctgctgctg tacggagcaa ccacaagagc cacaggactg 660
cccggcaggt tttccggatc tggaagcggc accgagaata tcctgacaat cagctccctg 720
cagtctgagg acttcgccat ctatttttgc cagcagtaca aggattggcc attcaccttt 780
ggccccggca gcaaggtgga catcaag 807
<210> 231
<211> 807
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 231
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaaga 60
cctcaggtga ccctgaagga gtccggccct gtgctggtga agccaacaga gaccctgaca 120
ctgacctgca cagtgtctgg cttcagcctg tccaacgcaa ggatgggcgt gagctggatc 180
aggcagcccc ctggcaaggc cctggagtgg ctgggccaca tctttagcac cgacgagaag 240
tcttacagca catccctgag aggcaggatc accatctcta aggatacaag cagaggcctg 300
gtggtgctga ccctgacaaa catggacccc gtggataccg ccacatacta ttgcgccagg 360
gacagctcca attacgaggg ctatttcgat ttttggggcc ctggcttcct ggtgaccgtg 420
tctagcggcg gcggcggctc tggaggagga ggaagcggag gaggaggatc cggcggcggc 480
ggctctgaga tcgtgatgac ccagtcccct gccacactgt ctgtgagccc aggcgagaga 540
gccaccctgt cttgtagggt gtcccagtct atcggcgcca atctggcctg gtaccagcag 600
aagttcggcc aggccccaag gctgctgatc tatggagcat ccaccagagc cacaggaatc 660
cccgtgaggt tctccggagg aggatctgga accgagttta ccctgacaat ctcctctctg 720
cagagcgagg actttgccat ctactcctgc cagcagtaca tctattggcc cttcacattt 780
ggccctggca ccacagtgga tatcaag 807
<210> 232
<211> 807
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 232
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaaga 60
ccacaggtga ccctgaagga gagcggaccc gtgctggtga agcctacaga gaccctgaca 120
ctgacctgca cagtgagcgg cttctccctg aacaatgcaa ggatgggcgt gtcctggatc 180
aggcagcccc ctggcaaggc cctggagtgg ttcgcccaca tctttagcac cgacgagaag 240
tcctttcgca catctctgag aagcaggctg accctgagca aggatacaag caagtcccag 300
gtggtgctga ccatgacaaa catggacccc gtggataccg ccacatacta ttgcgccaga 360
gacagctcca attacgaggg ctatttcgat tactggggcc agggcatcct ggtgaccgtg 420
tctagcggcg gcggcggctc tggaggagga ggaagcggag gaggaggatc cggcggcggc 480
ggctctgaga tcgtgatgac ccagtctccc gccacactgt ctgtgagccc tggcgagaga 540
gccacactga gctgtagggc ctcccagtct gtgagcaaca atctggcctg gtatcagcag 600
aagccaggcc aggcaccaag gctgctgatc tacggagcat ccaccagagc cacaggagtg 660
ccagcaaggt tctccggatc tgacagcggc accgagttta gcctgacaat ctcctctctg 720
cagtccgagg acttcgccgt gtatttttgc cagcagtaca aggattggcc attcaccttt 780
ggccccggca caaaggtgga gatcaag 807
<210> 233
<211> 819
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 233
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
ccagaggtga acctggtgga gtccggcggc ggcctggtga agcctggcgg atccctgagg 120
ctgtcttgcg aggcaagcgg cttcaccttc agctacgcct ggatgtcctg ggtgcgccag 180
gcccccggca agggactgga gtgggtggga cggatcaagt ccatcgcaga cggaggagca 240
accgattacg cagcccctgt gagaaacagg ttcacaatct ccagagacga ttctaggaat 300
accctgtatc tggagatgca ctctctgaag acagaggaca ccgccgtgta ctattgcacc 360
acaatccctg gcaacgacgc ctttgatatg tggggccagg gcacaatggt gaccgtgagc 420
tccggcggcg gcggctctgg aggaggagga agcggaggag gaggaagcgg gggcggcggc 480
tctgacatcg tgctgacaca gtccccactg tccctgtctg tgacccccgg cgagcctgca 540
agcatctcct gtagatctag ccagagcctg ctgtactcca acggcaagaa ttatctggat 600
tggttcctgc acaagccagg ccagtctccc cagctgctga tctacctggg atctaatagg 660
gcaagcggag tgccagaccg gttctctgga agcggatccg gcatcgactt catcctgaag 720
atcagcaggg tggaggccga ggatgtgggc gtgtactatt gcatgcaggc ccagcagaca 780
cccatcacct tcggccaggg cacaagactg gagatcaag 819
<210> 234
<211> 819
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 234
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
ccagaggtga acctggtgga gtccggcggc ggcctggtga agcctggcgg atccctgagg 120
ctgtcttgcg aggcaagcgg cttcaccttc agctacgcct ggatgtcctg ggtgcgccag 180
gcccccggca agggactgga gtgggtgggc cggatcaagt ccatcaccga cggaggcgtg 240
atcgattacg cagcacctgt gagaaacagg tgcacaatct ccagagacga ttctaggaat 300
accctgtatc tggagatgca ctctctgaag acagaggaca ccgccgtgta ctattgtacc 360
acaatccctg gcaacgacga tttcgatatg tggggccagg gcagaatggt gaccgtgagc 420
tccggcggcg gcggctctgg aggaggagga agcggaggag gaggaagcgg gggcggcggc 480
tctgacatcg tgctgacaca gtccccactg tccctgtctg tgacccccgg cgagcctgca 540
agcatctcct gtaggtctag ccagagcctg ctgtactcca acggcaagaa ttatctggat 600
tggtttctgc acaagccagg ccagtctccc cagctgctga tctacctggg atctaatagg 660
gcaagcggag tgccagaccg gttctctgga agcggatccg gcatcgactt catcctgaag 720
atcagccgcg tggaggcaga ggacgtgggc gtgtactatt gcatgcaggc ccagcagaca 780
cccatcacct tcggccaggg cacaagactg gagatcaag 819
<210> 235
<211> 816
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 235
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
ccagaggtgc agctggtgga gtcttggggc gtgctggtga agcctggcgg atctctgagg 120
ctgagctgcg cagcatccgg cttcatcttt aacaatgcct ggatgtcctg ggtgcgccag 180
gcccccggca agggactgga gtggatcggc cggatcaaga gcaagtccga cggaggaacc 240
acagattacg cagcacctgt gaaggaccgc ttcacaatct ctcgggacga tagcaaggat 300
accctgtatc tgcagatgaa cggcctgaag acagaggaca ccgccgtgta cttctgcacc 360
acagcccctg gcggcccttt tgattattgg ggccagggca cactggtgac cgtgagctcc 420
ggaggaggag gaagcggcgg aggaggcagc ggcggcggcg gctctggcgg cggcggcagc 480
gacatcgtgc tgacacagag ccctctgtcc ctgccagtga cccccggcga gcctgcctct 540
atcagctgtc gctctagcca gagcctgctg caccgggacg gcttcaatta cctggattgg 600
tttctgcaga agccaggcca gtccccccag ctgctgatct atctggcctc ctctagagcc 660
tctggcgtgc cagacaggtt ctccggctct gacagcggca cagacttcac cctgaagatc 720
agcagagtgg aggccgagga tgtgggcgtg tactattgca tgcaggccct gcagacaccc 780
atcaccttcg gccagggcac aagactggag atcaag 816
<210> 236
<211> 813
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 236
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
cctgaggtgc agctggtgga gagcggcggc ggcctggtga agcctggcgg atccctgagg 120
ctgtcttgcg aggcaagcgg cttcaccttt agcgacgcat ggatgtcctg ggtgcgccag 180
gcccctggca agggactgga gtgggtggga cggatcaaga gcaagacaga cggcggcacc 240
acagattacg tggtgccact gaacggccgc ttcatcatct cccgcgacga ttctcggaat 300
accctgtatc tgcagctgaa caatctgaag acagaggata ccgccgtgta ctattgcacc 360
acagtgccag gctcctacgg ctattggggc cagggcacac tggtgaccgt gagctccggc 420
ggcggcggct ctggaggagg aggaagcgga ggaggaggaa gcgggggcgg cggctctgac 480
atcgtgatga cacagtctcc actgagcctg ccagtgaccc ctggcgagcc agcctccatc 540
tcttgtcgct ctagccagag cctgctgcac aacaagcgga acaattacct ggattggttt 600
ctgcagaagc ctggccagtc ccctcagctg ctgatctatc tggccagcaa tagagcctcc 660
ggagtgccag acaggttctc tggaggagga agcggaacag acttcaccct gaagatcagc 720
agagtggagg ccgaggacgt gggcgtgtac tattgcatgc aggcccagca gacacctatc 780
accttcggcc agggcacaag actggagatc aag 813
<210> 237
<211> 837
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 237
atggctctgc ccgtcaccgc tctgctgctg cctctggccc tgctgctgca cgcagcaagg 60
ccacaggtgc agctggtgca gtccggagca gaggtgaaga agcctggcag ctccgtgaag 120
gtgagctgca aggcctccgg cgacacattc tctagcaacg caatcagctg ggtgcgccag 180
gcccctggcc agggactgga gtggatgggc gtgatcatcc ctatcttcgg caccgccgac 240
tatgcccaga agtttcaggg ccgggtgaca atcaccgccg atgagtctac aagcaccgcc 300
tacatggagc tgtcctctct gagatccgag gacacagccg tgtactattg tgccaggcac 360
acctatcacg agtacgcagg aggatactat ggaggagcaa tggatccttg gggacagggc 420
acactggtga ccgtgagctc cggcggcggc ggctctggag gaggaggaag cggaggagga 480
ggaagcgggg gcggcggctc tgagctgcag agcgtgctga cccagccacc ttccgcctct 540
ggaacaccag gccagagggt gaccatcagc tgctccggat ctagctccaa catcggctcc 600
aattacgtgt attggtacca gcagctgcca ggcacagccc ccaagatcct gatctaccgc 660
aacaatcagc ggccttctgg cgtgccagat agattctctg gcagcaagtc cggcacctct 720
gccagcctgg caatctccgg cctgaggtct gaggacgagg ccgattacta ttgcgccgcc 780
tgggacgata acctgagcgg ctgggtgttt ggcacaggca ccaagctgac agtgctg 837
<210> 238
<211> 1476
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 238
atggctctgc ccgtcaccgc tctgctgctg cccctggctc tgctgctgca cgctgctcgc 60
cctgatgtgg tcatgactca gtctcccctg tctctgcccg tcaccctggg acagcccgcc 120
agcatctcct gcaagagctc ccagagcctg ctgtactcca acggcaagac ctatctgaat 180
tggttccagc agagacccgg ccagagccct cggagactga tctaccaggt gtctaagctg 240
gacagcggcg tgcctgatcg cttctctgga agcggatccg gaaccgactt tacactgaag 300
atcagccggg tggaggcaga ggacgtgggc gtgtactatt gcggccagga tacccacttc 360
ccactgacat ttggcggcgg caccaaggtg gagatcaagg gaggaggagg aagcggagga 420
ggaggaagcg gcggcggcgg ctctggcggc ggcggcagcc aggtgcagct ggtgcagagc 480
ggagcagagg tgaagaagcc tggcgcctcc gtgaaggtgt cttgtaaggc cagcggctac 540
acattcaccg attatacact gcactgggtg cggcaggccc ctggccaggg actggagtgg 600
atgggaggaa tctggcctat caccggagga accacataca accagaagtt taagggcaga 660
gtgacaatga ccagggacac atctaccagc acagtgtata tggagctgtc tagcctgcgc 720
tccgaggata cagccgtgta ctattgcgcc agaggcgagg cacagggatc ttggggacag 780
ggcaccctgg tgacagtgtc ctctaccaca accccagcac caagaccacc tacccctgca 840
ccaacaatcg cctcccagcc tctgtctctg cgcccagagg catgtaggcc agcagcagga 900
ggagcagtgc acaccagggg cctggacttt gcctgcgata tctacatctg ggcaccactg 960
gcaggaacat gtggcgtgct gctgctgagc ctggtcatca ccctgtactg caagcgcggc 1020
cggaagaagc tgctgtatat cttcaagcag cccttcatga gacccgtgca gacaacccag 1080
gaggaggacg gctgctcctg taggttccca gaagaagagg agggcggctg tgagctgaga 1140
gtgaagtttt ccaggtctgc cgatgcacca gcataccagc agggacagaa tcagctgtat 1200
aacgagctga atctgggcag gcgcgaggag tatgacgtgc tggataagag gagaggaagg 1260
gaccctgaga tgggaggcaa gcctaggcgc aagaacccac aggagggcct gtacaatgag 1320
ctgcagaagg ataagatggc cgaggcctat tccgagatcg gcatgaaggg cgagcggaga 1380
aggggcaagg gccacgacgg gctgtaccag ggactgtcaa ccgctaccaa ggatacttac 1440
gacgccctgc atatgcaggc actgcctcca aggtga 1476
<210> 239
<211> 1479
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 239
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
cctcaggtga ccctgaagga gagcggccct gtgctgctga agccaacaga gaccctgaca 120
ctgacctgca cagtgtctgg cttcagcctg tccaaccccc ggatgggcgt gagctggatc 180
agacagcccc ctggcaaggc cctggagtgg ttcgcccaca tcttttctac cgatgagaag 240
agcctgaagc tgtccctgag atctaggctg accctgagca aggacacatc taagagccag 300
gtggtgctga ccatgacaaa catggcccct gtggactccg ccacatacta ttgcgccaga 360
gacagctcca attacgaggg ctatttcgac ttttggggcc agggcaccct ggtgacagtg 420
tctagcggcg gaggaggatc cggaggagga ggatctggcg gcggcggctc cggcggcggc 480
ggctccgagg tggtgctgac ccagagccct gccacactgt ccgtgtctcc aggcgagaga 540
gccaccctgt cttgtagggc cagccagtcc gtgcgcagca atctggcctg gtaccagcag 600
aagtccggcc aggccccaag actgctgatc tatggctcca ccatcagggc cacaggagtg 660
ccagcacgct tctctggaag cggatccggc acagagttta ccctgacaat ctcctctctg 720
cagtccgagg atttcgccgt gtactattgc cagcagtact ctgactggcc cttcaccttt 780
ggccctggca caaaggtgga tatcaagacc acaacccctg caccaaggcc accaacccca 840
gcacctacaa tcgcaagcca gccactgtcc ctgagacccg aggcctgtag gcctgcagca 900
ggaggagcag tgcacacccg cggcctggac tttgcctgcg atatctatat ctgggcacca 960
ctggcaggaa cctgtggcgt gctgctgctg agcctggtca tcaccctgta ctgcaagcgc 1020
ggccggaaga agctgctgta tatcttcaag cagcccttca tgcggcccgt gcagacaacc 1080
caggaggagg atggctgctc ctgtagattc cctgaggagg aggagggagg atgtgagctg 1140
agggtgaagt tttctcggag cgccgacgca ccagcatacc agcagggaca gaaccagctg 1200
tataacgagc tgaatctggg ccggagagag gagtacgacg tgctggataa gaggagggga 1260
agagacccag agatgggagg caagccacgg agaaagaacc cccaggaggg cctgtacaat 1320
gagctgcaga aggataagat ggccgaggcc tattctgaga tcggcatgaa gggagagagg 1380
cgccggggca agggacacga cggactgtac cagggactgt ccaccgcaac aaaggacacc 1440
tatgatgccc tgcatatgca ggcactgcct ccaaggtga 1479
<210> 240
<211> 1479
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 240
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
ccacaggtga ccctgaagga gtccggcccc gtgctggtga agcctacaga gaccctgaca 120
ctgacctgca cagtgtccgg cttctctctg agcaacgccc gcatgggcgt gtcttggatc 180
aggcagcccc ctggcaaggc cctggagtgg ctggcccaca tcttttccac cgacgagaag 240
tctatccgga gaagcctgcg ctccaggctg accctgagca aggatacatc caagtctcag 300
gtggtgctga ccatgacaaa catggacccc gtggataccg ccacatactt ctgcgccaga 360
gacagctcca attacgaggg ctattttgat tactggggcc agggcaccct ggtgacagtg 420
tctagcggag gaggaggaag cggaggagga ggatctggcg gcggcggctc tggcggcggc 480
ggcagcgagg tggtcatgac ccagagccca gccacactga gcgtgtcccc tggcgagagg 540
gtgaccctgt cctgtagggc atctcagagc gtgtcctcta acttcgcctg gtatcagcag 600
agaccaggcc aggcaccaag gctgctgctg tacggagcaa ccacaagagc cacaggactg 660
cccggcaggt tttccggatc tggaagcggc accgagaata tcctgacaat cagctccctg 720
cagtctgagg acttcgccat ctatttttgc cagcagtaca aggattggcc attcaccttt 780
ggccccggca gcaaggtgga catcaagacc acaacccctg caccaagacc accaacccca 840
gcacctacaa tcgcctctca gcctctgagc ctgcgcccag aggcatgtag gccagcagca 900
ggaggagcag tgcacacaag gggcctggac ttcgcctgcg atatctatat ctgggcacct 960
ctggcaggaa cctgtggcgt gctgctgctg agcctggtca tcaccctgta ttgcaagaga 1020
ggcaggaaga agctgctgta catcttcaag cagcctttta tgcgcccagt gcagacaacc 1080
caggaggagg acggctgcag ctgtcggttc cctgaagagg aggagggcgg ctgtgagctg 1140
agagtgaagt tttccaggtc tgccgatgcc ccagcctatc agcagggcca gaatcagctg 1200
tacaacgagc tgaatctggg caggcgcgag gagtacgacg tgctggataa gaggagagga 1260
agggatccag agatgggagg caagcctagg cgcaagaacc cacaggaggg cctgtataat 1320
gagctgcaga aggacaagat ggccgaggcc tactccgaga tcggcatgaa gggagagcgg 1380
agaaggggca agggacacga tggcctgtat cagggcctgt ctaccgccac aaaggacacc 1440
tacgatgccc tgcatatgca ggcactgcct ccaaggtga 1479
<210> 241
<211> 1479
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 241
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaaga 60
cctcaggtga ccctgaagga gtccggccct gtgctggtga agccaacaga gaccctgaca 120
ctgacctgca cagtgtctgg cttcagcctg tccaacgcaa ggatgggcgt gagctggatc 180
aggcagcccc ctggcaaggc cctggagtgg ctgggccaca tctttagcac cgacgagaag 240
tcttacagca catccctgag aggcaggatc accatctcta aggatacaag cagaggcctg 300
gtggtgctga ccctgacaaa catggacccc gtggataccg ccacatacta ttgcgccagg 360
gacagctcca attacgaggg ctatttcgat ttttggggcc ctggcttcct ggtgaccgtg 420
tctagcggcg gcggcggctc tggaggagga ggaagcggag gaggaggatc cggcggcggc 480
ggctctgaga tcgtgatgac ccagtcccct gccacactgt ctgtgagccc aggcgagaga 540
gccaccctgt cttgtagggt gtcccagtct atcggcgcca atctggcctg gtaccagcag 600
aagttcggcc aggccccaag gctgctgatc tatggagcat ccaccagagc cacaggaatc 660
cccgtgaggt tctccggagg aggatctgga accgagttta ccctgacaat ctcctctctg 720
cagagcgagg actttgccat ctactcctgc cagcagtaca tctattggcc cttcacattt 780
ggccctggca ccacagtgga tatcaagacc acaacccctg caccaaggcc accaacccca 840
gcacctacaa tcgcaagcca gccactgtcc ctgagaccag aggcatgtag gcctgcagca 900
ggaggagccg tgcacaccag aggcctggac tttgcctgcg atatctatat ctgggcacca 960
ctggcaggaa cctgtggcgt gctgctgctg agcctggtca tcaccctgta ctgcaagcgc 1020
ggccggaaga agctgctgta tatcttcaag cagcccttca tgcgccccgt gcagacaacc 1080
caggaggagg acggctgcag ctgtcggttc cctgaagagg aggagggagg atgtgagctg 1140
agggtgaagt ttagccggtc cgccgatgca ccagcatacc agcagggcca gaaccagctg 1200
tataacgagc tgaatctggg ccggagagag gagtacgacg tgctggataa gaggagggga 1260
agagacccag agatgggagg caagccacgg agaaagaacc cccaggaggg cctgtacaat 1320
gagctgcaga aggacaagat ggccgaggcc tatagcgaga tcggcatgaa gggagagagg 1380
cgccggggca agggacacga tggcctgtac cagggcctgt ccaccgccac aaaggacacc 1440
tatgatgccc tgcatatgca ggcactgcct ccaaggtga 1479
<210> 242
<211> 1479
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 242
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaaga 60
ccacaggtga ccctgaagga gagcggaccc gtgctggtga agcctacaga gaccctgaca 120
ctgacctgca cagtgagcgg cttctccctg aacaatgcaa ggatgggcgt gtcctggatc 180
aggcagcccc ctggcaaggc cctggagtgg ttcgcccaca tctttagcac cgacgagaag 240
tcctttcgca catctctgag aagcaggctg accctgagca aggatacaag caagtcccag 300
gtggtgctga ccatgacaaa catggacccc gtggataccg ccacatacta ttgcgccaga 360
gacagctcca attacgaggg ctatttcgat tactggggcc agggcatcct ggtgaccgtg 420
tctagcggcg gcggcggctc tggaggagga ggaagcggag gaggaggatc cggcggcggc 480
ggctctgaga tcgtgatgac ccagtctccc gccacactgt ctgtgagccc tggcgagaga 540
gccacactga gctgtagggc ctcccagtct gtgagcaaca atctggcctg gtatcagcag 600
aagccaggcc aggcaccaag gctgctgatc tacggagcat ccaccagagc cacaggagtg 660
ccagcaaggt tctccggatc tgacagcggc accgagttta gcctgacaat ctcctctctg 720
cagtccgagg acttcgccgt gtatttttgc cagcagtaca aggattggcc attcaccttt 780
ggccccggca caaaggtgga gatcaagacc acaacccctg caccaagacc accaacccca 840
gcacctacaa tcgcatccca gcctctgtct ctgagaccag aggcatgtag gccagcagca 900
ggaggagcag tgcacaccag gggcctggac tttgcctgcg atatctatat ctgggcacct 960
ctggcaggaa cctgtggcgt gctgctgctg agcctggtca tcaccctgta ttgcaagcgc 1020
ggccggaaga agctgctgta catcttcaag cagcctttta tgcgcccagt gcagacaacc 1080
caggaggagg acggctgctc ctgtcggttc cctgaagagg aggagggagg atgtgagctg 1140
agggtgaagt tttcccggtc tgccgatgcc ccagcctatc agcagggcca gaaccagctg 1200
tacaacgagc tgaatctggg ccggagagag gagtacgacg tgctggataa gaggagggga 1260
agagatccag agatgggagg caagcctcgg agaaagaacc cacaggaggg cctgtataat 1320
gagctgcaga aggacaagat ggccgaggcc tactccgaga tcggcatgaa gggagagagg 1380
cgccggggca agggacacga tggcctgtat cagggcctgt ctaccgccac aaaggacacc 1440
tacgatgccc tgcatatgca ggcactgcct ccaaggtga 1479
<210> 243
<211> 1491
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 243
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
ccagaggtga acctggtgga gtccggcggc ggcctggtga agcctggcgg atccctgagg 120
ctgtcttgcg aggcaagcgg cttcaccttc agctacgcct ggatgtcctg ggtgcgccag 180
gcccccggca agggactgga gtgggtggga cggatcaagt ccatcgcaga cggaggagca 240
accgattacg cagcccctgt gagaaacagg ttcacaatct ccagagacga ttctaggaat 300
accctgtatc tggagatgca ctctctgaag acagaggaca ccgccgtgta ctattgcacc 360
acaatccctg gcaacgacgc ctttgatatg tggggccagg gcacaatggt gaccgtgagc 420
tccggcggcg gcggctctgg aggaggagga agcggaggag gaggaagcgg gggcggcggc 480
tctgacatcg tgctgacaca gtccccactg tccctgtctg tgacccccgg cgagcctgca 540
agcatctcct gtagatctag ccagagcctg ctgtactcca acggcaagaa ttatctggat 600
tggttcctgc acaagccagg ccagtctccc cagctgctga tctacctggg atctaatagg 660
gcaagcggag tgccagaccg gttctctgga agcggatccg gcatcgactt catcctgaag 720
atcagcaggg tggaggccga ggatgtgggc gtgtactatt gcatgcaggc ccagcagaca 780
cccatcacct tcggccaggg cacaagactg gagatcaaga ccacaacccc agcaccaagg 840
ccacctacac ctgcaccaac catcgcatcc cagccactgt ctctgaggcc tgaggcatgt 900
cggccagcag caggaggagc agtgcacacc cgcggcctgg actttgcctg cgatatctac 960
atctgggcac cactggcagg aacatgtggc gtgctgctgc tgagcctggt catcaccctg 1020
tactgcaagc gcggccggaa gaagctgctg tatatcttca agcagccttt tatgagacca 1080
gtgcagacaa cccaggagga ggacggctgc tcctgtaggt tccctgaaga ggaggagggc 1140
ggctgtgagc tgagagtgaa gttttctagg agcgccgatg caccagcata ccagcaggga 1200
cagaatcagc tgtataacga gctgaatctg ggccggagag aggagtatga cgtgctggat 1260
aagaggaggg gaagggaccc tgagatggga ggcaagcccc ggagaaagaa ccctcaggag 1320
ggcctgtaca atgagctgca gaaggacaag atggccgagg cctatagcga gatcggcatg 1380
aagggagaga ggcgccgggg caagggacac gatggcctgt accagggcct gtccacagcc 1440
accaaggaca cctatgatgc cctgcatatg caggcactgc ctccaaggtg a 1491
<210> 244
<211> 1491
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 244
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
ccagaggtga acctggtgga gtccggcggc ggcctggtga agcctggcgg atccctgagg 120
ctgtcttgcg aggcaagcgg cttcaccttc agctacgcct ggatgtcctg ggtgcgccag 180
gcccccggca agggactgga gtgggtgggc cggatcaagt ccatcaccga cggaggcgtg 240
atcgattacg cagcacctgt gagaaacagg tgcacaatct ccagagacga ttctaggaat 300
accctgtatc tggagatgca ctctctgaag acagaggaca ccgccgtgta ctattgtacc 360
acaatccctg gcaacgacga tttcgatatg tggggccagg gcagaatggt gaccgtgagc 420
tccggcggcg gcggctctgg aggaggagga agcggaggag gaggaagcgg gggcggcggc 480
tctgacatcg tgctgacaca gtccccactg tccctgtctg tgacccccgg cgagcctgca 540
agcatctcct gtaggtctag ccagagcctg ctgtactcca acggcaagaa ttatctggat 600
tggtttctgc acaagccagg ccagtctccc cagctgctga tctacctggg atctaatagg 660
gcaagcggag tgccagaccg gttctctgga agcggatccg gcatcgactt catcctgaag 720
atcagccgcg tggaggcaga ggacgtgggc gtgtactatt gcatgcaggc ccagcagaca 780
cccatcacct tcggccaggg cacaagactg gagatcaaga ccacaacccc agcaccaagg 840
ccacctacac ctgcaccaac catcgcatcc cagccactgt ctctgaggcc tgaggcatgt 900
aggccagcag caggaggagc agtgcacacc agaggcctgg actttgcctg cgatatctac 960
atctgggcac cactggcagg aacatgtggc gtgctgctgc tgagcctggt catcaccctg 1020
tactgcaagc gcggccggaa gaagctgctg tatatcttca agcagccttt tatgagacca 1080
gtgcagacaa cccaggagga ggacggctgc tcctgtaggt tccctgaaga ggaggagggc 1140
ggctgtgagc tgagagtgaa gttttctagg agcgccgatg caccagcata ccagcaggga 1200
cagaatcagc tgtataacga gctgaatctg ggccggagag aggagtatga cgtgctggat 1260
aagaggaggg gaagggatcc tgagatggga ggcaagcccc ggagaaagaa ccctcaggag 1320
ggcctgtaca atgagctgca gaaggacaag atggccgagg cctatagcga gatcggcatg 1380
aagggagaga ggcgccgggg caagggacac gatggcctgt accagggcct gtccacagcc 1440
accaaggaca cctatgatgc cctgcatatg caggcactgc ctccaaggtg a 1491
<210> 245
<211> 1488
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 245
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
ccagaggtgc agctggtgga gtcttggggc gtgctggtga agcctggcgg atctctgagg 120
ctgagctgcg cagcatccgg cttcatcttt aacaatgcct ggatgtcctg ggtgcgccag 180
gcccccggca agggactgga gtggatcggc cggatcaaga gcaagtccga cggaggaacc 240
acagattacg cagcacctgt gaaggaccgc ttcacaatct ctcgggacga tagcaaggat 300
accctgtatc tgcagatgaa cggcctgaag acagaggaca ccgccgtgta cttctgcacc 360
acagcccctg gcggcccttt tgattattgg ggccagggca cactggtgac cgtgagctcc 420
ggaggaggag gaagcggcgg aggaggcagc ggcggcggcg gctctggcgg cggcggcagc 480
gacatcgtgc tgacacagag ccctctgtcc ctgccagtga cccccggcga gcctgcctct 540
atcagctgtc gctctagcca gagcctgctg caccgggacg gcttcaatta cctggattgg 600
tttctgcaga agccaggcca gtccccccag ctgctgatct atctggcctc ctctagagcc 660
tctggcgtgc cagacaggtt ctccggctct gacagcggca cagacttcac cctgaagatc 720
agcagagtgg aggccgagga tgtgggcgtg tactattgca tgcaggccct gcagacaccc 780
atcaccttcg gccagggcac aagactggag atcaagacca caaccccagc accaaggcca 840
cctacacctg caccaaccat cgcatcccag ccactgtctc tgagacctga ggcctgtagg 900
ccagcagcag gaggagcagt gcacaccagg ggcctggact ttgcctgcga tatctacatc 960
tgggcacctc tggcaggaac atgtggcgtg ctgctgctga gcctggtcat caccctgtac 1020
tgcaagagag gcaggaagaa gctgctgtat atcttcaagc agccttttat gagaccagtg 1080
cagacaaccc aggaggagga cggctgctcc tgtaggttcc ctgaagagga ggagggagga 1140
tgtgagctga gggtgaagtt ttcccggtct gccgatgcac cagcatacca gcagggacag 1200
aaccagctgt ataacgagct gaatctgggc cggagagagg agtacgacgt gctggataag 1260
aggcgcggca gagatccaga gatgggcggc aagccccgga gaaagaaccc tcaggagggc 1320
ctgtacaatg agctgcagaa ggacaagatg gccgaggcct atagcgagat cggcatgaag 1380
ggagagaggc gccggggcaa gggacacgat ggcctgtacc agggcctgtc cacagccacc 1440
aaggacacct atgatgccct gcatatgcag gcactgcctc caaggtga 1488
<210> 246
<211> 1485
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 246
atggctctgc ccgtcaccgc tctgctgctg ccactggccc tgctgctgca cgcagcaagg 60
cctgaggtgc agctggtgga gagcggcggc ggcctggtga agcctggcgg atccctgagg 120
ctgtcttgcg aggcaagcgg cttcaccttt agcgacgcat ggatgtcctg ggtgcgccag 180
gcccctggca agggactgga gtgggtggga cggatcaaga gcaagacaga cggcggcacc 240
acagattacg tggtgccact gaacggccgc ttcatcatct cccgcgacga ttctcggaat 300
accctgtatc tgcagctgaa caatctgaag acagaggata ccgccgtgta ctattgcacc 360
acagtgccag gctcctacgg ctattggggc cagggcacac tggtgaccgt gagctccggc 420
ggcggcggct ctggaggagg aggaagcgga ggaggaggaa gcgggggcgg cggctctgac 480
atcgtgatga cacagtctcc actgagcctg ccagtgaccc ctggcgagcc agcctccatc 540
tcttgtcgct ctagccagag cctgctgcac aacaagcgga acaattacct ggattggttt 600
ctgcagaagc ctggccagtc ccctcagctg ctgatctatc tggccagcaa tagagcctcc 660
ggagtgccag acaggttctc tggaggagga agcggaacag acttcaccct gaagatcagc 720
agagtggagg ccgaggacgt gggcgtgtac tattgcatgc aggcccagca gacacctatc 780
accttcggcc agggcacaag actggagatc aagaccacaa ccccagcacc aaggccacct 840
acacctgcac caaccatcgc ctcccagcct ctgtctctga gaccagaggc atgtaggcca 900
gcagcaggag gagcagtgca caccaggggc ctggactttg cctgcgatat ctacatctgg 960
gcacctctgg caggaacatg tggcgtgctg ctgctgagcc tggtcatcac cctgtactgc 1020
aagagaggca ggaagaagct gctgtatatc ttcaagcagc ccttcatgag acccgtgcag 1080
acaacccagg aggaggacgg ctgctcttgt aggttcccag aagaggagga gggaggatgt 1140
gagctgaggg tgaagtttag ccggtccgcc gatgcaccag cataccagca gggacagaac 1200
cagctgtata acgagctgaa tctgggccgg agagaggagt acgacgtgct ggataagagg 1260
aggggaaggg atccagagat gggaggcaag cctcggagaa agaacccaca ggagggcctg 1320
tacaatgagc tgcagaagga caagatggcc gaggcctatt ctgagatcgg catgaaggga 1380
gagaggcgcc ggggcaaggg acacgatggc ctgtaccagg gcctgagcac agccaccaag 1440
gacacctatg atgccctgca tatgcaggca ctgcctccaa ggtga 1485
<210> 247
<211> 1509
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 247
atggctctgc ccgtcaccgc tctgctgctg cctctggccc tgctgctgca cgcagcaagg 60
ccacaggtgc agctggtgca gtccggagca gaggtgaaga agcctggcag ctccgtgaag 120
gtgagctgca aggcctccgg cgacacattc tctagcaacg caatcagctg ggtgcgccag 180
gcccctggcc agggactgga gtggatgggc gtgatcatcc ctatcttcgg caccgccgac 240
tatgcccaga agtttcaggg ccgggtgaca atcaccgccg atgagtctac aagcaccgcc 300
tacatggagc tgtcctctct gagatccgag gacacagccg tgtactattg tgccaggcac 360
acctatcacg agtacgcagg aggatactat ggaggagcaa tggatccttg gggacagggc 420
acactggtga ccgtgagctc cggcggcggc ggctctggag gaggaggaag cggaggagga 480
ggaagcgggg gcggcggctc tgagctgcag agcgtgctga cccagccacc ttccgcctct 540
ggaacaccag gccagagggt gaccatcagc tgctccggat ctagctccaa catcggctcc 600
aattacgtgt attggtacca gcagctgcca ggcacagccc ccaagatcct gatctaccgc 660
aacaatcagc ggccttctgg cgtgccagat agattctctg gcagcaagtc cggcacctct 720
gccagcctgg caatctccgg cctgaggtct gaggacgagg ccgattacta ttgcgccgcc 780
tgggacgata acctgagcgg ctgggtgttt ggcacaggca ccaagctgac agtgctgacc 840
acaacccctg caccaagacc accaacacca gcacctacca tcgcaagcca gccactgtcc 900
ctgagacccg aggcctgtag gcctgcagca ggaggagcag tgcacaccag gggcctggac 960
tttgcctgcg atatctatat ctgggcacca ctggcaggaa catgtggcgt gctgctgctg 1020
agcctggtca tcaccctgta ttgcaagaga ggcaggaaga agctgctgta catcttcaag 1080
cagcccttta tgcgccctgt gcagacaacc caggaggagg acggctgcag ctgtcggttc 1140
ccagaagagg aggagggagg atgtgagctg agggtgaagt tttcccggtc tgccgatgca 1200
ccagcatatc agcagggaca gaatcagctg tacaacgagc tgaatctggg ccggagagag 1260
gagtacgacg tgctggataa gaggagggga agggaccctg agatgggagg caagccacgg 1320
agaaagaacc cccaggaggg cctgtataat gagctgcaga aggacaagat ggccgaggcc 1380
tactctgaga tcggcatgaa gggagagagg cgccggggca agggacacga tggcctgtat 1440
cagggcctga gcacagccac caaggacacc tacgatgccc tgcatatgca ggcactgcct 1500
ccaaggtga 1509
<210> 248
<211> 529
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 248
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu
20 25 30
Val Lys Pro Thr Glu Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe
35 40 45
Ser Leu Asn Asn Ala Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro
50 55 60
Gly Lys Ala Leu Glu Trp Phe Ala His Ile Phe Ser Thr Asp Glu Lys
65 70 75 80
Ser Phe Arg Thr Ser Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr
85 90 95
Ser Lys Ser Gln Val Val Leu Thr Met Thr Asn Met Asp Pro Val Asp
100 105 110
Thr Ala Thr Tyr Tyr Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Ile Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser
165 170 175
Pro Gly Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser
180 185 190
Asn Asn Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu
195 200 205
Leu Ile Tyr Gly Ala Ser Thr Arg Ala Thr Gly Val Pro Ala Arg Phe
210 215 220
Ser Gly Ser Asp Ser Gly Thr Glu Phe Ser Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Ser Glu Asp Phe Ala Val Tyr Phe Cys Gln Gln Tyr Lys Asp Trp
245 250 255
Pro Phe Thr Phe Gly Pro Gly Thr Lys Val Glu Ile Lys Gly Ser Gly
260 265 270
Gly Gly Gly Ser Cys Pro Tyr Ser Asn Pro Ser Leu Cys Ser Gly Gly
275 280 285
Gly Gly Ser Cys Pro Tyr Ser Asn Pro Ser Leu Cys Ser Gly Gly Gly
290 295 300
Gly Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
305 310 315 320
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
325 330 335
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
340 345 350
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
355 360 365
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
370 375 380
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
385 390 395 400
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
405 410 415
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
420 425 430
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
435 440 445
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
450 455 460
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
465 470 475 480
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
485 490 495
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
500 505 510
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
515 520 525
Arg
<210> 249
<211> 529
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 249
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Gln Val Thr Leu Lys Glu Ser Gly Pro Val Leu
20 25 30
Val Lys Pro Thr Glu Thr Leu Thr Leu Thr Cys Thr Val Ser Gly Phe
35 40 45
Ser Leu Ser Asn Ala Arg Met Gly Val Ser Trp Ile Arg Gln Pro Pro
50 55 60
Gly Lys Ala Leu Glu Trp Leu Ala His Ile Phe Ser Thr Asp Glu Lys
65 70 75 80
Ser Ile Arg Arg Ser Leu Arg Ser Arg Leu Thr Leu Ser Lys Asp Thr
85 90 95
Ser Lys Ser Gln Val Val Leu Thr Met Thr Asn Met Asp Pro Val Asp
100 105 110
Thr Ala Thr Tyr Phe Cys Ala Arg Asp Ser Ser Asn Tyr Glu Gly Tyr
115 120 125
Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
130 135 140
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Glu Val Val Met Thr Gln Ser Pro Ala Thr Leu Ser Val Ser
165 170 175
Pro Gly Glu Arg Val Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser
180 185 190
Ser Asn Phe Ala Trp Tyr Gln Gln Arg Pro Gly Gln Ala Pro Arg Leu
195 200 205
Leu Leu Tyr Gly Ala Thr Thr Arg Ala Thr Gly Leu Pro Gly Arg Phe
210 215 220
Ser Gly Ser Gly Ser Gly Thr Glu Asn Ile Leu Thr Ile Ser Ser Leu
225 230 235 240
Gln Ser Glu Asp Phe Ala Ile Tyr Phe Cys Gln Gln Tyr Lys Asp Trp
245 250 255
Pro Phe Thr Phe Gly Pro Gly Ser Lys Val Asp Ile Lys Gly Ser Gly
260 265 270
Gly Gly Gly Ser Cys Pro Tyr Ser Asn Pro Ser Leu Cys Ser Gly Gly
275 280 285
Gly Gly Ser Cys Pro Tyr Ser Asn Pro Ser Leu Cys Ser Gly Gly Gly
290 295 300
Gly Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala Pro Thr
305 310 315 320
Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg Pro Ala
325 330 335
Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys Asp Ile
340 345 350
Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu Leu Ser
355 360 365
Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu Leu Tyr
370 375 380
Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu
385 390 395 400
Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu
405 410 415
Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln
420 425 430
Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu
435 440 445
Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly
450 455 460
Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln
465 470 475 480
Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu
485 490 495
Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr
500 505 510
Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro
515 520 525
Arg
<210> 250
<211> 532
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 250
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Val Glu Ser Trp Gly Val Leu
20 25 30
Val Lys Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
35 40 45
Ile Phe Asn Asn Ala Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Ile Gly Arg Ile Lys Ser Lys Ser Asp Gly Gly Thr
65 70 75 80
Thr Asp Tyr Ala Ala Pro Val Lys Asp Arg Phe Thr Ile Ser Arg Asp
85 90 95
Asp Ser Lys Asp Thr Leu Tyr Leu Gln Met Asn Gly Leu Lys Thr Glu
100 105 110
Asp Thr Ala Val Tyr Phe Cys Thr Thr Ala Pro Gly Gly Pro Phe Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly
130 135 140
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
145 150 155 160
Asp Ile Val Leu Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
165 170 175
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Arg
180 185 190
Asp Gly Phe Asn Tyr Leu Asp Trp Phe Leu Gln Lys Pro Gly Gln Ser
195 200 205
Pro Gln Leu Leu Ile Tyr Leu Ala Ser Ser Arg Ala Ser Gly Val Pro
210 215 220
Asp Arg Phe Ser Gly Ser Asp Ser Gly Thr Asp Phe Thr Leu Lys Ile
225 230 235 240
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
245 250 255
Leu Gln Thr Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
260 265 270
Gly Ser Gly Gly Gly Gly Ser Cys Pro Tyr Ser Asn Pro Ser Leu Cys
275 280 285
Ser Gly Gly Gly Gly Ser Cys Pro Tyr Ser Asn Pro Ser Leu Cys Ser
290 295 300
Gly Gly Gly Gly Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro
305 310 315 320
Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys
325 330 335
Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala
340 345 350
Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu
355 360 365
Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys
370 375 380
Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr
385 390 395 400
Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly
405 410 415
Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala
420 425 430
Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg
435 440 445
Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu
450 455 460
Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn
465 470 475 480
Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met
485 490 495
Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly
500 505 510
Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala
515 520 525
Leu Pro Pro Arg
530
<210> 251
<211> 531
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 251
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 10 15
His Ala Ala Arg Pro Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
20 25 30
Val Lys Pro Gly Gly Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Phe
35 40 45
Thr Phe Ser Asp Ala Trp Met Ser Trp Val Arg Gln Ala Pro Gly Lys
50 55 60
Gly Leu Glu Trp Val Gly Arg Ile Lys Ser Lys Thr Asp Gly Gly Thr
65 70 75 80
Thr Asp Tyr Val Val Pro Leu Asn Gly Arg Phe Ile Ile Ser Arg Asp
85 90 95
Asp Ser Arg Asn Thr Leu Tyr Leu Gln Leu Asn Asn Leu Lys Thr Glu
100 105 110
Asp Thr Ala Val Tyr Tyr Cys Thr Thr Val Pro Gly Ser Tyr Gly Tyr
115 120 125
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp
145 150 155 160
Ile Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly Glu
165 170 175
Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Asn Lys
180 185 190
Arg Asn Asn Tyr Leu Asp Trp Phe Leu Gln Lys Pro Gly Gln Ser Pro
195 200 205
Gln Leu Leu Ile Tyr Leu Ala Ser Asn Arg Ala Ser Gly Val Pro Asp
210 215 220
Arg Phe Ser Gly Gly Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile Ser
225 230 235 240
Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala Gln
245 250 255
Gln Thr Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Gly
260 265 270
Ser Gly Gly Gly Gly Ser Cys Pro Tyr Ser Asn Pro Ser Leu Cys Ser
275 280 285
Gly Gly Gly Gly Ser Cys Pro Tyr Ser Asn Pro Ser Leu Cys Ser Gly
290 295 300
Gly Gly Gly Ser Thr Thr Thr Pro Ala Pro Arg Pro Pro Thr Pro Ala
305 310 315 320
Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu Ala Cys Arg
325 330 335
Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp Phe Ala Cys
340 345 350
Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly Val Leu Leu
355 360 365
Leu Ser Leu Val Ile Thr Leu Tyr Cys Lys Arg Gly Arg Lys Lys Leu
370 375 380
Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln
385 390 395 400
Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly
405 410 415
Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr
420 425 430
Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg
435 440 445
Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met
450 455 460
Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu
465 470 475 480
Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys
485 490 495
Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu
500 505 510
Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu
515 520 525
Pro Pro Arg
530
<210> 252
<211> 1590
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 252
atggcactgc cagtgaccgc cctgctgctg cctctggccc tgctgctgca cgcagccaga 60
ccccaggtga cactgaagga gagcggcccc gtgctggtga agcctacaga gacactgacc 120
ctgacctgca cagtgagcgg cttctccctg aacaatgcaa ggatgggcgt gtcctggatc 180
aggcagccac ctggcaaggc cctggagtgg ttcgcccaca tctttagcac cgacgagaag 240
tcctttcgca catctctgag aagcaggctg accctgagca aggatacaag caagtcccag 300
gtggtgctga ccatgacaaa catggaccct gtggataccg ccacatacta ttgtgcccgg 360
gacagctcca attacgaggg ctatttcgat tactggggcc agggcatcct ggtgaccgtg 420
tctagcggcg gcggcggctc tggaggagga ggaagcggag gaggaggatc cggcggcggc 480
ggctctgaga tcgtgatgac ccagtcccca gccacactgt ctgtgagccc aggagagaga 540
gccaccctgt cttgcagggc ctcccagtct gtgagcaaca atctggcctg gtatcagcag 600
aagcctggcc aggccccaag gctgctgatc tacggagcaa gcaccagagc aacaggagtg 660
cctgcaaggt tctccggatc tgacagcggc accgagtttt ctctgacaat ctcctctctg 720
cagagcgagg acttcgccgt gtatttttgt cagcagtaca aggattggcc attcaccttt 780
ggccccggca caaaggtgga gatcaagggc tccggaggag gaggatcctg cccctattcc 840
aacccttctc tgtgcagcgg aggaggagga agctgtccat actccaatcc ctccctgtgc 900
tccggcggcg gaggatccac cacaacccca gcacctagac caccaacccc agcaccaaca 960
atcgcatccc agcctctgtc tctgcggccc gaggcatgca ggccagcagc aggcggcgcc 1020
gtgcacacca ggggcctgga ctttgcctgc gatatctata tctgggcacc actggcagga 1080
acctgtggcg tgctgctgct gagcctggtc atcaccctgt attgcaagcg cggccggaag 1140
aagctgctgt acatcttcaa gcagcctttt atgcgcccag tgcagacaac ccaggaggag 1200
gacggctgct cctgtcggtt ccctgaagag gaggagggag gatgtgagct gcgcgtgaag 1260
ttttcccggt ctgccgatgc cccagcctat cagcagggcc agaaccagct gtacaacgag 1320
ctgaatctgg gccggagaga ggagtacgac gtgctggata agaggagggg aagagatccc 1380
gagatgggag gcaagcctcg gagaaagaac ccacaggagg gcctgtataa tgagctgcag 1440
aaggacaaga tggccgaggc ctactctgag atcggcatga agggagagag gcgccggggc 1500
aagggacacg atggcctgta tcagggcctg tccaccgcca caaaggacac ctacgatgcc 1560
ctgcacatgc aggccctgcc tccaaggtga 1590
<210> 253
<211> 1590
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 253
atggcactgc cagtgaccgc cctgctgctg cctctggccc tgctgctgca cgcagccaga 60
ccccaggtga cactgaagga gtccggcccc gtgctggtga agcctacaga gacactgacc 120
ctgacctgca cagtgagcgg cttctctctg agcaacgcaa ggatgggcgt gtcctggatc 180
aggcagccac ctggcaaggc cctggagtgg ctggcccaca tcttttccac cgacgagaag 240
tctatccgga gaagcctgcg ctcccggctg accctgagca aggatacatc caagtctcag 300
gtggtgctga ccatgacaaa catggaccct gtggataccg ccacatactt ctgtgcccgg 360
gacagctcca attacgaggg ctattttgat tactggggcc agggcaccct ggtgacagtg 420
tctagcggag gaggaggaag cggaggagga ggatcaggcg gcggcggctc tggcggcggc 480
ggcagcgagg tggtcatgac ccagtctcca gccacactga gcgtgtcccc aggagagcgc 540
gtgaccctga gctgccgggc ctctcagagc gtgtcctcta acttcgcctg gtatcagcag 600
cggcccggac aggcaccaag gctgctgctg tacggagcaa ccacaagagc aacaggcctg 660
cctggcaggt tttccggctc tggcagcggc accgagaata tcctgacaat cagctccctg 720
cagagcgagg acttcgccat ctatttttgt cagcagtaca aggattggcc attcaccttt 780
ggccccggct ccaaggtgga catcaaggga tccggaggag gaggatcttg cccctattct 840
aaccctagcc tgtgctccgg aggaggagga tcctgtccat actctaatcc atccctgtgc 900
agcggaggag gaggatctac cacaacccca gcacctagac caccaacccc agcacccaca 960
atcgcctctc agcctctgag cctgcgccca gaggcatgca ggccagcagc aggaggagca 1020
gtgcacacca ggggcctgga cttcgcctgc gatatctata tctgggcacc actggcagga 1080
acctgtggcg tgctgctgct gtccctggtc atcaccctgt attgcaagag aggcaggaag 1140
aagctgctgt acatcttcaa gcagcctttt atgcgcccag tgcagacaac ccaggaggag 1200
gacggctgca gctgtcggtt ccctgaagag gaggagggcg gctgtgagct gagagtgaag 1260
ttttccaggt ctgccgatgc cccagcctat cagcagggcc agaatcagct gtacaacgag 1320
ctgaatctgg gcaggcgcga ggagtacgac gtgctggata agaggagagg acgcgatccc 1380
gagatgggag gcaagcctag gcgcaagaac ccacaggagg gcctgtataa tgagctgcag 1440
aaggacaaga tggccgaggc ctactctgag atcggcatga agggagagcg gagaaggggc 1500
aagggacacg atggcctgta tcagggcctg agcaccgcca caaaggacac ctacgatgcc 1560
ctgcacatgc aggccctgcc tccaaggtga 1590
<210> 254
<211> 1599
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 254
atggccctgc cagtgaccgc cctgctgctg ccactggccc tgctgctgca cgccgccaga 60
cctgaggtgc agctggtgga gagctggggc gtgctggtga agccaggagg ctctctgagg 120
ctgagctgcg cagcatccgg cttcatcttt aacaatgcct ggatgtcctg ggtgagacag 180
gcaccaggca agggcctgga gtggatcggc aggatcaaga gcaagtccga cggaggaacc 240
acagattacg cagcacccgt gaaggaccgc ttcacaatct ctcgggacga tagcaaggat 300
accctgtatc tgcagatgaa cggcctgaag acagaggaca ccgccgtgta cttctgcacc 360
acagccccag gcggcccctt tgattattgg ggccagggca cactggtgac cgtgagctcc 420
ggaggaggag gaagcggcgg aggaggcagc ggcggcggcg gctctggcgg cggcggcagc 480
gacatcgtgc tgacacagag cccactgtcc ctgcctgtga ccccaggaga gcccgcctct 540
atcagctgtc gctctagcca gagcctgctg caccgggacg gcttcaatta cctggattgg 600
tttctgcaga agcctggcca gagcccacag ctgctgatct atctggcctc ctctagagca 660
tccggagtgc ctgacaggtt ctccggatct gacagcggca cagacttcac cctgaagatc 720
tcccgcgtgg aggcagagga tgtgggcgtg tactattgca tgcaggccct gcagacacca 780
atcaccttcg gccagggcac acggctggag atcaagggat ccggaggagg aggatcttgc 840
ccctactcta accctagcct gtgcagcggc ggaggaggat cttgtccata ttctaatcca 900
agcctgtgca gcgggggagg aggaagcacc acaacccctg caccaagacc ccctacacca 960
gcacctacca tcgcatccca gccactgtct ctgcggcccg aggcatgtag gccagcagca 1020
ggaggagcag tgcacaccag gggcctggac tttgcctgcg atatctacat ctgggcacca 1080
ctggcaggaa catgtggcgt gctgctgctg agcctggtca tcaccctgta ctgcaagaga 1140
ggcaggaaga agctgctgta tatcttcaag cagcctttta tgcgcccagt gcagacaacc 1200
caggaggagg acggctgctc ctgtaggttc ccagaagagg aggagggagg atgtgagctg 1260
cgcgtgaagt tttcccggtc tgccgatgca cctgcatacc agcagggaca gaaccagctg 1320
tataacgagc tgaatctggg ccggagagag gagtacgacg tgctggataa gaggagggga 1380
cgcgatcctg agatgggagg caagccccgg agaaagaacc ctcaggaggg cctgtacaat 1440
gagctgcaga aggacaagat ggccgaggcc tattccgaga tcggcatgaa gggagagagg 1500
cgccggggca agggacacga tggcctgtac cagggcctgt ctacagccac caaggacacc 1560
tatgatgccc tgcacatgca ggccctgcca ccaaggtga 1599
<210> 255
<211> 1596
<212> DNA
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 255
atggcactgc cagtgacagc cctgctgctg cctctggccc tgctgctgca cgcagccaga 60
ccagaggtgc agctggtgga gtccggagga ggcctggtga agccaggagg ctccctgagg 120
ctgtcttgcg aggccagcgg cttcaccttt agcgacgcct ggatgtcctg ggtgagacag 180
gcaccaggca agggcctgga gtgggtgggc aggatcaaga gcaagacaga cggcggcacc 240
acagattacg tggtgcctct gaacggccgg ttcatcatct cccgcgacga ttctcggaat 300
accctgtatc tgcagctgaa caatctgaag acagaggata ccgccgtgta ctattgcacc 360
acagtgcctg gctcctacgg ctattggggc cagggcacac tggtgaccgt gagctccggc 420
ggcggcggct ctggaggagg aggaagcgga ggaggaggaa gcgggggcgg cggctctgac 480
atcgtgatga cacagtctcc actgagcctg ccagtgaccc caggagagcc tgcctccatc 540
tcttgtcgct ctagccagtc cctgctgcac aacaagcgga acaattacct ggattggttc 600
ctgcagaagc caggccagtc tccccagctg ctgatctatc tggccagcaa tagagcctcc 660
ggagtgccag acaggttctc tggaggagga agcggaacag acttcaccct gaagatcagc 720
cgcgtggagg cagaggacgt gggcgtgtac tattgcatgc aggcccagca gacacccatc 780
acctttggcc agggaacccg gctggagatc aagggctccg gaggaggagg atcctgccct 840
tactccaacc catctctgtg cagcggagga ggaggatctt gtccatattc caatccttcc 900
ctgtgctccg gaggaggagg aagcaccaca acccctgcac caagaccccc tacaccagca 960
cctaccatcg catcccagcc tctgtctctg cggcccgagg catgtaggcc agcagcaggc 1020
ggcgccgtgc acaccagggg cctggacttt gcctgcgata tctacatctg ggcaccactg 1080
gcaggaacat gtggcgtgct gctgctgtct ctggtcatca ccctgtactg caagagaggc 1140
aggaagaagc tgctgtatat cttcaagcag cccttcatgc ggcccgtgca gacaacccag 1200
gaggaggacg gctgcagctg tcggttccct gaagaggagg agggaggatg tgagctgcgc 1260
gtgaagttta gccggtccgc cgatgcacca gcataccagc agggccagaa ccagctgtat 1320
aacgagctga atctgggccg gagagaggag tacgacgtgc tggataagag gaggggacgc 1380
gatcctgaga tgggaggcaa gcctcggaga aagaacccac aggagggcct gtacaatgag 1440
ctgcagaagg acaagatggc cgaggcctat agcgagatcg gcatgaaggg agagaggcgc 1500
cggggcaagg gacacgatgg cctgtaccag ggcctgtcca cagccaccaa ggacacctat 1560
gatgccctgc acatgcaggc cctgccacca aggtga 1596
<210> 256
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Synthetic Construct
<400> 256
Cys Pro Tyr Ser Asn Pro Ser Leu Cys
1 5
- 还没有人留言评论。精彩留言会获得点赞!