一种来自棉花抗病耐旱蛋白基因GhSNAP33及其应用的制作方法
本发明设及一种来自棉花抗病、耐旱蛋白基因ghsnap33及其应用,属于植物基因工程领域。用于通过植物基因工程技术改善植物抗病、耐旱以及其它有益棉花生产的农艺性状。
背景技术:
植物本身无法主动逃避不利环境,为适应病害以及干旱胁迫,植物细胞不断调整并最终形成了特有的内膜系统和膜泡运输机制以完成细胞内和细胞间的物质交流。植物细胞内可溶性物质的运输主要是通过细胞内的囊泡在不同细胞器膜结构间穿梭运输而实现。植物细胞囊泡运输过程中的膜融合主要是由snare(solublen-ethyl-maleimidesensitivefactorattachmentproteinreceptor)蛋白介导的,snare蛋白定位于运输囊泡与靶位膜上,分为vesicle-membranesnare(v-snare)和target-membranesnare(t-snare),v-snare与t-snare之间的特异性识别与snare复合体的形成诱导膜融合,形成snare核心复合体,它是由q-snares和r-snares组成的4个snare结构域结合在一起形成的四螺旋束,其中3个螺旋是位于靶位膜上。除了在囊泡运输中至关重要的作用外,snare家族蛋白在植物生长发育和抵御生物胁迫和非生物胁迫中也有重要的作用。例如,(1)它们参与拟南芥细胞板的形成(heesem,ganselx,sticherl,wickp,grebem,granierf,etal.,2001,functionalcharacterizationoftheknolle-interactingt-snareatsnap33anditsroleinplantcytokinesis.jcellbiol.155(2):239-49;zhengh,bednareksy,sanderfootaa,alonsoj,eckerjr,raikhelnv,2002,npsn11isacellplate-associatedsnareproteinthatinteractswiththesyntaxinknolle.plantphysiol,129(2):530-9);(2)与离子通道蛋白相互作用,介导植物的逆境胁迫响应(grefenc,chenz,honsbeina,donaldn,hillsa,blattmr,2010,anovelmotifessentialforsnareinteractionwiththek+channelkc1andchannelgatinginarabidopsis.plantcell,22(9):3076-92;zhuj,gongz,zhangc,songcp,damszb,inang,etal.,2002,osm1/syp61:asyntaxinproteininarabidopsiscontrolsabscisicacid-mediatedandnon-abscisicacid-mediatedresponsestoabioticstress.plantcell,14(12):3009-28);(3)参与了多种植物对病原菌的防御反应(kaldem,nuhsets,findlayk,pecksc,2007,thesyntaxinsyp132contributestoplantresistanceagainstbacteriaandsecretionofpathogenesis-relatedprotein1.proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica.104(28):11850-5;luoj,zhangh,heww,zhangy,caowl,zhanghs,etal.,2016,ossnap32,asnap25-typesnareprotein-encodinggenefromrice,enhancedresistancetoblastfungus.plantgrowthregulation,80(1):37-45)。
突触体结合蛋白(snap25)来源于哺乳动物神经元,是第一个同时含有qb-和qc-snare结构域t-snare蛋白,它与另外两个snare蛋白相互作用介导神经元突触膜融合(oylerga,higginsga,hartra,battenberge,billingsleym,bloomfe,etal.,1989,theidentificationofanovelsynaptosomal-associatedprotein,snap-25,differentiallyexpressedbyneuronalsubpopulations.jcellbiol.109(61):3039-52)。因此,含有qb-和qc-串联结构域的snare家族蛋白也被称作为snap25类型的蛋白。atsnap33是第一个分离和鉴定的snap25同源蛋白基因,研究结果表明它参与了拟南芥细胞分裂过程中细胞板的形成(heesem,ganselx,sticherl,wickp,grebem,granierf,etal.,2001,functionalcharacterizationoftheknolle-interactingt-snareatsnap33anditsroleinplantcytokinesis.jcellbiol.155(2):239-49)而且atsnap33表达受到多种病害(peronosporaparasitica,pseudomonassyringaepvtomato)及机械损伤的诱导,参与依赖于sa和不依赖于sa的两种抗病信号途径(wickp,ganselx,ouleveyc,pagev,studeri,durstm,etal.,2003,theexpressionofthet-snareatsnap33isinducedbypathogensandmechanicalstimulation.plantphysiol.132(1):343-51)。ossnap32是水稻中snap25类型的蛋白,ossnap32可以受多种生物和非生物胁迫的诱导,参与了水稻对于稻瘟病的防御反应(luoj,zhangh,heww,zhangy,caowl,zhanghs,etal.,2016,ossnap32,asnap25-typesnareproteinencodinggenefromrice,enhancedresistancetoblastfungus.plantgrowthregulation.80(1):37-45.)。hvsnap34与ror2相互作用介导大麦在细胞壁水平上对与白粉病的抗性(collinsnc,thordal-christensenh,lipkav,baus,kombrinke,qiujl,etal.,2003,snareproteinmediateddiseaseresistanceattheplantcellwall.nature,425(6961):973-7.)。由kolle/pen1/syp122/syp132-snap33-
vamp721/722组成snare复合物是植物中一个默认的胞外分泌通路(bednarekp,kwonc,schulze-lefertp,2010,notaperipheralissue:secretioninplant-microbeinteractions.curropinplantbiol.,13,378-387)。它能够将植物抗病或抗逆所需的蛋白质或相关信号分子如pr1,pmr4(胼胝质合成酶)等分泌到胞外,提高植物对病害的抗性和对逆境的耐受性。
棉花是世界范围内种植的经济作物,随着气候逐渐变暖的变化,棉花黄萎病、干旱等非生物胁迫对棉花产量和质量造成了严重危害。由于棉花抗黄萎病、耐旱品种培育研究面临的难题之一是抗病基因、耐旱基因的缺乏,以及有限的棉花基因操作工具,所以棉花抗病基因功能的揭示仍旧非常具有挑战性。随着棉花基因组序列的可用性为后基因时代棉花基因的功能基因组分析奠定了基础。本发明ghsnap33是一个snap25类型的抗病、耐旱蛋白。结合沉默ghsnap33基因棉花植株及转ghsnap33基因植物的病害监测和抗旱水平分析,证明ghsnap33能显著提高植物对黄萎病抗性和对干旱胁迫的耐受性。
技术实现要素:
本发明的目的在于公开一种来自棉花抗病、耐旱蛋白基因ghsnap33。本发明公开ghsnap33在抗黄萎病方面的作用,提供基因ghsnap33抗病基因工程应用。本发明公开ghsnap33在植物抵御干旱胁迫方面的作用。ghsnap33基因转入植物,提高植物的抗病性和耐旱性,进行棉花品种分子改良。
本发明的目的可通过如下技术方案实现:
棉花基因ghsnap33,该基因的cdna序列如seqidno.1所示。
本发明所述的棉花基因ghsnap33编码的棉花抗病、耐旱蛋白,其氨基酸序列如seqidno.2所示。
含有所述的棉花基因ghsnap33的表达载体。
所述的表达载体优选将所述的棉花基因ghsnap33插入表达载体pcambia1300的xbai和sali酶切位点所得。
本发明所述的棉花基因ghsnap33的基因工程应用。
本发明所述的棉花ghsnap33基因在通过基因工程手段培育黄萎病抗性品种和耐干旱品种的应用。
利用所述的棉花ghsnap33基因在通过基因工程手段培育抗病、耐旱植物品种,通常先利用本发明ghsnap33基因作为目的基因构建植物表达载体,再将含ghsnap33基因的重组表达载体转化目标植物。
利用本发明ghsnap33基因作为目的基因构建植物表达载体,其中可用任何一种启动子,如花椰菜花叶病毒(camv)35s启动子、泛素(ubiquitin)启动子或其它诱导性启动子,表达载体中必要时可包括增强子。转化方法可用农杆菌介导法、基因枪法、花粉管通道法或其它方法转化植物。
棉花ghsnap33的基因工程应用,优选包括以下步骤:
1)提取陆地棉中植棉2号的总rna。
2)棉花基因ghsnap33的克隆
将步骤1)获得的总rna反转录成cdna第一链,以此链为模板,p1:seqidno.3和p2:seqidno.4为引物,用高保真酶进行pcr扩增,连接到pmdtm18-t载体,测序获得棉花基因ghsnap33的cdna序列seqidno.1;
3)植物表达载体的构建
根据棉花ghsnap33基因的cdna序列seqidno.1,设计扩增出完整编码阅读框的引物,并且在上游引物p3和下游引物p4上分别引物限制性酶切位点xbai和sali。引物序列分别为:p9:seqidno.10和p10:seqidno.11;以步骤2)中获得的阳性克隆质粒为模板,经pcr扩增后,回收pcr产物,将其连接到pcambia1300植物表达载体上,测序鉴定表达载体中编码区阅读框正确。
4)转基因植株的获得
将步骤3)获得的表达载体pcambia1300-35s::ghsnap33转入农杆菌,进一步转入拟南芥或新陆早33号(棉花品种),对获得的转基因植株进行定量pcr验证后进行植物抗黄萎病和干旱耐受性监测评价,得到抗黄萎病和耐干旱胁迫的品种。
本发明的有益效果为:
1.本发明公开了一种来自棉花抗病、耐旱蛋白基因ghsnap33及其所编码的蛋白质。棉花snare家族蛋白基因ghsnap33为棉花中首次报道。棉花胁迫处理分析表明ghsnap33基因受棉花黄伟病菌接种和peg6000模拟干旱处理明显诱导。因此作为目的基因导入植物,提高植物抗病性,以进行植物品种改良。
2.构建病毒诱导的基因沉默载体获得ghsnap33基因沉默的植株,ghsnap33基因转录水平明显下降的植株对黄萎病的抗性减弱。而构建ghsnap33基因过表达载体,获得过表达的转ghsnap33基因植株,对黄萎病的抗性明显增强,说明ghsnap33基因与棉花黄萎病抗性相关,具有基因工程应用价值。
3.过表达植株的干旱胁迫处理,表明过表达植株对干旱的耐受性明显增强。叶片脱水处理表明,转ghsnap33基因植株的叶片失水率显著低于对照组植株。说明ghsnap33基因与植物干旱胁迫响应机制有关,可利用基因工程技术提高植物的干旱耐受性。
4.本发明的ghsnap33基因来自棉花,具有适合于棉花等双子叶植物表达的优化密码子,其基因工程受体植物除了单子叶植物,如水稻、玉米、小米等单子叶植物以外,更加适合于棉花、大豆、烟草等双子叶植物。
附图说明
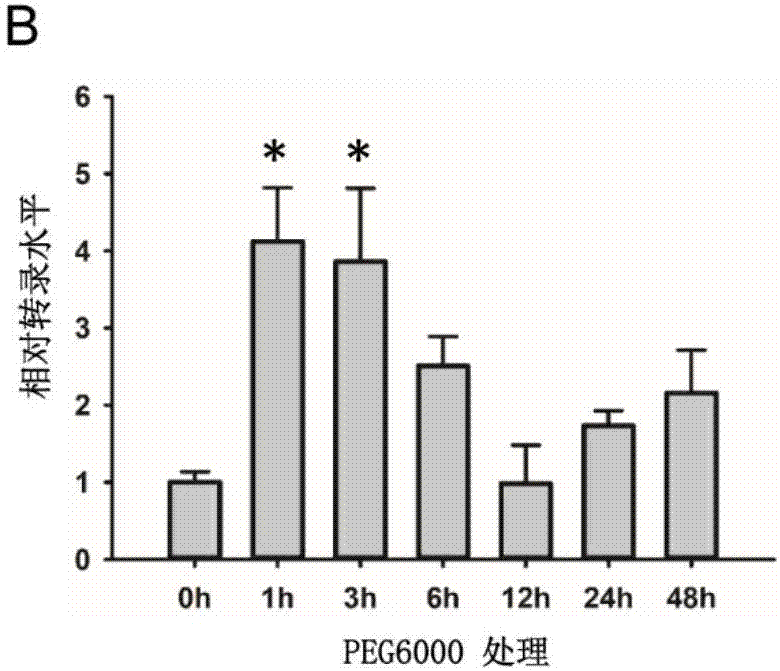
图1a-1b是棉花中ghsnap33的表达水平。其中,图1a表示黄萎病菌侵染后ghsnap33的表达水平;图1b表示peg6000模拟干旱条件下ghsnap33的表达水平。数据平均值代表3个生物学重复。误差线表示标准误差(se)。显著性分析*表示p<0.05,**表示p<0.01。
图2a-2b是病毒诱导的基因沉默后的棉花植株。其中,图2a表示基因沉默后棉花植株的表型;图2b表示半定量pcr鉴定棉花植株的基因沉默效率。
图3a-3d是ghsnap33沉默植株的病害严重性分析。其中,图3a是黄萎病菌侵染12天后的植株;图3b是黄萎病菌侵染12天后,棉花真叶的发病情况;图3c是黄萎病菌侵染14天后的病情指数;图3d是黄萎病菌侵染14天后,棉花茎的横切面图。数据平均值代表3个生物学重复。误差线表示标准误差(se)。显著性分析*表示p<0.05,**表示p<0.01。
图4a-4b是转基因拟南芥植株的鉴定。其中,图4a是ghsnap33在基因组水平的分析;图4b是ghsnap33转录水平的分析。m:dnamarker;l1-l13:不同的转基因株系;p:pcambia1300-ghsnap33质粒;wt:野生型对照。
图5a-5b是ghsnap33的亚细胞定位。其中,图5a是转基因拟南芥根部的ghsnap33-gfp荧光。图5b是质壁分离后,根部的ghsnap33-gfp荧光。箭头所示的是成功实现质壁分离的细胞。
图6a-6c是转基因拟南芥植株的黄萎病病害严重性分析。其中,图6a是接种黄萎病菌20天后的拟南芥植株;图6b是黄萎病菌接种12天和18天后,拟南芥植株的病情指数;图6c是接种20天后,植物中黄萎病菌的生物量。数据平均值代表3个生物学重复。误差线表示标准误差(se)。显著性分析*表示p<0.05,**表示p<0.01。
图7a-图7b是转基因棉花植株和非转基因棉花植株在病圃的病害抗性对比。其中,图7a为转基因棉株;图7b为非转基因棉株。
图8a-8c是转基因拟南芥植株的干旱耐受性分析。其中,图8a上面:正常生长条件下的拟南芥植株;中间:干旱胁迫20天后,拟南芥植株的生长状况;下面;覆水两天后,拟南芥植物的生长情况;图8b为正常生长条件和干旱胁迫覆水两天后,拟南芥植株的成活率。图8c为拟南芥地上部分的失水率。
具体实施方式
实施例1棉花ghsnap33基因的克隆
1)本发明中的棉花品种是来自陆地棉中植棉2号(我国审定的高抗枯、黄萎病的品种,可作为抗病虫害基因克隆的合适材料)。将棉花种植播种于营养土:蛭石1:1混合介质中,用塑料薄膜在花盆表层保持营养介质处于湿润状态,在25℃培养一周后,移去薄膜,继续培养2周。收集棉花幼苗样品,采用easyspin植物rna快速提取试剂盒(博迈德)提取rna,保存于-80℃备用。
2)采用原位杂交技术筛选出棉花ghsnap33基因的cdna序列,经酶切鉴定,获得阳性克隆,鉴定为snap33基因部分序列。检索棉花基因组数据库,获得915bp的ghsnap33基因的全长序列。设计全长两端引物p1和p2。
p1:5’-atgtcgggtttaaagagatc-3’(seqidno.3),
p2:5’-tctttccgagtaatcgacga-3’(seqidno.4),
将步骤1)获得总rna采用高效反转录酶fastquantrtenzyme(天根)合成的cdna为模板,以p1和p2为引物,采用选用taqdna聚合酶(天根)扩增出目的基因。pcr反应条件:94℃预变性5min;94℃变性30s,52℃复性30s,72℃延伸60s,33个循环;72℃延伸10min。将产物片段采用pmdtm18-tvectorcloningkit(大连宝生物)连接到pmdtm18-t载体,委托北京诺赛基因组研究中心有限公司测序获得ghsnap33的cdna序列seqidno.1。
3)分析上述获得的棉花ghsnap33的cdna序列seqidno.1及其编码蛋白序列seqidno.2。该基因编码的蛋白氨基酸序列。
实施例2ghsnap33在棉花中的诱导表达分析
用实施例1中的方法培育2周大小的棉花幼苗,将其轻轻从土里移出,用清水洗干净后,吸干水分。用1×106孢子/ml棉花黄萎病菌vd991侵染棉花幼苗处理,收集侵染后0、0.5、12、24、72、120和168h样品保存备用;同时,用10%(w/v)peg6000模拟干旱处理,收集处理0、1、3、6、12、24和48h样品保存备用。用实施例1中方法提取不同处理不同时间点的rna,合成cdna。设计real-timepcr引物p3和p4。
p3:5’-gctgaggagactacaaagactg-3’(seqidno.5),
p4:5’-acatacctccaaggcttccaag-3’(seqidno.6),
按照
p5:5’-gaaggcattccacctgaccaac-3’(seqidno.7),
p6:5’-cttgaccttcttcttcttgtgcttg-3’(seqidno.8),
采用2-δδct算法进行ghsnap33转录水平分析。结果表明,接种黄萎病菌vd99124h后,ghsnap33表达显著升高,然后逐渐下降(图1a);peg6000处理1h和3h时,ghsnap33表达增强而后逐渐下降(图1b)。表明ghnsap33表达与黄萎病侵染和干旱胁迫有关。
实施例3基因沉默载体构建和ghsnap33基因沉默植株的抗病性分析
设计棉花基因沉默载体引物p7和p8。
p7:5’-cgacgacaagaccgtgaccatgactgtcaataactgtgtg-3’(seqidno.9),
p8:5’-gaggagaagagccgtcattagagtaatcgacgagcacgttg-3’(seqidno.10),
以实施例1中cdna为模板,扩增沉默所需的基因片段。利用载体融合技术将目的片段连接到ptrv2载体上。选择序列正确的克隆转化农杆菌gv3010。按照病毒诱导基因沉默的方法(gaox,wheelert,liz,kenerleycm,hep,shanl,2011,silencingghndr1andghmkk2compromisescottonresistancetoverticilliumwilt.plantj.66,293-305.)进行ghsnap33表达下调的棉花植株培育(图3a)。用棉花ghubq为参照基因,进行基因沉默效率鉴定,结果表明在基因沉默之后,ghsnap33和ghcla1基因的转录水平都明显下降(图3b)。
ghsnap33基因沉默棉株抗病性检测。在ghsnap33基因沉默两周后,用蘸根法对棉花植株接种黄萎病菌vd991。在黄萎病菌接种2周后,调查植株真叶的发病情况,并计算病情指数(diseaseindex,di)。病情分级标准:0级:健株,无任何病症;1级:1片真叶变黄或坏死;2级:两片真叶变黄或坏死;3级:3片真叶变黄或坏死;4级:全株真叶变黄或坏死,甚至光杆或死亡。计算鉴定材料的相对病情指数。病情指数=[σ(各级病株数×相应病级)/调查总株数×最高病级(4)]×100。结果表明ghsnap33沉默的棉花植株对黄萎病菌更加敏感(图3a和b)。ghsnap33沉默植株的病情指数显著提高(图3c),棉花茎部维管组织褐化水平明显加深(图3d)。
实施例4转ghsnap33基因植物获得
设计带xbai和sali酶切位点的正反向引物。
p9:5’-aggctctagaatgtcgggtttaaagagatc-3’(seqidno.11),
p10:5’-aagcgtcgactctttccgagtaatcgacga-3’(seqidno.12),
实施例1中获得的阳性pmdtm18-t-ghsnap33质粒为模板,进行pcr反应。pcr反应条件:94℃预变性5min;94℃变性30s,52℃复性30s,72℃延伸60s,33个循环;72℃延伸10min。将pcr产物酶切回收连接构建到启动子为camv35s植物表达载体pcambia1300。通过农杆菌gv3010介导的花粉管通道转化法,将ghsnap33基因整合到拟南芥或棉花植株基因组中,从转基因拟南芥筛选到11个转ghsnap33基因超表达拟南芥株系(图4a),选取7株进行rt-pcr鉴定表明ghsnap33在植物中能够正确表达(图4b)。
实施例5ghsnap33的亚细胞定位
ghsnap33不含信号肽,没有跨膜域。借助转基因拟南芥ghsnap33-gfp融合蛋白进行激光共聚焦扫描显微镜观察,发现在ghsnap33过表达的拟南芥根部,绿色荧光信号在细胞膜或细胞壁上(图5a)。通过质壁分离,结果证明ghsnap33定位在植物细胞膜(图5b)。
实施例6转ghsnap33基因植物的抗病性检测
(1)利用蘸根法对拟南芥进行黄萎病菌接种。在黄萎病菌接种两周后调查病情指数。病情分级标准:0级:健株,无任何病症;1级:25%叶片变黄或坏死;2级:25%-50%叶片变黄或坏死;3级:50%-75%叶片黄或坏死;4级:75%-100%叶片变黄或坏死,甚至植株死亡。计算鉴定材料的相对病情指数。病情指数=[σ(各级病株数×相应病级)/调查总株数×最高病级(4)]×100,结果表明与野生型植株相比,ghsnap33转基因拟南芥植物的抗病性增强(图6a),病情指数明显降低(图6b)。设计引物p9和p10鉴定黄萎病菌。设计内参基因atef1-α引物p11和p12。
p11:5’-aaagttttaatggttcgctaaga-3’(seqidno.13),
p12:5’-cttggtcatttagaggaagtaa-3’(seqidno.14),
以已接种病原菌植株的基因组为模板,采用上述引物进行real-timepcr,评估不同植物系中的黄萎病菌的生物量。结果表明,转ghsnap33基因植物中留存的黄萎病原菌生物量显著低于野生型(图6c)。
(2)在病圃进行转ghsnap33基因棉花抗病性监测。与非转基因植株相比,转基因棉花植株的病害严重性明显减弱(图7a-7b)。
实施例7转ghsnap33基因植物的干旱耐受性检测
(1)转ghsnap33拟南芥的干旱耐受性检测
对3周大小的野生型和转基因拟南芥植物停止浇水,进行干旱胁迫处理。在干旱胁迫20d后,转基因植株表现出明显的耐干旱性状(图8a)。覆水两天后,转ghsnap33基因植株的成活率明显高于对照植株(图8b)。取4周大小的野生型和转基因拟南芥植株地上部分进行脱水试验,检测在脱水后0、1、2、3、4、5和6h后,不同植株的脱水率;结果表明转ghsnap33基因植株的失水率显著低于野生型(图8c)。
(2)转ghsnap33基因棉花的抗旱分析
选用t2代25d的转ghsnap33基因棉花株系(t-1、t-3)和未转基因棉花干旱胁迫(未浇水10d)后测定叶片中的相对水分含量,方法如lv等(2007)所述。生长4周的转ghsnap33基因棉花和野生型棉花对于脱水处理表现出不同的性状,缺水10d野生型棉花已表现出叶子枯黄症状,而转基因棉花没有异常。响应干旱胁迫,转ghsnap33基因棉花的初生根比野生型长,次生根也更加茂盛。10d干旱处理后,转基因棉花的相对含水量rwc减少小于野生型,如表1所示。
表1干旱处理条件下的棉花叶片中的相对水分rwc的含量
注:干旱胁迫10d后的测定值.*代表与wt相比存在显著性差异(p<0.05)
选取4周的转ghsnap33基因和对照棉花植株进行干旱处理,并采用bates等(1963)方法进行脯氨酸含量的测定。叶片用3%磺基水杨酸沸水浴中煮沸10min,提取溶液用茚三铜的方法在520nm波长下测定。采用l-脯氨酸做标准曲线,计算不同株系中的脯氨酸含量μgg-1fw。在干旱胁迫10d后,转ghsnap33基因株系比野生型的脯氨酸水平上升12倍,高于野生型(表2)。
表2干旱胁迫下转基因和野生型棉花叶片中脯氨酸的含量
注:干旱胁迫10d后的测定值.*代表与wt相比存在显著性差异(p<0.05)。
序列表
<110>中国农业大学
<120>一种来自棉花抗病耐旱蛋白基因ghsnap33及其应用
<160>2
<170>siposequencelisting1.0
<210>1
<211>915
<212>dna
<213>gossypiumhirsutum
<400>1
atgtcgggtttaaagagatctccattgaagactgtgaagctcaattctgtagatcctggg60
cgtgttgctgctcagaccaacccttttgattccgatgatgaattggacaacatgcaaacc120
cttaaacctaccagaacaacttcttctaagcctaccttgaatcctccaaatttcaggacc180
aatccgtttgatgatgatgaagagaaagtgaactcttcttcttcctcttcgtattggcaa240
acttcggcattgcagaacaagtataagaatgatttccgtgactgtggaggaattgagaac300
caaccagtgcaagaattggagaactatgctgtctacaaggctgaggagactacaaagact360
gtcaataactgtgtgaagattgcagaagaaatgagagaaggtgccaccaagactttgatt420
accttacatcagcagggtgagcaaattaccaggacacatgatgcggctgctggcatcgat480
catgaccttagtatgggtgagaagcttcttggaagccttggaggtatgttctctaaaact540
tggaagccaaagaagactcgtgcaatcattggccccattgtcacaagagatgattcacaa600
agaaggggtgcccatttggaacaaagggagaagcttggattgactcctgttcctcatgga660
ggttcaaagtctcgaacaccacccccggagcccacaaatacatctcaaaaagttgaggtt720
gaaaaatcaaagcaagacattgccctctccgatttaagtgatctactaggagaactgaag780
gatatggccattgacatgggatctgaaattgagaggcaaaacaaaagtctgaatggtctc840
caagatgacgtggatgagttaaatttccgagtccaaggtgcaaaccaacgtgcgcgtcga900
ttactcggaaagtag915
<210>2
<211>304
<212>prt
<213>gossypiumhirsutum
<400>2
metserglyleulysargserproleulysthrvallysleuasnser
151015
valaspproglyargvalalaalaglnthrasnpropheaspserasp
202530
aspgluleuaspasnmetglnthrleulysprothrargthrthrser
354045
serlysprothrleuasnproproasnpheargthrasnpropheasp
505560
aspaspgluglulysvalasnsersersersersersertyrtrpgln
65707580
thrseralaleuglnasnlystyrlysasnasppheargaspcysgly
859095
glyilegluasnglnprovalglngluleugluasntyralavaltyr
100105110
lysalaglugluthrthrlysthrvalasnasncysvallysileala
115120125
gluglumetarggluglyalathrlysthrleuilethrleuhisgln
130135140
glnglygluglnilethrargthrhisaspalaalaalaglyileasp
145150155160
hisaspleusermetglyglulysleuleuglyserleuglyglymet
165170175
pheserlysthrtrplysprolyslysthrargalaileileglypro
180185190
ilevalthrargaspaspserglnargargglyalahisleuglugln
195200205
argglulysleuglyleuthrprovalprohisglyglyserlysser
210215220
argthrproproprogluprothrasnthrserglnlysvalgluval
225230235240
glulysserlysglnaspilealaleuseraspleuseraspleuleu
245250255
glygluleulysaspmetalaileaspmetglysergluilegluarg
260265270
glnasnlysserleuasnglyleuglnaspaspvalaspgluleuasn
275280285
pheargvalglnglyalaasnglnargalaargargleuleuglylys
290295300
- 还没有人留言评论。精彩留言会获得点赞!