曼氏无针乌贼Spef1及其用途的制作方法
本发明涉及基因工程技术领域,尤其是涉及曼氏无针乌贼spef1及其用途。
技术背景
曼氏无针乌贼(sepiellajaponica)隶属于软体动物门(mollusca)、头足纲(cephalopoda)、十腕目(decapoda)、乌贼科(sepiidae)、无针乌贼属(sepiella),又名日本无针乌贼、墨鱼。作为曾经的东海“四大海产”之一,是一种重要的经济头足类物种。上世纪80年代以来人为的过度捕捞以及海洋生态环境的逐步恶化,导致其自然种群资源已经遭到严重破坏。众多学者对曼氏无针乌贼的资源分布、生物学特性、繁殖生物学以及增养殖方面进行了大量的研究,这些基础性的研究对于人工育苗奠定了坚实的基础,积累了丰富的经验。近年来在沿海各地开展的人工规模化养殖以及增殖放流已初见成效,并创造了一定的生态效益,目前已经具备规模化人工繁育技术,并已形成一定规模的曼氏无针乌贼养殖产业。
众所周知,生物体的繁殖是使种群不断延续的重要过程,精卵细胞的结合又包含了很多复杂的生物化学反应,众多的生物化学因子在这个过程中发挥独特的作用并直接或者间接的影响着受精过程的顺利进行。精子鞭毛蛋白1(spef1)是存在于雄性个体生殖系统中的特异性蛋白,截至目前的研究认为spef1可能参与了精子的发生,尤其是在精子鞭毛结构的形成以及组装过程中发挥了重要作用。
技术实现要素:
本发明的目的之一在于提供曼氏无针乌贼精子鞭毛蛋白spef1,分子生物学手段分析了曼氏无针乌贼精子鞭毛蛋白spef1的基因序列,通过基因表达组织特异性分析、基因组织表达定位分析研究了spef1的表达与定位,分析过程中组织切片与原位杂交是对组织与细胞的固定效果较好,提高了表达与定位的精确度。
本发明的目的之二在于提供曼氏无针乌贼精子鞭毛蛋白spef1的用途,spef1在精巢精小叶、初级精母细胞以及次级精母细胞中均由显著的表达,因此spef1可用于在头足类养殖过程中利用精子鞭毛蛋白及其拮抗剂进行生殖调控。
本发明针对
背景技术:
中提到的问题,采取的技术方案为:
曼氏无针乌贼精子鞭毛蛋白spef1基因的cdna全长共1135bp,其中5′utr178bp,3′utr165bp,开放阅读框(orf)区共792bp,编码263个氨基酸;编码蛋白相对分子质量为30.5677kda,等电点(pi)为7.03。spef1蛋白无跨膜结构,不含信号肽序列,因此曼氏无针乌贼精子鞭毛蛋白spef1不是分泌蛋白。
曼氏无针乌贼精子鞭毛蛋白spef1基因的cdna全序列为:
gggcaaatttgcaaagtgctcgcttcgaaaagacacaaataattttcttctaaaaaatagtatcaattgatactaaatttaaaattaaacacggaccaaggtttaatgttgacattatttactaattagaatttaaatataaataataattgcatacaataattaccagacatcaaccatggaattagatgatgacactttggaaaatatatacagttgggttgaccaacatcaactttctcgaccaaaaaggaatattgcaagagattttagtgacggagtcttaattgctgagattatcaaatgtcaccaaccaaaattagtggaaattcataattatactccagccaatgctactaaaccaaaaatggaaaattggtaccttcttaacaggcgagtatttgccaaaatgaaattcgaattggctgatgagattattcgttcagttgtgcagtgcaaaccatttgccattgaaagagttttattactgcttcaaagtcgtcttgaagagatgagccataaagaggaattaagtactatcaatttgaggatggagaaagatatccaggatgggaatacatctggtgctgcaagtcgtacaaaaccccctcaagtgcaacgtccagaccaacgagcattgcctaatcagaaagaaaggccacagcaatcggttcctttgagaagtgatcgcccagaagcagatcagggaatttatgataattatggccgttcagaaaaagagctcggagcatcttcacaagagtacagttcacagaagaagaagatggctggaattgatctgagagaatcaaatgttgttcctcgttcattacttgaagaaaaacaaaaagaaacacttgccaaagatgaaacaattgctattcttcaatccaagattcaacgtttggaatacttaaaccatttgaaagatttgagaattgaagatctacaaaagcatatacctaactgatgtttccattttccatgagcagtttctgctctttgaccgattcctacattctggattgacagaattttagaatgtattattccattacacaaatgcattcttctaaaatctttttttttttaaattgatgagccttaaaggggaaaaaaaaaaaaaaaaaaaaaa。
曼氏无针乌贼精子鞭毛蛋白spef1可参与调控曼氏无针乌贼生殖活动的分子生物学分析步骤包括:spef1基因克隆和表达分析、spef1基因表达组织特异性分析、spef1基因的组织表达定位分析,具体包括以下步骤:
一、spef1基因克隆和表达分析:
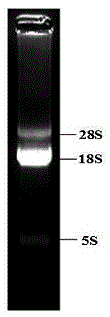
选取成熟健康的曼氏无针乌贼,用trizol试剂从曼氏无针乌贼的脑组织中提取总rna,将总rna利用核酸蛋白检测仪测定其在260nm以及280nm处的吸收波长,260/280值在1.8~2.0之间显示所提取的rna质量较纯,说明总rna有效;测定完成后,取2~3ml上样于1%琼脂糖凝胶电泳,电泳条件设定为135v和150ma,电泳15min,在凝胶成像分析仪中观察rna电泳条带,提取质量较好的rna能够观察到明显的28s和18s条带,亮度之比为1.9~2.1:1;使用m-mlvreversetranscriptase试剂盒,以总rna为模板,合成制备cdna第一链,以cdna为模板,以spef1-1f、spef1-1r为引物进行spef1基因核心片段的克隆与扩增,用1%琼脂糖凝胶电泳检测pcr扩增产物,对纯化后的目标片段按照试剂盒操作进行回收;分别以总rna获得扩增3′race、5′race所需的模板cdna,分别经过pcr扩增、检测、回收、连接、测序制得3′端、5′端序列,利用dnaman软件将spef1基因核心序列以及3′race和5′race得到的部分片段进行拼接得到曼氏无针乌贼精子鞭毛蛋白spef1基因全长及序列。
使用orffinder在线工具预测基因的开放阅读框(orf)位置,然后用dnaman软件将orf区核苷酸序列翻译为相应的氨基酸序列,预测spef1蛋白的相对分子质量和等电点使用的是在线工具expasy-protparam,氨基酸亲/疏水性分析采用在线工具expasy-protscale;netphos3.1server进行氨基酸磷酸化位点分析,scratchproteinpredictor分析氨基酸序列中二硫键位置,蛋白质二级结构预测采用antheprot5.0软件分析,应用在线工具signaip对编码的氨基酸序列进行信号肽预测,并用tmhmmserverv.2.0进行跨膜区结构预测。应用ncbi在线工具对spef1蛋白的氨基酸序列进行blastx和blastn分析,并用clustalw2对spef1蛋白及其同源氨基酸序列进行比对,保守结构域分析采用的是ncbi的在线搜索工具conserveddomainsearch。系统进化分析所选取的其它44条sp17同源氨基酸序列均来自于ncbi数据库,使用mega5.0软件比对并用clustalw功能对这些氨基酸序列进行初步的处理,然后使用phylogeny.fr在线网站的gblooks功能对氨基酸序列进行保守氨基酸的分析,导出相应结果后,应用modelgenerator软件对氨基酸替代模型进行预测,确定进化树模型参数,最后使用mrbayes软件基于贝叶斯理论构建系统进化树。pest序列分析采用在线网站pestfind工具。最后利用在线工具swissmodel采用同源建模的方法构建其蛋白部分序列的三维空间模型。
克隆spef1基因核心片段以及3′与5′端序列所需的引物为:
spef1-1f:agccaatgctactaaacc
spef1-1r:tgattaggcaatgctcg
3′-spef1-inner:ctcaagtgcaacgtccagac
3′-spef1-outer:ggtgctgcaagtcgtacaaa
3′-adptor:gcgagcacagaattaatatttttttttttt
5′-spef1-inner:gcaaatactcgcctgtta
5′-spef1-outer:aattcctctttatggctca
5′-adptor:ggccaggcgtcgactagtacgggggggggg
曼氏无针乌贼精子鞭毛蛋白spef1基因的cdna全长共1135bp,其中5′utr178bp,3′utr165bp,开放阅读框(orf)区共792bp,编码263个氨基酸;编码蛋白相对分子质量为30.5677kda,等电点(pi)为7.03。spef1蛋白无跨膜结构,不含信号肽序列,因此曼氏无针乌贼精子鞭毛蛋白spef1不是分泌蛋白。曼氏无针乌贼精子鞭毛蛋白spef1蛋白结构中含有49%的螺旋结构(heilx),5%的折叠结构(sheet),7%的转角结构(turn)以及38%的无规则卷曲结构(coil)。位于n端的14个左右的氨基酸是作为结合肌动蛋白的区域,该蛋白中ch2结构域主要由四个螺旋结构组成(helixⅰ、ⅱ、ⅲ、ⅳ),其中的三个螺旋结构helixⅱ、ⅲ以及ⅳ构成了比较宽松的一个螺旋结构束,彼此之间由三个loop结构串联起来。
曼氏无针乌贼spef1与加州双斑蛸(xp_014768591.1)、太平洋牡蛎(ekc38895.1)、眼斑雀鳝(lepisosteusoculatusxp_006629185.1)、矛尾鱼(latimeriachalumnaexp_014349819.1)相似性分别为59.49%、36.45%、39.45%和40.52%,而且该蛋白在进化中并不是很保守。保守结构域分析发现,不同物种的spef1氨基酸序列中均在靠近n端的位置处存在一个ch2结构域,如图5所示,该结构在进化中比较保守。
对44条spef1同源氨基酸序列构建进化树之前所有序列经过mega5.0软件比对以及gblooks在线工具整理,并使用modelgenerator软件预测最佳的氨基酸替代模型,最后采用mrbayes软件基于贝叶斯理论分析其系统进化关系,如图6所示。结果表明在进化上曼氏无针乌贼与加州双斑蛸的亲缘关系最近,其次是太平洋牡蛎,与人以及大猩猩的进化关系最远。
二、spef1基因表达组织特异性分析:
取健康无损、活力较强的熟雄性曼氏无针乌贼活体解剖,取13个组织包括:脑、视叶、胃、鰓、肝、心、精巢、输精管、储精囊、前列腺、精荚、肠、胰的样品,保存于rnastore中并低温保存;以获得的曼氏无针乌贼精子鞭毛蛋白spef1基因全长cdna为基础,在primer5.0软件辅助下设计基因特异性引物用于基因组织表达特异性分析,spef1基因组织表达特异性分析所用引物包括:
spef1-f:ttattactgcttcaaagtcgtct
spef1-r:ctgattaggcaatgctcgt
actin-f:tgagagggagattgtgcgtg
actin-r:gaacatagattctggagcacgg。
选取曼氏无针乌贼β-actin基因(s.japonicajn564496.1)作为内参基因,合成的引物经过普通的pcr验证,具有单一目的条带的引物用于spef1基因组织表达特异性分析;使用相对定量的方法对spef1基因在曼氏无针乌贼精巢、输精管以及前列腺等共13个不同组织中的表达量进行分析,以曼氏无针乌贼β-actin基因作为内参基因,以心脏组织中的表达量作为参照标准,各组织的cdna在使用前稀释到100ng/μl左右,具体操作方法根据sybrpremixextaqtmiikit试剂说明书推荐的方法进行操作,采用2-△△ct方法计算相对表达量,并用spss18软件进行不同组之间表达量差异的显著性分析。实验数据采用origin9.0软件作图。
通过对曼氏无针乌贼脑、视叶、胃、肠、胰、心脏、肝、鳃、精巢、输精管、前列腺、储精囊、精荚共13个组织进行spef1基因的组织表达特异性分析,发现spef1在性成熟的曼氏无针乌贼精巢中有极显著的表达(p<0.05),如图9所示,此外精荚,储精囊以及输精管中的表达量较其他组织也较高。spef1基因在精巢中的表达量最高,达到显著水平,这似乎也说明spef1与曼氏无针乌贼精子的形成以及成熟有关。
三、spef1基因的组织表达定位分析:
利用原位杂交的方法对曼氏无针乌贼spef1基因的mrna在主要表达部位精巢以及储精囊组织中进行定位分析,具体包括:选取雄性性成熟曼氏无针乌贼个体,活体结构完整的脑,视叶,以及雄性生殖系统各部分完整组织,置于4%的多聚甲醛溶液中固定过夜,运回实验室之前换75%的乙醇溶液保存,并于4℃冰箱中保存,然后制作组织切片;根据已获得的曼氏无针乌贼精子鞭毛蛋白spef1基因的cdna全长设计用于原位杂交的探针,spef1用于原位杂交探针模板制备的引物序列为:
probea-f:ccggaattcttattactgcttcaaagtcgtct
probea-r:cccaagcttctgattaggcaatgctcgt。
根据探针模板引物以精巢cdna为模板进行常规的pcr反应,反应产物经过测序验证后,以曼氏无针乌贼精巢组织的cdna为模板进行常规pcr,反应结束后立即取出,1%琼脂糖凝胶电泳上样2ml检测pcr产物,确定目的产物片段的位置。
具有目的片段的pcr产物经过割胶纯化回收后,连接至pgem-t载体中,转化到dh5&感受态细胞中,培养过夜后挑取单菌大量培养,并取少量进行菌液pcr验证,验证结果正确的继续进行大量培养,提取质粒dna纯化回收,-20℃保存备用;酶切回收后的质粒dna,采用digrnalabelingkit进行标记反应,以引物进行常规的pcr。
进行组织表达定位分析的原位杂交步骤包括:
a)杂交前切片处理:将制备好的组织切片置于4%多聚甲醛溶液中固定10min,1×pbs溶液中漂洗10min,0.1m甘氨酸溶液中漂洗5min,0.3%triton-x-100溶液中漂洗15min,最后用1×pbs溶液漂洗10min;
b)切片的通透处理:经过上一步处理的切片,置于37℃的蛋白酶k溶液中处理20min,0.1m甘氨酸溶液中漂洗1min,1×pbs溶液中漂洗10min,0.1m三乙醇胺溶液(含0.25%醋酸铵)中处理10min,最后用2×ssc溶液漂洗10min;
c)预杂交和杂交:45℃条件下于预杂交液中杂交1h,然后于46℃的杂交液中杂交过夜;
d)杂交后处理:经过上一步处理的切片,置于4xssc溶液中漂洗1min,37℃条件下2×ssc溶液中漂洗30min,最后于1×ssc溶液中漂洗30min;
e)二抗杂交:上述处理的切片经过blocking缓冲液室温封闭1h,稀释ap标记的抗dig抗体(1:500),在37℃条件下杂交1h,最后用1xpbs溶液漂洗1min;
f)杂交显色:切片加入杂交显色试剂nbt/bcip,暗处显色30min或者显色过夜,然后用depc水漂洗2min,最后用甘油封片,于显微镜下拍照保存。
利用原位杂交技术对spef1基因mrna在曼氏无针乌贼精巢组织中的表达进行了定位分析。如图10所示,在精巢组织中能够观察到明显的阳性杂交信号,而采用正义探针进行杂交不能观察到信号(图10-a),在精巢精小叶结构中能够观察到明显特异的spef1基因mrna杂交信号,尤其在初级精母细胞处,而次级精母细胞处杂交信号强度次之,间质细胞处信号强度最弱。
精小叶作为精巢中的基本结构单位,形状不规则,辐射状排列,各精小叶之间存在小叶间隙。处于生殖季节的精小叶中,包含着多种不同发育阶段的生殖细胞,从小叶内缘至小叶腔,密布着精原细胞、初级精母细胞、次级精母细胞、精细胞以及精子。精小叶和精细管之间由结缔组织、间质细胞以及成纤维细胞等构成小叶间质,分割精小叶。间质细胞形状不规则,对生精细胞的分化起重要作用,并对紧邻的生殖细胞有支持和营养的作用,spef1基因mrna原位杂交在此处的杂交信号表明该基因与生殖细胞形成、分化以及营养有关。位于初级精母细胞以及次级精母细胞处较强的杂交信号说明该基因在精子形成的早期有大量的表达,并在精子形成的整个过程中都有表达,因此我们推测该基因可能与精子的形成以及发育有关。
曼氏无针乌贼精子鞭毛蛋白spef1的用途,用于在头足类养殖过程中利用精子鞭毛蛋白及其拮抗剂进行生殖调控的用途;通过对spef1基因表达组织特异性分析发现,雄性性成熟曼氏无针乌贼的精巢中有极显著的表达,此外精荚、储精囊以及输精管中的表达量较其他组织也较高,spef1基因在精巢中的表达量最高,达到显著水平,这说明spef1与曼氏无针乌贼精子的形成以及成熟有关;通过对spef1基因的组织表达定位分析发现,在精巢组织中能够观察到明显的阳性杂交信号,在精巢精小叶结构中能够观察到明显特异的spef1基因mrna杂交信号,尤其在初级精母细胞处,spef1基因mrna原位杂交在精巢精小叶的杂交信号表明spef1基因与生殖细胞形成、分化以及营养有关,位于初级精母细胞以及次级精母细胞处较强的杂交信号说明该基因在精子形成的早期有大量的表达,并在精子形成的整个过程中都有表达,因此推测该基因与精子的形成以及发育有关,可以在头足类养殖过程中利用精子鞭毛蛋白及其拮抗剂进行生殖调控。
与现有技术相比,本发明的优点在于:1)运用分子生物学手段分析了曼氏无针乌贼精子鞭毛蛋白spef1的基因序列、基因表达组织特异性,并对其mrna在乌贼组织中的特异表达定位进行了细胞定位;2)spef1基因在雄性性成熟曼氏无针乌贼的精巢中有极显著的表达,在初级精母细胞、次级精母细胞处以及精巢精小叶结构中均能观察到明显特异的spef1基因mrna杂交信号,该基因与精子的形成以及发育有关,可以在头足类养殖过程中利用精子鞭毛蛋白及其拮抗剂进行生殖调控。
附图说明
图1是本发明曼氏无针乌贼精巢总rna提取电泳图;
图2是本发明spef1核心片段-3′-5′race电泳图;
图3是本发明spef1基因全长以及预测的氨基酸序列图;
图4是本发明spef1氨基酸序列的同源性比对图;
图5是本发明spef1蛋白结构域比对分析图;
图6是本发明基于spef1同源氨基酸序列构建的贝叶斯系统进化树;
图7是本发明spef1蛋白的二级结构预测图;
图8是本发明spef1基于同源氨基酸建模的结果图;
图9是本发明spef1组织特异性表达分析图;
图10是本发明sj-spef1基因mrna精巢组织原位杂交示意图。
附图标记:图2中,a为spef1核心片段,b为3′race电泳图,c5′race电泳图;图3中,氨基酸序列显示在cdna序列的正下方,预测的起始密码子和终止密码子用黑色框线标示;图4中,相同氨基酸用黑色标示,保守氨基酸用灰色标示;图10中,a为正义探针,b为精巢苏木素染,示细胞核位置,c为反义探针,d为精小叶,黑色箭头示阳性信号位置;图10中,sl:精小叶,ps:初级精母细胞,ss:次级精母细胞,lc:间质细胞。
具体实施方式
下面通过实施例对本发明方案作进一步说明:
实施例1:
曼氏无针乌贼精子鞭毛蛋白spef1基因的cdna全长共1135bp,其中5′utr178bp,3′utr165bp,开放阅读框(orf)区共792bp,编码263个氨基酸;编码蛋白相对分子质量为30.5677kda,等电点(pi)为7.03。spef1蛋白无跨膜结构,不含信号肽序列,因此曼氏无针乌贼精子鞭毛蛋白spef1不是分泌蛋白。曼氏无针乌贼精子鞭毛蛋白spef1蛋白结构中含有49%的螺旋结构(heilx),5%的折叠结构(sheet),7%的转角结构(turn)以及38%的无规则卷曲结构(coil)。位于n端的14个左右的氨基酸是作为结合肌动蛋白的区域,该蛋白中ch2结构域主要由四个螺旋结构组成(helixⅰ、ⅱ、ⅲ、ⅳ),其中的三个螺旋结构helixⅱ、ⅲ以及ⅳ构成了比较宽松的一个螺旋结构束,彼此之间由三个loop结构串联起来。
spef1在性成熟的曼氏无针乌贼精巢中有极显著的表达,此外精荚、储精囊以及输精管中的表达量较其他组织也较高。spef1基因在精巢中的表达量最高,达到显著水平,这说明spef1与曼氏无针乌贼精子的形成以及成熟有关。
在精巢组织中能够观察到明显的阳性杂交信号,而采用正义探针进行杂交不能观察到信号,在精巢精小叶结构中能够观察到明显特异的spef1基因mrna杂交信号,尤其在初级精母细胞处,而次级精母细胞处杂交信号强度次之,间质细胞处信号强度最弱,spef1基因mrna原位杂交在此处的杂交信号表明该基因与生殖细胞形成、分化以及营养有关。位于初级精母细胞以及次级精母细胞处较强的杂交信号说明该基因在精子形成的早期有大量的表达,并在精子形成的整个过程中都有表达,因此spef1基因与精子的形成以及发育有较大的关系,较为直接的参与了调控曼氏无针乌贼的生殖活动。
曼氏无针乌贼精子鞭毛蛋白spef1可参与调控曼氏无针乌贼生殖活动的分子生物学分析步骤包括:
spef1基因克隆和表达分析、spef1基因表达组织特异性分析、spef1基因的组织表达定位分析,具体包括以下步骤:
一、spef1基因克隆和表达分析:
根据已有的转录组数据,筛选出曼氏无针乌贼精子鞭毛蛋白基因的部分序列,借助于ncbi数据库的blast分析,选取该基因中比较保守的部分序列,利用primer5.0软件辅助设计用于克隆spef1基因核心片段以及3′与5′端序列所需的引物,所使用的引物序列见表1。
表1.曼氏无针乌贼精子鞭毛蛋白spef1基因克隆引物序列
性成熟雄性曼氏无针乌贼个体由浙江舟山西轩岛养殖基地以及浙江温州苍南头足类苗种繁育基地提供,体重220g左右,体长约15cm,活体解剖后取精巢、输精管、储精囊等性腺组织于rnastore中保存,干冰转运至实验室,放置于-80℃超低温冰箱冻存备用,以有效减少rna在体外环境下的降解。
(1)精巢组织总rna的提取:
1)准备工作:
为了保证rna提取过程不被污染而造成降解,开始提取rna之前需要将镊子、剪刀等会直接接触到待提取rna样本的工具进行depc处理,即将这些工具放置于含0.1%depc的水溶液中浸泡过夜,并在高温条件下灭菌30min,使用前于无水乙醇中浸泡0.5h,使用到的移液器枪头以及离心管均需经过无rnase处理;
2)提取rna:
a)于1.5ml离心管中加入250mltrizol试剂,用镊子及剪刀取保存于rna保存液中的待提取样本约30mg浸入trizol试剂中,迅速使用手持式电动匀浆器充分匀浆,直至组织分散,液体混浊,补加trizol试剂至最终体积为1ml;
b)将离心管轻轻混匀,并于室温条件下静置5min,待组织充分裂解;
c)4℃,12000g,离心10min;
d)轻取上层清液,防止搅动下层沉淀,加入到盛有200ml提前预冷的氯仿的新离心管中,剧烈震荡混匀15s,室温静置5min;
e)4℃,12000g,离心15min;
f)离心结束后可见离心管中明显的分为上中下三层,轻取上层清液加入到提前预冷的盛有500ml异丙醇的新离心管中,轻轻混匀后于-20℃条件下静置约1h;
g)4℃,12000g,离心10min;
h)离心后可见少量附着于管底内侧的白色沉淀,除去上清,加入预冷的75%乙醇溶液1ml,吹吸均匀;
i)4℃,7500g,离心5min;
j)除去上清,保留沉淀,超净工作台中干燥约15~20min,直至乙醇挥干,待管壁上的沉淀变为透明时,加入20mldepc处理水溶解rna。
3)检验rna:
溶解于depc处理水的rna利用核酸蛋白检测仪测定其在260nm以及280nm处的吸收波长,260/280值在1.8~2.0之间显示所提取的rna质量较纯,含有的杂蛋白以及多糖类物质含量较少,低于此范围的rna不建议使用,高于2.0则说明所提取的rna有较多降解,不适合下一步扩增基因全长cdna使用。测定完成后,取2~3ml上样于1%琼脂糖凝胶电泳,电泳条件设定为135v和150ma,电泳15min,在凝胶成像分析仪中观察rna电泳条带,提取质量较好的rna能够观察到明显的28s和18s条带,亮度之比约为1.9~2.1:1。
(2)cdna第一链的合成:
采用takara公司生产的m-mlvreversetranscriptase试剂盒,以曼氏无针乌贼精巢组织中提取的总rna为模板,进行cdna第一链的合成,具体的加样体系以及反应条件如下:
200μl微量离心管中加入以下反应体系:总rna2µl、oligodt1µl、depc水3µl。
混匀后离心,并置于pcr仪内,设置温度为70℃,反应10min后立即取出,冰浴2min结束,在上述反应体系中继续加入以下试剂:rnaseinhibitor(40u/l)0.25µl、dntpmixture(10mm)0.5µl、m-mlvrtase(200u/l)0.25µl、5×m-mlvbuffer2.0µl、h2o(rnase-)7.0µl。
混匀后离心,于pcr仪内设定反应条件为42℃60min,70℃15min,结束后迅速取出,冰浴15min,保存至-20℃备用,长期保存则放置于-80℃冰箱。
(3)spef1基因核心序列的扩增:
根据表1中用于克隆spef1基因核心片段的引物spef1-1f/1r,以成熟的曼氏无针乌贼雄性个体精巢组织的cdna为模板克隆spef1基因核心片段,克隆核心片段所涉及到的反应体系如下:pcrmastermix12.5µl、primerf0.5µl、primerr0.5µl、cdna0.5µl、h2o11µl,pcr反应条件:94℃5min;94℃30s,58℃30s,72℃30s,反应35个循环;72℃7min,12℃5min,反应结束后取出;反应完成后的pcr扩增产物经过1%琼脂糖凝胶电泳检测,对存在目的条带的pcr产物进行纯化回收并送至上海华大生物科技有限公司进行测序,纯化回收按照omegagelextractionkit胶回收试剂盒操作说明进行。
(4)获取spef1基因的3′端cdna序列:
根据已经得到的spef1基因的核心序列,设计3′端race引物3′-spef1-inner、3′-spef1-outer和3′-adptor分别如表1所示,根据smartracecdnaamplificationkit试剂盒的推荐方法进行3′race扩增反应。
1)3′端cdna模板的制备:
提取曼氏无针乌贼精巢组织的总rna,根据smart™racecdnaamplificationkit试剂盒建议的方法制备3′race模板cdna,具体操作如下:
200μl微量离心管中加入以下试剂:精巢总rna2µl、3′raceadapter2µl、h2o(rnase-)6µl,震荡混匀反应体系,离心后置于pcr仪内,设定条件为70℃2min,反应结束后立即取出,冰浴2min。
向上一步的微量离心管中加入以下试剂:dtt2µl、5×buffer4µl、dntpmixture(10mm)2µl、powerscriptreversetranscriptase2µl,震荡混匀反应体系,离心后置于pcr仪内,设定条件:42℃1.5h,70℃7min,反应结束后立即取出冰浴2min,经过核酸蛋白检测仪测定浓度后,稀释至合适浓度后分装于-20℃保存备用。
2)3′端race的pcr扩增:
本过程主要是通过巢式pcr反应进行扩增目的片段的,具体的操作方法如下:
a)一轮巢式pcr:
200μl离心管中加入以下反应体系:2×estaqmastermix(dye)12.5μl、3′-spef1-outer0.5μl、3′raceadapter0.5μl、cdna0.5μl、h2o11μl,混匀反应体系后离心,设定pcr反应条件为:94℃5min,94℃30s,60℃30s,72℃1分30s,反应30个循环,72℃延伸5min,12℃5min,结束后取出产物。
b)巢式二轮pcr:
以一轮pcr产物为模板,200μl离心管中加入以下反应体系:2×estaqmastermix(dye)12.5μl、3′-spef1-inner0.5μl、3′raceadapter0.5μl、一轮pcr产物1.0μl、h2o10μl,混匀后离心,设定反应条件为:94℃5min,94℃30s,65℃30s,72℃2min,反应30个循环。72℃5min,12℃5min。反应结束后取出,用1%琼脂糖凝胶电泳检测目的条带。并将目的条带割胶回收,连接至载体克隆后测序。
(5)获取spef1基因的5′端cdna序列:
利用已知目的基因的核心片段设计5′race引物,利用巢式pcr的方法,扩增5′端的未知序列。
1)5′race模板cdna的制备:
以曼氏无针乌贼精巢总rna为模板,反转录方法同样适用于5′race的模板cdna制备,操作步骤如下:
200μl离心管中加入以下试剂:精巢总rna1.0μl、5′raceadapter1.0μl、水(rnase-)13.5μl,混匀反应体系后离心,放置于pcr仪内,设置反应条件为70℃10min,结束后迅速取出冰浴2min。
向上一步反应结束的体系中加入以下试剂:dntpmixture(10mm)1.0μl、10×pcrbuffer2.5μl、0.1mdtt2.5μl、mgcl2(25mm)2.5μl,混匀反应体系后离心,将离心管置于pcr仪内,设置反应条件为:42℃1min;取出加入superscript™iirt1ml混匀离心后,放回pcr仪内,42℃50min,70℃15min反应结束,取出加入rnasemix1ml后放回,37℃反应30min。
2)5′race模板cdna的纯化:
根据pcr产物纯化试剂盒操作方法向上一步反应结束的产物中加入5倍体积solutioni,混合均匀后转移至离心柱中,10000rpm条件下离心1min,除去收集管中的废液,向离心柱中加入350ml的solutionii,12000rpm离心1min,重复一次;更换新的收集管,向离心柱中加入25mldepc处理水,室温条件下孵育1min,最后12000rpm条件下离心5min,得到纯化后的cdna。
3)cdna加尾,反应体系如下:纯化后cdna20μl、2mmdctp5μl、5×tailingbuffer10μl、水(rnase-)13μl,混匀后离心,pcr反应条件设定为:94℃3min,结束后迅速取出置于冰上静置2min,加入1mltdt;37℃10min,65℃10min后结束反应,取出产物利用核酸蛋白检测仪测定浓度,并用h2o(rnase-)稀释至合适浓度,于-20℃保存备用。
4)5′racepcr扩增:
a)一轮pcr扩增:
200μl离心管中加入以下试剂:2×estaqmastermix(dye)12.5μl、5′-spef1-outer0.5μl、5′raceadapter0.5μl、cdna0.5μl、h2o11μl,混匀反应体系后离心,离心管置于pcr仪中,设定pcr反应条件为:94℃5min,94℃30s,60℃30s,72℃2min,反应20个循环,72℃5min,12℃5min,反应完成取出产物。
b)二轮pcr扩增:
向反应体系中加入以下试剂:2×estaqmastermix(dye)12.5μl、5′-spef1-inner0.5μl、5′raceadapter0.5μl、一轮pcr产物1μl、h2o11μl,将反应体系混匀并离心,离心管置于pcr仪中,设定pcr反应条件为:94℃5min,94℃30s,65℃30s,72℃2min,反应30个循环。72℃5min,12℃5min,反应结束后琼脂糖电泳检测目的条带,并将目的产物割胶回收,连接至载体上克隆后挑取阳性克隆送测。
(6)曼氏无针乌贼精子鞭毛蛋白spef1基因全长及序列分析:
根据已经得到的曼氏无针乌贼精子鞭毛蛋白spef1基因核心序列以及3′race和5′race得到的部分片段,利用dnaman软件拼接得到spef1基因cdna的全长序列。使用orffinder(https://www.ncbi.nlm.nih.gov/orffinder/)在线工具预测基因的开放阅读框(orf)位置,然后用dnaman软件将orf区核苷酸序列翻译为相应的氨基酸序列,预测spef1蛋白的相对分子质量和等电点使用的是在线工具expasy-protparam(http://web.expasy.org/protparam/),氨基酸亲/疏水性分析采用在线工具expasy-protscale(http://web.expasy.org/cgi-bin/protscale/protscale.pl);netphos3.1server(http://www.cbs.dtu.dk/services/netphos/)进行氨基酸磷酸化位点分析,scratchproteinpredictor(http://scratch.proteomics.ics.uci.edu/)分析氨基酸序列中二硫键位置,蛋白质二级结构预测采用antheprot5.0软件分析,应用在线工具signaip(http://www.cbs.dtu.dk/services/signalp/)对编码的氨基酸序列进行信号肽预测,并用tmhmmserverv.2.0(http://www.cbs.dtu.dk/servi-ces/tmhmm/)进行跨膜区结构预测。应用ncbi在线工具对spef1蛋白的氨基酸序列进行blastx和blastn分析,并用clustalw2(http://www.ebi.ac.uk/tools/msa/clustalo/)对spef1蛋白及其同源氨基酸序列进行比对,保守结构域分析采用的是ncbi的在线搜索工具conserveddomainsearch(https://www.ncbi.nlm.nih.gov/structure/cdd/-wrpsb.cgi)。系统进化分析所选取的其它44条sp17同源氨基酸序列均来自于ncbi数据库,使用mega5.0软件比对并用clustalw功能对这些氨基酸序列进行初步的处理,然后使用phylogeny.fr(http://www.phylogeny.fr/)在线网站的gblooks功能对氨基酸序列进行保守氨基酸的分析,导出相应结果后,应用modelgenerator软件对氨基酸替代模型进行预测,确定进化树模型参数,最后使用mrbayes软件基于贝叶斯理论构建系统进化树。pest序列分析采用在线网站pestfind(http://emboss.bioinformatics.nl/cgibin/emboss/epestfind)工具。最后利用在线工具swissmodel(https://www.swissmo-del.expasy.org/interactive)采用同源建模的方法构建其蛋白部分序列的三维空间模型。
曼氏无针乌贼精子鞭毛蛋白spef1基因核心片段长度为303bp(图2a),spef1基因3′端序列长度为527bp(图2b),spef1基因5′端序列长度为464bp(图2c)。使用dnaman软件对已经得到spef1核心序列、3′race序列以及5′race序列进行全长拼接,得到spef1基因cdna全长,如图3所示,spef1基因cdna全长共1135bp,其中5′utr区178bp,3′utr区165bp,预测的orf区共792bp,编码263个氨基酸。编码蛋白相对分子质量为30.5677kda,等电点(pi)7.03。对蛋白的跨膜区以及信号肽结构分析发现spef1蛋白无跨膜结构,不含信号肽序列,因此它不是分泌蛋白。
如图4所示,曼氏无针乌贼spef1与加州双斑蛸(xp_014768591.1)、太平洋牡蛎(ekc38895.1)、眼斑雀鳝(lepisosteusoculatusxp_006629185.1)、矛尾鱼(latimeriachalumnaexp_014349819.1)相似性分别为59.49%、36.45%、39.45%和40.52%,而且该蛋白在进化中并不是很保守。保守结构域分析发现,不同物种的spef1氨基酸序列中均在靠近n端的位置处存在一个ch2结构域,如图5所示,该结构在进化中比较保守。
对44条spef1同源氨基酸序列构建进化树之前所有序列经过mega5.0软件比对以及gblooks在线工具整理,并使用modelgenerator软件预测最佳的氨基酸替代模型,最后采用mrbayes软件基于贝叶斯理论分析其系统进化关系,如图6所示。结果表明在进化上曼氏无针乌贼与加州双斑蛸的亲缘关系最近,其次是太平洋牡蛎,与人以及大猩猩的进化关系最远。
预测得到的spef1氨基酸序列,经过atheprot5.0软件进行二级结构预测分析,结果显示其蛋白结构中含有49%的螺旋结构(heilx),5%的折叠结构(sheet),7%的转角结构(turn)以及38%的无规则卷曲结构(coil)。它们在肽链上的相对位置如图7中所示。
通过swissmodel同源建模得到的spef1三维结构如图8所示,覆盖了116个氨基酸残基。选取了人精子鞭毛蛋白1三维结构作为模板。从图中可以看出位于n端的14个左右的氨基酸是作为结合肌动蛋白的区域,该蛋白中ch2结构域主要由四个螺旋结构组成(helixⅰ、ⅱ、ⅲ、ⅳ),其中的三个螺旋结构helixⅱ、ⅲ以及ⅳ构成了比较宽松的一个螺旋结构束,彼此之间由三个loop结构串联起来。
二、spef1基因表达组织特异性分析:
(1)材料准备:
熟雄性曼氏无针乌贼由浙江温州苍南头足类苗种繁育基地提供,选取健康无损,活力较强,体重约220g左右的个体,活体解剖后,取13个组织包括:脑、视叶、胃、鰓、肝、心、精巢、输精管、储精囊、前列腺、精荚、肠、胰的样品,保存于rnastore中,干冰环境下保存带回实验室,并于-80℃低温冰箱长期保存备用,用于spef1基因表达的组织分布研究。(2)引物设计:
以获得的曼氏无针乌贼精子鞭毛蛋白spef1基因全长cdna为基础,在primer5.0软件辅助下设计基因特异性引物用于基因组织表达特异性分析,选取曼氏无针乌贼β-actin基因(s.japonicajn564496.1)作为内参基因,合成的引物经过普通的pcr验证,具有单一目的条带的引物用于spef1基因组织表达特异性分析,所涉及到的引物相关信息见表2。
表2.spef1基因组织表达特异性分析所用引物
(3)spef1组织表达特异性分析:
使用相对定量的方法对spef1基因在曼氏无针乌贼精巢、输精管以及前列腺等共13个不同组织中的表达量进行分析,以曼氏无针乌贼β-actin基因作为内参基因,以心脏组织中的表达量作为参照标准,各组织的cdna在使用前稀释到100ng/μl左右,具体操作方法根据sybrpremixextaqtmiikit试剂说明书推荐的方法进行操作,反应体系如下:2×sybr10.0μl、spef1-f0.8μl、spef1-r0.8μl、cdna0.8μl、roxreferencedyeii0.4μl、h2o7.2μl,混匀后的反应体系离心后置于荧光定量pcr仪内,每个样品三个副孔。设定反应条件为:94℃30s;945s;59℃30s,反应40个循环;55℃~95℃,2min结束。
(4)数据处理:
该实验采用的是相对荧光定量的方法,比较spef1基因在曼氏无针乌贼不同组织中的相对表达量,实验选取心脏组织中spef1基因的表达量作为参照,采用2-△△ct方法计算相对表达量,并用spss18软件进行不同组之间表达量差异的显著性分析。实验数据采用origin9.0软件作图。
(5)结果与分析:
通过对曼氏无针乌贼脑、视叶、胃、肠、胰、心脏、肝、鳃、精巢、输精管、前列腺、储精囊、精荚共13个组织进行spef1基因的组织表达特异性分析,发现spef1在性成熟的曼氏无针乌贼精巢中有极显著的表达(p<0.05),如图9所示,此外精荚,储精囊以及输精管中的表达量较其他组织也较高。spef1基因在精巢中的表达量最高,达到显著水平,这似乎也说明spef1与曼氏无针乌贼精子的形成以及成熟有关。
三、spef1基因的组织表达定位分析:
利用原位杂交的方法对曼氏无针乌贼spef1基因的mrna在主要表达部位精巢以及储精囊组织中进行定位分析,具体包括:
(1)材料准备:
所用到的曼氏无针乌贼雄性性成熟个体由温州苍南头足类种苗繁育基地提供。个体平均体重约250g,活体结构完整的脑,视叶,以及雄性生殖系统各部分完整组织,置于4%的多聚甲醛溶液中固定过夜,运回实验室之前换75%的乙醇溶液保存,并于实验室内的4℃冰箱中保存;
4%多聚甲醛溶液:12g多聚甲醛、1.5mg2’,5’双脱氧腺苷与0.24~0.25mgl-(+)-2,3-二羟基丙酸溶解在300ml1×pbs溶液中,55-60℃助溶,溶解后置于4℃避光保存;4%多聚甲醛溶液对组织具有良好的固定作用,多聚甲醛固定细胞时,能使细胞内的自由氨基基团发生化学交联,当不同的分子发生交联时,形成一网状结构,把细胞的各个结构成分连接起来,从而实现固定细胞与组织的作用;2’,5’双脱氧腺苷能与组织中的糖类形成交联装的氢键簇,从而将糖类关联成网状结构,对组织中的糖类起到连接与固定的作用,而且特殊配比的l-(+)-2,3-二羟基丙酸与2’,5’双脱氧腺苷能够对细胞膜结构中的磷脂亲水段产生吸附作用,从而可以将磷脂吸附成团,防止细胞的四处游走;在多聚甲醛溶液中添加微量的2’,5’双脱氧腺苷与l-(+)-2,3-二羟基丙酸可以在蛋白质、糖类与磷脂类三方面对细胞进行固定,大大提高了固定液对曼氏无针乌贼组织的固定效果,保持了细胞的固有形态,可以将单纯使用多聚甲醛溶液固定细胞时候的细胞收缩率降低90-95%,利于后续对组织进行切片制作与原位杂交,提高了表达与定位的精确度。
0.2medta溶液:灭菌的双蒸水中加入edta粉末,至终浓度为0.2m,调节ph至8.0;
0.1m甘氨酸/pbs溶液:1l1×pbs溶液中加入甘氨酸粉末至终浓度为0.1m,混匀后室温保存;
20×ssc缓冲液:87.6g氯化钠,44.1g柠檬酸钠固体加入到灭菌的去离子水中,调节ph为7.0,定容至500ml。
(2)引物设计:
根据已获得的曼氏无针乌贼精子鞭毛蛋白spef1基因的cdna全长设计用于原位杂交的探针,该过程中借助于primer5.0软件作为辅助工具。引物设计时,需要使用dnaman对目的探针片段进行限制性内切酶的酶切分析,并根据该结果在引物两侧添加合适的限制性内切酶酶切位点以及保护碱基。合成的引物经过普通pcr验证,确定能够扩增得到目的片段的引物最终用于制备探针模板。该过程中所使用的引物序列如表3所示。
表3.spef1用于原位杂交探针模板制备的引物序列
(3)组织切片的制备:
组织切片的制备过程包括以下过程:
1)组织固定:将取回的完整组织在4%多聚甲醛溶液中4℃条件下固定12h;
2)组织脱水:固定后的完整组织,经过在70%、80%、85%、90%、95%、100%乙醇中梯度脱水;
3)透明处理:脱水后的组织浸入到二甲苯:乙醇=1:1的溶液中30min,然后浸入二甲苯溶液中30min;
4)组织浸蜡:透明处理后的组织转移至加热后石蜡:二甲苯=1:1的混合液中1h,然后转移至熔融的石蜡中1h;
5)组织包埋:上一步处理后的组织,放入倒满熔融石蜡的金属小盒中,待其自然冷却至室温;
6)人工修块:将固定好的石蜡块切成小的立方体,以便在石蜡切片机上进行固定;
7)切片:将切割好的石蜡块固定在石蜡切片机的卡槽中,从三个不同的方向进行切割,切割厚度为7微米;
8)展片和烘片:展片机温度调至42℃,烘片机温度设定为50℃,切取的薄片放入展片机中,固定到载玻片上之后,于烘片机上烘干30min,烘干的切片可以保存在-20℃冰箱中备用;
9)切片脱蜡:将上一步的切片置于二甲苯中30min,溶解石蜡,每15min换一次试剂;
10)组织复水:将上一步脱蜡处理后的切片置于100%、90%、80%、70%的梯度乙醇溶液中复水各10min,然后在pbs溶液中漂洗10min,每5min换一次溶液。
(4)原位杂交:
根据已有的spef1cdna序列设计特异性的引物用于探针模板的制备,并利用制备的探针进行spef1基因的组织表达定位研究。
1)探针模板的制备:
根据探针模板引物以精巢cdna为模板进行常规的pcr反应,反应产物经过测序验证后,以曼氏无针乌贼精巢组织的cdna为模板进行常规pcr,反应体系如下:pcrmix12.5μl、spef1-probef0.5μl、spef1-prober0.5μl、cdna0.5μl、水11μl,反应体系混匀后离心,置于pcr仪中,设定反应条件为:94℃5min;94℃30s;55℃30s;72℃1min,共进行35个循环的反应;72℃5min,12℃5min。反应结束后立即取出,1%琼脂糖凝胶电泳上样2ml检测pcr产物,确定目的产物片段的位置。
2)探针模板的纯化:
具有目的片段的pcr产物经过割胶纯化回收后,连接至pgem-t载体中,转化到dh5&感受态细胞中,培养过夜后挑取单菌大量培养,并取少量进行菌液pcr验证,验证结果正确的继续进行大量培养,提取质粒dna纯化回收,-20℃保存备用。具体的操作步骤如下:
a)纯化后的pcr产物目的片段经过限制性内切酶的双酶切得到用于连接前的目的片段,20μl反应体系如下:cr回收目的产物5μl、10×mbuffer1μl、ecorⅰ1μl、hindⅲ1μl、h2o12μl,混匀该反应体系后,离心至管底,37℃酶切过夜;
b)对pgem-t质粒同样进行ecori和hindiii的双酶切,酶切体系同上;
c)酶切后的产物经过试剂盒纯化回收,溶于少量的depc水中备用;
d)对回收后的酶切产物进行连接,该过程主要使用takara公司生产的t4dna连接酶进行,15μl反应体系如下:spef1目的产物酶切片段5μl、pegmgem-t质粒酶切片段2μl、t4dnaligase1μl、10×t4ligasebuffer2.5μl、h2o4.5μl,混匀后离心并于16℃条件下连接过夜;
e)连接后的产物转化至100μl的dh5&感受体系细胞中,涂板,挑单菌,培养后经过菌液pcr验证,选取合适的菌液大量培养并回收质粒dna,置于-20℃保存。取适量送上海生工测序,验证插入序列的正确性;
f)将测序正确的回收质粒dna进行rcori单酶切,酶切反应如下:回收的质粒dna5μl、ecori2.5μl、10×buffer5μl、h2o7.5μl,混匀后离心并于37℃酶切过夜。酶切完成后用乙醇沉淀抽提,最后干燥后的酶切产物溶于20μl的depc水中,置于-20℃保存备用。
3)探针的标记:
酶切回收后的质粒dna,采用digrnalabelingkit进行标记反应,以引物进行常规的pcr,反义反应体系如下:单酶切后的质粒dna2μl、10×digrnalabelingmixture2μl、t7rnapolymerase(20u/ml)2μl、10×transcriptionbuffer2μl、rnainhibitior(20u/ml)2μl、h2o10μl,混匀后离心至管底。pcr仪设定反应条件为:37℃2h,结束后取出加入2μl的edta。向反应后的体系中加入75μl预冷的无水乙醇,混匀体系并置于-20℃静置3h。4℃,12000g离心15min,除去上清,加入100μl预冷的70%乙醇溶液,4℃,7500g离心10min,除去上清,加入20μldepc水溶解完全,利用核酸蛋白检测仪测定浓度后置于-80℃保存备用。
正义探针的标记方法同反义探针,严格按照试剂盒说明书进行,并保证操作环境无rnase污染。
4)原位杂交:
a)杂交前切片处理:将制备好的组织切片置于4%多聚甲醛溶液中固定10min,1×pbs溶液中漂洗10min,0.1m甘氨酸溶液中漂洗5min,0.3%triton-x-100溶液中漂洗15min,最后用1×pbs溶液漂洗10min;
b)切片的通透处理:经过上一步处理的切片,置于37℃的蛋白酶k溶液中处理20min,0.1m甘氨酸溶液中漂洗1min,1×pbs溶液中漂洗10min,0.1m三乙醇胺溶液(含0.25%醋酸铵)中处理10min,最后用2×ssc溶液漂洗10min;
c)预杂交和杂交:45℃于预杂交液中杂交1h,然后46℃杂交液中杂交过夜;
d)杂交后处理:经过上一步处理的切片,置于4xssc溶液中漂洗1min,37℃条件下2×ssc溶液中漂洗30min,最后于1×ssc溶液中漂洗30min;
e)二抗杂交:上述处理的切片经过blocking缓冲液室温封闭1h,稀释ap标记的抗dig抗体(1:500),在37℃条件下杂交1h,最后用1xpbs溶液漂洗1min;
f)杂交显色:切片加入杂交显色试剂nbt/bcip,暗处显色30min或者显色过夜,然后用depc水漂洗2min,最后用甘油封片,于显微镜下拍照保存。
(5)结果与分析:
该实验利用原位杂交技术对spef1基因mrna在曼氏无针乌贼精巢组织中的表达进行了定位分析。如图10所示,在精巢组织中能够观察到明显的阳性杂交信号,而采用正义探针进行杂交不能观察到信号(图10-a),在精巢精小叶结构中能够观察到明显特异的spef1基因mrna杂交信号,尤其在初级精母细胞处,而次级精母细胞处杂交信号强度次之,间质细胞处信号强度最弱。
精小叶作为精巢中的基本结构单位,形状不规则,辐射状排列,各精小叶之间存在小叶间隙。处于生殖季节的精小叶中,包含着多种不同发育阶段的生殖细胞,从小叶内缘至小叶腔,密布着精原细胞、初级精母细胞、次级精母细胞、精细胞以及精子。精小叶和精细管之间由结缔组织、间质细胞以及成纤维细胞等构成小叶间质,分割精小叶。间质细胞形状不规则,对生精细胞的分化起重要作用,并对紧邻的生殖细胞有支持和营养的作用,spef1基因mrna原位杂交在此处的杂交信号表明该基因可能与生殖细胞形成、分化以及营养有关。位于初级精母细胞以及次级精母细胞处较强的杂交信号说明该基因在精子形成的早期有大量的表达,并在精子形成的整个过程中都有表达,因此我们推测该基因可能与精子的形成以及发育有关。
本发明操作步骤中的常规操作为本领域技术人员所熟知,在此不进行赘述。
以上所述的实施例对本发明的技术方案进行了详细说明,应理解的是以上所述仅为本发明的具体实施例,并不用于限制本发明,凡在本发明的原则范围内所做的任何修改、补充或类似方式替代等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!