一种应用于人群复杂亲缘关系鉴定的无创高通量检测方法与流程
本发明属于基因工程领域和法庭科学领域,具体涉及一种应用于人群复杂亲缘关系鉴定的无创高通量检测方法,可用于无创亲子关系鉴定,同时能应用于同胞/非同胞兄弟姐妹的母系追踪,因此,特别地,本方法能适用于第三代试管婴儿(非卵子供体受孕)的特殊的无创亲子关系鉴定(仅用于为法庭纠纷提供基因证据)。同时,本方法集中捕获中国人群的高频snp位点,更适合中国人群的亲缘关系鉴定。
背景技术:
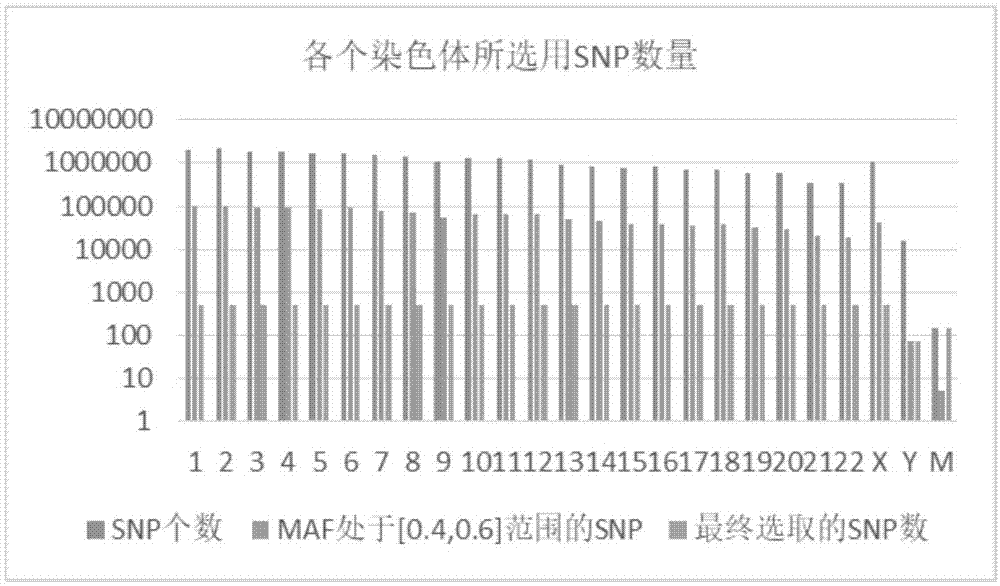
1.亲权鉴定亲权鉴定指应用医学、生物学和人类学的方法检测遗传标记,并依据遗传学理论进行分析,从而对被检者之间是否存在生物学亲缘关系所作的科学判定。亲权鉴定涉及的范围非常广泛,即包括两代直系间亲缘关系的判定,也包括同胞间、隔代直系间,以及旁系个体(叔侄、姨甥等)间亲缘关系的判定。2.snp及分型snp主要是指在基因组水平上由单个核苷酸的变异所引起的dna序列多态性。它是人类可遗传的变异中最常见的一种。占所有已知多态性的90%以上。snp在人类基因组中广泛存在,平均每500~1000个碱基对中就有1个,估计其总数可达300万个甚至更多。snp全称singlenucleotidepolymorphisms,是指在基因组上单个核苷酸的变异,包括转换、颠换、缺失和插入,形成的遗传标记,其数量很多,多态性丰富。从理论上来看每一个snp位点都可以有4种不同的变异形式,但实际上发生的只有两种,即转换和颠换,二者之比为2:1。snp在cg序列上出现最为频繁,而且多是c转换为t,原因是cg中的胞嘧啶常被甲基化,而后自发地脱氨成为胸腺嘧啶。一般而言,snp是指变异频率大于1%的单核苷酸变异。在人类基因组中大概每1000个碱基就有一个snp,人类基因组上的snp总量大概是3×10^6个。因此,snp成为第三代遗传标志,人体许多表型差异、对药物或疾病的易感性等等都可能与snp有关。3.线粒体dna与母系遗传线粒体中有遗传物质dna,称为母系遗传。线粒体dna与存在于细胞核的dna不同,它们遗传自双亲,而线粒体dna只来源于母体基因。线粒体只能通过母亲遗传给孩子得从精子与卵子的生成说起。卵子和精子都是由生殖细胞(卵原细胞)经过减数分裂而成,卵原细胞通过增殖和分化形成初级卵母细胞。一个初级卵母细胞经过减数第一次分裂形成一个次级卵母细胞和一个极体(第一极体),次级卵母细胞减数第二次分裂形成一个卵细胞和一个极体(第二极体),最后两个极体都死亡,只留下卵细胞。卵原细胞在两次分裂时大部分细胞中的物质都集中在卵细胞中,只有很少的一部分留在极体中,所以极体是没有用的,会死亡,留下卵细胞,卵细胞中细胞核,线粒体等遗传物质和细胞器,还有营养物质,用于胎儿最初的发育。而精子的减数分裂和卵细胞是不一样的,虽然也经历两次减数分裂,但是精子在分裂过程中是均匀分裂,分为四个精子,而没有极体生产,而且在分裂过程中所有的细胞器和营养物质都会被遗弃,只留下细胞核,就是遗传物质。简单来说就是,女性的的生殖细胞虽然分裂的四个细胞,但只留下了一个卵子,并留下了线粒体和遗传物质以及营养物质,卵子比较大;而男性的生殖细胞经过两次分裂留下4个精子,但是精子中只有遗传物质。所以卵子比较少,比较大,精子比较多比较小。4.高通量目标区域测序高通量测序技术(high-throughputsequencing)又称“下一代”测序技术("next-generation"sequencingtechnology),以能一次并行对几十万到几百万条dna分子进行序列测定和一般读长较短等为标志。全基因组测序可获得整个基因组的突变、插入、缺失以及拷贝数目等结构变异。然而,由于全基因组数据量巨大,以30x进行测序为例,人类全基因组就会产生超过9og的测序数据量。而肿瘤等相关的低突变频率,或者孕妇外周血的胎儿游离dna检测,则需要至少5000x层次以上的覆盖度,全基因组测序会产生15t以上的测序数据量。特别是体液标本中肿瘤相关的游离dna,或者孕妇外周血中源自胎儿的游离dna含量极低,全基因组的测序量将超过200t。这样大规模的测序数据,显著增加测序的成本,对数据的分析工作造成极大的困难,进而制约测序的应用。因此,在不做任何信号扩增就进行ngs高通量测序,得到的绝大多数信息是正常细胞基因组的信息。在这么强大的背景噪音下,检测的特异性和敏感性就都成问题。不但如此,因为花费大量的人力和财力来做测序得到的99.99%都是无用的信息,相当于高通量地产生垃圾。为解决这个难题,针对高通量测序平台而建立的多重pcr目标区域的捕获与富集技术应运而生。现在亲缘关系鉴定主要存在的缺点有:1)传统一代测序技术没法同时检测多个snp位点(需要付出很大的代价,和极大的dna量)。2)目前已有的无创亲子鉴定试剂盒,选用千人基因组中的人群频率较高的snp位点组合,但是中国人群和国外人群的频率仍有大部分的差异。3)目前已有亲权关系鉴定的高通量测序试剂盒,没法适用在同胞/非同胞姐妹等的亲权关系鉴定上。4)第三代试管婴儿(非卵子供体受孕)虽不受政策允许,涉嫌违法,但是相应纠纷在各大新闻平台还是屡见不鲜。该亲子鉴定涉及卵子供体女性、受孕女性、精子供体和胎儿的四方关系,比寻常的三联体亲权鉴定更为复杂,目前的无创亲子鉴定试剂盒暂时不适合解决这个问题。技术实现要素:为了解决上述存在的问题,本发明提供一种基于高通量测序平台的可同时检测11714个snp位点和37个特殊基因的无创性亲缘关系鉴定方法。本发明的目的在于一种应用于人群复杂亲缘关系鉴定的无创高通量检测方法。本发明所采取的技术方案是:一种鉴定亲缘关系的无创高通量检测方法,包括以下步骤:1)将人类全基因组24条染色体chrn,n={1-22,x,y}中所有snp在对应基因组数据库中的频率maf全部比对出来,此时24条染色体中所有snp的总集合为s1chrn;2)挑选出24条染色体中maf值为0.4~0.6的snp,符合该条件的snp集合为s2chrn;3)找出集合s2chrn中所有snp在每条染色体上的位置;针对每条染色体,截取位置最小snp与位置最大snp之间的区域,对该区域平均分为500等份,从每1个等份中选取1个tagsnp,从而得到snp集合为s3chrn;4)选取142个线粒体snp,与s3chrn合并得到snp集合为s4chrn;5)再选取37个线粒体dna区域作为检测目标区域的一部分,作为s4chrn集合的补充区域;6)设计能够检测上述s4chrn集合snp和37个线粒体dna区域的高通量测序多重pcr引物;7)提取待检样品的dna,利用上述设计好的引物进行多重pcr构建文库,并对文库进行高通量测序;8)对上述测序结果进行分析,得到待测样品中s4chrn集合snp和37个线粒体dna区域的具体基因型;9)根据具体亲缘关系的鉴定方式,结合对待测样品上述具体基因型的比对分析,判定是否具有相应亲缘关系。进一步的,所述37个线粒体dna区域在线粒体基因组中的具体位置如下所示:进一步的,步骤1)中,所述基因组数据库为千人基因组数据库。进一步的,步骤1)中,所述频率maf为东亚人群中的频率maf。进一步的,步骤3)中,选取tagsnp的具体方法为:将500等份中每一等份作为子区域使用软件计算最大的“snp群落”,选取该“snp群落”中“max(1-r2)”值最大的snp,作为每一等份的tagsnp。进一步的,r2是snp连锁关系的数值化表示。进一步的,所述s4chrn集合中snp数目为11714个。进一步的,所述142个线粒体snp的名称分别为:进一步的,亲缘关系的鉴定包括亲子鉴定、母系同源鉴定、姐妹关系鉴定、祖孙关系鉴定、祖先关系鉴定。进一步的,步骤8)中,测序结果进行分析过程中,对测序数据进行质控,并去除低质量的测序。本发明的有益效果是:1)snp选取数量的原则:24个染色体上,每个染色体选取500个snp,且snp的maf的频率在0.4到0.6之间。2)snp选取位置的原则:每个染色体平均分成500份,在每份区域里面选取1个maf值在预设范围的tagsnp。3)在检测过程中特殊地加入了142对线粒体snp的扩增引物对和37个特殊基因的扩增引物对,有利于复杂亲缘关系的鉴定。且snp位点数高达11714个,大大提高了亲权鉴定的有效snp数量。4)snp位点覆盖整个基因组区域的不同区域,避免了个体可能的微缺失微重复导致的大量snp信息丢失的情况。5)本发明方法可以适用在同胞/非同胞姐妹等的亲权关系鉴定上。6)三代试管婴儿(非卵子供体受孕)虽不受政策允许,涉嫌违法,但是相应纠纷在各大新闻平台还是屡见不鲜。该亲子鉴定涉及卵子供体女性、受孕女性、精子供体和胎儿的四方关系,比寻常的三联体亲权鉴定更为复杂。本检测方法可以推断胎儿和受检女性是否来自同一母系,为可能的法庭纠纷提供直接基因证据。附图说明图1为s4chrn集合中snp在染色体中的分布,图中m表示线粒体基因组。具体实施方式下面结合具体实施例对本发明作进一步的说明。实施例1一种鉴定亲缘关系的无创高通量检测方法步骤一:选取目标区域及设计引物1)使用annovar软件把全基因组24个染色体chrn,n={1-22,x,y}的snp所对应的千人基因组数据库(2015年8月版本)中的东亚人群频率maf全部比对出来,此时24条染色体的snp的总集合为s1chrn。2)挑选出每个染色体中maf处于0.4到0.6之间的snp,符合该条件的snp集合为s2chrn。3)对于每个染色体chrn,将集合s2chrn的snp位置从小到大进行排序,平均分为500等份,从每个500等份中选取1个tagsnp,从而得到snp集合为s3chrn。选取tagsnp的具体操作为:每个chrn的子区域(500份切分后的每一份区域)作为输入文件,使用wclustagv2软件(http://www.math.hkbu.edu.hk/~mng/wclustag/wclustag.html)计算最大的“snp群落(cluster)”,选取该“snp群落(cluster)”中”max(1-r2)”值最大的snp(r2是snp连锁关系的数值化表示),作为该chrn子区域的tagsnp。本计算步骤是基于东亚人群的snp频率数据进行计算,在其他人种计算时,可以选取其他人群的snp频率数据进行计算,获得不同人群的tagsnp(参考文献combiningfunctionalandlinkagedisequilibriuminformationintheselectionoftagsnps)。4)提取142个线粒体snp(见表1),与s3chrn合并得到snp集合为s4chrn。snp数目为11714个,其在各个染色体上的分布如图1所示。表1142个线粒体snp的具体信息5)特殊地,把下表2中37个线粒体dna区域作为目标区域的一部分,作为s4chrn集合的补充区域。表237个线粒体dna区域在线粒体基因组中的具体位置6)把集合s4chrn的snp和线粒体补充目标区域(上表2),合并为一个适用于本检测方案的bed文件。7)所述bed文件用于ionampliseqdesigner网站(用其他测序引物设计软件代替也可)设计用于高通量测序文库构建的多重pcr引物,作为网站输入文件。8)本所述bed文件也用于高通量测序目标区域测序质量评估的输入文件。步骤二:提取待检样本的样品dna。步骤三:将提取好的dna样本利用设计好的引物进行多重pcr文库构建并进行高通量测序。步骤四:对测序数据进行质控,并去除低质量的测序reads。1)利用fastqc工具对原始数据rawdata进行质控,得到fastqc的结果报告文件;2)根据fastqc结果报告文件,用cutadapt/trimmomatic对有需要的rawreads进行精细过滤,得到cleanreads,以便开展后续分析。步骤五:对上一步所得的测序cleanreads进行基因组定位:1)利用bowtie2把clean_raw_datas比对至参考基因组hg19,得到比对结果sam格式文件;2)利用samtools工具进行sam格式到bam格式的转换,并对bam文件进行排序;3)利用samtools工具的rmdup功能,去除pcrduplication,并建立bam文件索引;步骤六:检测待测样品在11714个snp和37个特殊基因的具体基因型:1)使用samtoolsmpileup和/或gatk开源软件对处理好的bam文件进行检索变异位点,得到snp信息初始文件;2)使用开源软件bcftools转换为vcf格式的变异位点结果文件,从而得到所有待测样品在11714个snp上的具体基因型;步骤七:根据具体的亲缘关系鉴定的形式,根据相应的snp结果计算进行亲缘关系鉴定。实施例2一种鉴定亲缘关系(无创亲子鉴定)的无创高通量检测方法案情简述:1)委托事项:检测送检标记为“徐女士”的样本中胎儿dna与标记为“李先生”和“郑先生”的样本中dna,dna来源者之间是否存在生物学亲子关系。2)样品及信息收集如表3所示:表3样品及信息收集样本人种样本信息样本类型性别样本编号徐女士亚洲孕周10w+全血女k207李先生亚洲怀疑生父口腔拭子男k208检测步骤:步骤一:分离待检样本k207血液样品的血浆和棕黄色层。1)预冷低速离心机,温度设置为4℃,待温度稳定后,放入采血管,1,600g离心10分钟,吸取上清血浆至ep管中,用于第2)步操作。把中间白细胞棕黄色层转移到2.0mlep管中,立即放入-80℃冰箱中保存。2)预冷高速离心机,温度设置为4℃,待温度稳定后,放入第1)步所得的血浆,16,000g离心10分钟,吸取上清血浆(注意避免吸取沉淀),分装至ep管中,立即放入-80℃冰箱中保存。步骤二:提取母体dna和孕妇游离dna(含胎儿dna)。1)向离心管(自备)中加入20μlproteinasek。2)加入200μl样本。3)加入160μlbuffercl,颠倒混匀,剧烈震荡至少30秒。4)60℃孵育30分钟,其间颠倒混匀数次。注意:200μl血清/血浆样本60℃孵育10-15分钟即可。5)加入360μlbuffercb(使用前检查是否加入异丙醇),震荡至彻底混匀。6)冰浴5分钟,短暂离心,使管壁和壁盖上的液体集中到管底。7)将步骤6所得溶液全部加入到已装入收集管的吸附柱(spincolumnsdf)中,若一次不能加完溶液,可分多次转入。12,000rpm离心1分钟,倒掉收集管中的废液,将吸附柱重新放回收集管中。8)向吸附柱中加入500μlbuffergw1(使用前检查是否加入无水乙醇),12,000rpm离心30秒,倒掉收集管中的废液,将吸附柱重新放回收集管中。9)向吸附柱中加入750μlbuffergw2(使用前检查是否加入无水乙醇),12,000rpm离心30秒,倒掉收集管中的废液,将吸附柱重新放回收集管中。10)向吸附柱中加入750μl无水乙醇,12,000rpm离心30秒,倒掉收集管中的废液,将吸附柱重新放回收集管中。11)12,000rpm离心2分钟,倒掉收集管中的废液。将吸附柱置于室温数分钟,以彻底晾干。注意:这一步的目的是将吸附柱中残余的乙醇去除,乙醇的残留会影响后续的酶促反应。12)将吸附柱置于新的离心管中,向吸附柱的中间部位悬空加入20-100μlbufferebl或灭菌水,室温放置2-5分钟,12,000rpm离心1分钟,收集dna溶液,-20℃保存dna。步骤三:提取待检样本k208的dna。1)用剪刀将棉签部分从其杆上剪下,放入2ml离心管中,加入400μl裂解液ml。再加入20μl的蛋白酶k(20mg/ml)溶液,立刻涡旋振荡充分混匀,2)可选步骤(一般不需要做):56℃放置1小时,期间每10分钟涡旋混匀10秒。3)加入400μl结合液cb,立刻涡旋振荡充分混匀,70℃放置10分钟。此时溶液应变清亮,简短离心以去除管盖内壁的液滴,然后挤压去除拭子,将尽可能多的裂解液转移至新的离心管。4)冷却后加200μl无水乙醇,立刻涡旋振荡充分混匀。简短离心以除去管盖内壁的液滴,收集所有的液体到管底。5)将上一步混合物加入一个吸附柱ac中,(吸附柱放入收集管中)12,000rpm离心30-60秒,倒掉收集管中的废液。6)加入500μl抑制物去除液ir,12,000rpm离心30秒,弃废液。7)加入500μl漂洗液wb(请先检查是否已加入无水乙醇!),12,000rpm离心30秒,弃掉废液。8)加入500μl漂洗液wb,12,000rpm离心30秒,弃掉废液。9)将吸附柱ac放回空收集管中,13,000rpm离心2分钟,尽量除去漂洗液,以免漂洗液中残留乙醇抑制下游反应。10)取出吸附柱ac,放入一个干净的离心管中,在吸附膜的中间部位加20-50μl洗脱缓冲液eb(洗脱缓冲液事先在65-70℃水浴中预热效果更好),室温放置1分钟,12,000rpm离心1分钟。将得到的溶液重新加入离心吸附柱中,室温放置1分钟,12,000rpm离心1分钟。步骤四:将提取好的dna样本利用设计好的引物进行多重pcr文库构建并进行高通量测序。对测序数据进行质控,并去除低质量的测序cleanreads。1)利用fastqc工具对原始数据rawdata进行质控,得到fastqc的结果报告文件;2)根据fastqc结果报告文件,用cutadapt/trimmomatic对有需要的rawreads进行精细过滤,得到cleanreads,以便开展后续分析。步骤五:对上一步所得的测序cleanreads进行基因组定位:1)利用bowtie2把cleanreads比对至参考基因组hg19,得到比对结果sam格式文件;2)利用samtools工具进行sam格式到bam格式的转换,并对bam文件进行排序;3)利用samtools工具的rmdup功能,去除pcrduplication,并建立bam文件索引;4)统计比对信息见表4。表4统计比对信息样品编号cleanreadsmappedreadsusefulreadspairedreadssereadsk20821,927,778213596242121985817983898139766k207棕黄层21,086,226204861442034345617362826142688k207血浆71,400,538643367526388134252880508455410步骤六:检测待测样品在11714个snp和37个特殊基因的具体基因型:1)借助本检测方案的bed文件,使用samtoolsmpileup和/或gatk开源软件对处理好的bam文件进行检索变异位点,得到snp信息初始文件;2)使用开源软件bcftools转换为vcf格式的变异位点结果文件,从而得到所有待测样品在11714个snp上的具体基因型,并获取37个线粒体基因区域的所有snv(非snp的单核苷酸变异)信息;步骤七:snp位点筛选和位点分析。通过以上检测和分析,从目标区域中筛选出139个位点存在胎儿dna与疑似父亲dna无法匹配的情况(见表5)。表5胎儿dna与疑似父亲dna无法匹配的位点步骤八:亲权指数计算。根据本次检测结果分析,在排除遗传变异,同卵多胞胎,近亲和外源干扰(如造血干细胞移植)等前提条件下,样本“徐女士”中胎儿dna和样本“李先生”dna之间存在139个snp位点不符合以上所描述的孟德尔遗传规律,累积亲权指数(cpi)小于0.0001,亲权概率(rcp)小于0.0001。因此,从遗传学角度不支持标记的“徐女士”样本中胎儿dna来源者与送检标记为“李先生”样本来源者存在生物亲子关系。实施例3一种鉴定亲缘关系(姐妹鉴定)的无创高通量检测方法案情简介:由于早年失散的原因,多年后相遇的陈婆婆和黄婆婆怀疑双方是亲生姐妹,现年分别为75岁和86岁。由于父母均以离世,所以需要进行疑似亲姐妹鉴定。检测步骤:分别对陈婆婆和黄婆婆进行采血,提取dna,参照上述实施例1和2所述的方法进行操作。由于本案例需要对样品推断是否来源相同母系,因此要从线粒体角度进行分析,把分析结果中的有效线粒体dna信息展示如下。根据本次检测结果分析,两个样本dna在线粒体基因组上存在18个位点不符合母系遗传规律(表6),因此,从遗传学角度不支持标记的“陈婆婆”样本中dna来源者与送检标记为“黄婆婆”样本来源者存在同胞/半同胞亲姐妹关系。表618个不符合母系遗传规律的位点本发明方法能应用的亲缘关系鉴定领包括无创产前亲子鉴定、亲子鉴定、母系同源鉴定、姐妹关系鉴定、祖孙关系鉴定、祖先关系判断。因此可广泛应用于亲子鉴定、祖籍追踪、人口失踪查询(打拐)、嫌疑排查等领域。本发明方法同时能应用于同胞/非同胞兄弟姐妹的母系追踪,因此,特别地,本方法能适用于第三代试管婴儿(非卵子供体受孕)的特殊的无创亲子关系鉴定(仅用于为法庭纠纷提供基因证据)。另外,本方法可以集中捕获中国人群的高频snp位点,更适合中国人群的亲缘关系鉴定。本发明选用中国人群等位基因频率在40%-60%的位点,更加适合于中国人群的亲权关系鉴定。本发明snp位点覆盖整个基因组区域的不同区域(因为选取的时候是把基因组进行整体平均切割,再在每个区域选代表性snp),避免了个体可能的微缺失微重复导致的大量snp信息丢失的情况。上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1