大籽猕猴桃EST-SSR分子标记及其应用的制作方法
本发明涉及分子标记与药用植物鉴定技术领域,尤其涉及一种大籽猕猴桃est-ssr分子标记及其应用。
背景技术
分子标记在种质资源多样性研究中有着广泛的应用。dna水平的多态性主要表现核苷酸序列的差异,对于任何类型的核苷酸序列的差异都可用分子标记技术对其进行检测。其中简单重复序列(simplesequencerepeat,ssr)又称微卫星序列,是由1~6个核苷酸的串联重复单元组成的一类dna序列。ssr标记呈现出以下优点:(1)广泛存在于真核生物的基因组中、多态性丰富;(2)其产物进行测序凝胶电泳分离时单碱基分辨率高、共显性标记遗传信息量大;(3)呈孟德尔式遗传,具有很好的稳定性,dna用量少,技术要求低,成本低廉,pcr扩增的可重复性高。因此,ssr标记是目前最具应用价值的分子标记之一,已经被广泛应用于基因的快速定位、指纹图谱和遗传图谱的构建以及分子标记辅助选育及种质鉴定等研究。然而,ssr标记物种特异性一定程度上限制了其引物的通用性,有限的基因序列资源依然是ssr标记开发的瓶颈。
2005年以来,第二代高通量测序技术的发展为规模化遗传变异检测和标记位点开发带来了新机遇。ssr可从表达序列标签(expresssequencetag,est)库或转录组测序数据中挖掘,称之为est-ssr。与基因组ssr相比,est-ssr开发相对较易,但其多态性相对较低。筛选高多态性的est-ssr分子标记对于遗传研究与种质资源评价均具有重要的价值。目前,基于转录组信息进行ssr标记引物开发在忍冬(lonicerajaponica)、丹参(salviamiltiorrhiza)、红豆杉(taxuscuspidata)、三七(panaxnotoginseng)、紫苏(perillafrutescens)和杜仲(eucommiaulmoides)等药用植物上得到有效应用。
中国是猕猴桃属植物的起源、进化及分布中心,其遗传资源极为丰富,占全世界猕猴桃遗传资源种属总数的96%。由于猕猴桃属植物现行分类的主要依据是少数一些表观性状,而形态性状本身易受环境的影响;加之该属物种间自然杂交和种质渗入频繁,且染色体倍性复杂,致使对一些种的界定模糊,系统位置难于确定。其中,大籽猕猴桃(actinidiamacrosperma)特产于我国,主产于浙江、江西、安徽等地,为华东地区常用大宗药材“猫人参”的基源植物(俗称“红货”),收载于《中药大辞典》等中药典籍,具有清热解毒、消肿疖等功效,临床用于治疗骨髓炎、肺癌深部脓肿、消化系统肿瘤和肝硬化黄疸腹水等,连续数年位居抗肿瘤配方中的前十名。市场调查结果显示,大籽猕猴桃药材资源的巨大消耗,市场上正宗“红货”几乎告罄,除临床用量需加倍的“白货”对萼猕猴桃(a.valvata)外,黑蕊猕猴桃(a.melanandra)、中华猕猴桃(a.chinensis)、小叶猕猴桃(a.lanceolata)、毛花猕猴桃(a.eriantha)等同属近缘种、甚至其它不同科植物被纷纷混充作猫人参流入市场,成为其在生产和临床上面临的较突出问题。
为解决“猫人参”种质混淆问题,很多学者对其基源植物进行了多方面的鉴定研究,主要集中在形态鉴别、显微鉴别、化学成分鉴别等方面,鲜见分子手段鉴定报道。利用est-ssr分子标记构建大籽猕猴桃核心种质尚属空白。相比于rapd、aflp以及issr这一类显性分子标记而言,est-ssr作为共显性分子标记,能够与植物物种某些表达不同的功能基因相联系,因此可以更加明确物种种间和种内的遗传差异。开发新的est-ssr引物,对于构建高密度遗传图谱和品种指纹图谱具有重要意义,对于大籽猕猴桃的分子鉴定工作将有现实的指导及实践意义。因此,本发明针对现有技术的不足,提供大籽猕猴桃具有高多态性的est-ssr分子标记及其引物与应用,以满足大籽猕猴桃这一重要药用植物资源遗传多样性系统分析和种质评价的需要。
技术实现要素:
本发明的目的在于针对现有技术的不足,提供一种大籽猕猴桃est-ssr分子标记及其应用。
本发明的目的是通过以下技术方案来实现的:一组大籽猕猴桃est-ssr分子标记,包括3个具有多态性的est-ssr分子标记am-es03、am-es06和am-es09,其序列如seqidno.1至seqidno.6所示。
本发明还提供了一种权利要求1所述大籽猕猴桃est-ssr分子标记在大籽猕猴桃及其分类学相近的种质资源遗传传多样性分析中的应用。
本发明与现有技术相比,具有以下技术效果:
(1)本发明从所用的大量引物中筛选出3个可完全鉴别大籽猕猴桃种质的引物作为核心分子标记,这些标记具有带型清晰、重复性好、可靠性强等特点。
(2)本发明标记的应用,具有准确性高、结果稳定、不受环境条件及发育时期的影响、统计简便等优点,可以快速并准确地检验大籽猕猴桃的真伪。
(3)本发明的检测方法在4小时之内即可完成品种真伪和纯度鉴定、及遗传多样性评价工作,具有高效、准确、低成本和操作简便等优势。
附图说明
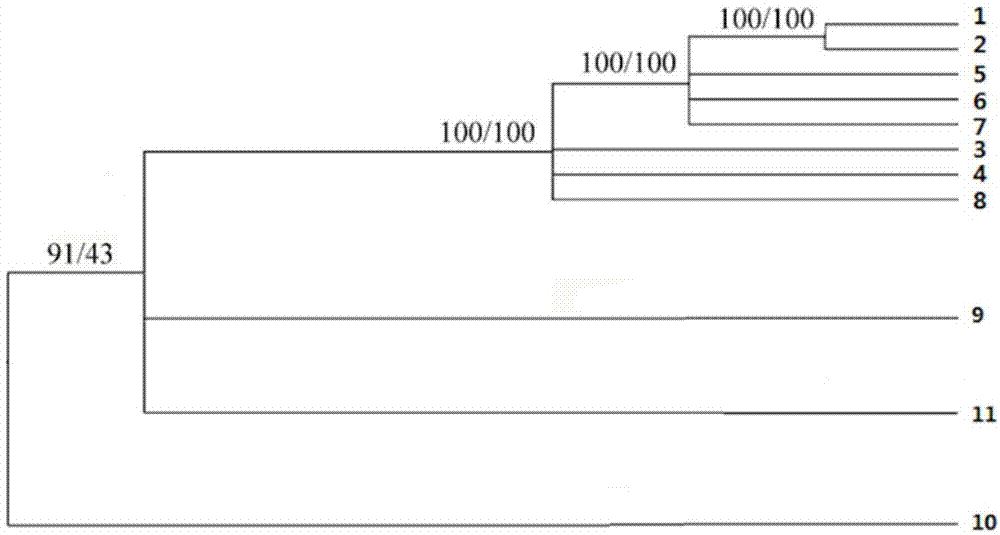
图1为基于3个est-ssr标记构建的不同产地大籽猕猴桃与3个同属外类群种质资源遗传相似系数的upgma聚类图;
图2是猕猴桃属基因组dna完整性电泳图谱。
具体实施方式
本发明结合附图和实施例作进一步的说明。下述实例用以进一步说明本发明,但并不由此限制本发明范围。
本发明提供的3个大籽猕猴桃est-ssr标记am-es03、am-es06和am-es09,其特征如表1所示。
表1:大籽猕猴桃est-ssr标记特征(seqidno:1~6)
本发明的3个大籽猕猴桃est-ssr标记,具体通过以下步骤实现:
(1)采用ctab法或plantzol试剂盒法提取大籽猕猴桃基因组dna。
(2)利用先前研究获得的大籽猕猴桃est序列和primer5.0软件设计引物,引物设计时也选用了ssr重复次数较高的est,从而增大引物在不同材料间的多态性扩增率。具体标准为:10次以上重复的双碱基重复序列,7次以上重复的三碱基序列,5次以上重复的四碱基序列和五碱基序列,以及4次以上重复的六碱基序列。pcr产物长度控制在125~300bp;gc含量40%~70%,最适合为50%;tm值控制在58℃左右;引物长度18~24bp;避免出现引物二聚体。
(3)采用ssr分子标记方法进行大籽猕猴桃分子标记的筛选。以大籽猕猴桃的基因组dna为模板进行pcr扩增:在20μl反应体系含2.0mmol·l-1mg2+,0.25mmol·l-1dntp,0.3μmol·l-1的引物,60ngdna,taqdnapolymerase1u。反应程序为:94℃预变性3min,然后94℃(30s)/60℃(30s)/72℃(45s)循环35次;之后72℃延伸15min;于4℃保存。扩增产物用8wt%丙烯酰胺凝胶电泳检测,硝酸银染色显影,并扫描图像记录结果;
(4)将初步筛选的有扩增产物的引物在不同产地大籽猕猴桃样本上进行扩增,于abi遗传分析仪上进行分析,并使用powermarkerv3.25,tree32和genale×6.501读取数据并分析结果。
本发明的另一个目的是提供所述的3个大籽猕猴桃est-ssr标记在大籽猕猴桃及其分类学相近的种质资源遗传传多样性分析中的应用。
本发明通过rna-seq得到大籽猕猴桃est序列,用misa软件寻找ssr位点,合成了16对引物,并对大籽猕猴桃及同属外类群样本开展pcr扩增、测序和片段长度分析,获得3个未被报道的具有高多态性的est-ssr标记。本发明的作用在于:(1)本发明的3个分子标记在8个大籽猕猴桃样本及3个同属样本中有多态性,且遗传多样性分析与猕猴桃属植物分类常识相吻合(图1),是稳定存在的新标记,可以直接将本发明所提供的3个引物对应用于更多猕猴桃属植物材料上,进行种质资源和遗传多样性分析;(2)本发明的3个分子标记均来自大籽猕猴桃est序列,可应用于大籽猕猴桃的品种鉴定、遗传多样性分析以及分子辅助选育等工作。
图1所示为基于3个est-ssr标记构建的不同产地大籽猕猴桃与3个同属外类群种质资源遗传相似系数的upgma聚类图。图中,基于邻接法(neighbor-joining,nj)和最大简约法(maximumparsimony,mp)进行测算,1为大籽猕猴桃(浙江富阳环山)、2为大籽猕猴桃(浙江临安西天目山)、3为大籽猕猴桃(浙江磐安大盘山)、4为大籽猕猴桃(浙江宁波天童)、5为大籽猕猴桃(安徽歙县三阳)、6为大籽猕猴桃(安徽黄山九龙瀑)、7为大籽猕猴桃(安徽休宁)、8为大籽猕猴桃(江西武陵岩)、9为黑蕊猕猴桃(江西武宁)、10为小叶猕猴桃(浙江临安西天目山)、11为对萼猕猴桃(江西武宁)。
实施例1:样本材料的收集
采用踏查和线路调查相结合的方式,完成资源调查和群体取样(主要集中在华东地区),共采集到大籽猕猴桃野生群体8个及对萼猕猴桃群体1个、黑蕊猕猴桃群体1个、小叶猕猴桃群体1个。
表1:野生大籽猕猴桃及外类群的采样策略
实施例2:基因组dna的提取
(1)ctab法提取基因组dna
配制ctab缓冲液(十六烷基三乙基溴化铵,hexadecyltrimethylammoniumbromide)的配方为:2%ctab,0.1mtris,20mmedta,1.4mnacl,ph值8.0,te缓冲液(10mmtris,1mmedta,ph8.0)。采大籽猕猴桃幼嫩叶片,放入液氮中充分研磨至粉末状,称取0.7g左右转入盛有4mlctab溶液与80μlβ-巯基乙醇(65℃预热)的10ml离心管种,65℃水浴1h;加入4ml氯仿/异戊醇(24:1,v/v),混匀,12000rpm离心10min。吸取上清液,加入2ml5mnacl,4ml异丙醇(-20℃预冷),混匀,放入-20℃2h。12000rpm离心15min,弃上清,沉淀用75%乙醇漂洗2次,晾干。加入100μl含10μg/mlrnasea的te缓冲液溶解沉淀,37℃水浴1h以降解rna。用分光光度计法检测dna浓度,用电泳法检测质量,保存于-20℃。
(2)plantzol试剂盒法提取dna基因组
采大籽猕猴桃幼嫩叶片,将叶片初步夹碎后,至于2ml离心管中,加入小磁珠和少量pvp,用磨样仪研磨2次(速度4m/s,时间30s)。每管加1ml洗涤液,震荡仪混匀,4℃12000rpm离心5min进行dna清洗,弃上清,取沉淀。而后每管加plantdnazol1ml和β-巯基乙醇混合液2μl,震荡仪混匀;65℃水浴60min,每15min轻轻颠倒10次。将水浴后的样品移入圆底离心管,每管加氯仿异戊醇(24:1)750μl,混匀后,于4℃12000rpm离心5min,取上清并加异丙醇630μl混匀,置于-20℃静置30min以上,离心(4℃,12000rpm,10min)取dna沉淀。而后每管加入75%乙醇1ml,清洗数次;挥干乙醇后,加30μlddh2o于4℃溶解dna4小时以上,最后保存于-20℃冰箱中。
(3)dna完整性检测
为评价dna质量,将提取到的猕猴桃属基因组dna采用紫外分光光度计检测,其od260/od280比值均介于1.8~2.0之间,适于后续试验,经1%琼脂凝胶电泳检测基因组dna完整性符合要求(图2)。
实施例3:est-ssr引物的设计与合成
本发明通过rna-seq得到大籽猕猴桃est序列并开发est-ssr引物。引物设计软件为:ncbiprimer-blast(http://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?link_loc=blasthomead)。引物设计时选用ssr重复次数较高的est,从而增大引物在不同材料间的多态性扩增率,提高est-ssr标记在资源分析中的效率。引物设计的具体条件为:pcr产物长度控制在125~300bp;引物长度18~24bp;避免出现引物二聚体。gc含量40%~70%,最适合为50%;tm值控制在58℃左右。共开发了16对est-ssr引物,序列如表2所示。其中,二核苷酸重复聚首(5个),占全部ssr的31.3%;三核苷酸则为11个,占比68.8%。可见在大籽猕猴桃est-ssr中,三核苷酸占主导地位。同时,tc/ct是二核苷酸重复中的优势基元,占二核苷酸重复基元的60%;三核苷酸中出现频率较高的为gaa/aga和aag/gag,均占三核苷酸重复基元的18.2%。
表2:大籽猕猴桃est-ssr标记特征(seqidno:1~32)
实施例4:有多态性引物筛选
用实例2中抽提的大籽猕猴桃叶片dna为模板,用实施例3中设计的16对引物进行扩增。
(1)pcr反应体系:在20μl反应体系含2.0mmol·l-1mg2+,0.25mmol·l-1dntp,0.3μmol·l-1的引物,60ngdna,taqdnapolymerase1u。
(2)利用eppendorfmastercycler(eppendorfscientific,inc.)pcr仪扩增。反应程序为:94℃预变性3min,然后94℃(30s)/60℃(30s)/72℃(45s)循环35次;之后72℃延伸15min。
(3)电泳检测:取5μlpcr产物用8%丙烯酰胺凝胶电泳检测,硝酸银染色显影,并扫描图像记录结果。
(4)str片段长度分析:将上述电泳检测中条带长度在合理范围内的pcr产物进行str片段长度分析,得到不同产地大籽猕猴桃的ssr位点的具体长度,收集3个引物多态性信息
(5)est-ssr的多态性分析:信息含量pic通过公式(1)计算:
pi为第i个等位基因的频率,k为等位基因的数量。pic≥0.10为多态性ssr,pic≥0.70为高多态性ssr。本实验采用16对est-ssr引物对10份大籽猕猴桃种质dna及同属近缘种进行扩增(大籽猕猴桃野生群体8个及对萼猕猴桃群体1个、黑蕊猕猴桃群体1个、小叶猕猴桃群体1个)(见表1)。舍弃产生条带不清晰、不稳定、多态性差或者条带较为复杂而无法读带的引物,最终筛选出6对est-ssr引物在10%的聚丙烯酰胺凝胶上能够获得清晰的扩增条带,成功率为37.5%(见表3)。其中,三核苷酸重复ssr的成功率最高,为66.67%。6对引物共扩增出36种条带,平均每对引物能扩增出6种条带。通过pic计算,3个为多态性est-ssr(pic≥0.10),多态性扩增率为50%,对10份供试材料的区分率达100%,占总引物数的18.75%。三个多态性引物扩增的多态性条带为23条,占6对引物总扩增条带的63.89%;共检测到37个等位基因,每对引物可检测到的等位基因数平均为12.3个。
表3:6对大籽猕猴桃est-ssr引物在猕猴桃属中的扩增结果
(6)upgma聚类图的构建:使用powermarker等软件构建upgma聚类图。大籽猕猴桃est-ssr分子标记在猕猴桃属种质遗传多样性分析中的应用。3个est-ssr分子标记用于大籽猕猴桃野生群体8个及对萼猕猴桃群体1个、黑蕊猕猴桃群体1个、小叶猕猴桃群体1个的基因型分析。聚类分析采用ntsyspc2.1软件,该分析基于upgma法(unweightedpairgroupmethodanalysis)计算遗传相似性。选取根据upgma聚类结果,基于邻接法(neighbor-joining,nj)和最大简约法(maximumparsimony,mp)进行测算:在遗传相似系数为0.73的水平上可将10份猕猴桃群体材料聚为2类,其结果与传统的形态分类大体一致(即净果组sect.leiocarpaedunn与星毛组sect.stellataeli),详见表4。
表4:大籽猕猴桃及其近缘种传统分类群
净果组中,大籽猕猴桃先聚为一类:来自富阳环山与浙江临安的大籽猕猴桃被聚成一支,与安徽歙县三阳、安徽黄山九龙瀑及安徽休宁的大籽猕猴桃较为接近;浙江磐安大盘山、浙江宁波天童与江西武陵岩的大籽猕猴桃则稍远。上述结果与地理分布的区块基本一致。而后,江西武宁的黑蕊猕猴桃、江西武宁的对萼猕猴桃、最终与8份大籽猕猴桃聚为一类。而属于星毛组的来自浙江临安西天目山的小叶猕猴桃被明显的分开,提示该份种质与其它种质亲缘关系较远(见图1)。该结果表明本发明所筛选的est-ssr能够将大籽猕猴桃种质按照区域分开,并明显区别于大籽猕猴桃药材资源混淆品的常见同属近缘种,研究结果从分子水平上解释了这些种质资源的遗传多样性水平,展示了大籽猕猴桃est-ssr在大籽猕猴桃的种质鉴定及评价上的良好通用性。
序列表
<110>浙江树人学院
<120>大籽猕猴桃est-ssr分子标记及其应用
<160>32
<170>siposequencelisting1.0
<210>1
<211>19
<212>dna
<213>人工序列(unknown)
<400>1
gatggctgacgaacaacga19
<210>2
<211>18
<212>dna
<213>人工序列(unknown)
<400>2
tctgtcacgatcacctcc18
<210>3
<211>18
<212>dna
<213>人工序列(unknown)
<400>3
agcgttcacttcagtttc18
<210>4
<211>19
<212>dna
<213>人工序列(unknown)
<400>4
tcaggtgagtccgagattc19
<210>5
<211>18
<212>dna
<213>人工序列(unknown)
<400>5
ctgggtgcctgtttctgg18
<210>6
<211>18
<212>dna
<213>人工序列(unknown)
<400>6
acctgggttggtcagtgg18
<210>7
<211>23
<212>dna
<213>人工序列(unknown)
<400>7
tgtaaaacgacggccagtgttca23
<210>8
<211>20
<212>dna
<213>人工序列(unknown)
<400>8
gtatctcccgctccgacttg20
<210>9
<211>23
<212>dna
<213>人工序列(unknown)
<400>9
tgtaaaacgacggccagtgggaa23
<210>10
<211>23
<212>dna
<213>人工序列(unknown)
<400>10
gagatgacactactgattctgga23
<210>11
<211>20
<212>dna
<213>人工序列(unknown)
<400>11
tctcgctctcagtcctccat20
<210>12
<211>20
<212>dna
<213>人工序列(unknown)
<400>12
aaatcagacctgaaccaccg20
<210>13
<211>20
<212>dna
<213>人工序列(unknown)
<400>13
aggaagatggaggagccatt20
<210>14
<211>20
<212>dna
<213>人工序列(unknown)
<400>14
ccacgactcgacgtcataca20
<210>15
<211>20
<212>dna
<213>人工序列(unknown)
<400>15
aggtgtattcgtgccctttg20
<210>16
<211>20
<212>dna
<213>人工序列(unknown)
<400>16
aaagacgagacttcctcgca20
<210>17
<211>20
<212>dna
<213>人工序列(unknown)
<400>17
caaacagctaatccccgaac20
<210>18
<211>20
<212>dna
<213>人工序列(unknown)
<400>18
tttcgaatcggcaatacaca20
<210>19
<211>20
<212>dna
<213>人工序列(unknown)
<400>19
tttccttcgcgagtcagaat20
<210>20
<211>20
<212>dna
<213>人工序列(unknown)
<400>20
ccttggactgggaccttaca20
<210>21
<211>22
<212>dna
<213>人工序列(unknown)
<400>21
tgtaaaacgacggccagttcca22
<210>22
<211>22
<212>dna
<213>人工序列(unknown)
<400>22
tcggcgttgaaggggacgtagt22
<210>23
<211>20
<212>dna
<213>人工序列(unknown)
<400>23
atttcgattctcaagggcaa20
<210>24
<211>20
<212>dna
<213>人工序列(unknown)
<400>24
cggtgagtcgaattggagat20
<210>25
<211>20
<212>dna
<213>人工序列(unknown)
<400>25
ccccaagagcatcaacatct20
<210>26
<211>20
<212>dna
<213>人工序列(unknown)
<400>26
agttgccttcaccatgaacc20
<210>27
<211>22
<212>dna
<213>人工序列(unknown)
<400>27
aattctggtccattcttctgtg22
<210>28
<211>19
<212>dna
<213>人工序列(unknown)
<400>28
ttccgtggctcccttatct19
<210>29
<211>20
<212>dna
<213>人工序列(unknown)
<400>29
caccaaacttcaccaccctc20
<210>30
<211>20
<212>dna
<213>人工序列(unknown)
<400>30
aaacgagccatctcgacaat20
<210>31
<211>20
<212>dna
<213>人工序列(unknown)
<400>31
tgaaggaggttgcaagtgtg20
<210>32
<211>20
<212>dna
<213>人工序列(unknown)
<400>32
ccaaaacccacaaagcagat20
- 还没有人留言评论。精彩留言会获得点赞!