一种快速制备单分子光学图谱标记文库的方法和试剂盒与流程
本发明涉及一种快速制备单分子光学图谱标记文库的方法,以及适用于此方法的试剂盒。
背景技术:
人类许多疾病是由于染色体结构变异引起的。现在常用的染色体结构分析的方法有核型分析、芯片杂交等。然而,这些技术均存在一定局限性,比如分辨率低、成本高、不能覆盖全基因组等。长片段单分子光学图谱技术的发展弥补了上述技术的局限。bionano公司推出的irys和saphyr系统利用限制性切刻酶或甲基化转移酶对dna进行识别并标记荧光1,再利用纳米通道把dna单分子线性化展开,并进行超长单分子高分辨率荧光成像,即可生成一幅特异酶位点分布图。单分子光学图谱技术可以辅助基因组组装、检测染色体结构变异和基因遗传病的检测2。然而,现阶段的单分子光学图谱标记文库的制备存在耗时长、制备过程繁琐等缺点,并不适用于临床检测。
此外,光学图谱技术的优势是可以在单分子水平上标记dna,因此基因组dna片段的长度是决定光学图谱标记效果的重要因素之一。一般要求dna片段的平均长度大于200kb,最长的dna片段达到mb级别。目前一般采用基于低熔点琼脂糖凝胶包埋的固相方法获得用于制备光学图谱标记文库的长片段基因组dna3-6。该方法可提取长达mb级别的高纯度基因组dna,但是包括将细胞包埋成琼脂糖胶块、蛋白酶k消化处理、胶块熔解、胶块酶解和基因组dna透析等步骤,操作过程非常繁琐,耗时超过24小时。并且,这种方法提取的长片段基因组dna产率往往不稳定,非常黏稠,均一性差导致难以测度浓度,从而影响后续光学图谱标记的质量。这种固相长片段基因组dna提取加上bionano公司的直接标记染色(directlabelingandstaining,dls),使得制备光学图谱标记文库总共需要长达3.5天的时间。这些因素都不利于光学图谱技术的大规模推广使用。
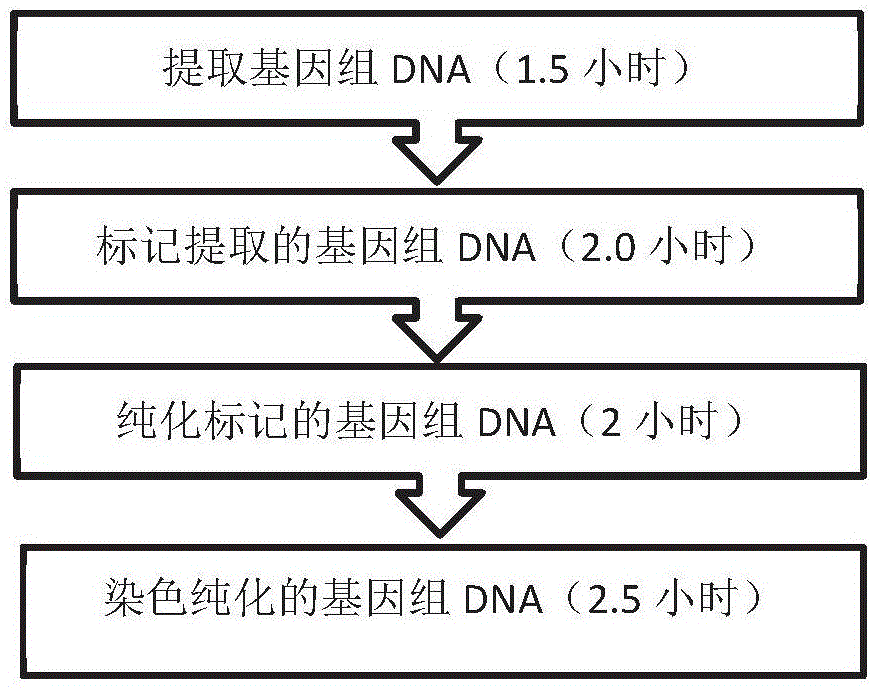
为解决以上问题,本发明提供了一种基于液相系统的快速制备光学图谱标记文库的方法,仅包含提取长片段基因组dna、标记基因组dna、纯化标记的基因组dna和将基因组dna染色四个步骤。该方法简化了光学图谱标记的流程,将时间由原来的3.5天缩短为8小时,并且最终获得的标记文库质量较高,重复性强,有利于单分子光学图谱技术在临床检测上的应用。
技术实现要素:
本发明的目的在于解决现阶段单分子光学图谱文库制备耗时长、过程繁琐等问题。通过液相提取长片段基因组dna并简化单分子光学图谱标记流程,本发明能够实现快速单分子光学图谱标记文库的快速制备。
因此,第一个方面,本发明提供一种快速制备单分子光学图谱标记文库的方法,包括以下步骤:
(1)提取长片段基因组dna;
(2)标记提取的基因组dna;
(3)纯化标记的基因组dna,和
(4)将基因组dna的骨架染色,获得单分子光学图谱标记文库。
在一个实施方案中,提取长片段基因组dna的步骤包括细胞裂解、基因组dna沉淀、基因组dna清洗和基因组dna溶解。在一个优选的实施方案中,提取长片段基因组dna的步骤包括以下子步骤:
a)向样品中加入裂解液和蛋白酶k,使细胞裂解并释放基因组dna;
b)向步骤a)的反应体系中加入沉淀剂,获得基因组dna的沉淀;
c)用清洗液洗涤所得的基因组dna的沉淀;
d)用溶解液溶解基因组dna。
在一个优选的实施方案中,提取长片段基因组dna的步骤由以上四个子步骤a)-d)组成。
在本文中,“长片段基因组dna”是指平均长度大于200kb,且最长可达mb级别的基因组dna。
在一个实施方案中,样品可以是动物来源的任何组织或细胞,例如来自胚胎组织、肝脏组织、胸腺组织、脾脏组织、体液的细胞,或体外培养的细胞系等。体液的实例包括但不限于血液、血清、血浆、关节液、精液、尿液、汗液、唾液、粪便、脑脊液、腹水、胸水、胆汁、胰腺液等。
在一个实施方案中,裂解液包含nacl、tris-hcl(ph7.4-8.0)、edta(ph8.0)和sds。在另一个实施方案中,裂解液由nacl、tris-hcl(ph7.4-8.0)、edta(ph8.0)和sds组成。裂解液中nacl、tris-hcl(ph7.4-8.0)和edta(ph8.0)的浓度可以根据具体实验要求通过常规实验进行调整和确定,例如,nacl的浓度为0.1m-0.5m,tris-hcl(ph7.4-8.0)的浓度为10mm-200mm,edta(ph8.0)的浓度为10mm-100mm。但是,sds的浓度对提取的基因组dna的质量非常关键。具体而言,sds的浓度过低,例如低于0.1%,则会导致细胞裂解不充分;sds的浓度过高,例如高于8%,则过量的sds会随着基因组dna沉淀,影响后续实验。因此,在本发明中,sds的浓度优选为0.1%-8%,更优选0.1%-5%。
在一个实施方案中,裂解液还可以包含rnasea,其作用是除去系统中的rna杂质。rnasea的含量可以由本领域技术人员根据需要确定,例如为20μg/ml。
在一个实施方案中,蛋白酶k的终浓度为0.005-4mg/ml,优选0.005-2.5mg/ml。蛋白酶k的作用在于降解膜蛋白和与基因组dna结合的蛋白质,从而使基因组dna充分游离。当蛋白酶k的含量过低,例如低于0.005mg/ml,则不能充分降解膜蛋白和基因组dna结合的蛋白质,可能会影响基因组dna后续应用,例如影响基因组dna的bionano光学图谱标记;当蛋白酶k的含量过高,例如高于4mg/ml,则过量的蛋白酶k随着基因组dna沉淀,会降解后续应用中的酶切体系,例如影响基因组dna的bionano光学图谱标记。
在一个实施方案中,蛋白酶k和裂解液可以依次加入,也可以同时加入。
上述步骤a)的处理时间和温度可以由本领域技术人员根据实验需求进行调整和确定。例如,可以在30-65℃的温度下处理1-2小时。
在一个实施方案中,本发明的沉淀剂包含至少一种无机盐和至少一种醇类。在另一个实施方案中,本发明的沉淀剂由无机盐和醇类组成。在本发明的方法中,无机盐的实例包括但不限于醋酸铵、醋酸钠、氯化钠、氯化锂等;醇类的实例包括但不限于无水乙醇、异丙醇等。在本发明中适用于沉淀基因组dna的无机盐的浓度为300-500mm,优选350-450mm,更优选约450mm,醇类的浓度为50-80%,更优选60-70%,更优选约65%。
上述步骤b)的沉淀时间可以由本领域技术人员根据实验需求进行调整和确定。
在一个实施方案中,清洗液包含70%-80%乙醇。在另一个实施方案中,清洗液由70%-80%乙醇组成。
在一个实施方案中,溶解液选自去离子水、tris-hcl、te缓冲液等。其他已知的可以用于溶解基因组dna的溶液也适用于本发明。
在一个实施方案中,在标记酶存在下用染料对提取的基因组dna进行标记。具体地,标记酶可以是商品化或纯化的限制性切刻酶或甲基化转移酶。染料可以选自bodipy、fitc、罗丹明、香豆素、呫吨、花青素、芘和酞菁。对提取的基因组dna进行标记的条件和方法可以由本领域技术人员根据实际需要进行调节。例如,可以在标记酶存在下,将染料与基因组dna在适于标记酶活性的温度下孵育足够时间以使标记反应充分发生。
纯化标记的基因组dna的目的主要是去除在标记反应中多余的或未反应的标记酶和染料。在一个实施方案中,用高温失活法或蛋白酶k消化法去除多余的标记酶;并用沉淀法或透析法去除多余的染料。适用于本发明的纯化方法是本领域技术人员已知的。例如,可以将标记的基因组dna与蛋白酶k一起孵育一段时间后,通过滤膜透析,达到纯化目的。
在一个实施方案中,用染料对纯化的基因组dna骨架进行染色,使基因组dna的骨架可视化。用于该步骤的染料可以是,但不限于,yoyo-1或dapi染料。本领域技术人员可以根据需要调节该染色步骤的条件和时间。
在另一个实施方案中,可以采用市售的试剂盒,例如由bionano公司提供的那些(例如,bionanoprepdls试剂盒)进行上述标记、纯化和染色步骤中的一个或多个步骤。
在一个实施方案中,本发明的制备单分子光学图谱标记文库的方法均在液相中进行。
在第二个方面,本发明还提供一种快速制备单分子光学图谱标记文库的试剂盒,包含用于提取长片段基因组dna的试剂、标记酶、至少两种染料和任选的蛋白酶k。
在一个实施方案中,用于提取长片段基因组dna的试剂进一步包含裂解液、蛋白酶k、任选的rnasea、沉淀剂、清洗液和溶解液。
在一个实施方案中,裂解液包含nacl、tris-hcl(ph7.4-8.0)、edta(ph8.0)和sds。在另一个实施方案中,裂解液由nacl、tris-hcl(ph7.4-8.0)、edta(ph8.0)和sds组成。裂解液中nacl、tris-hc(ph7.4-8.0)和edta(ph8.0)的浓度可以根据具体实验要求通过常规实验进行调整和确定,例如,nacl的浓度为0.1m-0.5m,tris-hcl(ph7.4-8.0)的浓度为1omm-200mm,edta(ph8.0)的浓度为10mm-100mm。在本发明中,优选的sds浓度为0.1%-8%,更优选0.1-5%。
优选地,用于提取长片段基因组dna的蛋白酶k的浓度为0.005-4mg/ml,更优选0.005-2.5mg/ml。当存在rnasea时,其含量为20μg/ml。
在一个实施方案中,沉淀剂包含至少一种无机盐和至少一种醇类。在另一个实施方案中,本发明的沉淀剂由无机盐和醇类组成。在本发明的方法中,无机盐的实例包括但不限于醋酸铵、醋酸钠、氯化钠等;醇类的实例包括但不限于无水乙醇、异丙醇等。在本发明中适用于沉淀基因组dna的无机盐的浓度为300-500mm,优选350-450mm,更优选约450mm。醇类的浓度为50-80%,更优选60-70%,更优选约65%。
在一个实施方案中,清洗液包含70%-80%乙醇。在另一个实施方案中,清洗液由70%-80%乙醇组成。
在一个实施方案中,溶解液选自去离子水、tris-hcl、te缓冲液等。
在一个实施方案中,标记酶是限制性切刻酶或甲基化转移酶。
在一个实施方案中,染料的实例包括但不限于yoyo-1、bodipy、fitc、罗丹明、香豆素、呫吨、花青素、芘、酞菁等。
根据本发明所述的方法和试剂盒可在液相中快速制备单分子光学图谱标记文库,其适用于例如bionanogenomics的irys和saphyr等平台。本发明的优异技术效果主要依赖于以下几个方面:
(1)在提取长片段基因组dna方面,本发明方法的所有操作均在单个反应管中完成,不涉及到基因组dna在不同反应管中的转移,从而在节省时间和成本的同时,还避免了基因组dna的损失和可能的污染。同时,整个基因组dna的提取过程只包含裂解、沉淀、清洗和溶解四个步骤,耗时1-2小时,大大缩短了提取时间。此外,本发明的提取方法比较安全,不会对操作人员的身体健康产生危害。本发明的提取方法使用的试剂均安全无毒,同时还避免使用传统基因组dna沉淀方法中所用的剧毒有机溶剂如酚氯仿。最后,提取的基因组dna长度和质量可靠,重复性强。相对于传统的硅胶膜柱和磁珠纯化方法,本发明避免了基因组dna的机械损伤。相对于经典的基于低熔点琼脂糖凝胶包埋的方法提取长片段基因组dna,本发明在实验的可操作性和重复性上有了显著的提高。
(2)根据本发明所述的方法和试剂盒,提取的长片段基因组dna均一性好且总量固定,无需测定浓度即可进行后续实验;由此获得的光学图谱标记文库无需定量浓度即可直接用于bionanogenomics公司的irys和saphyr平台。
(3)根据本发明所述的方法和试剂盒制备光学图谱标记文库的整个流程仅需8小时左右,相比于传统方法所需的3-4天的时间而言大大缩短了制备时间。
下面将结合附图和实施例进一步阐述本发明。应当理解,以下实施例仅仅是说明性的,而不意欲限制本发明的范围。
附图说明
图1.根据本发明方法制备长片段基因组dna的单分子光学图谱标记文库的流程。
图2.根据实施例1制备的单分子光学图谱标记文库的脉冲场电泳结果。
图3.使用不同蛋白酶k浓度提取的长片段基因组dna的标记结果。
图4.使用不同sds浓度提取的长片段基因组dna的标记结果。
图5.实施例3的染色体结构变异分析结果,示出了组装的两条染色体与5号参考染色体和14号参考染色体的比对结果。
具体实施方式
实施例1.根据本发明的方法制备长片段基因组dna的单分子光学图谱标记文库
步骤1.根据以下步骤提取长片段基因组dna
取0.3x106个白细胞,室温2000g离心2分钟,去除上清。加入30μl裂解液(100mmnacl,10mmtris-hcl(ph8.0),25mmedta(ph8.0),0.50%sds)重悬细胞,再加入2μl2mg/ml蛋白酶k混匀,并置于50℃金属浴处理1小时。然后加入12μl5m醋酸铵和90μl无水乙醇,颠倒10次后,置于摇床室温20rpm缓慢混匀5-10分钟,以便使dna完全沉淀并形成小团。用200μl70%乙醇洗涤dna2次,并用42μlte缓冲液(10mmol/ltris-hcl(ph8.0);1mmol/ledta(ph8.0))溶解dna,获得长片段基因组dna。
步骤2.标记提取的基因组dna
使用bionanoprepdlskit(catno.80005),在避光条件下制备如下表1所示的标记混合物:
表1:
然后将标记混合物于37℃孵育2h,然后瞬离。
步骤3.纯化标记的基因组dna
在步骤2所得的标记混合物中缓慢加入5μl蛋白酶k以去除多余的标记酶dle-1,并瞬离样品,然后于50℃孵育0.5h,再次瞬离样品。
然后,通过透析去除多余的染料dl-green。具体地,将25μl1×dle1缓冲液(5μl5×dle1缓冲液+20μlh2o,来自bionanoprepdlskit)加入24孔板的孔中,然后将滤膜置于缓冲液上,待其湿润后,加入去除了多余标记酶的基因组dna,并密封孔。室温避光放置20分钟。重复该透析步骤两次。
步骤4.染色纯化的基因组dna
从步骤3所得的基因组dna中取出20μldna放入2ml圆底避光管中,并加入40μl预染色混合物(15μl4×flowbuffer,12μl5×dtt,13μl不含核酸酶的h2o,均来自bionanoprepdlskit)。混匀后,加入yoyo-1染料(每300ng基因组dna加3.2μl染料),于室温下旋转2小时,然后瞬离样品,获得长片段基因组dna的单分子光学图谱标记文库。
取20μl标记文库进行脉冲场电泳,以检测dna大小,结果如图2所示。从图2可以看出,通过本发明的方法获得的单分子光学图谱标记文库中基因组dna的平均长度为200kb以上,且最长可达mb级别。
另取20μl标记文库在bionano公司的saphyr平台上机收集数据,结果如下表2所示。
表2:
从以上结果可以看出,根据本发明制备的单分子光学图谱标记文库质量较好,满足标记的质量要求,可用于进行基因组组装以及进一步的染色体分析。
实施例2.提取基因组dna时不同浓度的蛋白酶k和sds对光学图谱标记效果的影响
为了检测提取基因组dna时不同浓度的蛋白酶k和sds裂解对光学图谱标记效果的影响,根据实施例1的步骤1,使用不同浓度的蛋白酶k和sds提取白细胞的长片段基因组dna。然后根据实施例1的步骤2-4制备单分子光学图谱标记文库,并在bionanogenomics公司的saphyr平台上进行数据收集。
如图3所示,当蛋白酶k浓度过高,例如为5mg/ml时,提取的长片段dna的标记密度和比对率均太低,假阴性标记比例太高,达不到理想的标记要求(根据参考范围确定)。而当蛋白酶k浓度过低,例如为0.003125mg/ml时,提取的长片段dna的标记密度和比对率仍然太低,同样达不到理想的标记要求。
如图4所示,当sds浓度过高,例如为10%时,提取的长片段dna的标记密度和比对率均极低,假阴性标记比例太高,达不到理想的标记要求(根据参考范围确定)。而当sds浓度过低,例如为0.05%时,提取的长片段dna的标记密度和比对率仍然太低,并且分子长度n50也远远超过参考范围,同样达不到理想的标记要求。
因此,为使提取的长片段dna的质量足够高从而满足后续对其直接进行测序和组装的要求(例如在bionano的单分子光学图谱技术的情况下,长片段dna的质量要足够高以达到理想的标记要求,之后才能有效进行基因组组装而不影响其完成度和精确度),在提取长片段dna时应将蛋白酶k浓度和sds浓度控制在一定的范围内。例如,蛋白酶浓度优选0.005-4mg/ml,更优选0.005-2.5mg/ml;sds浓度优选0.1-8%,更优选0.1-5%。
实施例3.根据本发明制备的单分子光学图谱标记文库在染色体结构变异分析中的用途
本实施例采用的是带有平衡易位的血液样本,通过核型分析,其核型为46,xx,t(5;14)(q11;q32)。根据实施例1所述的方法从该血液样本中提取长片段基因组dna并制备单分子光学图谱标记文库,然后在saphyr平台上收集数据,并利用bionanosolve软件对收集的数据进行基因组组装,获得n50为75.6mb的基因组图。对该基因组图进行染色体结构变异分析,发现在chr5:50217079-50228607和chr14:90511140-90522191存在染色体断裂(图5),这与核型分析结果一致。
因此,根据本发明的方法和试剂盒可以快速制备单分子光学图谱标记文库,并能够成功地进行基因组组装和染色体结构变异分析。
需要说明的是,虽然已通过以上实施例阐明了本发明的一些特征,但不能用于限制本发明。对于本领域的技术人员来说,本发明可以有各种更改和变化。因此对于本领域技术人员来说,在不脱离本发明的构思和原则之内,还可做出若干简单替换,这些均应包含在本发明的保护范围之内。
参考文献
[1]lametetal.genomemappingonnanochannelarraysforstructuralvariationanalysisandsequenceassembly.natbiotechnol.2012aug;30(8):771-6.
[2]barseghyanhetal.next-generationmapping:anovelapproachfordetectionofpathogenicstructuralvariantswithapotentialutilityinclinicaldiagnosis.genomemed.2017oct25;9(1):90.
[3]grunwaldetal.reducedrepresentationopticalmethylationmapping(r2om2).biorxxiv,2017mar3.
[4]daiyetal.single-moleculeopticalmappingenablesaccuratemoleculardiagnosisoffacioscapulohumeralmusculardystrophy(fshd)genomemappingonnanochannelarraysforstructuralvariationanalysisandsequenceassembly.biorxiv,2018mar21.
[5]chanekfetal.opticalmappingrevealsahigherlevelofgenomicarchitectureofchainedfusionsincancer.genomeres.2018may;28(5):726-738.
[6]keeble-gagnèregetal.opticalandphysicalmappingwithlocalfinishingenablesmegabase-scaleresolutionofagronomicallyimportantregionsinthewheatgenome.genomebiol.2018aug17;19(1):112.
- 还没有人留言评论。精彩留言会获得点赞!