用于指导RNA设计和使用的方法和系统与流程
用于指导rna设计和使用的方法和系统
交叉引用
1.本申请要求2018年5月16日提交的美国临时申请号62/672,437的权益,该临时申请通过引用并入本文。
背景技术:
2.设计用于靶向和操作特定dna序列的工程化核酸酶技术正在迅速用作许多不同应用的有用技术,这些应用包括细胞和整个生物体的遗传操作、靶向的基因删除、替换和修复,以及外源序列(转基因)向基因组中的插入。基因组编辑技术的示例包括锌指、转录激活因子样效应物(tale)和成簇的规律间隔的短回文重复序列(crispr)/crispr相关的(cas)(“crispr/cas”)系统。
3.crispr/cas系统可以用作多种不同生物体中的基因编辑工具,用于在靶位点产生断裂,随后在基因座引入突变。基因编辑过程可能需要两个主要组分:核酸内切酶(例如cas酶)和识别特定dna靶序列的短rna分子。crispr/cas系统可以依靠定制的短rna分子将cas酶募集到新的dna靶位点上,而不是为每个dna靶标设计核酸酶。cas酶的实例包括cas9和cpf1。
4.crispr/cas系统可以在原核和真核系统中用于基因组编辑和转录调控。在一些情况下,crispr/cas系统会产生不必要的脱靶基因组编辑,并在不同基因靶标之间产生不同的编辑效率。
技术实现要素:
5.本公开内容描述了与设计识别用于crispr/cas介导的基因操作的相应靶寡核苷酸序列的一种或多种寡核苷酸(例如rna分子)有关的技术,并且更具体地,本公开内容描述了在目标物种的整个基因组中确定脱靶值的方法,用于最小化脱靶基因组编辑并提高编辑效率。本公开内容描述了用于执行此类寡核苷酸的设计和验证的软件和硬件配置。
6.在某些实施方案中,本文描述了用于鉴定可与基因组中的目标基因组区域杂交的指导rna(grna)集的方法,该方法包括:设计grna集,该grna集中的每个grna:可与来自目标基因组区域内的多个靶位点中的靶位点杂交,该靶位点与来自该指导rna集的至少一个其他指导rna的多个靶位点中的不同靶位点相距至少30个碱基。在一些实施方案中,靶位点与不同靶位点相距最多170个碱基。
7.在一些实施方案中,grna集中至少一个grna的序列与目标基因组区域互补。在一些实施方案中,grna集中至少一个grna的序列与目标基因组区域部分互补。在一些实施方案中,grna集中与目标基因组区域部分互补的至少一个grna的序列相对于目标基因组区域包含1个、2个、3个、4个、5个或大于5个错配。在一些实施方案中,grna集中的每个grna的长度为约17个至约42个碱基。在一些实施方案中,grna集中的每个grna的长度为约20个碱基。在一些实施方案中,grna集中的每个grna包含约20个碱基的指导序列,并且还包含长度为约22个至约80个碱基的恒定区。在一些实施方案中,grna集中的每个grna的指导序列选择
性地与目标基因组区域杂交。在一些实施方案中,初始grna集中的每个grna的长度为约100个碱基。
8.在一些实施方案中,目标基因组区域包含基因的编码区域。在一些实施方案中,目标基因组区域包含基因的外显子。在一些实施方案中,目标基因组区域包含基因家族。在一些实施方案中,目标基因组区域包含来自基因家族的一个或多个编码区域。在一些实施方案中,目标基因组区域包含基因组的非编码区域。在一些实施方案中,非编码区域是调控元件。在一些实施方案中,调控元件是顺式调控元件或反式调控元件。在一些实施方案中,顺式调控元件选自:启动子、增强子和沉默子。
9.在一些实施方案中,目标基因组区域跨越大于5kb、大于10kb、大于15kb、大于20kb、大于50kb或大于100kb。在一些实施方案中,grna集包含至少1个、至少2个、至少3个或至少4个grna。在一些实施方案中,来自指导rna集的至少一个grna包含修饰。在一些实施方案中,该修饰选自:2
’‑
o
‑
c1
‑
4烷基如2
’‑
o
‑
甲基(2
’‑
ome)、2
’‑
脱氧基(2
’‑
h)、2
’‑
o
‑
c1
‑
3烷基
‑
o
‑
c1
‑
3烷基如2
’‑
甲氧基乙基(2
’‑
moe)、2
’‑
氟(2
’‑
f)、2
’‑
氨基(2
’‑
nh2)、2
’‑
阿拉伯糖(2
’‑
阿糖)核苷酸、2
’‑
f
‑
阿拉伯糖(2
’‑
f
‑
阿糖)核苷酸、2’锁核酸(lna)核苷酸、2’解锁核酸(2
’‑
unlocked nucleic acid,ulna)核苷酸、l型糖(l
‑
糖)和4
’‑
硫代核糖核苷酸。在一些实施方案中,该修饰是选自硫代磷酸酯、膦基羧酸酯、硫代膦基羧酸酯、烷基膦酸酯和二硫代磷酸酯的核苷酸间键修饰。在一些实施方案中,该修饰选自:2
‑
硫尿嘧啶(2
‑
硫u)、2
‑
硫胞嘧啶(2
‑
硫c)、4
‑
硫尿嘧啶(4
‑
硫u)、6
‑
硫鸟嘌呤(6
‑
硫g)、2
‑
氨基腺嘌呤(2
‑
氨基a)、2
‑
氨基嘌呤、假尿嘧啶、次黄嘌呤、7
‑
脱氮鸟嘌呤、7
‑
脱氮
‑8‑
氮杂鸟嘌呤、7
‑
脱氮腺嘌呤、7
‑
脱氮
‑8‑
氮杂腺嘌呤、5
‑
甲基胞嘧啶(5
‑
甲基c)、5
‑
甲基尿嘧啶(5
‑
甲基u)、5
‑
羟甲基胞嘧啶、5
‑
羟甲基尿嘧啶、5,6
‑
脱氢尿嘧啶、5
‑
丙炔基胞嘧啶、5
‑
丙炔基尿嘧啶、5
‑
乙炔基胞嘧啶、5
‑
乙炔基尿嘧啶、5
‑
烯丙基尿嘧啶(5
‑
烯丙基u)、5
‑
烯丙基胞嘧啶(5
‑
烯丙基c)、5
‑
氨基烯丙基尿嘧啶(5
‑
氨基烯丙基u)、5
‑
氨基烯丙基胞嘧啶(5
‑
氨基烯丙基c)、无碱基核苷酸、z碱基、p碱基、非结构化核酸(una)、异鸟嘌呤(异g)、异胞嘧啶(异c)和5
‑
甲基
‑2‑
嘧啶。
10.在一些实施方案中,多个靶位点中的靶位点与用于核酸酶的pam位点相邻,核酸酶选自:cas9、c2c1、c2c3和cpf1。在一些实施方案中,核酸酶是cas9。在一些实施方案中,核酸酶是失活的cas9。在一些实施方案中,将grna集设计为敲除细胞中的目标基因组区域中的基因。在一些实施方案中,该细胞选自:人类原代细胞、人类永生化细胞、人类诱导的多能干细胞、小鼠胚胎干细胞和中国仓鼠卵巢细胞。在一些实施方案中,设计是由计算机执行的。在一些实施方案中,本文描述了包含指导rna(grna)集的试剂盒,该grna集中的每个grna均通过本文描述的任何方法设计。
11.在某些实施方案中,本文描述了包含可与基因组中的目标基因组区域杂交的grna集的试剂盒,其中该grna集中的每个grna:可与来自目标基因组区域内的多个靶位点中的靶位点杂交,该靶位点与来自该指导rna集的至少一个其他指导rna的多个靶位点中的不同靶位点相距至少30个碱基。在一些实施方案中,靶位点与不同靶位点相距最多170个碱基。在一些实施方案中,grna集包含至少2个、至少3个或至少4个grna。在一些实施方案中,试剂盒还包含一种或多种选自cas9、c2cl、c2c3和cpf1的核酸酶。在一些实施方案中,试剂盒还包含多个grna集,每个grna集可与基因组中不同的目标基因组区域杂交。在一些实施方案中,将一种或多种核酸酶与至少一种grna偶联。
12.在某些实施方案中,本文描述了用于选择一个或多个指导rna(grna)来与物种的基因组基因杂交的方法,该方法包括:对于与该基因杂交的初始指导rna集的多个指导rna中的每一个,通过列举与基因组中潜在的指导rna杂交位点的错配数目来计算脱靶值。在一些实施方案中,多个grna中的每个grna的长度为100个碱基。在一些实施方案中,多个grna中的每个grna的约20个碱基与目标基因组区域内的不同靶位点杂交。在一些实施方案中,错配数目是0。在一些实施方案中,错配数目是1。在一些实施方案中,错配数目是2。在一些实施方案中,错配数目是3。在一些实施方案中,该计算列举了初始指导rna集中的每个grna的错配数目的累计总和。在一些实施方案中,该计算将错配数目组织成分片(shard)。
13.在一些实施方案中,相对于参考基因组计算脱靶值。在一些实施方案中,参考基因组是人类参考基因组。在一些实施方案中,参考基因组选自:智人(homo sapiens)、小家鼠(mus musculus)、黑线仓鼠(cricetulus griseus)、褐家鼠(rattus norvegicus)、斑马鱼(danio rerio)和秀丽隐杆线虫(caenorhabditis elegans)。在一些实施方案中,脱靶值是在参考基因组的1,000,000bp或整个参考基因组上确定的。在一些实施方案中,脱靶值是相对于核酸酶结合位点数据库计算的。在一些实施方案中,核酸酶选自:cas9、c2c1、c2c3和cpf1。在一些实施方案中,核酸酶是cas9。在一些实施方案中,数据库包含多于10,000个、多于50,000个、多于100,000个、多于150,000个、多于200,000个、多于250,000个、多于300,000个、多于350,000个、多于400,000个、多于450,000个、多于500,000个、多于550,000个、多于600,000个、多于650,000个、多于700,000个、多于750,000个、多于800,000个、多于850,000个、多于900,000个、多于950,000个或多于1,000,000个核酸酶结合位点。在一些实施方案中,核酸酶结合位点数据库包含多于2500万个、多于5000万个、多于7500万个、多于1亿个、多于1.25亿个、多于1.5亿个、多于1.75亿个、多于2亿个、多于2.25亿个、多于2.5亿个、多于2.75亿个或多于3亿个核酸酶结合位点。在一些实施方案中,通过列举错配数目来计算脱靶值是由计算机执行的。
14.在某些实施方案中,本文描述了用于设计用于与物种的基因组基因杂交的一个或多个指导rna(grna)的方法,该方法包括:从该基因的多个转录物中选择转录物;以及鉴定初始grna集,其中该初始grna集中的每个grna与选择的转录物的基因中的不同靶位点杂交。在一些实施方案中,初始grna集中的每个grna的长度为约17个至约42个碱基。在一些实施方案中,初始grna集中的每个grna的长度为约20个碱基。在一些实施方案中,初始grna集中的每个grna包含约20个碱基的指导序列和长度为约22个至约80个碱基的恒定区。在一些实施方案中,初始grna集中的每个grna的指导序列选择性地与靶位点杂交。在一些实施方案中,初始grna集中的每个grna的长度为约100个碱基。在一些实施方案中,选择的转录物是数据库中该基因的最丰富的转录物。在一些实施方案中,选择的转录物是该基因的多个转录物中最长的转录物。
15.在一些实施方案中,该方法还包括选择存在于选择的转录物中的基因中的编码区域。在一些实施方案中,选择的编码区域是早期位置外显子。在一些实施方案中,早期位置外显子在该基因的前半部分。在一些实施方案中,早期位置外显子是该基因的第一、第二、第三、第四、第五或第六外显子。在一些实施方案中,选择的编码区域是选择的外显子,该外显子是基因的多个转录物中具有最高丰度的转录物。在一些实施方案中,选择的外显子长于多个转录物中的一个或多个其他外显子。在一些实施方案中,选择的外显子为至少50bp、
至少55bp、至少60bp、至少65bp、至少70bp或至少75bp。在一些实施方案中,选择的外显子是基于长度和丰度两者在多个转录物中选择的。
16.在一些实施方案中,该方法还包括确定初始grna集的每个grna的脱靶值。在一些实施方案中,在物种的整个基因组上确定脱靶值。在一些实施方案中,基因组是物种的参考基因组。在一些实施方案中,物种的参考基因组是含有染色体和未定位重叠群的完整参考装配体。在一些实施方案中,该方法还包括通过列举初始grna集中每个grna与基因组中多个靶位点相比的错配数目来确定脱靶值。在一些实施方案中,多个靶位点包括整个基因组上所有可能的cas核酸酶结合位点。在一些实施方案中,多个靶位点包括至少1000个、10,000个、100,000个、200,000个、300,000个、400,000个、500,000个、600,000个、700,000个、800,000个、900,000个、1,000,000个、2,000,000个或3,000,000个靶位点。在一些实施方案中,多个靶位点包括至少100,000,000个、200,000,000个、300,000,000个、400,000,000个、500,000,000个、600,000,000个、700,000,000个、800,000,000个、900,000,000个、1,000,000,000个或1,500,000,000个靶位点。在一些实施方案中,该列举包括确定初始指导rna集的每个grna的具有0个、1个、2个、3个或4个错配数目的脱靶杂交区域。
17.在一些实施方案中,不同靶位点中的靶位点与用于核酸酶的pam位点相邻,该核酸酶选自:cas9、c2c1、c2c3和cpf1。在一些实施方案中,核酸酶是cas9。在一些实施方案中,pam位点是ngg。在一些实施方案中,核酸酶是失活的cas。在一些实施方案中,物种选自:智人、小家鼠、黑线仓鼠、褐家鼠、斑马鱼和秀丽隐杆线虫。
18.在一些实施方案中,该方法还包括基于中靶效率阈值和脱靶阈值从初始grna集中选择指导rna的子集。在一些实施方案中,通过计算方位(azimuth)得分来确定初始grna集的每个指导rna的中靶效率阈值。在一些实施方案中,方位得分大于0.4。在一些实施方案中,鉴定是基于方位得分和脱靶杂交值的阈值。在一些实施方案中,初始grna集敲除细胞中的基因。在一些实施方案中,初始grna集将突变敲入细胞中的基因中。
19.在一些实施方案中,该细胞选自:人类原代细胞、人类永生化细胞、人类诱导的多能干细胞、小鼠胚胎干细胞和中国仓鼠卵巢细胞。在一些实施方案中,来自初始指导rna集中至少一个指导rna的至少一个核苷酸包含修饰。在一些实施方案中,该修饰选自:2
’‑
o
‑
c1
‑
4烷基如2
’‑
o
‑
甲基(2
’‑
ome)、2
’‑
脱氧基(2
’‑
h)、2
’‑
o
‑
c1
‑
3烷基
‑
o
‑
c1
‑
3烷基如2
’‑
甲氧基乙基(2
’‑
moe)、2
’‑
氟(2
’‑
f)、2
’‑
氨基(2
’‑
nh2)、2
’‑
阿拉伯糖(2
’‑
阿糖)核苷酸、2
’‑
f
‑
阿拉伯糖(2
’‑
f
‑
阿糖)核苷酸、2’锁核酸(lna)核苷酸、2’解锁核酸(ulna)核苷酸、l型糖(l
‑
糖)和4
’‑
硫代核糖核苷酸。在一些实施方案中,该修饰是选自硫代磷酸酯、膦基羧酸酯、硫代膦基羧酸酯、烷基膦酸酯和二硫代磷酸酯的核苷酸间键修饰。在一些实施方案中,该修饰选自:2
‑
硫尿嘧啶(2
‑
硫u)、2
‑
硫胞嘧啶(2
‑
硫c)、4
‑
硫尿嘧啶(4
‑
硫u)、6
‑
硫鸟嘌呤(6
‑
硫g)、2
‑
氨基腺嘌呤(2
‑
氨基a)、2
‑
氨基嘌呤、假尿嘧啶、次黄嘌呤、7
‑
脱氮鸟嘌呤、7
‑
脱氮
‑8‑
氮杂鸟嘌呤、7
‑
脱氮腺嘌呤、7
‑
脱氮
‑8‑
氮杂腺嘌呤、5
‑
甲基胞嘧啶(5
‑
甲基c)、5
‑
甲基尿嘧啶(5
‑
甲基u)、5
‑
羟甲基胞嘧啶、5
‑
羟甲基尿嘧啶、5,6
‑
脱氢尿嘧啶、5
‑
丙炔基胞嘧啶、5
‑
丙炔基尿嘧啶、5
‑
乙炔基胞嘧啶、5
‑
乙炔基尿嘧啶、5
‑
烯丙基尿嘧啶(5
‑
烯丙基u)、5
‑
烯丙基胞嘧啶(5
‑
烯丙基c)、5
‑
氨基烯丙基尿嘧啶(5
‑
氨基烯丙基u)、5
‑
氨基烯丙基胞嘧啶(5
‑
氨基烯丙基c)、无碱基核苷酸、z碱基、p碱基、非结构化核酸(una)、异鸟嘌呤(异g)、异胞嘧啶(异c)和5
‑
甲基
‑2‑
嘧啶。
20.在一些实施方案中,选择和鉴定是由计算机执行的。在一些实施方案中,初始grna集中的每个grna可与靶位点杂交,该靶位点与来自初始指导rna集的至少一个其他指导rna的靶位点相距至少30个碱基。在一些实施方案中,本文描述了包含指导rna(grna)集的试剂盒,该grna集中的每个grna均通过本文描述的任何方法设计。
21.在某些实施方案中,本文描述了用于编辑目标基因组区域的方法,该方法包括:使包含目标基因组区域的细胞群体与以下项接触:(i)包含靶向目标基因组区域的至少两个grna的grna集和(ii)核酸酶;其中包含至少两个grna的grna集的编辑效率高于至少两个grna中的每一个的单独编辑效率。在一些实施方案中,目标基因组区域是基因的编码区域。在一些实施方案中,编码区域是基因的外显子。在一些实施方案中,目标基因组区域是基因组中的非编码区域。在一些实施方案中,非编码区域是调控元件。在一些实施方案中,调控元件是顺式调控元件或反式调控元件。在一些实施方案中,顺式调控元件选自:启动子、增强子和沉默子。在一些实施方案中,该方法还包括使细胞与供体多核苷酸接触。在一些实施方案中,供体多核苷酸包含相对于细胞的野生型基因型的点突变、等位基因、标签或外源性外显子。
22.在一些实施方案中,编辑效率是接触后细胞群体中包含非野生型基因型的细胞的比例。在一些实施方案中,非野生型基因型是基因的敲除。在一些实施方案中,非野生型基因型是相对于野生型基因型的插入或缺失。在一些实施方案中,细胞群体中至少50%、至少60%、至少70%、至少80%、至少90%或至少95%的细胞包含非野生型基因型。在一些实施方案中,至少两个grna中的每个grna与目标基因组区域中的不同靶位点杂交。在一些实施方案中,至少两个grna中的每个grna可与靶位点杂交,该靶位点与来自指导rna集的至少一个其他指导rna的靶位点相距至少30个碱基。
23.在一些实施方案中,该方法还包括引入靶向多个目标基因组区域的多个grna集。在一些实施方案中,将多个grna集的每一个与细胞群体的多个子集的每一个接触。在一些实施方案中,多个grna集中的每一个靶向多个目标基因组区域中的不同目标基因组区域。在一些实施方案中,在细胞群体的多个子集的至少50%中,至少50%、至少60%、至少70%、至少80%、至少90%或至少95%的细胞包含非野生型基因型。在一些实施方案中,在细胞群体的多个子集的至少70%中,至少50%、至少60%、至少70%、至少80%、至少90%或至少95%的细胞包含非野生型基因型。在一些实施方案中,在细胞群体的多个子集的至少90%中,至少50%、至少60%、至少70%、至少80%、至少90%或至少95%的细胞包含非野生型基因型。
24.在一些实施方案中,该方法还包括筛选细胞群体的表型。
25.在某些实施方案中,本文描述了用于设计一个或多个用于与物种的基因组的基因杂交的指导rna(grna)的计算机系统,该系统包括:一个或多个计算机处理器;和非暂时性计算机可读介质,该介质包括可操作的指令,当由一个或多个计算机处理器执行时,该指令使系统:从基因的多个转录物中选择转录物,并鉴定与选择的转录物的基因内的多个靶位点中的不同靶位点杂交的初始grna集。在一些实施方案中,初始grna集中的每个grna的长度为约17个至约42个碱基。在一些实施方案中,初始grna集中的每个grna的长度为约20个碱基。在一些实施方案中,初始grna集中的每个grna包含约20个碱基的指导序列,并且还包含长度为约22个至约80个碱基的恒定区。在一些实施方案中,初始grna集中的每个grna的
指导序列选择性地与基因杂交。在一些实施方案中,初始grna集中的每个grna的长度为约100个碱基。在一些实施方案中,选择的转录物是数据库中该基因的最丰富的转录物。在一些实施方案中,选择的转录物是该基因的多个转录物中最长的转录物。
26.在一些实施方案中,所述指令还可操作以使系统选择存在于选择的转录物中的基因中的编码区域,从而得到选择的编码区域。在一些实施方案中,选择的编码区域是早期位置外显子。在一些实施方案中,早期位置外显子在该基因的前半部分。在一些实施方案中,早期位置外显子是该基因的第一、第二、第三、第四、第五或第六外显子。在一些实施方案中,选择的编码区域是选择的外显子,该外显子是基因的多个转录物中具有最高丰度的转录物。在一些实施方案中,选择的外显子长于多个转录物中的一个或多个其他外显子。在一些实施方案中,选择的外显子为至少50bp、至少55bp、至少60bp、至少65bp、至少70bp或至少75bp。在一些实施方案中,选择的外显子是基于长度和丰度两者在多个转录物中选择的。在一些实施方案中,所述指令还可操作以使系统确定初始grna集中的每个grna的脱靶值。在一些实施方案中,所述指令还可操作以使系统在物种的整个基因组上进行确定。
27.在一些实施方案中,基因组是物种的参考基因组。在一些实施方案中,物种的参考基因组是包含染色体和未定位重叠群的完整参考装配体。在一些实施方案中,所述指令还可操作以使系统通过列举初始grna集中每个grna与基因组中多个靶位点相比的错配数目来确定脱靶值。在一些实施方案中,多个靶位点包括整个基因组上所有可能的cas核酸酶结合位点。在一些实施方案中,多个靶位点包括至少1000个、10,000个、100,000个、200,000个、300,000个、400,000个、500,000个、600,000个、700,000个、800,000个、900,000个、1,000,000个、2,000,000个或3,000,000个靶位点。在一些实施方案中,多个靶位点包括至少100,000,000个、200,000,000个、300,000,000个、400,000,000个、500,000,000个、600,000,000个、700,000,000个、800,000,000个、900,000,000个、1,000,000,000个或1,500,000,000个靶位点。在一些实施方案中,列举包括确定初始指导rna集的每个grna的具有0个、1个、2个、3个或4个错配数目的脱靶杂交区域。在一些实施方案中,多个靶位点中的靶位点与用于核酸酶的pam位点相邻,核酸酶选自:cas9、c2c1、c2c3和cpf1。在一些实施方案中,核酸酶是cas9。在一些实施方案中,pam位点是ngg。在一些实施方案中,物种选自:智人、小家鼠、黑线仓鼠、褐家鼠、斑马鱼和秀丽隐杆线虫。
28.在一些实施方案中,所述指令还可操作以基于中靶效率阈值和脱靶阈值从初始grna集中选择指导rna的子集。在一些实施方案中,通过计算方位得分来确定初始grna集的每个指导rna的中靶效率阈值。在一些实施方案中,方位得分大于0.4。在一些实施方案中,所述指令还可操作以使系统基于方位得分和脱靶杂交值的阈值来鉴定初始grna集。在一些实施方案中,来自初始指导rna集中至少一个指导rna的至少一个核苷酸包含修饰。在一些实施方案中,该修饰选自:2
’‑
o
‑
c1
‑
4烷基如2
’‑
o
‑
甲基(2
’‑
ome)、2
’‑
脱氧基(2
’‑
h)、2
’‑
o
‑
c1
‑
3烷基
‑
o
‑
c1
‑
3烷基如2
’‑
甲氧基乙基(2
’‑
moe)、2
’‑
氟(2
’‑
f)、2
’‑
氨基(2
’‑
nh2)、2
’‑
阿拉伯糖(2
’‑
阿糖)核苷酸、2
’‑
f
‑
阿拉伯糖(2
’‑
f
‑
阿糖)核苷酸、2’锁核酸(lna)核苷酸、2’解锁核酸(ulna)核苷酸、l型糖(l
‑
糖)和4
’‑
硫代核糖核苷酸。在一些实施方案中,该修饰是选自硫代磷酸酯、膦基羧酸酯、硫代膦基羧酸酯、烷基膦酸酯和二硫代磷酸酯的核苷酸间键修饰。在一些实施方案中,该修饰选自:2
‑
硫尿嘧啶(2
‑
硫u)、2
‑
硫胞嘧啶(2
‑
硫c)、4
‑
硫尿嘧啶(4
‑
硫u)、6
‑
硫鸟嘌呤(6
‑
硫g)、2
‑
氨基腺嘌呤(2
‑
氨基a)、2
‑
氨基嘌呤、假尿嘧啶、次黄嘌呤、
7
‑
脱氮鸟嘌呤、7
‑
脱氮
‑8‑
氮杂鸟嘌呤、7
‑
脱氮腺嘌呤、7
‑
脱氮
‑8‑
氮杂腺嘌呤、5
‑
甲基胞嘧啶(5
‑
甲基c)、5
‑
甲基尿嘧啶(5
‑
甲基u)、5
‑
羟甲基胞嘧啶、5
‑
羟甲基尿嘧啶、5,6
‑
脱氢尿嘧啶、5
‑
丙炔基胞嘧啶、5
‑
丙炔基尿嘧啶、5
‑
乙炔基胞嘧啶、5
‑
乙炔基尿嘧啶、5
‑
烯丙基尿嘧啶(5
‑
烯丙基u)、5
‑
烯丙基胞嘧啶(5
‑
烯丙基c)、5
‑
氨基烯丙基尿嘧啶(5
‑
氨基烯丙基u)、5
‑
氨基烯丙基胞嘧啶(5
‑
氨基烯丙基c)、无碱基核苷酸、z碱基、p碱基、非结构化核酸(una)、异鸟嘌呤(异g)、异胞嘧啶(异c)和5
‑
甲基
‑2‑
嘧啶。在一些实施方案中,该集中的每个grna:可与目标基因组区域中的不同靶位点杂交;且可与与该指导rna集中至少一个其他指导rna的靶位点相距至少30个碱基靶位点杂交。
29.在某些实施方案中,本文描述了用于设计一个或多个与个体的基因组区域杂交的指导rna的方法,该方法包括:使用个体的基因组,确定潜在的grna靶位点;对于潜在的grna靶位点中的每个潜在的grna靶位点,确定预期的指导rna的脱靶值;以及鉴定一个或多个具有改进的效用指数的指导rna。在一些实施方案中,一个或多个grna中的每个grna的长度为约100个碱基。在一些实施方案中,一个或多个grna中的每个grna的约20个碱基可与潜在的grna靶位点中的每个潜在的grna靶位点杂交。在一些实施方案中,效用指数是治疗指数。在一些实施方案中,治疗指数包括脱靶结合的减少、增加的中靶效率、增加的敲除效率、增加的敲入效率或crispr干扰的调节。在一些实施方案中,个体是人类。在一些实施方案中,个体患有病症。在一些实施方案中,个体是患有一种或多种病症的群体队列的一部分。在一些实施方案中,一种或多种病症包括一种或多种类型的癌症。在一些实施方案中,病症是癌症。
30.在一些实施方案中,将一个或多个指导rna设计为敲除个体细胞基因组区域中的基因。在一些实施方案中,将一个或多个指导rna设计为在个体细胞基因组区域中敲入突变。在一些实施方案中,所述方法还包括用具有改进的效用指数的一个或多个指导rna编辑细胞。在一些实施方案中,潜在的grna靶位点的确定和一个或多个指导rna的鉴定是由计算机执行的。
31.在某些实施方案中,本文描述了评估crispr试剂对个体的脱靶效应的方法,包括:使用个体的基因组,由计算机系统通过列举与个体基因组中的潜在靶位点的错配数目来确定crispr试剂的脱靶值。在一些实施方案中,crispr试剂是治疗剂。在一些实施方案中,crispr试剂是长度为约100个碱基的指导rna(grna)。在一些实施方案中,grna包含可与靶标杂交的20个碱基。在一些实施方案中,针对可与靶标杂交的20个碱基中的每一个独立地计算错配数目。
32.在一些实施方案中,列举包括分别列举与潜在靶位点的1个、2个、3个、4个或5个错配中的至少两个。在一些实施方案中,潜在靶位点的数目为至少1000个、10,000个、100,000个、200,000个、300,000个、400,000个、500,000个、600,000个、700,000个、800,000个、900,000个、1,000,000个、2,000,000个或3,000,000个。在一些实施方案中,该方法还包括输出报告,该报告列举与个体基因组中的潜在靶位点的错配数目。在一些实施方案中,输出显示在屏幕上。在一些实施方案中,crispr试剂的脱靶效应的评估用作伴随诊断。
33.在某些实施方案中,本文描述了用于验证预期的grna的方法,该方法包括:在计算机系统上确定预期的grna在基因组或基因组一部分中的多个脱靶位点;使用计算机系统针对多个脱靶位点中的每个脱靶位点计算预期的grna的脱靶值;以及使用计算机系统,使用
脱靶值预测预期的grna的活性。在一些实施方案中,预测列出了中靶杂交位点或脱靶杂交位点的潜力。在一些实施方案中,基因组或基因组的一部分超过1,000,000bp。在一些实施方案中,通过计算grna与多个脱靶位点的错配数目来确定脱靶值。在一些实施方案中,错配数目为0个、1个、2个、3个和/或4个。在一些实施方案中,多个脱靶位点包括至少100,000,000个脱靶位点。
34.在某些实施方案中,本文描述了计算机系统,该系统包括:被配置为选择目标物种和来自目标物种的目标基因的用户界面系统;被配置为鉴定目标基因的一个或多个指导rna(grna)序列的与用户界面集成的设计模块;被配置为示出选择的grna的输出系统;以及被配置为启动rna合成仪合成一个或多个grna的激活单元。在一些实施方案中,每个grna的长度为约20个碱基。在一些实施方案中,用户界面系统包括超过100个、1000个、10,000个、100,000个、500,000个不同参考基因组的选择。在一些实施方案中,设计模块被配置为基于脱靶值和脱靶效率得分选择grna。在一些实施方案中,设计模块被配置为访问云端的参考基因组。在一些实施方案中,设计模块被配置为访问超过10,000个、20,000个、30,000个、40,000个、50,000个、60,000个、70,000个、80,000个、90,000个、100,000个、110,000个或120,000个参考基因组。在一些实施方案中,用户界面包括:用于获取个体基因组的输入的基因组数据接收模块。在一些实施方案中,基因组数据接收模块被配置为从服务器或用户上传的文件中获取个体的基因组。
35.在某些实施方案中,本文描述了系统,其包含:被配置为向用户提供对超过10,000个参考基因组的访问的界面;被配置为对超过50,000个参考基因组的任何一个中的基因选择一个或多个指导rna的软件;以及配置为示出选择的指导rna的输出系统。在一些实施方案中,该系统包含超过20,000个、30,000个、40,000个、50,000个、60,000个、70,000个、80,000个、90,000个、100,000个、110,000个或120,000个参考基因组。在一些实施方案中,该系统还包括被配置为激活和启动至少一个或多个指导rna的合成的脚本。
36.在某些实施方案中,本文描述了用于设计指导rna(grna)的方法,该方法包括:通过计算机系统鉴定基因的初级转录物;通过计算机系统鉴定初级转录物与多个替代转录物之间的共同外显子;通过计算机系统鉴定共同外显子内的核酸酶靶位点;通过计算机系统计算核酸酶的参考基因组序列中的脱靶结合位点的数目,从而得出核酸酶脱靶结合位点的计算出的数目;通过计算机系统计算中靶效率得分,从而得出计算出的中靶效率得分;以及通过计算机系统输出至少一个grna序列,其中该至少一个grna序列包含计算出的中靶效率高于阈值并且计算出的核酸酶脱靶结合位点数目为零的序列。在一些实施方案中,该方法还包括指导与靶位点具有部分互补性的核酸的合成。在一些实施方案中,本文描述了包含指导rna(grna)集的试剂盒,通过本文所述的任何方法设计grna集中的每个grna。
37.在某些实施方案中,本文描述了用于处理通过网络来自用户的生物聚合物合成请求的系统,该系统包括:被配置为通过网络与用户的数字计算机进行通信的通信接口;被配置为存储一个或多个参考基因组的参考基因组数据库;包括一个或多个可操作地偶联至通信接口和数据库的计算机处理器的计算机,其中一个或多个计算机处理器被单独或共同配置为:(a)通过网络从通信接口接收来自用户的数字计算机的生物聚合物合成请求,该生物聚合物合成请求包括靶基因组信息;(b)相对于来自数据库的一个或多个参考基因组处理
靶基因组信息,以鉴定与靶基因组信息对应的靶序列;(c)执行算法以生成与靶序列至少部分互补的第一指导核糖核酸(grna)序列集,并计算第一grna序列集中每个grna序列的脱靶互补性得分;(d)输出第二grna序列集,以示出在用户的数字计算机的图形用户界面上,其中第二grna序列集中的每一个均具有低于阈值的计算出的脱靶互补性得分;以及(e)从用户的数字计算机接收对第二grna序列集中的给定grna序列的选择。
38.在一些实施方案中,一个或多个计算机处理器被单独地或共同地编程以将给定的grna序列引导到用于合成grna序列的伫列中。在一些实施方案中,参考基因组数据库中的至少一个基因组是个体的个性化基因组。在一些实施方案中,参考基因组数据库中的至少一个基因组是受病症困扰人群的个性化基因组集。在一些实施方案中,参考基因组是智人参考基因组。在一些实施方案中,该系统还包括输出预测的基因组序列,其中该预测的基因组序列代表使用来自第二grna序列集的一个或多个grna编辑靶基因组信息的预测输出。在一些实施方案中,预测的基因组序列包含基因组缺失。在一些实施方案中,预测的基因组序列包含基因组插入。在一些实施方案中,该计算得到方位得分。在一些实施方案中,第二grna序列集示出高于某个阈值的至少两个grna。在一些实施方案中,参考基因组数据库包括至少5万个参考基因组。在一些实施方案中,参考基因组数据库包括至少12万个参考基因组。
39.在某些实施方案中,本文描述了用于处理通过网络来自用户的生物聚合物合成请求的方法,该方法包括:(a)由计算机系统通过网络接收来自用户的数字计算机的生物聚合物合成请求,该生物聚合物合成请求包括靶基因组信息;(b)由计算机系统相对于来自参考基因组数据库的一个或多个参考基因组处理靶基因组信息,以鉴定与靶基因组信息对应的靶序列;(c)使用一个或多个计算机处理器执行算法以(i)生成与靶序列至少部分互补的第一指导核糖核酸(grna)序列集,并(ii)对每个grna序列计算第一grna序列集中每个grna序列的脱靶互补性得分;(d)由计算机系统输出第二grna序列集,以示出在用户的数字计算机的图形用户界面上,其中第二grna序列集中的每一个均包含低于阈值的计算出的脱靶互补性得分;以及(e)从用户的数字计算机接收合成第二grna序列集中的给定grna序列的请求。
40.在一些实施方案中,一个或多个计算机处理器被单独地或共同地编程以指导在合成仪中合成第二grna序列集中的给定grna序列。在一些实施方案中,参考基因组数据库中的至少一个基因组是个体的个性化基因组。在一些实施方案中,参考基因组数据库中的至少两个基因组是受病症困扰人群的个性化基因组。在一些实施方案中,参考基因组是智人参考基因组。在一些实施方案中,该方法还包括输出预测的基因组序列,其中该预测的基因组序列代表使用来自第二grna序列集的一个或多个grna编辑靶基因组信息的预测输出。在一些实施方案中,预测的基因组序列包含基因组缺失。在一些实施方案中,预测的基因组序列包含基因组插入。在一些实施方案中,该计算得到方位得分。在一些实施方案中,第二grna序列集示出高于某个阈值的至少两个grna。在一些实施方案中,参考基因组数据库包括至少5万个参考基因组。在一些实施方案中,参考基因组数据库包括至少12万个参考基因组。
41.在一些实施方案中,本文描述了非暂时性计算机可读介质,该介质包括可操作的指令,当由一个或多个计算机处理器执行时,该指令使一个或多个计算机处理器执行本文所述的任何方法。
42.在某些实施方案中,本文描述了包括机器可执行代码的非暂时性计算机可读介质,在由一个或多个计算机处理器执行后,该机器可执行代码实现用于处理通过网络来自用户的生物聚合物合成请求的方法。该方法包括:(a)通过网络接收来自用户的数字计算机的生物聚合物合成请求,该生物聚合物合成请求包括靶基因组信息;(b)相对于来自参考基因组数据库的一个或多个参考基因组处理靶基因组信息,以鉴定与靶基因组信息对应的靶序列;(c)执行算法以生成与靶序列至少部分互补的第一指导核糖核酸(grna)序列集,并计算第一grna序列集中每个grna序列的脱靶互补性得分;(d)输出第二grna序列集,以示出在用户的数字计算机的图形用户界面上,其中第二grna序列集中的每一个均具有低于阈值的计算出的脱靶互补性得分;以及(e)从用户的数字计算机接收对第二grna序列集中的给定grna序列的选择。援引并入
43.本说明书中提到的所有出版物、专利和专利申请均通过引用并入本文,其程度如同明确且单独地指出每个单独的出版物、专利或专利申请通过引用并入。在通过引用并入的出版物和专利或专利申请与说明书中包含的公开内容相抵触的程度上,本说明书旨在取代和/或优先于任何此类矛盾的物质。
附图说明
44.本发明的新颖性特征在所附的权利要求书中具体阐述。通过参考以下对利用到本发明原理的说明性实施方案加以阐述的详细描述以及附图,将获取对本发明的特征和优点的更好的理解。在这些附图中:
45.图1显示了设计用于与物种的基因组基因杂交的一个或多个指导物的方法的流程图的示例。
46.图2显示了物种基因组基因的多个转录物的表格的示例。
47.图3显示了一个或多个grna靶向的转录物的早期编码区域的示例。
48.图4a显示了来自转录物的多个外显子的相对表现度和外显子长度的图的示例。图4b显示了指导物的示例以及它们的脱靶和中靶活性分析。
49.图5显示了用于计算多个指导物在整个基因组上的脱靶值的数据处理架构。
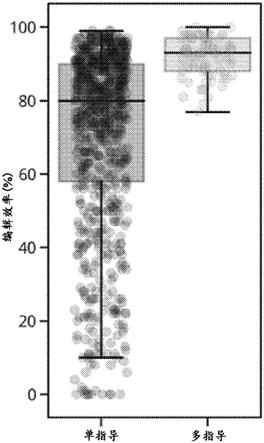
50.图6显示了验证用于与物种的基因组基因杂交的一个或多个指导物的方法的流程图的示例。
51.图7a
‑
图7d示出了用于选择目标基因组和目标基因以请求设计用于与基因组基因杂交的一个或多个指导物的图形用户界面(gui)的窗口的示例。图7a示出了在选择目标基因组和目标基因之前的窗口。图7b示出了显示与键入的输入匹配的基因组列表的窗口。图7c示出了显示与键入的输入匹配的基因列表的窗口。图7d示出了选择目标基因组、目标基因和核酸酶之后的窗口。
52.图8示出了gui窗口的示例,其用于展示设计用于与目标基因组的基因杂交的一个或多个指导物的进程。
53.图9a
‑
图9d示出了gui窗口的示例,其用于展示被设计为与目标基因组的基因杂交的一个或多个指导物。图9a示出了显示设计一个或多个grna的概述的窗口。图9b示出了对排序最高的grna的选择。图9c示出了显示关于选择的grna的信息的窗口。图9d示出了显示
被设计为与目标基因组的基因杂交的其他grna的窗口。
54.图10a
‑
图10e示出了gui窗口的示例,其用于展示关于设计的指导物的详细信息。图10a示出了选择的grna的性能的概述。图10b示出了cas
‑
grna复合物与目标靶区域相互作用的示意图,其中选择了rna指导序列。图10c示出了cas
‑
grna复合物与目标靶区域相互作用的示意图,其中选择了pam区域。图10d示出了cas
‑
grna复合物与目标靶区域相互作用的示意图,其中选择了靶序列。图10e示出了选择的grna的脱靶位点的列表。
55.图11a
‑
图11b示出了gui窗口的示例,其用于选择和购买被设计为与目标基因组的基因杂交的一个或多个指导物的子集。图11a示出了显示选择grna子集的窗口。图11b示出了显示选择的grna的窗口,该窗口具有订购修饰的或未修饰的grna的附加选择。
56.图12a
‑
图12b示出了gui窗口的示例,其用于选择目标物种的基因组并输入先前生成的指导序列以请求验证指导性能。图12a示出了在选择目标基因组和指导序列之前的窗口。图12b示出了在选择目标基因组和指导序列之后的窗口。
57.图13a
‑
图13b示出了gui窗口的示例,其用于显示关于指导物的验证的详细信息。图13a示出了预定grna的性能的概述。图13b示出了预定grna的脱靶位点的列表。
58.图14示出了可以被编程或以其他方式配置为实现本文提供的方法的计算机系统。
59.图15示出了单指导rna与多指导rna的编辑效率。对于单指导rna,每个数据点代表一个转染的sgrna的百分比编辑效率或ko得分。对于多指导rna,每个数据点代表三个共转染的sgrna的ko得分。
60.图16示出了相对于多grna集中的指导rna的间隔的百分比编辑效率。
61.图17示出了对于每对靶向的基因,使用多个grna进行双敲除的百分比编辑效率。
62.图18a
‑
图18b示出了使用多指导敲除设计进行的阵列化文库筛选。图18a示出了针对功能测定的文库筛选。图18b示出了针对编辑效率的文库筛选。
具体实施方式
63.尽管本文已经示出并描述了本发明的各种实施方案,但对于本领域技术人员显而易见的是,这些实施方案仅以示例的方式提供。在不脱离所公开的实施方案的情况下,本领域技术人员现将会想到很多变化、改变和替代。应当理解,可采用本文所述的本发明实施方案的各种替代方案。
64.本文使用的术语仅为了描述具体情况,而不意在限制。除了本领域技术人员对这些术语的理解之外,讨论以下术语以说明其在本说明书中使用时的含义。如本文和所附权利要求书中所用,单数形式的“一个”、“一种”和“该”包括复数指示物,除非上下文另有明确规定。还应注意的是,权利要求书可以被撰写为排除任何可选要素。照此,该陈述旨在作为与权利要求要素的叙述相关联地使用诸如“仅”、“只”等排他性术语的先行基础,或作为“否定”限制的使用的先行基础。
65.本文提供了某些范围,其中数值之前带有术语“约”。术语“约”在本文中用于为其后的确切数字以及与该术语后的数字接近或近似的数字提供文字支持。在确定数字是否接近或近似于具体列举的数字时,接近或近似的未列举数字可以是在给出该数字的上下文中提供了具体列举数字的基本等同形式的数字。在提供数值范围的情况下,应理解的是,在该范围的上限与下限之间的每个中间值(至下限的单位的十分之一,除非上下文另有明确规
定)以及在规定范围内的任何其他规定或中间值均包括在本文所述的方法和组合物中。这些较小范围的上限和下限可以独立地包括在该较小范围内,并且也包括在本文所述的方法和组合物中,但受到在规定范围内的任何特别排除的限制。在规定的范围包括一个或两个极限的情况下,排除其中一个或两个极限的范围也包括在本文所述的方法和组合物中。
66.如本文可互换使用的,术语“多核苷酸”或“核酸”通常可以指任何长度的核苷酸的聚合形式,即核糖核苷酸和/或脱氧核糖核苷酸。因此,这些术语包括但不限于单链、双链或多链dna或rna、基因组dna、互补dna(cdna)、指导rna(grna)、信使rna(mrna)、dna
‑
rna杂合体,或包含嘌呤和嘧啶碱基或其他天然的、化学或生物化学修饰的、非天然的或衍生的核苷酸碱基的聚合物。如本文所用,术语“寡核苷酸”通常可以指约5个至约100个核苷酸之间的单链或双链dna或rna的多核苷酸。然而,出于本公开内容的目的,寡核苷酸的长度可能没有上限。在一些情况下,寡核苷酸可以被称为“寡聚物(oligomer)”或“寡聚体(oligo)”,并且可以从基因中分离或通过本领域已知的方法化学合成。术语“多核苷酸”和“核酸”应理解为包括单链(如有义或反义)和双链多核苷酸。
67.如本文所用,术语“修饰的核苷酸”通常可以指相对于天然存在的碱基、糖和/或磷酸二酯键或骨架部分,对碱基、糖和/或磷酸二酯键或骨架部分(包括核苷酸磷酸)中的一个或多个的化学结构具有修饰的核苷酸。
68.如本文所用,术语“杂交”通常可指其中完全或部分互补的多核苷酸链在合适的杂交条件下结合在一起以形成其中两个组成链通过氢键连接的双链结构或区域的过程。在一些情况下,修饰的核苷酸可以形成允许或促进杂交的氢键。在一些情况下,可以认为主题dna靶向的rna分子的蛋白质结合区段的鸟嘌呤(g)与尿嘧啶(u)互补,反之亦然。
69.如本文所用,术语“切割”通常可以指多核苷酸的核糖基磷酸二酯骨架中的共价磷酸二酯键的断裂。术语“切割”可涵盖导致单链断裂和双链断裂的切割。在一些情况下,切割可能会导致平整末端或交错(或黏性)末端的产生。
70.如本文所用,术语“crispr/cas”可指包含指导rna(grna)和crispr相关的(cas)核酸内切酶的核糖核蛋白复合物。术语“crispr”是指成簇的规律间隔的短回文重复序列及其相关系统。crispr被发现是一种适应性防御系统,能够使细菌和古菌检测并沉默外来核酸(例如,来自病毒或质粒),它可以适用于多种细胞类型,以允许序列特异方式的多核苷酸编辑。在一些情况下,crispr系统的一个或多个元件可以衍生自i型、ii型或iii型crispr系统。在crispr ii型系统中,指导rna可以与cas相互作用并将cas酶的核酸酶活性引导至靶区域。靶区域可以包含“原间隔子”和“原间隔子相邻基序”(pam),并且两个结构域对于cas酶介导的活性(例如,切割)可能都是必需的。原间隔子可以被称为靶位点(或基因组靶位点)。grna可以与原间隔子的相对链(结合位点)配对(或杂交),以将cas酶引导至靶区域。pam位点通常是指被cas酶识别并且在一些情况下为cas酶活性所必需的短序列。pam位点的序列和核苷酸数目可以根据cas酶的类型而有所不同。
71.如本文所用,术语“cas”通常可以指野生型cas蛋白、其片段或其突变体或变体。
72.cas蛋白可以包含crispr/cas i型、ii型或iii型系统的蛋白或其衍生蛋白,该蛋白可以具有rna指导的多核苷酸结合或核酸酶活性。合适的cas蛋白的示例包括casx、cas3、cas4、cas5、cas5e(或casd)、cas6、cas6e、cas6f、cas7、cas8a1、cas8a2、cas8b、cas8c、cas9(也称为csnl和csxl2)、cas10、cas10d、casf、casg、cash、csy1、csy2、csy3、cse1(或casa)、
cse2(或casb)、cse3(或case)、cse4(或casc)、csc1、csc2、csa5、csn2、csm2、csm3、csm4、csm5、csm6、cmr1、cmr3、cmr4、cmr5、cmr6、csb1、csb2、csb3、csx17、csx14、csx10、csx16、csax、csx3、csz1、csx15、csf1、csf2、csf3、csf4、cu1966、其同系物及其修饰形式。在一些情况下,cas蛋白可包含crispr/cas v型或vi型系统的蛋白或其衍生蛋白,如cpf1(cas12a)、c2c1(cas12b)、c2c2、其同系物及其修饰形式。在一些情况下,cas蛋白可以是催化死亡的或失活的cas(dcas)。
73.在一些情况下,cas蛋白可以是cas9蛋白。在一些情况下,cas9蛋白识别的pam序列可以是ngg,其中“n”是任何核苷酸。
74.如本文所用,术语“指导rna”或“grna”通常可以指可以与cas蛋白结合并帮助将cas蛋白靶向靶多核苷酸(例如,dna)内的特定位置的rna分子(或一组rna分子的总称)。指导rna可以包含crispr rna(crrna)区段和反式激活crrna(tracrrna)区段。如本文所用,术语“crrna”或“crrna区段”可以指包含多核苷酸靶向的指导序列、茎序列和任选的5
’‑
突出端序列的rna分子或其部分。术语“tracrrna”或“tracrrna区段”可以指包括蛋白质结合区段(例如,蛋白质结合区段能够与crispr相关的蛋白如cas9相互作用)的rna分子或其部分。术语“指导rna”可以涵盖单指导rna(sgrna),其中crrna区段和tracrrna区段位于同一rna分子中。术语“指导rna”也可以共同涵盖一组两个或更多个rna分子,其中crrna区段和tracrrna区段位于不同的rna分子中。
75.在一些情况下,crispr/cas活性可用于需要以位点特异性(靶向)方式修饰核酸的任何体外或体内应用,例如基因敲除(ko)、基因敲入(ki)、基因编辑、基因标记等,例如在基因治疗中使用。核酸可以是dna或rna。基因治疗的示例包括治疗疾病或作为抗病毒、抗病原或抗癌疗法;农业中转基因生物体的生产;为治疗、诊断或研究目的由细胞大规模生产蛋白质;诱导多能干细胞(ips细胞或ipsc)的诱导;以及靶向病原体基因以进行删除或替换。在一些情况下,cas可以是催化死亡的或失活的cas(dcas),并且所得的crispr/dcas系统可用于基因表达的序列特异性抑制(crispr干扰)或激活(crispr激活)。
76.如本文所用,术语“受试者”、“个体”或“患者”通常可以指可能需要和/或接受了治疗的所有生物体或其集合,如农场动物、伴侣动物或人类或其集合。在一些情况下,术语“受试者”可以是细胞或其细胞系。
77.如本文所用,术语“基因”通常可以指编码功能性遗传信息的核苷酸序列,例如,举例而言,编码多肽(例如,蛋白质)、转移rna(trna)或核糖体rna(rrna)的核苷酸序列。基因可以包含dna、rna或其他核苷酸。设计寡核苷酸的方法
78.一方面,本公开内容提供了一种用于设计一个或多个用于与目标基因组区域杂交的指导rna(grna)的方法。目标基因组区域可以是某物种的基因组的基因。该方法可以包括从基因的多个转录物中选择转录物。该方法可以包括鉴定与选择的转录物的基因中的不同靶位点杂交的初始grna集。该基因可以是目标基因。目标基因组区域可以是基因组的非编码区域。非编码区域可以是调控元件。调控元件可以是顺式调控元件或反式调控元件。顺式调控元件可以是启动子、增强子或沉默子。
79.可以从多个数据库获取包括物种的基因组和/或物种的参考基因组的信息。在一些情况下,多个数据库可以包括包含来自dna(dna
‑
seq)和/或rna(rna
‑
seq)的测序数据的
基因和/或基因组数据库。此类基因组数据库的示例包括gencode、ncbi、ensembl、{appris}和nih人类微生物组计划。替代地或附加地,可以从个性化基因组数据库检索个体的基因组信息,包括但不限于23andme、decode genetics、gene by gene、gene planet、dna ancestry、ubiome和医疗服务提供者。在一些情况下,包括目标物种的基因组的至少一部分的必要信息可以由用户提供(例如,经由诸如个人计算机之类的用户设备上的用户界面)。
80.物种的基因组可以包含物种(例如,细胞或生物体)中存在的一些或完整的遗传物质集。物种的示例包括但不限于哺乳动物(例如,智人、小家鼠、黑线仓鼠、褐家鼠、倭黑猩猩(pan paniscus))、鱼(例如,斑马鱼、白条双锯鱼(amphiprion frenatus))、昆虫(例如,黑腹果蝇(drosophila melanogaster))、植物(例如,拟南芥(arabidopsis thaliana))、线虫(例如,秀丽隐杆线虫)和微生物,包括细菌(例如,大肠杆菌(escherichia coli)、保加利亚乳杆菌(lactobacillus bulgaricus))。在一些情况下,细菌可以包括由个体作为补充剂(例如作为酸奶中的介质)和/或作为治疗剂(例如抑制或改善病状)消耗的菌株。在一些情况下,细菌可以包括在个体体内存在的菌株(例如,人微生物组)。
81.基因组的遗传物质可以是dna和/或rna。遗传物质可包括基因和基因间区域中的核酸序列。在一些情况下,遗传物质可以表示为染色体的单位。在一些情况下,遗传物质可以表示为已从基因转录的一个或多个转录物。基因及其相应的一个或多个转录物可包含一个或多个编码区域(即外显子)。在一些情况下,基因及其相应的一个或多个转录物可包含一个或多个基因内非编码区域(即内含子)。一个或多个基因内非编码区域可位于编码区域之间。在一些情况下,基因可以编码一个转录物。在一些情况下,基因编码多个转录物,每个转录物包含来自基因的外显子和内含子的不同变化。在一个示例中,rela基因编码转录因子p65,并且智人的rela基因编码至少18种不同长度的已知转录物:rela
‑
202、rela
‑
207、rela
‑
226、rela
‑
205、rela
‑
201、rela
‑
208、rela
‑
220、rela
‑
207、rela
‑
215、rela
‑
204、rela
‑
222、rela
‑
213、rela
‑
225、rela
‑
211、rela
‑
219、rela
‑
221和rela
‑
212。因此,多个转录物可以具有不同数目核苷酸碱基(多核苷酸长度)。替代地或附加地,可以将多个转录物翻译成具有不同数目氨基酸(多肽长度)的多肽(例如,蛋白质)。在一些情况下,多个转录物中的每一个可以相对于一个或多个其他转录物具有不同的表达水平(丰度)。
82.为了鉴定用于基因杂交的初始grna集,可以从基因的多个转录物中选择转录物。在一些情况下,选择的转录物可以比多个转录物中的一个或多个其他转录物具有更高的报告丰度。在一些实施方案中,从数据库确定基因的多个转录物的丰度。选择的转录物可以具有在多个转录物中的第一、第二、第三、第四或第五高的报告丰度。在一些情况下,选择的转录物可以具有与多个转录物中的一个或多个其他转录物相比的至少一个额外的核苷酸。选择的转录物可以具有在多个转录物中的第一、第二、第三、第四、第五多的核苷酸。在一些情况下,与从多个转录物中的一个或多个其他转录物翻译的一种或多种多肽相比,来自选择的转录物的翻译的多肽(例如,蛋白质)可以具有至少一个额外的氨基酸。来自选择的转录物的翻译的多肽可以具有在多个转录物中的第一、第二、第三、第四或第五多的氨基酸。在一些情况下,多个转录物的丰度可以是用于从基因的多个转录物中确定选择的转录物的第一标准。
83.为了鉴定与基因杂交的初始grna集,可以选择存在于选择的转录物中的基因中的编码区域。如果基因是dna,则选择的编码区域可以更靠近该基因的启动子(上游)而不是该
基因的终止子(下游)。如果基因是rna,则选择的编码区域可以更靠近该基因的5’端而不是该基因的3’端。在一些情况下,选择的编码区域可以是选择的转录物内的早期位置外显子。早期位置外显子可以是位于基因前半部分的外显子。早期位置外显子可以是该基因的第一、第二、第三、第四、第五或第六外显子。
84.在一些情况下,选择的转录物的选择的编码区域可以是比该基因的多个转录物中的一个或多个中存在的一个或多个其他外显子具有更高普遍性的外显子。在一些情况下,选择的转录物的选择的外显子可以包含(常见)于多个转录物中的约50%的其他转录物中。选择的转录物的选择的外显子可以包含于多个转录物中的至少约40个百分比(%)、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%或更多的其他转录物中。选择的转录物的选择的外显子可以包含于多个转录物中的最多约95%、90%、85%、80%、75%、70%、65%、60%、55%、50%、45%、40%或更少的其他转录物中。在一些情况下,选择的转录物的选择的外显子可以具有与选择的转录物中的其他外显子相比的至少一个额外的核苷酸。在一些情况下,选择的外显子可以具有至少40个、45个、50个、55个、60个、65个、70个、75个、80个、85个、90个、95个、100个、105个、110个、115个、120个或更多个核苷酸。在一些情况下,外显子的普遍性和核苷酸数均可以作为确定选择的转录物的选择的外显子的标准。
85.可以将初始grna集设计为与靶区域(在本文中也称为结合位点)杂交。靶区域可以在物种的基因组中的基因或基因的一部分中。在一些情况下,基因的一部分可以是基因的外显子。外显子可以是在基因的每个转录物中均存在的外显子。外显子可以是基因的多个转录物中根据上述标准得到的选择的转录物的选择的外显子。在一些情况下,初始grna集中的一个或多个grna可以是单指导rna(sgrna)。在一些情况下,sgrna可以是单多核苷酸链。sgrna可包含杂交多核苷酸序列和第二多核苷酸序列。
86.杂交多核苷酸序列可以与基因的一部分(例如,基因的多个转录物中选择的转录物的选择的外显子)杂交。sgrna的杂交多核苷酸序列可以在17个至23个核苷酸之间。sgrna的杂交多核苷酸序列可以是至少17个、18个、19个、20个、21个、22个、23个或更多个核苷酸。sgrna的杂交多核苷酸序列可以是最多23个、22个、21个、20个、19个、18个、17个或更少的核苷酸。在一个示例中,grna的杂交多核苷酸序列是20个核苷酸。杂交多核苷酸序列可以与靶区域互补或部分互补。与靶区域互补的杂交多核苷酸序列可包含与靶区域的序列具有100%互补性的序列。与靶区域部分互补的grna包含的序列可相对于与靶区域100%互补的序列具有至少1个、至少2个、至少3个、至少4个或至少5个错配。
87.单多核苷酸链sgrna的第二多核苷酸序列可以与cas酶相互作用(结合)。第二多核苷酸序列可以是约80个核苷酸。第二多核苷酸序列可以是80个核苷酸。第二多核苷酸序列可以是至少80个或更多个核苷酸。第二多核苷酸序列可以是至多80个或更少的核苷酸。
88.总体而言,单多核苷酸链sgrna的范围可介于97个至103个核苷酸之间。单多核苷酸链sgrna可以是至少97个、98个、99个、100个、101个、102个、103个或更多个核苷酸。单多核苷酸链sgrna可以是最多103个、102个、101个、100个、99个、98个、97个或更少的核苷酸。在一个示例中,单多核苷酸链sgrna可以是100个核苷酸。
89.在一些情况下,初始grna集中的一个或多个grna可以是crispr rna(crrna)区段和反式激活crrna(tracrrna)区段的复合物(例如,通过氢键)。crrna可以包含杂交多核苷酸序列和tracrrna结合多核苷酸序列。杂交多核苷酸序列可以与基因的一部分(例如,基因
的多个转录物中选择的转录物的选择的外显子)杂交。crrna的杂交多核苷酸序列的范围可以是17个至23个核苷酸。crrna的杂交多核苷酸序列可以是至少17个、18个、19个、20个、21个、22个、23个或更多个核苷酸。crrna的杂交多核苷酸序列可以是最多23个、22个、21个、20个、19个、18个、17个或更少的核苷酸。在一个示例中,crrna的杂交多核苷酸序列是20个核苷酸。crrna的tracrrna结合多核苷酸序列可以是22个核苷酸。crrna的tracrrna结合多核苷酸序列可以是至少22个或更多个核苷酸。crrna的tracrrna结合多核苷酸序列可以是最多22个或更少的核苷酸。总体而言,crrna的范围可以是39个至45个核苷酸。crrna可以是至少39个、40个、41个、42个、43个、44个、45个或更多个核苷酸。crrna可以是最多45个、44个、43个、42个、41个、40个、39个或更少的核苷酸。tracrrna的范围可以是60个至80个核苷酸。tracrrna可以是至少60个、61个、62个、63个、64个、66个、68个、70个、72个、74个、76个、78个、80个或更多个核苷酸。tracrrna可以是最多80个、79个、78个、77个、76个、74个、72个、70个、68个、66个、64个、62个、60个或更少的核苷酸。在一个示例中,tracrrna可以是72个核苷酸。在另一个示例中,crrna的杂交多核苷酸序列是20个核苷酸,crrna是42个核苷酸,并且相应的tracrrna是72个核苷酸。
90.在一些情况下,初始grna集可以包含一个或多个sgrna以及crrna和tracrrna的一个或多个复合物两者。替代地或附加地,在初始grna集中的一个或多个grna可以是三条或更多rna链的复合物。三条或更多rna链的复合物中的至少一条rna链可包含杂交多核苷酸序列。三条或更多rna链的复合物中的至少一条rna链可包含cas酶结合序列。
91.当grna与目标基因组区域的靶区域杂交时,目标基因组区域的杂交部分可以是包含原间隔子(靶位点)、被cas酶识别的原间隔子相邻基序(pam)和原间隔子的相反链(结合位点)的靶区域(或靶基因座)。原间隔子的相反链可以是grna杂交基因组区域(序列)。基因中的grna杂交序列范围可以是17个至23个核苷酸。基因中的grna杂交序列可以是至少17个、18个、19个、20个、21个、22个、23个或更多个核苷酸。基因中的grna杂交序列可以是最多23个、22个、21个、20个、19个、18个、17个或更少的核苷酸。
92.可以将初始grna集中的每个grna设计为结合其在目标基因组区域中的相应结合位点(例如,选择的外显子中的结合位点)。但是,在一些情况下,每个grna还可结合其他包含pam位点的cas靶区域,从而导致与脱靶杂交区域的不需要的脱靶结合。照此,对于初始grna集中的每个grna,可以确定脱靶值。脱靶值可以在物种的整个基因组上确定。在一些情况下,脱靶值可以在物种的整个参考基因组(例如,人类参考基因组、微生物组基因组等)上确定。物种的参考基因组可以是从对来自一批供体的dna(或rna)测序而装配成的基因集。参考基因组可包含来自一个或多个染色体的遗传物质。参考基因组可包含一个或多个重叠群(例如,未定位序列重叠群)。每个重叠群可以是代表dna连续区域的重叠的多核苷酸克隆集。在一个示例中,每个重叠群可以是连续的dna序列。脱靶值可以通过列举初始grna集中每个grna与基因组中多个靶位点相比的错配数目来确定。多个靶位点可以包括基因组上所有可能的cas核酸酶靶区域的原间隔子。
93.在一些情况下,多个靶位点中的每一个可以与pam位点相邻。在一些情况下,多个靶位点的每一个可以与用于核酸酶的pam位点相邻,该核酸酶选自:cas9、c2c1、c2c3、cpf1、cas13b和cas13c。在一个示例中,cas核酸酶是来自酿脓链球菌(streptococcus pyogenes)的cas9(spcas9),并且多个靶位点包括与spcas9的pam位点(ngg,其中“n”是任何核苷酸)相
邻的所有核苷酸序列。在另一个示例中,cas核酸酶是来自脑膜炎奈瑟菌(neisseria meningitidis)的cas9(nmcas9),并且多个靶位点包括与nmcas9的pam位点(gatt)相邻的所有核苷酸序列。为了定向至此类靶位点,可以将一种或多种核酸酶(例如,cas9、c2c1、c2c3、cpf1、cas13b、cas13c等)偶联至至少一个grna。可以将至少一个grna设计为与至少一个是靶位点(原间隔子)的相反链的结合位点杂交。
94.在一个示例中,针对细菌报告的多个靶位点可以包括至少100个、1,000个、10,000个、20,000个、30,000个、40,000个、50,000个、60,000个、70,000个、80,000个、90,000个、100,000个或更多个靶位点。在另一个示例中,针对人类报告的多个靶位点可以包括至少1000个、10,000个、100,000个、1,000,000个、10,000,000个、20,000,000个、30,000,000个、40,000,000个、50,000,000个、60,000,000个、70,000,000个、80,000,000个、90,000,000个、100,000,000个、200,000,000个、300,000,000个或更多个靶位点。在另一个示例中,针对植物报告的多个靶位点可以包括至少10,000个、100,000个、1,000,000个、10,000,000个、100,000,000个、200,000,000个、300,000,000个、400,000,000个、500,000,000个、600,000,000个、700,000,000个、800,000,000个、900,000,000个、1,000,000,000个、1,100,000,000个、1,200,000,000个、1,300,000,000个、1,400,000,000个、1,500,000,000个或更多个靶位点。
95.在一些情况下,列举每个grna与多个靶位点相比的错配数目可包括确定具有0个、1个、2个、3个、4个、5个或更多个错配数目的脱靶杂交区域。可以在grna所设计针对的整个基因组或此基因组的一部分中确定该区域。基因组可以是参考基因组。在一些情况下,基因组的一部分可以是至少1条、2条、3条、4条、5条、6条、7条、8条、9条、10条或更多条染色体。可以对可与靶标杂交的每个grna(例如,包含20个碱基的杂交多核苷酸序列的grna)独立地计算错配数目。列举的错配数目可以为0。列举的错配数目可以为1。列举的错配数目可以为2。列举的错配数目可以为3。列举的错配数目可以为4。在一些情况下,确定(例如,计算)脱靶值包括列举初始grna集中每一个的错配数目的累计总和。在一些情况下,列举可包括分别列举与靶位点(潜在靶位点)的0个、1个、2个、3个、4个或5个错配中的至少两个。在一个示例中,对于设计的grna,可以存在1个具有0个错配的脱靶杂交区域(例如,grna的每个核苷酸与脱靶杂交区域的相应的核苷酸全部配对)、3个具有1个错配的脱靶杂交区域、5个具有2个错配的脱靶杂交区域、7个具有3个错配的脱靶杂交区域和9个具有4个错配的脱靶杂交区域。因此,设计的grna的所得脱靶值可以表示为[1,3,5,7,9]。在另一个示例中,对于另一个设计的grna,可以存在0个具有0个错配的脱靶杂交区域(例如,grna的每个核苷酸与脱靶杂交区域的相应的核苷酸全部配对)、0个具有1个错配的脱靶杂交区域、15个具有2个错配的脱靶杂交区域、50个具有3个错配的脱靶杂交区域和90个具有4个错配的脱靶杂交区域。因此,设计的grna的所得脱靶值可以表示为[0,0,15,50,90]。
[0096]
脱靶值可以用作从初始grna集中选择grna子集的标准。在一些情况下,错配数目之一可以用作从初始grna集生成grna子集的阈值。在一个示例中,grna子集不能具有任何具有0个错配的脱靶杂交区域。在此种情况下,每个grna子集可以具有[0,#,#,#,#]的脱靶值,其中“#”表示大于等于0的任何整数。在另一个示例中,grna子集不能具有任何具有0个和1个错配的脱靶杂交区域。在此种情况下,每个grna子集可以具有[0,0,#,#,#]的脱靶值,其中“#”表示大于等于0的任何整数。不希望受到理论的束缚,提高阈值可以产生体外或体
内脱靶效应的可能性更低的grna子集。
[0097]
可以确定初始grna集中每个grna的中靶效率值。可以通过计算方位得分来确定grna的脱靶效率值。方位得分可以基于doench“规则集2”评分模型。规则集2评分模型可以使用一种或多种机器学习算法来计算每个grna对其相应靶区域的中靶活性。一种或多种机器学习算法使用的参数的示例包括:单核苷酸的位置;双核苷酸的位置;单核苷酸和双核苷酸的频率;grna中g和c碱基的数目;grna在基因中的位置;以及grna的前5个、中8个和后5个核苷酸的解链温度。计算后,最终的中靶活性(方位得分)的范围可以为从0到1。
[0098]
在一些情况下,中靶效率值(方位得分)可以作为从初始grna集中选择grna子集的标准。来自初始grna集的grna子集可以具有至少约0.2的中靶效率值。来自初始grna集的grna子集可以具有至少约0.2、0.3、0.4、0.5、0.6、0.7、0.8、0.9或更大的中靶效率值。在一个示例中,来自初始grna集的grna子集可以具有大于0.4的中靶效率值。
[0099]
在一些情况下,grna的中靶效率和脱靶值均可作为从初始grna集中选择grna子集的标准。在一个示例中,鉴定grna子集可以基于中靶效率的阈值(例如,方位得分大于0.4)和脱靶值的阈值(例如,没有具有0或1个错配的脱靶杂交位点)。基于这两个标准,可以对初始grna集中的每个grna进行排序。来自初始grna集的grna子集可以包含1个至10个排序最高的grna。来自初始grna集的grna子集可以包含至少1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个排序最高的grna。来自初始grna集的grna子集可以包含最多10、9、8、7、6、5、4、3、2或更少的排序最高的grna。
[0100]
初始grna集各自被设计为与物种基因组基因的一部分杂交,可用于敲除(ko)细胞中的基因。ko可以是靶ko。替代地或附加地,初始grna集各自被设计为与物种基因组的基因的一部分杂交,可用于将突变敲入(ki)细胞中的基因中。ki可以是靶插入。靶插入可以是供体多核苷酸的插入。在一些情况下,可以通过至少一个特异性grna将至少一种crispr/cas复合物引导至靶区域并切割该靶区域。在一些示例中,切割可以导致插入和/或缺失(“indel”)突变或通过非同源末端连接(nhej)过程的移码,从而导致靶基因特异性ko。在一些情况下,可以通过特异性grna与共同施用的供体多核苷酸(单链或双链)一起将crispr/cas复合物引导至靶基因组区域。切割靶区域后,同源性介导修复(hdr)过程可以使用一种或多种供体多核苷酸作为一种或多种模板,用于(a)修复切割的靶核苷酸序列和(b)将遗传信息从供体多核苷酸转移到靶dna。根据遗传信息的性质,hdr过程可以产生靶基因特异性的ko或ki。hdr介导的基因ki的应用示例包括向基因添加(插入或替换)编码蛋白质的核酸物质、mrna、小干扰rna(sirna)、标签(例如,6xhis)、报告蛋白(例如,绿色荧光蛋白)和调控序列(例如,启动子、聚腺苷酸化信号)。
[0101]
对于hdr过程,供体多核苷酸可包含要复制的所需基因编辑(序列),以及与紧接切割靶位点的上游和下游同源的在两端的额外核苷酸序列(同源臂)。在一些情况下,hdr过程的效率可取决于基因编辑的大小和/或同源臂的大小。替代地或附加地,hdr过程的效率可取决于包含pam位点的cas靶区域的可用性。因此,包括确定(a)来自多个基因和/或基因组数据库的初始rna序列集、(b)中靶效率和/或(c)脱靶值的方法可以用于设计用于hdr的供体多核苷酸集。
[0102]
根据本公开内容的crispr/cas系统可以用于多种细胞中。细胞可以是任何原核或真核活细胞、源自这些生物体以用于体外培养的细胞系、动物或植物来源的原代细胞。真核
细胞可以指真菌、植物、藻类或动物细胞或源自以下所列生物体并用于体外培养而建立的细胞系。真菌的属可以是曲霉属(aspergillus)、青霉属(penicillium)、枝顶孢霉属(acremonium)、木霉属(trichoderma)、金孢霉属(chrysoporium)、被孢霉属(mortierella)、克鲁维酵母属(kluyveromyces)或毕赤酵母属(pichia);更优选地,真菌的种是黑曲霉(aspergillus niger)、构巢曲霉(aspergillus nidulans)、米曲霉(aspergillus oryzae)、土曲霉(aspergillus terreus)、产黄青霉(penicillium chrysogenum)、橘青霉(penicillium citrinum)、产黄头孢霉(acremonium chrysogenum)、里氏木霉(trichoderma reesei)、高山被孢霉(mortierella alpine)、拉克悼金孢霉(chrysosporium lucknowense)、乳酸克鲁维酵母(kluyveromyceslactis)、巴斯德毕赤酵母(pichia pastoris)或西弗毕赤酵母(pichia ciferrii)。植物的属可以是拟南芥属(arabidospis)、烟草属(nicotiana)、茄属(solanum)、莴苣属(lactuca)、芥属(brassica)、稻属(oryza)、天门冬属(asparagus)、豌豆属(pisum)、苜蓿属(medicago)、玉蜀黍属(zea)、大麦属(hordeum)、黑麦属(secale)、小麦属(triticum)、辣椒属(capsicum)、黄瓜属(cucumis)、南瓜属(cucurbita)、西瓜属(citrullis)、柑橘属(citrus)、高粱属(sorghum)。植物的种可以是拟南芥(arabidospis thaliana)、普通烟草(nicotiana tabaccum)、番茄(solanum lycopersicum)、马铃薯(solanum tuberosum)、金银茄(solanum melongena)、番茄(solanum esculentum)、莴苣(lactuca saliva)、欧洲油菜(brassica napus)、甘蓝(brassica oleracea)、芜菁(brassica rapa)、光稃稻(oryza glaberrima)、水稻(oryza sativa)、芦笋(asparagus officinalis)、豌豆(pisumsativum)、紫花苜蓿(medicago sativa)、玉米(zea mays)、大麦(hordeum vulgare)、黑麦(secale cereal)、普通小麦(triticuma estivum)、硬粒小麦(triticum durum)、辣椒(capsicum sativus)、西葫芦(cucurbitapepo)、西瓜(citrullus lanatus)、甜瓜(cucumis melo)、来檬(citrus aurantifolia)、柚子(citrus maxima)、枸橼(citrus medica)和柑橘(citrus reticulata)。动物细胞的属可以是人属(homo)、大鼠属(rattus)、小鼠属(mus)、仓鼠属(cricetulus)、灵长属(pan)、猪属(sus)、牛属(bos)、丹鱼属(danio)、犬属(canis)、猫属(felis)、马属(equus)、鲑属(salmo)、大麻哈鱼属(oncorhynchus)、原鸡属(gallus)、吐绶鸡属(meleagris)、果蝇属(drosophila)、隐杆线虫属(caenorhabditis)。动物细胞的种可以是智人、褐家鼠、小家鼠、黑线仓鼠、倭黑猩猩、欧亚野猪(sus scrofa)、家牛(bos taurus)、灰狼(canis lupus)、黑线仓鼠、斑马鱼、家猫(felis catus)、家马(equus caballus)、褐家鼠、大西洋鲑(salmo salar)、虹鳟(oncorhynchus mykiss)、原鸡(gallus gallus)、火鸡(meleagris gallopavo)、黑腹果蝇和秀丽隐杆线虫。
[0103]
示例细胞系可以选自cho细胞(例如,cho
‑
k1);hek293细胞;caco2细胞;u2
‑
os细胞;nih 3t3细胞;nso细胞;sp2细胞;dg44细胞;k
‑
562细胞,u
‑
937细胞;mc5细胞;imr90细胞;jurkat细胞;hepg2细胞;hela细胞;ht
‑
1080细胞;hct
‑
116细胞;hu
‑
h7细胞;huvec细胞和molt 4细胞。适用于本公开内容范围的其他细胞的示例可以包括干细胞、胚胎干细胞(esc)和诱导多能干细胞(ipsc)。可以通过本发明的方法修饰所有这些细胞系和/或细胞,以提供细胞系模型用于产生、表达、定量、检测和/或研究目标基因或蛋白质;和/或筛选在研究和生产以及诸如作为非限制性示例的化学、生物燃料、治疗和农学的各个领域中的目标生物活性分子。
[0104]
在一些情况下,可以修饰来自初始指导rna集中至少一个指导rna的至少一个核苷酸。至少一个核苷酸的修饰的示例可以包括:(a)末端修饰,包括5’末端修饰或3’末端修饰;(b)核碱基(或“碱基”)的修饰,包括碱基的替换或去除;(c)糖修饰,包括在2’、3’和/或4’位置的修饰;以及(d)骨架修饰,包括磷酸二酯键的修饰或替换。不希望受到理论的束缚,至少一个核苷酸的修饰可以提供例如:(a)改善的靶特异性;(b)降低的crispr/cas复合物有效浓度;(c)改善的grna稳定性(例如,对核糖核酸酶(rna酶)和/或脱氧核糖核酸酶(dna酶)的抗性);以及(d)降低的免疫原性。在一个示例中,来自初始指导rna集中至少一个指导rna的至少一个核苷酸可以是2
’‑
o
‑
甲基核苷酸。这种修饰可以提高grna针对rna酶和/或dna酶攻击的稳定性。
[0105]
在一些情况下,并入指导rna中的核苷酸糖修饰选自2
’‑
o
‑
c1‑4烷基如2
’‑
o
‑
甲基(2
’‑
ome)、2
’‑
脱氧基(2
’‑
h)、2
’‑
o
‑
c1‑3烷基
‑
o
‑
c1‑3烷基如2
’‑
甲氧基乙基(2
’‑
moe)、2
’‑
氟(2
’‑
f)、2
’‑
氨基(2
’‑
nh2)、2
’‑
阿拉伯糖(2
’‑
阿糖)核苷酸、2
’‑
f
‑
阿拉伯糖(2
’‑
f
‑
阿糖)核苷酸、2’锁核酸(lna)核苷酸、2’解锁核酸(ulna)核苷酸、l型糖(“l
‑
糖”)和4
’‑
硫代核糖核苷酸。在一些情况下,并入指导rna中的核苷酸间键修饰选自:硫代磷酸酯“p(s)”(p(s))、膦基羧酸酯(p(ch2)
n
coor)如膦基乙酸酯“pace”(p(ch2coo
‑
))、硫代膦基羧酸酯((s)p(ch2)
n
coor)如硫代膦基乙酸酯“硫pace”((s)p(ch2)
n
coo
‑
)、烷基膦酸酯(p(c1‑3烷基)如甲基膦酸酯
‑
p(ch3)、硼酸膦酸酯(p(bh3))和二硫代磷酸酯(p(s)2)。
[0106]
在一些情况下,并入指导rna中的核碱基(“碱基”)修饰选自:2
‑
硫尿嘧啶(“2
‑
硫u”)、2
‑
硫胞嘧啶(“2
‑
硫c”)、4
‑
硫尿嘧啶(“4
‑
硫u”)、6
‑
硫鸟嘌呤(“6
‑
硫g”)、2
‑
氨基腺嘌呤(“2
‑
氨基a”)、2
‑
氨基嘌呤、假尿嘧啶、次黄嘌呤、7
‑
脱氮鸟嘌呤、7
‑
脱氮
‑8‑
氮杂鸟嘌呤、7
‑
脱氮腺嘌呤、7
‑
脱氮
‑8‑
氮杂腺嘌呤、5
‑
甲基胞嘧啶(“5
‑
甲基c”)、5
‑
甲基尿嘧啶(“5
‑
甲基u”)、5
‑
羟甲基胞嘧啶、5
‑
羟甲基尿嘧啶、5,6
‑
脱氢尿嘧啶、5
‑
丙炔基胞嘧啶、5
‑
丙炔基尿嘧啶、5
‑
乙炔基胞嘧啶、5
‑
乙炔基尿嘧啶、5
‑
烯丙基尿嘧啶(“5
‑
烯丙基u”)、5
‑
烯丙基胞嘧啶(“5
‑
烯丙基c”)、5
‑
氨基烯丙基尿嘧啶(“5
‑
氨基烯丙基u”)、5
‑
氨基烯丙基胞嘧啶(“5
‑
氨基烯丙基c”)、无碱基核苷酸、z碱基、p碱基、非结构化核酸(“una”)、异鸟嘌呤(“异g”)、异胞嘧啶(“异c”)和5
‑
甲基
‑2‑
嘧啶。
[0107]
在一些情况下,在核苷酸糖、核碱基、磷酸二酯键和/或核苷酸磷酸上引入一种或多种同位素修饰。此类修饰包括包含一个或多个
15
n、
13
c、
14
c、氘、3h、
32
p、
125
i、
131
i原子或用作示踪剂的其他原子或元素的核苷酸。
[0108]
在一些情况下,并入指导rna中的“末端”修饰选自:peg(聚乙二醇)、烃接头(包括:杂原子(o、s、n)取代的烃间隔基;卤素取代的烃间隔基;含酮基、羧基、酰胺基、亚硫酰基、氨基甲酰基、硫代氨基甲酰基的烃间隔基)、精胺接头、染料(包括连接至接头的荧光染料(例如荧光素、罗丹明、花菁),诸如6
‑
荧光素己基)、猝灭剂(例如dabcyl、bhq)和其他标记(例如生物素、洋地黄毒苷、吖啶、链霉亲和素、亲和素、肽和/或蛋白质)。在一些情况下,“末端”修饰包括指导rna与另一个分子的缀合(或连接),该另一个分子包括寡核苷酸(包含脱氧核苷酸和/或核糖核苷酸)、肽、蛋白质、糖、寡糖、类固醇、脂质、叶酸、维生素和/或其他分子。在一些情况下,并入指导rna中的“末端”修饰通过接头(诸如2
‑
(4
‑
丁氨基荧光素)丙烷
‑
1,3
‑
二醇双(磷酸二酯)接头)定位于指导rna序列内部,该接头作为磷酸二酯键并入并且可以并入指导rna中的任何两个核苷酸之间的位置。
[0109]
在一些情况下,可以使用计算机来执行用于设计用于与目标基因组区域杂交的一个或多个指导rna(grna)的方法。
[0110]
]图1显示了设计用于与物种的基因组基因杂交的一个或多个指导物(例如,grna)的方法的流程图100的示例。该方法包括:(a)从一个或多个数据库105获取目标基因的细节;(b)定位靶区域(例如,该基因的多个转录物中选择的转录物的外显子)110;(c)获取潜在的指导物(例如,具有杂交多核苷酸序列的一个或多个grna)115;(d)计算每个指导物的中靶值(例如,计算每个指导物的方位得分)120;(e)计算每个指导物的脱靶命中125;(f)获取关于可能的脱靶的更多细节(例如,列举每个grna与整个基因组上的多个可能的cas靶区域相比的错配数目)130;以及(g)输出指导物的排序列表135。在(g)中,可以通过中靶值和/或脱靶值对指导物进行排序。在一些情况下,步骤(d)和步骤(e
‑
f)的顺序可以互换。
[0111]
图2显示了物种的基因组基因的多个转录物的表格200的示例。该信息可以从一个或多个数据库获取。在该示例中,目标基因是智人210的rela 220。已知rela的多个转录物230。多个rela转录物可以具有不同数目的核苷酸碱基232。替代地或附加地,可以将多个转录物翻译成具有不同数目氨基酸234的多肽(例如,蛋白质)。此外,转录物可以比多个转录物中的一个或多个其他转录物具有更高的报告丰度(未示出)。基于一个或多个上述因素来选择初级转录物240。
[0112]
图3显示了一个或多个grna靶向的转录物的早期编码区域的示例。在人类基因rela的多个转录物中,分析初级转录物240以选择包含外显子310的早期编码区域。外显子310同样存在于多个转录物中的超过50%的其他转录物中。随后,可以鉴定出整个外显子310上所有可能的cas靶区域以设计用于靶向和杂交的一种或多种grna。
[0113]
图4a显示了来自转录物的多个外显子的相对表现度410和外显子长度420的图的示例。多个外显子(位置1至位置11)来自初级转录物240。选择的外显子310的相对表现度值超过0.9,表明它存在于同一基因的超过90%的其他转录物中。选择的外显子310的长度也超过100个核苷酸。
[0114]
图4b显示了grna 450、452、454和460的示例以及它们的脱靶和中靶活性分析。将每个grna的分析总结为推荐括号[a,b,c,d],其中a表示grna是否被设计为靶向基因的早期编码区域;b表示基因的预期靶位点在该基因的多个转录物中是否是共同的(即,在该基因的多个转录物中的超过50%的其他转录物中发现);c表示grna的中靶活性是否高于阈值(即,方位得分大于0.4);并且d表示grna的脱靶活性是否高于阈值(即,没有任何具有0个和1个错配的脱靶杂交区域)。对于要被选择使用的grna,从a到d的所有四个因素都必须为“真”(与“假”相对)。在图4b中,仅grna 460被视为四个因素都为真。计算机系统
[0115]
本公开内容的另一方面提供了一种计算机系统,该计算机系统用于执行上述方法,以设计一种或多种与目标基因组区域杂交的指导rna(grna)。本公开内容的另一方面提供了一种计算机系统,该计算机系统用于执行上述方法,以设计用于与多个目标基因组区域中的每一个杂交的一个或多个指导rna(grna)。目标基因组区域可以是物种的基因组基因。该计算机系统可以包括用于从基因的多个转录物中选择转录物的计算机可读介质。该计算机系统可以包括用于鉴定与选择的转录物的基因中的不同靶位点杂交的初始grna集的计算机可读介质。
[0116]
计算机的计算机可读介质可以接收(例如,通过用户设备上的用户界面从用户接收)目标基因和物种的输入。该计算机可读介质可以与多个数据库通信以获取包括该物种的基因组和/或该物种的参考基因组的信息。在一些情况下,计算机可读介质可以与包括基因和/或基因组数据库在内的多个数据库通信,该数据库包括来自dna(dna
‑
seq)和/或rna(rna
‑
seq)的测序数据。基于这样的信息,计算机可读介质可以从基因的多个转录物中选择转录物。该计算机可读介质可以鉴定与选择的转录物基因中的不同靶位点杂交的初始grna集。替代地或附加地,计算机可读介质可以被配置为执行与上述设计用于与物种的基因组基因杂交的一个或多个grna的方法有关的一种或多种任务(例如,计算一个或多个grna的脱靶值)。此外,计算机可读介质还可以包括用于自动激活由设计工具的用户选择的生物聚合物(例如,rna)合成仪的指令。
[0117]
图5示出了用于计算多个grna在整个基因组中的脱靶值的数据处理架构500。初始grna集(或者替代地,每个grna的相应靶位点序列集)510输入到计算平台(例如,无服务器计算平台)的“主”查询520中。同时,将基因组上所有可能的cas靶区域(例如,每个结构域包括原间隔子序列和pam位点)的数据库划分为较小的子集(或“分片”)505。为了获取脱靶值,主查询520调用附加的“从属”查询525,每个分片一个,并且将每个从属查询与每个分片进行比较530以确定错配和每个grna的总脱靶值。在脱靶搜索之后,来自从属查询
‑
分片比较的结果被收集到结果聚合器540中。在一个示例中,约3.2亿个cas靶区域的数据库可以被划分为161个分片,每个分片包括约200万个cas靶区域。照此,主查询将调用161个从属查询进行脱靶搜索。通过使用数据处理架构500并同时运行比较,可以减少分析的输出时间。多grna系统
[0118]
在一些实施方案中,本公开内容提供了一种用于鉴定靶向目标基因组区域的指导rna(grna)集的方法。grna集可以包含至少2个、至少3个、至少4个、至少5个、至少10个、至少20个、至少50个、至少100个或至少200个grna。grna集可以由2个grna集成。grna集可以由3个grna集成。grna集可以由4个grna集成。该方法可以包括在计算机中设计grna集。该集中的每个grna可以与目标基因组区域内的不同靶位点杂交(例如,基因、基因簇、外显子)。
[0119]
在靶向相同目标基因组区域的grna集中,每个grna的靶位点之间的距离在本文中也可以被称为指导物间间隔。指导物间间隔可以是从第一grna的目标基因组区域中的第一靶位点的3’端到grna集中的第二个grna的目标基因组区域中的第二靶位点的5’端的距离(以碱基对表示)。指导物间间隔可以不包括包含第一grna的目标基因组区域中的靶位点和第二grna的目标基因组区域中的靶位点的碱基对。可以根据参考基因组确定指导物间间隔。可以确定目标基因组区域中顺序的靶位点之间的指导物间间隔。在一个示例中,grna集中的grna的目标基因组区域中的靶位点与该grna集中至少一个其他grna的目标基因组区域中的靶位点之间的最小距离为至少30个碱基。在另一个示例中,grna集中的每个grna的目标基因组区域中的靶位点与该grna集中的每个其他grna的目标基因组区域中的靶位点之间的最小距离为至少30个碱基。在另一个示例中,grna集中的grna的目标基因组区域中的靶位点与该grna集中的至少一个其他grna的目标基因组区域中的靶位点之间的最大距离为至多150个碱基。在一些实施方案中,多个grna集中的至少50%、至少60%、至少70%、至少80%、至少90%或至少95%的grna集包含至少3个grna。
[0120]
编辑效率可以指示在目标基因组区域处包含编辑的基因型的细胞的比例。该细胞
可以是与至少一个grna集、核酸酶和任选的供体多核苷酸接触的细胞群体。编辑的基因型可以是任何非野生型基因型。相对于野生型基因型,编辑的基因型可以包括插入或缺失。编辑的基因型可能是修复靶位点上的crispr/cas复合物引起的双链断裂的结果。编辑的基因型可导致目标基因组区域的敲除。在一些实施方案中,在具有30个或更多碱基的grna集中每个grna的目标基因组区域中的靶区域之间具有最小距离的grna集产生大于50%、60%、70%或80%的编辑效率。在一些实施方案中,多个grna集中的至少50%、60%、70%、80%、90%或95%的grna集包含大于50%的平均编辑效率。在一些实施方案中,多个grna集中的至少50%、60%、70%、80%、90%或95%的grna集包含大于70%的平均编辑效率。可以通过测序确定编辑效率。测序可以是sanger测序。测序可以是高通量测序。
[0121]
该集中的每个grna可以与靶位点杂交,该靶位点与grna集中至少一个其他grna的目标基因组区域中的靶位点相距至少10个碱基。该集中的每个grna可以与目标基因组区域中的靶位点杂交,该靶位点与grna集中至少一个其他grna的目标基因组区域中的靶位点相距至少30个碱基。该集中的每个grna可与目标基因组区域中的靶位点杂交,该靶位点与该指导rna集中至少一个其他指导rna的目标基因组区域中的靶位点相距最多170个碱基。该集中的每个grna可以与目标基因组区域中的靶位点杂交,该靶位点与该指导rna集中至少一个其他指导rna的目标基因组区域中的靶位点相距最多1000个碱基。优选地,来自grna集的每个grna的目标基因组区域中的靶位点与该集中的任何其他grna的目标基因组区域中的靶位点相距约10
‑
170个、30
‑
170个、10
‑
150个、30
‑
150个、10
‑
100个、30
‑
100个或30
‑
1000个碱基。这种安排可以导致改善的ko特性和各种crispr酶之间的协同效应。在一些实施方案中,实现该目标基因组区域的敲除所使用的grna集中的每种grna的量小于单独使用每个grna来实现目标基因组区域的敲除所需的量。实现敲除目标基因组区域所需的grna集中每个grna的量可以是单独使用每个grna来实现敲除目标基因组区域所需的量的1/3。实现敲除目标基因组区域所需的grna集中每个grna的量可以是单独使用每个grna来实现敲除目标基因组区域所需的量的1/2。
[0122]
在某些实施方案中,本文进一步描述了用于鉴定多个指导rna(grna)集的方法,多个grna集中的每个grna集靶向多个目标基因组区域中的不同目标基因组区域。多个目标基因组区域可以包含2个、3个、4个、5个、6个、7个、8个、9个、10个或超过10个目标基因组区域。该方法可以包括在计算机中为多个目标基因组区域的每一个设计不同的grna集。grna集中的每个grna均可与目标基因组区域内的不同靶位点(例如,基因、基因簇、外显子)杂交。grna集中的每个grna可以与目标基因组区域中的靶位点杂交,该靶位点与grna集中的至少一个其他grna的目标基因组区域中的靶位点相距至少10个碱基。该集中的每个grna可以与目标基因组区域中的靶位点杂交,该靶位点与grna集中至少一个其他grna的目标基因组区域中的靶位点相距至少30个碱基。grna集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与该指导rna集中至少一个其他指导rna的目标基因组区域中的靶位点相距最多170个碱基。grna集中的每个grna可与目标基因组区域中的靶位点杂交,该靶位点与grna集中至少一个其他grna的目标基因组区域中的靶位点相距最多1000个碱基。优选地,来自grna集的每个grna的目标基因组区域中的靶位点与该集中的任何其他grna的目标基因组区域中的靶位点相距约10
‑
170个、30
‑
170个、10
‑
150个、30
‑
150个、10
‑
100个、30
‑
100个或30
‑
1000个碱基。这种安排可以导致改善的ko特性和各种crispr酶之间的协同效应。
[0123]
计算机可以是用于执行设计一种或多种与目标基因组区域杂交的grna的方法的前述计算机系统。目标基因组区域可以是物种的基因组基因。目标基因组区域可以是基因组的非编码区域。非编码区域可以是调控元件。调控元件可以是顺式调控元件或反式调控元件。顺式调控元件可以是启动子、增强子或沉默子。所鉴定的grna集可以是用于与物种的基因组基因杂交的一个或多个grna的子集。在一些情况下,grna集中的一个或多个grna可以是单指导rna(sgrna)。在一些情况下,grna集中的一个或多个grna可以是crispr rna(crrna)区段和反式激活crrna(tracrrna)区段的复合物(例如,通过氢键)。
[0124]
grna集中的每个grna可包含与目标基因组区域内的不同靶位点杂交的多核苷酸序列(杂交多核苷酸序列)。grna的杂交多核苷酸序列的范围可以是17个至23个核苷酸。grna的杂交多核苷酸序列可以是至少17个、18个、19个、20个、21个、22个、23个或更多个核苷酸。grna的杂交多核苷酸序列可以是至多23个、22个、21个、20个、19个、18个、17个或更少的核苷酸。在一个示例中,grna的杂交多核苷酸序列是20个核苷酸。
[0125]
该物种基因组的基因可以具有一个或多个转录物。在一个示例中,该基因可以被转录成一个或多个转录物。该一个或多个转录物可以包含一个或多个编码区域(即外显子)和/或一个或多个基因内非编码区域(即内含子)。在一些情况下,目标基因组区域可以包含基因的编码区域。在一些情况下,目标基因组区域可包含基因的非编码区域。在一些情况下,目标基因组区域可包含基因的编码区域和基因的非编码区域。如果基因是dna,则基因的编码区域可以更靠近该基因的启动子(上游)而不是该基因的终止子(下游)。如果基因是rna,则基因的编码区域可以更靠近该基因的5’端而不是该基因的3’端。在一些情况下,目标基因组区域可以包含基因的外显子。目标基因组区域可以是基因内的早期位置外显子。早期位置外显子可以是位于基因前半部分的外显子。早期位置外显子可以是该基因的第一、第二、第三、第四、第五或第六外显子。
[0126]
该物种的基因组基因可以是基因家族中的一个,并且由grna集靶向的目标基因组区域可以包括基因家族。在一个示例中,该基因是nf
‑
κb(rel)基因家族,其包括rela、relb、rel、nfkb1和nfkb2,并且目标基因组区域可以是包含五个基因的nf
‑
κb(rel)基因家族。在另一个示例中,过氧化物氧还蛋白基因家族包括prdx1、prdx2、prdx3、prdx4、prdx5和prdx6,并且目标基因组区域可以是包含六个基因的过氧化物氧还蛋白基因家族。
[0127]
该物种的基因组基因可以是假基因之一。假基因可以是加工的假基因、未加工的假基因、单一假基因和伪假基因。
[0128]
grna集所靶向的目标基因组区域可以包含来自基因家族的一个或多个编码区域。一个或多个编码区域的每个编码区域可以由0%至100%的基因家族表现(包含)。一个或多个编码区域中的每个编码区域可以由至少0%、10%、20%、30%、40%、50%、60%、70%、80%、90%、95%或更多的基因家族表现。一个或多个编码区域中的每个编码区域可以由最多100%、90%、80%、70%、60%、50%、40%、30%、20%、10%、5%或更少的基因家族表现。在一个示例中,目标基因组区域包括一个在基因家族的所有基因中表现的编码区域。
[0129]
在一些情况下,基因组区域是基因的连续多核苷酸区段。目标基因组区域的范围可以从1,000个碱基或核苷酸(1kb)到500kb。目标基因组区域可以是至少1kb、5kb、10kb、15kb、20kb、50kb、100kb、500kb或更大。目标基因组区域可以是最多500kb、100kb、50kb、20kb、15kb、10kb、5kb、1kb或更小。
[0130]
靶向目标基因组区域的鉴定的grna集可以包含2个至200个grna。靶向目标基因组区域的鉴定的grna集可以包含至少2个、3个、4个、5个、6个、7个、8个、9个、10个、15个、20个、30个、40个、50个、60个、70个、80个、90个、100个、200个或更多个grna。靶向目标基因组区域的鉴定的grna集可以包含至少2个grna。靶向目标基因组区域的鉴定的grna集可包含至少3个grna。靶向目标基因组区域的鉴定的grna集可以包含最多200个、100个、90个、80个、70个、60个、50个、40个、30个、20个、15个、10个、9个、8个、7个、6个、5个、4个、3个或更少的grna。靶向目标基因组区域的鉴定的grna集可包含最多4个grna。靶向目标基因组区域的鉴定的的grna集可包含最多3个grna。
[0131]
靶向目标基因组区域的grna集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的至少一个其他grna的目标基因组区域中的靶位点相距约10个至200个碱基(核苷酸)。靶向目标基因组区域的grna集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的至少一个其他grna的目标基因组区域中的靶位点相距约30个至1000个碱基(核苷酸)。靶向目标基因组区域的grna集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的至少一个其他grna的目标基因组区域中的靶位点相距至少30个碱基(核苷酸)。grna集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的至少一个其他grna的目标基因组区域中的靶位点相距至少10个、15个、20个、25个、30个、35个、40个、45个、50个、60个、70个、80个、90个、100个、120个、140个、160个、180个、200个或更多个碱基。grna集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的至少一个其他grna的目标基因组区域中的靶位点相距最多2000个、1500个、1000个、500个、200个、180个、160个、140个、120个、100个、90个、80个、70个、60个、50个、45个、40个、35个、30个、25个、20个、15个、10个或更少的碱基。
[0132]
可以设计grna集,以使grna集将crispr/cas复合物集引导至细胞中目标基因组区域内的不同靶位点。crispr/cas复合物可在靶位点产生核酸序列的断裂。断裂可以是双链断裂。断裂可以是单链断裂。在一个示例中,可以设计grna集以指导crispr/cas复合物集敲除(ko)细胞中目标基因组区域内的一个或多个不同靶位点。敲除可能是由于修复由crispr/cas复合物引起的断裂而引入的移码突变的结果。敲除可能是由于目标基因中外显子的删除而发生。敲除可能是由于目标基因组区域中至少1个、至少2个、至少3个、至少4个、至少5个、至少6个、至少7个、至少8个、至少9个、至少10个、至少100个、至少1000个或至少10,000个碱基对的删除而发生。敲除可以消除基因的功能。
[0133]
在另一个示例中,可以设计grna集来指导crispr/cas复合物集在细胞中目标基因组区域内不同靶位点敲入(ki)一个或多个突变。crispr/cas复合物集可以与供体多核苷酸集共同施用用于ki。敲入的一个或多个突变可将点突变、等位基因、标签或外源外显子引入基因组。点突变、等位基因、标签或外源性外显子可以位于供体多核苷酸上。如本文所述,可使用同源性介导修复(hdr)将供体多核苷酸掺入基因组。敲入的一个或多个突变可以恢复先前无功能的基因的功能。敲入的一个或多个突变可以改善基因的功能。基因功能的改善可以是基因产生的蛋白质的量的增加。敲入可以敲除基因的功能。
[0134]
在一些实施方案中,敲入的一个或多个突变可将标签引入基因组。标签可以是可检测标签。可检测标签可以是荧光标签。可检测标签可以是限制性片段长度多态性(rflp)。
[0135]
在一些实施方案中,敲入的一个或多个突变可将点突变引入基因组。点突变可以是目标基因组区域中核酸的插入、删除或置换。在一些实施方案中,敲入的一个或多个突变可将等位基因引入基因组。等位基因可以是转基因。
[0136]
在一些实施方案中,敲入的一个或多个突变可以将外源外显子引入基因组。外显子可以与靶基因的内源性外显子至少80%、85%、90%、95%或99%相同。内源性外显子可以是野生型外显子。外源性外显子可以是相对于靶基因的内源性外显子包含至少一个突变的外显子。在一些实施方案中,敲入的一个或多个突变可以用外源性外显子代替内源性外显子。相对于野生型外显子,外源性外显子可包含至少一种突变。外源外显子可以在供体多核苷酸中。
[0137]
在一些实施方案中,该方法还包括设计至少第二初始grna集,以产生多个初始grna集。该方法可以包括鉴定靶向多个基因的多个初始grna集,其中多个初始grna集中的每个初始grna集与多个基因中的基因中的不同靶位点杂交。多个grna的试剂盒
[0138]
本公开内容的另一方面提供了一种试剂盒,其包含通过上述方法产生的多个grna,该grna用于鉴定靶向目标基因组区域的指导rna(grna)集。该试剂盒可以包含grna集。该集中的每个grna均可与目标基因组区域内的不同靶位点杂交。该集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的至少一个其他grna的目标基因组区域中的靶位点相距至少10个碱基。该集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的至少一个其他grna的目标基因组区域中的靶位点相距至少30个碱基。该集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的至少一个其他grna的目标基因组区域中的靶位点相距最多170个碱基。该集中的每个grna均可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的每个其他grna的目标基因组区域中的靶位点相距30个至1000个碱基。在一些实施方案中,该grna集包含至少2个、至少3个、至少4个、至少5个、至少10个、至少15个、至少20个或至少30个grna。
[0139]
在一些实施方案中,试剂盒包含用于多个目标基因组区域中的每一个的一个grna集。本文所述的试剂盒可用于敲除目标基因组区域或多个目标基因组区域。本文所述的试剂盒可用于将供体多核苷酸引入目标基因组区域。本文所述的试剂盒可用于将多个供体多核苷酸引入多个目标基因组区域。
[0140]
在一些实施方案中,试剂盒包含至少一种供体多核苷酸。在一些实施方案中,试剂盒包含用于多个目标基因组区域的每个的至少一种供体多核苷酸。在一些实施方案中,试剂盒包含核酸酶。核酸酶可以是cas蛋白。cas蛋白可以是本文所述的任何cas蛋白,例如cas9、c2c1、c2c3或cpf1。在一些实施方案中,试剂盒包含试剂,例如缓冲液。缓冲液可以是tris缓冲液、tris
‑
edta(te)缓冲液、tris/硼酸盐/edta(tbe)缓冲液或tris
‑
醋酸盐
‑
edta(tae)缓冲液。该试剂盒可以包含无rna酶的h2o。在一些实施方案中,试剂盒包含转染试剂。转染剂的示例包括但不限于lipofectamine
tm
和oligofectamine
tm
。
[0141]
在一些实施方案中,试剂盒包括被分隔开以容纳一个或多个容器(如小瓶、管等)的载体、包装或容器,每个容器包含一个用于本文描述的方法分开的要素。合适的容器包括例如瓶、小瓶、注射器和试管。在一些实施方案中,容器由多种物质(如玻璃或塑料)形成。该
试剂盒可包含多孔板。多孔板可以是4孔板、6孔板、12孔板、24孔板、48孔板、96孔板或384孔板。在一些实施方案中,多孔板中的每个孔包含一个grna。在一些实施方案中,多孔板中的每个孔包含靶向单个目标基因组区域的一个grna集。在一些实施方案中,多孔板中的每个孔包含靶向多个目标基因组区域的多个grna。
[0142]
在一些实施方案中,试剂盒包含一个或多个另外的容器,每个容器具有一种或多种从商业和用户的角度来看对于本文中描述的用途有用的各种物质(如试剂,任选地以浓缩形式,和/或装置)。此类物质的非限制性示例包括但不限于缓冲液、引物、酶、稀释剂、过滤器、载体、包装、容器、小瓶和/或试管标签,其中列出了内容物和/或使用说明以及具有使用说明的包装插页。在一些情况下,包含一组说明。在一些情况下,标签在容器上或与容器相关联。当将组成标签的字母、数字或其他字符粘贴、模制或蚀刻到容器本身中时,标签可以位于容器上。当标签(例如,作为包装插页)存在于同样容纳该容器的贮器或载体中时,该标签可以与容器相关联。标签可用于指示内容物将用于特定的治疗应用。标签可以指示(如本文所述的方法中的)内容物的使用说明。
[0143]
本公开内容的另一方面提供了一种试剂盒,该试剂盒包含通过上述方法产生的单个grna,所述方法用于鉴定靶向目标基因组区域的指导rna(grna)。该grna可以与目标基因组区域内的靶位点杂交。
[0144]
本公开内容的另一方面提供了一种试剂盒,该试剂盒包含多个修饰的细胞,该修饰的细胞在目标基因组区域包含修饰。多个修饰的细胞可通过使多个细胞与grna集以及核酸酶和任选的供体多核苷酸接触而产生,该grna集由用于鉴定靶向目标基因组区域的指导rna(grna)集的上述方法鉴定。计算机系统算法
[0145]
本公开内容的另一方面提供一种计算机系统,该计算机系统包括用于执行上述方法的算法,该方法用于鉴定靶向目标基因组区域的指导rna(grna)集。该算法可以包括鉴定grna集的步骤。该算法可以包括为多个目标基因组区域的每一个鉴定grna集的步骤。该集中的每个grna均可与目标基因组区域内的不同靶位点杂交。该集中的每个grna可以与目标基因组区域中的靶位点杂交,该靶位点与grna集中至少一个其他grna的目标基因组区域中的靶位点相距至少10个碱基。该集中的每个grna可以与目标基因组区域中的靶位点杂交,该靶位点与grna集中至少一个其他grna的目标基因组区域中的靶位点相距至少30个碱基。该集中的每个grna可以与目标基因组区域中的靶位点杂交,该靶位点与grna集中所有其他grna的目标基因组区域中的靶位点相距至少30个碱基。该集中的每个grna可与目标基因组区域中的靶位点杂交,该靶位点与该指导rna集中至少一个其他指导rna的目标基因组区域中的靶位点相距最多170个碱基。该组中的每个grna可以与目标基因组区域中的靶位点杂交,该靶位点与该指导rna集中每个其他指导rna的目标基因组区域中的靶位点相距30个至1000个碱基之间。计算脱靶效率
[0146]
本公开内容的另一方面提供了一种用于选择至少一个指导rna(grna)以与目标基因组区域杂交的方法。目标基因组区域可以是物种的基因组基因。对于与该基因杂交的初始grna集中的多个grna中的每一个,该方法可以包括通过列举与基因组中的潜在grna杂交位点的错配数目来计算脱靶值。
[0147]
该方法可以利用包括计算机可读介质的前述计算机系统来执行用于设计一种或多种与目标基因组区域杂交的grna的方法。目标基因组区域可以是物种的基因组基因。计算机可读介质可以通过列举与基因组中潜在的grna杂交位点的错配数目来计算脱靶值。
[0148]
在一些情况下,计算机系统的计算机可读介质可以计算脱靶值并将错配数目组织在分片中。在计算初始grna集的脱靶值时,可以将包含物种中基因组和/或参考基因组中所有可能的核酸酶(例如,cas核酸酶)靶区域的数据库分区域(划分)为可能的cas核酸酶靶区域的多个“分片”(子集)。可以将初始grna集与每个分片进行比较,以列举0、1、2、3和/或4个的错配。相对于将初始grna集与包含所有可能的cas核酸酶靶区域的一个数据库进行比较,同时将初始grna集与包含可能的cas核酸酶靶区域子集的每个分片进行比较可以提高计算脱靶值的吞吐量、速度和整体性能。
[0149]
脱靶值可以在参考基因组的100,000个碱基对(bp)或核苷酸至3,000,000,000bp上或者在整个参考基因组上确定。脱靶值可以在参考基因组的至少100,000bp、500,000bp、1,000,000bp、5,000,000bp、10,000,000bp、50,000,000bp、100,000,000bp、500,000,000bp、1,000,000,000bp、2,000,000,000bp、3,000,000,000bp或更多上或者在整个参考基因组上确定。脱靶值可以在参考基因组的最多3,000,000,000bp、2,000,000,000bp、1,000,000,000bp、500,000,000bp、100,000,000bp、50,000,000bp、10,000,000bp、5,000,000bp、1,000,000bp、500,000bp、100,000bp或更少上或者在整个参考基因组上确定。在一个示例中,脱靶值可以在参考基因组的1,000,000bp上或者在整个参考基因组上确定。
[0150]
包含多个基因组和/或参考基因组的可能的核酸酶(例如,cas核酸酶)靶区域的数据库可以具有1,000至1,000,000个核酸酶结合位点。该数据库可以具有至少1,000个、10,000个、50,000个、100,000个、150,000个、200,000个、250,000个、300,000个、350,000个、400,000个、450,000个、500,000个、550,000个、600,000个、650,000个、700,000个、750,000个、800,000个、850,000个、900,000个、950,000个、1,000,000个或更多的核酸酶结合位点。该数据库可以具有最多1,000,000个、950,000个、900,000个、850,000个、800,000个、750,000个、700,000个、650,000个、600,000个、550,000个、500,000个、450,000个、400,000个、350,000个、300,000个、250,000个、200,000个、150,000个、100,000个、50,000个、10,000个、1,000个或更少的核酸酶结合位点。
[0151]
包含多个基因组和/或参考基因组的可能的核酸酶(例如,cas核酸酶)靶区域的数据库可以具有1000万个至3亿个核酸酶结合位点。数据库可以具有至少1000万个、2500万个、5000万个、7500万个、1亿个、1.25亿个、1.5亿个、1.75亿个、2亿个、2.25亿个、2.5亿个、2.75亿个、3亿个或更多个核酸酶结合位点。该数据库可以具有最多3亿个、2.75亿个、2.5亿个、2.25亿个、2亿个、1.75亿个、1.5亿个、1.25亿个、1亿个、7500万个、5000万个、2500万个、1000万个或更少的核酸酶靶位点。个性化治疗
[0152]
本公开内容的另一方面提供了一种用于设计用于与个体中的目标基因组区域杂交的一个或多个指导rna(grna)的方法。该方法可以包括使用个体的基因组来确定潜在的grna靶位点。该方法可以包括针对每个潜在的grna靶位点确定预期的指导rna的脱靶值。该方法可以包括鉴定具有改进的效用指数的一个或多个grna。
[0153]
在一些情况下,该方法可以包括使用个体群体的基因组来确定潜在的grna靶位
点。个体人群的示例包括在年龄范围内的个体集(例如,青少年、65岁或更大年龄等)、被诊断患有相同疾病的个体集(例如,患有肌营养不良症、帕金森氏症疾病等的患者群体)、接受相同疾病治疗的个体集(例如,被诊断和/或治疗过乳腺癌、前列腺癌等的受试者队列)等。此类方法可以为每个个体、来自个体群体的个体子集和/或整个个体群体鉴定潜在的grna靶位点。
[0154]
在一些情况下,使用本文描述的软件和方法进行grna的选择和/或推荐,该grna可用于整个患者队列和/或特定患者人群中的crispr系统。例如,可以将包含具有激活的或失活的核酸内切酶的crispr系统的治疗剂施用于已经使用本文的方法和系统选择的受试者。如果确定grna产生的脱靶结合的数目超过阈值,则将导致无法选择患者进行治疗,或推荐其他治疗方法。如果确定grna产生的脱靶结合的数目少于阈值,将导致选择患者进行治疗或向患者推荐这种治疗。
[0155]
在一些情况下,本文的任何方法和系统都用于鉴定存在于群体中的一个、一些或所有受试者中或能够结合群体中的一个、一些或所有受试者中的靶位点的grna,其优选地具有降低的脱靶值。这也将涵盖选择的grna针对群体中所有受试者计算脱靶值。
[0156]
可以使用来源于如本文所述的临床研究的信息或使用来自多个个体(例如,至少10个、100个、1,000个、10,000个或100,000个)的基因组信息来评估由参考装配体设计的grna。例如,临床研究可涉及对患有待治疗病症和/或正常的受试者集的基因组进行测序,并确定测试grna在上述个体的整个基因组上的脱靶值。在一些情况下,可以评估从单个参考基因组(例如,一个个体)设计的grna在群体中的一个、一些或所有受试者中的脱靶活性。该评估可以用于例如使用参考基因组设计新的治疗剂,并使用本文所述的方法和系统在受试者、受试者集或整个个体群体或人群中评估其可能的效率或功效。可以对grna进行进一步修饰,以提高稳定性、pk、递送,降低脱靶值或降低脱靶影响。
[0157]
在一个示例中,临床研究可涉及对患有待治疗病症和/或正常的受试者集中至少一个受试者的基因组进行测序,根据至少一个受试者的基因组设计grna,并确定设计的grna对受试者集中一个、一些或所有受试者的脱靶值。
[0158]
用于设计用于与个体的基因组区域杂交的一个或多个grna的方法可以利用上述用于设计一种或多种用于与目标基因组区域杂交的grna的方法。目标基因组区域可以是物种的基因组基因。该方法可以还包括接收个体的基因组和/或个体群体的基因组(例如,来自通过用户设备上的用户界面的用户输入,或者来自数据库)。该方法可以还包括从基因组中鉴定所有可能的靶区域(或靶基因座),该靶区域包括原间隔子(靶位点)、被一种或多种类型的cas酶识别的原间隔子相邻基序(pam)以及原间隔子的相反链(结合位点)。该方法可以还包括在目标基因组区域中分离所鉴定的靶区域。分离的靶区域可以是潜在靶位点。在一个示例中,目标基因组区域可以是个体或个体群体的免疫细胞的t细胞基因组内的特定位点,以降低基因在不正确或不希望的位置插入的风险。
[0159]
该方法可以还包括鉴定与个体或个体群体的潜在靶位点杂交的初始grna集。该方法可以还包括计算初始grna集中每个grna的脱靶值和/或中靶效率,以确定每个grna的效用指数。在一些情况下,效用指数可以是治疗指数。在一些情况下,治疗指数包括可以通过脱靶值和/或脱靶效率评估的脱靶结合的减少。因此,在一些情况下,脱靶值和开放靶效率的不同阈值可用于鉴定效用指数提高的一个或多个grna。因此,该方法可以还包括用具有
提高的效用指数的一个或多个grna编辑细胞。
[0160]
在一些情况下,治疗指数不仅包括脱靶结合的减少,还包括在个体或个体群体的至少一个细胞中的中靶效率的提高、敲除(ko)效率的提高、敲入(ki)效率的提高或crispr干扰的调节。在一个示例中,可以设计一个或多个grna以在个体或个体群体的细胞的基因组区域中ko基因。在另一个示例中,可以将一个或多个grna设计为在个体或个体群体的细胞的基因组区域中ki突变。
[0161]
在一些情况下,个体可以是人类。在一些情况下,个体可以是非人类(例如,小鼠、大鼠等)。在一些情况下,个体(或个体群体中的个体)可能患有病症。病症可以是已知或预测与多种疾病相关基因有关,并且本公开内容的一个或多个grna可以将一种或多种crispr/cas系统引导至多种疾病相关的基因。多种疾病相关的基因可以包括与非疾病对照的组织或细胞相比,在源自受疾病影响的组织的细胞中以异常水平或异常形式产生转录或翻译产物的任何基因或多核苷酸。一个示例可以是以异常高水平表达的基因。另一个示例可以是以异常低水平表达的基因,其中改变的表达与疾病的发生和/或发展相关。替代地或附加地,多种疾病相关的基因可以包括任何具有突变的基因。
[0162]
多种疾病相关的基因的示例包括阿尔茨海默病、帕金森病、多发性硬化症、脊髓性肌营养不良症、肌营养不良症、影响髓样细胞的疾病、慢性淋巴细胞性白血病、多发性骨髓瘤、恶性肿瘤、黑素瘤、囊性纤维化、血友病、镰状细胞病以及各种器官的癌症,包括乳腺癌、肠道癌、前列腺癌、中枢神经系统癌、胶质母细胞瘤和肉瘤。
[0163]
确定潜在的grna靶位点和鉴定一个或多个grna的方法可以通过计算机进行。该计算机可以是前述的计算机系统,其包括用于执行用于设计一种或多种与目标基因组区域杂交的grna的方法的可计算介质。个性化诊断
[0164]
本公开内容的另一方面提供了一种用于评估crispr试剂对个体的脱靶效应的方法。该方法包括使用个体的基因组,通过计算机列举与个体基因组中潜在靶位点的错配数目,从而确定crispr试剂的脱靶值。
[0165]
这在例如临床试验环境中可能有用,以选择要包括在临床试验或治疗中或从中排除的患者。例如,个人基因组的脱靶结合位点数目多于阈值(例如0个、1个、2个、3个等)的患者被排除在临床试验或治疗方案之外,而个人基因组具有较少或没有脱靶结合位点的患者被纳入临床试验或接受治疗。
[0166]
用于评估crispr试剂对个体的脱靶效应的方法可以利用上述计算机系统,该计算机系统包括计算机可读介质,用于执行用于设计一种或多种与目标基因组区域杂交的grna的方法。目标基因组区域可以是物种的基因组基因。该方法可以还包括输出报告,该报告列举与个体基因组中潜在靶位点的错配数目。在一些情况下,输出可以示出在屏幕上(例如,经由诸如个人计算机之类的用户设备上的用户界面)。
[0167]
crispr试剂可以是治疗剂。治疗剂可以是来自一个或多个用于与物种的基因组基因杂交的grna的grna。grna可以将crispr/cas复合物引导至靶区域。治疗剂可具有多种用途,包括修饰(例如,删除、插入、转移、灭活、激活)多种细胞类型中的靶区域。这样,治疗剂可具有广泛的应用,包括但不限于基因治疗、药物筛选、疾病诊断和预后。
[0168]
个体基因组中潜在靶位点的数目范围可以为1,000个至3,000,000个。个体基因组
中潜在靶位点的数目可以为至少1,000个、10,000个、100,000个、200,000个、300,000个、400,000个、500,000个、600,000个、700,000个、800,000个、900,000个、1,000,000个、2,000,000个、3,000,000个或更多。个体基因组中潜在靶位点的数目可以为最多3,000,000个、2,000,000个、1,000,000个、900,000个、800,000个、700,000个、600,000个、500,000个、400,000个、300,000个、200,000个、100,000个、10,000个、1,000个或更少。高效和精确的编辑方法
[0169]
本公开内容的一个方面提供了用于编辑细胞或细胞群体的方法,该方法包括:使细胞或细胞群体与一个或多个grna集以及核酸酶和任选的供体多核苷酸接触,以产生修饰的细胞群体。修饰的细胞群体可以在至少一个目标基因组区域中包含至少一个编辑。可以通过本文描述的任何方法设计一个或多个grna集。至少一个编辑可以导致目标基因组区域中的基因敲除或在基因组的目标基因组区域处敲入点突变、等位基因、标签或外源外显子。本公开内容的另一方面提供了一种用于筛选细胞或修饰的细胞群体的方法,该细胞或修饰的细胞群体包含在至少一个目标基因组区域中的由至少一个grna集产生的至少一个编辑。包含至少两个grna的一个或多个grna集的编辑效率可以高于至少两个grna中的每一个的单独编辑效率。
[0170]
在一些实施方案中,用于编辑细胞群体中的多个目标基因组区域的方法包括:使细胞群体与以下各项接触:(i)靶向多个目标基因组区域的多个grna集和(ii)核酸酶;其中在接触后,细胞群体中至少50%的细胞在每个目标基因组区域包含与野生型基因型不同的经编辑的基因型。在一些实施方案中,用于编辑细胞群体中多个目标基因组区域的方法包括使该细胞群体的子集的每一个与以下各项接触:(i)来自多个grna集的grna集,其中多个grna集中的每个grna集靶向来自多个目标基因组区域的不同目标基因组区域,和(ii)核酸酶。在一些实施方案中,在接触后,细胞群体的至少50%的子集中的至少80%的细胞在目标基因组区域包含不同于野生型基因型的经编辑的基因型。在一些实施方案中,在接触后,细胞群体的每个子集中的至少70%的细胞在目标基因组区域包含不同于野生型基因型的经编辑的基因型。每个grna集可以包含三个grna。在一些情况下,至少50%、60%、60%、70%、80%、90%或95%的grna集包含至少三个grna。grna集中的每个grna可以包含至少30个碱基的指导物间间隔。grna集中的每个grna可与目标基因组区域中的靶位点杂交,该靶位点与来自该grna集的所有其他grna的靶位点相距至少30个碱基。
[0171]
该方法可以包括设计一个或多个用于与目标基因组区域杂交的指导rna(grna)集,如本文所述。修饰的细胞群体可以包含至少一个细胞。至少一个细胞可以是哺乳动物细胞、鱼细胞、昆虫细胞、植物细胞或微生物。微生物可以是细菌。至少一个细胞可以是如本文所述的细胞系中的细胞。至少一个细胞可以是肿瘤细胞。至少一个细胞可以衍生自个体。
[0172]
该方法可以包括使细胞或细胞群体与一个或多个grna集和核酸酶接触,以产生修饰的细胞或修饰的细胞群体。该方法可以还包括使细胞或细胞群体与供体多核苷酸接触。接触可以包括将一个或多个grna集、核酸酶或编码核酸酶的多核苷酸或其组合转染到细胞或细胞群体中。在一些实施方案中,在转染之前,将一个或多个grna集中的每个grna与cas蛋白复合以产生cas
‑
grna复合物,在本文中也称为crispr/cas复合物或crispr/cas系统。在一些实施方案中,该方法还包括将至少一种供体多核苷酸转染到细胞或细胞群体中。转染可以是非病毒转染或病毒转染。非病毒转染可以是电穿孔、脂质转染或显微注射。病毒转
染可包括使用病毒载体。病毒载体可以是逆转录病毒载体、腺病毒载体、腺相关病毒(aav)载体、α病毒载体、牛痘病毒载体、单纯疱疹病毒(hsv)载体、慢病毒载体或逆转录病毒载体。病毒载体可以是具有复制能力的病毒载体或没有复制能力的病毒载体。
[0173]
目标基因组区域可以是基因。该基因可以是目标途径中的基因。该方法可以包括靶向多个目标基因组区域。多个目标基因组区域可以包含在目标途径中的多个基因。多个目标基因组区域可以包含在多个目标途径中的多个基因。目标途径可以是代谢途径、信号转导途径或基因调节途径。目标途径可以是与疾病有关的途径。该疾病可能是癌症。目标途径可以是涉及目标分子产生的途径。目标分子可以是具有药理活性的分子。目标基因组区域可以是基因组的非编码区域。非编码区域可以是调控元件。调控元件可以是顺式调控元件或反式调控元件。顺式调控元件可以是启动子、增强子或沉默子。
[0174]
在一些实施方案中,该方法包括使靶向目标基因组区域的grna集与修饰的细胞群体的子集接触。在一些实施方案中,该方法包括使靶向目标基因组区域的多个grna集的每一个与修饰的细胞群体的多个子集的每一个接触。在一个示例中,可以将修饰的细胞群体的多个子集放置在多孔板的每个孔中。多孔板可以是4孔板、6孔板、12孔板、24孔板、48孔板、96孔板或384孔板。修饰的细胞群体的子集可以包含至少102个、至少103个、至少104个、至少105或至少106个细胞。多孔板的每个孔可进一步包含靶向目标基因组区域的grna集。多孔板的每个孔中的每个grna集均可靶向不同的目标基因组区域。多个grna集可以靶向至少5个、至少10个、至少20个、至少50个或至少100个不同的目标基因组区域。在一些实施方案中,接触发生在多孔板的每个孔中。
[0175]
在一些实施方案中,该方法包括使修饰的细胞群体或修饰的细胞群体的子集与刺激物接触。刺激物可以是附加试剂。附加剂可以是治疗剂(例如:抗生素、生物或小分子药物)或在修饰的细胞中诱导疾病状态的试剂。
[0176]
在一些实施方案中,该方法包括检测修饰的细胞群体或修饰的细胞群体的子集的表型。该表型可以是细胞活力。该表型可以是grna集的编辑效率。该表型可以是修饰的细胞群体或修饰的细胞群体的子集产生的目标分子的量。目标分子可以是蛋白质或编码蛋白质的转录物。在一些实施方案中,该方法包括检测修饰的细胞群体或修饰的细胞群体的子集的标签。验证grna
[0177]
本公开内容的另一方面提供了用于验证预期的grna的方法。该方法可以包括确定在基因组或基因组的一部分中预期grna的脱靶命中数。该方法可以包括使用脱靶命中数计算预期grna的脱靶值。该方法可以包括使用脱靶值来预测预期的grna的活性。
[0178]
用于验证预期的grna的方法可以利用上述方法来设计一个或多个与目标基因组区域杂交的grna。目标基因组区域可以是物种的基因组基因。替代地或附加地,用于验证预期的grna的方法可以进一步利用上述计算机系统,该计算机系统包括计算机可读介质以用于执行(1)设计一个或多个用于与目标基因组区域杂交的grna的方法,(2)用于鉴定靶向目标基因组区域的grna集的方法,以及(3)选择至少一个用于与目标基因组区域杂交的grna的方法。
[0179]
图6显示了验证用于与物种的基因组基因杂交的一个或多个指导物(例如,grna)的方法的流程图600的示例。该方法包括:(a)确认该指导物存在(例如,确认物种的基因组
中存在用户提供的grna的互补序列)605;(b)查看切割信息(例如,确认基因组中grna的互补序列与原间隔子相邻基序(pam)位点相邻)610;(c)计算grna的中靶值(例如,计算grna的方位得分)615;(d)计算每个指导物620的脱靶命中;(e)获取关于可能的脱靶的更多细节(例如,列举每个grna与整个基因组中多个可能的cas靶区域相比的错配数目)625;和(f)输出grna活性的预测630。在一些情况下,步骤(c)和步骤(d
‑
e)的顺序可以互换。用户界面
[0180]
本公开内容的另一个附加方面提供了一种计算机系统。该计算机系统可以包括用户界面系统,用于选择目标物种和从目标物种中选择目标基因。该计算机系统可以包括与用户界面集成的设计模块,用于鉴定目标基因的一个或多个小指导rna(grna)序列。该计算机系统可以包括输出系统,用于示出选择的小grna或包含小grna的grna。每个小grna可以是每个grna的约20个碱基或核苷酸。该计算机系统可以包括激活单元,用于启动通过rna合成仪合成一个或多个小grna。
[0181]
计算机系统的设计模块可以执行用于验证预期grna的上述方法,可以利用上述方法来设计一种或多种与目标基因组区域杂交的grna。
[0182]
用户界面系统可以包括100个至500,000个不同参考基因组的选择。用户界面系统可以包括至少100个、1,000个、10,000个、100,000个、500,000个或更多个不同参考基因组的选择。用户界面系统可以包括最多500,000个、100,000个、10,000个、1,000个、100个或更少的不同参考基因组的选择。可以将不同的参考基因组存储在云端(例如,amazon web services could中的一个或多个数据库)。计算机系统的设计模块可以访问云端的参考基因组。
[0183]
计算机系统的设计模块可以访问10,000个至120,000个参考基因组。计算机系统的设计模块可以访问至少10,000个、20,000个、30,000个、40,000个、50,000个、60,000个、70,000个、80,000个、90,000个、100,000个、110,000个、120,000个或更多个参考基因组。计算机系统的设计模块可以访问最多120,000个、110,000个、100,000个、90,000个、80,000个、70,000个、60,000个、50,000个、40,000个、30,000个、20,000个、10,000个或更少的参考基因组。
[0184]
计算机系统的用户界面还可以包括用于获取个体基因组输入的基因组数据接收模块。基因组数据接收模块可以从服务器(例如,个体基因组服务的服务器)或用户上传的文件中获取个体基因组或用户基因组的一部分。
[0185]
图7a
‑
图7d示出了用于选择目标物种的基因组基因以请求设计用于杂交该基因的一个或多个grna的图形用户界面(gui)的窗口的示例。可以设计一个或多个grna来指导crispr/cas系统敲除目标基因。图7a示出了用于敲除指导物设计的gui 700的窗口,其允许用户键入目标基因组710和基因720的名称或标识符。在该gui中,使用的核酸酶730被预先选择为酿脓链球菌cas9(spcas9)。在一些情况下,用户可以选择目标cas酶。图7b示出了用于敲除指导物设计700的gui的窗口。当用户键入目标基因组的双名法的一部分(例如,“homo”)时,gui 700的软件可以向用户建议在属名中包含“homo”(例如智人(homo sapiens),712)或在种名中包含“homo”(同型腐酒乳杆菌(lactobacillus homohiochii),714)的可用基因组的列表。gui 700的软件还可以建议不同类型的相同基因组(例如,来自“genecode版本26”的智人基因组712和来自“genecode版本21”的智人基因组716)。然后,用
户可以选择正确的目标双名法。替代地或附加地,用户可以完整地键入目标双名法。图7c示出了用于敲除指导物设计700的gui的窗口。当用户键入目标基因的缩写和/或全名的一部分(例如,“re”)时,gui 700的软件可以向用户建议包含键入的输入的可用基因的列表(例如“rela”722或“alyref”724)。然后,用户可以选择正确的目标基因。替代地或附加地,用户可以完整地键入目标基因的名称。图7d示出了用于敲除指导物设计700的gui的窗口。一旦选择了基因组710、基因720和核酸酶730,用户可以单击搜索按钮740以指导gui的软件来启动设计用于与目标物种的基因组基因杂交的一个或多个grna的方法。
[0186]
图8示出了用于展示与目标基因组的基因杂交的一个或多个grna的设计进程的gui的窗口800的示例。窗口800示出了用于设计一个或多个grna的方法的步骤列表。该窗口可以通过标记已进行的步骤810并留下未标记的步骤820来示出进程。
[0187]
图9a
‑
图9d示出了gui窗口的示例,其用于展示一种或多种被设计为与目标基因组的基因杂交的grna。图9a示出了gui的窗口900。窗口900提供了设计一个或多个grna的结果的总结910(例如,目标基因中的多个cas靶位点,可用于基因敲除的多个排序最高的grna等)。窗口900提供了排序最高的grna 920的杂交多核苷酸序列。窗口900还提供了用于产生一个或多个grna的基因的选择的编码区域930(例如rela基因的外显子3)以及基因的选择区域930内排序最高的grna 920的杂交位置940的示意图。图9b示出了gui的窗口900。当用户从排序最高的grna 920中选择grna 922时,gui会在基因的选择的编码区域930中突出示出选择的grna922的相应杂交位置942。此外,如图9c所示,窗口900还示出关于选择的grna 922的细节944,包括靶多核苷酸序列、物种基因组内的cas切割位点(断裂位点)、选择的编码区域位置、中靶值和脱靶值。图9d示出了gui的窗口905。窗口905示出来自一个或多个grna的另外的grna,这些grna被设计为与目标基因组的基因杂交。用户可以从其他grna中选择至少一个,以进行进一步的分析和/或购买。
[0188]
图10a
‑
图10e示出了gui的窗口的示例,其用于展示关于设计为与目标基因组的基因杂交的grna的详细信息。当用户选择设计的grna(例如,图9c中的grna 922)时,用户被引导到新的gui窗口1000,如图10a所示。窗口1000提供了选择的grna的性能的总结1010(例如,grna的靶基因、基因组内切割位点的位置等)。窗口1000还示出了其他细节1020,包括选择用于分析的grna序列、基因组、基因和核酸酶。此外,窗口1000示出了cas
‑
grna复合物的示意图1030,其绘制成与目标基因的靶区域相互作用。窗口1000还示出了选择的grna的脱靶位点1040的示例,以及附带的信息,包括选择的grna与每个脱靶位点之间的错配数目,基因组内脱靶位点的位置和包含脱靶位点的基因的名称(图10e所示的选择的grna的其他脱靶位点1045的列表)。当用户选择示意图1030的不同部分时,gui可以通知用户示意图1030的哪个部分正在示出rna指导序列(图10b,1032)、靶位点中的原间隔子相邻基序(pam)位点(图10c,1034)和靶位点序列(图10d,1036)。
[0189]
图11a
‑
图11b示出了gui窗口的示例,其用于选择和购买被设计为与目标基因组的基因杂交的一个或多个grna的子集。图11a示出了gui的窗口900。用户可以从排序最高的grna 920中选择grna 1110的子集,并前往1120购买grna 1110的子集的合成分子。图11b示出了当用户前往1120购买grna 1110的子集的合成分子时向用户示出的窗口1100。窗口1100示出了选择的grna的总结1130(例如,选择的grna的数目及其预期的靶基因和基因组)。窗口1100还要求用户在修饰的grna 1140或未修饰的grna 1145之间进行选择以供合
成。另外,窗口1100示出被选择用于合成的grna的最终总结1150。用户可以前往付款。
[0190]
图12a
‑
图12b示出了gui的窗口的示例,其用于选择目标物种的基因组并输入先前生成的grna序列以请求验证指导性能。之前可能已经设计了grna来指导crispr/cas系统进行基因编辑。图12a示出了用于grna验证的gui 1200的窗口。gui允许用户键入基因组1210的名称或标识符号以及先前确定的grna 1220的序列。在该gui中,使用的核酸酶1230被预先选择为酿脓链球菌cas9(spcas9)。在一些情况下,用户可以选择目标cas酶。如图12b所示,一旦确定了基因组1215、grna序列1225和核酸酶1230,则用户可以前往1240验证grna序列。
[0191]
图13a
‑
图13b示出了gui的窗口的示例,其用于展示关于被设计为与目标基因组的基因杂交的grna的验证的详细信息。当用户请求验证预定的grna(例如,图12b中的grna序列1225)时,用户被引导到新的gui窗口1300,如图13a所示。窗口1300提供预定grna的性能的总结1310(例如,grna的预测靶基因,基因组内切割位点的位置等)。窗口1300还示出了其他细节1320,包括选择用于分析的预定grna序列,基因组和核酸酶。此外,窗口1300示出了cas
‑
grna复合物的示意图1330,其绘制成与预测的靶基因的靶区域相互作用。窗口1300还示出了预定grna的脱靶位点1340的示例,附带的信息包括预定grna与每个脱靶位点之间的错配数目、基因组内脱靶位点的位置以及包含脱靶位点的基因的名称,如图13b所示。系统
[0192]
本公开内容的另一个不同方面提供了一种系统,该系统包括:(1)向用户提供对超过10,000个参考基因组的访问的界面;以及(2)软件,用于从50,000多个参考基因组的任何一个中的基因选择一个或多个grna(grna);以及(3)输出系统,示出选择的指导rna。
[0193]
系统可以利用包括计算机可读介质的前述计算机系统来执行设计一种或多种与目标基因组区域杂交的grna的方法。
[0194]
该系统可包含20,000个至120,000个参考基因组。该系统可包含至少20,000个、20,000个、30,000个、40,000个、50,000个、60,000个、70,000个、80,000个、90,000个、100,000个、110,000个、120,000个或更多参考基因组。该系统可包含最多120,000个、110,000个、100,000个、90,000个、80,000个、70,000个、60,000个、50,000个、40,000个、30,000个、20,000个或更少的参考基因组。
[0195]
该系统可以包括合成多核苷酸的机器(例如,合成仪)。该软件可以与机器通信。替代地或附加地,该系统可以与合成多核苷酸的外部机器通信。在一些情况下,系统可以进一步包含激活和启动grna合成的脚本。grna的合成可以基于用户对一个或多个grna的选择。
[0196]
本公开内容进一步提供了一种用于设计指导rna(grna)的方法。用于设计grna的方法可以包括鉴定基因的初级转录物。用于设计grna的方法可以包括鉴定初级转录物与多个替代转录物之间的共同外显子。用于设计grna的方法可以包括鉴定共同外显子内的核酸酶靶位点。用于设计grna的方法可以包括相对于参考基因组序列计算核酸酶靶标的脱靶结合位点的数目,从而得出计算出的核酸酶脱靶结合位点的数目。用于设计grna的方法可以包括计算中靶效率得分,从而得出计算出的中靶效率得分。用于设计grna的方法可以包括输出至少一个grna序列,其中grna包含的序列的计算中靶效率高于阈值且核酸酶脱靶结合位点的计算数目为零。
[0197]
在一些情况下,用于设计grna的方法可以包括指导与靶位点具有部分互补性的核
酸的合成。grna与靶位点之间的部分互补性可以包含1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或超过10个核苷酸的错配。
[0198]
本公开内容进一步提供一种用于处理通过网络来自用户的生物聚合物合成请求的系统。该系统可以包括被配置为通过网络与用户的数字计算机进行通信的通信接口。该系统可以包括存储一个或多个参考基因组的参考基因组数据库。该系统可以包括计算机,该计算机包括可操作地偶联至通信接口和数据库的一个或多个计算机处理器。一个或多个计算机处理器可以被单独地或共同地编程为:(a)通过网络从通信接口接收来自用户的数字计算机的生物聚合物合成请求,该生物聚合物合成请求包括靶基因组信息;(b)相对于来自数据库的一个或多个参考基因组处理靶基因组信息,以鉴定与靶基因组信息对应的靶序列;(c)执行算法以生成与靶序列至少部分互补的第一指导核糖核酸(grna)序列集,并计算第一grna序列集中每个grna序列的脱靶互补性得分;(d)输出第二grna序列集,以示出在用户的数字计算机的图形用户界面上,其中第二grna序列集中的每一个均具有低于阈值的计算出的脱靶互补性得分;以及(e)从用户的数字计算机接收对第二grna序列集中的给定grna序列的选择。
[0199]
在一些情况下,可以对一个或多个计算机处理器进行单独或共同编程,以将给定的grna序列引导到用于合成grna序列的伫列中。在一些情况下,参考基因组数据库中的至少一个基因组可以是个体的个性化基因组。在一些情况下,参考基因组数据库中的至少一个基因组可以是受病症困扰的群体的个性化基因组集。在一些情况下,参考基因组可以是智人参考基因组。
[0200]
在一些情况下,可以对一个或多个计算机处理器进行单独或共同编程以输出预测的基因组序列。预测的基因组序列可以代表使用来自第二grna序列集的一个或多个grna编辑靶基因组信息的预测输出。预测的基因组序列可以包含基因组缺失。预测的基因组序列包含基因组插入。
[0201]
在一些情况下,计算脱靶互补性得分包括计算方位得分。在一些情况下,第二grna序列集可以示出高于特定阈值的至少两个grna。
[0202]
在一些情况下,参考基因组数据库可包含至少5万个参考基因组。在一些情况下,参考基因组数据库可包含至少12万个参考基因组。
[0203]
本公开内容还提供一种用于处理通过网络来自用户的生物聚合物合成请求的方法。该方法可以包括:(a)通过网络接收来自用户的数字计算机的生物聚合物合成请求,该生物聚合物合成请求包括靶基因组信息;(b)相对于来自参考基因组数据库的一个或多个参考基因组处理靶基因组信息,以鉴定与靶基因组信息对应的靶序列;(c)使用一个或多个计算机处理器执行算法以(i)生成与靶序列至少部分互补的第一指导核糖核酸(grna)序列集,并(ii)对每个grna序列计算第一grna序列集中每个grna序列的脱靶互补性得分;(d)输出第二grna序列集,以示出在用户的数字计算机的图形用户界面上,其中第二grna序列集中的每一个均具有低于阈值的计算出的脱靶互补性得分;以及(e)从用户的数字计算机接收合成第二grna序列集中的给定grna序列的请求。
[0204]
在一些情况下,计算机程序(例如,计算机可读介质)可被配置为指示计算机执行处理通过网络来自用户的生物聚合物合成请求的方法。
[0205]
在一些情况下,可以单独地或共同地对接收合成请求的一个或多个计算机处理器
进行编程,以指导在合成仪中合成第二grna序列集中的给定grna序列。在一些情况下,参考基因组数据库中的至少一个基因组可以是个体的个性化基因组。在一些情况下,参考基因组数据库中的至少两个基因组可以是受病症困扰的群体的个性化基因组。在一些情况下,参考基因组可以是智人参考基因组。
[0206]
在一些情况下,该方法可以还包括输出预测的基因组序列。预测的基因组序列可以代表用来自第二grna序列集的一个或多个grna编辑靶基因组信息的预测输出。在一些情况下,预测的基因组序列可包含基因组缺失。在一些情况下,预测的基因组序列可包含基因组插入。在一些情况下,计算可以计算方位得分。在一些情况下,第二grna序列集可以示出高于特定阈值的至少两个grna。在一些情况下,参考基因组数据库可包含至少5万个参考基因组。在一些情况下,参考基因组数据库可包含至少12万个参考基因组。
[0207]
本公开内容进一步提供了一种包括机器可执行代码的非暂时性计算机可读介质,该机器可执行代码在被一个或多个计算机处理器执行时,实现了用于处理通过网络来自用户的生物聚合物合成请求的方法。该方法可以包括通过网络从用户的数字计算机接收生物聚合物合成请求,该生物聚合物合成请求包括靶基因组信息。该方法可以包括相对于来自参考基因组数据库的一个或多个参考基因组处理靶基因组信息,以鉴定对应于靶基因组信息的靶序列。该方法可以包括执行算法以生成与靶序列至少部分互补的第一指导核糖核酸(grna)序列集,并为第一grna序列集中的每个grna序列计算脱靶互补性得分。该方法可以包括输出第二grna序列集以在用户的数字计算机的图形用户界面上示出,其中第二grna序列集中的每一个具有低于阈值的计算出的脱靶互补性得分。该方法可以包括从用户的数字计算机接收对第二grna序列集中的给定grna序列的选择。计算机系统
[0208]
本公开内容提供了被编程为实现本公开内容的方法的计算机系统。本公开内容的计算机系统可以用于设计一种或多种用于与目标基因组区域杂交的指导rna。目标基因组区域可以是物种的基因组基因。来自此处描述的任何计算机系统的信息都可以向远程计算机提供报告。
[0209]
图14示出了被编程或以其他方式配置成与本公开内容的计算机系统通信并调节本公开内容的计算机系统的各个方面的计算机系统1401。
[0210]
计算机系统1401可以调节本公开内容的各个方面,例如,设计用于与物种的基因组基因杂交的一个或多个指导rna,或者通过列举目标基因组中潜在的指导rna杂交位点的错配数目来计算脱靶值。。计算机系统1401可以是用户的电子设备或相对于该电子设备位于远程的计算机系统。该电子设备可以是移动电子设备。
[0211]
计算机系统1401包括中央处理单元(cpu,在本文中也称为“处理器”和“计算机处理器”)1405,其可以是单核或多核处理器,或者是用于并行处理的多个处理器。计算机系统1401还包括存储器或存储器位置1410(例如,随机存取存储器、只读存储器、闪存)、电子存储单元1415(例如,硬盘)、与一个或多个其他系统通信的通信接口1420(例如,网络适配器)以及外围设备1425,例如高速缓存、其他存储器、数据存储和/或电子示出适配器。存储器1410、存储单元1415、接口1420和外围设备1425通过诸如主板的通信总线(实线)与cpu 1405通信。存储单元1415可以是用于存储数据的数据存储单元(或数据存储库)。计算机系统1401可以借助于通信接口1420可操作地偶联至计算机网络(“网络”)1430。网络1430可以
是因特网、互联网和/或外联网或与因特网通信的内联网和/或外联网。在一些情况下,网络1430是电信和/或数据网络。网络1430可以包括一个或多个计算机服务器,其可以实现分布式计算,例如云计算。在一些情况下,网络1430可以在计算机系统1401的帮助下实现对等网络,该对等网络可以使偶联至计算机系统1401的设备能够充当客户端或服务器。
[0212]
cpu 1405可以执行一系列机器可读指令,该指令可以体现在程序或软件中。指令可以被存储在诸如存储器1410的存储器位置中。指令可以被定向到cpu 1405,其可以随后对cpu 1405进行编程或以其他方式配置以实现本公开内容的方法。cpu 1405执行的操作的示例可以包括获取、解码、执行和写回。
[0213]
cpu 1405可以是电路的一部分,例如集成电路。系统1401的一个或多个其他组件可以包括在电路中。在一些情况下,该电路是专用集成电路(asic)。
[0214]
存储单元1415可以存储文件,例如驱动程序、文库和保存的程序。存储单元1415可以存储用户数据,例如用户偏好和用户程序。在一些情况下,计算机系统1401可以包括计算机系统1401外部的一个或多个附加数据存储单元,例如位于通过内部网或因特网与计算机系统1401通信的远程服务器上。
[0215]
算机系统1401可以通过网络1430与一个或多个远程计算机系统通信。例如,计算机系统1401可以与用户的远程计算机系统通信。远程计算机系统的示例包括个人计算机(例如,便携式pc)、平板或平板计算机(例如,ipad,galaxy tab)、电话、智能电话(例如,iphone、支持安卓的设备、)或个人数字助理。用户可以通过网络1430访问计算机系统1401。
[0216]
本文所述的方法可以通过存储在计算机系统1401的电子存储位置(例如,存储器1410或电子存储单元1415上)的机器(例如计算机处理器)可执行代码的方式来实现。机器可执行或机器可读代码可以以软件的形式提供。在使用期间,代码可以由处理器1405执行。在一些情况下,可以从存储单元1415检索代码并将其存储在存储器1410中,以供处理器1405随时访问。在一些情况下,可以不包括电子存储单元1415,并且将机器可执行指令存储在存储器1410中。
[0217]
代码可以被预编译并配置为与具有适于执行代码的处理器的机器一起使用,或者可以在运行时被编译。可以以编程语言的形式提供代码,可以选择编程语言以使代码能够以预编译或即时编译的方式执行。
[0218]
本文提供的系统和方法的方面,例如计算机系统1401,可以体现为编程。可以将技术的各个方面视为“产品”或“制造品”,其通常以机器(或处理器)可执行代码和/或关联数据的形式承载或体现在一类机器可读介质中。机器可执行代码可以存储在电子存储单元上,例如存储器(例如,只读存储器、随机存取存储器、闪存)或硬盘。“存储”类型的介质可以包括计算机、处理器等的任何或所有有形存储器,或其相关模块,例如各种半导体存储器、磁带驱动器、磁盘驱动器等,它们可以提供非暂时性存储以供随时进行软件编程。软件的全部或部分有时可以通过因特网或其他各种电信网络进行通信。这样的通信例如可以使得能够将软件从一个计算机或处理器加载到另一计算机或处理器,例如从管理服务器或主机加载到应用服务器的计算机平台。因此,可以承载软件元件的另一种介质包括光波、电波和电磁波,例如通过有线和光学陆线网络以及通过各种空中链路跨本地设备之间的物理接口使
用的。诸如有线或无线链路、光学链路等携带这些波的物理元件也可以视为承载软件的介质。如本文所使用的,除非限于非暂时性的有形“存储”介质,否则诸如计算机或机器“可读介质”的术语是指参与向处理器提供指令以供执行的任何介质。
[0219]
因此,诸如计算机可执行代码的机器可读介质可以采取许多形式,包括但不限于有形存储介质、载波介质或物理传输介质。非易失性存储介质包括例如光盘或磁盘,例如任何计算机中的任何存储设备等,诸如其可用于实现附图中所示的数据库等。易失性存储介质包括动态存储器,例如这种计算机平台的主存储器。有形的传输介质包括同轴电缆;铜线和光纤,包括构成计算机系统内总线的电线。载波传输介质可以采用电信号或电磁信号或声波或光波的形式,例如在射频(rf)和红外(ir)数据通信期间生成的那些。因此,计算机可读介质的常见形式包括:软盘、柔性盘、硬盘、磁带、任何其他磁介质、cd
‑
rom、dvd或dvd
‑
rom、任何其他光学介质、穿孔卡片纸带、带孔图案的任何其他物理存储介质、ram、rom、prom和eprom、flash
‑
eprom、任何其他存储芯片或盒带、用于传输数据或指令的载波、用于传输此类载波的电缆或链接,或计算机可以从中读取编程代码和/或数据的任何其他介质。这些形式的计算机可读介质中的许多可以涉及将一个或多个指令的一个或多个序列传送给处理器以执行。
[0220]
计算机系统1401可以包括电子显示器1435或与之通信,该电子显示器包括用户界面(ui)1440,用于提供例如选择目标物种和从目标物种选择目标基因的能力。ui的示例包括但不限于图形用户界面(gui)和基于网络的用户界面。
[0221]
可以通过一种或多种算法来实现本公开内容的方法和系统。可以在中央处理单元1405执行时通过软件来实现算法。该算法可以例如设计用于与物种的目标基因组区域杂交的一个或多个grna,并激活和启动一个或多个grna中的至少一个的合成。实施例实施例1:使用多个指导rna比使用单指导rna具有更高的编辑效率
[0222]
总共设计了228个指导rna来杂交76个基因,每个基因设计了三个指导rna。设计每一个三指导rna的集的以使指导物间间隔至少为30bp。将指导rna导入hek293和mcf7细胞中,该细胞以每孔35,000个细胞的密度接种在96孔板上。用于单指导编辑的指导rna以4.5μmol转染,而多指导rna中的指导rna的以各2.25μmol转染。通过核转染将所有指导rna都作为核糖核蛋白(rnp)转染。转染前,将0.5μmol的cas9与rnp进行复合。转染后第2天,通过sanger测序查询细胞的所得基因型,并使用crispr编辑推理(ice)分析总体编辑效率。使用百分比编辑效率来指示在设计grna进行编辑的位置处包含非野生型基因型的细胞的百分比。针对每个基因使用单grna以及每个基因使用三grna集评估编辑效率(图15)。数据点后面的方框图表示中位数,25/75
th
百分位数和5/95
th
百分位数。这些结果表明,使用具有指定的指导物间间隔的三grna可实现比使用与目标基因杂交的单grna更高的编辑效率(p<1e
‑
15,mann
‑
whitney u检验)。
[0223]
进一步分析了使用多指导sgrna的编辑结果中的指导物间隔的影响。设计了537个grna,可杂交179个基因,每个基因设计了三个grna。设计的这些grna的指导物间间隔在
‑
20bp(即完全重叠)至80bp之间。用于多指导的指导rna各自以2.25μmol转染。通过核转染将所有grna均作为核糖核蛋白(rnp)转染。转染前,将0.5μmol cas9与rnp进行复合。转染后第2天,通过sanger测序询问细胞的基因型,并分析总体编辑效率。随着指导物间间隔(即,指
导物之间的末端到起点的距离)增加到超过30个碱基对(bp),总体编辑效率得到改善,从而未观察到小于75%的效率(图16)。实施例2:多种指导rna试剂盒的组合
[0224]
设计了九个sgrna,每三个sgrna靶向人类基因组中的三个靶区域中的每一个:两个基因——蛋白质精氨酸甲基转移酶5(prmt5)和甲硫腺苷磷酸化酶(mtap),以及腺相关病毒整合位点1(aavs1)中的一个位点。在每个sgrna集中,指导物间间隔至少为30bp。通过sanger测序确定每个成对组合中的每个基因的编辑效率(图17)。
[0225]
将每孔5000个hep3b细胞接种在96孔板中。使用nucleofector
tm
技术(lonza),将这些细胞用靶向该对的多个rnp转染,并在转染后24、48和72个小时测定细胞滴度。这些结果表明,可以在一次转染中同时在多个基因组位点创建编辑。实施例3:阵列化文库筛选
[0226]
在96孔板的92孔中每孔接种35,000个u2os细胞。92个孔中的每个孔还包含靶向基因的2或3个sgrna的集(指导物间间隔至少为30bp,总共有92个被筛选试验靶向的不同基因)和cas9核酸内切酶。使用nucleofector
tm
技术(lonza)转染细胞。随后在转染后6天使用发光细胞活力测定法评估细胞活力(图18a)。另外,在转染后2天用sanger测序对这些细胞进行基因分型,然后使用crispr编辑推理(ice)进行分析以确定编辑效率(图18b)。评估相同细胞群体的所得基因型的能力可能是阵列筛选方法优于合并筛选方法的优势。
[0227]
尽管本文中已经示出并描述了本发明的各种实施方案,但对于本领域技术人员容易理解的是,这些实施方案仅以示例的方式提供。并非意图通过说明书中提供的特定示例来限制本发明。尽管已经参考前述说明书描述了本发明,但是本文中的实施方案的描述和图示并无意于以限制性的意义来解释。本领域技术人员在不脱离本发明的情况下可想到多种变化、改变和替代。此外,应当理解,本发明的所有方面不限于本文所阐述的具体描述、构造或相对比例,其取决于各种条件和变量。应当理解,可采用本文所述的本发明实施方案的各种替代方案。因此可以理解,本发明还将涵盖任何这样的替代、修改、变化或等同形式。所附权利要求旨在限定本发明的范围,并且由此涵盖这些权利要求范围内的方法和结构及其等同物。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1