一种H7N9病毒特异性识别抗体P52H12及检测试剂盒的制作方法
本发明涉及病毒诊断技术领域,具体涉及一种h7n9型禽流感病毒的特异性识别抗体p52h12及该病毒的检测试剂盒。
背景技术:
急性呼吸道感染是人类最普遍的感染性疾病。多数呼吸道感染是由病毒引起的,临床上常见的病原体有腺病毒、流感病毒、副流感病毒、呼吸道合胞体病毒和鼻病毒,而支原体和衣原体均为呼吸道致病细菌。由于针对不同细菌和病毒感染采取的治疗方案有所不同,所以在感染后就需要尽快做出诊断。同时由于高致病性流感病毒的强传播性和高致死性,需对其进行严格的隔离与防护。由于一般的临床诊断方法不能鉴别病原体的差别,因此就需要采取准确可靠的实验室方法进行病原体的鉴别诊断。
为实现针对流感病毒抗原的检测,需要得到针对流感病毒的高亲和力抗体,目前市场上的检测抗体多为动物来源,例如:杂交瘤技术发展出的鼠源抗体和兔源抗体。但由于人血清成分的复杂性,因此很容易产生假阳性的检测情况,即未感染病毒的血清仍可能存在针对鼠源或兔源抗体的抗体,这时的血清学诊断结果即为假阳性。这将为临床未发病的疑似病例诊断带来很大的困难。为了解决这一问题,目前已有方案是对得到的鼠源抗体或兔源抗体进行种属改变,即对其进行人源化,但在这过程中,将大概率会导致抗体的亲和力和特异性的严重丢失,最终得到的人源化抗体也失去了其应用的价值。因此我们利用噬菌体展示技术,筛选针对流感病毒抗原的人源的高亲和力和高特异性的单克隆抗体用于临床的诊断。
技术实现要素:
本研究通过构建人源抗体的噬菌体展示文库,筛选针对流感病毒h7n9抗原的高亲和力和高特异性的人源单克隆抗体,用于临床的诊断,以解决目前临床诊断上普遍存在的假阳性情况,为流感病毒的防控提供准确的检测结果。
下面详述本发明的技术方案:
第一方面,本发明提供了一种h7n9病毒特异性识别抗体p52h12,重链及轻链的决定簇互补区(complementarity-determiningregion,cdr)氨基酸序列如下:
决定簇互补区为高变区,是抗体与抗原的结合位置,cdr3具有更高的高变程度,重链在于抗原结合中起重要的作用。
优选的,上述抗体p52h12中,重链氨基酸序列如seqidno.1所示,轻链氨基酸序列如seqidno.2所示。
第二方面,本发明提供了上述抗体p52h12的编码基因。
优选的,上述编码基因中,重链核苷酸序列如seqidno.3所示,轻链核苷酸序列如seqidno.4所示。
第三方面,本发明提供了含上述编码基因的载体。
第四方面,本发明提供了含上述编码基因的宿主细胞。
第五方面,本发明提供了上述抗体p52h12在制备h7n9病毒的诊断试剂中的应用。
第六方面,本发明提供了一种h7n9病毒特异性检测试剂盒,含有上述抗体p52h12。
优选的,上述检测试剂盒中,还含有包被有抗体p52h12的微孔板、经修饰的抗体p52h12作为二抗、缓冲溶液、样品稀释液、显色液、阴性对照血清和阳性对照液。缓冲溶液、样品稀释液、显色液和阳性对照液等均采用本领域常规试剂即可,无特殊成分要求。例如,缓冲溶液和样品稀释液可使用pbs,酶标二抗可以采用hrp标记,使用tmb显色液或abts显色液。阳性对照液中含有h7n9灭活抗原和pbs。
与现有技术相比,本发明具有如下有益效果:
本发明的抗体p52h12为人源单克隆抗体,能够特异性识别h7n9流感病毒抗原,具有高亲和力和高特异性,抗原抗体结合的ec50值较小,远高于目前的大部分抗体(ec50值一般在五十至几百纳摩尔),亲和力非常高,而且相比于现有的rt-pcr检测方法,使用该抗体直接进行免疫检测更加简单快速、低成本,可有效解决目前临床诊断上普遍存在的假阳性情况,为流感病毒的防控提供准确的检测结果。
另外,使用本发明的抗体进行免疫检测,相较于以往的rt-pcr方法检测,更加简单快速。
附图说明
图1为p52h12蛋白表达产物电泳图。
图2为通过elisa对p52h12igg形式抗体进行亲和力测定结果。
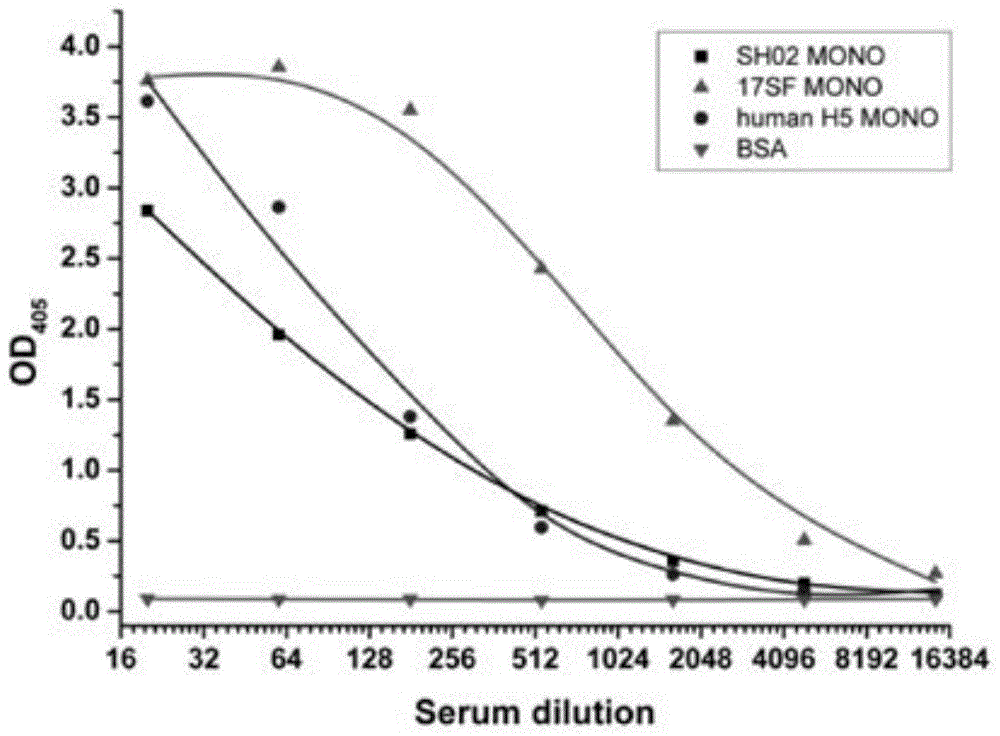
图3为恢复期病人血清与sh02ha毒株(a/shanghai/2/2013h7n9)、17sfha毒株(a/guangdong/17sf003/2016)、humanh5rha单体蛋白(sh02mono,17sfmono,humanh5mono)结合验证实验结果,bsa为阴性对照。
具体实施方式
下面结合具体实施例对本发明作进一步说明,以使本领域的技术人员可以更好地理解本发明并能予以实施,但所举实施例不作为对本发明的限定。
以下实施例中所使用的技术,除非特别说明,均为本领域的技术人员已知的常规技术;所使用的仪器设备、试剂等,除非是本说明书特别说明,均为本领域的研究和技术人员可以通过公共途径获得的。
实施例1h7n9病毒特异性识别抗体的筛选
(1)构建人源fab抗体的噬菌体展示文库
以感染h7n9病毒后恢复期的病人血清与sh02ha、17sfha、humanh5rha单体蛋白(sh02mono、17sfmono、humanh5mono)进行结合实验,实验结果如图3所示,说明恢复期病人血清中含有能h7或h5抗原结合的抗体,bsa作为阴性对照,没有非特异性结合。选用有特异性结合的病人血液作为建库样本。
以能够与上述病毒蛋白进行特异性结合的病人血液为样本,分离淋巴细胞,提取总rna,经逆转录合成cdna,以cdna为模板,利用特异性引物对抗体的fab片段进行扩增。将得到的片段连接到噬菌粒载体上,电转化到商业化的tg1感受态中,加入辅助噬菌体以产生人源fab抗体噬菌体展示文库。
(2)针对h7n9的ha抗原进行初步筛选
将流感病毒的ha基因构建在杆状病毒表达载体上,经sf9细胞表达得到h7n9的囊膜蛋白ha,并以此为靶标(靶蛋白)进行筛选,得到若干候选抗体。对这些候选抗体进行亲和力测定(elisa,生物传感器)和表位鉴定,综合考虑多种因素后确定若干候选克隆做进一步优化。
(3)候选克隆的亲和力成熟
以筛选到的候选抗体为模板做随机突变,构建亲和力成熟的噬菌体展示文库。继续利用上述靶蛋白进行筛选,经过几轮筛选-突变,得到针对ha亲和力较高的克隆。
(4)人源抗体亲和力和特异性分析
挑取若干候选克隆,将它们连入真核表达载体中,在293f细胞表达并纯化igg形式的候选克隆。对这些克隆进行亲和力测定(elisa,生物传感器)、特异性分析(双抗夹心法验证不同ha抗原和其他无关抗原)。
最终筛选获得特异性、灵敏度高的抗体片段,命名为p52h12,其重链和轻链核苷酸序列如seqidno.3-4所示,其重链和轻链氨基酸序列如seqidno.1-2所示。igg蛋白表达电泳结果如图1所示,p52h12蛋白约在55kd左右。
实施例2抗体p52h12性质检测
参见图2,为通过elisa对p52h12igg抗体进行亲和力测定的结果,其中●表示17sfha毒株(a/guangdong/17sf003/2016),■表示sh02ha毒株(a/shanghai/2/2013h7n9),▲表示bsa阴性对照。
结果显示,p52h12对h7n9的两个毒株sh02ha以及17sfha蛋白的ec50值分别为7.67nm和34.4nm,具有较高的亲和力,对h7亚型的ha蛋白具有一定的交叉活性;同时elisa结果显示该抗体与对照bsa没有结合,没有非特异性。该抗体无中和抑制活性。
以上所述实施例仅是为充分说明本发明而所举的较佳的实施例,本发明的保护范围不限于此。本技术领域的技术人员在本发明基础上所作的等同替代或变换,均在本发明的保护范围之内。本发明的保护范围以权利要求书为准。
序列表
<110>武汉珈创生物技术有限公司
<120>一种h7n9病毒特异性识别抗体p52h12及检测试剂盒
<130>wh2006143-1
<160>9
<170>siposequencelisting1.0
<210>1
<211>237
<212>prt
<213>人工序列(artificialsequence)
<400>1
gluvalglnleuvalglnserglyalagluvallyslysproglyser
151015
servallysvalsercyslysalaserglyglythrpheserasntyr
202530
alailesertrpvalargglnalaproglyglnglyleuglutrpmet
354045
glyglyileileproilepheglythralaasntyralaglnlysphe
505560
glnglyargvalthrilethralaaspgluserthrserthralatyr
65707580
metgluleuserserleuargsergluaspthralavaltyrtyrcys
859095
alaleuprocysserserthrsercystyrleuvalvalglnglyala
100105110
tyrtyrtyrglymetaspvaltrpglyglnglythrthrvalthrval
115120125
serseralaserthrlysglyproservalpheproleualaproser
130135140
serlysserthrserglyglythralaalaleuglycysleuvallys
145150155160
asptyrpheprogluprovalthrvalsertrpasnserglyalaleu
165170175
thrserglyvalhisthrpheproalavalleuglnserserglyleu
180185190
tyrserleuserservalvalthrvalproserserserleuglythr
195200205
glnthrtyrilecysasnvalasnhislysproserasnthrlysval
210215220
asplyslysvalgluprolyssercysasplysthrser
225230235
<210>2
<211>219
<212>prt
<213>人工序列(artificialsequence)
<400>2
aspvalvalmetthrglnserproleuserleuprovalthrprogly
151015
gluproalaserilesercysargserserglnserleuleuhisser
202530
asnglytyrasntyrleuasptrptyrleuglnlysproglyglnser
354045
proglnleuleuiletyrleuglyserasnargalaserglyvalpro
505560
aspargpheserglyserglyserglythraspphethrleulysile
65707580
serargvalglualagluaspvalglyvaltyrtyrcysmetglnala
859095
leuglnthrproprothrpheglyglyglythrlysvalgluvallys
100105110
argthrvalalaalaproservalpheilepheproproseraspglu
115120125
glnleulysserglythralaservalvalcysleuleuasnasnphe
130135140
tyrproargglualalysvalglntrplysvalaspasnalaleugln
145150155160
serglyasnserglngluservalthrgluglnaspserlysaspser
165170175
thrtyrserleuserserthrleuthrleuserlysalaasptyrglu
180185190
lyshislysleutyralacysgluvalthrhisglnglyleuserser
195200205
provalthrlysserpheasnargglyglucys
210215
<210>3
<211>711
<212>dna
<213>人工序列(artificialsequence)
<400>3
gaggtgcagctggtgcagtctggggctgaggtgaaaaagcctgggtcctcggtgaaggtc60
tcctgcaaggcttctggaggcaccttcagcaactatgctatcagctgggtgcgacaggcc120
cctgggcaagggcttgagtggatgggagggatcatccctatctttggtacagcaaactac180
gcacagaagttccagggcagagtcacgattaccgcggacgaatccacgagcacagcctac240
atggagctgagcagcctgagatctgaggacacggccgtgtattactgtgcactaccttgt300
agtagtaccagctgctatttggttgtacaaggagcttactactacggtatggacgtctgg360
ggccaagggaccacggtcaccgtctcctcagcctccaccaagggcccatcggtcttcccc420
ctggcaccctcctccaagagcacctctgggggcacagcggccctgggctgcctggtcaag480
gactacttccccgaaccggtgacggtgtcgtggaactcaggcgccctgaccagcggcgtg540
cacaccttcccggctgtcctacagtcctcaggactctactccctcagcagcgtggtgacc600
gtgccctccagcagcttgggcacccagacctacatctgcaacgtgaatcacaagcccagc660
aacaccaaggtggacaagaaagttgagcccaaatcttgtgacaaaactagt711
<210>4
<211>660
<212>dna
<213>人工序列(artificialsequence)
<400>4
gatgttgtgatgactcagtctccactctccctgcccgtcacccctggagagccggcctcc60
atctcctgcaggtctagtcagagcctcctgcatagtaatggatacaactatttggattgg120
tacctgcagaagccagggcagtctccacagctcctgatctatttgggttctaatcgggcc180
tccggggtccctgacaggttcagtggcagtggatcaggcacagattttacactgaaaatc240
agcagagtggaggctgaggatgttggggtttattactgcatgcaagctctacaaactcct300
cccactttcggcggagggaccaaggtggaggtcaaacgaactgtggctgcaccatctgtc360
ttcatcttcccgccatctgatgagcagttgaaatctggaactgcctctgttgtgtgcctg420
ctgaataacttctatcccagagaggccaaagtacagtggaaggtggataacgccctccaa480
tcgggtaactcccaggagagtgtcacagagcaggacagtaaggacagcacctacagcctc540
agcagcaccctgacgctgagcaaagcagactacgagaaacacaaactctacgcctgcgaa600
gtcacccatcagggcctgagctcgcccgtcacaaagagcttcaacaggggagagtgttaa660
<210>5
<211>8
<212>prt
<213>人工序列(artificialsequence)
<400>5
glyglythrpheserasntyrala
15
<210>6
<211>8
<212>prt
<213>人工序列(artificialsequence)
<400>6
ileileproilepheglythrala
15
<210>7
<211>23
<212>prt
<213>人工序列(artificialsequence)
<400>7
alaleuprocysserserthrsercystyrleuvalvalglnglyala
151015
tyrtyrtyrglymetaspval
20
<210>8
<211>11
<212>prt
<213>人工序列(artificialsequence)
<400>8
glnserleuleuhisserasnglytyrasntyr
1510
<210>9
<211>9
<212>prt
<213>人工序列(artificialsequence)
<400>9
metglnalaleuglnthrproprothr
15
- 还没有人留言评论。精彩留言会获得点赞!