基于可见光和近红外高光谱成像的桑螟幼虫及其对桑叶损害的快速识别方法与流程
本发明涉及桑螟幼虫及桑叶损伤的快速识别方法领域,具体涉及一种基于可见光和近红外高光谱成像的桑螟幼虫及其对桑叶损害的快速识别方法。
背景技术:
中国的蚕桑生产已有几千年的历史,已形成一个具有一定经济价值的完整产业。作为蚕桑最重要的生产材料之一,桑叶是蚕的唯一饲料来源。桑叶的产量和品质直接制约了蚕桑的发展。2017年,中国桑园总面积达到788.73万hm2,比2000年的632.5万hm2增加了24.7%。在过去十年中,尽管桑树种植面积有所增加,但桑叶的产量因害虫而遭受巨大损失。桑螟是一种寄生虫,是桑树常见的重要害虫之一,桑螟啃食叶子并对桑叶的生产构成巨大威胁。桑螟的排泄也会严重影响桑叶的质量,对蚕茧和桑蚕的产生构成严重威胁。桑树害虫的防治是当前桑树田间管理的重点。蚕桑业需要开发一种快速无损的桑螟检测技术,从而为蚕提供优质的桑叶作为饲料。
目视检测是有害生物检测的常用方法。然而,人工观察是费力的,昂贵的和乏味的。此外,由于幼虫很小并且它们的颜色与桑叶的颜色非常相似,因此在基于肉眼的害虫检测中容易出错。另一方面,计算机视觉技术可以以低成本的方式衡量识别害虫及其损害的客观结果。与目视检测不同,图像检测不受人为因素的干扰,因此可以减少主观因素造成的干扰。然而,由于它主要检测可见光谱范围内的单色或彩色图像,计算机视觉技术无法区分具有相似颜色的样本。桑螟和桑叶的颜色相似,因此很难通过目视观察或计算机视觉技术来区分害虫和叶子。
作为计算机视觉技术的延伸,高光谱成像(hsi)被广泛用于各种食品和农产品的质量检测以及植物病虫害检测。hsi可以同时获得样本的光谱和空间信息,并形成三维的(3-d)数据立方体。因此,hsi可以使用光谱信息来区分害虫和叶子,并通过立方体中包含的空间信息来定位害虫的空间位置。然而,到目前为止,基于hsi技术,对于快速检测桑螟及其对桑叶的损害的报道很少。
本发明利用可见光和近红外(vis-nir)高光谱反射成像技术来区分桑螟幼虫和桑叶,并以快速和客观的方式识别桑螟对桑叶的损害。实验在可见光和近红外(400-1700nm)光谱范围内测量高光谱图像,并提取桑螟幼虫和桑叶的特征光谱,之后通过多变量分析建立健康叶片与损伤叶片、健康叶片和桑螟幼虫、损伤叶片和桑螟幼虫、及所有样品之间的分类模型,确定对分类最有用的几个重要波长,最后是分别对在400-1000nm和900-1700nm范围内工作的两个高光谱成像系统进行比较。这项实验的成功结果非常有利于为蚕农提供优质的桑叶,从而提高蚕的产量,提高蚕丝的质量。
技术实现要素:
本发明提供一种基于可见光和近红外高光谱成像的桑螟幼虫及其对桑叶损害的快速识别方法,可在对桑树的管理中,实现为蚕农提供优质的桑叶,从而提高蚕的产量,提高蚕丝的质量。
一种基于可见光和近红外高光谱成像的桑螟幼虫及其对桑叶损害的快速识别方法,包括如下步骤:
步骤1,采摘桑叶作为待测样本,获得每片叶子的可见光和近红外高光谱;
从当地桑园采摘桑叶,然后选择除桑螟幼虫损伤外没有其他缺陷的叶子;获得三种叶子,即健康叶片,具有幼虫损伤的叶子和具有幼虫的叶子;
步骤2,将步骤1获得的原始图像校正为高光谱图像,并对校正后高光谱图像进行识别样品的roi,获得叶脉、健康叶肉、轻微损伤叶肉和严重损伤叶肉以及幼虫的五类roi;
桑叶样本放置在传送带上并逐行在镜头下移动,获得每片叶子的可见光(400-1000nm)范围数据和nir(900-1700nm)范围数据;在完成数据收集之后,获得包含光谱(λ)和空间(x,y)信息的3-d数据立方体。
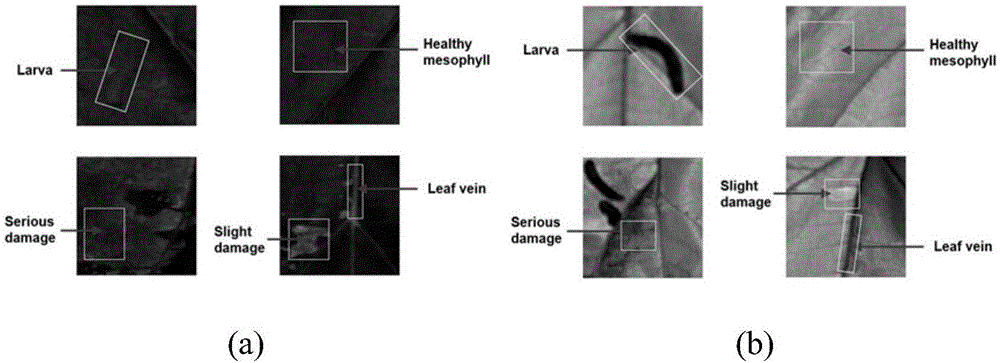
步骤3,将步骤2中获得的每个roi都是作为样本,计算每个roi中所有像素的平均值以获得该roi的代表性光谱,如图2中(a)和(b)所示,将每个样本的代表性光谱组合成光谱矩阵x,使用不同自然数来表示上述五类roi,并将所有样本的类别编号组合成列向量y;
矩阵的每列是变量并代表一个波长。矩阵的每一行代表一个样本。
本技术方案中个,有五类roi,即叶脉,健康叶肉,轻微损伤叶肉,严重损害叶肉和桑螟幼虫,使用主成分分析(pca)判别分析五类的可分性,结果如图3所示。使用自然数1,2,3,4,5来表示上述五个类别。
步骤4,使用全变量以及多变量分析建立矩阵x和向量y之间的定性分析模型;
步骤5,重新采集桑叶,获取桑叶的高光谱图,经步骤2和步骤3处理后,带入步骤4所建模型中,进行桑螟幼虫及其对桑叶损害的快速识别。
本技术方案中,手动在enviv4.6软件的感兴趣区域(roi)功能中对步骤3获得的校正后图像进行识别样品的roi,获得叶脉、健康叶肉、轻微损伤叶肉和严重损伤叶肉以及幼虫的五类roi。
作为优选的,步骤1及步骤5中挑选在相同的种植环境且长势良好的桑树上的叶位一致、除桑螟幼虫损伤外没有其他缺陷的桑叶。即尽可能选择相同叶位,树龄一致,长势近似,大小近似的叶片。
作为优选的,步骤2中,使用基于白色和暗图像参考将原始图像校正为高光谱图像。步骤2中,在收集数据时黑盒子被关闭,以避免外部光线的影响。在可见光范围数据通过光谱仪用si检测器(imspectorv10e,spectralimagingltd.,oulu,finland)测量。nir范围数据通过光谱仪用ingaas检测器(imspectorn17e,spectralimagingltd.,oulu,finland)测量。测量所有片叶子的高光谱图像。
步骤2中,收集的原始图像信号是信号强度,而不是样品的反射光谱;因此,使用基于白色和暗图像参考将原始图像校正为高光谱图像,校正后结果如图1中(a)和(b)所示;
作为优选的,白色参考用聚四氟乙烯白板,具有接近99%的反射率。为了捕捉暗图像,关闭灯并用不透明的盖子盖住镜头。进一步优选的,暗图像和白图像的数据采集过程与样本的图像采集过程一致。
步骤2中可见光高光谱图像中roi中的像素点对于叶脉通常为100至400,对于健康叶肉为4000和12,000,对于轻微损伤叶肉为400和2000,对于严重损害叶肉为1200和4800,对于桑螟幼虫为200和800。另一方面,近红外高光谱图像中roi的像素点一般在叶脉为25到100之间,健康叶肉为1000和3000,轻度损伤叶肉为100和500,严重损害叶肉为300和1200,桑螟幼虫为50和200。
作为优选的,步骤4中,针对全变量分析用偏最小二乘判别分析pls-da,最小二乘支持向量机ls-svm和线性判别分析lda算法分别建立了用于鉴定五类样本的定性分析模型。
作为优选的,步骤4中,针对多变量分析分别使用连续投影算法spa,非信息变量消除uve,uve-spa和竞争自适应重加权抽样cars方法选择可以区分昆虫和叶片损害的波长,之后建立五类样本的定性分析模型。
作为优选的,将所有样本随机分成一组校准(四分之三样本)和另一个独立的预测集(四分之一样本)。
本发明获取叶脉,健康叶肉,轻微损伤叶肉,严重损害叶肉和桑螟幼虫高光谱图;对高光谱图进行校正处理;对校正处理后的图进行识别样品的roi;计算一个roi中所有像素的平均值以获得该roi(样本)的代表性光谱,将每个样品的代表性光谱组合成光谱矩阵(x);分为五类roi,即叶脉,健康叶肉,轻微损伤叶肉,严重损害叶肉和桑螟幼虫,使用不同自然数来表示上述五个类别,将所有样品的类别编号组合成列向量(y);使用全变量以及多变量分析建立矩阵x和向量y之间的定性分析模型,根据模型结果分析基于可见光和近红外高光谱成像的桑螟幼虫及其对桑叶损害的快速识别方法的可行性与精确度。
附图说明
图1为波长为660nm(a)和1250nm(b)时叶脉、健康叶肉、轻微损伤叶肉、严重损伤叶肉以及桑螟幼虫的高光谱图;其中,(a)为波长660nm的高光谱图;(b)为波长为1250nm的高光谱图;
图2为2400-1000nm(a)以及900-1700nm(b)波长范围内叶脉、健康叶肉、轻微损伤叶肉、严重损伤叶肉以及桑螟幼虫的代表性光谱图;其中,(a)为波长在2400-1000nm代表性光谱图,(b)为波长在900-1700nm的代表性光谱图;
图3为五类roi使用主成分分析(pca)分析图;
图4为基于可见光和近红外范围数据的矩阵,用于识别五类样品(叶脉,健康叶肉,轻微损伤,严重损伤和桑螟幼虫)的分类模型的结果图;
具体实施方式
下面结合实施例和附图来详细说明本发明,但本发明并不仅限于此。
本实施例中,基于可见光和近红外高光谱成像的桑螟幼虫及其对桑叶损害的快速识别方法,包括以下步骤:
(1)样本制备:从当地桑园采摘相同叶位,树龄一致,长势近似,大小近似的桑叶,然后选择除桑螟幼虫损伤外没有其他缺陷的叶子。获得三种叶子(120片叶子),即健康叶片(35片叶子),具有幼虫损伤的叶子(70片叶子)和具有幼虫的叶子(15片叶子)。
(2)数据采集:高光谱成像系统由两部分组成。一个是光谱采集单元,它位于一个大的黑盒子里。光谱采集单元有两个摄像头,两个成像摄谱仪和两个可调150瓦石英卤钨灯。两个光谱仪分别测量400-1000nm(可见光范围)和900-1700nm(nir范围)的高光谱图像。在收集数据时黑盒子被关闭,以避免外部光线的影响。另一部分是控制单元,它有一个输送带,一个步进电机和一台电脑。在数据采集期间,将桑叶放置在传送带上并逐行在镜头下移动。获得每片叶子的可见光范围数据(400-1000nm)和nir范围数据(900-1700nm)。可见光范围数据通过光谱仪用si检测器(imspectorv10e,spectralimagingltd.,oulu,finland)测量。nir范围数据通过光谱仪用ingaas检测器(imspectorn17e,spectralimagingltd.,oulu,finland)测量。测量了所有120片叶子的高光谱图像。
(3)图像校正:在完成数据收集之后,获得包含光谱(λ)和空间(x,y)信息的3-d数据立方体。收集的原始图像信号是信号强度,而不是样品的反射光谱。因此,使用基于白色和暗图像参考的以下等式将原始图像校正为高光谱图像。
其中ir是原始高光谱图像,id是反射率接近0的暗图像,iw是白色图像,其基于聚四氟乙烯白板测量并且具有接近99%的反射率。为了捕捉暗图像,关闭灯并用不透明的盖子盖住镜头。暗参考也可以消除原始数据中的暗电流。暗图像和白图像的数据采集过程与样本的图像采集过程一致。校正后的图像用于后续计算,包括光谱提取,变量选择和模型建立。
(4)数据分析:手动在enviv4.6软件的感兴趣区域(roi)功能中对校正后图像进行识别样品的roi,获得叶脉、健康叶肉、轻微损伤叶肉和严重损伤叶肉以及幼虫的五类roi。每个roi都是一个样本,计算一个roi中所有像素的平均值以获得该roi(样本)的代表性光谱。将每个样品的代表性光谱组合成光谱矩阵(x),矩阵的每列是变量并代表一个波长。矩阵的每一行代表一个样本。有五类roi,即叶脉,健康叶肉,轻微损伤叶肉,严重损害叶肉和桑螟幼虫,使用主成分分析(pca)判别分析五类的可分性,结果如图3所示。使用自然数1,2,3,4,5来表示上述五个类别,将所有样品的类别编号组合成列向量(y)。本实施例中,矩阵(x)的列可见光范围数据有428个变量,近红外范围数据有256个变量。
使用全变量以及多变量分析建立矩阵x和向量y之间的定量关系模型。将所有样本随机分成一组校准(四分之三样本)和另一个独立的预测集(四分之一样本)。针对全变量用偏最小二乘判别分析(pls-da),最小二乘支持向量机(ls-svm)和线性判别分析(lda)算法分别建立了用于鉴定五类样品的分类模型。针对多变量分别使用连续投影算法(spa),非信息变量消除(uve),uve-spa和竞争自适应重加权抽样(cars)方法选择可以区分昆虫和叶片损害的波长,之后建立五类样品的分类模型。
其结果可见图4。根据模型结果对比,所有的模型都能在一定程度上实现识别五类roi,其中用于识别所有五个roi的最佳模型是基于可见范围数据的uve-spa-ls-svm模型,其具有97.30%的正确预测率(crp)值。说明采用可见光和近红外高光谱成像能较准确的识别鉴定出桑螟幼虫及其对桑叶损害,在农业检测植物病虫害上具有重要的推广价值。
以上所述仅为本发明的较佳实施举例,并不用于限制本发明,凡在本发明精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!