基于变步长最小均方误差自适应滤波器的基因识别系统的制作方法
本发明涉及生物信息学中对测序获得的dna序列进行可编码基因区域的识别和定位领域,具体涉及一种基于变步长最小均方误差(lms)自适应滤波器的基因识别系统。
背景技术:
现阶段基因识别的研究取得了很多优秀的成果,形成了许多成熟的预测系统,但这些系统主要都依托于一些经典的机器学习标记方法,如hmm和crf等,一般模型结构复杂,调节参数繁多,训练耗时且不易存储,同时系统特异性过强,不利于物种间的迁移通用。另外,针对短基因序列随机噪声大、特征信息稀少而识别率低等问题,也不能进行有效的抑制和解决。
为了克服上述种种问题,国内外研究者们在数字信号处理技术、基因序列长程相关性以及统计特征等方面都做了大量的研究和分析,取得了不错的研究成果。
技术实现要素:
本发明的目的在于提供一种基于变步长最小均方误差(lms)自适应滤波器的基因识别系统,提出了变步长lms自适应滤波器改进算法和多特征加权融合算法,并将二者集成到同一个基因识别系统中,进一步改善其识别性能。
为实现上述目的,本发明采取的技术方案为:
一种基于变步长最小均方误差自适应滤波器的基因识别系统,包括变步长lms自适应滤波器算法改进单元和基因结构多特征加权融合算法处理单元;所述的变步长lms自适应滤波器算法改进单元,用于通过变步长lms自适应改进算法对基因序列进行滤波处理,得到随机噪声较少、周期3行为较强的基因特征;所述的基因结构多特征加权融合算法处理单元,用于通过多特征加权融合策略,对基因序列进行特征提取,从而得到表达能力更强的特征向量。
研究表明,外显子编码区域对应的功率谱曲线在其π/3处通常可以观测到明显的波峰,而非编码区序列对应的功率谱曲线则观测不到如此波峰。假设系统输出为y(n),分别定义其一阶、二阶变量为
e1(n)=y(n)-y(n-1)
e2(n)=e1(n)-e1(n-1)
因为输出y(n)中的波峰部分通常表示算法预测的外显子区域,对算法识别性能而言至关重要,所以着重改善算法对波峰曲线上升部分的预测十分必要。在波峰曲线处于上升阶段时,可以适当减小步长因子,以提高算法识别的准确性;而在其他部分,可以适当增大步长因子,以促使系统快速收敛并具备更强的实时追踪能力。依据函数微分的几何定义,曲线从波谷上升到波峰的阶段其斜率必有e1(n)>0,而其曲线斜率的变化趋势可能通常要经历一个由小变大再变小的过程。在曲线斜率增长变快时,希望调整步长相应越小,而曲线斜率增长变慢,则调整步长相应逐渐增大。为了满足这一要求,提出系统输出反馈与步长因子新的函数关联关系,公式定义如下
其中,u0为初始步长,k为常数,且有1≤k<1/u0λmax,λmax为输入信号自相关矩阵最大的特征值。
另外,一般lms自适应滤波器基因识别算法研究中,通常定义a、c、g、t四种碱基对应的滤波器输出信号的平方和为y(n),有
y(n)=|ya(n)|2+|yc(n)|2+|yg(n)|2+|yt(n)|2,
在实际情况中,四种碱基对外显子的预测结果的影响可能不尽相同,造成
ya(n),yc(n),yg(n),yt(n)对总输出y(n)的贡献比率也应该各有不同。由仿真研究表明,碱基g、c的影响贡献率往往大于碱基a、t的影响,同时引入的噪声也相对更少。为了清晰表示出这种不同,提出根据基因序列gc含量与at含量的比值作为权重重新定义系统的输出,即
其中tb>0,分别对应四种碱基的加权系数,且默认有tg=tc=1,ta=tt=1。
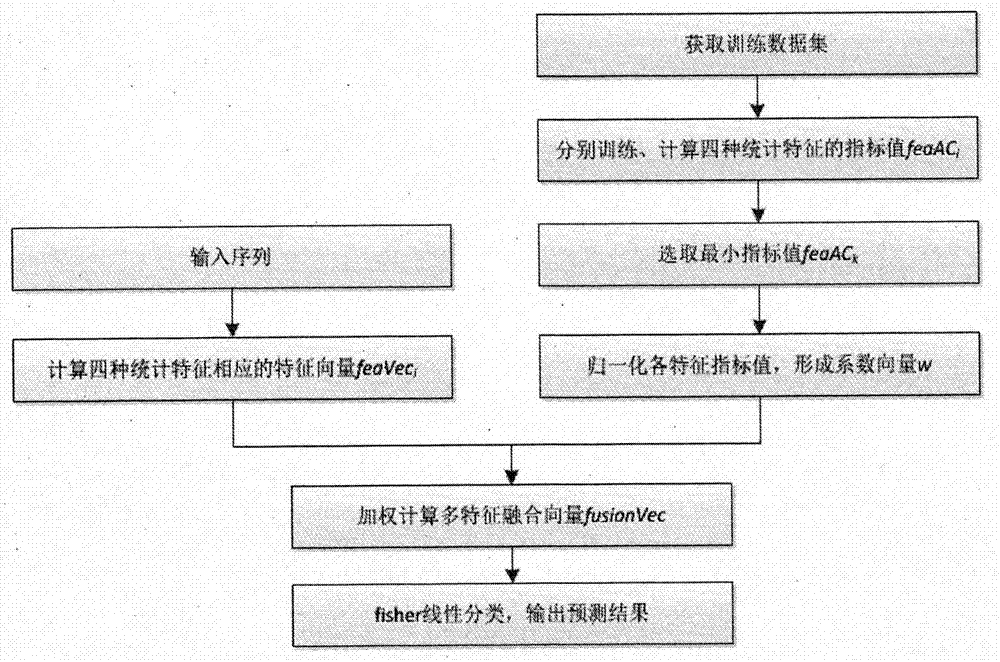
基因结构多特征加权融合算法处理单元的主要设计思想是依据单一特征在同一统计分析方法下,对基因编码区识别的表征能力的强弱或者说影响贡献的大小,即ac指标,对所选择的基因结构多种特征进行加权融合,形成可统一维度大小的复合特征向量。本系统主要涉及四种编码区特征,分别为:碱基组成成分、碱基位置相关性、密码子使用频率以及周期3行为。具体实现步骤如下:
1)、在同一统计分析方法下,分别单独使用上述提到的四种统计特征,训练判别模型并计算其对应的近似相关系数(ac)的算法评价指标,可记作feaaci,0≤i≤3。
2)、选取四种单一特征指标最小的值,令各个特征的ac指标值分别除以最小指标值做归一化处理,其结果值作为对应特征的权值,表达式记为
其中,feaaci分别对应上述四种统计特征的ac评估指标值,wi表示四种特征归一化后对应的权系数。
3)、将四种特征中的每一个特征量与其对应的权值相乘,组合成一个75维的多特征融合向量继续用于基因识别的算法研究,表达式可记为
其中,feaveci表示第i个特征向量,wi表示第i个特征对应的权系数,fusionvec表示加权融合后的特征向量。注意,这里所有的单一特征分量维数不尽相同,融合前,需为每一特征量设定固定填充位置,其余位置用0填充,统一扩充到75维。
本发明具有以下有益效果:
提出了变步长lms自适应滤波器改进算法和多特征加权融合算法,并将二者集成到同一个基因识别系统中,进一步改善其识别性能。
附图说明
图1为本发明实施例中变步长lms改进算法处理过程小结。
图2为本发明实施例中改进的多特征加权融合算法相应的处理框图。
图3为本发明实施例中结合上述两种算法改进的基因识别系统的单序列识别流程图。
具体实施方式
为了使本发明的目的及优点更加清楚明白,以下结合实施例对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
研究表明,外显子编码区域对应的功率谱曲线在其π/3处通常可以观测到明显的波峰,而非编码区序列对应的功率谱曲线则观测不到如此波峰。假设系统输出为y(n),分别定义其一阶、二阶变量为
e1(n)=y(n)-y(n-1);
e2(n)=e1(n)-e1(n-1);
因为输出y(n)中的波峰部分通常表示算法预测的外显子区域,对算法识别性能而言至关重要,所以着重改善算法对波峰曲线上升部分的预测十分必要。在波峰曲线处于上升阶段时,可以适当减小步长因子,以提高算法识别的准确性;而在其他部分,可以适当增大步长因子,以促使系统快速收敛并具备更强的实时追踪能力。依据函数微分的几何定义,曲线从波谷上升到波峰的阶段其斜率必有e1(n)>0,而其曲线斜率的变化趋势可能通常要经历一个由小变大再变小的过程。在曲线斜率增长变快时,希望调整步长相应越小,而曲线斜率增长变慢,则调整步长相应逐渐增大。为了满足这一要求,提出系统输出反馈与步长因子新的函数关联关系,公式定义如下
其中,u0为初始步长,k为常数,且有1≤k<1/u0λmax,λmax为输入信号自相关矩阵最大的特征值。
另外,一般lms自适应滤波器基因识别算法研究中,通常定义a、c、g、t四种碱基对应的滤波器输出信号的平方和为y(n),有
y(n)=|ya(n)|2+|yc(n)|2+|yg(n)|2+|yt(n)|2,
在实际情况中,四种碱基对外显子的预测结果的影响可能不尽相同,造成ya(n),yc(n),yg(n),yt(n)对总输出y(n)的贡献比率也应该各有不同。由仿真研究表明,碱基g、c的影响贡献率往往大于碱基a、t的影响,同时引入的噪声也相对更少。为了清晰表示出这种不同,提出根据基因序列gc含量与at含量的比值作为权重重新定义系统的输出,即
其中tb>0,分别对应四种碱基的加权系数,且默认有tg=tc=1,ta=tt=1。该部分完整的算法处理过程如附图1所示。
基因结构多特征加权融合算法处理单元的主要设计思想是依据单一特征在同一统计分析方法下,对基因编码区识别的表征能力的强弱或者说影响贡献的大小,即ac指标,对所选择的基因结构多种特征进行加权融合,形成可统一维度大小的复合特征向量。本系统主要涉及四种编码区特征,分别为:碱基组成成分、碱基位置相关性、密码子使用频率以及周期3行为。具体实现步骤如下:
1)、在同一统计分析方法下,分别单独使用上述提到的四种统计特征,训练判别模型并计算其对应的近似相关系数(ac)的算法评价指标,可记作feaaci,0≤i≤3。
2)、选取四种单一特征指标最小的值,令各个特征的ac指标值分别除以最小指标值做归一化处理,其结果值作为对应特征的权值,表达式记为
其中,feaaci分别对应上述四种统计特征的ac评估指标值,wi表示四种特征归一化后对应的权系数。
3)、将四种特征中的每一个特征量与其对应的权值相乘,组合成一个75维的多特征融合向量继续用于基因识别的算法研究,表达式可记为
其中,feaveci表示第i个特征向量,wi表示第i个特征对应的权系数,fusionvec表示加权融合后的特征向量。注意,这里所有的单一特征分量维数不尽相同,融合前,需为每一特征量设定固定填充位置,其余位置用0填充,统一扩充到75维。算法的相应处理过程如附图2所示。
实施例
以单条dna序列输入为例,默认系统已经预先从配置文件中加载了相关模型的训练参数值,其识别流程如图3所示。系统开始每次读取一条未知的输入序列,先统计其中a、c、g、t四种碱基的组成成份,计算gc含量与at含量的比值,更新各碱基对应滤波器输出的权值并保存,以便于在计算序列n/3处总功率谱或信噪比值时,对单一碱基的滤波器功率谱输出加权。再以单个碱基为步长滑动处理,每向前滑动一个碱基位置,判断是否抵达序列的终点,如果反馈结果为是,说明整条序列已经处理完备,系统会自动对其进行一些必要的后处理,再输出预测结果,并退出程序;如果反馈为否,会以该位置为中心,截取长度为m的序列片段(假如长度不足m,系统默认会以对称和随机原则生成相关碱基加以补充),可以用多线程同时计算上述四种特征向量,其中对于周期3特征向量的计算,从流程图可以看到,是由lms自适应滤波处理之后,再由第一步保存的滤波器输出权值加权求和而成(参照图1所示算法处理过程)。然后加权融合四种特征量(加权融合算法参照图2所示框图),获得对应的多维特征向量,计算fisher线性分类结果,与训练的阈值比较,如果大于,则相应位置碱基用1替代,否则用0替代。之后,向前滑动一个碱基位置,继续上述处理。
关于系统模型参数的训练,图3表示的系统中因为涉及到多特征融合和fisher线性判别分类器,需要训练保存的参数值比较多,如上述四种特征融合的权值参数,75维的fisher分类器的权值向量以及一个用于二值化预测结果的阈值参数。其训练策略采用5折交叉验证,即获取足够量的特定种类的基因标记序列数据集,随机均分为5个互不相交的子集,每次抽取一组子集作为测试数据集,其余混合用于训练模型。按标记抽取训练集中的编码区,作为外显子训练数据集,其他剩余的非编码部分统一作为内含子训练数据集。测试集也依此处理。依照图2所示的多特征提取和加权融合的算法处理过程,训练、保存模型参数,再在测试集上进行评估,记录其对应的近似相关系数指标ac值。5次循环操作之后,选择ac指标最大的那组,即平均测试误差最小的组作为系统的模型参数,写入系统配置文件。
关于fisher线性分类阈值模型的选择,采用已有的概率平均法,即分别求出训练集中所有外显子和内含子对应的特征评估值的平均值与标准偏差,如功率谱、信噪比等,按类别相乘再求和,并除以外显子和内含子的平均值之和,其比值即为所得到的阈值参数。
为了完整说明我们实现的基因识别系统,在上述两种改进单元之外,需要再引入两个处理单元,分别为基因数据预处理单元和预测结果输出单元。所述的基因数据预处理单元,主要是对获取的dna序列进行必要的预处理操作,如序列中含有未知碱基n或者n,则通过随机概率生成的方式,将其以a、c、g、t四种碱基的某一项作替换;如序列长度低于某一阈值,则通过不断复制拼接原序列的方式扩展序列长度,最后拼接进去的原序列,如果使总长度大于阈值,则多余的部分直接截断舍弃,如果总长度尚小于阈值,不足的部分则以随机概率生成的方式进行补充。然后将处理后的基因数据作为系统的输入数据。所述的预测结果输出单元,主要利用提取到的特征向量进行预测,并做必要的后处理操作,即先给定特定阈值,用0、1元素二值化预测结果(1表示是外显子编码区中碱基,0表示是非编码区中的碱基),再把预测结果中,两处连1之间相夹的长度小于30bp的连0重置为1,最后将长度小于30bp的连1重置为0,统计记录各个连1区间的起始和终止位置,作为匹配的外显子预测范围输出
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以作出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!