基因变异数据的GDS-Huffman压缩方法与流程
本发明涉及生命组学分析
技术领域:
,更具体地,涉及一种基因变异数据的gds-huffman压缩方法。
背景技术:
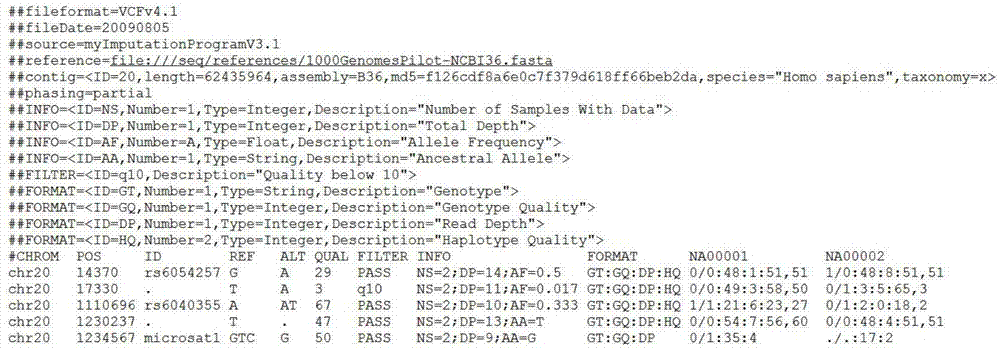
:随着生命组学分析样本数的增多,基因组和全外显子组分析产生的基因突变数据vcf文件越来越大。例如,在精准医疗计划中研究的样本数可达到上万个,这么多样本的全外显子分析产生的vcf可能达到tb量级,而大文件读写速度慢,处理起来是很困难的,严重降低了分析速度,成为计算瓶颈。研究新的基因突变数据vcf文件组织方法,降低文件大小是解决这个问题的有效途径。一个典型的vcf格式如图1所示。从图中的示例可以看到,vcf文件一般可以分成两部分内容:以“#”开头的注释部分;没有“#”开头的数据部分。注释部分解释了vcf文件产生的一些原始信息以及出现在数据部分中各个字段的含义。数据部分每行代表了一个变异位点的详细信息。表1对vcf文件数据部分各列含义进行了详细描述。数据部分的最后两列是两个样本的基因型信息,这些基因型信息是与格式(format)中定义的字段格式相对应的。各个字段含义在注释部分有相关说明。表1vcf数据部分各列描述gds格式是一种分层的数据格式,用来储存可伸缩的面向数组的数据集。它在处理较大的数据集上有优势,特别是针对比内存大得多的数据集。在此基础上开发了名为gdsfmt的r包。gdsfmt针对小于一个字节的的整数数据提供了高效的操作,因为一个二倍体基因型,通常占用不到一个字节的空间。gdsfmt采用的压缩为无损压缩,并提供高效的随机访问。利用r中的parallel包,实现了对gds文件的并行读取。gds利用分层结构储存vcf文件,针对vcf文件不同的区域采取相应的压缩方式进行储存。对于vcf文件中的dp,gq,pl等整型字段gds采用变长编码的方式进行编码储存,例如,对于一个-64到63的32位的整数,只需要用一个字节储存而不是四个字节。32位有符号整型变长编码的示意图如图2所示,bi表示每个字节向量中第i个位置的比特值,bi取0或1。b1表示符号位,每个字节的最高位表示下个字节是否需要,比如b8=0,那么说明这个整数只占据这一个字节,后面的字节是不需要的。48被编码为01100000,64被编码为1000000000000001。采取这种方式可以有效降低整数储存空间。对于vcf中的基因型(gt字段),gds采用一个2-bit的数组来储存,比如一个二倍体基因型的储存数组m2x3x4。m2x3x4是一个三维数组,第一维表示倍型,比如人类基因型就是2,第二维表示样本个数,第三维表示变异位点所占的空间。如果一个2bits不能表示一个位点的所有等位基因,那么就要进行扩充。针对gds文件,有很多r工具包对其进行后续的下游分析,比如snprelate,seqvartools和genesis,这些工具可以在bioconductor中获取。这些工具可以直接处理gds文件完成下游的统计分析,从而形成一套完整的从上游到下游的分析流程。gvcf格式是在多样本全外显子分析过程中常用的一种储存变异信息的格式,如果能将上述gds方法应用在gvcf上,就能有效降低中间文件大小,提高分析效率。而gds面向的是标准的vcf格式,它核心是对整数和基因型的编码。而gvcf文件中的基因型的分布是非常不均的,如基因型0/0占到98%。gds并未考虑基因型分布的特点。若根据基因型频率特点采用新的编码方式,将进一步提高压缩率。技术实现要素:本发明为解决现有技术没有考虑基因型分布而导致的gvcf文件的中间文件过大的技术缺陷,提供了一种基因变异数据的gds-huffman压缩方法。为实现以上发明目的,采用的技术方案是:一种基因变异数据的gds-huffman压缩方法,对于gvcf文件,基于gds压缩方法根据基因型频率采用huffman编码对gvcf文件中的基因型进行编码,而采用变长整型编码的方式对gvcf文件中的整型字段进行编码,得到压缩后的gds文件。与现有技术相比,本发明的有益效果是:本发明提供的gds-huffman压缩方法充分考虑了gvcf中基因型频率的特点,利用huffman编码基因型,更加高效地压缩gvcf文件。与传统的分析方法相比,利用gds-huffman压缩方法压缩gvcf文件,然后直接处理压缩后gds-huffman文件的方式大大降低了分析过程中中间文件的大小,解决了分析过程中大文件处理的问题。附图说明图1为vcf格式范例图。图2为32位有符号整型变长编码的示意图。图3为huffman编码树的示意图。图4为编码演示样例图。图5为基于gds-huffman的全外显子分析流程图。具体实施方式附图仅用于示例性说明,不能理解为对本专利的限制;以下结合附图和实施例对本发明做进一步的阐述。实施例1本实施例针对gvcf文件,基于gds压缩方法,利用基因型频率特点,采用huffman编码对基因型进行编码,从而更加高效地压缩gvcf文件。根据基因型频率构建的huffman编码树如图3所示。编码如表2所示。基因型将根据编码表进行编码。对于gvcf文件中的整型字段采用变长整型编码的方式进行编码。表2基因型huffman编码基因型编码0/011/1010/1001./.00001/00001使用上述方法对图4中的变异信息进行编码,结果为:gt:100101gq:000101000001010000010100dp:01000000011000001000000000000001本实施例将gds-huffman压缩方法应用在全外显子分析过程中,采用如图5所示的分析流程。对于产生的gvcf,首先采用gds-huffman压缩方法将其转换为gds格式的文件。重新实现genotypegvcfs和catvariants两个过程,直接处理gds格式的gvcf文件,产生各个染色体区域对应的gds格式文件,最终直接合并这些gds文件,产生最终结果。gds-huffman压缩方法以及genotypegvcfs和catvariants的实现都是采用c++和r语言混合编译的方式进行的,这里用到了rcpp包,它将这两种语言集成起来,提供相互调用的方法。最终将这些方法封装在r包中。实施例2表3是对gds-huffman压缩方法压缩性能的测试。该测试针对gvcf文件,原始数据来自于千人基因组项目(the1000genomesproject)中的全外显子测序数据,样本详细信息可以在千人基因组官网查询,这些数据是已经比对到参考基因组并且通过cram进行压缩的cram格式文件。首先通过samtools对cram文件进行预处理,得到原始序列的fastq格式文件,然后按照全外显子分析流程处理这些样本,得到每个样本的gvcf文件。将gvcf文件作为输入,使用gds-huffman压缩方法进行压缩。平均压缩率达到5.1%,压缩速率为4.1m/s。表3gds-huffman压缩性能测试基于gds-huffman的全外显子分析测试如表4所示,测试集包含的样本编号如表5所示。从图中可以看到当样本数较少,样本总量较少时,基于gds-huffman的工作流的时间比常规分析还要多,但随着样本数增多,处理文件的总量增大,常规分析由于io等问题,消耗时间会迅速增多,而基于gds-huffman的工作流由于大大降低了gvcf文件大小,并在这基础上处理文件,因此消耗时间比常规时间短。表4基于gds-huffman的全外显子分析测试表5测试集包含的样本编号显然,本发明的上述实施例仅仅是为清楚地说明本发明所作的举例,而并非是对本发明的实施方式的限定。对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动。这里无需也无法对所有的实施方式予以穷举。凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明权利要求的保护范围之内。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1