一种蛋白质功能注释方法及系统与流程
本发明涉及生物信息领域,特别是涉及一种蛋白质功能注释方法及系统。
背景技术:
在蛋白质鉴定和检测中,itraq(isobarictagforrelativeandabsolutequantitation)和tmt(tandemmasstags)技术是最常规有效的。和核酸测序不同的是,蛋白质测序需要有背景序列作为参考进行质谱图的匹配,这样的结果使每一个鉴定的蛋白序列都有明确来源。如果背景序列是公开数据库(ncbi,uniport,ensembl等)下载处理得到的序列,或者是已经做过功能注释的转录本预测序列,那么鉴定结果就有充足的先验知识背景。
在此背景下,常规的蛋白注释流程是借鉴成熟的核酸注释流程,其中包括go功能注释,kegg通路注释和cog/kog功能注释。
目前对于蛋白功能注释主要借鉴于包装核酸注释流程,其中一些重要的组成部件有blast2go,kobas等。
采用blast办法能提高序列之间比对的精确度,但是比对时间长,5000条序列与nr全库比对时间平均超过10小时;整套blast2go流程需要复杂的数据搜集和软硬件(比如关系型数据库mysql)配合,导致调试和更新数据的难度变大,部分公司和产品线的数据更新频率少于2次/年,导致蛋白注释的结果偏老旧。其中一个结果是,得到的鉴定有效的蛋白序列在最新的资料显示并不是相同的功能。
kobas和blast2go一样,需要复杂的安装调试;或者在线上运行,但对于批处理数据不适用(操作步骤多)。kobas的本地数据更新依赖于kegg的付费下载。
此外,学术界对于蛋白质的研究进展是日新月异的,用上几个月甚至前几年的数据进行蛋白质的功能注释,而不用最新,最前沿的数据库来支持功能注释,可能导致研究者得不到精确的实验目标。现有的蛋白功能注释工具在注释用数据库的更新上尚有不足。
而kegg数据库支持对物种进行细分,数据库下有多个子数据库。在物种区分方面,不区分物种的通路id有ko和map,区分物种的有人hsa,小鼠mmu等;在keggontology方面,不区分物种的有k和人hsa,小鼠mmu等。区分物种导致流程复杂度增大。
技术实现要素:
为了克服现有技术的缺陷,本发明提供了一种蛋白质功能注释方法及系统。
本发明首先提供了一种蛋白质功能注释方法,包括下列步骤:
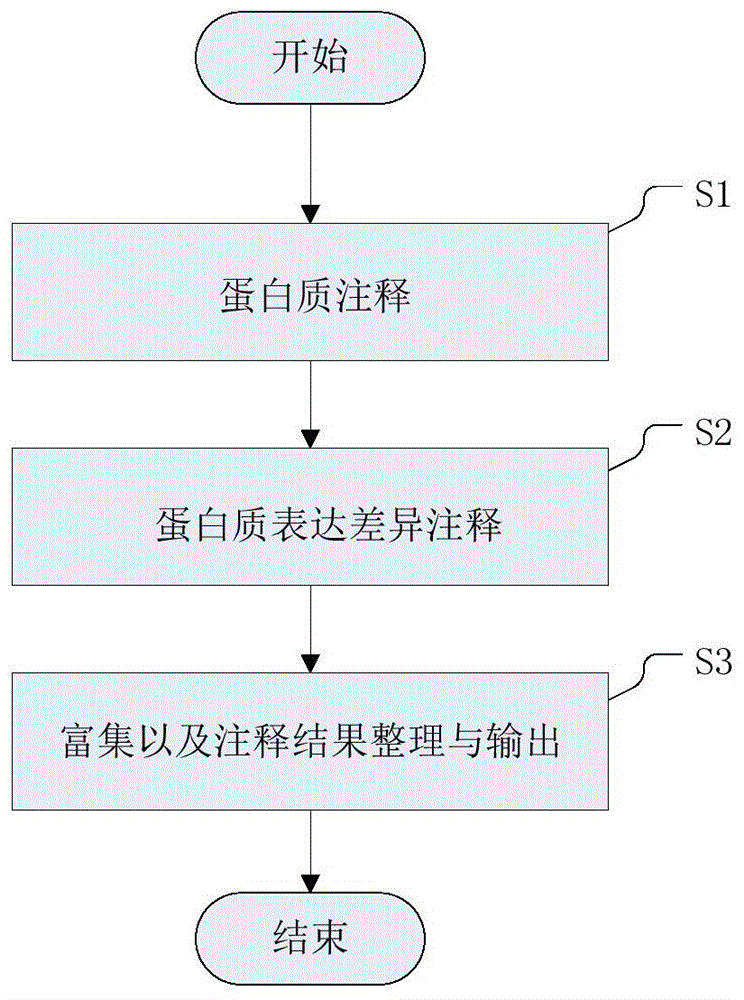
步骤s1、蛋白质注释:结合序列比对与id匹配,完成go功能注释及kegg通路注释;
步骤s2、蛋白质表达差异注释;
步骤s3、富集以及注释结果整理与输出。
其中,所述go功能注释的步骤包括:
1)提供nr数据库、pir数据库及go数据库;
2)序列比对:将待注释蛋白的序列利用比对工具diamond比对nr数据库,得到nr注释;利用nr注释中的相关信息,通过与pir数据库的对应匹配,获得待注释蛋白序列的序列数据编号与go的id的匹配关系;将待注释蛋白的序列比对go数据库,获得待注释蛋白序列的序列数据编号与go的id的匹配关系;
3)id匹配:将待注释蛋白序列的序列数据编号与uniprot数据库和/或pir数据库直接进行id匹配,得到待注释蛋白序列的序列数据编号与go的id的匹配关系;
4)将序列比对结果与id匹配结果合并,根据对应待注释蛋白序列的goid号,从go数据库中获得对应的go功能注释信息并提供go功能注释结果。
所述kegg通路注释步骤包括:
a)提供kegg比对数据库,所述kegg比对数据库区分物种,每一物种至少包含通路分类和描述信息表、序列数据库、通路图片及通路图信息文件;提供uniprot数据库和/或pir数据库;
b)id匹配:将待注释蛋白序列的序列数据编号与uniprot数据库直接进行id匹配,得到待注释蛋白序列的序列数据编号与kegg的id的匹配关系;
c)序列比对:将待注释蛋白的序列利用比对工具blast或比对工具diamond比对kegg比对数据库,得到待注释蛋白序列的序列数据编号与keggid的匹配关系;
d)将序列比对结果与id匹配结果合并,根据kegg的id号从kegg比对数据库中获得对应的kegg功能注释信息。
在较佳的实施方式中,go功能注释及kegg通路注释所用的数据库均定期(如每月)更新并留存于服务器。
在较佳的实施方式中,本发明还进一步增设了筛选参数,如用于筛选表达差异蛋白的差异倍数和/或显著性、筛选差异富集柱状图个数等。
本发明还提供了一种蛋白质功能注释系统,包括:
蛋白质注释模块,用于结合序列比对与id匹配,完成go功能注释及kegg通路注释;
蛋白质表达差异注释模块,用于利用蛋白质注释结果,获得差异表达的蛋白质对应的go功能注释及kegg通路注释;
富集以及注释结果整理与输出模块,用于对蛋白质表达差异注释结果进行富集以及整理,并输出报告。
进一步的,本发明还提供了一种服务终端,包括:
通信器,用于与外部通信;
存储器,存储有计算机程序;
处理器,用于运行所述计算机程序以实现前述蛋白质功能注释方法。
进一步的,本发明还提供了一种计算机可读存储介质,存储有计算机程序,用于运行以实现前述蛋白质功能注释方法。
采用本发明的蛋白质功能注释方法及系统相比采用现有nr全库blast的方法可以减少注释时间。由于本发明注释时采用了序列比对与id匹配相结合的方式,因此在不损失准确度的前提下,可让更多的序列匹配数据库信息,增加了数据的覆盖度。多个数据库同时使用,增加了鉴定序列注释的广度(在保证准确度的前提下,在go和kegg注释方面有更多的蛋白有功能注释)。在优选的方案中,由于本发明比对用数据库更新周期短,因此可减少已经和最新数据冲突的注释,提高数据的保鲜度。利用本发明的蛋白质功能注释系统可减少操作,以降低错误率和提高效率。相比于传统借鉴核酸注释流程,本发明的流程每月下载最新数据保证注释数据保持和主流数据库同步更新。本发明还可减少使用人员准备数据和整理报告的时间,提高效率。
附图说明
图1显示为本发明实施例中的蛋白质功能注释流程图
图2显示为本发明实施例中的go和kegg注释信息转换流程图
图3显示为本发明实施例中的通路分类和描述信息表
图4显示为本发明实施例中的样本go二级上下调蛋白数图
图5显示为本发明实施例中的样本差异蛋白go二级富集柱状图
图6显示为本发明实施例中的样本上下调蛋白kegg通路注释图
图7显示为本发明实施例中的样本差异蛋白kegg通路富集柱状图
图8显示为本发明实施例中的蛋白质功能注释系统的模块示意图
图9显示为本发明优选实施例中的蛋白质功能注释系统的模块示意图
图10显示为本发明实施例中的服务终端的结构示意图
具体实施方式
以下通过特定的具体实例说明本发明的实施方式,本领域技术人员可由本说明书所揭露的内容轻易地了解本发明的其他优点与功效。本发明还可以通过另外不同的具体实施方式加以实施或应用,本说明书中的各项细节也可以基于不同观点与应用,在没有背离本发明的精神下进行各种修饰或改变。
请参阅图1,该实施例中的蛋白质功能注释方法包括下列步骤:
步骤s1、蛋白质注释:结合序列比对与id匹配,完成go功能注释及kegg通路注释;
步骤s2、蛋白质表达差异注释;
步骤s3、富集以及注释结果整理与输出。
本发明的蛋白质功能注释方法,特别适用于对蛋白质谱搜库结果进行蛋白质功能注释,尤其适用于itraq|tmt质谱搜库结果。蛋白质谱搜库结果的获得属于现有技术,例如可以采用itraq(isobarictagsforrelativeandabsolutequantitation)技术或tmt(tandemmasstag)技术结合lc-ms蛋白质组分析技术获得。蛋白质谱搜库结果中的信息包括:蛋白的氨基酸序列、蛋白的序列数据编号(accession)、蛋白相对丰度、差异倍数(foldchange)、可信度(t-testpvalue),蛋白质谱搜库结果可呈现为文本模式,例如为txt文件,第一列为鉴定蛋白accession,之后的列为不同样本中该蛋白的表达量。
在优选的实施方式中,蛋白质功能注释方法中的go功能注释如图2所示,包括:
1)提供nr数据库、pir数据库、go数据库,可选择地提供uniprot数据库或ncbi数据库;
2)序列比对:将待注释蛋白的序列利用比对工具diamond比对nr数据库,得到nr注释;利用nr注释中的相关信息,通过与pir数据库的对应匹配,获得待注释蛋白序列的序列数据编号与go的id的匹配关系;将待注释蛋白的序列比对go序列数据库,获得待注释蛋白序列的序列数据编号与go的id的匹配关系;
3)id匹配:将待注释蛋白序列的序列数据编号与uniprot数据库和/或pir数据库直接进行id匹配,得到待注释蛋白序列的序列数据编号与go的id的匹配关系;
4)将序列比对结果与id匹配结果合并,根据对应待注释蛋白序列的goid号,从go数据库中获得对应的go功能注释信息并提供go功能注释结果。
nr数据库、pir数据库、go数据库(如go序列数据(go_weekly-seqdb.fasta))及uniprot数据库均为现有的在线数据库,可在线下载获取。
所述“可选择地”含义为可以有,也可以没有。
在go功能注释的序列比对中,将待注释蛋白的序列利用比对工具diamond比对nr数据库中的序列信息,可获得对应的nr注释,nr注释包括pir数据库相关信息(在pir提供的数据表格中,每行表示某蛋白在不同的数据库中的不同id),所述pir数据库相关信息具体可为refseq信息,根据pir数据库中与相应refseq信息对应的goid信息,获得待注释蛋白序列的序列数据编号与go的id的匹配关系。序列比对结果优选为tab格式,以方便数据后期检查。
可采用blastp将待注释蛋白的序列比对go序列数据库,获得待注释蛋白序列的序列数据编号与go的id的匹配关系。
uniprot、pir中含有序列数据编号信息,在go功能注释的id匹配中,可通过字符匹配实现待注释蛋白序列的序列数据编号与数据库uniprot和/或数据库pir的id匹配。例如,可利用uniprot网站提供的idmapping窗口实现待注释蛋白序列的序列数据编号与数据库uniprot和/或数据库pir的id匹配。
从ncbi数据库中可下载nr全序列数据库,通过序列比对步骤获得待注释蛋白序列的序列数据编号与go的id的匹配关系。
序列比对结果与id匹配结果可合并生成go.list文件,根据对应待注释蛋白序列的goid号,获得对应的go功能注释信息。
go功能注释信息包括:细胞组分(cellularcomponent,cc)、分子功能(molecularfunction,mf)及生物学过程(biogicalprocess,bp)。go数据库包含goid号及对应的go功能注释信息,在获得待注释蛋白的goid号后,即可利用goid在go数据库中获得对应的go功能注释信息。
go功能注释结果包括但不限于:go二级分类柱形图,go二级分类饼图,二级三级四级分类九饼图。在优选的实施方式中,除了提供golevel2,3,4的三大功能分类,还提供各个level的bp、mf、cc的饼图(数值形式和百分比形式),以丰富可视化结果。饼图可采用python-metplotlib,r-ggplot,excel等工具获得。
在优选的实施方式中,go功能注释用的nr、pir、uniprot及go数据库定期更新并留存于服务器,例如每月更新。每次更新后的数据库均留存于服务器,以便使用者根据需求选取。
本发明流程中的go注释相对于blast2go,一方面减少了软件环境配置时间,第二方面本发明披露的利用diamond的序列比对流程,相比blast,完成同样的比对任务时,所耗时间仅为1/3。
在优选的实施方式中,摒弃了常规kegg通路注释用的kobas注释,对kegg通路注释进行了优化,优选的kegg通路注释如图2所示包括:
1)提供kegg比对数据库,所述kegg比对数据库区分物种,每一物种至少包含通路分类和描述信息表、序列数据库、通路图片及通路图信息文件;提供uniprot数据库和/或pir数据库;
2)id匹配:将待注释蛋白序列的序列数据编号与uniprot数据库直接进行id匹配,得到待注释蛋白序列的序列数据编号与kegg的id的匹配关系;
3)序列比对:将待注释蛋白的序列利用比对工具blast或比对工具diamond比对kegg比对数据库,得到待注释蛋白序列的序列数据编号与keggid的匹配关系;
4)将序列比对结果与id匹配结果合并,根据kegg的id号从kegg比对数据库中获得对应的kegg功能注释信息。
在另一实施方式中,步骤2)和3)中,首先进行id匹配,无法通过id匹配获得keggid的,再进行序列比对获得keggid号。在kegg通路注释的id匹配中,同样可通过字符匹配实现待注释蛋白序列的序列数据编号与数据库uniprot的id匹配。
所述的keggid号至少包括:蛋白ko编号及通路ko编号。
具体的,kegg比对数据库可采用下列步骤构建:
a)获取kegg物种通路信息、kegg物种通路图片及kegg物种通路图片对应的网页信息、kegg序列信息;
b)利用kegg物种通路信息及kegg物种通路图片对应的网页信息构建各物种的通路分类和描述信息表;
c)区分物种,合并同一物种下的kegg序列信息,得到kegg对物种的序列数据库;
d)区分物种,对通路分类和描述信息表、kegg物种通路图片及kegg物种通路图片对应的网页信息、kegg对物种的序列数据库整合,获得kegg比对数据库。
在进一步优选的实施方式中,所述kegg物种通路信息、kegg物种通路图片及kegg物种通路图片对应的网页信息、kegg序列信息定期更新,优选每月更新。每次更新后获得的比对数据库均留存于服务器,对应新更新时间新建文件夹,原来文件夹不删除,以便使用者根据需求选取。
所述kegg物种通路信息、kegg物种通路图片及kegg物种通路图片对应的网页信息均可从通路官方网址(https://www.kegg.jp)获得、kegg序列信息可通过keggapi下载工具(http://rest.kegg.jp)获取。kegg物种通路信息包含通路大类(如metabolism)、通路小类(如carbohydratemetabolism)、通路编号(如hsa:00010)及通路描述(如glycolysis/gluconeogenesis);通路图片对应的网页信息包括通路包含的蛋白ko编号信息(如has:127等),通路包含的蛋白ko编号信息来自通路图片对应的源代码文件;kegg序列信息包含蛋白ko(keggorthology)编号及对应的序列信息。所述通路分类和描述信息表如图3所示,至少包括:通路编号及与该通路编号对应的通路大类、通路小类、通路描述及通路包含的各蛋白ko编号信息。kegg比对数据库可为文本文件。
在kegg功能注释的序列比对中,可将待注释蛋白的序列利用比对工具blast或比对工具diamond比对kegg比对数据库中的序列,进而根据kegg中序列比对、id匹配对应关系,获得待注释蛋白序列的序列数据编号与keggid的匹配关系。
kegg功能注释的序列比对结果与id匹配结果可合并生成pathway.txt文件,进而根据kegg的id获得对应的kegg功能注释信息。
在优选的实施方式中,可通过python对位图的像素点rgb值进行修改,改变kegg图片中框,点,线的颜色。
kegg功能注释信息包括:蛋白对应的keggid以及蛋白参与的kegg通路;注释到的每个kegg通路图的图片png和html;注释到kegg通路的统计(含有蛋白数量的前20个通路)和分类。
优选的kegg注释流程相对于kobas,一方面减少了软件环境配置时间,二方面使得单个物种分析更加具体。在此基础上,与kegg同步更新的数据使得ko和accession的比对结果更加前卫(表现为就是当前的数据,不是上个月,半年前的数据),通路图在单个物种的研究也更加具体,避免了部分通路和部分蛋白实际不存在于某具体物种、部分蛋白实际不属于某具体通路而造成通路图被注释的情况。
蛋白质功能注释方法中,蛋白质表达差异注释为:在蛋白质谱搜库结果中筛选出表达差异蛋白,利用步骤s1获得的蛋白质注释结果,获得差异表达的蛋白质对应的go功能注释及kegg通路注释。
在蛋白质谱搜库结果中筛选表达差异蛋白时,可通过设置筛选参数来筛选,可设筛选参数包括但不限于:差异倍数>1.2或<0.83,显著性pvalue<0.05等
筛选出的差异蛋白,可采用python和r来实现蛋白质表达差异注释。
本实施例的富集以及注释结果可选自:go上下调蛋白表达柱状图和表格;kegg上下调表达通路图和表格;go差异富集柱状图和表格;kegg差异富集图和表格。上述富集及注释结果均属于现有技术,例如可采用goatools,kobas等完成上述富集以及注释结果整理。
在优选的实施方式中,只在绘制富集柱状图中,设置筛选差异富集柱状图个数功能,富集柱少于10不绘制,多余60则取top60进行绘制。
在优选的实施方式中,如图4-图7,同时提供差异蛋白上调、下调、上下调结合的go和kegg的注释和富集,方便查阅分析结果。
在优选的实施方式中,还可新增多个可视化图标,丰富表现方式,使得注释结果表现力加强。
常规核酸功能注释流程一般调用goatools进行富集,在优选的实施方式中,利用python语言提供统计计算函数,完善差异显著性pvalue校验(确认为fdr),完善差异富集的超几何分布计算,使用python的科学计算包scipy提供的scipy.stats.hypergeom.pmf进行超几何分布/富集率计算。此举优点在于客户可根据富集率和显著性来挑选在其研究中可能有重要生物学功能的通路。
富集以及注释结果可以结题报告输出。结题报告可以是word版也可为pdf版,在优选的实施方式中,注释运行结束后生成office-docx的结题报告,word版结题报告的生成能减少人力手动重复填充,统一格式,相比pdf版更方便后期修改增删。
请进一步参阅图8,本实施例还提供了一种蛋白质功能注释系统,由于本实施例中的系统与前述方法实施例的原理基本相同,实施例间的特征可以互用,因此,本实施例中仅对具体实现的部分技术细节进行阐述,而不全部重复赘述。
本实施例提供的蛋白质功能注释系统包括:
蛋白质注释模块101,用于结合序列比对与id匹配,完成go功能注释及kegg通路注释;
蛋白质表达差异注释模块102,用于利用蛋白质注释结果,获得差异表达的蛋白质对应的go功能注释及kegg通路注释;
富集以及注释结果整理与输出模块103,用于对蛋白质表达差异注释结果进行富集以及整理,并输出报告。
进一步的,如图9所示,蛋白质功能注释系统还包括数据库更新及存储模块104,用于定期下载数据库相关信息并存储数据库以用于蛋白质功能注释分析。
下载的数据库相关信息选自但不限于:nr、pir、uniprot、go数据库。
在优选的实施方式中,定期下载数据库相关信息的频率优选为每月下载,每次获得的新数据库均留存于服务器,以便使用者根据需求选取。
在优选的实施方式中,所述数据库更新及存储模块还用于kegg比对数据库的构建及存储。
为了构建kegg比对数据库,定期下载数据库相关信息还包括:kegg物种通路信息、kegg物种通路图片及kegg物种通路图片对应的网页信息、kegg序列信息。在优选的实施方式中,上述定期下载数据库相关信息的频率优选为每月下载。每次更新后构建获得的比对数据库均留存于服务器,对应新更新时间新建文件夹,原来文件夹不删除,以便使用者根据需求选取。
蛋白质功能注释系统的蛋白质注释模块包括:go功能注释子模块及kegg通路注释子模块。
其中,go功能注释子模块用于执行下列步骤:
1)调取nr数据库、pir数据库、go数据库,可选择地调取uniprot数据库或ncbi数据库;
2)序列比对:将待注释蛋白的序列利用比对工具diamond比对nr数据库,得到nr注释;利用nr注释中的相关信息,通过与pir数据库的对应匹配,获得待注释蛋白序列的序列数据编号与go的id的匹配关系;将待注释蛋白的序列比对go序列数据库,获得待注释蛋白序列的序列数据编号与go的id的匹配关系;
3)id匹配:将待注释蛋白序列的序列数据编号与uniprot数据库和/或pir数据库直接进行id匹配,得到待注释蛋白序列的序列数据编号与go的id的匹配关系;
4)将序列比对结果与id匹配结果合并,根据对应待注释蛋白序列的goid号,从go数据库中获得对应的go功能注释信息并提供go功能注释结果。
在优选的实施方式中,kegg通路注释子模块用于执行下列步骤:
1)调取kegg比对数据库,所述kegg比对数据库区分物种,每一物种至少包含通路分类和描述信息表、序列数据库、通路图片及通路图信息文件;调取uniprot数据库和/或pir数据库;
2)id匹配:将待注释蛋白序列的序列数据编号与uniprot数据库直接进行id匹配,得到待注释蛋白序列的序列数据编号与kegg的id的匹配关系;
3)序列比对:将待注释蛋白的序列利用比对工具blast或比对工具diamond比对kegg比对数据库,得到待注释蛋白序列的序列数据编号与keggid的匹配关系;
4)将序列比对结果与id匹配结果合并,根据kegg的id号从kegg比对数据库中获得对应的kegg功能注释信息。
在较佳的实施方式中,步骤2)和3)中,首先进行id匹配,无法通过id匹配获得keggid的,再进行序列比对获得keggid号。
在优选的实施方式中,kegg通路注释子模块还用于kegg图片色彩编辑。具体的,可根据来自用户端的参数设置对位图的像素点rgb值进行修改,改变kegg图片中框,点,线的颜色。可利用python对位图的像素点rgb值进行修改。
蛋白质功能注释系统的蛋白质表达差异注释模块用于在蛋白质谱搜库结果中筛选出表达差异蛋白,利用蛋白质注释结果,获得差异表达的蛋白质对应的go功能注释及kegg通路注释。对于筛选出的差异蛋白,可采用python和r来实现蛋白质表达差异注释。
在优选的实施方式中,蛋白质表达差异注释模块根据用户端设置的表达差异蛋白筛选参数筛选出表达差异蛋白。对应的,用户端可设筛选参数包括但不限于:差异倍数>1.2或<0.83,显著性pvalue<0.05等。
蛋白质功能注释系统的富集以及注释结果整理与输出模块用于富集以及注释结果整理与输出。
富集以及注释结果可选自:go上下调蛋白表达柱状图和表格;kegg上下调表达通路图和表格;go差异富集柱状图和表格;kegg差异富集图和表格。
在优选的实施方式中,富集以及注释结果整理与输出模块根据用户端设置的差异富集柱状图个数绘制富集柱状图。对应的,用户端可设参数包括筛选差异富集柱状图个数(即筛选差异富集柱状图中富集柱个数)。例如,可以设置富集柱少于10不绘制,多余60则取top60进行绘制。
需要说明的是,应理解以上系统的各个模块的划分仅仅是一种逻辑功能的划分,实际实现时可以全部或部分集成到一个物理实体上,也可以物理上分开。这些模块可以全部以软件通过处理元件调用的形式实现;也可以全部以硬件的形式实现;还可以部分模块通过处理元件调用软件的形式实现,部分模块通过硬件的形式实现。例如,蛋白质注释模块可以为单独设立的处理元件,也可以集成在某一个芯片中实现,此外,也可以以程序代码的形式存储于存储器中,由某一个处理元件调用并执行以上蛋白质注释模块的功能。其它模块的实现与之类似。此外这些模块全部或部分可以集成在一起,也可以独立实现。这里所述的处理元件可以是一种集成电路,具有信号的处理能力。在实现过程中,上述方法的各步骤或以上各个模块可以通过处理器元件中的硬件的集成逻辑电路或者软件形式的指令完成。
例如,以上这些模块可以是被配置成实施以上方法的一个或多个集成电路,例如:一个或多个特定集成电路(applicationspecificintegratedcircuit,简称asic),或,一个或多个微处理器(digitalsingnalprocessor,简称dsp),或,一个或者多个现场可编程门阵列(fieldprogrammablegatearray,简称fpga)等。再如,当以上某个模块通过处理元件调度程序代码的形式实现时,该处理元件可以是通用处理器,例如中央处理器(centralprocessingunit,简称cpu)或其它可以调用程序代码的处理器。再如,这些模块可以集成在一起,以片上系统(system-on-a-chip,简称soc)的形式实现。
如图10所示,展示一种服务终端的结构示意图。
所述服务终端200包括:处理器201、存储器202、及通信器203;所述通信器203,用于与外部通信;所述存储器202存储有计算机程序;所述处理器201,用于运行所述计算机程序以实现所述的蛋白质功能注释方法。
所述服务终端200可以通过其通信器203与具备网络通信能力的用户终端通信,从而提供蛋白质功能注释服务。
在图10实施例中的存储器202,可能包括但不限于高速随机存取存储器、非易失性存储器。例如一个或多个磁盘存储设备、闪存设备或其他非易失性固态存储设备;在图10实施例中的处理器201,可能包括但不限于中央处理器(centralprocessingunit,简称cpu)、网络处理器(networkprocessor,简称np)等;还可以是数字信号处理器(digitalsignalprocessing,简称dsp)、专用集成电路(applicationspecificintegratedcircuit,简称asic)、现场可编程门阵列(field-programmablegatearray,简称fpga)或者其他可编程逻辑器件、分立门或者晶体管逻辑器件、分立硬件组件。
在图10实施例中的通信器203,可以是有线或无线制式的网络通信电路模块。
在本发明的一些实施例中,本发明还可提供计算机可读存储介质,存储有计算机程序,用于运行以实现所述蛋白质功能注释方法。所述计算机可读存储介质可包括,但不限于,软盘、光盘、cd-rom(只读光盘存储器)、磁光盘、rom(只读存储器)、ram(随机存取存储器)、eprom(可擦除可编程只读存储器)、eeprom(电可擦除可编程只读存储器)、磁卡或光卡、闪存、或适于存储机器可执行指令的其他类型的介质/机器可读介质。所述计算机可读存储介质可以是未接入计算机设备的产品,也可以是已接入计算机设备使用的部件。
在具体实现上,所述计算机程序为执行特定任务或实现特定抽象数据类型的例程、程序、对象、组件、数据结构等等。
上述实施例仅例示性说明本发明的原理及其功效,而非用于限制本发明。任何熟悉此技术的人士皆可在不违背本发明的精神及范畴下,对上述实施例进行修饰或改变。因此,举凡所属技术领域中具有通常知识者在未脱离本发明所揭示的精神与技术思想下所完成的一切等效修饰或改变,仍应由本发明的权利要求所涵盖。
- 还没有人留言评论。精彩留言会获得点赞!