基于深度稀疏表示网络的DNA结合蛋白识别方法
基于深度稀疏表示网络的dna结合蛋白识别方法
技术领域
1.本发明涉及一种基于深度稀疏表示网络的dna结合蛋白识别方法。
背景技术:
2.dna结合蛋白是一种能够与dna结合和交互的特殊蛋白质。dna结合蛋白参与到许多生物进程中,例如:特异性核苷酸的鉴定,转录调控和基因表达调控。同时,dna结合蛋白是抗癌药物、抗生素和类固醇的重要成分,在抗癌药物的研究和遗传疾病的治疗中发挥着重要作用。早期的dna结合蛋白识别方法一般是生物实验方法,比如过滤器结合法,基因分析法,染色质免疫共沉淀技术和x-射线结晶法。生物实验方法既耗时又耗力,不能满足大规模蛋白质序列检测的需要。
3.现有技术中常见的检测方法为科学计算方法,如专利号202111056316.9所述的基于xgboost算法的dna结合蛋白识别研究方法、系统、存储介质及设备。方法过程:获取处理的dna结合蛋白特征数据集;采用不同的提取算法提取dna结合蛋白数据集的特征数据;将不同的特征提取算法提取的序列特征拼接起来,得到拼接后的特征矩阵;然后对生成的特征矩阵进行规范化处理;使用mrmd算法矩阵进行降维处理;使用xgboost算法构建并训练dna结合蛋白识别分类器模型。
4.这种方法各个步骤的训练目标不一致,与宏观目标存在偏差,这样训练出来的模型很难达到最优的结果;且每一个步骤都有误差,前一个步骤的误差会影响到下一步骤训练的结果,误差的积累最终导致最终结果很差。
技术实现要素:
5.本发明要解决的技术问题是一种基于深度稀疏表示网络的dna结合蛋白识别方法。
6.为了解决上述技术问题,本发明采用的技术方案是:一种基于深度稀疏表示网络的dna结合蛋白识别方法,包括以下具体步骤:s1,获取dna结合蛋白序列数据集,所述dna结合蛋白序列数据集分为训练集和测试集;s2,采用psl-blast软件计算所述dna结合蛋白序列数据集内所有序列的特异性打分矩阵;s3,将所有的所述特异性打分矩阵分别填充或裁剪成相同尺寸的新特异性打分矩阵;s4,采用深度稀疏表示网络构建并训练dna结合蛋白识别分类器模型;s5,将步骤s3中的所述新特异性打分矩阵输入所述dna结合蛋白识别分类器模型内,完成对所述dna结合蛋白序列的识别。构建并训练出的dna结合蛋白识别分类器模型是一种端到端的网络,能够明显的提高预测精度,误差较小,采用卷积自编码器可以鲁棒地学习特异性打分矩阵的潜在特征,并通过稀疏表示层进行分类,提高了模型的泛化能力。
7.优选的,步骤s4具体包括:s41,将步骤s1中的所述训练集和所述测试集进行行拼接得到拼接数据集其中x
train
为所述训练集,所述训练集x
test
为所述测试集,所述测试集其中,d0表示所述训练集和所述测
试集的维度,n表示训练集样本个数,m表示测试集样本个数;s42,将所述拼接数据集x输入包含编码器、解码器和稀疏表示层的深度稀疏表示网络模型中,所述拼接数据集x经过所述编码器输出编码z,z=[z
train
,z
test
],其中,z
train
表示训练集编码,z
test
表示测试集编码,所述编码z经过所述稀疏表示层得到其中即可定义in是单位矩阵,0n×m,0m均为0矩阵,a为稀疏表示矩阵,通过所述深度稀疏表示网络的目标函数其中,是所述解码器的输出,表示训练集解码,表示测试集解码,所述λ0=0.1和λ1=0.1,λ0和λ1均为正则化参数,即可计算出所述稀疏表示矩阵a;s43,取所述测试集x
test
中任意一个测试样本其对应的测试样本编码为其对应的在所述稀疏表示矩阵a中的相关稀疏编码列为αi,定义新向量中的非0元素是和αi相关的k类元素,任意一个测试样本的分类公式为完成对所述dna结合蛋白识别分类器模型的构建和训练。
[0008]
优选的,步骤s2中,采用psi-blast软件生成特异性打分矩阵矩阵,生成的命令为blast+options:-num_iterations 3-db nr-inclusion_ethresh0.001。
[0009]
优选的,步骤s2中,所述特异性打分矩阵矩阵为pssm,具体公式为诶其中,每个元素表示特定位置特定序列被取代的可能性,l为蛋白质的序列长度。
[0010]
优选的,所述步骤s3具体为:当所述序列的pssm行数大于70时,对底部多出来的行裁剪掉;当所述序列的pssm行数小于70时,对底部缺少的的行用0补充。
[0011]
由于上述技术方案运用,本发明与现有技术相比具有下列优点:本发明提供一种基于深度稀疏表示网络的dna结合蛋白识别方法,构建并训练出的dna结合蛋白识别分类器模型是一种端到端的网络,能够明显的提高预测精度,误差较小,采用卷积自编码器可以鲁棒地学习特异性打分矩阵的潜在特征,并通过稀疏表示层进行分类,提高了模型的泛化能力。
附图说明
[0012]
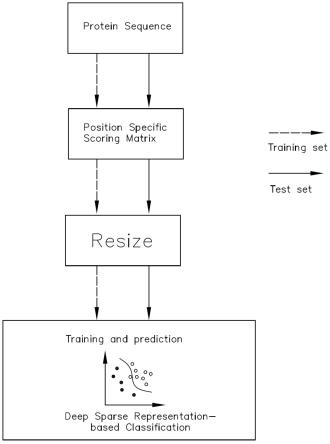
图1为本发明流程步骤示意图;
[0013]
图2为深度稀疏表示网络结构示意图;
具体实施方式
[0014]
现在将参考附图来详细描述本发明的示例性实施方式。应当理解,附图中示出和描述的实施方式仅仅是示例性的,意在阐释本发明的原理和精神,而并非限制本发明的范围。
[0015]
本发明提供了如各图所示的基于深度稀疏表示网络的dna结合蛋白识别方法,包括以下具体步骤:
[0016]
步骤s1,获取dna结合蛋白序列数据集,所述dna结合蛋白序列数据集分为训练集和测试集。
[0017]
本实施例中,dna结合蛋白序列数据集是从protein data bank中下载到的pdb186,pdb1075,pdb2272和pdb14189数据集,其中pdb186中有93个dna结合蛋白数量,93个非dna结合蛋白数量;pdb1075中有525个dna结合蛋白数量,550个非dna结合蛋白数量;pdb2272中有7192个dna结合蛋白数量,7060个非dna结合蛋白数量;pdb14189中有1153个dna结合蛋白数量,1119个非dna结合蛋白数量,其中pdb186和pdb2272为测试集,pdb1075和pdb14189为训练集。
[0018]
步骤s2,采用psl-blast软件计算所述dna结合蛋白序列数据集内所有序列的特异性打分矩阵,生成的命令为blast+options:-num_iterations 3-dbnr-inclusion_ethresh 0.001,特异性打分矩阵即为pssm,具体公式为其中,l为蛋白质的序列长度。
[0019]
步骤s3,将所有的所述特异性打分矩阵pssm分别填充或裁剪成相同尺寸的新特异性打分矩阵,当某一序列的pssm行数大于70时,对底部多出来的行裁剪掉;当某一序列的pssm行数小于70时,对底部缺少的的行用0补充。
[0020]
步骤s4,采用深度稀疏表示网络构建并训练dna结合蛋白识别分类器模型,构建并训练出的dna结合蛋白识别分类器模型是一种端到端的网络,能够明显的提高预测精度,误差较小,采用卷积自编码器可以鲁棒地学习特异性打分矩阵的潜在特征,并通过稀疏表示层进行分类,提高了模型的泛化能力。
[0021]
步骤s4具体步骤s4具体包括:s41,将步骤s1中的所述训练集和所述测试集进行行拼接得到拼接数据集其中x
train
为所述训练集,所述训练集x
test
为所述测试集,所述测试集其中,d0表示所述训练集和所述测试集的维度,n表示训练集样本个数,m表示测试集样本个数;s42,将所述拼接数据集x输入包含编码器、解码器和稀疏表示层的深度稀疏表示网络模型中,所述拼接数据集x经过所述编码器输出编码z,z=[z
train
,z
test
],其中,z
train
表示训练集编码,z
test
表示测试集编码,所述编码z经过所述稀疏表示层得到其中即
可定义in是单位矩阵,0n×m,0m均为0矩阵,a为稀疏表示矩阵,通过所述深度稀疏表示网络的目标函数其中,是所述解码器的输出,表示训练集解码,表示测试集解码,所述λ0=0.1和λ1=0.1,λ0和λ1均为正则化参数,即可计算出所述稀疏表示矩阵a;s43,取所述测试集x
test
中任意一个测试样本其对应的测试样本编码为其对应的在所述稀疏表示矩阵a中的相关稀疏编码列为αi,定义新向量中的非0元素是和αi相关的k类元素,任意一个测试样本的分类公式为完成对所述dna结合蛋白识别分类器模型的构建和训练。
[0022]
通过向卷积自编码器内输入拼接数据集x进行预训练,如图2所示,可以得到编码器和解码器的参数,再对编码器和解码器的参数进行微调,而深度稀疏表示网络模型中的编码器和解码器继承了微调后的编码器和解码器参数,卷积自编码器可以鲁棒地学习特异性打分矩阵的潜在特征,稀疏表示层在卷积自编码器的中间用来学习稀疏表示系数。
[0023]
深度模型在小样本数据上学习时容易过拟合,而本模型是通过稀疏表示层进行分类,提高了模型的泛化能力,在小样本数据上取得了很好的效果。
[0024]
步骤s5,将步骤s3中的新特异性打分矩阵输入dna结合蛋白识别分类器模型内,完成对dna结合蛋白序列的识别。
[0025]
上述实施例只为说明本发明的技术构思及特点,其目的在于让熟悉此项技术的人士能够了解本发明的内容并据以实施,并不能以此限制本发明的保护范围。凡根据本发明精神实质所作的等效变化或修饰,都应涵盖在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1