一种基于miRNA-mRNA调控关系的合成致死基因组合的特征构建方法
一种基于mirna-mrna调控关系的合成致死基因组合的特征构建方法
技术领域
1.本发明属于生物信息领域,具体涉及一种基于mirna-mrna调控关系的合成致死基因组合的特征构建方法。
背景技术:
2.合成致死(synthetic lethality)效应,又称协同致死效应,是最近几年抗癌药研发的一种全新思路,因该治疗策略毒性小且效果好而备受关注。然而,如何获得一批高质量的具合成致死效应的基因组合是当前面临的主要挑战。
3.机器学习预测是获取大量合成致死基因组合的一种高效的方法。大部分采用机器学习预测的方法所使用的特征值来源有限,如基因表达、蛋白质网络拓扑结构和基因功能等方面。然而,在合成致死相关的mrna-mirna调控网络特征值研究仍然缺乏,特别是基于大数据的研究方法。
技术实现要素:
4.目的:为了克服现有技术中存在的不足,本发明提供一种基于mirna-mrna调控关系的合成致死基因组合的特征构建方法,基于本方法构建的特征值可以对机器学习预测作出有效的贡献,提高预测效果。
5.为解决上述技术问题,本发明采用的技术方案为:
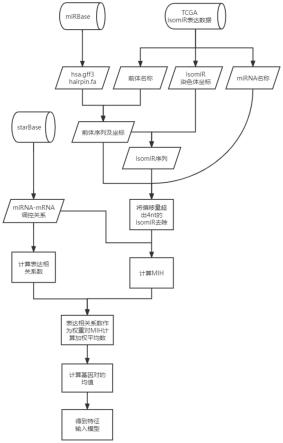
6.一种基于mirna-mrna调控关系的合成致死基因组合的特征构建方法,包括以下步骤:
7.获取isomir序列,利用信息熵量化mirna对应isomir序列的不确定性,作为mirna的特征;
8.获取mirna-mrna调控关系,找到对合成致死相关基因具有调控关系的mirna,用表达相关系数作为权重对mirna特征计算加权平均值;
9.合成致死基因组合的特征即为2个相关基因上述计算结果的均值,用于输入机器学习模型。
10.进一步的,根据isomir表达数据获取所有isomir的染色体坐标及其对应前体和成熟mirna的名称;根据前体的名称,从hsa.gff3中获得前体的染色体坐标,从hairpin.fa中获取前体的序列。
11.进一步的,获取所有isomir的染色体坐标,获取相应位置的序列。
12.进一步的,根据isomir的染色体坐标与前体坐标的相对位置,从前体序列中提取出isomir序列,其中正链的坐标起点在序列头部,负链的坐标起点在序列尾部。
13.进一步的,从hsa.gff3中前体对应成熟mirna中,根据成熟mirna名称找到isomir对应成熟mirna的染色体坐标。
14.进一步的,从hsa.gff3中前体对应成熟mirna中,根据成熟mirna名称找到isomir
对应成熟mirna的染色体坐标,将相对于成熟mirna坐标偏移量超出4nt的isomir去除。
15.进一步的,从starbase数据库下载所有mirna-mrna调控关系结果,下载的每一组调控关系应至少被三种预测软件预测到。
16.进一步的,对mirna利用信息熵量化mirna对应isomir5’端和3’端序列的不确定性,作为mirna的特征,记为mih:
[0017][0018]
mih表示mirna的特征,l表示经典mirna长度变化范围,其范围为
±
4nt,i为其中某位置,表示某位置碱基出现的频率,表示某位置碱基不出现的频率。
[0019]
进一步的,一对mirna-mrna调控关系,用tcga表达数据进行spearman相关性分析,保留显著负相关的结果。
[0020]
进一步的,对于所有调控某个mrna的mirna,对它们的mih用表达相关系数作为权重计算加权平均值,作为mrna的特征,记为m:
[0021][0022]
m表示mrna的特征,n为调控某个mrna的mirna数量,i表示其中一个mirna,r为表达相关系数,mih表示mirna的特征;
[0023]
合成致死基因组合的特征即为2个相关基因上述计算结果的均值。
[0024]
一种基于mirna-mrna调控关系的合成致死基因组合的特征构建方法,其特征在于,包括以下步骤
[0025]
步骤1:从tcga数据库isomir表达数据里获取所有isomir的染色体坐标,收集各mirna的isomir序列。将相对于成熟mirna坐标偏移量超出4nt的isomir去除。
[0026]
对这些mirna利用信息熵量化其对应isomir5’端和3’端序列的不确定性,作为mirna的特征,记为mih:
[0027][0028]
mih表示mirna的特征,l表示经典mirna长度变化范围(
±
4nt),i为其中某位置,表示某位置碱基出现的频率,表示某位置碱基不出现的频率。
[0029]
步骤2:从starbase数据库下载所有mirna-mrna调控关系结果,下载的每一组调控关系应至少被三种预测软件预测到。找到对合成致死相关基因具有调控关系的mirna。一对mirna-mrna调控关系,用tcga表达数据进行spearman相关性分析,用表达相关系数作为权重对mirna特征mih计算加权平均值,作为mrna的特征。
[0030]
步骤3:合成致死基因组合的特征即为2个相关基因上述计算结果的均值,用于输入机器学习模型。
[0031]
有益效果
[0032]
本发明提供的基于mirna-mrna调控关系的合成致死基因组合的特征构建方法,利用mirna调控及短小灵活的isomir深入探寻合成致死基因组合的特征,对机器学习预测作
出有效的贡献,提高预测效果。
附图说明
[0033]
图1是本发明实施例的方法流程图;
[0034]
图2是实施例中是否加入本发明所构建的特征时的模型预测效果比较。
具体实施方式
[0035]
下面结合附图和实施例对本发明作进一步描述。以下实施例仅用于更加清楚地说明本发明的技术方案,而不能以此来限制本发明的保护范围。
[0036]
实施例1
[0037]
从mirbase数据库下载人类mirna的染色体坐标文件hsa.gff3,下载所有前体的fasta格式序列文件hairpin.fa。
[0038]
从tcga数据库isomir表达数据获取所有isomir的染色体坐标及其对应前体和成熟mirna的名称。根据前体的名称,从hsa.gff3中获得前体的染色体坐标,从hairpin.fa中获取前体的序列。根据isomir的染色体坐标与前体坐标的相对位置,从前体序列中提取出isomir序列,其中正链的坐标起点在序列头部,负链的坐标起点在序列尾部。从hsa.gff3中前体对应成熟mirna中,根据成熟mirna名称找到isomir对应成熟mirna的染色体坐标,将相对于成熟mirna坐标偏移量超出4nt的isomir去除。
[0039]
从starbase数据库下载所有mirna-mrna调控关系结果,下载的每一组调控关系应至少被三种预测软件预测到。对于合成致死基因组合中涉及的mrna,从调控关系结果中提取出对其具有调控作用的mirna集合,对这些mirna利用信息熵量化mirna对应isomir5’端和3’端序列的不确定性,作为mirna的特征,记为mih:
[0040][0041]
mih表示mirna的特征,l表示经典mirna长度变化范围(
±
4nt),i为其中某位置,表示某位置碱基出现的频率,表示某位置碱基不出现的频率。
[0042]
一对mirna-mrna调控关系,用tcga表达数据进行相同样本集合下的spearman相关性分析,保留显著负相关的结果。对于所有调控某个mrna的mirna,用表达相关系数为权重计算加权平均值作为mrna的特征,记为m:
[0043][0044]
m表示mrna的特征,n为调控某个mrna的mirna数量,i表示其中一个mirna,r为表达相关系数,mih表示mirna的特征。
[0045]
合成致死基因组合的特征即为2个基因上述计算结果的均值。
[0046]
根据上述步骤得到的基于mirna-mrna调控关系所构建的合成致死基因组合的特征,结合多组学数据为实验验证过的合成致死基因组合构建特征矩阵。使用支持向量机模型(svm),90%的样本用于训练,10%的样本用于测试。重复十次,取测试结果的均值作为模型的最终评估结果。评估指标为auc(area under curve),定义为roc曲线下的面积。其中,
roc(receiver operating characteristic curve)是以真阳性率为纵坐标,假阳性率为横坐标绘制的曲线。对30种癌症类型进行了合成致死基因组合预测效果评估,同时也对仅使用多组学数据特征的预测模型进行了预测效果评估(表1,model1代表使用了本发明的特征的模型,model2代表仅使用多组学数据特征的模型,其他步骤一致):
[0047]
表1
[0048]
cancer typemodel1model2acc0.8530.681blca0.8580.818brca0.8500.807cesc0.8440.784chol0.9300.811coad0.8300.771dlbc0.9290.716esca0.8530.794gbm0.8650.812hnsc0.9080.664kich0.8370.777kirc0.8610.767kirp0.9330.854laml0.8920.832lgg0.8320.770lihc0.8680.831luad0.8350.781lusc0.8480.748ov0.9150.832paad0.8690.661pcpg0.8930.744prad0.8580.721read0.8680.784sarc0.8650.821skcm0.8910.847stad0.8820.753tgct0.8690.738thca0.9240.806thym0.9040.870ucec0.8780.731
[0049]
使用了本发明的特征的模型,在30种癌症中的auc值均高于仅使用多组学数据特征的模型。总体上,使用了本发明的特征的模型的auc值显著高于仅使用多组学数据特征的模型的auc值(图2)。
[0050]
在另一种机器学习模型随机森林中,基于mirna-mrna调控关系所构建的合成致死基因组合的特征的“meandecreaseaccuracy”指标达到40%,表明对本发明所构建的特征进行随机扰动后,对预测准确率会有显著的负面影响,证明了本特征构建方法的有效性与特征的重要性。
[0051]
以上所述仅是本发明的优选实施方式,应当指出:对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1