缺失突变的制作方法
自从20世纪10年代,产生溶剂的梭菌已用于丙酮、丁醇和乙醇的工业生产。在20世纪50年代期间,用来合成这些溶剂的更有效的石化技术的建立导致这样的大规模细菌发酵的放弃。然而,在当前环境中,随着利用可持续和可再生过程来进行化学品开发的压力越来越大,正重新开始对用于溶剂的生产的梭菌发酵感兴趣。这还受到对这些产溶剂梭菌(solventogenic clostridia)的生物理解的进步的帮助,其中借助于几个基因组的测序以及RNA测序和转录组学的应用。这些研究领域已打开了工程设计新菌株的可能性,其中上述新菌株能够过度产生丁醇,或除去竞争的副产物的生产,从而进一步提高溶剂型发酵的经济性。
为了利用基因组信息的这种流入,仍然需要用于产生重组梭菌和其它细菌菌株的快速和有效的方法,以促进研究和理解,其将导致商业相关的菌株的产生。
传统上很难产生重组梭菌菌株。低转化效率连同低重组效率一起已阻碍了制作稳定的表现出改善的溶剂相关表型的重组菌株的努力。在过去的几年中,已经开发了这样的技术,通过使用II型内含子,例如Targetron(Sigma)和Clostron(例如WO 2007/148091),其允许基因的插入失活;以及通过使用‘等位基因偶联交换’(ACE,例如WO 2009/101400),其允许新途径基因的整合;但通过这样的方法,DNA的部分的缺失是费力的并且这些方法并不适用于内源性质粒的完全去除。
已设计了等位基因交换法以通过同源重组方法来产生携带特定点突变或框内缺失的梭菌菌株。由于难以分离和选择成功的重组菌株,这些方法通常是低效的,虽然它们可能是高度特异性的(例如Cartman et al.,Appl.Environ.Microbiol.78(13),4683-90(2012))。另外。RNA敲除(knock-down)和干扰方法可以是有用的,如同用于产生重组菌株的转座子诱变。然而,虽然这些方法是有价值的研究工具,但这些方法针对非常特定的或狭窄范围的基因。
在一些情况下,基因组材料的较大的区域靶向的但非特定部分的缺失是优选的,因为这些方法可以用来产生菌株,其中完整操纵子被除去或其可以用来鉴定相对于非必要遗传物质的必要遗传物质(主要遗传物质,essential genetic material)。此外,在一些情况下,它是有用的以能够快速和有效地除去内源性质粒。内源性质粒的除去可以回答关于质粒的必要性的问题,例如巨大质粒(大质粒、巨型质粒)对于细胞存活可能是必要的,而小质粒可能是不必要的。当来源不相容性干扰有效转化方法时,迅速除去内源性质粒的能力也可以是有用的,从而制备对于研究和开发来说所必要的无质粒菌株。这些天然质粒的丧失‘天然地’可能需要相当大的努力(经过多轮的传代和选择)。
然后,得到的无质粒或‘携带缺失’菌株的基因组分析可以用来更多地理解关于微生物生理学,其可以潜在地与底物摄取机制和溶剂的生产相联。例如丙酮丁醇梭菌(C.acetobutylicum)携带巨大质粒,该巨大质粒编码α-淀粉酶和sol操纵子,其对于溶剂生产是必要的。在一些情况下,通过菌株降解,这种巨大质粒可能丢失。在丙酮丁醇梭菌中,巨大质粒的丧失提供了用于操作代谢通量的起始菌株(M5)。通过补回sol操纵子的仅所选基因,借助于质粒的丧失,其中上述sol操纵子被除去,这可以用来产生具有改变比率的丙酮:丁醇:乙醇溶剂的菌株(例如Nair et al.,J.Bacteriol.,176(18),5843-6,1994;Sillers et al.,Metab.Eng.,10(6),321-32,2008;Lee et al.,Biotechnol.J.,4(10),1432-1440,2009)。
可能会被用来进行这些基因组变化的方法是转录激活子样内切核酸酶(TALEN,例如US 8,420,782B2),但上述技术已开发用于编辑真核基因组,而尚未特别适用于工业相关的产溶剂型梭菌菌株。为每种基因靶工程设计TALEN的需要是昂贵和耗时的,以及在梭菌中上述技术将精确地如何工作的实用性,均不利于它在不远的将来变成广泛使用的工具。
因此总的来说,目前可用的用于产生高度特异性突变和缺失的方法都是复杂的、多阶段的和时间密集型流程。为了能够更快速地筛查或调查各种所选基因、操纵子、和质粒的重要性,以不需要是精确的方式,从细菌基因组DNA内或从内源性地存在于细菌中的质粒,简单和快速地除去遗传物质的片段的方法将显著更有用。
因此已开发了一种新方法,其是基于使用梭菌CRISPR/Cas系统(CRISPR是成簇的规则间隔的短回文重复序列的首字母缩略词)。这些系统通常被描述为‘原核适应性免疫系统’并且是这样的方式,借此方式细菌或古细菌细胞可以保护自己免受入侵DNA影响,通常为噬菌体或质粒DNA。
具有CRISPR/Cas系统的细胞能够选择性地将来自‘入侵’DNA的短片段整合进入Cas基因簇。每个片段被称为‘间隔区’并由同向重复序列(直接重复、正向重复,direct repeat)所侧接(flank)。如果细胞再次遭遇相同的入侵DNA,则细胞会将它识别为敌对物并会通过借助于Cas内切核酸酶来切割它以破坏它。
CRISPR/Cas系统在入侵DNA中识别的序列被称为‘原间隔区’并且与在基因组中的间隔区拷贝具有同一性。为了确保细胞不会意外攻击间隔区的基因组拷贝,在Cas I或Cas II系统中的原间隔区必须具有与它相关的短序列,称为PAM序列。PAM序列可以是原间隔区序列的上游或下游,其取决于系统的类型。如果它不存在或以任何方式被突变,则细胞将不再识别入侵DNA并且它将不会被破坏。
与来自酿脓链球菌(Streptococcus pyogenes)的cas9相关的PAM序列是众所周知的(Jiang et al.,Nature Biotech.(March 2013),vol.31,no.3,pp.233-239);然而,先前还没有确定与梭菌系统相关的PAM。
不是所有原核细胞都具有CRISPR/Cas基因同源物并且其中它们确实属于几个不同的类别(Makarova et al.,Nat.Rev.Microbiol.,9(6),467-77.2011)。关于来自酿脓链球菌和肺炎链球菌(Streptococcus pneumoniae)的II型cas 9系统,已发表了大量的工作(例如Jiang et al.,Nature Biotech.(March 2013),vol.31,no.3,pp.233-239)。这已经发展成为供用于真核细胞的基因组-编辑工具,其已成功用于例如酵母(DiCarlo et al.,Nucleic Acids Research,41(7),4336-4343,2013),斑马鱼(Hwang et al.,Nat.Biotechnol.,31(3),227-9.2013)和哺乳动物细胞(Ran et al.,Nature Protocols,8,2281-2308,2013)。
在真核细胞中,通过CRISPR/Cas系统所产生的dsDNA断裂可以用来生成缺失和其它INDEL(在DNA中的插入和缺失)以有效地敲除几乎任何基因,这是由于在真核细胞中非同源末端连接(NHEJ)途径的存在。然而,大多数细菌缺乏这些非同源末端连接(NHEJ)修复机制,认为借助于基因组序列的Cas9切割,仅导致细胞死亡,而不是基因缺失的产生。这在Barrangou和Marraffini,Molecular Cell,54(2),234–244,2014的综述中进行了总结。
现已开发了一种新方法,其使得能够在细菌基因组中以快速和有效的单一步骤过程来产生缺失。
此新方法也可以用来从细菌除去靶内源性质粒(包括巨大质粒)。通过,借助于缺失载体,靶向在质粒序列内的两个或更多个位置,上述系统应将质粒切割成两个或更多线性片段,其将易受破坏(通过天然细胞机制)。鉴于与典型的基因组相比,质粒的相对较小尺寸,所以在再结合可以发生以前更可能发生后者的完全降解,从而导致质粒的丧失。这可以用来确定是否内源性(巨大)质粒对于宿主生存是必要的,并且可以,关于质粒对宿主细胞的益处或代谢负担,提供洞察力。在实践中,为消除内源性质粒所需要的切割位点的最小数量可能依赖于靶质粒的尺寸,因为所有产生的片段应该足够小以致它们被降解而不是再结合。
因此,在一种实施方式中,本发明提供了用于在细菌基因组中产生缺失的方法,其中细菌包含CRISPR/Cas系统,上述方法包括以下步骤:
(a)用一种或多种缺失载体来转化细菌的群体,其中缺失载体能够引导产生第一crRNA和第二crRNA,其靶向在群体内的细菌的基因组内的第一和第二PAM/原间隔区;
(b)在由此产生第一crRNA和第二crRNA的条件下培养细菌的群体,其中它们靶向第一和第二PAM/原间隔区,以及其中第一crRNA和第二crRNA促进在群体内的一种或多种细菌中基因组的双重切割,以及其中再结合细菌基因组DNA的两个切割端;以及
(c)分离一种或多种细菌,其基因组包含在细菌基因组DNA中在第一和第二PAM/原间隔区之间的缺失。
在步骤(c)中,在细菌基因组DNA中的缺失优选包含至少包括和在第一和第二PAM/原间隔区之间的区。
在一些实施方式中,一种缺失载体能够引导产生第一crRNA和第二crRNA,其靶向在群体内的细菌的基因组内的第一和第二PAM/原间隔区。
优选地,缺失载体包含第一和第二Cas间隔元件,其侧接有Cas同向重复元件(直接重复元件、正向重复元件,direct repeat elements)。
第一和第二CRISPR间隔区可以是在缺失载体内的单个Cas阵列中或在单独的Cas阵列中。
在本实施方式中,缺失载体优选包含:
(i)Cas前导元件,
(ii)第一Cas同向重复元件,
(iii)第一Cas间隔元件,其能够引导产生第一crRNA,
(iv)第二Cas同向重复元件,
(v)第二Cas间隔元件,其能够引导产生第二crRNA,以及
(vi)第三Cas同向重复元件。
可替换地,缺失载体优选包含两个阵列,第一阵列包含:
(i)第一Cas前导元件,
(ii)第一Cas同向重复元件,
(iii)第一Cas间隔元件,其能够引导产生第一crRNA,以及
(iv)第二Cas同向重复元件,
以及第二阵列包含:
(v)第二Cas前导序列,
(vi)第三Cas同向重复元件,
(vii)第二Cas间隔元件,其能够引导产生第二crRNA,以及
(viii)第四Cas同向重复元件。
在其它实施方式中,第一缺失载体能够引导产生第一crRNA,其靶向在群体内的细菌的基因组内的第一PAM/原间隔区,以及第二缺失载体能够引导产生第二crRNA,其靶向在群体内的细菌的基因组内的第二PAM/原间隔区。
在本实施方式中,第一缺失载体优选包含:
(i)第一Cas前导元件,
(ii)第一Cas同向重复元件,
(iii)第一Cas间隔元件,其能够引导产生第一crRNA,
(iv)第二Cas同向重复元件,
和/或第二缺失载体优选包含:
(v)第二Cas前导元件,
(vi)第三Cas同向重复元件,
(vii)第二Cas间隔元件,其能够引导产生第二crRNA,
(viii)第四Cas同向重复元件。
在进一步的实施方式中,本发明提供了用于在细菌基因组中产生重排的方法,其中细菌包含CRISPR/Cas系统,上述方法包括以下步骤;
(a)用一种或多种缺失载体来转化细菌的群体,其中缺失载体能够引导产生第一crRNA和第二crRNA,其靶向在群体内的细菌的基因组内的第一和第二PAM/原间隔区;
(b)在由此产生第一crRNA和第二crRNA的条件下培养细菌的群体,其中它们靶向第一和第二PAM/原间隔区,以及其中第一crRNA和第二crRNA促进在群体内的一种或多种细菌中基因组的双重切割;以及
(c)分离一种或多种细菌,与对照非转化细菌相比,其基因组包含在细菌基因组DNA中的重排。
在另一种实施方式中,本发明提供了用于从在细菌中的内源性质粒除去或缺失DNA(其可能导致质粒的丧失)的方法,其中细菌包含CRISPR/Cas系统,上述方法包括以下步骤:
(a)用一种或多种缺失载体来转化细菌的群体,其中缺失载体能够引导产生两种或更多种crRNA,其靶向在群体内的细菌中的靶质粒内的两个或更多个PAM/原间隔区;
(b)在由此产生两种或更多种crRNA的条件下培养细菌的群体以及其中两种或更多种crRNA靶向一个或多个PAM/原间隔区,以及其中两种或更多种crRNA促进在两个或更多个位置处靶质粒的切割以在群体内的两种或更多种细菌中产生线性化DNA片段,以及其中使线性化片段经受通过内源性细胞机制的降解;以及
(c)分离缺乏靶质粒的一种或多种细菌。
优选地,在(低效)末端再结合过程能够修复质粒以前,使线性化片段经受通过内源性细胞机制的降解。
在细菌的群体中的细菌必须具有CRISPR/Cas系统。此CRISPR/Cas系统将是这样的一种系统,利用第一crRNA和第二crRNA或两种或更多种crRNA,其能够切割染色体DNA或其它靶DNA,例如靶质粒,优选细菌的内源性质粒。
将被接受的是,在一些情况下,在细菌群体内,可能存在污染。如在本文中所使用的,术语"细菌的群体"主要指期望用缺失载体来转化的细菌。
优选地,CRISPR/Cas系统是I型CRISPR/Cas系统。
在群体中的细菌可以具有内源性CRISPR/Cas系统或CRISPR/Cas系统可以是异源的。例如,异源性CRISPR/Cas系统可以是基于质粒的。
优选地,CRISPR/Cas系统是内源性CRISPR/Cas系统,即,它存在于野生型细菌中。在本发明的一些实施方式中,CRISPR/Cas系统不是基于质粒的系统。
在群体中的细菌可以,例如,是革兰氏阳性或革兰氏阴性细菌。优选地,细菌是革兰氏阳性细菌。
在一些实施方式中,细菌是生孢子细菌(spore-forming bacteria)。在其它实施方式中,细菌是解糖细菌(saccharolytic bacteria)。
细菌可以是需氧或厌氧细菌。优选地,细菌是厌氧菌。细菌可以是嗜热菌。
在还有其它实施方式中,细菌能够将基质转化成RCOOH,例如,转化成乙酸酯和/或丁酸酯。在这种情况下,R是脂族C1-C5,优选C1-3烷基或烯基。细菌还可以能够将RCOOH转化成溶剂,优选转化成丙酮、乙醇和/或丁醇的一种或多种。
在其它实施方式中,细菌是溶剂产生细菌。如在本文中所使用的,术语"溶剂产生"意思是,细菌是那些细菌,其能够产生溶剂,优选溶剂如丙酮、乙醇、丙醇和/或丁醇。在某些特别优选的实施方式中,细菌能够产生乙醇、丙酮和丁醇。优选地,细菌是丁醇产生细菌或丁醇耐受细菌。
在一些优选实施方式中,细菌属于梭菌属(Clostridium)。优选的梭菌属包括丙酮丁醇梭菌(C.acetobutylicum)、C.arbusti、金黄丁酸梭菌(C.aurantibutyricum)、拜氏梭菌(C.beijerinckii)、噬纤维梭菌(C.cellulovorans)、解纤维梭菌(C.cellulolyticum)、热纤维梭菌(C.thermocellum)、热丁酸梭菌(C.thermobutyricum)、巴氏梭菌(C.pasteurianum)、克氏梭菌(C.kluyveri)、诺氏梭菌(C.novyi)、糖丁酸梭菌(C.saccharobutylicum)、产琥珀酸梭菌(C.thermosuccinogenes)、热棕榈梭菌(C.thermopalmarium)、解糖梭菌(C.saccharolyticum)、糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)、酪丁酸梭菌(C.tyrobutyricum)、假破伤风梭菌(C.tetanomorphum)、大梭菌(C.magnum)、杨氏梭菌(C.ljungdahlii)、产乙醇梭菌(C.autoethanogenum)、丁酸梭菌(C.butyricum)、紫色梭菌(C.puniceum)、C.diolis、同型丙酸梭菌(C.homopropionicum)和玫瑰色梭菌(C.roseum)。
在本发明的一些优选实施方式中,细菌是糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)菌株N1,例如N1-4。在本发明的其它实施方式中,细菌是糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)N1-4(HMT)。在本发明的还有其它实施方式中,宿主细胞是糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)N1-504。
在其它优选实施方式中,细菌是巴氏梭菌(例如DSM 525)、酪丁酸梭菌(例如ATCC 52755)、或拜氏梭菌(例如NCP 258和NCP 262)或梭菌属DL-VIII。
在其它优选实施方式中,细菌属于芽孢杆菌属(杆菌属,Bacillus)。在其它优选实施方式中,细菌属于放线菌目(Actinomycetales)。在其它实施方式中,细菌优选不是链球菌(Streptococcus)或大肠杆菌(E.coli)。
细菌优选是这样的细菌,其具有内源性端连接酶或机制。在其它实施方式中,细菌是这样的细菌,其具有异源性端连接酶或途径。
靶质粒可以是内源性质粒(其自然发生在考虑之中的细菌中)。
待缺失的细菌DNA的核心区(基因组或质粒)侧接有两个CRISPR PAM/原间隔区,其能够由细菌的CRISPR/Cas系统所识别。
PAM/原间隔区是在细菌基因组或靶质粒中的序列,其包括PAM序列和原间隔区的功能组合。每种PAM/原间隔区序列能够由使用的CRISPR/Cas系统所识别,以及在生产适当的crRNA以后,它将通过CRISPR/Cas系统切割。
如在本文中所使用的,术语"功能CRISPR/PAM/原间隔区"是指CRISPR PAM/原间隔区,其能够由crRNA所识别,其中上述crRNA识别在细菌基因组或靶质粒中的CRISPR/PAM/原间隔区。在一些情况下,单突变(例如在PAM序列中)可能足以使得CRISPR/PAM/原间隔区无功能。
PAM是原间隔区-相邻基序的缩写词。PAM元件能够由细菌CRISPR/Cas系统所识别。PAM元件通常是3-6个核苷酸长并且对于每个细菌种类是特异的。
PAM元件相对于在细菌基因组或靶质粒中的原间隔区的取向是重要的。在一些细菌种类中,通常在原间隔区的5'端处或接近原间隔区的5'端处发现PAM元件。在其它种类中,通常在原间隔区的3'端处或接近原间隔区的3'端处发现PAM元件。
PAM元件可以是在细菌基因组或靶质粒的任一链上,但选为Cas间隔元件的序列应在和PAM元件相同的DNA链上(以致PAM元件和原间隔区是直接相邻的)。
一些研究已发现,在PAM元件中的几乎任何突变均消除通过CRISPR/Cas系统的识别(例如Jiang et al.,Nature Biotech(March 2013),vol.31,no.3,pp.233-239)。除功能原间隔区之外,PAM/原间隔区必须各自具有功能PAM元件。如在本文中所使用的,术语"功能PAM元件"或"CRISPR PAM元件,其在细菌中是有功能的"意思是,PAM元件能够由细菌的内源性CRISPR/Cas系统所识别,或者,如果细菌没有内源性CRISPR/Cas系统,则通过基于载体的异源性CRISPR/Cas系统,其已被引入细菌。
多于一种的序列可以能够充当在所选细菌种类中的PAM元件。例如,已知来自大肠杆菌K-12的I-E CRISPR-Cas系统具有四个功能PAM序列(Gomaa et al.(2014).mBio,5(1):e00928-13DOI:10.1128/mBio.00928-13),以及,使用在实施例2中描述的方法,已确定在糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)N1-4(HMT)中,至少四个有效的PAM序列(CCC、CCT、CCA和CCG)。
可以通过用质粒,其包含CRISPR间隔区、和相邻测试PAM元件,来转化具有CRISPR/Cas系统(它的内源性CRISPR/Cas系统或异源性质粒衍生系统)的细菌来测试PAM元件在特定细菌种类中发挥作用的能力。如果PAM元件在细菌中是有功能的,则CRISPR/Cas系统将破坏含PAM元件的质粒,以及转化效率将显著减小。在本文中此概念说明于实施例2。
CRISPR原间隔区是在细菌基因组或靶质粒内的序列,其被crRNA所靶向(条件是,也适当定位相容的PAM元件)。
在用于在细菌基因组中产生缺失的方法的步骤(a)中,用一种或多种缺失载体来转化细菌的群体,其中缺失载体能够引导产生第一crRNA和第二crRNA,其靶向在群体内的细菌的基因组内的第一和第二PAM/原间隔区。
此步骤(a)的目的是准备生产两种crRNA:第一crRNA将结合到在细菌基因组中的第一PAM/原间隔区;以及第二crRNA将结合到在细菌基因组中的第二PAM/原间隔区。
在用于除去在细菌中的靶质粒的方法的步骤(a)中,用一种或多种缺失载体来转化细菌的群体,其中缺失载体能够引导产生两种或更多种crRNA,其靶向在群体内的细菌中的靶质粒内的两个或更多个PAM/原间隔区。
此步骤(a)的目的是准备生产两种或更多种crRNA,其将结合到在靶质粒中的一个或多个PAM/原间隔区。
在用于除去在细菌中的靶(优选内源性)质粒的方法中,缺失载体可以能够引导产生一种、两种、三种、四种、五种或更多种crRNA。它们可以靶向在靶质粒内的一个、两个、三个、四个、五个或更多个PAM/原间隔区。
如在本文中所使用的,术语"转化(transformation)"和"转化(transforming)"是指借其将缺失载体插入细菌细胞的任何步骤。因此它尤其包括任何形式的电穿孔、接合或转染。
缺失可以是在框内或不在框内。PAM/原间隔区将两者/所有是在缺失的位点内。优选地,缺失包括至少部分的PAM或PAM/原间隔区,以致PAM/原间隔区变成不再有功能的(即不再由CRISPR/Cas系统所识别)。
缺失载体优选是环形载体。
缺失载体优选具有起始元件(起始点元件,origin element),最优选革兰氏阳性起始元件(例如“pBP1”)。
缺失载体还可以包含适当的选择标记(例如抗生素抗性基因)。
缺失载体不包含CRISPR PAM/原间隔区,其能够由crRNA所识别,其中上述crRNA识别在细菌基因组(或靶质粒)中的CRISPR/PAM/原间隔区。
在用于在细菌基因组中产生缺失的方法中,步骤(b)包括在由此产生第一crRNA和第二crRNA的条件下培养细菌的群体,其中它们靶向第一和第二PAM/原间隔区以及其中第一crRNA和第二crRNA促进在群体内的一种或多种细菌中的基因组的切割,以及其中细菌基因组DNA的两个切割端再结合。
在此步骤中,从细菌基因组(在缺失中)除去两个CRISPR PAM/原间隔区或使得两个CRISPR PAM/原间隔区不能由crRNA所识别,其中上述crRNA识别PAM/原间隔区。
在此步骤中,细菌的CRISPR/Cas系统(内源性或异源性)将在对应于第一PAM/原间隔区的基因组序列内的位点处以及在对应于第二PAM/原间隔区的基因组序列内的位点处切割细菌基因组DNA。因此,细菌基因组将被切割两次,线性化并且DNA的缺失部分将被释放。在第一和第二PAM/原间隔区内,切割的实际位点,因而缺失的程度,将依赖于在基因组中在两个PAM/原间隔区之间的距离。非常可能的是,在再结合以前,通过细胞机制,基因组DNA的切割端将被修剪回来(trim back)。在一些情况下,此修剪回来的程度可能是相当显著的,例如导致许多kb的侧接PAM/原间隔区对的序列的丧失。可以通过必要基因的邻近来确定修剪的最大程度,因为进一步修剪回来(进入这样的基因)将不是可行的,并且将与用于末端再结合的过程竞争。
在用于除去在细菌中的靶质粒的方法中,步骤(b)包括在由此产生两种或更多种crRNA的条件下培养细菌的群体以及其中两种或更多种crRNA靶向两个或更多个PAM/原间隔区,以及其中两种或更多种crRNA促进在两个或更多个位置处靶质粒的切割,以在群体内的一种或多种细菌中产生线性化DNA片段,以及其中通过内源性细胞机制,使线性化片段经受降解。在其中降解比DNA再结合更加快速地发生的情况下,质粒将从细胞丢失。
在此步骤中,在CRISPR PAM/原间隔区处或接近CRISPR PAM/原间隔区处切割靶质粒以产生质粒的两个或更多线性片段。在宿主修复机制能够再结合DNA以前,可以通过宿主细胞机制来降解这样的片段。
用于培养细菌的适宜条件将是本领域中容易已知的。这样的条件,例如,描述于“Clostridia:Biotechnology and Medical Applications”,Eds H.Bahl and P.Dürre,ISBN 3-527-30175-5,尤其是部分3.4“Growth conditions and nutritional requirements”。在Bergey’s Manual of Systematic Bacteriology,Volume appropriate to the chosen phylum of bacteria,e.g.Volume Three for the Firmicutes,ISBN 978-0-387-95041-9中还提供了详细信息。
术语"crRNA"是指CRISPR RNA。crRNA是短单链RNA分子,其由短同向重复序列组成,其中上述短同向重复序列侧接待通过CRISPR/Cas系统加以切割的靶间隔区序列。
“Cas前导元件”是DNA元件,其通常在同向重复序列簇中的第一重复序列的上游被发现。它有助于促进crRNA的生产,即,它充当RNA启动子。至今已确定许多Cas前导序列并且在任何特定的Cas系统中可以容易地建立它的序列。优选地,Cas前导序列是一种这样的序列,其对应于CRISPR/Cas系统,其存在于待转化的细菌群体中。
Cas同向重复序列是DNA元件,其由存在于细菌的群体中的CRISPR/Cas系统所识别。这些同向重复序列的长度通常为25-35个核苷酸,更通常约29或30个核苷酸的长度。
同向重复序列不需要是相同的序列(通常,Cas蛋白容忍1-2个核苷酸的差异)。同向重复序列通常具有能够形成发夹环的回文区。
依据CRISPR/Cas系统的CRISPR/Cas同向重复序列-间隔簇的任何检查,可以容易地找到适用于任何一种CRISPR/Cas系统的同向重复序列的DNA序列。
Cas间隔元件包含20-50个核苷酸(优选30-40个,更优选36-38个核苷酸)的序列,其具有相对于20-50个核苷酸(优选30-40个,更优选36-38个核苷酸)的高水平的序列同一性,相对于在PAM/原间隔区中的PAM元件,其存在(优选立即)于5'或相对于在PAM/原间隔区中的PAM元件,其存在(优选立即)于3',这取决于存在于感兴趣的细菌群体中的CRISPR/Cas系统的偏爱。
优选地,在细菌基因组或靶质粒中的PAM元件是直接相邻于原间隔区序列(在细菌基因组中)的起始。
在基因组DNA或靶质粒中的原间隔区和在缺失载体中的间隔元件序列之间的序列同一性的程度各自优选为至少80%,更优选至少85%、90%、95%、96%、97%、98%或99%,或是100%。
优选地,选择Cas间隔区序列,以致存在与非原间隔区元件的低概率的相互作用。
第一和第二Cas间隔元件结合于在细菌基因组中的第一和第二PAM/原间隔区。
类似地,Cas间隔元件结合于在靶质粒中的相应的位点。
通过在第一和第二PAM/原间隔区之间的距离来定义缺失的最小程度。
优选地,上述距离是10bp-1000kb,更优选100bp-100kb,甚至更优选200-500bp。
缺失载体可以包含抗生素抗性元件或其它选择标记,从而允许,例如在某些抗生素,例如氯霉素、红霉素、四环素、壮观霉素、链霉素等的存在下选择载体。优选地,缺失载体(当存在多于一种时)包含抗生素抗性元件,其允许它们的基于不同的抗生素的选择。
可以利用序列对比的BLAST方法来获得百分比氨基酸序列同一性和核苷酸序列同一性(Altschul et al.(1997),"Gapped BLAST and PSI-BLAST:a new generation of protein database search programs",Nucleic Acids Res.25:3389-3402;和http://www.ncbi.nlm.nih.gov/BLAST)。优选地,使用标准或默认序列对比参数。
标准蛋白质-蛋白质BLAST(blastp)可以用于在蛋白质数据库中发现类似的序列。像其它BLAST程序,blastp被设计用来找到相似性的局部区域。当序列相似性跨越整个序列时,blastp还将报告全局序列对比,对于蛋白质鉴定目的来说,其是优选的结果。优选地,使用标准或默认序列对比参数。在一些情况下,可以取掉"低复杂度滤器"。
还可以借助于BLASTN程序,得分=50,字长=3,来进行BLAST蛋白搜索。为了获得用于比较目的的有缺口的序列对比,可以采用Gapped BLAST(在BLAST 2.0中),如描述于Altschul et al.(1997)Nucleic Acids Res.25:3389。可替换地,PSI-BLAST(在BLAST 2.0中)可以用来进行迭代搜索,其检测在分子之间的远距离关系(远缘谱系关系,distant relationship)。(见Altschul et al.(1997)同上)。当采用BLAST、Gapped BLAST、PSI-BLAST时,可以使用各个程序的默认参数。
关于核苷酸序列比较,MEGABLAST、不连续的megablast、和blastn可以用来完成此目标。优选地,使用标准或默认序列对比参数。MEGABLAST是专门设计的以有效地找到在非常相似的序列之间的长序列对比。不连续的MEGABLAST可以用来查找与本发明的核酸类似但不同的核苷酸序列。
通过将查询分成称为字(word)的短子序列,BLAST核苷酸算法寻找类似的序列。上述程序首先识别与查询字的精确匹配(字命中(word hits))。然后在多个步骤中BLAST程序扩展这些字命中以生成最终有缺口的序列对比。在一些实施方式中,可以借助于BLASTN程序,得分=100,字长=12,来进行BLAST核苷酸搜索。
控制BLAST搜索的灵敏度的重要参数之一是字长。blastn比MEGABLAST更加敏感的最重要的原因在于,它使用较短默认字长(11)。由于这个原因,在查找与来自其它生物的相关核苷酸序列的序列对比中,blastn优于MEGABLAST。在blastn中字长是可调节的并且可以从默认值减小至为7的最小值以增加搜索灵敏度。
可以通过使用新引入的不连续的megablast页面(www.ncbi.nlm.nih.gov/Web/Newsltr/FallWinter02/blastlab.html)来实现更敏感搜索。此页面使用与由Ma et al.(Bioinformatics.2002Mar;18(3):440-5)报道的算法类似的算法。不是要求精确词匹配作为序列对比扩展的种子,不连续的megablast使用在模板的较长窗口内的非连续字。在编码模式下,通过专注于寻找在第一和第二密码子位置处的匹配同时忽略在第三位置处的错配来考虑第三碱基摆动(wobbling)。在不连续的MEGABLAST中,使用相同字长的搜索是比使用相同字长的标准blastn更加灵敏和有效的。不连续的megablast的独特的参数是:字长:11或12:模板:16、18、或21;模板类型:编码(0),非编码(1),或两者(2)。
通过细菌DNA修复机制,将再结合细菌基因组的两个切割端。(技术人员将容易地理解,在本文中提及“两个切割端”是指细菌基因组的两个双链DNA末端,其技术上包括四个DNA链)。
较小片段,例如生成自巨大质粒或质粒的切割,可能不再结合,而是在可能发生再结合以前,通过细胞机制降解。
缺失将经常是粗缺失(crude deletion),即,缺失往往不会是在已由Cas酶所切割的细菌基因组的两端之间的精确再结合。例如,细胞修复机制可以加工切割端,其导致一个或可能还有更多额外的核苷酸的缺失。例如,缺失可以包含在由第一和第二PAM/原间隔位点侧接的DNA的区的一侧或两侧上的1-5kb、5-10kb、10-30kb、30-50kb或50-100kb DNA的缺失。
在用于在细菌基因组中产生缺失的方法中,步骤(c)包括分离一种或多种细菌,其基因组包含在细菌基因组DNA中在第一和第二PAM/原间隔区之间以及可选地包括第一和第二PAM/原间隔区的缺失。优选地,除在由第一和第二PAM原间隔位点侧接的DNA的区的每侧上的至少1-10kb的侧接DNA之外,缺失还包含第一和第二PAM/原间隔区。
具有所期望的DNA缺失的细菌将容易失去缺失的DNA(在两个CRISPR/Cas切割位点之间以及可选地包括上述两个CRISPR/Cas切割位点),这是由于以下事实,它将是线性DNA并且因此容易出现通过宿主细胞机制的降解(例如通过内源性核酸酶的消化)。可能的是,DNA的缺失部分将不含有所有的用于在细胞(例如适当来源)中稳定维持的必需遗传元件,即使在降解以前它将被再结合。
分离的细菌是这样的细菌,其中基因组的两个切割端已被再结合。
在用于除去内源性质粒的方法中,步骤(c)包括分离一种或多种细菌,其缺乏完整或未切割的靶质粒,其以前携带一个或多个PAM/原间隔区.。
所选择或分离的细菌将是活细菌。
本发明进一步提供了用于制作突变细菌的方法,该方法包括通过本发明的方法在细菌基因组中产生缺失。
此外,本发明进一步提供了用于通过本发明的方法来制作缺乏靶巨大质粒或其它质粒(优选内源性质粒)的突变细菌的方法。
在质粒中,可能的是,上述方法将产生这样的缺失,其并不导致完整质粒的消除,而是导致产生较小质粒,其缺乏DNA的一部分,包括两个(或更多)靶位点。对于较大质粒,这更可能被看到,因为它们比较小质粒更可能能够容忍大缺失,因为存在较少的影响必需元件,如适宜的起点的缺失的机会。
本发明还提供了这样的细菌,其基因组已有通过本发明的方法所产生的缺失。
此外,本发明还提供了这样的细菌,通过本发明的方法,已引起其失去靶巨大质粒或质粒,优选内源性(巨大)质粒。
附图说明
图1示出来自若干梭菌种的同向重复序列的序列对比。
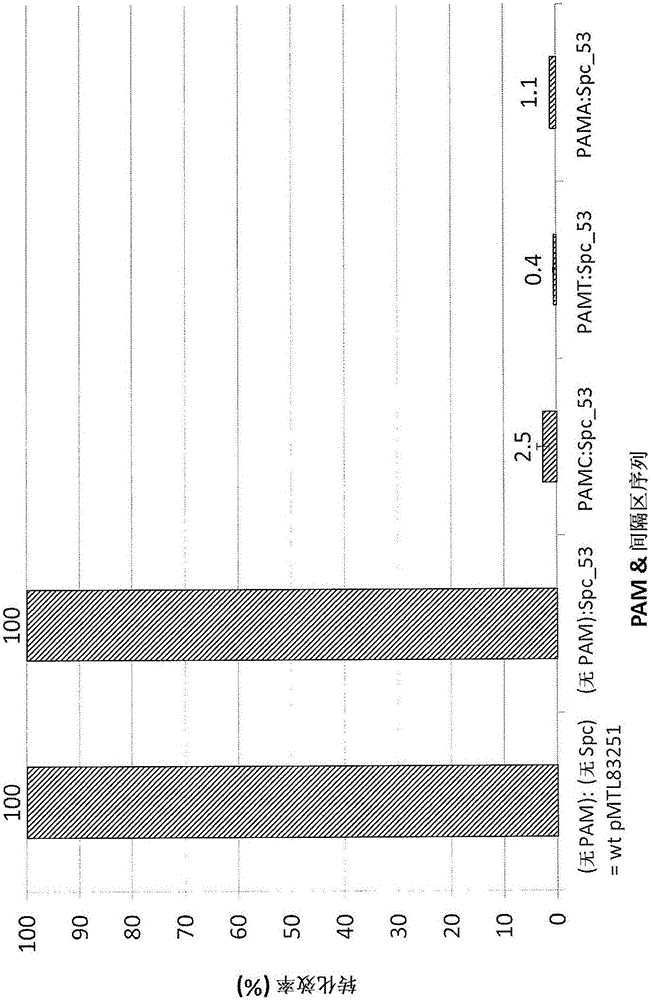
图2示出PAM序列对质粒到糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)N1-4(HMT)的转化效率的影响。
图3比较糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)N1-4(HMT)的野生型序列与来自在实施例3中产生的缺失菌株之一的基因组序列数据的相同区。该图是利用在https://www.sanger.ac.uk/resources/software/artemis/处的Artemis基因组浏览工具所制得。
实施例
实施例1:来自若干梭菌物种的同向重复序列的序列对比
目标:以鉴定可用于本发明的方法的一些同向重复序列。
方法:
利用CRISPRFinder程序Grissa,I.,Vergnaud,G.,&Pourcel,C.(2007)来寻找同向重复序列和间隔区序列。CRISPRFinder:一种用来鉴定聚簇规则间隔短回文重复序列(成簇的规律间隔的短回文重复序列)的网络工具。Nucleic Acids Res.,35,W52-7。
结果:
图1示出来自若干梭菌物种的同向重复序列的选择。在一些情况下,特定菌株具有多于一种的序列,所以这里包括最常用同向重复序列。缩写词如下:C_saccharoper=糖乙酸多丁醇梭菌(Clostridium saccharoperbutylacetonicum)N1-4(HMT)或N1-504),C_saccharob=糖丁酸梭菌(NCP258或NCP262,_1和_2是指2个主要DR簇),C_tyro=酪丁酸梭菌(ATCC 52755,_1和_2是指2个主要DR簇),C_pasteurianum=巴氏梭菌(DSM 525),C_autoethanogenum=产乙醇梭菌(Clostridium autoethanogenum)(DSM10061),C_sp_DLVIII=梭菌属(Clostridium sp.)(DL-VIII)。
实施例2:证实在糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)N1-4(HMT)中的PAM序列
目标:以说明如何测试假定PAM序列的有效性。
方法:
将来自糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)N1-4(HMT)的主要同向重复序列簇的间隔区_53的序列克隆到梭菌穿梭载体,pMTL83251。紧邻此间隔元件的5’端,加入各种不同的三核苷酸组合,包括预测的PAM序列CCC、CCG、CCT和非PAM序列GAC。当正确结合功能PAM序列时,间隔元件充当原间隔区。
利用标准电穿孔协议,接着在还含有5%葡萄糖的梭菌生长培养基(CGM)中过夜恢复阶段,将质粒转化成糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)N1-4(HMT)。然后将混合物扩散到含有5%葡萄糖和40μg/ml红霉素的CGM琼脂平板上并在32℃的厌氧箱中放置至少48小时。然后计数菌落,以和空载体的转化相比,确定转化效率的变化。
通过在750ml dH2O中溶解以下量来制备CGM培养基:5.0g酵母提取物、0.75g K2HPO4、0.75g KH2PO4、0.40g MgSO4.7H2O、0.01g FeSO4.7H2O、0.01g MnSO4.4H2O、1.0g NaCl、2.0g(NH4)2SO4、2.0g天冬酰胺(以及15g细菌琼脂1号,如果制作固体培养基),然后高压灭菌。不调节培养基的pH(通常在6.6的区域)。分别制备20%(w/v)葡萄糖溶液(50g葡萄糖,溶解于足够的dH2O,以给出250ml的最终容积)和高压灭菌。在冷却以后,合并葡萄糖和CGM溶液(根据需要)。
结果:
不同质粒的相对转化效率示于图2。空质粒pMTL83251和携带间隔区_53而没有PAM序列的质粒均产生菌落的菌苔(lawn)。携带相邻于5’CCC(PAMC)、CCT(PAMT)或CCA(PAMA)的间隔区_53的质粒产生明显更少的菌落。
实施例3:显示粗缺失方法
目标:以显示本发明的方法如何适于缺失DNA的较大片段。
方法:
在单靶基因中确定候选原间隔区元件(紧接地位于PAM序列的3’,已知其在此细菌系统中是功能性的)的选择,并从其中选择两种,其大约分开1kb(在基因组DNA中),对于此实施例,命名为原间隔区_1和原间隔区_2,以及分别位于靶基因中的5’和3’。基于pMTL82154骨架来设计和构建缺失载体。它携带前导序列、第一同向重复序列、间隔区_1(对应于潜在的原间隔区_1)、第二同向重复序列、间隔区_2(对应于潜在的原间隔区_2)和第三同向重复序列。
利用标准电穿孔方法,将这种缺失载体转化进入糖乙酸多丁醇梭菌(C.saccharoperbutylacetonicum)N1-4(HMT)。当被转化进入细胞时,缺失载体导致基因的Cas介导的切割以及遗传物质的随后丧失。
利用此方法,回收两个菌落,随后测序其中之一。这揭示了,相比于亲代菌株,本发明的方法已生成29205nt缺失。缺失部分包括在两个靶向PAM/原间隔位点之间的所有1kb区(见图3)。
在突变株的基因组序列中,读过缺失位点的序列数据如下:
用波浪线下划线的最初的27个核苷酸在位置5709460至5709486处匹配野生型基因组。用虚线下划线的最后的74个核苷酸在位置5738692至5738765处匹配野生型基因组。缺失的总长度是5738692-1-5709486=29205nt。
序列表
<110> 绿色生物制品有限公司(Green Biologics Ltd)
<120> 缺失突变
<130> 489.119750/01
<150> GB1406968.6
<151> 2014-04-17
<160> 12
<170> PatentIn version 3.5
<210> 1
<211> 30
<212> DNA
<213> 糖乙酸多丁醇梭菌(Clostridium saccharoperbutylacetonicum)
<400> 1
cttttaatat atccatattg gaatgtaaat 30
<210> 2
<211> 29
<212> DNA
<213> 糖乙酸多丁醇梭菌(Clostridium saccharoperbutylacetonicum)
<400> 2
gttttatatt aactatgagg aatgtaaat 29
<210> 3
<211> 29
<212> DNA
<213> 梭菌属 DLVIII(Clostridium sp DLVIII)
<400> 3
gttttatatt aactatgagg aatgtaaat 29
<210> 4
<211> 28
<212> DNA
<213> 糖丁酸梭菌(Clostridium saccharobutylicum)
<400> 4
gttttatctt aactagagga atgtaaat 28
<210> 5
<211> 30
<212> DNA
<213> 酪丁酸梭菌(Clostridium tyrobutyricum)
<400> 5
gttgaacctt aacatgagat gtatttaaat 30
<210> 6
<211> 30
<212> DNA
<213> 酪丁酸梭菌(Clostridium tyrobutyricum)
<400> 6
attgaacctt aacatgagat gtatttaaat 30
<210> 7
<211> 30
<212> DNA
<213> 糖丁酸梭菌(Clostridium saccharobutylicum)
<400> 7
gattaacatt aacatgagat gtatttaaat 30
<210> 8
<211> 30
<212> DNA
<213> 糖丁酸梭菌(Clostridium saccharobutylicum)
<400> 8
gttgaagatt aacattatat gttttgaaat 30
<210> 9
<211> 30
<212> DNA
<213> 糖丁酸梭菌(Clostridium saccharobutylicum)
<400> 9
gttgaagatt aacattatat gttttgaaat 30
<210> 10
<211> 30
<212> DNA
<213> 巴氏梭菌(Clostridium pasteurianum)
<400> 10
gttgaacctt aacataggat gtatttaaat 30
<210> 11
<211> 30
<212> DNA
<213> 产乙醇梭菌(Clostridium autoethanogenum)
<400> 11
gttgaacctc aacatgagat gtatttaaat 30
<210> 12
<211> 101
<212> DNA
<213> 糖乙酸多丁醇梭菌(Clostridium saccharoperbutylacetonicum)
<400> 12
aaaccctatt tccaaaaaaa atgaatgctt ttcatgcttt tttaaaattt cttctatcag 60
cttatttata caaaactctt ctatttttag ctcatatgct t 101
- 还没有人留言评论。精彩留言会获得点赞!
- 区分疫苗免疫和野毒感染动物的猪胸膜肺炎放线杆菌血清7型双基因缺失突变株的疫苗 ...的制作方法
- 迟缓爱德华氏菌基因工程弱毒株及其应用的制作方法
- 猪传染性胸膜肺炎放线杆菌血清1型锌离子结合蛋白a缺失株及其构建方法和应用的制作方法
- 一种不含抗性标记的鼠伤寒沙门氏菌基因缺失突变菌株、疫苗及应用的制作方法
- 一种不含抗性标记的猪胸膜肺炎放线杆菌血清1型双基因缺失突变株的疫苗及应用的制作方法
- 一种伪狂犬病tk的制作方法
- 猪传染性胸膜肺炎放线杆菌Apx I C基因缺失突变株、构建方法、疫苗及应用的制作方法
- L-乳酸脱氢酶基因缺失的工程菌及其构建方法与应用的制作方法
- 一种不含抗性标记的猪霍乱沙门氏菌基因缺失突变菌株及其疫苗的制作方法
- Bim基因缺失突变检测探针和液相芯片的制作方法