治疗剂的制作方法
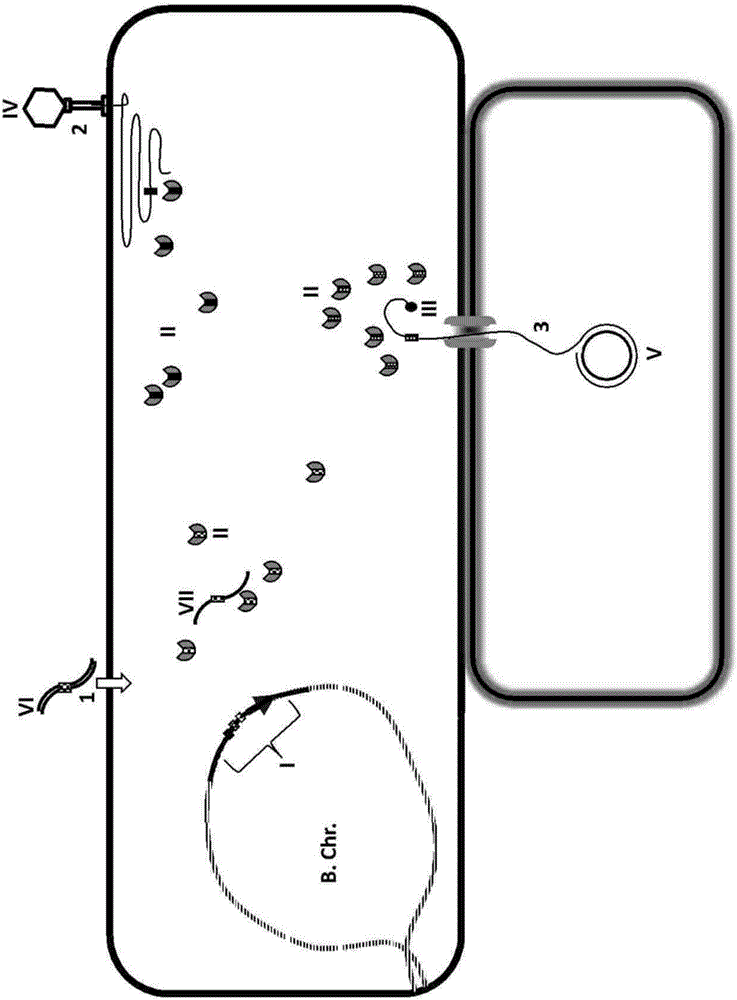
最初从微生物,如链霉菌(Streptomyces)中分离的抗生素是治疗感染性疾病的有效方式。然而,细菌响应选择压力非常迅速地获得对抗生素的抗微生物抗性(AMR)。AMR的一个常见途径是经由质粒载体上的水平传递获得在初始抗生素生产微生物中进化的抗性基因。此类质粒在一些情况下获得了由可转座元件和整合子携带的多种抗生素抗性基因。还发生了修饰细菌蛋白靶标或防止抗生素进入的宿主编码的突变。微生物如细菌病原体对抗生素的抗性是我们最严重的健康威胁之一。例如,来自抗性细菌的感染现在并不罕见,并且一些病原体甚至变得对多种类型或种类的抗生素具有抗性。有效的抗生素的损失破坏了我们抵抗传染病和管理易受攻击的患者中常见的传染性并发症的能力,例如那些正在经历用于癌症的化学疗法,用于肾衰竭的透析,和手术,特别是器官移植的患者(对于所述患者,治疗继发感染的能力是至关重要的)。当一线和二线治疗选项受到抗生素抗性限制或不可用时,医疗护理提供者被迫使用备选抗生素,其可能对患者更具毒性,更昂贵且效果更差。即使在存在备选治疗时,感染抗性的患者通常更可能死亡,而幸存者可以具有显著较长的住院期,延迟的恢复期和长期残疾。现代医学的许多成就受到AMR的威胁。没有用于护理和预防感染的有效抗生素,治疗如器官移植,癌症化学疗法和大手术的成功将受到损害。全球贸易和旅行的增长允许抗生素抗性微生物通过人类,其它动物和食物快速传播。抗性机制分成四种类别:(1)降解抗生素的酶,包括破坏青霉素抗生素家族的β-内酰胺环的β-内酰胺酶;(2)修饰抗生素的酶包括磷酸化氨基糖苷类抗生素,如卡那霉素的氨基糖苷磷酸转移酶;使氯霉素乙酰化的氯霉素乙酰转移酶(CAT);(3)外排泵,其将抗生素从胞质溶胶输出到细胞外部,如在四环素存在下表达的四环素流出泵,以及赋予多药抗性的其它泵,其能够输出一系列抗生素;和(4)改变抗生素的蛋白质靶标,使得它不再被所述靶标灭活的突变;例如β-内酰胺是杀细菌的,因为它们抑制肽聚糖生物合成和细菌细胞壁完整性需要的青霉素结合蛋白(PBP),并且不会抑制具有降低的与β-内酰胺的结合的PBP突变体。目前正在使用或开发几种方法来解决抗生素抗性的问题,包括以下。第一,开发了新的抗生素,如现有药物的衍生物。已经开发了较少的新的抗生素药物,并且许多更具毒性,因此在最后使用。微生物已经获得了对这两种类型的新抗生素的抗性。第二,已经尝试直接抑制抗性酶。实例包括克拉维酸(clavulanicacid),一种与阿莫西林(一种β-内酰胺抗生素(也称为力百汀(Augmentin))组合使用的β-内酰胺酶抑制剂。其它β-内酰胺酶抗生素抑制剂包括碳青霉烯类(carbapenems)。但即使在这里抗性已经出现。BlueberryTherapeutics和Avacta正在开发肽亲和体(affimer)来靶向抗性机制。开发抗生素抗性酶的新抑制剂需要药物开发的长管线(longpipeline)。EricBrown博士在加拿大麦克马斯特大学(McMasterUniversity)采用了一种备选方法,他正在针对具有无关靶标的已知药物(已经批准使用)筛选灭活抗体抗性机制的隐蔽活性。第三,已经使用非抗生素杀细菌剂。例如,噬菌体的感染是在20世纪20年代发展起来的,并且虽然在发现抗生素的情况下很大程度上中断,但是在某些国家已经得到保留。目前的方法使用杀死细菌,包括抗生素抗性细菌的有毒力的裂解性噬菌体,但这开辟了选择对噬菌体感染有抗性的细菌变体的方式。为了避免这点,正在使用含有不同噬菌体株的混合物的制剂。在患有败血症的患者中使用此类裂解噬菌体的另一个缺点是通过裂解噬菌体所致的细胞裂解和死亡可以将内毒素从细胞释放到血液中,并且可以引起内毒素休克。需要抗击抗生素抗性微生物的其它组合物和方法。根据本发明的第一方面,提供了重组多核苷酸,其包含具有或转录RNA引导分子(RNAguidemolecule)的聚簇规则间隔短回文重复(clusteredregularlyinterspacedshortpalindromicrepeat,CRISPR)阵列核酸序列(arraynucleicacidsequence),所述RNA引导分子具有与微生物中的抗生素抗性基因的靶序列充分互补的间隔区(spacer,也称为间隔物)序列,使得抗生素抗性基因在存在CRISPR相关(Cas)DNA结合多肽或其功能等同物或修饰形式的情况下被灭活,从而使微生物对抗生素敏感。在本发明的另一方面,提供了用于将多核苷酸导入微生物,诸如抗生素抗性微生物中的递送媒介物,其中所述递送媒介物包含用于灭活携带基因的DNA的重组多核苷酸,所述基因编码对微生物赋予抗生素抗性的抗生素抗性酶,并且其中所述重组多核苷酸包含具有或转录RNA引导分子的聚簇规则间隔短回文重复(CRISPR)阵列核酸序列,所述RNA引导分子具有与所述抗生素抗性基因的靶DNA序列充分互补的间隔区序列,使得所述抗生素抗性基因在存在CRISPR相关(Cas)DNA结合多肽或其功能等同物或修饰形式的情况下被靶向和灭活,从而使所述微生物对所述抗生素敏感,其中所述递送媒介物是非毒力或溶原性噬菌体。本发明的一般目的是使用CRISPR/Cas系统灭活携带编码抗生素抗性酶的基因的DNA。本发明的优点是一种或多种现有的抗生素可以用于治疗感染性疾病,因为微生物对抗生素变得重新敏感或被阻止获得抗生素抗性。抗生素抗性基因的靶序列可以是基因本身的侧翼序列,其在被破坏的情况下灭活抗生素抗性基因。例如,如果抗生素抗性基因位于质粒上,那么本发明可以涵盖质粒中的靶序列。与灭活抗生素抗性酶的现有技术方法形成对比,本发明不需要每种新药需要的新药开发和伴随的管理批准。相反,本发明提供了可以直接应用于靶向和灭活相关抗生素抗性基因而不是基因产物的工具。例如,可以靶向编码蛋白质的基因,所述蛋白质分别通过改变膜通透性和流出泵表达来调节抗生素的摄取和输出。CRISPR/Cas系统是RNA介导的基因组防御途径,其是类似于真核RNAi途径的针对核酸侵入物的天然细菌和古细菌免疫系统的一部分(参见例如Grissaetal.,2007,BMCInformatics8:172;Horvath&Barrangou,2010,Science,327:167–170;Gasiunasetal.,2012,Proc.NatlAcad.Sci.USA109:E2579;Marraffini&Sontheimer,2008,Science,322:1843-1845;Garneauet.al.,2010,Nature468:67)。天然CRISPR系统含有Cas基因以及能够编程CRISPR介导的核酸切割的特异性的非编码RNA元件的组合。迄今为止,在一大批细菌和古细菌宿主中鉴定了三种类型(I-III)CRISPR系统。每个CRISPR基因座由一系列以非重复间隔区序列分隔的短DNA同向重复(directrepeat,也称为直接重复)组成。间隔区序列本质上通常源自外来遗传元件,如噬菌体和质粒。如本文中使用的,重复加上非重复间隔区序列的系列称为CRISPR阵列。将CRISPR阵列转录并与重复互补的tracrRNA杂交,随后在同向重复序列内切割并加工成含有单独间隔区序列的短成熟双重tracrRNA:crRNA,其将Cas核酸酶导向靶位点(也称为“原间隔区(protospacer)”)。例如,II型CRISPR/Cas9系统(一个充分研究的例子)在四个步骤中实施靶向DNA双链断裂(“DSB”)。首先,两个RNA,前crRNA阵列(pre-crRNAarray)和tracrRNA,从CRISPR基因座转录。其次,tracrRNA与pre-crRNA的重复区杂交,并且介导将前-crRNA加工为成熟的crRNA(本文中也称为“RNA引导分子gRNA”,其含有个别或单体间隔区序列);第三,成熟的crRNA:tracrRNA复合物通过crRNA上的间隔区和靶DNA上的靶位点之间的碱基配对将Cas9蛋白以核糖核蛋白的形式导向靶DNA。最后,Cas9介导靶DNA的切割并创建DSB。在本发明中,如本文中详述的,使用经修饰的CRISPR构建体靶向抗生素抗性基因。使用此类构建体的本发明的重组多核苷酸在本文中也称为“刺客构建体(assassinconstruct)”,其用于实现此类基因的灭活。迄今为止使用CRISPR技术的主要焦点是用作反求遗传学的DNA编辑工具,主要在真核生物中。然而,WO2007/025097描述了CRISPR技术在调节细胞中针对入侵靶核酸或其转录产物,特别是针对入侵噬菌体的抗性中的用途。例如在WO2010/075424中已经提出了使用CRISPR技术下调原核基因表达以靶向由基因转录的靶mRNA的方法。WO2012/164565描述了来自乳球菌(Lactoccocus)的CRISPR系统和该系统在调节细胞针对侵入靶核酸或其转录产物的抗性中的用途。相比之下,本发明尤其关心抗生素抗性微生物中涉及赋予抗生素抗性的基因的灭活。根据本发明,RNA引导分子可以介导CasDNA结合多肽或其功能等同物或其修饰形式与抗生素抗性基因的结合。这反映了上文描述的天然系统。本发明的CasDNA结合多肽或其功能等同物或其修饰形式还可以结合RNA或其它核酸分子。换言之,对于能够结合DNA的CasDNA结合多肽或其功能等同物或其修饰形式的要求在本发明的一些方面中不排除能够结合RNA或其它核酸分子的多肽或其功能等同物或其修饰形式。在这些方面中,CasDNA结合多肽或其功能等同物或其修饰形式可以称为Cas核酸结合多肽或其功能等同物或其修饰形式。对于某些应用,微生物可以具有天然内源性或导入的工程化的CasDNA结合多肽或功能等同物或修饰形式。这意味着本发明的重组多核苷酸不需要编码CasDNA结合多肽或功能等同物或修饰形式。或者,本发明的重组多核苷酸可以进一步包含编码CasDNA结合多肽或其功能等同物或修饰形式的核酸序列。在另一方面,本发明的重组多核苷酸不编码CasDNA结合多肽或其功能等同物或修饰形式,但确实可以与分开的多核苷酸联合使用。可以使用将CasDNA结合多肽或其功能等同物或其修饰形式导入微生物中的其它手段。根据本发明的示例性CasDNA结合多肽是Cas9或其功能等同物或其修饰形式。在根据本发明的各个方面的重组多核苷酸中,CRISPR阵列核酸序列可以具有或转录额外的RNA引导分子,每个RNA引导分子包含与抗生素抗性基因或一种或多种另外的抗生素抗性基因的靶序列充分互补的间隔区序列。所述或每个RNA引导分子可以从其自身的启动子序列转录。或者,多个RNA引导分子的组可以从一个启动子序列转录并任选地与一个或多个其它此类组组合。例如,四个RNA引导分子的组可以从一个启动子序列转录,例如与引导分子的一个或多个其它此类组组合。具有多个RNA引导分子允许微生物中的不同抗生素抗性(或其它类型)基因被同时靶向和灭活。根据本发明的各个方面的重组多核苷酸可以另外或备选设计为包括靶向参与致病性或微生物代谢的其它方面的基因的RNA引导分子(如另外的RNA引导分子)。例如,某些病原体形成生物膜,其使得抗生素难以接近它们。可以靶向参与用于生物膜生成的细菌代谢的一个或多个基因。启动子远端的间隔区序列通常不太有效地转录。理想地,针对不同靶标的多个RNA引导分子应当或多或少地同等呈现。因此,可以使用转录每个RNA引导分子的一个启动子(而不是依赖于长的多顺反子RNA引导分子[或前体crRNA]转录)。例如,存在许多编码β-内酰胺酶的抗性基因(bla基因),其给予针对一大批不同β-内酰胺抗生素的抗性。表达多个RNA引导分子的DNA构建体可以用于靶向许多不同的bla基因,所述RNA引导分子可以各自从它们自己的此类启动子单独转录。因此,在本发明的方面中,CRISPR阵列核酸序列可以具有或转录一个或多个RNA引导分子,每个RNA引导分子包含与一个或多个β-内酰胺酶基因的靶序列充分互补的间隔区序列。例如,一个或多个RNA引导分子可以靶向选自下组的一种或多种或全部基因:NDM,VIM,IMP,KPC,OXA,TEM,SHV,CTX,OKP,LEN,GES,MIR,ACT,ACC,CMY,LAT和FOX。特别地,一个或多个RNA引导分子可以包含与抗生素抗性基因的β内酰胺家族的靶序列充分互补的间隔区序列,其包括一个或多个或全部以下各项:与NDM-1,-2,-10的靶序列充分互补的第一间隔区序列;与VIM-1,-2,-4,-12,-19,-26,-27-33,34的靶序列充分互补的第二间隔区;与IMP-32,-38,-48的靶序列充分互补的第三间隔区;与KPC-1,-2,-3,-4,-6,-7,-8,-11,-12,-14,-15,-16,-17的靶序列充分互补的第四间隔区;与OXA-48的靶序列充分互补的第五间隔区;与用于TEM-1,-1B,-3,-139,-162,-183,-192,-197,-198,-209的靶序列充分互补的第六间隔区;与SHV及其变体的靶序列充分互补的第七间隔区;以及与CTX及其变体的靶序列充分互补的第八间隔区。待灭活的抗生素抗性基因可以位于染色体上,或位于称为复制子并且包括质粒和噬菌体的染色体外复制DNA分子上。根据本发明的一个方面使用的CRISPR/Cas系统在靶序列中产生DSB。当靶序列位于染色体或复制子,如细菌染色体或质粒上的情况下,则DSB可以导致降解并因此导致遭受此类DSB的染色体或复制子的损失。如果靶序列位于细菌染色体上,那么细胞可以由于DSB而直接死亡。另外,一些质粒(包括天然质粒)携带杀伤功能,其仅在细胞丧失质粒的情况下变为有毒性的,这是确保质粒在分裂细胞中的忠实遗传力的天然机制。如果携带抗生素抗性基因的靶序列的质粒也携带此类杀伤功能,并且质粒由于生成的DSB而丧失,那么细胞可能死亡(参见Sengupta&Austin,2011,Infect.Immun.79:2502-2509)。在由此类DSB引起的细胞死亡增加根据本发明的各个方面的重组多核苷酸的抗性的选择压力的情况下,这可以通过例如采用经修饰的CasDNA结合多肽来减轻,所述经修饰的CasDNA结合多肽密封在产生缺失后的靶位点以使抗生素抗性基因的靶序列灭活,而不是产生DSB。因此,根据本发明的各个方面的CasDNA结合多肽在某些方面可以被包含重组酶催化域的经修饰的CasDNA结合多肽替代,其中经修饰的CasDNA结合多肽不产生DSB,但创建缺失并重新密封靶序列中的位点。经修饰的CasDNA结合多肽可以例如是包含重组酶催化域的经修饰的Cas9蛋白。根据本发明的各个方面的重组多核苷酸还可以包含编码对微生物赋予选择性优点的基因的核苷酸序列,例如从而提高CRISPR/Cas系统向靶微生物的递送效率。例如,该基因可以比非感染的同胞(sibling)或编码细菌素(这些是由细菌产生的杀死或抑制其它细菌的生长的蛋白毒素)的基因赋予生长优势,并且可以使用相应的免疫多肽。对微生物的选择性优点可以包括或者是下述选择性优点,其防止或减少由于由CasDNA结合多肽引起的DSB导致的复制子丧失的效应。例如,编码对微生物赋予选择性优点的基因的核苷酸序列可以编码中和由靶序列所在的复制子携带的毒素或杀伤功能的效应的抗毒素。此外,编码对微生物赋予选择性优点的基因的核苷酸序列可以编码由复制子编码的一种或多种蛋白质,所述复制子由于由CasDNA结合多肽引起的DSB而降解。在本发明的另一方面,提供了用于将多核苷酸导入微生物的递送媒介物,其中递送媒介物包含如本文中定义的重组多核苷酸。根据本发明的各种一些方面的递送媒介物可以是噬菌体。或者,递送媒介物可以是质粒,如接合质粒或其它质粒复制子,核苷酸载体,线性双链DNA(dsDNA),RNA噬菌体或RNA分子。递送媒介物可以是非毒力噬菌体,如噬菌体M13,丝状噬菌体,其感染大肠杆菌(Escherichiacoli)细胞,复制并从细菌宿主细胞分泌,而不杀死细菌宿主,该细菌宿主继续生长和更慢地分裂。另一种合适的丝状噬菌体是从淋病奈瑟氏球菌(Neisseriagonorrhoeae)分离的NgoPhi6,其能够感染多种革兰氏阴性细菌而不杀死宿主。或者,可以使用溶原性噬菌体,其不总是在感染后杀死宿主细胞,而是感染并变为休眠的原噬菌体。因此,本发明可以使用新的系统来灭活细菌中的抗生素抗性基因,主要将不杀死细菌的噬菌体,和/或接合质粒和/或直接DNA转化用作本发明的重组多核苷酸的递送机制。在本发明的另一方面,提供了包含如本文中定义的重组多核苷酸或如本文中定义的递送媒介物的组合物。组合物可以是药物组合物,非致病微生物,如例如在益生菌制剂(probioticformulation)或膳食补充剂中的共生细菌。如本文中使用,“药物组合物”是指一种或多种活性剂(如本文中所述的重组多核苷酸或递送媒介物)与其它化学组分如生理学合适的载体和赋形剂的制剂。药物组合物的目的是便于将活性剂施用于生物体。组合物可以配制用于局部(topical),肠或肠胃外施用。如果想要的话,本发明的组合物可以在包装,分配器装置或试剂盒中呈现,其中每一个可以含有一种或多种含有活性剂的单位剂型。包装,分配器装置或试剂盒可以伴随有施用说明书。根据本发明还提供了如本文中定义的组合物,其用作药物。在另一方面,根据本发明提供了如本文中定义的组合物,其用于治疗或预防由抗生素抗性微生物引起的感染,所述抗生素抗性微生物包含由重组多核苷酸的RNA引导分子靶向的抗生素抗性基因。本发明还提供了治疗或预防受试者由包含抗生素抗性基因的抗生素抗性微生物引起的感染的方法,其中所述方法包括下述步骤:将治疗有效量的如本文中定义的组合物导入微生物中,其中RNA引导分子靶向抗生素抗性基因,从而使抗生素抗性基因灭活并使微生物对抗生素敏感。组合物可以局部或口服施用。或者,组合物可通过静脉内,胃肠外,眼或肺施用来施用。用于局部施用的本发明组合物可以是适于局部使用的形式,如例如气溶胶,霜剂,软膏,洗剂或撒粉剂(dustingpowder)。本文中提供的组合物可以配制为通过吸入施用。例如,组合物可以是气溶胶,雾(mist)或粉末的形式。因此,本文中所述的组合物可以使用合适的推进剂诸如例如二氯二氟甲烷,三氯氟甲烷,二氯四氟乙烷,二氧化碳或其它合适的气体从加压包装或喷雾器以气溶胶喷雾呈现形式递送。在使用加压气溶胶的情况下,可以通过提供阀门以递送计量的量来确定剂量单位。在该方法中,受试者可以是植物,鱼,鸟,爬行类动物或哺乳动物(如人)。根据该方法,可以通过质粒接合或噬菌体感染将递送媒介物从微生物直接转移到另一种微生物(如抗生素抗性微生物)中。该方法还可包括同时或随后对受试者施用微生物已致敏的抗生素的步骤。本发明的另一方面提供了灭活微生物中抗生素抗性的方法,所述方法包括将如本文中定义的重组多核苷酸或如本文中定义的递送媒介物导入微生物中。该方法可以是应用于受试者的体内方法,或体外方法。根据本发明还提供了包含本文中定义的重组多核苷酸的宿主细胞。宿主细胞可以是例如共生细菌。通过本发明的各个方面靶向的微生物可以在体表,局部化(例如,包含在器官内,在手术创伤或其它创伤的部位,在脓肿内),或者可以是全身的。包括治疗细菌性感染,其适于通过局部施用,例如使用本发明的噬菌体进行的治疗。本发明还涵盖用本发明的重组多核苷酸,递送媒介物或组合物涂覆除体表外的表面(例如伤口敷料或医疗装置表面)。现在将参考以下附图描述本发明的其它方面,特征和非限制性实施方案:图1:CRISPR/Cas9介导的针对质粒上的抗生素抗性基因的细菌免疫:I,CRISPR/Cas基因座,其中框表示靶向不同抗生素抗性基因的不同间隔区序列;II,gRNA-Cas9,其中框表示靶向每个抗生素抗性基因的不同gRNA;III,含有抗生素抗性基因的质粒;IV,Cas9的靶标识别和定位;V,切割的质粒,VI:噬菌体。ApR=氨苄青霉素抗性,CmR=氯霉素抗性,KmR=卡那霉素抗性。ApS=氨苄青霉素敏感,CmS=氯霉素敏感,KmS=卡那霉素敏感。Bla=β-内酰胺酶,CAT=氯霉素乙酰转移酶,APH=氨基糖苷磷酸转移酶。1.注射:通过细菌特异性噬菌体感染将CRISPR/Cas9与噬菌体DNA一起注射到细菌细胞中。2.溶原化:将噬菌体DNA溶原化并整合入细菌宿主染色体(B.chr.)。3.crRNA生物发生(biogenesis)和Cas9的装配:Pre-crRNA被转录并与tracrRNA杂交并加工以生成成熟的crRNA:tracrRNA(一种RNA引导分子,或“gRNA”),其与Cas9装配。4.识别:这些gRNA-Cas9复合物识别质粒上的靶DNA。5.切割:gRNA-Cas9复合物在由crRNA识别的位点切割DNA。6.灭活:这导致抗生素抗性酶生成的灭活;7.敏感:因此,细菌细胞变得对各种抗生素敏感。图2:防止非致病性细菌和无症状病原体未来遇到抗生素抗性基因:I,CRISPR/Cas基因座,在本文例示为存在于细菌染色体(B.Chr.)中,并且其中框表示靶向不同抗生素抗性基因的不同间隔区序列;II,gRNA-Cas9,其中框表示针对每个抗生素抗性基因的不同gRNA;III,松弛酶:在双链DNA中产生链特异性,序列特异性切口以启动质粒DNA的接合转移的酶;IV,噬菌体,V,质粒;VI,双链DNA;VII,单链DNA。显示了遇到未来抗生素抗性基因的三个进入途径。1.转化:携带抗生素抗性基因的裸DNA进入细菌细胞,2.噬菌体介导的转导:携带抗生素抗性基因的噬菌体感染细菌细胞。3.接合:携带抗生素抗性基因的质粒从供体细菌细胞接合转移至接受体细菌细胞。当将这些抗生素抗性基因导入“免疫的”细胞中时;它预先装备(pre-armed)有编码gRNA-Cas9的CRISPR/Cas基因座,所述gRNA-Cas9找到抗生素抗性基因上的特异性靶序列并将其切割以破坏基因。图3:使有症状的病原体对抗生素重新敏感:I,细菌染色体(B.Chr.)中的CRISPR/Cas基因座,其中框表示靶向每个抗生素抗性基因的不同间隔区序列;II,gRNA-Cas9,其中框表示靶向每个抗生素抗性基因的不同gRNA;III,抗生素;IV,通过切割相应的基因破坏蛋白质合成;V,质粒。此图显示无症状的病原体由于机会性感染而变得有症状。病原体含有提供各种抗生素抗性剂的质粒DNA,如1.外排泵:其能够从细菌细胞中泵出抗生素,2.抗生素降解酶和3.抗生素修饰酶。通过gRNA-Cas9介导的对相应基因的位点特异性切割破坏(X)每种抗生素抗性剂。图4:三种可能的CRISPR/Cas递送途径:I,CRISPR/Cas基因座,其中框表示靶向每个抗生素抗性基因的不同间隔区序列;II,松弛酶:在双链DNA中产生链特异性,序列特异性切口以启动质粒DNA的接合转移的酶;III,噬菌体;IV,质粒;V,双链DNA;VI,单链DNA。此图显示了CRISPR/Cas9表达构建体的三种可能的递送途径。1.转化:通过细胞表面上的DNA受体将携带CRISPR/Cas9的裸DNA进入细菌细胞,随后整合(4)入细菌染色体(B.Chr.),2.噬菌体介导的转导:携带CRISPR/Cas9的噬菌体感染细菌细胞,随后环化(5),然后噬菌体介导的整合(4)入细菌染色体(B.Chr.)。3.接合:携带CRISPR/Cas9的质粒从供体细菌细胞接合转移到受体细菌细胞,随后环化(5)和质粒复制。在所有情况下,CRISPR/Cas9基因座中的框表示靶向每个抗生素抗性基因的不同间隔区序列。图5:双向抗生素抗性传播模型的结构(还参见AmJEpidemiol.2013;178(4):508-520)和抗生素抗性基因灭活疗法的效果:A,抗生素抗性是占优势的;B,抗生素敏感性为占优势的;S,易感,AS,抗生素敏感;AR,抗生素抗性;T,传播;R,恢复,C,转化,CCD,CRISPR/Cas9递送。在抗生素抗性状态中的抗生素抗性基因和在敏感状态中抗生素抗性基因的对应物受到例如gRNA介导的Cas9切割破坏。抗生素抗性基因灭活疗法通过增加从抗生素抗性到敏感性的转化率和降低从抗生素敏感性到抗性的转化率,将双向模型(A)转化为几乎单向的转化模型(B)。每个正方形的面积代表每种状态的群体密度。箭头的宽度与每个参数(传播,恢复和转化)的量值成比例,即在此图中假设抗生素抗性细菌的传播高于抗生素敏感细菌。从对抗生素敏感的病原体的感染的恢复高于对抗生素抗性的病原体的恢复。在抗生素存在下,由于选择压力,抗生素敏感性病原体转化为抗生素抗性病原体远超过从抗性至敏感病原体的反向转化。治疗目的是通过将CRISPR/Cas9导入细菌中来逆转这些转化参数以将抗生素抗性病原体群体驱动至敏感状态。图6:超感染(superinfection)抗生素抗性传播模型的结构和抗生素抗性基因灭活疗法的效果:另外/或者,图5中描绘的双向抗生素抗性传播模型可以通过以下超感染抗生素抗性传播模型描述(还参见AmJEpidemiol.2013;178(4):508-520,如和图5一样)。当抗生素抗性基因受到例如gRNA介导的Cas9切割而破坏时,这允许群体通过超感染状态从抗生素抗性状态转变为抗生素敏感状态。抗生素抗性基因的破坏增加了从抗生素抗性到敏感状态的转化率。此图给出了应用CRISPR-Cas系统之前(图6A)和之后(图6B)的相对群体密度的比较。群体由S,易感;Iw,敏感;Iz,抗性;和Iwz,超感染状态构成。每个圆的面积与每种状态的相对群体密度成比例。在此模拟中,假定每个状态中的初始密度是相同的(各为0.25,由细线圆圈表示)。粗线圆圈代表在平衡状态下的群体密度。CRISPR-Cas系统显然有助于敏感状态中的群体的增加,因此平衡状态下的恢复速率(即S在B中扩大)和减少抗性状态中的群体(即,Iz在B中收缩)。箭头的宽度与每个参数(感染,恢复和状态转化)的量值成比例,即在此图中假定抗生素抗性细菌的感染低于抗生素敏感细菌的。从抗生素敏感性病原体的感染的恢复高于从抗生素抗性病原体的感染的恢复,因为抗生素对敏感性菌株有效。在抗生素存在下,由于抗生素选择压力,抗生素敏感性病原体被转化为抗生素抗性病原体远超过反向转化。治疗的目的是逆转这些转化参数并将抗生素抗性病原体群体驱动至敏感状态。图7:CRISPR基因座的结构:此图显示了包含靶向六个不同区域的六个间隔区的CRISPR基因座。以单顺反子方式从表示为P的相同启动子转录每个crRNA以控制对于每个靶标相同的转录水平。每个crRNA转录物起始于前导序列L并以终止子序列T终止。每个前-crRNA的转录物显示为箭头,并且含有不同间隔区序列的盒通过独特的阴影标示。图8:在bla基因序列上定位短细菌脱靶序列。该图显示了定位到β内酰胺酶基因的细菌短序列(SEQIDNO:3-5,8-14,17-23和26-33)的局部比对。β内酰胺酶序列(SEQIDNO:1-2,6-7,15-16和24-25)显示在每个图的顶部,PAM(原间隔区相邻基序)序列显示为黑色。具有crRNA的碱基配对区域加下划线,细菌基因组上的脱靶种子序列是斜体的。包括PAM序列的脱靶种子序列由两条垂直线指示。β内酰胺酶基因序列上的两个数字是预期的两个碱基位置,其中磷酸二酯键被Cas9切割(参见下面的实施例1)。图9:预测的crRNA二级结构。参考图8和实施例1,显示了使用mFold的crRNA序列预测的二级结构。图9中所示的CR05,CR30,CR70和CR90的序列分别对应于SEQIDNO:34-37。图10:来自pBR322的β-内酰胺酶基因(SEQIDNO:38)上的预期切割位置。与crRNA间隔区序列碱基配对的两个原间隔区序列加下划线(原间隔区链)。两个反原间隔区序列(anti-protospacersequence)(其在原间隔区序列与crRNA间隔区序列碱基配对时被置换)是粗斜体化的(反间隔区链)。相关的PAM序列用带框的小写字母(在原间隔区链上)或带框大写字母(在反原间隔区链上)标示。预期的切割位置由星号标示。前导序列从第1至第69个碱基加下划线,并以灰色突出显示。图11:在0.8%琼脂糖凝胶上电泳分离的DNA产物的照片,显示实施例2中产生的来自三个区域中每一个的PCR扩增子加上EcoRV消化的载体pACYC184。标记的泳道如下:1*-INB(N3232S)1Kb分子标志物,2μg/泳道;2-片段1,tracrRNA-Cas9区域:4758bp;3*-NEB(N3232S)1Kb分子标志物,0.75μg/泳道;4-pACYC184未切割;5-pACYC184EcoRV切割:4245bp;6-pACYC184EcoRV切割:4245bp;7*-NEB(N3232S)1Kb分子标志物,0.12μg/泳道;8-片段2,前导物和第一同向重复区:276bp;9*-INB(N3232S)1Kb分子标志物,0.2μg/泳道;和10-片段3,第二同向重复区:452bp。注意:*通过使用NIHImageJ1.48v软件(http://imagej.nih.gov/ij/)比较合适标志物带的强度来估计每个PCR扩增子的质量。图12:在实施例2中构建的pNB100的质粒图谱。通过SnapGeneviewerver.2.4.3免费版本(http://www.snapgene.com)绘出质粒图谱。两个同向重复(DR)显示为与前导序列的3’末端相邻的窄白色矩形框。图13:照片显示根据本发明的实施方案通过细菌细胞交配(bacterialcellmating)(参见实施例2)的“Nemesis共生活性”(NSA)的结果。左图显示Ap100Cm35中的JA200pNT3xDH5αpNB100,而右图显示Ap100Cm35中的JA200pNT3xDH5αpNB102,两者以5×107个细胞/ml铺板。图14:照片显示了通过质粒转化(参见实施例2),根据本发明另一个实施方案的NSA的结果。左:LBCm35板。菌落1-40是用pNB102转化的DH5αpBR322;菌落45-60是用pNB100转化的DH5αpBR322。所有菌落显示对质粒pNB100和pNB102携带的Cm的抗性;右:LBAp100板。注意,在用携带间隔区(所述间隔区靶向菌株NBEc001中的质粒pBR322上携带的β-内酰胺酶基因)的pNB102转化后,,菌落1-40已经失去ApR,从而证明了Nemesis共生活性。缺乏此间隔区但携带Cas9基因的pNB100不能使β-内酰胺酶基因灭活。图15:显示Ngophi6的噬菌粒。从Ngophi6基因组中删除Ngophi6的ORF11和ORF7。编码序列由指示每个ORF的翻译极性的箭头表示。每个Ngophi6噬菌体ORF至M13的相应基因命名为ORF1(gII),ORF2(gV),ORF4(gVIII),ORFV(gVIII),ORF8(gVI),ORF9(gI)。M13基因命名位于括号中。将推定的左整合位点L=CTTATAT的第二个T改变为A,以产生L’=CATATAT,以避免整合。MCS=多克隆位点。Ngophi6和M13mp18的位置由大的两个空心箭头指示。图16:显示一组间隔区序列(SEQIDNO:39-60),其编码20个指导RNA分子,靶向肺炎克雷伯氏菌(Klebsiellapneumoniae)的NCBIARDB数据库中鉴定的117种不同bla基因(参见实施例5)。鉴定候选间隔区序列以破坏在ARDB数据库中发现的所有肺炎克雷伯氏菌β-内酰胺酶基因。β-内酰胺酶基因序列以关键字肺炎克雷伯氏菌从ARDB数据库收集。除去冗余序列,并且使用网络程序ClustalOmega将独特的序列进行多重序列比对。从每个簇中选择一个规范序列,并收集由网络程序JackLin的CRISPR/Cas9gRNA寻找器(finder)预测的20nt间隔区序列。选择间隔区序列以使在属于相同分支的序列中发现的原间隔区序列的比率最大化。因此,第四列中所示的每个实例间隔区序列都具有破坏第三列中的基因的能力。在该分析中使用的β-内酰胺酶基因是:SHV-a=1,2,2a,5,5a,11,12,14,26,27,28,31,33,38,43,44,48,55,56,60,61,62,71,73,74,75,76,77,78,79,80,81,82,85,89,92,98,99,101,103,106,107,108,109,CTXM-b=1,3,10,12,15,22,32,54,60,62,68,CTXM-c=13,14,16,18,19,24,26,CTXM-d=2,35,59,CTXM-e=26,63,TEM-f=1,1b,3,ESBL,139,KPC-g=1,2,3,4,OKP-h=A11,A12,A16,A17,B13,B-SB613,6,LEN-i=2,17,18,19,20,21,22,24,GES-j=1,3,4,5,VIM-a=1,2,4,12,19,IMP-b=4,8,CMY-a=2,4,25,31,36,LAT-b=1,2,CMY-c=1,8b,10,19,FOX-d=1,5,7,OXA-a=1,30,47,OXA-2,OXA-9,OXA-b=10,17。β-内酰胺抗生素分为四类,即青霉烷类(penams),头孢烯类(cephems),碳青霉烯类(carbapenems)和单环β-内酰胺类(monobactams)。在每个类别下列举一个抗生素名称作为实例。可以打开β内酰胺环的β内酰胺酶由R表示。例如,KPC灭活碳青霉烯。如果想要使细菌对碳青霉烯重新敏感,则应当将间隔区序列5’-TTGTTGCTGAAGGAGTTGGG用于间隔区阵列中以灭活KPC基因。注意,CMY-a的间隔区序列也可用于LAT-b切割。从5'到3'方向显示了间隔区序列的实例。图17:显示了一组间隔区序列(SEQIDNO:61-77),其编码17个引导RNA分子,靶向肺炎克雷伯氏菌的CARD数据库中鉴定的154种不同的bla基因(参见实施例5)。鉴定候选间隔区序列以破坏在CARD数据库中发现的所有肺炎克雷伯氏菌β-内酰胺酶基因。此表是用与图16中的图例中说明的方法相同的方法创建的。间隔区序列的实例从5'到3'方向显示。图18:经修饰的CasDNA结合多肽Cas9R的图谱。Cas9和Tn3解离酶(resolvase)之间的遗传融合。解离酶和Cas9由箭头指示。箭头的方向表示转录极性。Cas9的功能域名显示在Cas9空心箭头下方的框中。此Cas9是内切核酸酶活性缺陷突变体dCas9,其中具有RuvCI域中的氨基酸取代D10A,HNH域中的H840A(如Tsaietal.[2014,NatureBiotechnology32:569–576]所述)。突变体Tn3解离酶(如Proudfootetal.[2011PLoSONE6(4):e19537]所述)通过12聚体多肽接头与此dCas9的N末端融合。这些取代的氨基酸残基中降低重组位点特异性的一些取代的位置由解离酶的N末端域中的短垂直条(残基1-148)指示。这些取代的完整列表是:R2A,E56K,G101S,D102Y,M103I,Q105L,V107F,E132A,L135R。在融合的Cas9区域中:RuvCI,II,III,HNH和PI(PAM相互作用)域是核酸酶域,REC1a和REC1b分别是重复和反重复RNA的识别域。REC2域与原间隔区-gRNA异双链体没有任何接触。四个CRISPR间隔区序列S1,S2,S3和S4在一个CRISPR前导序列的表达下排列,并且需要通过突变体Tn3解离酶引起Cas9R介导的重组事件,这导致靶序列的缺失和重新连接。Tn3R=Tn3分解酶,R=同向重复,L=前导物。图19:示意图,显示了通过gRNA指导的Cas9的解离酶的位点特异性定位。步骤I中的空心箭头是用于灭活的质粒上的靶抗生素抗性基因。每个重组位点A(A1,A2)和B(B1,B2)由gRNA独立识别,并且正确定位的解离酶在紧密邻近处二聚化(步骤II)。将每个重组位点A1+A2和B1+B2中的二聚体四聚化以形成突触(步骤III)。突触复合物(III)扩大,gRNA呈现为标为S1,S2,S3和S4的虚线箭头。大椭圆形代表dCas9,纵向椭圆形是通过接头肽连接的解离酶。白色和灰色纵向椭圆形分别是在重组位点B和A上二聚化的解离酶催化域。垂直箭头标示解离酶在重组位点上的切割位置。细水平平行箭头代表含有重组位点A1+A2的DNA,并且粗水平平行箭头代表含有重组位点B1+B2的DNA。箭头显示DNA序列的3'末端。短黑色箭头是每个PAM序列的位置。图20:示意图,显示了重组位点A1+A2和B1+B2的交换半位点,随后是链解析和密封断裂点。交换,并且连接和解析重组A1和B1的半位点。将靶抗生素基因的区域解析为环状DNA,而将其余的染色体或质粒复制子重新环化(步骤IV)。短黑色箭头是每个PAM序列在解析后的位置。图21:显示了编码8个指导RNA分子的一组间隔区序列(SEQIDNO:78-85),所述指导RNA分子靶向A类基因,SHV-a,CTX-M-b,TEM-c,KPC-d;B类基因VIM-e,IMP-f,NDM-g和D类基因,OXA-48,其中SHV-a=1,1a,2,2a,5,5a,11,12,14,18,20,21,22,23,26,27,28,31,32,33,38,43,44,48,52,55,56,60,61,62,71,73,74,75,76,77,78,79,80,81,82,85,89,92,98,99,100,101,103,106,107,108,109,110,111,121,136,134,137,140,143,144,147,148,149,150,151,152,153,154,155,157,158,159,160,161,162,163,164,165,168,172,173,178,179;CTXM-b=1,3,10,12,15,19,22,32,52,54,59,60,62,68,71,80,81,99,141,147;TEM-c=1,1B,3,139,162,183,192,197,198,209;KPC-d=1,2,3,4,6,7,8,11,12,14,15,16,17;VIM-e=1,2,4,19,26,27,33,34;IMP-f=4,8,32,38;和NDM-g=1,9,10(参见实施例7)。图22显示了给出了在构建质粒pNB200,202,203,104A,104B和108(参见实施例7)中使用的寡核苷酸的序列(SEQIDNO:86-98)的表。图23:实施例7中构建的pNB104A的质粒图谱。通过SnapGeneviewerver.2.4.3免费版本(http://www.snapgene.com)绘出质粒图谱。用BsaI(其限制性位点位于A1和A2中)消化四聚体间隔区多联体(图29A),并连接到pNB202上的BsaI间隔区克隆位点以产生pNB203。显示了pNB104A上的单个启动子和间隔区(6221-7001)。P=启动子,L=前导物,R=同向重复,S=间隔区,T=尾部。连环(concatenated)的间隔区(靶向NDM,IMP,VIM和KPC)位于单个启动子的下游。图24:实施例7中构建的pNB104B的质粒图谱。通过SnapGeneviewerver.2.4.3免费版本(http://www.snapgene.com)绘出质粒图谱。显示了pNB104B上的单个启动子区(6221-6987)。P=启动子,L=前导物,R=同向重复,S=间隔区,T=尾部。连环的间隔区(靶向OXA-48,SHV,TEM和CTX-M)位于来自单个启动子的表达下(arelocateddownstreamofthesinglepromoter)。图25:实施例7中构建的pNB108的质粒图谱。通过SnapGeneviewerver.2.4.3免费版本(http://www.snapgene.com)绘出质粒图谱。用BsaI(其限制性位点位于A1和A2中)消化八聚体间隔区多联体(图29B),并连接到pNB100上的BsaI间隔区克隆位点,以产生pNB108。显示了pNB108上的单个启动子和间隔区(6221-7225)。P=启动子,L=前导物,R=同向重复,S=间隔区,T=尾部。连环的间隔区(靶向NDM,IMP,VIM,KPC,OXA-48,SHV,TEM和CTX-M)位于单个启动子下。图26:在实施例7中构建的pNB200的质粒图谱。通过SnapGeneviewerver.2.4.3免费版本(http://www.snapgene.com)绘出质粒图谱。通过PCR用引物对NB018和NB019从模板pNB100合成双重启动子盒,将扩增子用BbvI消化并连接到pNB100的BsaI位点,以产生pNB100,pNB100的小BsaI片段(来自位置5776-5741)(参见图12)在该过程中被替换。显示了pNB200上的双重启动子和两个间隔区克隆区(6221-7382)。P=启动子,L=前导物,R=同向重复,S=间隔区,T=尾部。限制酶BsaI和SapI分别用于克隆上游和下游间隔区序列。图27:在实施例7中构建的pNB202的质粒图谱。通过SnapGeneviewerver.2.4.3免费版本(http://www.snapgene.com)绘出质粒图谱。用SapI(其限制性位点位于B1和B2中)消化四聚体间隔区多联体(图29A),并连接到pNB200上的SapI间隔区克隆位点,以产生pNB202。显示了pNB202上的双重启动子和间隔区(6221-7329)。P=启动子,L=前导物,R=同向重复,S=间隔区,T=尾部。连环的间隔区(靶向OXA-48,SHV,TEM和CTX-M)位于第二启动子的下游。图28:实施例7中构建的pNB203的质粒图谱。通过SnapGene观察器2.4.3免费版本(http://www.snapgene.com)绘出质粒图谱。用BsaI(其限制性位点位于A1和A2中)消化图29A所示的四间隔区多联体a+b+c+d,并连接到pNB202上的BsaI间隔区克隆位点,以产生pNB203。显示了pNB203上的双重启动子和间隔区(6221-7501)。P=启动子,L=前导物,R=同向重复,S=间隔区,T=尾部。连环的间隔区(靶向NDM,IMP,VIM和KPC)位于第一启动子的下游。连环的间隔区(靶向OXA-48,SHV,TEM和CTX-M)位于第二启动子的下游。图29A:实施例7中的四聚体间隔区连环化。寡聚物相关的数目对应于图22中列出的引物编号。寡聚物分别经由a,c,e和g独特间隔区(I)在26和27,28和34,35和31,32和36之间成对退火,并在单独的管中延伸(II)。来自26和27的二聚体多联体连环间隔区a和b。来自28和34的二聚体多联体连环间隔区b,c和d。来自35和31的二聚体多联体连环间隔区e和f。来自32和36的二聚体多联体连环间隔区f,g和h(II)。连环的二聚体a+b和b+c+d,e+f和f+g+h分别经由b和f间隔区进一步杂交并延伸以使四个间隔区a,b,c和d或e,f,g和h(III)连环。用SapI(其限制性位点位于B1和B2中)消化四聚体间隔区多联体e+f+g+h,,并连接到pNB200上的SapI间隔区克隆位点,以产生pNB202。用BsaI(其限制性位点位于A1和A2中)消化四间隔区多联体a+b+c+d,并连接到pNB202上的BsaI间隔区克隆位点,以产生pNB203。a=用于NDM的20聚体间隔区,b=用于IMP的20聚体间隔区,c=用于VIM的20聚体间隔区,d=用于KPC的20聚体间隔区,e=用于OXA-48的20聚体间隔区,f=用于SHV的20聚体间隔区,g=用于TEM的20聚体间隔区,h=用于CTX-M的20聚体间隔区。图29B:实施例7中的八聚体间隔区连环化。分别用引物对NB026和NB029,NB030和NB033(V)扩增四聚体间隔区多联体a+b+c+d和e+f+g+h,并且经由间隔区d区杂交四多联体,随后延伸以产生八聚体间隔区a+b+c+d+e+f+g+h。将此八聚体用BsaI消化并与BsaI(其限制性位点位于A1和A2中)连接,之后与pNB100上的BsaI间隔区克隆位点连接以产生pNB108。a=用于NDM的20聚体间隔区,b=用于IMP的20聚体间隔区,c=用于VIM的20聚体间隔区,d=用于KPC的20聚体间隔区,e=用于OXA-48的20聚体间隔区,f=用于SHV的20聚体间隔区,g=用于TEM的20聚体间隔区,h=用于CTX-M的20聚体间隔区。图30:照片,显示通过细菌细胞交配根据本发明的实施方案的“Nemesis共生活性”(NSA)的结果(参见实施例7)。图30A,左上图显示Ap100Cm35中的JA200pNT3xDH5αpNB100,而右上图显示Ap100Cm35中的JA200pNT3xDH5αpNB102。图30B显示NCTC13440xDH5αpNB100,左上图和NCTC13353xDH5αpNB100,右上图;和NCTC13440xDH5αpNB104A,左下图和NCTC13353xDH5αpNB104B,右下图,全部在Ap100Cm35中。图30C显示JA200pNT3xDH5αpNB100(5-),NCTC13440xDH5αpNB100(2-)和NCTC13353xDH5αpNB100(4-),左上图;和JA200pNT3xDH5αpNB108(5/8),NCTC13440xDH5αpNB108(2/8)和NCTC13353xDH5αpNB108(4/8),右上图和JA200pNT3xDH5αpNB100(5+),底图,都在Ap100Cm35中。如本文中使用,术语“抗生素”是指由微生物产生的对其它微生物生长拮抗的经典抗生素,并且更一般地还涵盖能够杀死或抑制微生物生长的抗微生物剂,包括化学合成的形式和天然存在的抗生素的变体。术语“充分互补”是指间隔区序列和靶序列的序列同一性使得包含间隔区序列的RNA引导分子能够与靶序列杂交,优选特异性和选择性杂交,从而使得经由本文中所述的CRISPR/Cas系统灭活包含靶序列的抗生素抗性基因。例如,间隔区序列可以在其整个长度上与靶序列具有至少60%,65%,70%,75%,80%,85%,90%,95%,96%,97%,98%,99%或100%的序列同一性。如本文中使用的术语“功能等同物”是指能够具有与CasDNA结合多肽(或如本文中使用,Cas核酸结合多肽)相同活性的多肽。“功能等同物”可以具有与CasDNA结合多肽相同的定性生物学性质。“功能等同物”包括但不限于天然CasDNA结合多肽及其片段的片段或衍生物,条件是等同物与相应的天然序列多肽具有共同的生物学活性。尽管结构同一性不是常见生物活性所必需的,但在一个方面,功能等同物可以在其整个长度上与CasDNA结合多肽,例如Cas9具有至少50%,55%,60%,65%,70%,75%,80%,85%,90%,95%,96%,97%,98%,99%或100%的序列同一性(Ferrettietal,2001,PNAS,98No.8:4658-4663,GeneID:901176,Cas9GI:15675041;SEQIDNO:99)。术语“CasDNA结合多肽”涵盖全长Cas多肽,Cas多肽的酶活性片段和Cas多肽或其片段的酶活性衍生物。Cas多肽或其片段的合适衍生物包括但不限于Cas蛋白或其片段的突变体,融合体,共价修饰。术语“经修饰的”CasDNA结合多肽涵盖如上所定义的CasDNA结合多肽,除了多肽的DSB催化功能由于例如存在重组酶催化域而替代为DNA密封功能。本文描述了此类修饰的CasDNA结合多肽的其它特征。核苷酸或氨基酸序列之间的序列同一性可以通过比较序列的比对来确定。当在比较的序列中的等同位置被相同的碱基或氨基酸占据时,则分子在该位置是相同的。将比对作为同一性的百分比评分是由比较的序列共享的位置处的相同氨基酸或碱基的数目的函数。当比较序列时,最佳比对可能需要将缺口导入一个或多个序列以考虑序列中可能的插入和缺失。序列比较方法可以采用缺口罚分,使得对于在比较的序列中相同数量的相同分子,具有尽可能少的缺口(反映两个比较序列之间的较高相关性)的序列比对将实现比具有许多缺口的比对更高的评分。考虑到缺口罚分,计算最大百分比同一性涉及产生最佳比对。用于进行序列比较的合适的计算机程序在商业和公共部门中广泛可用。实例包括MatGat(Campanellaetal.,2003,BMCBioinformatics4:29;程序可获自http://bitincka.com/ledion/matgat),Gap(Needleman&Wunsch,1970,J.Mol.Biol.48:443-453),FASTA(Altschuletal.,1990,J.Mol.Biol.215:403-410;程序可获自http://www.ebi.ac.uk/fasta),ClustalW2.0和X2.0(Larkinetal.,2007,Bioinformatics23:2947-2948;程序可获自http://www.ebi.ac.uk/tools/clustalw2)和EMBOSS成对比对算法(Needleman&Wunsch,1970,同上;Kruskal,1983,In:Timewarps,stringeditsandmacromolecules:thetheoryandpracticeofsequencecomparison,Sankoff&Kruskal(编),pp1-44,AddisonWesley;程序可获自http://www.ebi.ac.uk/tools/emboss/align)。所有程序都可以使用缺省参数运行。例如,可以使用EMBOSS成对比对算法的“针”方法进行序列比较,所述方法当在其全长上考虑时确定两个序列的最佳比对(包括缺口)并提供百分比同一性评分。氨基酸序列比较(“蛋白质分子”选项)的缺省参数可以是缺口延伸罚分:0.5,缺口开放罚分:10.0,矩阵:Blosum62。核苷酸序列比较(“DNA分子”选项)的缺省参数可以是缺口延伸罚分:0.5,缺口开放罚分:10.0,矩阵:DNAfull。在本发明的一个方面,可以在参照序列的全长上进行序列比较。如本文中使用,术语“基因”是指编码多肽或转录非编码的功能性RNA的DNA序列。术语“抗生素抗性基因”涵盖编码赋予抗生素抗性的产物或转录功能性RNA的基因或其编码部分。例如,抗生素抗性基因可以是有助于上述四种抗性机制中的任一种的基因或其编码部分。抗生素抗性基因可以例如编码(1)降解抗生素的酶,(2)修饰抗生素的酶,(3)泵,如流出泵,或(4)抑制抗生素的效应的突变靶标。术语“多核苷酸”是指任何长度的核苷酸的聚合形式,例如RNA(如mRNA)或DNA。特别对于寡核苷酸标志物,该术语还包括已知类型的修饰,例如本领域已知的标记物,甲基化,“帽”,用类似物取代一个或多个天然存在的核苷酸,核苷酸间修饰,如具有不改变连接的那些修饰,例如磷酸甲酯,磷酸三酯,氨基磷酸酯,氨基甲酸酯等,和具有带电荷的连接的那些修饰。如本文中使用,术语“多肽”是指氨基酸的聚合物。该术语不是指聚合物的特定长度,因此肽,寡肽和蛋白质包括在多肽的定义内。术语“多肽”可以包括表达后修饰,例如糖基化,乙酰化,磷酸化等。包括在“多肽”的定义中的是例如含有一个或多个氨基酸类似物(包括例如非天然氨基酸)的多肽,具有取代的连接的多肽以及本领域中已知的其它修饰(天然存在和非天然存在)。术语“微生物”包括原核生物,如细菌和古细菌(例如,属于阔古细菌界(Euryarchaeota)和圆齿古细菌界(Crenarchaeota)的那些古细菌)。细菌包括革兰氏阳性菌和革兰氏阴性菌二者。某些种类的临床上重要的致病性真菌包括在微生物的定义中,例如假丝酵母属(Candida),曲霉属(Aspergillus),隐球菌属(Cryptococcus),组织胞浆菌属(Histoplasma),肺囊虫属(Pneumocystis)和葡萄状穗霉属(Stachybotrys)的成员。本发明的各个方面在附图中示出并在下面描述。在本发明中,使用包含本发明的重组多核苷酸的噬菌体或接合性质粒的优点在于,各自充当在感染噬菌体后或在质粒接合后的“特洛伊木马(Trojanhorse)”,导致将“刺客构建体”插入靶细菌或其它微生物中。刺客构建体一旦插入靶微生物中,也提供了细胞针对含有抗生素抗性基因的质粒的未来到达的免疫(参见图2),另外,当然破坏已经存在的此类基因(参见图1和图3)。然后,刺客构建体开始进行抗生素抗性基因的降解过程。如果由本发明的CasDNA结合蛋白创建的DSB破坏携带此类抗生素抗性基因的复制子,则具有抗生素抗性基因的微生物可以被刺杀者直接杀死。如果微生物幸免于DSB,则抗性基因将被灭活,然后可以现在对微生物致敏的抗生素治疗患者。重要的是,应该没有作用于此事件的直接选择压力或有减少的直接选择压力,若随后用抗生素治疗患者的话并且直到随后用抗生素治疗患者。因此,病原性细菌或其它微生物应当具有对噬菌体(或质粒)抗性较少的直接选择或没有直接选择,因此没有或很少建立“进化性军备竞赛(evolutionaryarmsrace)”–这有时是噬菌体直接作为杀细菌剂的已知用途的重大限制性特征。在DSB诱导的微生物杀伤增加对噬菌体或接合质粒递送剂的抗性的选择压力的情况下,可以通过例如使用如本文中定义的修饰的CasDNA多肽来减轻该问题。本发明提供用于口服,局部和益生性,膳食补充剂递送的潜在药剂以及用于沉默灭活病原性细菌或其它微生物中的抗生素抗性基因的流行病学工具(参见图3)。计划进行手术或在医院中的其它治疗的患者完全可以在医院收治前用携带靶向抗生素抗性基因的CRISPR/Cas9(或其它)刺客构建体的重组噬菌体进行预防性处理。以这种方式,在预期可能发生需要用抗生素治疗的任何手术后感染的情况下,可以直接杀死存在于它们的微生物组(microbiome)中的病原体或清除抗生素抗性基因。因此,本发明提供了用于沉默灭活病原菌中的抗生素抗性基因的流行病学工具。为了实现本发明的例示,可以构建靶向所选抗生素的一组CRISPR/Cas9刺客。变量是噬菌体(在本文中也同义简称为“噬菌体”)或接合质粒或DNA递送剂:可以开发对一批重要的细菌病原体特异的一批噬菌体和质粒药剂。需要修饰单一概括(singlegeneralised)的噬菌体或质粒递送剂以靶向不同细菌病原体的程度取决于参与噬菌体生命周期的噬菌体蛋白质或质粒生物学和致病性细菌靶标的相互作用。可以开发感染宿主并成为原噬菌体而变得休眠的溶原性噬菌体,以及复制但不杀死宿主的非毒力噬菌体。关于溶原性噬菌体特异性,仅需要噬菌体的溶原性生命周期,因此需要(i)噬菌体进入细菌细胞和(ii)其随后整合到靶染色体中的附着位点所涉及的特异性。可以不需要整合,并且可以通过使用噬菌体递送质粒替换,所述质粒然后可以例如通过cre-lox重组切割,并且在细胞中独立地复制。可以使用非毒力M13噬菌体及其衍生物。可以保留溶原噬菌体中的功能裂解循环,使得在溶原化细菌中进入裂解循环中的低水平将产生新的噬菌体,其可以随后继续感染其它细菌(致病性细菌或非致病性细菌,以提供免疫)。从噬菌体在致病性群体中的流行病学传播的观点来看,这可能不是必需的,并且单一的初始感染可能就足够了。这可以通过实验测试。鉴定了用于有效感染和适当的感染复数的最佳条件。实验可以在模型系统中进行(参见图1)。例如,大肠杆菌携带多种药物抗性,例如,对氨苄青霉素,氯霉素和卡那霉素,或针对抗性普遍的常用抗生素。还可以用靶标病原体,例如肺炎克雷伯氏菌碳青霉烯酶(“KPC”)进行实验。KPC造成英国医院中的感染和死亡。噬菌体递送:噬菌体递送系统(参见例如图2,途径2)可以适用于治疗由抗生素抗性细菌感染引起的创伤和烧伤。可以使用通过两种不同的大肠杆菌溶原性噬菌体:噬菌体λ和噬菌体Mu递送靶向这些抗生素的刺客构建体。噬菌体λ在宿主染色体中具有称为λ附着位点的特异性整合位点。噬菌体Mu能够随机整合到宿主基因组中并且具有不需要细菌中的特定附着位点的优点。噬菌体也可用于递送含有本发明的重组多核苷酸的质粒复制子,其如上讨论在感染后通过cre-lox重组,或通过使用作为附加体复制的噬菌体P1切除。成功感染中涉及的特异性是对细菌细胞表面上的膜蛋白的识别。在λ的情况下,这是麦芽糖通透酶蛋白,其将麦芽糖运输到细菌细胞中。在噬菌体Mu的情况下,受体是LPS。感染携带F-因子质粒的大肠杆菌细胞的雄性特异性噬菌体M13也可以用于递送靶向一种或多种抗生素抗性基因的CRISPR/Cas9构建体(或本发明的其它刺客构建体)。M13是一个充分研究的噬菌体,其复制,但不杀死细菌宿主。评估在通过重组噬菌体的溶原化或者携带针对这些抗生素抗性基因的刺客构建体的M13重组体的感染后对靶抗生素的抗性的成功消除。可以进行对携带对相同抗生素的抗性的其它密切相关的肠杆菌科(Enterobacteriaceae)的研究。在上文讨论的肺炎克雷伯氏菌外,这些可以包括鼠伤寒沙门氏菌(Salmonellatyphimurium)和弗氏志贺氏菌(Shigellaflexneri)。为了做到这点,用编码噬菌体的尾纤维蛋白的不同基因构建修饰的噬菌体,以使其与存在于细菌表面上的不同受体相互作用。可以使用溶原性噬菌体或雄性特异性噬菌体,如M13,它们是这些细菌的天然宿主。还可以利用相变异(phasevariation)的现象,其中噬菌体能够通过以低频率倒位DNA区段以控制基因表达并表达替代的尾纤维蛋白转变其宿主特异性。并且,以这种方式开发通用的噬菌体递送系统。例如噬菌体Mu携带可倒转的G区段(由Mu编码的位点特异性重组酶,Gin调节),产生两种噬菌体,能够感染大肠杆菌K12的G(+)和能够感染阴沟肠杆菌(Enterobactercloacae),弗氏柠檬酸杆菌(Citrobacterfreundii),粘质沙雷氏菌(Serratiamarcescens)和胡萝卜软腐欧文氏菌(Erwiniacarotovora)的G(-)。另一个实例是淋病奈瑟氏球菌丝状噬菌体NgoPhi6,或其修饰形式。天然(野生型)噬菌体具有革兰氏阴性菌的广泛的宿主范围(α,β和γ蛋白菌),参见Piekarowiczetal.(2014J.Virol.88:1002-1010)。天然噬菌体不是裂解性的,而是溶原性的,并且在宿主基因组上具有整合位点。在本发明的一个方面,除去噬菌体整合到宿主染色体中的能力,并且工程化改造噬菌体以独立地复制。用L’,CATATAT替换左侧整合位点L,CTTATAT将消除整合事件。为了在染色体外复制噬菌体基因组,M13ori和M13基因II可以用于模拟M13噬菌体复制。可以保持最小的修饰以维持受感染细菌的后代产生的能力。虽然最大包装尺寸是未知的,但大约12Kb的噬菌体DNA被实验证明是可包装的,并且由感染这种噬菌体的细菌产生的噬菌体后代是可感染的。噬菌体的长度是约4微米,其是M13噬菌体颗粒的4.4倍,并且指示比M13更长的DNA的较高的包装容量-即假定DNA的包装大小与噬菌体颗粒长度的大小成比例,NgoPhi6噬菌体的DNA包装容量将为4微米/0.9微米x6407nt=28475nt,大得足以包装噬菌体基因组和本发明的重组多核苷酸,包括例如CRISPR-Cas9构建体(如果需要的话)。噬菌体载体(噬菌粒)的示例结构满足要求-参见图15。通过接合质粒的递送:可以评价通过选择的宽宿主范围接合质粒递送靶向这些抗生素的刺客构建体(参见图4,途径3)。这里,可以通过良性非致病宿主递送质粒。本文的申请可以作为益生菌用于GI道并且可以预防性使用。在这方面,由CasDNA结合蛋白引起的DSB的可能致死作用是不关注的,因为质粒不会转移到受体。类似地,可以评价在携带刺客货物的质粒接合转移后成功消除对三种靶抗生素的抗性。通过转化线性DNA的递送:也可以进行通过线性双链DNA转化递送靶向这些抗生素的刺客构建体(参见图4,途径1)。这里,DNA经由来自细菌周围环境的细菌表面上的DNA受体递送。实施例通过以下非限制性实施例例示本发明。实施例1实施例1的目的是通过在大肠杆菌的非致病性细菌菌株中导入CRISPR/Cas9构建体来显示抗生素功效的复苏(resurrection)的概念证据(proof-of-concept)。CRISPR/Cas9构建体直接从分离自酿脓链球菌(Streptococcuspyogenes)菌株SF370的基因组DNA制备,所述菌株SF370获自:http://www.straininfo.net/strains/117800/browser;jsessionid=A07A638D6D2472EA2FEDBD3A1928F347.straininfo2。这里,通过PCR,使用序列特异性DNA引物,提取Cas9编码区加邻近调节区和编码tracrRNA的DNA,并克隆到合适的质粒克隆载体中。类似地,通过PCR提取CRISPR阵列的单位重复(其包含在间隔区序列侧翼的同向重复),并修饰以通过克隆位点替换间隔区序列,从而允许随后导入设计为靶向所选择的靶DNA区域,如抗生素抗性基因的间隔区序列。对携带期望的重组体的细菌转化体具有阳性选择是有用的,其中此类间隔区序列已经成功克隆到克隆位点中。附录3给出了此类阳性选择的例子。或者,从pCas9质粒获得等同的CRISPR/Cas9基因靶向构建体,虽然缺乏对重组体的阳性选择,例如可从Addgene获得:http://www.addgene.org/42876/。此质粒携带Cas9基因加上编码具有独特克隆位点的tracrRNA和CRISPR阵列的DNA序列,以便导入期望的间隔区序列以靶向给定的DNA序列而被Cas9内切核酸酶切割。为了举例的目的,下面描述了此种现有的pCas9质粒的用途。实施例1显示携带赋予对β-内酰胺抗生素,氨苄青霉素的抗性的β-内酰胺酶(bla)基因的细菌在导入靶向bla基因的修饰的CRISPR/Cas9构建体后对氨苄青霉素变得敏感:CRISPR/Cas9/反bla构建体。材料和方法A.细菌菌株,质粒和噬菌体1.细菌菌株:DH5α是(F-endA1glnV44thi-1recA1relA1gyrA96deoRnupGΦ80dlacZΔM15Δ(lacZYA-argF)U169,hsdR17(rK-mK+),λ–)。JM109是(endA1glnV44thi-1relA1gyrA96recA1mcrB+Δ(lac-proAB)e14-[F'traD36proAB+lacIqlacZΔM15]hsdR17(rK-mK+))。2.质粒:pUC18(OripMB1);pCas9(基于pACYC184的载体,具有Orip15A,CRISPR基因座加Cas9基因,CMR);pCRISPR(OripMB1,CRISPR,KnR)。3.噬菌体:M13mp18在实施例1A中,在一个菌株大肠杆菌DH5α中并携带pBR322(一种中等拷贝质粒或备选地低拷贝质粒),通过在质粒pCas9中裸DNA转化递送CRISPR/Cas9/反-bla(anti-bla)构建体,命名为pCas9::反-bla。已经存在的质粒pBR322或低拷贝质粒表达源自细菌转座子Tn3的β-内酰胺酶;pBR322还携带对四环素的抗性。由于pCas9::反-bla携带对氯霉素的抗性,通过对生长培养基添加氯霉素来实现维持pCas9::反-bla的细胞的选择。在分开的阴性对照实验中,将具有pBR332的DH5α细胞或低拷贝质粒用pCas9(其是在所有方面与pCas9::反-bla一样,但缺乏作为靶向bla基因的间隔区序列的反bla的质粒:预测pCas9在缺乏反bla中不能攻击和灭活bla基因。再次,通过氯霉素的存在维持pCas9。可以通过作为头孢菌素的显色衍生物的头孢硝噻(nitrocefin)来检测β-内酰胺酶活性,当β-内酰胺环被水解时,完整的头孢硝噻的紫外吸收被转移到约500nm,这允许目测检测β内酰胺酶的存在。让对氯霉素有抗性的细菌总数为N0,并且β-内酰胺酶抗性细菌的数为Nr。Nr的生长在存在头孢硝噻的情况下表现为红色菌落。让β内酰胺酶敏感细菌的数量为Nw。Nw菌落的生长在存在头孢硝噻的情况下表现为白色菌落。因此,通过测量白色菌落的分数(即Nw/N0=(N0-Nr)/N0)计算CRISPR/Cas9/反bla介导的bla基因灭活效率的功效。或者,可以看到β内酰胺酶活性直接攻击含有氨苄青霉素的LB平板上的细菌。对氯霉素有抗性的总细菌是N0,氨苄青霉素抗性细菌是Na,然后可以通过测量氨苄青霉素敏感性菌落的分数1-Na/N0来定义CRISPR/Cas9/反bla介导的bla基因灭活效率。在实施例1B中,使用M13噬菌体递送系统将CRISPR/Cas9/反-bla构建体导入到表达来自驻留质粒的β内酰胺酶基因的大肠杆菌菌株JM109中。如下制备M13::CRISPR/Cas9/反bla噬菌体重组体:通过用SalI和XbaI消化该构建体,从pCas9::反bla分离CRISPR/Cas9/反bla构建体。消化的片段大小为5796bp。将该片段克隆在M13mp18RFI的SalI(GTCGAC)和XbaI(TCTAGA)位点处。含有CRISPR/Cas9/反-bla构建体的M13mp18的整个大小为13035bp。将该M13重组噬菌体DNA转化至大肠杆菌JM109(携带F'质粒的细菌菌株),纯化从细菌中挤出的重组M13噬菌体,并将其用于将CRISPR/Cas9构建体导入含有pBR322的大肠杆菌JM109。作为阴性对照,通过限制性酶SalI和XbaI从pCas9制备缺少反bla区域的等同的M13::CRISPR/Cas9噬菌体重组体,并且将扩增子克隆在M13mp18RF1的SalI和XbaI位点处。当M13感染细菌细胞时,它不会杀死它们,但噬菌体DNA在细胞内复制并表达噬菌体基因。因此,使用M13::CRISPR/Cas9/反bla噬菌体重组体的M13噬菌体感染应该导致bla基因的灭活,相反,在阴性对照中,导致缺少反-bla区的M13::CRISPR/Cas9噬菌体重组体的感染。由于M13噬菌体感染减缓细胞生长的速率,此时在细胞的菌苔上培养的感染细胞产生缓慢生长的感染细胞的浑浊噬菌斑。如果将头孢硝噻添加到生长培养基中,则当挑选噬菌斑并作为菌落培养以除去背景菌苔时,通过测量白色菌落与红色红色菌落的分数来计算CRISPR/Cas9/反bla介导的bla基因灭活效率的功效。也可以挑取噬菌斑,并铺板到具有/不含氨苄青霉素的LB平板上,以评分将产生的氨苄青霉素敏感性菌落与氨苄青霉素抗性菌落的比率。B.从靶序列中选择间隔区序列我们可以使用来自BlueHeron的引导RNA靶设计工具(参见https://wwws.blueheronbio.com/external/tools/gRNASrc.jsp)从靶标中选择间隔区序列。此程序简单地返回在3’末端具有适当的PAM序列(原间隔区相邻基序)的20nt间隔区序列和GC含量。它不考虑二级结构稳定性和序列特异性。二级结构预测和特异性搜索是手动执行的。我们从上述程序中获得的候选序列中选择实际的间隔区序列,其应该满足以下两个标准:1)形成稳定的crRNA二级结构的低趋势,2)在宿主基因组DNA上没有靶DNA。可能很难找到满足标准2)(No.2)的独特序列。考虑来JineketalScience337,816(2012)中图3E的不匹配的靶标数据,放松标准2)以允许匹配的序列多达靶序列中的第12个核苷酸位置(第一个核苷酸位置从刚好与PAM序列邻近起计数)。换言之,当靶序列的第一个12聚体间隔区序列与3'末端中的crRNA间隔区序列的12聚体序列完全匹配,但序列的其余部分不匹配时,假定靶dsDNA不被切割。沿着大肠杆菌K12基因组序列的原间隔区序列的特异性检查通过BLAST进行。针对主题序列大肠杆菌菌株亚株MG1655搜索bla序列(http://www.ncbi.nlm.nih.gov/nucleotide/556503834?report=genbank&log$=nuclalign&blast_rank=1&RID=JUYB76FX014),并且任何匹配的染色体序列中的每一个针对bla序列进行定位以计数来自规范PAM(NGG)序列的种子序列。二级结构可以通过mFold预测(http://mfold.rna.albany.edu/?q=mfold/RNA-Folding-Form)以选择其G尽可能大的序列,优选对于crRNA间隔区二级结构是阳性的。以下是从bla序列中选择合适的间隔区序列的方式。表1:候选反原间隔区序列。使用来自BlueHeron的引导RNA靶设计工具(https://wwws.blueheronbio.com/external/tools/gRNASrc.jsp)获得来自bla基因的所有靶序列。返回98个目标候选序列。将细菌脱靶染色体短相似序列相对于bla基因定位,随后计数来自规范PAM序列的种子序列。选择种子序列号小于8且吉布斯自由能相对较大的序列。所选靶序列的性质的概述显示在表1中。该表还显示pCas9和pBR322上的脱靶序列的种子序列的核苷酸长度。用于间隔区序列的寡聚物序列以下四个间隔区序列是靶向作为宿主菌株的大肠杆菌中的pBR322上的β-内酰胺酶的crRNA产生盒,其满足上述两个标准。在β-内酰胺酶基因上,crRNACR05切割第762位碱基C和第763位碱基C之间的磷酸二酯键,CR30切割第198位碱基G和第199位碱基A之间的磷酸二酯键,CR70切割第575位碱基T和第576位碱基A之间的磷酸二酯键,并且CR90切断第221位碱基T和第222位碱基A之间的磷酸二酯键。用于β-内酰胺酶基因上的单一靶标的衔接头。CR055’-AAACTAGATAACTACGATACGGGAgSEQIDNO:115atctattgatgctatgccctcAAAA-5’SEQIDNO:116CR305’-AAACACTTTAAAAGTGCTCATCATgSEQIDNO:117tgaaattttcacgagtagtacAAAA-5’SEQIDNO:118DraICR705’-AAACACGTTGCGCAAACTATTAACgSEQIDNO:119tgcaacgcgtttgataattgcAAAA-5’SEQIDNO:120AclICR905’-AAACACTTTTAAAGTTCTGCTATGgSEQIDNO:121tgaaaatttcaagacgataccAAAA-5’SEQIDNO:122DraI用于β-内酰胺酶基因上的双重靶标的衔接头。内部同向重复序列是斜体化并且加下划线的。每个寡聚物的5'端被磷酸化并准备在BsaI位点克隆。六碱基切割剂限制性内切核酸酶位点是加下划线的,其可用于筛选重组体。我们也可以使用盒寡聚物之一作为引物,连同对于质粒载体的另一种独特引物通过PCR一起筛选重组体。附录1pCas9质粒序列(SEQIDNO:125)Cas9基因,CRISPR表达基因座和tracrRNA(均来自酿脓链球菌)tracrRNA的反向互补体为小写字母,Cas9编码序列(SEQIDNO:126)是框示的,并且CRISPR中的同向重复序列为粗体。通过神经网络算法(http://www.fruitfly.org/seq_tools/promoter.html)预测启动子序列。以粗斜体黑色突出显示的两个独特位点SalI(GTCGAC)和XbaI(TCTAGA)用于分离用于克隆到M13mp18中的CRISPR/Cas9构建体。pACYC184主链序列为斜体。或者,附录1的信息如下呈现:pCas9质粒序列(SEQIDNO:125)Cas9基因,CRISPR表达基因座和tracrRNA(均来自酿脓链球菌)主链载体pACYC184序列是斜体的,序列位置从加有下划线的EcoRI位点的G起编号。tracrRNA的反向互补体为斜体化粗体,从1844到1929定位,Cas9起始和终止密码子用粗体三字母标示,从核苷酸号2225开始和在6331结束,然后是由斜体化粗体字母标示的前导序列6389-6483,第一,第二和第三同向重复序列加下划线,在间隔区克隆区位于其中的第一和第二同向重复之间。此间隔区克隆区含有由粗斜体字母5’-GAGACC-3’和5’-GGTCTC-3’标示的两个反向BsaI位点,用于分别创建5’四碱基突出间隔区克隆位点5’-GTTT-3’和5’-TTTT-3’。如上预测启动子序列,由Cas9的正向启动子和前导序列的小写字母和tracrRNA的反向启动子的斜体小写字母标示,推定的转录起始位点由粗体大写字母标示。以粗斜体黑色突出显示的两个独特位点SalI(GTCGAC)和XbaI(TCTAGA)用于分离用于克隆到M13mp18中的CRISPR/Cas9构建体。附录2pCas9上间隔区克隆位点的修饰此系统利用下述事实,即小基因(minigene)的表达产生某种寡肽,其当诱导表达时消耗宿主细胞中的tRNA合并物,这导致蛋白质合成的破坏(TensonT,Vega-HerreraJ,KlossP,GuarnerosG,MankinAS.J.Bacteriol.1999;181:1617-1622)。将此构思应用于克隆间隔区序列:当插入间隔区序列时,这中断了小基因,并且不发生寡肽表达,因此tRNA不被消耗,因此只有含有具有插入物的质粒的细菌能在诱导的表达的条件下存活;而含有无插入物的质粒的细菌不能生长。以下(SEQIDNO:129)是序列结构。为了构建此结构,细菌应该组成性表达lacI阻抑物以关闭小基因转录。重组选择应在IPTG存在下进行以诱导转录。BsaI识别位点加下划线。lac操纵基因3和1是粗斜体的。Tac启动子为粗体字母,启动子序列中的-35和-10区域加下划线,Shine-Dalgarno序列为框示的,第一个二肽为粗体M和R两者,后面是三个连续终止密码子--斜体的TAA。采用色氨酸终止子信号序列,以斜体化字母标示。附录3使用CR90间隔区序列在靶序列上预期的切割位点第一加工事件使用tracrRNA的加下划线的序列与以大写字母指示的前-crRNA的同向重复区杂交。细菌RNA酶III在指定位置“|”处切割双链RNA区,即第一个加工事件。第一个加工事件后的结构第一个加工事件也可以描述如下:由小写字母指示的tracrRNA与由大写粗体黑色字母指示的前-crRNA的同向重复区杂交。细菌RNA酶III在具有箭头的指示位置处切割双链RNA区,其是第一个加工事件。在第一和第二同向重复中的第22和第23个碱基之间的磷酸二酯键被切割。斜体间隔区序列CR90是加框的。第一个加工事件后的结构第二个加工事件第二切割点(*)距离间隔区序列的3'末端约20nt。“注意,第二个加工事件发生在重复内的第一个切割的特定距离处。考虑到间隔区序列彼此不相同,因此可能的是,间隔区内的第二个加工事件是距离依赖性的而不是序列依赖性的”(参见Nature.Mar31,2011;471(7340):602-607中的补充图2图注)。尽管上述提及的文章没有具体规定涉及此第二加工事件的酶和/或机制,但是很可能Cas9参与此第二切割。考虑到间隔区序列内的此切割位置,整个间隔区序列不促成靶识别,相反,仅20nt间隔区序列用于杂交。第二加工事件也可以描述如下:在上图中,由距离间隔区序列的3'末端大约20nt的箭头指示的第二切割点。适用以上引自Nature的注释和我们的评论。斜体间隔区序列CR90在框中。第二个加工事件后的结构靶DNA切割显示了含有CR90的靶标β-内酰胺酶DNA序列的部分。crRNA与其靶区域的互补序列杂交,Cas9切割点用点“。”表示,PAM序列tgg在框中表示。靶DNA切割的备选描述如下所示。这里,含有CR90原间隔区序列的靶β-内酰胺酶DNA序列的部分以小写粗体黑色字母显示。crRNA与原间隔区链的互补序列杂交,Cas9切割点以点箭头标示,并且PAM序列tgg加下划线。与原间隔区DNA的退火和切割点实施例2以下实验描述了为了证明CRISPR-Cas9系统可以用于灭活细菌中的抗生素抗性而进行的一些概念验证实验。它们描述了实施例中描述的通常适用的DNA盒的构建,用于递送CRISPR-Cas9系统加上携带靶向抗生素抗性基因的间隔区序列的衍生物,通过裸DNA转化和噬菌体感染递送,并且用于证明抑制通过质粒接合的抗生素抗性扩散。pNB100的构建pNB100是在大肠杆菌中表达CRISPR-Cas9系统的载体,具有适当的独特限制性位点BsaI,以在CRISPR基因座中的两个同向重复序列之间克隆任何期望的间隔区序列。载体的主链衍生自pACYC184,并且将CRISPR-cas9基因座插入载体的EcoRV位点中。通过PCR从购自ATCC的酿脓链球菌菌株SF370的基因组DNA中扩增CRISPR-cas9基因座的三个区域,并通过Gibson装配(GibsonDG,et.al.NatureMethods2009;6:343-345)与反应中的pACYC184载体一起装配。通过Sanger测序验证最终构建体的序列。使用携带靶向细菌转座子Tn3和Tn1的β内酰胺酶基因的间隔区序列的pNB100衍生物pNB102来证实CRISPR-Cas9活性。三个区域的扩增序列和凝胶上的扩增子图像以下序列是通过PCR扩增的三个区域。下划线序列是模板特异性引物序列,粗体字母是用于Gibson装配的重叠序列。1.片段1(SEQIDNO:140),tracrRNA-cas9:扩增子长度=4758bp在酿脓链球菌SF370基因组DNA上,正向引物为854170至854193,而反向引物为858867至858848。CCAGAAGTCTTTTTCTTGCACTGTTTCCTTTTCTTTATGATAGTTTACGAAATCATCCTGTGGAGCTTAGTAGGTTTAGCAAGATGGCAGCGCCTAAATGTAGAATGATAAAAGGATTAAGAGATTAATTTCCCTAAAAATGATAAAACAAGCGTTTTGAAAGCGCTTGTTTTTTTGGTTTGCAGTCAGAGTAGAATAGAAGTATCAAAAAAAGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATGCTGTTTTGAATGGTTCCAACAAGATTATTTTATAACTTTTATAACAAATAATCAAGGAGAAATTCAAAGAAATTTATCAGCCATAAAACAATACTTAATACTATAGAATGATAACAAAATAAACTACTTTTTAAAAGAATTTTGTGTTATAATCTATTTATTATTAAGTATTGGGTAATATTTTTTGAAGAGATATTTTGAAAAAGAAAAATTAAAGCATATTAAACTAATTTCGGAGGTCATTAAAACTATTATTGAAATCATCAAACTCATTATGGATTTAATTTAAACTTTTTATTTTAGGAGGCAAAAATGGATAAGAAATACTCAATAGGCTTAGATATCGGCACAAATAGCGTCGGATGGGCGGTGATCACTGATGAATATAAGGTTCCGTCTAAAAAGTTCAAGGTTCTGGGAAATACAGACCGCCACAGTATCAAAAAAAATCTTATAGGGGCTCTTTTATTTGACAGTGGAGAGACAGCGGAAGCGACTCGTCTCAAACGGACAGCTCGTAGAAGGTATACACGTCGGAAGAATCGTATTTGTTATCTACAGGAGATTTTTTCAAATGAGATGGCGAAAGTAGATGATAGTTTCTTTCATCGACTTGAAGAGTCTTTTTTGGTGGAAGAAGACAAGAAGCATGAACGTCATCCTATTTTTGGAAATATAGTAGATGAAGTTGCTTATCATGAGAAATATCCAACTATCTATCATCTGCGAAAAAAATTGGTAGATTCTACTGATAAAGCGGATTTGCGCTTAATCTATTTGGCCTTAGCGCATATGATTAAGTTTCGTGGTCATTTTTTGATTGAGGGAGATTTAAATCCTGATAATAGTGATGTGGACAAACTATTTATCCAGTTGGTACAAACCTACAATCAATTATTTGAAGAAAACCCTATTAACGCAAGTGGAGTAGATGCTAAAGCGATTCTTTCTGCACGATTGAGTAAATCAAGACGATTAGAAAATCTCATTGCTCAGCTCCCCGGTGAGAAGAAAAATGGCTTATTTGGGAATCTCATTGCTTTGTCATTGGGTTTGACCCCTAATTTTAAATCAAATTTTGATTTGGCAGAAGATGCTAAATTACAGCTTTCAAAAGATACTTACGATGATGATTTAGATAATTTATTGGCGCAAATTGGAGATCAATATGCTGATTTGTTTTTGGCAGCTAAGAATTTATCAGATGCTATTTTACTTTCAGATATCCTAAGAGTAAATACTGAAATAACTAAGGCTCCCCTATCAGCTTCAATGATTAAACGCTACGATGAACATCATCAAGACTTGACTCTTTTAAAAGCTTTAGTTCGACAACAACTTCCAGAAAAGTATAAAGAAATCTTTTTTGATCAATCAAAAAACGGATATGCAGGTTATATTGATGGGGGAGCTAGCCAAGAAGAATTTTATAAATTTATCAAACCAATTTTAGAAAAAATGGATGGTACTGAGGAATTATTGGTGAAACTAAATCGTGAAGATTTGCTGCGCAAGCAACGGACCTTTGACAACGGCTCTATTCCCCATCAAATTCACTTGGGTGAGCTGCATGCTATTTTGAGAAGACAAGAAGACTTTTATCCATTTTTAAAAGACAATCGTGAGAAGATTGAAAAAATCTTGACTTTTCGAATTCCTTATTATGTTGGTCCATTGGCGCGTGGCAATAGTCGTTTTGCATGGATGACTCGGAAGTCTGAAGAAACAATTACCCCATGGAATTTTGAAGAAGTTGTCGATAAAGGTGCTTCAGCTCAATCATTTATTGAACGCATGACAAACTTTGATAAAAATCTTCCAAATGAAAAAGTACTACCAAAACATAGTTTGCTTTATGAGTATTTTACGGTTTATAACGAATTGACAAAGGTCAAATATGTTACTGAAGGAATGCGAAAACCAGCATTTCTTTCAGGTGAACAGAAGAAAGCCATTGTTGATTTACTCTTCAAAACAAATCGAAAAGTAACCGTTAAGCAATTAAAAGAAGATTATTTCAAAAAAATAGAATGTTTTGATAGTGTTGAAATTTCAGGAGTTGAAGATAGATTTAATGCTTCATTAGGTACCTACCATGATTTGCTAAAAATTATTAAAGATAAAGATTTTTTGGATAATGAAGAAAATGAAGATATCTTAGAGGATATTGTTTTAACATTGACCTTATTTGAAGATAGGGAGATGATTGAGGAAAGACTTAAAACATATGCTCACCTCTTTGATGATAAGGTGATGAAACAGCTTAAACGTCGCCGTTATACTGGTTGGGGACGTTTGTCTCGAAAATTGATTAATGGTATTAGGGATAAGCAATCTGGCAAAACAATATTAGATTTTTTGAAATCAGATGGTTTTGCCAATCGCAATTTTATGCAGCTGATCCATGATGATAGTTTGACATTTAAAGAAGACATTCAAAAAGCACAAGTGTCTGGACAAGGCGATAGTTTACATGAACATATTGCAAATTTAGCTGGTAGCCCTGCTATTAAAAAAGGTATTTTACAGACTGTAAAAGTTGTTGATGAATTGGTCAAAGTAATGGGGCGGCATAAGCCAGAAAATATCGTTATTGAAATGGCACGTGAAAATCAGACAACTCAAAAGGGCCAGAAAAATTCGCGAGAGCGTATGAAACGAATCGAAGAAGGTATCAAAGAATTAGGAAGTCAGATTCTTAAAGAGCATCCTGTTGAAAATACTCAATTGCAAAATGAAAAGCTCTATCTCTATTATCTCCAAAATGGAAGAGACATGTATGTGGACCAAGAATTAGATATTAATCGTTTAAGTGATTATGATGTCGATCACATTGTTCCACAAAGTTTCCTTAAAGACGATTCAATAGACAATAAGGTCTTAACGCGTTCTGATAAAAATCGTGGTAAATCGGATAACGTTCCAAGTGAAGAAGTAGTCAAAAAGATGAAAAACTATTGGAGACAACTTCTAAACGCCAAGTTAATCACTCAACGTAAGTTTGATAATTTAACGAAAGCTGAACGTGGAGGTTTGAGTGAACTTGATAAAGCTGGTTTTATCAAACGCCAATTGGTTGAAACTCGCCAAATCACTAAGCATGTGGCACAAATTTTGGATAGTCGCATGAATACTAAATACGATGAAAATGATAAACTTATTCGAGAGGTTAAAGTGATTACCTTAAAATCTAAATTAGTTTCTGACTTCCGAAAAGATTTCCAATTCTATAAAGTACGTGAGATTAACAATTACCATCATGCCCATGATGCGTATCTAAATGCCGTCGTTGGAACTGCTTTGATTAAGAAATATCCAAAACTTGAATCGGAGTTTGTCTATGGTGATTATAAAGTTTATGATGTTCGTAAAATGATTGCTAAGTCTGAGCAAGAAATAGGCAAAGCAACCGCAAAATATTTCTTTTACTCTAATATCATGAACTTCTTCAAAACAGAAATTACACTTGCAAATGGAGAGATTCGCAAACGCCCTCTAATCGAAACTAATGGGGAAACTGGAGAAATTGTCTGGGATAAAGGGCGAGATTTTGCCACAGTGCGCAAAGTATTGTCCATGCCCCAAGTCAATATTGTCAAGAAAACAGAAGTACAGACAGGCGGATTCTCCAAGGAGTCAATTTTACCAAAAAGAAATTCGGACAAGCTTATTGCTCGTAAAAAAGACTGGGATCCAAAAAAATATGGTGGTTTTGATAGTCCAACGGTAGCTTATTCAGTCCTAGTGGTTGCTAAGGTGGAAAAAGGGAAATCGAAGAAGTTAAAATCCGTTAAAGAGTTACTAGGGATCACAATTATGGAAAGAAGTTCCTTTGAAAAAAATCCGATTGACTTTTTAGAAGCTAAAGGATATAAGGAAGTTAAAAAAGACTTAATCATTAAACTACCTAAATATAGTCTTTTTGAGTTAGAAAACGGTCGTAAACGGATGCTGGCTAGTGCCGGAGAATTACAAAAAGGAAATGAGCTGGCTCTGCCAAGCAAATATGTGAATTTTTTATATTTAGCTAGTCATTATGAAAAGTTGAAGGGTAGTCCAGAAGATAACGAACAAAAACAATTGTTTGTGGAGCAGCATAAGCATTATTTAGATGAGATTATTGAGCAAATCAGTGAATTTTCTAAGCGTGTTATTTTAGCAGATGCCAATTTAGATAAAGTTCTTAGTGCATATAACAAACATAGAGACAAACCAATACGTGAACAAGCAGAAAATATTATTCATTTATTTACGTTGACGAATCTTGGAGCTCCCGCTGCTTTTAAATATTTTGATACAACAATTGATCGTAAACGATATACGTCTACAAAAGAAGTTTTAGATGCCACTCTTATCCATCAATCCATCACTGGTCTTTATGAAACACGCATTGATTTGAGTCAGCTAGGAGGTGACTGATGGC2.片段2(SEQIDNO:141),前导物和第一同向重复:扩增子长度=276bp在酿脓链球菌基因组DNA上,正向引物是860648至860671,并且反向引物是860862至860806。GGCCACGTGAACTATATGATTTTCCGCAGTATATTTTAGATGAAGATTATTTCTTAATAACTAAAAATATGGTATAATACTCTTAATAAATGCAGTAATACAGGGGCTTTTCAAGACTGAAGTCTAGCTGAGACAAATAGTGCGATTACGAAATTTTTTAGACAAAAATAGTCTACGAGGTTTTAGAGCTATGCTGTTTTGAATGGTCCCAAAACTG3.片段3(SEQIDNO:144),第二同向重复:扩增子长度=452bp在酿脓链球菌基因组DNA上,正向引物是从861221到861276,而反向引物是从861613到861594。来自861246-861255GGTCTCCATT(SEQIDNO:142)的十聚物序列(其含有基因组DNA上的BsaI识别序列)被GGTCCCAAAA(SEQIDNO:143)替换以破坏BsaI识别序列,并将基因组上的CRISPR阵列中的第7个截短的同向重复转化为此载体中的规范的第2个同向重复序列。CGTTTTAGAGCTATGCTGTTTTGAATGGTCCCAAAACAACATTGCCGATGATAACTTGAGAAAGAGGGTTAATACCAGCAGTCGGATACCTTCCTATTCTTTCTGTTAAAGCGTTTTCATGTTATAATAGGCAAAAGAAGAGTAGTGTGATGGAACAAACATTTTTTATGATTAAGCCATATGGGGTTAAGCAAGGGGAGGTAGTTGGAGAGGTTTTACGGTGGATTGAACGCCTAAGATTTACGTTTAAGCGATTCGAGCTAAGACAAGCTAGTTCGAAATACTTGGCTAAGCACGACGAGGCCTTGGTGATAAACCTTTTGATCCTAAACTTAAAGCTTACATGACAAGTGGTCCTGTTTTAATTGGGATAATTCTTGGGGACTAAGGTGGT产生三个片段的PCR条件是:在图11中提供了显示PCR扩增子的结果。从三个PCR扩增子,tracrRNA-cas9,前导物和第一个同向重复,第二个同向重复;加上用EcoRV消化的pACYC184装配pNB100我们采用来自NEB(E5510)的Gibson装配试剂盒,并遵循制造商提供的方案来装配上述三种PCR扩增子以及pACYC184。装配反应中每个片段的组分显示在下表中。0.1pmol/μL片段10.2pmol0.2pmol/μL片段20.2pmol0.2pmol/μL片段30.2pmol0.01pmol/μLpACYC1840.04pmol片段1::片段2:片段3:载体5:5:5:1Gibson装配主混合物(2X)1x温育50℃达1hr将2μL装配反应物转化到DH5α感受态细胞(购自NewEnglandBiolabs),随后在氯霉素(35μg/mL)LB平板上选择。使用用于获得初始三个片段的三个引物组通过PCR筛选重组体。从候选克隆中分离给出三种期望的扩增子的质粒模板,并进行序列分析。pNB100的最终构建体的序列(SEQIDNO:145)核苷酸的总数为9578bp。主链载体pACYC184序列是斜体的,序列位置从下划线的EcoRI位点(GAATTC)的G编号。tracrRNA位于核苷酸号1889至1974的指定粗体字母,Cas9起始和终止密码子用粗体三字母标示,从核苷酸2270号开始并且在6376号结束,其后是由斜体粗体字母指示的前导序列6462-6556,第一和第二同向重复序列加下划线,在其之间定位间隔区克隆区(30聚体)。此间隔区克隆区含有由粗斜体字母5'-GAGACC-3'和5'-GGTCTC-3'指示的两个反向BsaI位点,用于分别创建5'四碱基突出间隔区克隆位点5'-GTTT-3'和5'-TTTT-3'和也用粗斜体指示的一个独特的AatII(5'-GACGTC-3')位点,以在不完全BsaI消化的情况下减少自身连接。注意,由Sanger测序检测到的转化和颠换碱基改变G6573A,A6779T分别以粗体字母显示。然而,这些点突变不影响CRISPR-Cas9活性,这将在后面的部分中显示。用以粗斜体字母突出显示的两个独特位点SalI(GTCGAC)和XbaI(TCTAGA)用于分离CRISPR/Cas9构建体以克隆到M13mp18中。pNB100的质粒图谱显示于图12中。可以在BsaI位点之间沿顺时针方向克隆期望的间隔区序列。此载体含有1393-848处的p13A起点和219-9137处的cat(氯霉素抗性)基因。括号中指出的每种限制酶的切割位置是指识别序列内顶链上5'切割位点的位置。pNB102的构建用BsaI和AatII消化pNB100,然后使用Agencourtampure珠纯化。从在上文材料和方法部分中B.从靶序列中选择间隔区序列的讨论中采用间隔区序列CR30。CR30序列如下:5’-AAACACTTTAAAAGTGCTCATCATg(SEQIDNO:117)tgaaattttcacgagtagtacAAAA-5’(SEQIDNO:118)在激酶反应后,通过在95C变性1分钟并在1xT4连接酶缓冲液(50mMTris-HCl(pH7.5at25C),10mMMgCl2,1mMATP10mMDTT)加50mMNaCl中每分钟-1度至30℃再退火产生此双链DNA盒,以在每个寡聚物的5'末端中添加磷酸部分。退火的盒在通过BsaI消化在pNB102上产生的位点的两端含有5’突出的四碱基相容碱基。通过T4DNA连接酶将此CR30盒与pNB100连接,并转化到DH5α感受态细胞(购自NewEnglandBiolabs)。在氯霉素LB板上选择转化体,并通过PCR筛选转化体,所述PCR使用CR30盒的底部序列作为反向引物和正向引物CF1:5’-acgttgacgaatcttggagc,其在重组质粒上的6209-6228区域退火以产生409bpPCR扩增子。对PCR阳性克隆进行测序以确认CR30间隔区序列,并将该重组克隆命名为pNB102并用于体外β-内酰胺酶基因破坏实验。CR30间隔区与β内酰胺酶基因的有义链退火并切割有义链和反义链上第188和189位核苷酸之间的磷酸二酯键。M13mp18::NB102的构建用独特的限制性位点SalI和XbaI消化pNB102以产生6044bp和3524bp的两个片段。从限制性识别位点内的上链中的限制性位点的5'端计算片段长度。在CRISPR阵列中含有CRISPR基因座和CR30间隔区序列的的6044bp片段与3524bp片段分离,并在制备型1%琼脂糖凝胶上纯化。用SalI-XbaI消化M13mp18,随后进行Agencourtampure纯化以从反应中除去6个碱基的SalI-XbaI片段。将这两个纯化的片段组合,并通过T4DNA连接酶连接,并转化到DH5αF’Iq感受态细胞(购自NewEnglandBiolabs)。将转化的细胞与作为噬菌体指示剂的新鲜生长的DH5αF’Iq细胞以及作为蓝白色指示剂的IPTG/X-gal溶液一起在熔化的顶层琼脂的情况下铺板到LB平板上。通过PCR筛选从菌苔收集的白色噬菌斑中的CR30间隔区序列的存在。通过两次单噬菌斑分离纯化获得正确的噬菌体构建体。最终构建体的整个序列长度为13288bp。此间隔区CR30阳性重组体命名为M13mp18::NB102,并用于由M13噬菌体递送介导的bla基因灭活实验。将CRISPR-Cas9构建体递送至细菌构建了CRISPR-Cas9质粒pNB100和衍生物质粒pNB102(其携带靶向由细菌转座因子Tn1和Tn3编码的β-内酰胺酶(bla)基因的间隔区插入),然后我们试图使用三种递送方法(i)质粒接合,(ii)质粒DNA转化,(iii)噬菌体感染证明携带具有bla间隔区插入的CRISPR-Cas9构建体的细菌细胞将不能在β-内酰胺抗生素氨苄青霉素的存在下生长。能够经由CRISPR-Cas系统灭活靶基因(包括抗生素抗性基因)并且是本发明的一个方面的构建体在本文中也称为“Nemesis共生体(symbiotics)”。通过质粒接合的“Nemesis共生体”(NSA)测定法:预防实验我们在这里证明,Nemesis共生体可以通过抑制携带抗生素抗性基因的接合质粒从供体细胞接合转移到携带Nemesis共生体的受体细胞来防止抗生素抗性的传播。为此,我们将携带Nemesis共生体的受体细胞与携带接合质粒,加上携带编码氨苄青霉素抗性的bla基因的多拷贝可移动质粒的供体细胞交配。在成功的交配中,接合质粒转移自身加上携带氨苄青霉素抗性的可移动质粒至受体。可以选择对存在于受体中不可移动的质粒上的氯霉素和从供体接受的氨苄青霉素两者具有抗性的接合后体。成功的Nemesis共生活动将降低氨苄青霉素抗性转移的效率。受体细胞DH5α(F-endA1glnV44thi-1recA1relA1gyrA96deoRnupGΦ80dlacZΔM15Δ(lacZYA-argF)U169,hsdR17(rK-mK+),λ–)购自NewEnglandBiolabs,并用质粒pNB100或pNB102或pACYC184转化,其中质粒编码氯霉素抗性,并且pNB100和pNB102两者携带CRISPR-Cas9,但是仅pNB102携带靶向β-内酰胺酶基因的间隔区序列。质粒pACYC184是用于如上所述pNB100和pNB102的构建中的不可移动的亲本质粒。也携带质粒pNT3的供体菌株JA200(F+thr-1,leu-6,DE(trpE)5,recA,lacY,thi,gal,xyl,ara,mtl)由Sakaetal.DNAResearch12,63–68(2005)描述。质粒pNT3是携带Tn1的bla基因的可移动质粒。从含有100μg/mL氨苄青霉素的Luria肉汤(LB)平板中挑取供体JA200pNT3的单个菌落,并在含有100μg/mL氨苄青霉素的1mLLB培养基中在37℃下振荡培养过夜。从含有35μg/mL氯霉素的LB板中挑取每个受体DH5αpNB100和DH5αpNB102的单菌落,并在具有35μg/mL氯霉素的1mLLB中在37℃下振荡培养过夜。为了洗涤细胞以除去抗生素,将50μL细胞加入到Eppendorf管中的1mLLB中,并以12500rpm离心60秒。将细胞重悬于50μLLB中。为了建立交配,将JA200pNT3点样到LB平板上,然后将2μL携带pNB100和pNB102的每个DH5α加入到该点。还进行了供体和受体的分开的2μL点样(即不交配)。将板在37℃下温育4小时。取出细胞,重悬于LB中,并将100μL铺板在含有100μg/mL氨苄青霉素和35μg/mL氯霉素两者的LB平板(LBApCm)上。还将100μL的10,000倍(10^-4)稀释液涂布在LB平板上并在37℃下温育过夜。如下表所示计数所得菌落。图13中的照片显示以下之间的交配的铺板:(A)JA200pNT3xDH5αpNB100(如预期缺乏Nemesis共生活性);和(B)JA200pNT3xDH5αpNB102(显示Nemesis共生活性)。在LB平板上铺板的10^-4稀释液产生约500个菌落,计数为5×10^7个细胞/mL,而对于JA200pNT3xDH5αpNB102,仅3.7×10^2细胞/mL能够在LBAp100Cm35平板上生长。因此,假设一半细胞是受体/接合后体,则:3.7×10^2个细胞/ml除以2.5×10^7给出携带pNB102的受体的交配效率为1.2×5×10^-5。该实验证明相对于pNB100xJA200pNT3,与携带pNB102的DH5α交配的CmRApR接合后体显著减少。为了更准确地测量相对交配效率,在将液体交配细胞接种在LB平板上以滴定所有细胞后,铺板LBAp100以滴定供体加上接合后体,铺板LBCm以滴定受体加上接合后体,并且铺板LBAp100Cm35以仅滴度接合后体。将10μLJA200pNT3的液体交配过夜培养物与10μL受体DH5αpNB100或DH5αpNB102混合,加入200μLLB,并将管在37℃下温育过夜。将交配混合物在LB中以10^-1,10^-3,10^-5稀释,并且在LB,LBAp100Cm35,LBAp100和LBCm35板上铺板50μL稀释液,并将平板在37℃温育过夜。下表总结了所获得的细胞滴度。LBCm加LBAp平板上的细胞数目应等于LB平板上的细胞数目。对于pNB100,1.40×10^8(Cm板)加上2.64×10^8(Ap板)=4.04×10^8,其与在LB板上的4.14×10^8非常一致。对于pNB102,1.78×10^8(Cm板)加上3.8210^8(Ap板)=5.6×10^8,其与在LB板上的5.16×10^8非常一致。总之,这些数据显示,在液体培养物中过夜交配后,将具有间隔区的pNB102与缺乏间隔区的pNB100进行比较,每个受体的交配效率降低5000倍。通过质粒转化的NSA测定法在此实验中,我们证明通过DNA转化将Nemesis共生体导入受体细胞灭活转化体中的抗生素抗性。为了获得测试菌株,将购自NewEnglandBiolabs的DH5α感受态细胞用pBR322(携带源自Tn3的bla基因)转化并在LBAp100平板上选择。然后使用如Sambrook和Russell在MolecularCloning:ALaboratoryManual(第3版,2001)中所述的CaCl2方案25(1.116)制备衍生菌株DH5αpBR322的感受态细胞,随后用质粒pNB100,pNB102和pACYC184在选择CmR的情况下转化所述感受态细胞。然后将转化体菌落挑取到LBCm35和LBAp100平板上。用牙签将初级转化体同时挑到LBCm35和LBAp100平板两者上并在37℃温育过夜。图14所示的结果显示,从pNB100转化的DH5αpBR322(缺乏bla基因靶向间隔区序列)中用牙签挑出的所有菌落仍然对氨苄青霉素有抗性。相比之下,从由pNB102转化的DH5αpBR322中用牙签挑出的所有菌落丧失氨苄青霉素抗性,因此证明了Nemesis共生活性。上述实验没有给出其中NSA已经灭活bla基因的初级转化体的分数的值。为了解决这点,将来自初级转化体的单个菌落挑取到1mLLB中并在LB中以10^-3稀释。然后如下将100μL铺板到板上,并记录结果。结果显示了用pNB102转化DH5αpBR322后,少于10^-6个细胞保留ApR。Nemesis共生活动非常有效。通过噬菌体M13感染的NSA测定法在此实验中,我们证明通过噬菌体感染将Nemesis共生体导入受体细胞使转化体中的抗生素抗性灭活。我们选择雄性特异性丝状噬菌体M13作为Nemesis共生构建体的递送剂。携带Cas9CRISPR加pNB102的bla基因靶标间隔区的M13衍生物M13mp18::NB102用于通过感染F+菌株JA200(其在质粒pNT3上携带氨苄青霉素抗性)来递送Nemesis共生体。将0.2mL该菌株的新鲜培养物加入到3mLLB顶层琼脂(具有0.7%琼脂的Luria肉汤)中并倒在LB平板上。然后将2μL的M13mp18::NB102(10^8pfu/mL)的噬菌体储液和作为对照的M13mp18点样在菌苔上,并且将板在37℃下温育8小时。将噬菌斑挑取到1.5mLLB中,并在37℃下振荡生长o/n。将来自单菌落的缺乏氨苄青霉素抗性的对照菌株DH5α也挑取到1.5mLLB中培养过夜。进行β-内酰胺酶活性的头孢硝噻测定法:将1mL细胞培养物在微量离心机中以12,500rpm离心60秒,然后向1mL细胞上清液中加入2μL储备头孢硝噻(10mg/mL,在DMSO中),在分光光度计中在添加头孢硝噻之后的几个时间点在482nm处测量头孢硝噻的降解产物的吸光度。下表总结了结果。上面报道的实验提供了概念证明,即在模式生物大肠杆菌中,携带Cas9CRISPR区域加上具有靶向β-内酰胺酶基因的靶区域的序列的间隔区的DNA构建体可以在通过裸DNA转化和噬菌体感染递送时灭活氨苄青霉素抗性,以及通过质粒接合防止氨苄青霉素抗性的转移。显然,本发明的Nemesis共生体可以应用于致病细菌和其它抗生素抗性基因。实施例3实施例3的目的是通过在肺炎克雷伯氏菌的致病性细菌菌株中导入CRISPR/Cas9构建体来扩展抗生素效力恢复的概念证明。对较新的一类β-内酰胺抗生素碳青霉烯的抗性已经通过获得编码能够甚至降解1-碳青霉烯(Carbpenems)的β-内酰胺酶的新bla基因而出现在肠杆菌科中。具有对碳青霉烯类抗性的肺炎克雷伯氏菌菌株KPC是在医院获得的和长期护理相关感染中的发病率和死亡率的重要原因。值得注意的是在具有菌血症或肺部感染的患者中存在的非常高的死亡率(约30-70%)。实施例3显示携带赋予对β-内酰胺抗生素氨苄青霉素抗性的β-内酰胺酶(bla)基因的肺炎克雷伯氏菌在导入经修饰的靶向bla基因的CRISPR/Cas9构建体后对氨苄青霉素变得敏感。使用与实施例1或实施例2相同的CRISPR/Cas9/反bla构建体,并通过与携带具有CRISPR/Cas9/反-bla的接合质粒的供体菌株接合而递送至肺炎克雷伯氏菌。在适当的培养基上在针对供体细胞和未能接受质粒的肺炎克雷伯氏菌细胞的反选择的情况下选择携带具有CRISPR/Cas9/反-bla构建体的质粒的接合后体受体肺炎克雷伯氏菌。对于实施例1和实施例2,通过与接受具有CRISPR/Cas9的质粒的肺炎克雷伯氏菌接合后体菌株的阴性对照(即缺乏反bla区)相比较评价CRISPR/Cas9/反bla构建体的效力。与在实施例1和实施例2中一样,可以通过头孢硝噻计数白色对红色菌落检测β-内酰胺酶活性。同样如实施例1和实施例2,直接攻击具有/不含氨苄青霉素的LB平板上的细菌,并测量氨苄青霉素敏感性菌落相对于氨苄青霉素抗性菌落的分数,可以看到β-内酰胺酶活性。还取而代之使用CRISPR/Cas9/反-blaCb来评估与上述实验相同的实验,其中CRISPR/Cas9/反-blaCb构建体携带靶向编码β-内酰胺酶的bla基因的区域反blaCb,所述β-内酰胺酶能够分解碳青霉烯并存在于特定的肺炎克雷伯氏菌菌株中。实施例4实施例4提供了治疗性构建体的递送途径。这些途径都适用于口服,局部,益生性递送的兽医以及人类应用,以及用于手术灌洗流体和伤口敷料中。口服:含有刺客构建体作为活性成分的噬菌体可以作为稳定化的治疗制剂口服施用:在抗生素疗法之前施用或以与抗生素复合的佐剂的形式施用。含有刺客构建体作为活性成分的接合质粒可以作为携带这些质粒的共生细菌的培养物施用,以将这些质粒传递到肠道菌群,从而产生针对抗生素抗性细菌病原体的未来感染的预防性保护。局部:含有刺客构建体作为活性成分的噬菌体可以作为稳定化药物(软膏剂,喷雾剂,粉末等)局部施用:在抗生素局部药物前施用;或以与抗生素药物的复合物的形式施用。含有刺客构建体作为活性成分的接合质粒的局部应用可以经由携带这些质粒的共生细菌的稳定化培养物,以将质粒传递到肠道菌群,从而产生针对抗生素抗性细菌病原体的未来感染的预防性保护。益生性:含有刺客构建体作为活性成分的噬菌体可作为携带这些质粒的共生细菌(例如乳杆菌属物种)的稳定化培养物益生性施用,以将质粒传递到肠道菌群,从而产生针对抗生素抗性细菌病原体的未来感染的预防性保护。这点的一个实例可以是在具有抗生素抗性细菌病原体的高风险感染的背景(如医院,护理院(carehomes),学校,移植中心等)中对受试者益生性预防性施用此类制剂。另一个实例可以是经由动物饲料对家畜施用此类制剂,从而限制抗生素抗性细菌的上升和水平传播。因此,本发明在家畜中的使用代表了用于(间接)预防性治疗人类中抗生素抗性细菌的一种手段。手术灌洗流体:含有刺客构建物作为活性成分的噬菌体可以加入到手术灌洗流体,还可以加入用于消毒污染物的喷雾剂。含有掺入刺客构建体的接合质粒的稳定化共生细菌培养物可以作为涂层材料添加到手术擦拭物和伤口敷料。实施例5在该实施例中,构建体被设计为包括多个RNA引导分子,其中每个RNA引导分子由其自身的启动子转录。图16例示了我们已经鉴定的一组间隔区序列,其编码靶向肺炎克雷伯氏菌的NCBIARDB数据库中鉴定的117种不同bla基因的20个引导RNA分子。显示了从NCBIARDB数据库获得的肺炎克雷伯氏菌中的β-内酰胺酶基因类型,间隔区序列和抗生素抗性谱。从含有关键字肺炎克雷伯氏菌的ARDB数据库收集β-内酰胺酶基因序列。除去冗余序列,并且使用网络程序ClustalOmega使用独特的序列进行多重序列比对。从每个簇中选择一个规范序列,并且收集通过网络程序JackLin的CRISPR/Cas9gRNA寻找器预测的20nt间隔区序列。选择间隔区序列以使属于相同分支的序列中发现的原间隔区序列的比率最大化。在第4列中显示的每个实例间隔区序列具有破坏第三列中的基因的能力。在该分析中使用的β-内酰胺酶基因是:SHV-a=1,2,2a,5,5a,11,12,14,26,27,28,31,33,38,43,44,48,55,56,60,61,62,71,73,74,75,76,77,78,79,80,81,82,85,89,92,98,99,101,103,106,107,108,109,CTXM-b=1,3,10,12,15,22,32,54,60,62,68,CTXM-c=13,14,16,18,19,24,26,CTXM-d=2,35,59,CTXM-e=26,63,TEM-f=1,1b,3,ESBL,139,KPC-g=1,2,3,4,OKP-h=A11,A12,A16,A17,B13,B-SB613,6,LEN-i=2,17,18,19,20,21,22,24,GES-j=1,3,4,5,VIM-a=1,2,4,12,19,IMP-b=4,8,CMY-a=2,4,25,31,36,LAT-b=1,2,CMY-c=1,8b,10,19,FOX-d=1,5,7,OXA-a=1,30,47,OXA-2,OXA-9,OXA-b=10,17。β-内酰胺抗生素分为四类,即青霉烷类,头孢烯类,碳青霉烯类和单环β-内酰胺类。在图16中在每个类别下列举一个抗生素名称作为实例。可以打开β内酰胺环的β内酰胺酶由R表示。例如,KPC灭活碳青霉烯。为了使细菌对碳青霉烯重新敏感,应当将间隔区序列5’-TTGTTGCTGAAGGAGTTGGG(SEQIDNO:45)用于间隔区阵列中并且灭活KPC基因。注意,CMY-a的间隔区序列可用于LAT-b切割。类似地,图17例示了一组间隔区序列,其编码17种引导RNA分子,靶向肺炎克雷伯氏菌的CARD数据库中鉴定的154种不同bla基因。显示了从IIDRCARD数据库获得的肺炎克雷伯氏菌中的β-内酰胺酶基因类型,间隔区序列和抗生素抗性谱。通过用关键字肺炎克雷伯氏菌过滤所有收集的β-内酰胺酶基因并使用网络程序ClustalOmega进行多重序列比对,收集β内酰胺酶基因序列。选择来自每个簇的一个规范序列,并且收集通过网络程序JackLin的CRISPR/Cas9gRNA发现器预测的20nt间隔区序列。选择间隔区序列以使属于相同分支的序列中发现的原间隔区序列的比率最大化。在第4个行中显示的每个实例间隔区序列具有破坏第三列中的基因的能力。在该分析中使用的β-内酰胺酶基因是:SHV-a=1a,5,2a,11,18,20,21,22,23,28,31,32,52,55,98,99,100,106,107,110,111,121,134,136,137,140,143,144,147,148,149,150,151,152,153,154,155,157,158,159,160,161,162,163,164,165,168,172,173,178,179,CTXM-b=10,12,15,52,54,62,71,80,CTXM-c=19,81,99,147,TEM-f=1,162,183,192,197,198,209,KPC-g=3,4,6,7,8,11,12,14,15,16,17,OKP-h=5,6,A1,A2,A4,A5,A6,A7,A8,A9A10,A11,A12,A13,A14,A15,A16,B1,B2,B3,B4,B5,B6,B7,B8,B9,B10,B11,B12,B13,B17,B18,B19,B20,LEN-i=5,6,7,8,9,10,11,12,13,18,19,20,21,22,23,24,GES-j=1,3,4,VIM-a=4,26,27,28,33,34,IMP-b=32,38,NDM-c=1,9,10,ACT-3,CMY-a=25,31,56,87,FOX-d=8,9,OXA-a=1,30,47,OXA-1,OXA-a=181,204,247,OXA-9。β-内酰胺抗生素分为四类,即青霉烷类,头孢烯类,碳青霉烯类和单环β-内酰胺类。在图17中,在每个类别下列举一个抗生素名称作为实例。可以打开β内酰胺环的β内酰胺酶由R表示。例如,KPC灭活碳青霉烯。为了使细菌对碳青霉烯重新敏感,应当将间隔区序列5’-TTGTTGCTGAAGGAGTTGGG(SEQIDNO:45)用于间隔区阵列中并且灭活KPC基因。实施例6在此实施例中,创建经修饰的CasDNA结合多肽,其删除和重新密封而不是留下DSB。如上所述,由CasDNA结合多肽,如CRISPR-Cas9引起的DSB可以对靶向的复制子是致死的,并且如果复制子中的抗生素抗性基因是靶向灭活的,那么可以导致细胞死亡,而不是例如对抗生素的再致敏。针对随后被抗生素杀伤的立即的细胞死亡,而不是对抗生素的此类再致敏可以增加针对靶向构建体递送的选择压力。代替DSB,我们可以修饰Cas9基因以允许通过导入缺失或倒位在基因灭活后重新密封靶定的序列。Tsai等人(2014)已经构建了无催化活性的Cas9(dCas9)蛋白与限制性内切核酸酶Fok1的野生型核酸酶域之间的融合,以增加在真核细胞中切割的特异性。Proudfoot等人(2011)已经类似地将锌指DNA识别域融合至重组酶的催化域以在指定的DNA序列处编程位点特异性重组。我们在这里添加适当的重组酶域到dCas9以催化重组反应,而不是产生在靶基因上的CRISPR介导的RNA引导的期望位置处的DSB。在图18中称为Cas9R的Cas9解离酶融合物被导向由Cas9域:指导RNA间隔区序列决定的期望的位点;并且融合的解离酶位于重组位点处。为了在两个重组位点:A和B之间产生缺失,解离酶必须在每个重组位点处二聚化。因此,两个Cas9R需要在由间隔区S1-S4编码的序列所指定的每个重组位点A1A2和B1B2处紧密定位。A1相对于A2和B1相对于B2的正确取向将需要通过实验确定。在dCas9融合的FokI(Tsai等,2014)的情况下已经证明如图19所示的gRNA的取向-即重组位点中每个原间隔区序列的PAM序列直接位于全长重组位点的外边界。因此,我们采用退火gRNA极性的相同的配置。图19和20显示了此Cas9R融合解离酶如何解析突触和亲本复制子DNA如何再环化的示意性过程。实施例7以下实验描述了一些概念验证实验,其经实施以证明CRISPR-Cas9系统可以用于单个构建体中以灭活大量不同的β-内酰胺酶基因,所述基因可以在微生物病原体中以及微生物组的非致病性成员中找到。在这些例证中,构建质粒,其携带CRISPR-Cas9系统加上携带侧翼为同向重复的间隔区序列的衍生物,所述间隔区序列靶向多至8个抗性基因的以下β-内酰胺酶家族:SHV,CTX-M,TEM,KPC,VIM,IMP,NDM和OXA。对于这8个β-内酰胺酶基因家族中的每一个,设计单个间隔区,其将靶向该家族的许多基因成员。这些是:SHV-a=1,1a,2,2a,5,5a,11,12,14,18,20,21,22,23,26,27,28,31,32,33,38,43,44,48,52,55,56,60,61,62,71,73,74,75,76,77,78,79,80,81,82,85,89,92,98,99,100,101,103,106,107,108,109,110,111,121,136,134,137,140,143,144,147,148,149,150,151,152,153,154,155,157,158,159,160,161,162,163,164,165,168,172,173,178,179;CTXM-b=1,3,10,12,15,19,22,32,52,54,59,60,62,68,71,80,81,99,141,147;TEM-c=1,1B,3,139,162,183,192,197,198,209;KPC-d=1,2,3,4,6,7,8,11,12,14,15,16,17;VIM-e=1,2,4,19,26,27,33,34;IMP-f=4,8,32,38;和NDM-g=1,9,10。图21显示了设计用于靶向抗性基因的8个β-内酰胺酶家族的八个间隔区序列:SHV,CTX-M,TEM,KPC,VIM,IMP,NDM和OXA-48。用于构建质粒的引物序列列于图22的表中。在一个例示中,在实施例中描述了pNB100(图12)的质粒衍生物pNB104A(图23),一种普遍适用的DNA盒,其携带CRISPR-Cas9系统加上携带侧翼为同向重复的间隔区序列的衍生物并且从一个启动子表达出来,所述间隔区序列靶向细菌中抗生素抗性基因的四个β-内酰胺酶家族:NDM,IMP,VIM和KPC。在另一个例示中,在实施例中描述了pNB100的质粒衍生物pNB104B(图24),一种通常适用的DNA盒的质粒衍生物,其携带CRISPR-Cas9系统加上携带侧翼为同向重复的间隔区序列的衍生物并且从一个启动子表达出来,所述间隔区序列靶向在细菌中的4个β-内酰胺酶抗生素抗性基因家族:OXA-48,SHV,TEM和CTX-M。在另一个例示中,在实施例中描述了pNB100的质粒衍生物,pNB108(图25),一种通常适用的DNA盒,其携带CRISPR-Cas9系统加上携带侧翼为同向重复的间隔区序列的衍生物并且从一个启动子表达出来,所述间隔区序列靶向在细菌中的8个β-内酰胺酶抗生素抗性基因家族:SHV,CTX-M,TEM,KPC,VIM,IMP,NDM和OXA-48。在另一个例示中,构建pNB100的质粒衍生物,其中间隔区序列可以从两种不同启动子的选择中表达。此质粒pNB200(图26)在每个启动子的下游携带不同的独特限制性位点,以克隆任何期望的间隔区序列,其侧翼有CRISPR基因座中两个同向重复之间的其自身同向重复。描述了pNB200的衍生物,其携带CRISPR-Cas9系统加上携带侧翼为同向重复的间隔区序列的衍生物,所述间隔区序列靶向8个β-内酰胺酶抗性基因家族:SHV,CTX-M,TEM,KPC,VIM,IMP,NDM和OXA-48。在pNB202(图27)中,第二启动子用于表达侧翼为同向重复的间隔区,以靶向4个β-内酰胺酶抗性基因家族:OXA-48,SHV,TEM和CTX-M:。并且在源自pNB202的pNB203(图28)中,使用两种启动子:第一种表达侧翼有同向重复的间隔区以靶向四个β-内酰胺酶抗性基因家族:NDM,IMP,VIM和KPC,以及第二启动子表达侧翼为同向重复的间隔区以靶向4种β-内酰胺酶抗性基因家族:OXA-48,SHV,TEM和CTX-M。pNB104A的构建用BsaI(其限制性位点位于A1和A2中)消化图29A所示的四聚体间隔区多联体a+b+c+d,并连接到pNB100上的BsaI间隔区克隆位点。在图23中显示了pNB104A上单个启动子和间隔区(6221-7001)的结构。四间隔多联体从单个启动子的近端到远端含有靶向NDM,IMP,VIM和KPC的间隔区序列。pNB104B的构建用SapI(其限制性位点位于B1和B2中)消化图29A中所示的四聚体间隔区序列多联体e+f+g+h,并连接到pNB200上的SapI间隔区克隆位点。在筛选pNB202筛选时,发现此构建体并通过测序证实。发生了在与前导序列相邻的同向重复之间的缺失事件,并恢复了同向重复序列以产生pNB104B。图24中显示了pNB104B上的单个启动子区(6221-6987)。连环的间隔区(靶向OXA-48,SHV,TEM和CTX-M)位于单个启动子下。pNB108的构建从亚克隆载体pCRBluntII-TOPO_SpacerA和pCRBluntII-TOPO_SpacerB中分别用引物组NB026和NB029,NB030和NB033扩增连环的间隔区阵列序列A和B。在间隔区A的扩增子的3'末端和间隔区B的扩增子的5'末端是来自KPC间隔区序列的重叠序列的20个碱基。将这两种扩增子凝胶纯化并用于在不存在引物的情况下基于PCR的成对循环延伸反应。使用引物组NB037(5’-GGGCTGGCAAGCCACGTTTGGTG-3’;SEQIDNO.150)和NB038(5’-CCGGGAGCTGCATGTGTCAGAGG-3’;SEQIDNO.151)再扩增延伸的材料以产生完全的8间隔区阵列多联体。将该8-间隔连接体克隆到pCRBluntII-TOPO载体中,并确认序列。此pCRBluntII-TOPO亚克隆的BsaI消化从pCRBluntII-TOPO载体(其在两端含有5'突出的四碱基相容碱基,用于通过BsaI消化在pNB100上创建的位点)中以亚克隆取出完全8间隔区阵列多联体。然后用BsaI消化pNB100,随后进行琼脂糖凝胶纯化。通过T4DNA连接酶将从pCRBluntII-TOPO释放的8间隔区多连体盒连接到pNB100中,并转化到DH5α感受态细胞(购自NewEnglandBiolabs)。在氯霉素LB平板上选择转化体,并用反向引物NB021:5’-GGTGACTGATGGCCACGT(SEQIDNO:149)和正向引物NB020:5’-CCAACTACCTCCCCTTGCTTAAC(SEQIDNO:148)(其分别在重组质粒上的6368-6385区域和7203-7225区域退火以产生858bpPCR扩增子)通过PCR进行筛选。对PCR阳性克隆进行测序以确认8个间隔区多连体序列,并且该重组克隆被命名为pNB108,并用于证明在将DNA递送至携带这些基因的细菌菌株后,靶向的β内酰胺酶基因的CRISPR/Cas9介导的灭活。pNB108的质粒图谱显示于图25中。pNB200的构建质粒pNB200含有两个相同的用于gRNA表达的启动子并且在每个启动子下游携带不同的独特限制性位点BsaI和SapI,以克隆任何期望的间隔区序列,其侧翼有在CRISPR基因座中两个同向重复之间的其自身同向重复。pNB200中的CRISPR阵列基因座的期望结构包含两组盒,其以串联的方式含有启动子-前导物-同向重复-间隔区克隆区-同向重复尾部。正向引物NB018在前导序列的5'端与pNB100中的启动子退火,并引入第一间隔区克隆区,第二同向重复和尾部序列。反向引物NB019在pNB100中前导序列的3'端退火,并引入第三同向重复,第二间隔区克隆区。将该扩增子克隆在pNB100上的BsaI位点之间以产生pNB200。以pNB100作为模板用引物NB018和NB019扩增的序列(SEQIDNO:146)正向引物NB018和反向引物NB019加下划线,并且对于第一个PCR循环,初始退火位点分别用粗体字母和斜体粗体字体标示。将该扩增子用BbvI(5'-GCAGCN8/N12)消化以创建与BsaI消化的pNB100相容的4碱基突出的5'端。Bbv1识别位点通过粗体小写字母描绘。产生双重启动子-前导物盒的PCR条件是:组分无核酸酶水41.25μL10XPCR缓冲液5μL10mMdNTP1μL10μM正向引物NB0181μL10μM反向引物NB0191μLpNB1000.5μLQIAGENHotStartTaq0.25μL循环条件STEP温度时间初始变性95℃15min35个循环94℃30sec55℃30sec72℃30sec最终延伸72℃10minpNB200(SEQIDNO:147)中修饰的CRISPR阵列的序列pNB200的总核苷酸数为9919bp。修饰区域中的序列如下所示,其替换了pNB100中的小BsaI片段。第一和第二启动子序列加下划线。前导物序列为粗体字。同向重复序列是斜体化的,并且同向重复序列之间的间隔区克隆区用斜体和下划线标示。第一间隔区阵列克隆区含有以粗斜体字下划线字母标示的两个反向BsaI位点5’-GAGACC-3’和5’-GGTCTC-3’,用于分别在载体上创建5'四碱基突出间隔区克隆位点5'-GTTT-3'和5'-TTTT-3',和也以粗斜体下划线字母标示的一个独特的AatII(5'-GACGTC-3')位点,其用于在不完全BsaI消化的情况下减少自身连接。第二间隔克隆区含有以粗斜体字下划线字母标示的两个反向SapI位点5'-GAAGAGC-3'和5'-GCTCTTC-3',用于分别在载体上创建5'三碱基突出间隔区克隆位点5'-GTT-3'和5'-TTT-3'。pNB200的质粒图谱显示于图26中。对于每个启动子,可以分别在两个BsaI位点和两个SapI位点之间以顺时针方向克隆期望的间隔区阵列序列。pNB202的构建质粒pNB201含有由其衍生的pNB200的双重启动子,但是靶向OXA-48,SHV,TEM和CTX-Mβ内酰胺酶基因家族的间隔区阵列序列从第二启动子表达。为了构建pNB202,用BsaI消化pNB200,随后进行琼脂糖凝胶纯化。通过PCR介导的成对寡聚物连结(concatenation)产生连结的间隔区序列序列B。将该四间隔区多连体阵列B克隆到购自LifeTechnologies的pCRBluntII-TOPO载体,并确认序列。SapI从TOPO载体以亚克隆切出间隔区阵列序列B,并且通过SapI消化在pNB200上创建的位点的两端产生5'突出的三碱基相容碱基。通过T4DNA连接酶将该四间隔区多连体B盒与pNB200连接,并转化到DH5α感受态细胞(购自NewEnglandBiolabs)。在氯霉素LB平板上选择转化体,并使用反向引物NB021:5’-GGTGACTGATGGCCACGT(SEQIDNO:149)和正向引物NB020:5’-CCAACTACCTCCCCTTGCTTAAC(SEQIDNO:148)(其分别在重组质粒上的6368-6385区和7307-7329区退火,以产生962bpPCR扩增子)通过PCR进行筛选。对PCR阳性克隆进行测序以确认四间隔区多连体B序列,并将该重组克隆命名为pNB202,并用于证明在将DNA递送至携带此类基因的细菌菌株后,CRISPR/Cas9介导的靶向β内酰胺酶基因的灭活。pNB202的质粒图谱显示于图27中。pNB203的构建质粒pNB203含有pNB200的双启动子,但每个启动子表达四个间隔区。第一个启动子表达靶向NDM,IMP,VIM和KPC的间隔区序列,第二个启动子表达OXA,SHV,TEM和CTX-Mβ内酰胺酶基因。质粒pNB202用BsaI消化,随后进行琼脂糖凝胶纯化。从含有具有BsaI的间隔区多连体A的pCRBluntII-TOPO载体切出连结的间隔区阵列序列A。BsaI切割出间隔区,其在通过BsaI消化的pNB202上创建的位点的两端含有5'突出的四碱基相容碱基。通过T4DNA连接酶将该四间隔区多连体A盒与pNB202连接,并转化到DH5α感受态细胞(购自NewEnglandBiolabs)。在氯霉素LB平板上选择转化体,并通过PCR用反向引物NB021:5’-GGTGACTGATGGCCACGT和正向引物NB020:5’-CCAACTACCTCCCCTTGCTTAAC(其分别在重组质粒上的6368-6385区域和7479-7501区域退火,以产生1134bpPCR扩增子)进行筛选。对PCR阳性克隆进行测序以证实四间隔区多连体A和B序列,并且将该重组克隆命名为pNB203,并用于证明在将DNA递送至携带此类基因的细菌菌株后,被靶向的β内酰胺酶基因通过CRISPR/Cas9介导的灭活。pNB203的质粒图谱显示于图28中。连结的间隔区阵列的结构的示意图确定间隔区序列以最大化靶β-内酰胺酶基因家族的覆盖。每个单元寡聚物含有在每个末端在合适的间隔区序列侧翼的同向重复。连结反应在成对寡聚物之间进行,即最近邻单元寡聚物首先连结以产生两个单位长度寡聚物,然后两个单位长度寡聚物连结以产生四个单位长度寡聚物等。四聚体和八聚体间隔区结构的示意性结构显示于图29中。S:间隔区,R:同向重复,A和B含有BsaI位点以创建用于克隆到pNB100中和pNB200的第一启动子下游的连接相容位点。C和D含有SapI位点以创建连接,用于pNB200的第二启动子下游克隆的相容位点。在此实施例中,我们采用了靶向NDM的间隔区序列S1,靶向IMP的S2,靶向VIM的S3,靶向KPC的S4,靶向OXA的S5,靶向SHV的S6,靶向靶向TEM的S7和靶向CTX-M的S8。间隔区连结反应每个寡聚物在3'和5'末端具有重叠序列以退火到除第一个和最后一个寡聚物之外的最近邻寡聚物。第一个和最后一个寡聚物具有与仅在5'端的第二个和倒数第二个寡聚物重叠的序列。为了连结四个间隔区,合成四个寡聚物。换言之,寡聚物No.1在5'和3'末端包含间隔区1和2。寡聚物No.2在3'和5'末端包含间隔区2和3的反向互补物。寡聚物No.3在5'和3'末端包含间隔区3和4。寡聚物No.4在3'末端包含间隔区4的反向互补物。因此,寡聚物No.2可以连接寡聚物No.1和寡聚物No.3,寡聚物4与寡聚物No.3的3'端退火。使用以下PCR反应条件将寡聚物No.1和寡聚物No.2,寡聚物No.3和寡聚物No.4在单独的管中连结。组分A1A2B1B2x4无核酸酶的水33.5μL33.5μL33.5μL33.5μL1345XQ5反应缓冲液10μL10μL10μL10μL4010mMdNTPs1μL1μL1μL1μL4Q5高保真性DNA聚合酶0.5μL0.5μL0.5μL0.5μL210μM正向引物NB0262.5μL10μM反向引物NB0272.5μL10μM正向引物NB0282.5μL10μM反向引物NB0342.5μL10μM正向引物NB0352.5μL10μM反向引物NB0312.5μL10μM正向引物NB0322.5μL10μM反向引物NB0362.5μL循环条件步骤温度时间初始变性98℃60sec35个循环98℃10sec55℃10sec72℃20sec最终延伸72℃2分钟保持4℃在此实施例中,将NB026和NB027,NB028和NB034,NB035和NB031,NB032和NB036连结。凝胶纯化每个连结的产物A1,A2,B1和B2,并在以下PCR条件下使用纯化的A1和A2,B1和B2二聚体产物建立第二连结反应。循环条件步骤温度时间初始变性95℃15min35个循环94℃30secA:55℃30sec72℃30sec最终延伸72℃10min通过NB037和NB038用Q5DNA聚合酶扩增这些延伸产物。将最终的扩增子克隆到pCRBluntIITOPO载体中,并确认多连体序列。在8间隔区连结的情况下,分别用引物对NB026和NB029,NB030和NB033扩增pCRBluntIITOPO载体上的间隔区多连体A和间隔区多连体B,并将扩增子凝胶纯化。在以下循环延伸反应中,使用纯化的间隔区A和B作为长引物。组分A无核酸酶的水35.75μL10XPCR缓冲液5μL10mMdNTPs1μLQIAGENHotStartTaq0.25μL凝胶提取A4μL凝胶提取B4μL循环条件步骤温度时间初始变性95℃15min25个循环94℃30sec55℃30sec72℃30sec最终延伸72℃10min保持4℃o/n通过NB037和NB038用Q5DNA聚合酶扩增这些延伸产物。将最终的扩增子克隆到pCRBluntIITOPO载体中,并确认多连体序列。将CRISPR-Cas9构建体递送至细菌能够经由CRISPR-Cas系统灭活靶基因(包括抗生素抗性基因)并且是本发明的一个方面的构建体在本文中也称为“Nemesis共生体”。CRISPR-Cas9质粒衍生物pNB104A,pNB104B,pNB202和pNB203都携带靶向所述选定的β-内酰胺酶(bla)基因家族的间隔区插入,因此提供范例证明单个质粒构建体拥有Nemesis共生活性(NSA),并能够灭活来自β-内酰胺抗生素的所有8个不同家族的代表性基因。对质粒pNB104A(参见图23)测试其灭活NDM,IMP,VIM和KPC家族的代表性基因成员的能力;对pNB104B(参见图24)和pNB202(见图27)测试其灭活OXA,SHV,TEM和CTX-M家族的代表性基因成员的能力;并且对pNB108(参见图25)和pNB203(参见图28)测试其灭活SHV,CTX-M,TEM,KPC,VIM,IMP,NDM和OXA家族的代表性基因成员的能力。通过质粒转化的Nemesis共生活性(NSA)测定法实施例2中描述的NSA测定法显示了用质粒pNB102对大肠杆菌菌株DH5α(其也在质粒pBR322上携带TEM-3β内酰胺酶基因)的DNA转化将转化体转化为氨苄青霉素敏感性(ApS)。这里,质粒pNB102编码对氯霉素的抗性,并且在LBCm平板上选择现在携带pNB102的DH5α(pBR322)转化体,然后筛选ApS(参见图14)。这里,质粒pNB102在表达具有编码靶向TEM-3基因的gRNA的间隔区序列的CRISPR/Cas9系统中,使TEM-3基因灭活。相反,在阴性对照实验中,当用携带表达CRISPR/Cas9系统但缺乏靶向TEM-3基因的gRNA的亲本质粒pNB100转化DH5α(pBR322)时,没有发生向ApS的转化。为了例示的目的,描述了等同实验,其中构建pBR322的质粒衍生物,其中TEM-3被来自其它7个不同的β-内酰胺抗生素家族的代表性基因替代:SHV,CTX-M,KPC,VIM,IMP,NDM和OXA。此类基因获自携带此类基因的合适的细菌菌株。这允许与同基因遗传背景中的实施例2中所述概念验证实验的直接比较。携带来自这7个不同的β-内酰胺抗生素家族的代表性基因的一组大肠杆菌和肺炎克雷伯氏菌菌株购自CultureCollections,PublicHealthEngland,PortonDown,Salisbury,SP4OJG,UK。这些是:NCTC13368,携带SHV-18基因的肺炎克雷伯氏菌株;NCTC13353,携带CTX-M-15基因的大肠杆菌菌株;NCTC13438,携带KPC-3基因的肺炎克雷伯氏菌株;NCTC13440,携带VIM-1基因的肺炎克雷伯氏菌株;NCTC13476,携带未表征的IMP基因的大肠杆菌菌株;NCTC13443,携带NDM-1基因的肺炎克雷杆菌菌株和NCTC13442,携带OXA-48基因的肺炎克雷伯氏菌菌株。所有七个基因编码β内酰胺酶,其也能够降解和灭活青霉烷类抗生素(参见图16,17)。测试所有菌株,并且如所预期的,发现对青霉烷类抗生素氨苄青霉素具有抗性。使用如下所示的合适的正向和反向引物组从细胞扩增β内酰胺酶编码序列:每个正向引物含有17个碱基序列以恢复pBR322上的β-内酰胺酶启动子,并且每个反向引物含有BsaI位点(对于NDM-1,OXA-48,SHV-18,KPC-3,IMP-4和CTX-M15)或FokI位点(对于VIM-1)以创建5'-ACCG四碱基突出的5'端。在用高保真DNA聚合酶,如Q5DNA聚合酶扩增每个β内酰胺酶基因后,用位于上文所述的反向引物中的合适的限制性酶消化扩增子。在除去TEM-3片段之后,消化的扩增子准备使用T4连接酶在质粒pBR322(购自NewEnglandBiolabs)上的SspI和BsaI位点之间进行连接。SspI产生平端,并且BsaI创建5'-CGGT突出末端。下文显示了在限制性消化后每个扩增子的编码序列的反向互补物。5'突出端加下划线,并且启动子序列的3'端为粗体小写字母。这七个基因的甲硫氨酸起始密码子ATG的反向互补物CAT也以粗体显示,产生七种其它β-内酰胺酶的编码区与pBR322的TEM-3β-内酰胺酶的翻译信号序列的精确融合。NDM-1(SEQIDNO:166)ACCGTCAGCGCAGCTTGTCGGCCATGCGGGCCGTATGAGTGATTGCGGCGCGGCTATCGGGGGCGGAATGGCTCATCACGATCATGCTGGCCTTGGGGAACGCCGCACCAAACGCGCGCGCTGACGCGGCGTAGTGCTCAGTGTCGGCATCACCGAGATTGCCGAGCGACTTGGCCTTGCTGTCCTTGATCAGGCAGCCACCAAAAGCGATGTCGGTGCCGTCGATCCCAACGGTGATATTGTCACTGGTGTGGCCGGGGCCGGGGTAAAATACCTTGAGCGGGCCAAAGTTGGGCGCGGTTGCTGGTTCGACCCAGCCATTGGCGGCGAAAGTCAGGCTGTGTTGCGCCGCAACCATCCCCTCTTGCGGGGCAAGCTGGTTCGACAACGCATTGGCATAAGTCGCAATCCCCGCCGCATGCAGCGCGTCCATACCGCCCATCTTGTCCTGATGCGCGTGAGTCACCACCGCCAGCGCGACCGGCAGGTTGATCTCCTGCTTGATCCAGTTGAGGATCTGGGCGGTCTGGTCATCGGTCCAGGCGGTATCGACCACCAGCACGCGGCCGCCATCCCTGACGATCAAACCGTTGGAAGCGACTGCCCCGAAACCCGGCATGTCGAGATAGGAAGTGTGCTGCCAGACATTCGGTGCGAGCTGGCGGAAAACCAGATCGCCAAACCGTTGGTCGCCAGTTTCCATTTGCTGGCCAATCGTCGGGCGGATTTCACCGGGCATGCACCCGCTCAGCATCAATGCAGCGGCTAATGCGGTGCTCAGCTTCGCGACCGGGTGCATAATATTGGGCAATTCOXA-48(SEQIDNO:167)ACCGCTAGGGAATAATTTTTTCCTGTTTGAGCACTTCTTTTGTGATGGCTTGGCGCAGCCCTAAACCATCCGATGTGGGCATATCCATATTCATCGCAAAAAACCACACATTATCATCAAGTTCAACCCAACCGACCCACCAGCCAATCTTAGGTTCGATTCTAGTCGAGTATCCAGTTTTAGCCCGAATAATATAGTCACCATTGGCTTCGGTCAGCATGGCTTGTTTGACAATACGCTGGCTGCGCTCCGATACGTGTAACTTATTGTGATACAGCTTTCTTAAAAAGCTGATTTGCTCCGTGGCCGAAATTCGAATACCACCGTCGAGCCAGAAACTGTCTACATTGCCCGAAATGTCCTCATTACCATAATCGAAAGCATGTAGCATCTTGCTCATACGTGCCTCGCCAATTTGGCGGGCAAATTCTTGATAAACAGGCACAACTGAATATTTCATCGCGGTGATTAGATTATGATCGCGATTCCAAGTGGCGATATCGCGCGTCTGTCCATCCCACTTAAAGACTTGGTGTTCATCCTTAACCACGCCCAAATCGAGGGCGATCAAGCTATTGGGAATTTTAAAGGTAGATGCGGGTAAAAATGCTTGGTTCGCCCGTTTAAGATTATTGGTAAATCCTTGCTGCTTATTCTCATTCCAGAGCACAACTACGCCCTGTGATTTATGTTCAGTAAAGTGAGCATTCCAACTTTTGTTTTCTTGCCATTCCTTTGCTACCGCAGGCATTCCGATAATCGATGCCACCAAAAACACAGCCGATAAGGCTAATACACGSHV-18(SEQIDNO:168)ACCGTTAGCGTTGCCAGTGCTCGATCAGCGCCGCGCCGATCCCGGCGATTTGCTGATTTCGCTCGGCCATGCTCGCCGGCGTATCCCGCAGATAAATCACCACAATCCGCTCTGCTTTGTTATTCGGGCCAAGCAGGGCGACAATCCCGCGCGCACCCCGTTTGGCAGCTCCGGTCTTATCGGCGATAAACCAGCCCGCCGGCAGCACGGAGCGGATCAACGGTCCGGCGACCCGATCGTCCACCATCCACTGCAGCAGCTGCCGTTGCGAACGGGCGCTCAGACGCTGGCTGGTCAGCAGCTTGCGCAGGGTCGCGGCCATGCTGGCCGGGGTAGTGGTGTCGCGGGCGTCGCCGGGAAGCGCCTCATTCAGTTCCGTTTCCCAGCGGTCAAGGCGGGTGACGTTGTCGCCGATCTGGCGCAAAAAGGCAGTCAATCCTGCGGGGCCGCCGACGGTGGCCAGCAGCAGATTGGCGGCGCTGTTATCGCTCATGGTAATGGCGGCGGCACAGAGTTCGCCGACCGTCATGCCGTCGGCAAGGTGTTTTTCGCTGACCGGCGAGTAGTCCACCAGATCCTGCTGGCGATAGTGGATCTTTCGCTCCAGCTGTTCGTCACCGGCATCCACCCGCGCCAGCACTGCGCCGCAGAGCACTACTTTAAAGGTGCTCATCATGGGAAAGCGTTCATCGGCGCGCCAGGCGGTCAGCGTGCGGCCGCTGGCCAGATCCATTTCTATCATGCCTACGCTGCCCGACAGCTGGCTTTCGCTTAGTTTAATTTGCTCAAGCGGCTGCGGGCTGGCGTGTACCGCCAGCGGCAGGGTGGCTAACAGGGAGATAATACACAGGCGAAAATAACGVIM-1(SEQIDNO:169)ACCGCTACTCGGCGACTGAGCGATTTTTGTGTGCTTTGACAACGTTCGCTGTGTGCTGGAGCAAGTCTAGACCGCCCGGTAGACCGTGCCCGGGAATGACGACCTCTGCTTCCGGGTAGTGTTTTTGAATCCGCTCAACGGAGGTGGGCCATTCAGCCAGATCGGCATCGGCCACGTTCCCCGCAGACGTGCTTGACAACTCATGAACGGCACAACCACCGTATAGCACGTTCGCTGACGGGACGTATACAACCAGATTGTCGGTCGAATGCGCAGCACCAGGATAGAAGAGCTCTACTGGACCGAAGCGCACTGCGTCCCCGCTCGATGAGAGTCCTTCTAGAGAATGCGTGGGAATCTCGTTCCCCTCTGCCTCGGCTAGCCGGCGTGTCGACGGTGATGCGTACGTTGCCACCCCAGCCGCCCGAAGGACATCAACGCCGCCGACGCGGTCGTCATGAAAGTGCGTGGAGACTGCACGCGTTACGGGAAGTCCAATTTGCTTTTCAATCTCCGCGAGAAGTGCCGCTGTGTTTTTCGCACCCCACGCTGTATCAATCAAAAGCAACTCATCACCATCACGGACAATGAGACCATTGGACGGGTAGACCGCGCCATCAAACGACTGCGTTGCGATATGCGACCAAACACCATCGGCAATCTGGTAAAGTCGGACCTCTCCGACCGGAATTTCGTTGACTGTCGGATACTCACCACTCGGCTCCCCGGAATGGGCTAACGGACTTGCGACAGCCATGACAGACGCGGTCATGTAGACCAATAAACTACTAATAACTTTTAAKPC-3(SEQIDNO:170)ACCGTTACTGCCCGTTGACGCCCAATCCCTCGAGCGCGAGTCTAGCCGCAGCGGCGATGACGGCCTCGCTGTACTTGTCATCCTTGTTAGGCGCCCGGGTGTAGACGGCCAACACAATAGGTGCGCGCCCAGTGGGCCAGACGACGGCATAGTCATTTGCCGTGCCATACACTCCGCAGGTTCCGGTTTTGTCTCCGACTGCCCAGTCTGCCGGCACCGCCGCGCGGATGCGGTGGTTGCCGGTCGTGTTTCCCTTTAGCCAATCAACAAACTGCTGCCGCTGCGGCGCAGCCAGTGCAGAGCCCAGTGTCAGTTTTTGTAAGCTTTCCGTCACGGCGCGCGGCGATGAGGTATCGCGCGCATCGCCTGGGATGGCGGAGTTCAGCTCCAGCTCCCAGCGGTCCAGACGGAACGTGGTATCGCCGATAGAGCGCATGAAGGCCGTCAGCCCGGCCGGGCCGCCCAACTCCTTCAGCAACAAATTGGCGGCGGCGTTATCACTGTATTGCACGGCGGCCGCGGACAGCTCCGCCACCGTCATGCCTGTTGTCAGATATTTTTCCGAGATGGGTGACCACGGAACCAGCGCATTTTTGCCGTAACGGATGGGTGTGTCCAGCAAGCCGGCCTGCTGCTGGCTGCGAGCCAGCACAGCGGCAGCAAGAAAGCCCTTGAATGAGCTGCACAGTGGGAAGCGCTCCTCAGCGCGGTAACTTACAGTTGCGCCTGAGCCGGTATCCATCGCGTACACACCGATGGAGCCGCCAAAGTCCTGTTCGAGTTTAGCGAATGGTTCCGCGACGAGGTTGGTCAGCGCGGTGGCAGAAAAGCCAGCCAGCGGCCATGAGAGACAAGACAGCAGAACTAGACGGCGATACAGTGAIMP-4(SEQIDNO:171)ACCGTTAGTTGCTTAGTTTTGATGGTTTTTTACTTTCGTTTAACCCTTTAACCGCCTGCTCTAATGTAAGTTTCAAGAGTGATGCGTCTCCAGCTTCACTGTGACTTGGAACAACCAGTTTTGCCTTACCATATTTGGATATTAATAATTTAGCGGACTTTGGCCAAGCTTCTAAATTTGCGTCACCCAAATTACCTAGACCGTACGGTTTAATAAAACAACCACCGAATAATATTTTCCTTTCAGGCAGCCAAACTACTAGGTTATCTGGAGTGTGTCCTGGGCCTGGATAAAAAACTTCAATTTTATTTTTAACTAGCCAATAGTTAACCCCGCCAAATGAATTTTTAGCTTGAACCTTACCGTCTTTTTTAAGCAGCTCATTAGTTAATTCAGACGCATACGTGGGGATGGATTGAGAATTAAGCCACTCTATTCCGCCCGTGCTGTCACTATGAAAATGAGAGGAAATACTGCCTTTTATTTTATAGCCACGTTCCACAAACCAAGTGACTAACTTTTCAGTATCTTTAGCCGTAAATGGAGTGTCAATTAGATAAGCTTCAGCATCTACAAGAACAACCAAACCATGTTTAGGAACAACGCCCCACCCGTTAACTTCTTCAAACGAAGTATGAACATAAACGCCTTCATCAAGTTTTTCAATTTTTAAATCTGGCAAAGACTCTGCTGCGGTAGCAATGCTACAAAACAAAAATATAAAGAATACAGATAACTTGCTCTX-M-15(SEQIDNO:172)ACCGTTACAAACCGTCGGTGACGATTTTAGCCGCCGACGCTAATACATCGCGACGGCTTTCTGCCTTAGGTTGAGGCTGGGTGAAGTAAGTGACCAGAATCAGCGGCGCACGATCTTTTGGCCAGATCACCGCGATATCGTTGGTGGTGCCATAGCCACCGCTGCCGGTTTTATCCCCCACAACCCAGGAAGCAGGCAGTCCAGCCTGAATGCTCGCTGCACCGGTGGTATTGCCTTTCATCCATGTCACCAGCTGCGCCCGTTGGCTGTCGCCCAATGCTTTACCCAGCGTCAGATTCCGCAGAGTTTGCGCCATTGCCCGAGGTGAAGTGGTATCACGCGGATCGCCCGGAATGGCGGTGTTTAACGTCGGCTCGGTACGGTCGAGACGGAACGTTTCGTCTCCCAGCTGTCGGGCGAACGCGGTGACGCTAGCCGGGCCGCCAACGTGAGCAATCAGCTTATTCATCGCCACGTTATCGCTGTACTGTAGCGCGGCCGCGCTAAGCTCAGCCAGTGACATCGTCCCATTGACGTGCTTTTCCGCAATCGGATTATAGTTAACAAGGTCAGATTTTTTGATCTCAACTCGCTGATTTAACAGATTCGGTTCGCTTTCACTTTTCTTCAGCACCGCGGCCGCGGCCATCACTTTACTGGTGCTGCACATCGCAAAGCGCTCATCAGCACGATAAAGTATTTGCGAATTATCTGCTGTGTTAATCAATGCCACACCCAGTCTGCCTCCCGACTGCCGCTCTAATTCGGCAAGTTTTTGCTGTACGTCCGCCGTTTGCGCATACAGCGGCACACTTCCTAACAACAGCGTGACGGTTGCCGTCGCCATCAGCGTGAACTGGCGCAGTGATTTTTTAAC然后,用这些连接物转化从NewEnglandBiolabs购买的DH5α感受态细胞,随后在LB氨苄青霉素(100μg/mL)平板上选择期望的重组体。从这些转化体中分离质粒DNA样品,并进行DNA序列分析以确认七个不同β内酰胺酶基因中每一个的正确序列存在于每个构建体中,产生质粒:如此衍生的这些pBR322衍生质粒命名为:i.携带SHV-18基因的pNB010;ii.携带CTX-M-15基因的pNB011;iii.携带KPC-3基因的pNB012;iv.携带VIM-1基因的pNB013;v.携带IMP基因的pNB014;vi.携带NDM-1基因的pNB015;vii.携带OXA-48基因的pNB016;除此之外,如所描述的,viii.携带TEM-3基因的pBR322然后,用质粒pNB104A,pNB104B,pNB108和pNB203转化7个受体大肠杆菌菌株DH5α,其中每个受体携带质粒pNB010-016上的这7个β内酰胺酶基因之一,并选择氯霉素抗性以选择获得这些Nemesis共生质粒,同时用阴性对照pNB100转化以及通过pNB102作为阳性对照转化DH5α(pBR322),然后如实施例2所述测试转化为氨苄青霉素敏感性(参见图14)。通过质粒接合测试携带靶向β内酰胺酶基因家族的多个间隔区的构建体的活性的“Nemesis共生活性”(NSA)测定法实施例2中描述的质粒接合测定法也可以用于测试携带靶向多个β内酰胺酶基因家族的间隔区序列的新CRISPR/Cas9质粒构建体的Nemesis共生活性。该测定法牵涉将在接合质粒上携带β内酰胺酶基因并且因此具有氨苄青霉素抗性的供体细胞与在编码氯霉素抗性的非可移动质粒上携带Nemesis共生体的受体细胞交配。在含有100μg/mL氨苄青霉素和35μg/mL氯霉素两者的LB平板上选择接合后体。在氨苄青霉素抗性基因转移效率的降低中观察到成功的Nemesis共生活性。作为供体,使用与实施例2中使用的相同的菌株。该菌株JA200(F+thr-1,leu-6,DE(trpE)5,recA,lacY,thi,gal,xyl,ara,mtl)也携带质粒pNT3,如Sakaetal.(DNAResearch12,63–68,2005)描述。质粒pNT3是携带Tn1的TEM-1β内酰胺酶基因的可移动质粒。通过共同驻留的F+质粒的转移功能实现pNT3的接合并因此转移氨苄青霉素抗性。其它潜在的供体是携带来自这7个不同的β-内酰胺抗生素家族的代表性基因的一组大肠杆菌和肺炎克雷伯氏菌菌株,它们购自如上所述的CultureCollections,PublicHealthEngland。这些菌株需要是氯霉素敏感的,以便允许选择接合后体,并且选择在LB氯霉素35μg/mL平板上的生长。NCTC13368,携带SHV-18基因的肺炎克雷伯氏菌株;NCTC13438,携带KPC-3基因的肺炎克雷伯氏菌株;NCTC13476,携带未表征的IMP基因的大肠杆菌菌株;NCTC13443,携带NDM-1基因的肺炎克雷杆菌菌株和NCTC13442,携带OXA-48基因的肺炎克雷伯氏菌菌株均被发现对氯霉素有抗性,但是NCTC13353,携带CTX-M-15基因的大肠杆菌菌株,和NCTC13440,携带VIM-1基因的肺炎克雷伯氏菌菌株被发现是氯霉素敏感的,并且在受体的交配中进行测试。作为受体,实施例2中使用的菌株充当测试新质粒构建体的对照。因此,阴性对照是DH5α(F-endA1glnV44thi-1recA1relA1gyrA96deoRnupGΦ80dlacZΔM15Δ(lacZYA-argF)U169,hsdR17(rK-mK+),λ,具有编码CRISPR/Cas9盒的质粒pNB100,但是没有靶向任何抗生素抗性基因的间隔区序列;并且阳性对照是DH5α,具有编码CRISPR/Cas9盒的质粒pNB102以及靶向TEMβ-内酰胺酶家族的间隔区序列。待测试的是具有质粒pNB104A,pNB104B和pNB108的DH5α菌株。除了间隔区序列(都从一个启动子驱动出来)外,这些质粒还编码CRISPR/Cas9盒,启动子的近端至远端按下列顺序(pNB104A):NDM–IMP–VIM–KPC;(pNB104B):OXA-48–SHV–TEM–CTX-M(pNB108):NDM–IMP–VIM–KPC–OXA-48–SHV–TEM–CTX-M。为了建立交配,将来自LBAp100(氨苄青霉素100μg/mL)的供体菌落和来自LBCm35(氯霉素35μg/mL)平板的受体挑取到200μLLB中,然后将100μL受体与35μL供体混合,然后将5μL混合物点样到LB平板上并在37℃温育5小时以允许交配。图30A显示来自交配携带TEM-1β内酰胺酶(JA200(F+thr-1,leu-6,DE(trpE)5,recA,lacY,thi,gal,xyl,ara,mtl))并在图中命名为(5)的供体与命名为(–)的接收体DH5αpNB100,命名为(+)的DH5αpNB102的结果。这里将一环的每个交配混合物重悬浮于220μLLB中,并将200μL细胞在LBAp100Cm35平板上铺板以选择接合后体。如从实施例2所预期的,TEM-1的供体与具有pNB100的受体之间的杂交给出了有效的交配和细胞的菌苔(图30A,左上,5-),并且TEM-1的供体与具有编码单个TEM间隔区的pNB102的受体的交配显示出对TEM-1基因的转移的强抑制(参见图30A,5+,右上,其中仅看到少数分离的菌落)。携带TEM-1基因的供体与携带pNB104的受体的交配(参见图的底板,5B)也显示出对TEM-1基因转移的强抑制,并且证明在携带四个间隔区OXA-48-SHV-TEM-CTX-M4的pNB104B构建体中,TEM间隔区是活性的。图30B显示来自交配菌株NCTC13440,携带VIM-1的肺炎克雷伯氏菌菌株(命名为(2))和菌株NCTC13353,携带CTX-M-15基因的大肠杆菌菌株(命名为(4))的结果。再次将一环每个此类交配混合物重悬在220μLLB中,并将200μL细胞在LBAp100Cm35平板上铺板以选择接合后体。结果显示,这些菌株能够作为供体起作用,并将VIM-1和CTX-M-15基因分别转移到缺乏Nemesis共生活性的DH5αpNB100阴性对照受体(分别参见图30B,左上和右上)。然而,发现VIM-1和CTX-M-15基因的转移在与携带靶向这些基因的间隔区的受体的交配中受到强烈抑制:pNB104A和pNB104B(分别参见板2A和4B,分别在下左和右侧)。在另一个实验中,完成等同交配以在携带质粒pNB108中的所有8个间隔区序列的受体中测试Nemesis共生活性。这里,直接从交配混合物中挑取细胞,并划线到LBAp100Cm35平板上。图30C板,左上,再次显示供体2,4和5与携带pNB100的受体的成功交配(参见图30C中的2-,4-和5-)。然而,在所有情况下,与携带pNB108(参见图30C,平板右上,2/8,4/8和5/8)的受体交配给出氨苄青霉素抗性基因转移的强抑制。并且与该测定法中的阳性对照相比较,具有5+的底板(参见图30C)显示出对携带pNB102的受体转移TEM-1氨苄青霉素抗性基因的强抑制。上面报道的实验提供了概念证明,即在模式生物体大肠杆菌中,携带Cas9CRISPR区域加上具有靶向β-内酰胺酶基因的靶区域的序列的间隔区的DNA构建体可以在通过裸DNA转化和噬菌体感染递送时灭活氨苄青霉素抗性,以及通过质粒接合防止氨苄青霉素抗性的转移。显然,本发明的Nemesis共生体可以应用于致病细菌和其它抗生素抗性基因。虽然已经参考优选或示例性实施方案描述了本发明,但是本领域技术人员将认识到,在不脱离本发明的精神和范围的情况下,可以对其完成各种修改和变化,并且此类修改是本文中明确涵盖的。就本文中公开的和所附权利要求中阐述的具体实施方案而言并不意图限制,也不应推断任何限制。本文引用的所有文献通过引用整体并入。序列表<110>内梅西斯生物有限公司<120>治疗剂<130>P/71337.WO01/MAR<150>GB1406674.0<151>2014-04-14<150>GB1413719.4<151>2014-08-01<150>GB1418508.6<151>2014-10-17<160>172<170>PatentInversion3.5<210>1<211>12<212>DNA<213>未知<220><223>CR05bla基因序列<400>1gatacgggaggg12<210>2<211>12<212>DNA<213>未知<220><223>CR05bla基因相反链<400>2ccctcccgtatc12<210>3<211>10<212>DNA<213>未知<220><223>CR05短细菌脱靶序列<400>3cgatacggga10<210>4<211>11<212>DNA<213>未知<220><223>CR05短细菌脱靶序列<400>4cgatacgggag11<210>5<211>11<212>DNA<213>未知<220><223>CR05短细菌脱靶序列<400>5gatacgggagg11<210>6<211>19<212>DNA<213>未知<220><223>CR30bla基因序列<400>6tgctcatcattggaaaacg19<210>7<211>19<212>DNA<213>未知<220><223>CR30bla基因序列相反链<400>7cgttttccaatgatgagca19<210>8<211>12<212>DNA<213>未知<220><223>CR30短细菌脱靶序列<400>8tcattggaaaac12<210>9<211>10<212>DNA<213>未知<220><223>CR30短细菌脱靶序列<400>9tgctcatcat10<210>10<211>11<212>DNA<213>未知<220><223>CR30短细菌脱靶序列-3<400>10gctcatcattg11<210>11<211>13<212>DNA<213>未知<220><223>CR30短细菌脱靶序列<400>11ctcatcattggaa13<210>12<211>17<212>DNA<213>未知<220><223>CR30短细菌脱靶序列<400>12ctcagcattgcaaaacg17<210>13<211>15<212>DNA<213>未知<220><223>CR30短细菌脱靶序列<400>13tcatcattgaaaaac15<210>14<211>12<212>DNA<213>未知<220><223>CR30短细菌脱靶序列<400>14tcattggaaaac12<210>15<211>19<212>DNA<213>未知<220><223>CR70bla基因序列<400>15tcgccagttaatagtttgc19<210>16<211>19<212>DNA<213>未知<220><223>CR70bla序列相反链<400>16agcggtcaattatcaaacg19<210>17<211>11<212>DNA<213>未知<220><223>CR30短细菌脱靶序列-1<400>17tcgccagttaa11<210>18<211>12<212>DNA<213>未知<220><223>CR70短细菌脱靶序列<400>18tcgccagttaat12<210>19<211>11<212>DNA<213>未知<220><223>CR70短细菌脱靶序列<400>19cgccagttaat11<210>20<211>12<212>DNA<213>未知<220><223>CR70短细菌脱靶序列<400>20cgccagttaata12<210>21<211>15<212>DNA<213>未知<220><223>CR70短细菌脱靶序列<400>21cgccagctaatagtt15<210>22<211>11<212>DNA<213>未知<220><223>CR70短细菌脱靶序列<400>22ccagttaatag11<210>23<211>11<212>DNA<213>未知<220><223>CR70短细菌脱靶序列<400>23taatagtttgc11<210>24<211>20<212>DNA<213>未知<220><223>CR90bla序列<400>24ccgcgccacatagcagaact20<210>25<211>20<212>DNA<213>未知<220><223>CR90bla序列相反链<400>25agttctgctatgtggcgcgg20<210>26<211>11<212>DNA<213>未知<220><223>CR90短细菌脱靶序列<400>26acatagcagaa11<210>27<211>11<212>DNA<213>未知<220><223>CR90短细菌脱靶序列<400>27cgcgccacata11<210>28<211>13<212>DNA<213>未知<220><223>CR90短细菌脱靶序列<400>28cgcgccacatagc13<210>29<211>11<212>DNA<213>未知<220><223>CR90短细菌脱靶序列<400>29gcgccacatag11<210>30<211>11<212>DNA<213>未知<220><223>CR90短细菌脱靶序列<400>30gccacatagca11<210>31<211>11<212>DNA<213>未知<220><223>CR90短细菌脱靶序列<400>31acatagcagaa11<210>32<211>11<212>DNA<213>未知<220><223>CR90短细菌脱靶序列<400>32catagcagaac11<210>33<211>16<212>DNA<213>未知<220><223>CR90短细菌脱靶序列<400>33gcgccatatagcagaa16<210>34<211>20<212>RNA<213>人工序列<220><223>人工RNA序列<220><221>stem_loop<222>(1)..(10)<400>34uagauaacuacgauacggga20<210>35<211>20<212>RNA<213>人工序列<220><223>人工RNA序列<220><221>stem_loop<222>(1)..(11)<400>35acuuuaaaagugcucaucau20<210>36<211>20<212>RNA<213>人工序列<220><223>人工RNA序列<220><221>stem_loop<222>(3)..(13)<400>36acguugcgcaaacuauuaac20<210>37<211>20<212>RNA<213>人工序列<220><223>人工RNA序列<220><221>茎_环<222>(9)..(17)<400>37acuuuuaaaguucugcuaug20<210>38<211>861<212>DNA<213>未知<220><223>来自pBR322的Β-内酰胺酶基因<400>38atgagtattcaacatttccgtgtcgcccttattcccttttttgcggcattttgccttcct60gtttttgctcacccagaaacgctggtgaaagtaaaagatgctgaagatcagttgggtgca120cgagtgggttacatcgaactggatctcaacagcggtaagatccttgagagttttcgcccc180gaagaacgttttccaatgatgagcacttttaaagttctgctatgtggcgcggtattatcc240cgtgttgacgccgggcaagagcaactcggtcgccgcatacactattctcagaatgacttg300gttgagtactcaccagtcacagaaaagcatcttacggatggcatgacagtaagagaatta360tgcagtgctgccataaccatgagtgataacactgcggccaacttacttctgacaacgatc420ggaggaccgaaggagctaaccgcttttttgcacaacatgggggatcatgtaactcgcctt480gatcgttgggaaccggagctgaatgaagccataccaaacgacgagcgtgacaccacgatg540cctgcagcaatggcaacaacgttgcgcaaactattaactggcgaactacttactctagct600tcccggcaacaattaatagactggatggaggcggataaagttgcaggaccacttctgcgc660tcggcccttccggctggctggtttattgctgataaatctggagccggtgagcgtgggtct720cgcggtatcattgcagcactggggccagatggtaagccctcccgtatcgtagttatctac780acgacggggagtcaggcaactatggatgaacgaaatagacagatcgctgagataggtgcc840tcactgattaagcattggtaa861<210>39<211>20<212>DNA<213>未知<220><223>间隔区序列<400>39ccgcgtaggcatgatagaaa20<210>40<211>20<212>DNA<213>未知<220><223>间隔区序列<400>40acgttaaacaccgccattcc20<210>41<211>20<212>DNA<213>未知<220><223>间隔区序列<400>41gcgctggagaaaagcagcgg20<210>42<211>20<212>DNA<213>未知<220><223>间隔区序列<400>42aagctgattgcccatctggg20<210>43<211>20<212>DNA<213>未知<220><223>间隔区序列<400>43acgctcaacaccgcgatccc20<210>44<211>20<212>DNA<213>未知<220><223>间隔区序列<400>44aactacttactctagcttcc20<210>45<211>20<212>DNA<213>未知<220><223>间隔区序列<400>45ttgttgctgaaggagttggg20<210>46<211>20<212>DNA<213>未知<220><223>间隔区序列<400>46agcgaaaaacaccttgccga20<210>47<211>20<212>DNA<213>未知<220><223>间隔区序列<400>47ctgggaaacggcactgaatg20<210>48<211>20<212>DNA<213>未知<220><223>间隔区序列<400>48tgggttgttggagagaaaac20<210>49<211>20<212>DNA<213>未知<220><223>间隔区序列<400>49aaacacagcggcacttctcg20<210>50<211>20<212>DNA<213>未知<220><223>间隔区序列<400>50aaaattgaagttttttatcc20<210>51<211>20<212>DNA<213>未知<220><223>间隔区序列<400>51tggcagccgcagtggaagcc20<210>52<211>20<212>DNA<213>未知<220><223>间隔区序列<400>52tcacagctacttgaaggttc20<210>53<211>20<212>DNA<213>未知<220><223>间隔区序列<400>53atcaaaactggcagccgcaa20<210>54<211>20<212>DNA<213>未知<220><223>间隔区序列<400>54atcaaaactggcagccgcaa20<210>55<211>20<212>DNA<213>未知<220><223>间隔区序列<400>55cagtactccaaccccagcat20<210>56<211>20<212>DNA<213>未知<220><223>间隔区序列<400>56tcacctggccgcaaatagtc20<210>57<211>20<212>DNA<213>未知<220><223>间隔区序列<400>57acaacggattaacagaagca20<210>58<211>20<212>DNA<213>未知<220><223>间隔区序列<400>58agaacatcagcgcttggtca20<210>59<211>20<212>DNA<213>未知<220><223>间隔区序列<400>59ataacggcttgacccagtca20<210>60<211>20<212>DNA<213>未知<220><223>间隔区序列<400>60ggcaaccagaatatcagtgg20<210>61<211>20<212>DNA<213>未知<220><223>间隔区序列<400>61ggatgccggtgacgaacagc20<210>62<211>20<212>DNA<213>未知<220><223>间隔区序列<400>62gctacagtacagcgataacg20<210>63<211>20<212>DNA<213>未知<220><223>间隔区序列<400>63gacgttgcgtcagcttacgc20<210>64<211>20<212>DNA<213>未知<220><223>间隔区序列<400>64aactacttactctagcttcc20<210>65<211>20<212>DNA<213>未知<220><223>间隔区序列<400>65ttgttgctgaaggagttggg20<210>66<211>20<212>DNA<213>未知<220><223>间隔区序列<400>66agcgaaaaacaccttgccga20<210>67<211>20<212>DNA<213>未知<220><223>间隔区序列<400>67acctttaaagtgctgctgtg20<210>68<211>20<212>DNA<213>未知<220><223>间隔区序列<400>68tgggttgttggagagaaaac20<210>69<211>20<212>DNA<213>未知<220><223>间隔区序列<400>69aaacacagcggcacttctcg20<210>70<211>20<212>DNA<213>未知<220><223>间隔区序列<400>70aaaattgaagttttttatcc20<210>71<211>20<212>DNA<213>未知<220><223>间隔区序列<400>71ggtttgatcgtcagggatgg20<210>72<211>20<212>DNA<213>未知<220><223>间隔区序列<400>72gtggattaacgttccgaaag20<210>73<211>20<212>DNA<213>未知<220><223>间隔区序列<400>73cagcgacagcaaagtggcat20<210>74<211>20<212>DNA<213>未知<220><223>间隔区序列<400>74cttgccacctacagtgcggg20<210>75<211>20<212>DNA<213>未知<220><223>间隔区序列<400>75cccccaaaggaatggagatc20<210>76<211>20<212>DNA<213>未知<220><223>间隔区序列<400>76caccaagtctttaagtggga20<210>77<211>20<212>DNA<213>未知<220><223>间隔区序列<400>77ataacggcttgacccagtca20<210>78<211>20<212>DNA<213>未知<220><223>间隔区序列<400>78ctgggaaacggaactgaatg20<210>79<211>20<212>DNA<213>未知<220><223>间隔区序列<400>79acgttaaacaccgccattcc20<210>80<211>20<212>DNA<213>未知<220><223>间隔区序列<400>80aactacttactctagcttcc20<210>81<211>20<212>DNA<213>未知<220><223>间隔区序列<400>81ttgttgctgaaggagttggg20<210>82<211>20<212>DNA<213>未知<220><223>间隔区序列<400>82aaacacagcggcacttctcg20<210>83<211>34<212>DNA<213>未知<220><223>间隔区序列<400>83ggctagttaaaaataaaattgaagttttttatcc34<210>84<211>20<212>DNA<213>未知<220><223>间隔区序列<400>84ggtttgatcgtcagggatgg20<210>85<211>20<212>DNA<213>未知<220><223>间隔区序列<400>85ataacggcttgacccagtca20<210>86<211>195<212>DNA<213>人工<220><223>引物序列<400>86ccaaaactgagacctgctgcggacgtccaaaggtctcgttttagagctatgctgttttga60atggtcccaaaacttgccgatgataacttgagaaagagggttaataccagcagtcggata120ccttcctattctttctgttaaagcgttttcatgttataataggcaaattttagatgaaga180ttatttcttaataac195<210>87<211>81<212>DNA<213>人工<220><223>引物序列<400>87tctaaaacgaagagcgctgcgtccgagacgctcttcagttttgggaccattcaaaacagc60atagctctaaaacctcgtaga81<210>88<211>54<212>DNA<213>人工<220><223>引物序列<400>88gggctggcaagccacgtttggtgggtctcgaaacggtttgatcgtcagggatgg54<210>89<211>90<212>DNA<213>人工<220><223>引物序列<400>89ggataaaaaacttcaattttatttttaactagccgttttgggaccattcaaaacagcata60gctctaaaacccatccctgacgatcaaacc90<210>90<211>90<212>DNA<213>人工<220><223>引物序列<400>90ggctagttaaaaataaaattgaagttttttatccgttttagagctatgctgttttgaatg60gtcccaaaacaaacacagcggcacttctcg90<210>91<211>76<212>DNA<213>人工<220><223>引物序列<400>91cccaactccttcagcaacaagttttgggaccattcaaaacagcatagctctaaaaccgag60aagtgccgctgtgttt76<210>92<211>76<212>DNA<213>人工<220><223>引物序列<400>92ttgttgctgaaggagttggggttttagagctatgctgttttgaatggtcccaaaacgtcc60atcccacttaaagact76<210>93<211>76<212>DNA<213>人工<220><223>引物序列<400>93cattcagttccgtttcccaggttttgggaccattcaaaacagcatagctctaaaacagtc60tttaagtgggatggac76<210>94<211>76<212>DNA<213>人工<220><223>引物序列<400>94ctgggaaacggaactgaatggttttagagctatgctgttttgaatggtcccaaaacaact60acttactctagcttcc76<210>95<211>111<212>DNA<213>人工<220><223>引物序列<400>95ccgggagctgcatgtgtcagaggggtctccaaaacggaatggcggtgtttaacgtgtttt60gggaccattcaaaacagcatagctctaaaacggaagctagagtaagtagtt111<210>96<211>111<212>DNA<213>人工<220><223>引物序列<400>96ccgggagctgcatgtgtcagaggggtctccaaaaccccaactccttcagcaacaagtttt60gggaccattcaaaacagcatagctctaaaaccgagaagtgccgctgtgttt111<210>97<211>54<212>DNA<213>人工<220><223>引物序列<400>97gggctggcaagccacgtttggtggctcttcaaacgtccatcccacttaaagact54<210>98<211>111<212>DNA<213>人工<220><223>引物序列<400>98ccgggagctgcatgtgtcagagggctcttcaaaacggaatggcggtgtttaacgtgtttt60gggaccattcaaaacagcatagctctaaaacggaagctagagtaagtagtt111<210>99<211>1368<212>PRT<213>酿脓链球菌M1GAS<400>99MetAspLysLysTyrSerIleGlyLeuAspIleGlyThrAsnSerVal151015GlyTrpAlaValIleThrAspGluTyrLysValProSerLysLysPhe202530LysValLeuGlyAsnThrAspArgHisSerIleLysLysAsnLeuIle354045GlyAlaLeuLeuPheAspSerGlyGluThrAlaGluAlaThrArgLeu505560LysArgThrAlaArgArgArgTyrThrArgArgLysAsnArgIleCys65707580TyrLeuGlnGluIlePheSerAsnGluMetAlaLysValAspAspSer859095PhePheHisArgLeuGluGluSerPheLeuValGluGluAspLysLys100105110HisGluArgHisProIlePheGlyAsnIleValAspGluValAlaTyr115120125HisGluLysTyrProThrIleTyrHisLeuArgLysLysLeuValAsp130135140SerThrAspLysAlaAspLeuArgLeuIleTyrLeuAlaLeuAlaHis145150155160MetIleLysPheArgGlyHisPheLeuIleGluGlyAspLeuAsnPro165170175AspAsnSerAspValAspLysLeuPheIleGlnLeuValGlnThrTyr180185190AsnGlnLeuPheGluGluAsnProIleAsnAlaSerGlyValAspAla195200205LysAlaIleLeuSerAlaArgLeuSerLysSerArgArgLeuGluAsn210215220LeuIleAlaGlnLeuProGlyGluLysLysAsnGlyLeuPheGlyAsn225230235240LeuIleAlaLeuSerLeuGlyLeuThrProAsnPheLysSerAsnPhe245250255AspLeuAlaGluAspAlaLysLeuGlnLeuSerLysAspThrTyrAsp260265270AspAspLeuAspAsnLeuLeuAlaGlnIleGlyAspGlnTyrAlaAsp275280285LeuPheLeuAlaAlaLysAsnLeuSerAspAlaIleLeuLeuSerAsp290295300IleLeuArgValAsnThrGluIleThrLysAlaProLeuSerAlaSer305310315320MetIleLysArgTyrAspGluHisHisGlnAspLeuThrLeuLeuLys325330335AlaLeuValArgGlnGlnLeuProGluLysTyrLysGluIlePhePhe340345350AspGlnSerLysAsnGlyTyrAlaGlyTyrIleAspGlyGlyAlaSer355360365GlnGluGluPheTyrLysPheIleLysProIleLeuGluLysMetAsp370375380GlyThrGluGluLeuLeuValLysLeuAsnArgGluAspLeuLeuArg385390395400LysGlnArgThrPheAspAsnGlySerIleProHisGlnIleHisLeu405410415GlyGluLeuHisAlaIleLeuArgArgGlnGluAspPheTyrProPhe420425430LeuLysAspAsnArgGluLysIleGluLysIleLeuThrPheArgIle435440445ProTyrTyrValGlyProLeuAlaArgGlyAsnSerArgPheAlaTrp450455460MetThrArgLysSerGluGluThrIleThrProTrpAsnPheGluGlu465470475480ValValAspLysGlyAlaSerAlaGlnSerPheIleGluArgMetThr485490495AsnPheAspLysAsnLeuProAsnGluLysValLeuProLysHisSer500505510LeuLeuTyrGluTyrPheThrValTyrAsnGluLeuThrLysValLys515520525TyrValThrGluGlyMetArgLysProAlaPheLeuSerGlyGluGln530535540LysLysAlaIleValAspLeuLeuPheLysThrAsnArgLysValThr545550555560ValLysGlnLeuLysGluAspTyrPheLysLysIleGluCysPheAsp565570575SerValGluIleSerGlyValGluAspArgPheAsnAlaSerLeuGly580585590ThrTyrHisAspLeuLeuLysIleIleLysAspLysAspPheLeuAsp595600605AsnGluGluAsnGluAspIleLeuGluAspIleValLeuThrLeuThr610615620LeuPheGluAspArgGluMetIleGluGluArgLeuLysThrTyrAla625630635640HisLeuPheAspAspLysValMetLysGlnLeuLysArgArgArgTyr645650655ThrGlyTrpGlyArgLeuSerArgLysLeuIleAsnGlyIleArgAsp660665670LysGlnSerGlyLysThrIleLeuAspPheLeuLysSerAspGlyPhe675680685AlaAsnArgAsnPheMetGlnLeuIleHisAspAspSerLeuThrPhe690695700LysGluAspIleGlnLysAlaGlnValSerGlyGlnGlyAspSerLeu705710715720HisGluHisIleAlaAsnLeuAlaGlySerProAlaIleLysLysGly725730735IleLeuGlnThrValLysValValAspGluLeuValLysValMetGly740745750ArgHisLysProGluAsnIleValIleGluMetAlaArgGluAsnGln755760765ThrThrGlnLysGlyGlnLysAsnSerArgGluArgMetLysArgIle770775780GluGluGlyIleLysGluLeuGlySerGlnIleLeuLysGluHisPro785790795800ValGluAsnThrGlnLeuGlnAsnGluLysLeuTyrLeuTyrTyrLeu805810815GlnAsnGlyArgAspMetTyrValAspGlnGluLeuAspIleAsnArg820825830LeuSerAspTyrAspValAspHisIleValProGlnSerPheLeuLys835840845AspAspSerIleAspAsnLysValLeuThrArgSerAspLysAsnArg850855860GlyLysSerAspAsnValProSerGluGluValValLysLysMetLys865870875880AsnTyrTrpArgGlnLeuLeuAsnAlaLysLeuIleThrGlnArgLys885890895PheAspAsnLeuThrLysAlaGluArgGlyGlyLeuSerGluLeuAsp900905910LysAlaGlyPheIleLysArgGlnLeuValGluThrArgGlnIleThr915920925LysHisValAlaGlnIleLeuAspSerArgMetAsnThrLysTyrAsp930935940GluAsnAspLysLeuIleArgGluValLysValIleThrLeuLysSer945950955960LysLeuValSerAspPheArgLysAspPheGlnPheTyrLysValArg965970975GluIleAsnAsnTyrHisHisAlaHisAspAlaTyrLeuAsnAlaVal980985990ValGlyThrAlaLeuIleLysLysTyrProLysLeuGluSerGluPhe99510001005ValTyrGlyAspTyrLysValTyrAspValArgLysMetIleAla101010151020LysSerGluGlnGluIleGlyLysAlaThrAlaLysTyrPhePhe102510301035TyrSerAsnIleMetAsnPhePheLysThrGluIleThrLeuAla104010451050AsnGlyGluIleArgLysArgProLeuIleGluThrAsnGlyGlu105510601065ThrGlyGluIleValTrpAspLysGlyArgAspPheAlaThrVal107010751080ArgLysValLeuSerMetProGlnValAsnIleValLysLysThr108510901095GluValGlnThrGlyGlyPheSerLysGluSerIleLeuProLys110011051110ArgAsnSerAspLysLeuIleAlaArgLysLysAspTrpAspPro111511201125LysLysTyrGlyGlyPheAspSerProThrValAlaTyrSerVal113011351140LeuValValAlaLysValGluLysGlyLysSerLysLysLeuLys114511501155SerValLysGluLeuLeuGlyIleThrIleMetGluArgSerSer116011651170PheGluLysAsnProIleAspPheLeuGluAlaLysGlyTyrLys117511801185GluValLysLysAspLeuIleIleLysLeuProLysTyrSerLeu119011951200PheGluLeuGluAsnGlyArgLysArgMetLeuAlaSerAlaGly120512101215GluLeuGlnLysGlyAsnGluLeuAlaLeuProSerLysTyrVal122012251230AsnPheLeuTyrLeuAlaSerHisTyrGluLysLeuLysGlySer123512401245ProGluAspAsnGluGlnLysGlnLeuPheValGluGlnHisLys125012551260HisTyrLeuAspGluIleIleGluGlnIleSerGluPheSerLys126512701275ArgValIleLeuAlaAspAlaAsnLeuAspLysValLeuSerAla128012851290TyrAsnLysHisArgAspLysProIleArgGluGlnAlaGluAsn129513001305IleIleHisLeuPheThrLeuThrAsnLeuGlyAlaProAlaAla131013151320PheLysTyrPheAspThrThrIleAspArgLysArgTyrThrSer132513301335ThrLysGluValLeuAspAlaThrLeuIleHisGlnSerIleThr134013451350GlyLeuTyrGluThrArgIleAspLeuSerGlnLeuGlyGlyAsp135513601365<210>100<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>100tagataactacgatacggga20<210>101<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>101gatcgttgtcagaagtaagt20<210>102<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>102actttaaaagtgctcatcat20<210>103<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>103tttactttcaccagcgtttc20<210>104<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>104attaatagactggatggagg20<210>105<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>105acaattaatagactggatgg20<210>106<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>106gcaacaattaatagactgga20<210>107<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>107cccggcaacaattaatagac20<210>108<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>108aactacttactctagcttcc20<210>109<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>109acgttgcgcaaactattaac20<210>110<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>110tgtaactcgccttgatcgtt20<210>111<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>111atgtaactcgccttgatcgt20<210>112<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>112aacttacttctgacaacgat20<210>113<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>113agtcacagaaaagcatctta20<210>114<211>20<212>DNA<213>未知<220><223>反原间隔区序列<400>114acttttaaagttctgctatg20<210>115<211>25<212>DNA<213>未知<220><223>衔接头序列<400>115aaactagataactacgatacgggag25<210>116<211>25<212>DNA<213>未知<220><223>衔接头序列<400>116aaaactcccgtatcgtagttatcta25<210>117<211>25<212>DNA<213>未知<220><223>衔接头序列<400>117aaacactttaaaagtgctcatcatg25<210>118<211>25<212>DNA<213>未知<220><223>衔接头序列<400>118aaaacatgatgagcacttttaaagt25<210>119<211>25<212>DNA<213>未知<220><223>衔接头序列<400>119aaacacgttgcgcaaactattaacg25<210>120<211>25<212>DNA<213>未知<220><223>衔接头序列<400>120aaaacgttaatagtttgcgcaacgt25<210>121<211>25<212>DNA<213>未知<220><223>衔接头序列<400>121aaacacttttaaagttctgctatgg25<210>122<211>25<212>DNA<213>未知<220><223>衔接头序列<400>122aaaaccatagcagaactttaaaagt25<210>123<211>81<212>DNA<213>未知<220><223>用于双重靶标的衔接头序列<400>123aaacactttaaaagtgctcatcatgttttagagctatgctgttttgaatggtcccaaaac60acttttaaagttctgctatgg81<210>124<211>81<212>DNA<213>未知<220><223>用于双重靶标的衔接头序列<400>124aaaaccatagcagaactttaaaagtgttttgggaccattcaaaacagcatagctctaaaa60catgatgagcacttttaaagt81<210>125<211>9326<212>DNA<213>人工<220><223>质粒序列<400>125gaattccggatgagcattcatcaggcgggcaagaatgtgaataaaggccggataaaactt60gtgcttatttttctttacggtctttaaaaaggccgtaatatccagctgaacggtctggtt120ataggtacattgagcaactgactgaaatgcctcaaaatgttctttacgatgccattggga180tatatcaacggtggtatatccagtgatttttttctccattttagcttccttagctcctga240aaatctcgataactcaaaaaatacgcccggtagtgatcttatttcattatggtgaaagtt300ggaacctcttacgtgccgatcaacgtctcattttcgccaaaagttggcccagggcttccc360ggtatcaacagggacaccaggatttatttattctgcgaagtgatcttccgtcacaggtat420ttattcggcgcaaagtgcgtcgggtgatgctgccaacttactgatttagtgtatgatggt480gtttttgaggtgctccagtggcttctgtttctatcagctgtccctcctgttcagctactg540acggggtggtgcgtaacggcaaaagcaccgccggacatcagcgctagcggagtgtatact600ggcttactatgttggcactgatgagggtgtcagtgaagtgcttcatgtggcaggagaaaa660aaggctgcaccggtgcgtcagcagaatatgtgatacaggatatattccgcttcctcgctc720actgactcgctacgctcggtcgttcgactgcggcgagcggaaatggcttacgaacggggc780ggagatttcctggaagatgccaggaagatacttaacagggaagtgagagggccgcggcaa840agccgtttttccataggctccgcccccctgacaagcatcacgaaatctgacgctcaaatc900agtggtggcgaaacccgacaggactataaagataccaggcgtttccccctggcggctccc960tcgtgcgctctcctgttcctgcctttcggtttaccggtgtcattccgctgttatggccgc1020gtttgtctcattccacgcctgacactcagttccgggtaggcagttcgctccaagctggac1080tgtatgcacgaaccccccgttcagtccgaccgctgcgccttatccggtaactatcgtctt1140gagtccaacccggaaagacatgcaaaagcaccactggcagcagccactggtaattgattt1200agaggagttagtcttgaagtcatgcgccggttaaggctaaactgaaaggacaagttttgg1260tgactgcgctcctccaagccagttacctcggttcaaagagttggtagctcagagaacctt1320cgaaaaaccgccctgcaaggcggttttttcgttttcagagcaagagattacgcgcagacc1380aaaacgatctcaagaagatcatcttattaatcagataaaatatttctagatttcagtgca1440atttatctcttcaaatgtagcacctgaagtcagccccatacgatataagttgtaattctc1500atgtttgacagcttatcatcgataagctttaatgcggtagtttatcacagttaaattgct1560aacgcagtcaggcaccgtgtatgaaatctaacaatgcgctcatcgtcatcctcggcaccg1620tcaccctggatgctgtaggcataggcttggttatgccggtactgccgggcctcttgcggg1680attacgaaatcatcctgtggagcttagtaggtttagcaagatggcagcgcctaaatgtag1740aatgataaaaggattaagagattaatttccctaaaaatgataaaacaagcgttttgaaag1800cgcttgtttttttggtttgcagtcagagtagaatagaagtatcaaaaaaagcaccgactc1860ggtgccactttttcaagttgataacggactagccttattttaacttgctatgctgttttg1920aatggttccaacaagattattttataacttttataacaaataatcaaggagaaattcaaa1980gaaatttatcagccataaaacaatacttaatactatagaatgataacaaaataaactact2040ttttaaaagaattttgtgttataatctatttattattaagtattgggtaatattttttga2100agagatattttgaaaaagaaaaattaaagcatattaaactaatttcggaggtcattaaaa2160ctattattgaaatcatcaaactcattatggatttaatttaaactttttattttaggaggc2220aaaaatggataagaaatactcaataggcttagatatcggcacaaatagcgtcggatgggc2280ggtgatcactgatgaatataaggttccgtctaaaaagttcaaggttctgggaaatacaga2340ccgccacagtatcaaaaaaaatcttataggggctcttttatttgacagtggagagacagc2400ggaagcgactcgtctcaaacggacagctcgtagaaggtatacacgtcggaagaatcgtat2460ttgttatctacaggagattttttcaaatgagatggcgaaagtagatgatagtttctttca2520tcgacttgaagagtcttttttggtggaagaagacaagaagcatgaacgtcatcctatttt2580tggaaatatagtagatgaagttgcttatcatgagaaatatccaactatctatcatctgcg2640aaaaaaattggtagattctactgataaagcggatttgcgcttaatctatttggccttagc2700gcatatgattaagtttcgtggtcattttttgattgagggagatttaaatcctgataatag2760tgatgtggacaaactatttatccagttggtacaaacctacaatcaattatttgaagaaaa2820ccctattaacgcaagtggagtagatgctaaagcgattctttctgcacgattgagtaaatc2880aagacgattagaaaatctcattgctcagctccccggtgagaagaaaaatggcttatttgg2940gaatctcattgctttgtcattgggtttgacccctaattttaaatcaaattttgatttggc3000agaagatgctaaattacagctttcaaaagatacttacgatgatgatttagataatttatt3060ggcgcaaattggagatcaatatgctgatttgtttttggcagctaagaatttatcagatgc3120tattttactttcagatatcctaagagtaaatactgaaataactaaggctcccctatcagc3180ttcaatgattaaacgctacgatgaacatcatcaagacttgactcttttaaaagctttagt3240tcgacaacaacttccagaaaagtataaagaaatcttttttgatcaatcaaaaaacggata3300tgcaggttatattgatgggggagctagccaagaagaattttataaatttatcaaaccaat3360tttagaaaaaatggatggtactgaggaattattggtgaaactaaatcgtgaagatttgct3420gcgcaagcaacggacctttgacaacggctctattccccatcaaattcacttgggtgagct3480gcatgctattttgagaagacaagaagacttttatccatttttaaaagacaatcgtgagaa3540gattgaaaaaatcttgacttttcgaattccttattatgttggtccattggcgcgtggcaa3600tagtcgttttgcatggatgactcggaagtctgaagaaacaattaccccatggaattttga3660agaagttgtcgataaaggtgcttcagctcaatcatttattgaacgcatgacaaactttga3720taaaaatcttccaaatgaaaaagtactaccaaaacatagtttgctttatgagtattttac3780ggtttataacgaattgacaaaggtcaaatatgttactgaaggaatgcgaaaaccagcatt3840tctttcaggtgaacagaagaaagccattgttgatttactcttcaaaacaaatcgaaaagt3900aaccgttaagcaattaaaagaagattatttcaaaaaaatagaatgttttgatagtgttga3960aatttcaggagttgaagatagatttaatgcttcattaggtacctaccatgatttgctaaa4020aattattaaagataaagattttttggataatgaagaaaatgaagatatcttagaggatat4080tgttttaacattgaccttatttgaagatagggagatgattgaggaaagacttaaaacata4140tgctcacctctttgatgataaggtgatgaaacagcttaaacgtcgccgttatactggttg4200gggacgtttgtctcgaaaattgattaatggtattagggataagcaatctggcaaaacaat4260attagattttttgaaatcagatggttttgccaatcgcaattttatgcagctgatccatga4320tgatagtttgacatttaaagaagacattcaaaaagcacaagtgtctggacaaggcgatag4380tttacatgaacatattgcaaatttagctggtagccctgctattaaaaaaggtattttaca4440gactgtaaaagttgttgatgaattggtcaaagtaatggggcggcataagccagaaaatat4500cgttattgaaatggcacgtgaaaatcagacaactcaaaagggccagaaaaattcgcgaga4560gcgtatgaaacgaatcgaagaaggtatcaaagaattaggaagtcagattcttaaagagca4620tcctgttgaaaatactcaattgcaaaatgaaaagctctatctctattatctccaaaatgg4680aagagacatgtatgtggaccaagaattagatattaatcgtttaagtgattatgatgtcga4740tcacattgttccacaaagtttccttaaagacgattcaatagacaataaggtcttaacgcg4800ttctgataaaaatcgtggtaaatcggataacgttccaagtgaagaagtagtcaaaaagat4860gaaaaactattggagacaacttctaaacgccaagttaatcactcaacgtaagtttgataa4920tttaacgaaagctgaacgtggaggtttgagtgaacttgataaagctggttttatcaaacg4980ccaattggttgaaactcgccaaatcactaagcatgtggcacaaattttggatagtcgcat5040gaatactaaatacgatgaaaatgataaacttattcgagaggttaaagtgattaccttaaa5100atctaaattagtttctgacttccgaaaagatttccaattctataaagtacgtgagattaa5160caattaccatcatgcccatgatgcgtatctaaatgccgtcgttggaactgctttgattaa5220gaaatatccaaaacttgaatcggagtttgtctatggtgattataaagtttatgatgttcg5280taaaatgattgctaagtctgagcaagaaataggcaaagcaaccgcaaaatatttctttta5340ctctaatatcatgaacttcttcaaaacagaaattacacttgcaaatggagagattcgcaa5400acgccctctaatcgaaactaatggggaaactggagaaattgtctgggataaagggcgaga5460ttttgccacagtgcgcaaagtattgtccatgccccaagtcaatattgtcaagaaaacaga5520agtacagacaggcggattctccaaggagtcaattttaccaaaaagaaattcggacaagct5580tattgctcgtaaaaaagactgggatccaaaaaaatatggtggttttgatagtccaacggt5640agcttattcagtcctagtggttgctaaggtggaaaaagggaaatcgaagaagttaaaatc5700cgttaaagagttactagggatcacaattatggaaagaagttcctttgaaaaaaatccgat5760tgactttttagaagctaaaggatataaggaagttaaaaaagacttaatcattaaactacc5820taaatatagtctttttgagttagaaaacggtcgtaaacggatgctggctagtgccggaga5880attacaaaaaggaaatgagctggctctgccaagcaaatatgtgaattttttatatttagc5940tagtcattatgaaaagttgaagggtagtccagaagataacgaacaaaaacaattgtttgt6000ggagcagcataagcattatttagatgagattattgagcaaatcagtgaattttctaagcg6060tgttattttagcagatgccaatttagataaagttcttagtgcatataacaaacatagaga6120caaaccaatacgtgaacaagcagaaaatattattcatttatttacgttgacgaatcttgg6180agctcccgctgcttttaaatattttgatacaacaattgatcgtaaacgatatacgtctac6240aaaagaagttttagatgccactcttatccatcaatccatcactggtctttatgaaacacg6300cattgatttgagtcagctaggaggtgactgaagtatattttagatgaagattatttctta6360ataactaaaaatatggtataatactcttaataaatgcagtaatacaggggcttttcaaga6420ctgaagtctagctgagacaaatagtgcgattacgaaattttttagacaaaaatagtctac6480gaggttttagagctatgctgttttgaatggtcccaaaactgagaccagtctcggaagctc6540aaaggtctcgttttagagctatgctgttttgaatggtcccaaaacttcagcacactgaga6600cttgttgagttccatgttttagagctatgctgttttgaatggactccattcaacattgcc6660gatgataacttgagaaagagggttaataccagcagtcggataccttcctattctttctgt6720taaagcgttttcatgttataataggcaaaagaagagtagtgtgatcgtccattccgacag6780catcgccagtcactatggcgtgctgctagcgctatatgcgttgatgcaatttctatgcgc6840acccgttctcggagcactgtccgaccgctttggccgccgcccagtcctgctcgcttcgct6900acttggagccactatcgactacgcgatcatggcgaccacacccgtcctgtggatcctcta6960cgccggacgcatcgtggccggcatcaccggcgccacaggtgcggttgctggcgcctatat7020cgccgacatcaccgatggggaagatcgggctcgccacttcgggctcatgagcgcttgttt7080cggcgtgggtatggtggcaggccccgtggccgggggactgttgggcgccatctccttgca7140tgcaccattccttgcggcggcggtgctcaacggcctcaacctactactgggctgcttcct7200aatgcaggagtcgcataagggagagcgtcgaccgatgcccttgagagccttcaacccagt7260cagctccttccggtgggcgcggggcatgactatcgtcgccgcacttatgactgtcttctt7320tatcatgcaactcgtaggacaggtgccggcagcgctctgggtcattttcggcgaggaccg7380ctttcgctggagcgcgacgatgatcggcctgtcgcttgcggtattcggaatcttgcacgc7440cctcgctcaagccttcgtcactggtcccgccaccaaacgtttcggcgagaagcaggccat7500tatcgccggcatggcggccgacgcgctgggctacgtcttgctggcgttcgcgacgcgagg7560ctggatggccttccccattatgattcttctcgcttccggcggcatcgggatgcccgcgtt7620gcaggccatgctgtccaggcaggtagatgacgaccatcagggacagcttcaaggatcgct7680cgcggctcttaccagcctaacttcgatcattggaccgctgatcgtcacggcgatttatgc7740cgcctcggcgagcacatggaacgggttggcatggattgtaggcgccgccctataccttgt7800ctgcctccccgcgttgcgtcgcggtgcatggagccgggccacctcgacctgaatggaagc7860cggcggcacctcgctaacggattcaccactccaagaattggagccaatcaattcttgcgg7920agaactgtgaatgcgcaaaccaacccttggcagaacatatccatcgcgtccgccatctcc7980agcagccgcacgcggcgcatctcgggcagcgttgggtcctggccacgggtgcgcatgatc8040gtgctcctgtcgttgaggacccggctaggctggcggggttgccttactggttagcagaat8100gaatcaccgatacgcgagcgaacgtgaagcgactgctgctgcaaaacgtctgcgacctga8160gcaacaacatgaatggtcttcggtttccgtgtttcgtaaagtctggaaacgcggaagtcc8220cctacgtgctgctgaagttgcccgcaacagagagtggaaccaaccggtgataccacgata8280ctatgactgagagtcaacgccatgagcggcctcatttcttattctgagttacaacagtcc8340gcaccgctgtccggtagctccttccggtgggcgcggggcatgactatcgtcgccgcactt8400atgactgtcttctttatcatgcaactcgtaggacaggtgccggcagcgcccaacagtccc8460ccggccacggggcctgccaccatacccacgccgaaacaagcgccctgcaccattatgttc8520cggatctgcatcgcaggatgctgctggctaccctgtggaacacctacatctgtattaacg8580aagcgctaaccgtttttatcaggctctgggaggcagaataaatgatcatatcgtcaatta8640ttacctccacggggagagcctgagcaaactggcctcaggcatttgagaagcacacggtca8700cactgcttccggtagtcaataaaccggtaaaccagcaatagacataagcggctatttaac8760gaccctgccctgaaccgacgaccgggtcgaatttgctttcgaatttctgccattcatccg8820cttattatcacttattcaggcgtagcaccaggcgtttaagggcaccaataactgccttaa8880aaaaattacgccccgccctgccactcatcgcagtactgttgtaattcattaagcattctg8940ccgacatggaagccatcacagacggcatgatgaacctgaatcgccagcggcatcagcacc9000ttgtcgccttgcgtataatatttgcccatggtgaaaacgggggcgaagaagttgtccata9060ttggccacgtttaaatcaaaactggtgaaactcacccagggattggctgagacgaaaaac9120atattctcaataaaccctttagggaaataggccaggttttcaccgtaacacgccacatct9180tgcgaatatatgtgtagaaactgccggaaatcgtcgtggtattcactccagagcgatgaa9240aacgtttcagtttgctcatggaaaacggtgtaacaagggtgaacactatcccatatcacc9300agctcaccgtctttcattgccatacg9326<210>126<211>4107<212>DNA<213>人工<220><223>Cas9编码序列<400>126atggataagaaatactcaataggcttagatatcggcacaaatagcgtcggatgggcggtg60atcactgatgaatataaggttccgtctaaaaagttcaaggttctgggaaatacagaccgc120cacagtatcaaaaaaaatcttataggggctcttttatttgacagtggagagacagcggaa180gcgactcgtctcaaacggacagctcgtagaaggtatacacgtcggaagaatcgtatttgt240tatctacaggagattttttcaaatgagatggcgaaagtagatgatagtttctttcatcga300cttgaagagtcttttttggtggaagaagacaagaagcatgaacgtcatcctatttttgga360aatatagtagatgaagttgcttatcatgagaaatatccaactatctatcatctgcgaaaa420aaattggtagattctactgataaagcggatttgcgcttaatctatttggccttagcgcat480atgattaagtttcgtggtcattttttgattgagggagatttaaatcctgataatagtgat540gtggacaaactatttatccagttggtacaaacctacaatcaattatttgaagaaaaccct600attaacgcaagtggagtagatgctaaagcgattctttctgcacgattgagtaaatcaaga660cgattagaaaatctcattgctcagctccccggtgagaagaaaaatggcttatttgggaat720ctcattgctttgtcattgggtttgacccctaattttaaatcaaattttgatttggcagaa780gatgctaaattacagctttcaaaagatacttacgatgatgatttagataatttattggcg840caaattggagatcaatatgctgatttgtttttggcagctaagaatttatcagatgctatt900ttactttcagatatcctaagagtaaatactgaaataactaaggctcccctatcagcttca960atgattaaacgctacgatgaacatcatcaagacttgactcttttaaaagctttagttcga1020caacaacttccagaaaagtataaagaaatcttttttgatcaatcaaaaaacggatatgca1080ggttatattgatgggggagctagccaagaagaattttataaatttatcaaaccaatttta1140gaaaaaatggatggtactgaggaattattggtgaaactaaatcgtgaagatttgctgcgc1200aagcaacggacctttgacaacggctctattccccatcaaattcacttgggtgagctgcat1260gctattttgagaagacaagaagacttttatccatttttaaaagacaatcgtgagaagatt1320gaaaaaatcttgacttttcgaattccttattatgttggtccattggcgcgtggcaatagt1380cgttttgcatggatgactcggaagtctgaagaaacaattaccccatggaattttgaagaa1440gttgtcgataaaggtgcttcagctcaatcatttattgaacgcatgacaaactttgataaa1500aatcttccaaatgaaaaagtactaccaaaacatagtttgctttatgagtattttacggtt1560tataacgaattgacaaaggtcaaatatgttactgaaggaatgcgaaaaccagcatttctt1620tcaggtgaacagaagaaagccattgttgatttactcttcaaaacaaatcgaaaagtaacc1680gttaagcaattaaaagaagattatttcaaaaaaatagaatgttttgatagtgttgaaatt1740tcaggagttgaagatagatttaatgcttcattaggtacctaccatgatttgctaaaaatt1800attaaagataaagattttttggataatgaagaaaatgaagatatcttagaggatattgtt1860ttaacattgaccttatttgaagatagggagatgattgaggaaagacttaaaacatatgct1920cacctctttgatgataaggtgatgaaacagcttaaacgtcgccgttatactggttgggga1980cgtttgtctcgaaaattgattaatggtattagggataagcaatctggcaaaacaatatta2040gattttttgaaatcagatggttttgccaatcgcaattttatgcagctgatccatgatgat2100agtttgacatttaaagaagacattcaaaaagcacaagtgtctggacaaggcgatagttta2160catgaacatattgcaaatttagctggtagccctgctattaaaaaaggtattttacagact2220gtaaaagttgttgatgaattggtcaaagtaatggggcggcataagccagaaaatatcgtt2280attgaaatggcacgtgaaaatcagacaactcaaaagggccagaaaaattcgcgagagcgt2340atgaaacgaatcgaagaaggtatcaaagaattaggaagtcagattcttaaagagcatcct2400gttgaaaatactcaattgcaaaatgaaaagctctatctctattatctccaaaatggaaga2460gacatgtatgtggaccaagaattagatattaatcgtttaagtgattatgatgtcgatcac2520attgttccacaaagtttccttaaagacgattcaatagacaataaggtcttaacgcgttct2580gataaaaatcgtggtaaatcggataacgttccaagtgaagaagtagtcaaaaagatgaaa2640aactattggagacaacttctaaacgccaagttaatcactcaacgtaagtttgataattta2700acgaaagctgaacgtggaggtttgagtgaacttgataaagctggttttatcaaacgccaa2760ttggttgaaactcgccaaatcactaagcatgtggcacaaattttggatagtcgcatgaat2820actaaatacgatgaaaatgataaacttattcgagaggttaaagtgattaccttaaaatct2880aaattagtttctgacttccgaaaagatttccaattctataaagtacgtgagattaacaat2940taccatcatgcccatgatgcgtatctaaatgccgtcgttggaactgctttgattaagaaa3000tatccaaaacttgaatcggagtttgtctatggtgattataaagtttatgatgttcgtaaa3060atgattgctaagtctgagcaagaaataggcaaagcaaccgcaaaatatttcttttactct3120aatatcatgaacttcttcaaaacagaaattacacttgcaaatggagagattcgcaaacgc3180cctctaatcgaaactaatggggaaactggagaaattgtctgggataaagggcgagatttt3240gccacagtgcgcaaagtattgtccatgccccaagtcaatattgtcaagaaaacagaagta3300cagacaggcggattctccaaggagtcaattttaccaaaaagaaattcggacaagcttatt3360gctcgtaaaaaagactgggatccaaaaaaatatggtggttttgatagtccaacggtagct3420tattcagtcctagtggttgctaaggtggaaaaagggaaatcgaagaagttaaaatccgtt3480aaagagttactagggatcacaattatggaaagaagttcctttgaaaaaaatccgattgac3540tttttagaagctaaaggatataaggaagttaaaaaagacttaatcattaaactacctaaa3600tatagtctttttgagttagaaaacggtcgtaaacggatgctggctagtgccggagaatta3660caaaaaggaaatgagctggctctgccaagcaaatatgtgaattttttatatttagctagt3720cattatgaaaagttgaagggtagtccagaagataacgaacaaaaacaattgtttgtggag3780cagcataagcattatttagatgagattattgagcaaatcagtgaattttctaagcgtgtt3840attttagcagatgccaatttagataaagttcttagtgcatataacaaacatagagacaaa3900ccaatacgtgaacaagcagaaaatattattcatttatttacgttgacgaatcttggagct3960cccgctgcttttaaatattttgatacaacaattgatcgtaaacgatatacgtctacaaaa4020gaagttttagatgccactcttatccatcaatccatcactggtctttatgaaacacgcatt4080gatttgagtcagctaggaggtgactga4107<210>127<211>69<212>DNA<213>人工<220><223>pCas9中的-10启动子<400>127agtatattttagatgaagattatttcttaataactaaaaatatggtataatactcttaat60aaatgcagt69<210>128<211>80<212>DNA<213>人工<220><223>pCas9中的前导序列<400>128aatacaggggcttttcaagactgaagtctagctgagacaaatagtgcgattacgaaattt60tttagacaaaaatagtctac80<210>129<211>194<212>DNA<213>人工<220><223>间隔区序列<400>129aaaactgagaccgggcagtgagcgcaacgcaattaacattaggcaccccaggcttgacaa60ttaatcatcggctcgtataatgtgtggaattgtgagcggataacaatttcacacggaggt120cacatatgagataataataactagctgaattccccagcccgcctaatgagcgggcttttt180tttggtctcgtttt194<210>130<211>86<212>RNA<213>人工<220><223>tracrRNA<400>130ggaaccauucaaaacagcauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaa60aguggcaccgagucggugcuuuuuuu86<210>131<211>91<212>DNA<213>人工<220><223>前-crRNA<400>131guuuuagagcuaugctguuuugaauggucccaaacacuuuuaaaguucugcuaugguuuu60agagcuaugctguuuugaauggucccaaaac91<210>132<211>11<212>RNA<213>人工<220><223>在第一个加工事件后切割的tracrRNA<400>132ggaaccauuca11<210>133<211>75<212>DNA<213>人工<220><223>切割的tracrRNA<400>133aaacagcauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccga60gucggugcuuuuuuu75<210>134<211>55<212>DNA<213>人工<220><223>切割的前-crRNA<400>134aauggucccaaacacuuuuaaaguucugcuaugguuuuagagcuaugctguuuug55<210>135<211>42<212>DNA<213>人工<220><223>在第二个加工事件后切割的tracrRNA<400>135acuuuuaaaguucugcuaugguuuuagagcuaugctguuuug42<210>136<211>20<212>DNA<213>未知<220><223>在靶β-内酰胺酶DNA序列内的crRNA互补序列<400>136acttttaaagttctgctatg20<210>137<211>38<212>DNA<213>未知<220><223>含有CR90的靶β-内酰胺酶DNA序列反原间隔区链<400>137ccaagtcaacttttaaagttctgctatgtggcgcggta38<210>138<211>38<212>DNA<213>未知<220><223>含有CR90的靶β-内酰胺酶DNA序列原间隔区链<400>138taccgcgccacatagcagaactttaaaagttgacttgg38<210>139<211>20<212>DNA<213>未知<220><223>靶β-内酰胺酶DNA序列内的crRNA互补序列-相反链<400>139catagcagaactttaaaagt20<210>140<211>4758<212>DNA<213>酿脓链球菌<400>140atgccggtactgccgggcctcttgcgggatccagaagtctttttcttgcactgtttcctt60ttctttatgatagtttacgaaatcatcctgtggagcttagtaggtttagcaagatggcag120cgcctaaatgtagaatgataaaaggattaagagattaatttccctaaaaatgataaaaca180agcgttttgaaagcgcttgtttttttggtttgcagtcagagtagaatagaagtatcaaaa240aaagcaccgactcggtgccactttttcaagttgataacggactagccttattttaacttg300ctatgctgttttgaatggttccaacaagattattttataacttttataacaaataatcaa360ggagaaattcaaagaaatttatcagccataaaacaatacttaatactatagaatgataac420aaaataaactactttttaaaagaattttgtgttataatctatttattattaagtattggg480taatattttttgaagagatattttgaaaaagaaaaattaaagcatattaaactaatttcg540gaggtcattaaaactattattgaaatcatcaaactcattatggatttaatttaaactttt600tattttaggaggcaaaaatggataagaaatactcaataggcttagatatcggcacaaata660gcgtcggatgggcggtgatcactgatgaatataaggttccgtctaaaaagttcaaggttc720tgggaaatacagaccgccacagtatcaaaaaaaatcttataggggctcttttatttgaca780gtggagagacagcggaagcgactcgtctcaaacggacagctcgtagaaggtatacacgtc840ggaagaatcgtatttgttatctacaggagattttttcaaatgagatggcgaaagtagatg900atagtttctttcatcgacttgaagagtcttttttggtggaagaagacaagaagcatgaac960gtcatcctatttttggaaatatagtagatgaagttgcttatcatgagaaatatccaacta1020tctatcatctgcgaaaaaaattggtagattctactgataaagcggatttgcgcttaatct1080atttggccttagcgcatatgattaagtttcgtggtcattttttgattgagggagatttaa1140atcctgataatagtgatgtggacaaactatttatccagttggtacaaacctacaatcaat1200tatttgaagaaaaccctattaacgcaagtggagtagatgctaaagcgattctttctgcac1260gattgagtaaatcaagacgattagaaaatctcattgctcagctccccggtgagaagaaaa1320atggcttatttgggaatctcattgctttgtcattgggtttgacccctaattttaaatcaa1380attttgatttggcagaagatgctaaattacagctttcaaaagatacttacgatgatgatt1440tagataatttattggcgcaaattggagatcaatatgctgatttgtttttggcagctaaga1500atttatcagatgctattttactttcagatatcctaagagtaaatactgaaataactaagg1560ctcccctatcagcttcaatgattaaacgctacgatgaacatcatcaagacttgactcttt1620taaaagctttagttcgacaacaacttccagaaaagtataaagaaatcttttttgatcaat1680caaaaaacggatatgcaggttatattgatgggggagctagccaagaagaattttataaat1740ttatcaaaccaattttagaaaaaatggatggtactgaggaattattggtgaaactaaatc1800gtgaagatttgctgcgcaagcaacggacctttgacaacggctctattccccatcaaattc1860acttgggtgagctgcatgctattttgagaagacaagaagacttttatccatttttaaaag1920acaatcgtgagaagattgaaaaaatcttgacttttcgaattccttattatgttggtccat1980tggcgcgtggcaatagtcgttttgcatggatgactcggaagtctgaagaaacaattaccc2040catggaattttgaagaagttgtcgataaaggtgcttcagctcaatcatttattgaacgca2100tgacaaactttgataaaaatcttccaaatgaaaaagtactaccaaaacatagtttgcttt2160atgagtattttacggtttataacgaattgacaaaggtcaaatatgttactgaaggaatgc2220gaaaaccagcatttctttcaggtgaacagaagaaagccattgttgatttactcttcaaaa2280caaatcgaaaagtaaccgttaagcaattaaaagaagattatttcaaaaaaatagaatgtt2340ttgatagtgttgaaatttcaggagttgaagatagatttaatgcttcattaggtacctacc2400atgatttgctaaaaattattaaagataaagattttttggataatgaagaaaatgaagata2460tcttagaggatattgttttaacattgaccttatttgaagatagggagatgattgaggaaa2520gacttaaaacatatgctcacctctttgatgataaggtgatgaaacagcttaaacgtcgcc2580gttatactggttggggacgtttgtctcgaaaattgattaatggtattagggataagcaat2640ctggcaaaacaatattagattttttgaaatcagatggttttgccaatcgcaattttatgc2700agctgatccatgatgatagtttgacatttaaagaagacattcaaaaagcacaagtgtctg2760gacaaggcgatagtttacatgaacatattgcaaatttagctggtagccctgctattaaaa2820aaggtattttacagactgtaaaagttgttgatgaattggtcaaagtaatggggcggcata2880agccagaaaatatcgttattgaaatggcacgtgaaaatcagacaactcaaaagggccaga2940aaaattcgcgagagcgtatgaaacgaatcgaagaaggtatcaaagaattaggaagtcaga3000ttcttaaagagcatcctgttgaaaatactcaattgcaaaatgaaaagctctatctctatt3060atctccaaaatggaagagacatgtatgtggaccaagaattagatattaatcgtttaagtg3120attatgatgtcgatcacattgttccacaaagtttccttaaagacgattcaatagacaata3180aggtcttaacgcgttctgataaaaatcgtggtaaatcggataacgttccaagtgaagaag3240tagtcaaaaagatgaaaaactattggagacaacttctaaacgccaagttaatcactcaac3300gtaagtttgataatttaacgaaagctgaacgtggaggtttgagtgaacttgataaagctg3360gttttatcaaacgccaattggttgaaactcgccaaatcactaagcatgtggcacaaattt3420tggatagtcgcatgaatactaaatacgatgaaaatgataaacttattcgagaggttaaag3480tgattaccttaaaatctaaattagtttctgacttccgaaaagatttccaattctataaag3540tacgtgagattaacaattaccatcatgcccatgatgcgtatctaaatgccgtcgttggaa3600ctgctttgattaagaaatatccaaaacttgaatcggagtttgtctatggtgattataaag3660tttatgatgttcgtaaaatgattgctaagtctgagcaagaaataggcaaagcaaccgcaa3720aatatttcttttactctaatatcatgaacttcttcaaaacagaaattacacttgcaaatg3780gagagattcgcaaacgccctctaatcgaaactaatggggaaactggagaaattgtctggg3840ataaagggcgagattttgccacagtgcgcaaagtattgtccatgccccaagtcaatattg3900tcaagaaaacagaagtacagacaggcggattctccaaggagtcaattttaccaaaaagaa3960attcggacaagcttattgctcgtaaaaaagactgggatccaaaaaaatatggtggttttg4020atagtccaacggtagcttattcagtcctagtggttgctaaggtggaaaaagggaaatcga4080agaagttaaaatccgttaaagagttactagggatcacaattatggaaagaagttcctttg4140aaaaaaatccgattgactttttagaagctaaaggatataaggaagttaaaaaagacttaa4200tcattaaactacctaaatatagtctttttgagttagaaaacggtcgtaaacggatgctgg4260ctagtgccggagaattacaaaaaggaaatgagctggctctgccaagcaaatatgtgaatt4320ttttatatttagctagtcattatgaaaagttgaagggtagtccagaagataacgaacaaa4380aacaattgtttgtggagcagcataagcattatttagatgagattattgagcaaatcagtg4440aattttctaagcgtgttattttagcagatgccaatttagataaagttcttagtgcatata4500acaaacatagagacaaaccaatacgtgaacaagcagaaaatattattcatttatttacgt4560tgacgaatcttggagctcccgctgcttttaaatattttgatacaacaattgatcgtaaac4620gatatacgtctacaaaagaagttttagatgccactcttatccatcaatccatcactggtc4680tttatgaaacacgcattgatttgagtcagctaggaggtgactgatggccacgtgaactat4740atgattttccgcagtata4758<210>141<211>276<212>DNA<213>酿脓链球菌<400>141attgatttgagtcagctaggaggtgactgatggccacgtgaactatatgattttccgcag60tatattttagatgaagattatttcttaataactaaaaatatggtataatactcttaataa120atgcagtaatacaggggcttttcaagactgaagtctagctgagacaaatagtgcgattac180gaaattttttagacaaaaatagtctacgaggttttagagctatgctgttttgaatggtcc240caaaactgagaccagtctcggacgtccaaaggtctc276<210>142<211>10<212>DNA<213>酿脓链球菌<400>142ggtctccatt10<210>143<211>10<212>DNA<213>酿脓链球菌<400>143ggtcccaaaa10<210>144<211>452<212>DNA<213>人工<220><223>片段3,从酿脓链球菌菌株SF370的基因组DNA扩增<400>144gagaccagtctcggacgtccaaaggtctcgttttagagctatgctgttttgaatggtccc60aaaacaacattgccgatgataacttgagaaagagggttaataccagcagtcggatacctt120cctattctttctgttaaagcgttttcatgttataataggcaaaagaagagtagtgtgatg180gaacaaacattttttatgattaagccatatggggttaagcaaggggaggtagttggagag240gttttacggtggattgaacgcctaagatttacgtttaagcgattcgagctaagacaagct300agttcgaaatacttggctaagcacgacgaggccttggtgataaaccttttgatcctaaac360ttaaagcttacatgacaagtggtcctgttttaattgggataattcttggggactaaggtg420gtatcgtccattccgacagcatcgccagtcac452<210>145<211>9578<212>DNA<213>人工<220><223>最终构建体pNB100的序列<400>145gaattccggatgagcattcatcaggcgggcaagaatgtgaataaaggccggataaaactt60gtgcttatttttctttacggtctttaaaaaggccgtaatatccagctgaacggtctggtt120ataggtacattgagcaactgactgaaatgcctcaaaatgttctttacgatgccattggga180tatatcaacggtggtatatccagtgatttttttctccattttagcttccttagctcctga240aaatctcgataactcaaaaaatacgcccggtagtgatcttatttcattatggtgaaagtt300ggaacctcttacgtgccgatcaacgtctcattttcgccaaaagttggcccagggcttccc360ggtatcaacagggacaccaggatttatttattctgcgaagtgatcttccgtcacaggtat420ttattcggcgcaaagtgcgtcgggtgatgctgccaacttactgatttagtgtatgatggt480gtttttgaggtgctccagtggcttctgtttctatcagctgtccctcctgttcagctactg540acggggtggtgcgtaacggcaaaagcaccgccggacatcagcgctagcggagtgtatact600ggcttactatgttggcactgatgagggtgtcagtgaagtgcttcatgtggcaggagaaaa660aaggctgcaccggtgcgtcagcagaatatgtgatacaggatatattccgcttcctcgctc720actgactcgctacgctcggtcgttcgactgcggcgagcggaaatggcttacgaacggggc780ggagatttcctggaagatgccaggaagatacttaacagggaagtgagagggccgcggcaa840agccgtttttccataggctccgcccccctgacaagcatcacgaaatctgacgctcaaatc900agtggtggcgaaacccgacaggactataaagataccaggcgtttccccctggcggctccc960tcgtgcgctctcctgttcctgcctttcggtttaccggtgtcattccgctgttatggccgc1020gtttgtctcattccacgcctgacactcagttccgggtaggcagttcgctccaagctggac1080tgtatgcacgaaccccccgttcagtccgaccgctgcgccttatccggtaactatcgtctt1140gagtccaacccggaaagacatgcaaaagcaccactggcagcagccactggtaattgattt1200agaggagttagtcttgaagtcatgcgccggttaaggctaaactgaaaggacaagttttgg1260tgactgcgctcctccaagccagttacctcggttcaaagagttggtagctcagagaacctt1320cgaaaaaccgccctgcaaggcggttttttcgttttcagagcaagagattacgcgcagacc1380aaaacgatctcaagaagatcatcttattaatcagataaaatatttctagatttcagtgca1440atttatctcttcaaatgtagcacctgaagtcagccccatacgatataagttgtaattctc1500atgtttgacagcttatcatcgataagctttaatgcggtagtttatcacagttaaattgct1560aacgcagtcaggcaccgtgtatgaaatctaacaatgcgctcatcgtcatcctcggcaccg1620tcaccctggatgctgtaggcataggcttggttatgccggtactgccgggcctcttgcggg1680atccagaagtctttttcttgcactgtttccttttctttatgatagtttacgaaatcatcc1740tgtggagcttagtaggtttagcaagatggcagcgcctaaatgtagaatgataaaaggatt1800aagagattaatttccctaaaaatgataaaacaagcgttttgaaagcgcttgtttttttgg1860tttgcagtcagagtagaatagaagtatcaaaaaaagcaccgactcggtgccactttttca1920agttgataacggactagccttattttaacttgctatgctgttttgaatggttccaacaag1980attattttataacttttataacaaataatcaaggagaaattcaaagaaatttatcagcca2040taaaacaatacttaatactatagaatgataacaaaataaactactttttaaaagaatttt2100gtgttataatctatttattattaagtattgggtaatattttttgaagagatattttgaaa2160aagaaaaattaaagcatattaaactaatttcggaggtcattaaaactattattgaaatca2220tcaaactcattatggatttaatttaaactttttattttaggaggcaaaaatggataagaa2280atactcaataggcttagatatcggcacaaatagcgtcggatgggcggtgatcactgatga2340atataaggttccgtctaaaaagttcaaggttctgggaaatacagaccgccacagtatcaa2400aaaaaatcttataggggctcttttatttgacagtggagagacagcggaagcgactcgtct2460caaacggacagctcgtagaaggtatacacgtcggaagaatcgtatttgttatctacagga2520gattttttcaaatgagatggcgaaagtagatgatagtttctttcatcgacttgaagagtc2580ttttttggtggaagaagacaagaagcatgaacgtcatcctatttttggaaatatagtaga2640tgaagttgcttatcatgagaaatatccaactatctatcatctgcgaaaaaaattggtaga2700ttctactgataaagcggatttgcgcttaatctatttggccttagcgcatatgattaagtt2760tcgtggtcattttttgattgagggagatttaaatcctgataatagtgatgtggacaaact2820atttatccagttggtacaaacctacaatcaattatttgaagaaaaccctattaacgcaag2880tggagtagatgctaaagcgattctttctgcacgattgagtaaatcaagacgattagaaaa2940tctcattgctcagctccccggtgagaagaaaaatggcttatttgggaatctcattgcttt3000gtcattgggtttgacccctaattttaaatcaaattttgatttggcagaagatgctaaatt3060acagctttcaaaagatacttacgatgatgatttagataatttattggcgcaaattggaga3120tcaatatgctgatttgtttttggcagctaagaatttatcagatgctattttactttcaga3180tatcctaagagtaaatactgaaataactaaggctcccctatcagcttcaatgattaaacg3240ctacgatgaacatcatcaagacttgactcttttaaaagctttagttcgacaacaacttcc3300agaaaagtataaagaaatcttttttgatcaatcaaaaaacggatatgcaggttatattga3360tgggggagctagccaagaagaattttataaatttatcaaaccaattttagaaaaaatgga3420tggtactgaggaattattggtgaaactaaatcgtgaagatttgctgcgcaagcaacggac3480ctttgacaacggctctattccccatcaaattcacttgggtgagctgcatgctattttgag3540aagacaagaagacttttatccatttttaaaagacaatcgtgagaagattgaaaaaatctt3600gacttttcgaattccttattatgttggtccattggcgcgtggcaatagtcgttttgcatg3660gatgactcggaagtctgaagaaacaattaccccatggaattttgaagaagttgtcgataa3720aggtgcttcagctcaatcatttattgaacgcatgacaaactttgataaaaatcttccaaa3780tgaaaaagtactaccaaaacatagtttgctttatgagtattttacggtttataacgaatt3840gacaaaggtcaaatatgttactgaaggaatgcgaaaaccagcatttctttcaggtgaaca3900gaagaaagccattgttgatttactcttcaaaacaaatcgaaaagtaaccgttaagcaatt3960aaaagaagattatttcaaaaaaatagaatgttttgatagtgttgaaatttcaggagttga4020agatagatttaatgcttcattaggtacctaccatgatttgctaaaaattattaaagataa4080agattttttggataatgaagaaaatgaagatatcttagaggatattgttttaacattgac4140cttatttgaagatagggagatgattgaggaaagacttaaaacatatgctcacctctttga4200tgataaggtgatgaaacagcttaaacgtcgccgttatactggttggggacgtttgtctcg4260aaaattgattaatggtattagggataagcaatctggcaaaacaatattagattttttgaa4320atcagatggttttgccaatcgcaattttatgcagctgatccatgatgatagtttgacatt4380taaagaagacattcaaaaagcacaagtgtctggacaaggcgatagtttacatgaacatat4440tgcaaatttagctggtagccctgctattaaaaaaggtattttacagactgtaaaagttgt4500tgatgaattggtcaaagtaatggggcggcataagccagaaaatatcgttattgaaatggc4560acgtgaaaatcagacaactcaaaagggccagaaaaattcgcgagagcgtatgaaacgaat4620cgaagaaggtatcaaagaattaggaagtcagattcttaaagagcatcctgttgaaaatac4680tcaattgcaaaatgaaaagctctatctctattatctccaaaatggaagagacatgtatgt4740ggaccaagaattagatattaatcgtttaagtgattatgatgtcgatcacattgttccaca4800aagtttccttaaagacgattcaatagacaataaggtcttaacgcgttctgataaaaatcg4860tggtaaatcggataacgttccaagtgaagaagtagtcaaaaagatgaaaaactattggag4920acaacttctaaacgccaagttaatcactcaacgtaagtttgataatttaacgaaagctga4980acgtggaggtttgagtgaacttgataaagctggttttatcaaacgccaattggttgaaac5040tcgccaaatcactaagcatgtggcacaaattttggatagtcgcatgaatactaaatacga5100tgaaaatgataaacttattcgagaggttaaagtgattaccttaaaatctaaattagtttc5160tgacttccgaaaagatttccaattctataaagtacgtgagattaacaattaccatcatgc5220ccatgatgcgtatctaaatgccgtcgttggaactgctttgattaagaaatatccaaaact5280tgaatcggagtttgtctatggtgattataaagtttatgatgttcgtaaaatgattgctaa5340gtctgagcaagaaataggcaaagcaaccgcaaaatatttcttttactctaatatcatgaa5400cttcttcaaaacagaaattacacttgcaaatggagagattcgcaaacgccctctaatcga5460aactaatggggaaactggagaaattgtctgggataaagggcgagattttgccacagtgcg5520caaagtattgtccatgccccaagtcaatattgtcaagaaaacagaagtacagacaggcgg5580attctccaaggagtcaattttaccaaaaagaaattcggacaagcttattgctcgtaaaaa5640agactgggatccaaaaaaatatggtggttttgatagtccaacggtagcttattcagtcct5700agtggttgctaaggtggaaaaagggaaatcgaagaagttaaaatccgttaaagagttact5760agggatcacaattatggaaagaagttcctttgaaaaaaatccgattgactttttagaagc5820taaaggatataaggaagttaaaaaagacttaatcattaaactacctaaatatagtctttt5880tgagttagaaaacggtcgtaaacggatgctggctagtgccggagaattacaaaaaggaaa5940tgagctggctctgccaagcaaatatgtgaattttttatatttagctagtcattatgaaaa6000gttgaagggtagtccagaagataacgaacaaaaacaattgtttgtggagcagcataagca6060ttatttagatgagattattgagcaaatcagtgaattttctaagcgtgttattttagcaga6120tgccaatttagataaagttcttagtgcatataacaaacatagagacaaaccaatacgtga6180acaagcagaaaatattattcatttatttacgttgacgaatcttggagctcccgctgcttt6240taaatattttgatacaacaattgatcgtaaacgatatacgtctacaaaagaagttttaga6300tgccactcttatccatcaatccatcactggtctttatgaaacacgcattgatttgagtca6360gctaggaggtgactgatggccacgtgaactatatgattttccgcagtatattttagatga6420agattatttcttaataactaaaaatatggtataatactcttaataaatgcagtaatacag6480gggcttttcaagactgaagtctagctgagacaaatagtgcgattacgaaattttttagac6540aaaaatagtctacgaggttttagagctatgctattttgaatggtcccaaaactgagacca6600gtctcggacgtccaaaggtctcgttttagagctatgctgttttgaatggtcccaaaacaa6660cattgccgatgataacttgagaaagagggttaataccagcagtcggataccttcctattc6720tttctgttaaagcgttttcatgttataataggcaaaagaagagtagtgtgatggaacata6780cattttttatgattaagccatatggggttaagcaaggggaggtagttggagaggttttac6840ggtggattgaacgcctaagatttacgtttaagcgattcgagctaagacaagctagttcga6900aatacttggctaagcacgacgaggccttggtgataaaccttttgatcctaaacttaaagc6960ttacatgacaagtggtcctgttttaattgggataattcttggggactaaggtggtatcgt7020ccattccgacagcatcgccagtcactatggcgtgctgctagcgctatatgcgttgatgca7080atttctatgcgcacccgttctcggagcactgtccgaccgctttggccgccgcccagtcct7140gctcgcttcgctacttggagccactatcgactacgcgatcatggcgaccacacccgtcct7200gtggatcctctacgccggacgcatcgtggccggcatcaccggcgccacaggtgcggttgc7260tggcgcctatatcgccgacatcaccgatggggaagatcgggctcgccacttcgggctcat7320gagcgcttgtttcggcgtgggtatggtggcaggccccgtggccgggggactgttgggcgc7380catctccttgcatgcaccattccttgcggcggcggtgctcaacggcctcaacctactact7440gggctgcttcctaatgcaggagtcgcataagggagagcgtcgaccgatgcccttgagagc7500cttcaacccagtcagctccttccggtgggcgcggggcatgactatcgtcgccgcacttat7560gactgtcttctttatcatgcaactcgtaggacaggtgccggcagcgctctgggtcatttt7620cggcgaggaccgctttcgctggagcgcgacgatgatcggcctgtcgcttgcggtattcgg7680aatcttgcacgccctcgctcaagccttcgtcactggtcccgccaccaaacgtttcggcga7740gaagcaggccattatcgccggcatggcggccgacgcgctgggctacgtcttgctggcgtt7800cgcgacgcgaggctggatggccttccccattatgattcttctcgcttccggcggcatcgg7860gatgcccgcgttgcaggccatgctgtccaggcaggtagatgacgaccatcagggacagct7920tcaaggatcgctcgcggctcttaccagcctaacttcgatcattggaccgctgatcgtcac7980ggcgatttatgccgcctcggcgagcacatggaacgggttggcatggattgtaggcgccgc8040cctataccttgtctgcctccccgcgttgcgtcgcggtgcatggagccgggccacctcgac8100ctgaatggaagccggcggcacctcgctaacggattcaccactccaagaattggagccaat8160caattcttgcggagaactgtgaatgcgcaaaccaacccttggcagaacatatccatcgcg8220tccgccatctccagcagccgcacgcggcgcatctcgggcagcgttgggtcctggccacgg8280gtgcgcatgatcgtgctcctgtcgttgaggacccggctaggctggcggggttgccttact8340ggttagcagaatgaatcaccgatacgcgagcgaacgtgaagcgactgctgctgcaaaacg8400tctgcgacctgagcaacaacatgaatggtcttcggtttccgtgtttcgtaaagtctggaa8460acgcggaagtcccctacgtgctgctgaagttgcccgcaacagagagtggaaccaaccggt8520gataccacgatactatgactgagagtcaacgccatgagcggcctcatttcttattctgag8580ttacaacagtccgcaccgctgtccggtagctccttccggtgggcgcggggcatgactatc8640gtcgccgcacttatgactgtcttctttatcatgcaactcgtaggacaggtgccggcagcg8700cccaacagtcccccggccacggggcctgccaccatacccacgccgaaacaagcgccctgc8760accattatgttccggatctgcatcgcaggatgctgctggctaccctgtggaacacctaca8820tctgtattaacgaagcgctaaccgtttttatcaggctctgggaggcagaataaatgatca8880tatcgtcaattattacctccacggggagagcctgagcaaactggcctcaggcatttgaga8940agcacacggtcacactgcttccggtagtcaataaaccggtaaaccagcaatagacataag9000cggctatttaacgaccctgccctgaaccgacgaccgggtcgaatttgctttcgaatttct9060gccattcatccgcttattatcacttattcaggcgtagcaccaggcgtttaagggcaccaa9120taactgccttaaaaaaattacgccccgccctgccactcatcgcagtactgttgtaattca9180ttaagcattctgccgacatggaagccatcacagacggcatgatgaacctgaatcgccagc9240ggcatcagcaccttgtcgccttgcgtataatatttgcccatggtgaaaacgggggcgaag9300aagttgtccatattggccacgtttaaatcaaaactggtgaaactcacccagggattggct9360gagacgaaaaacatattctcaataaaccctttagggaaataggccaggttttcaccgtaa9420cacgccacatcttgcgaatatatgtgtagaaactgccggaaatcgtcgtggtattcactc9480cagagcgatgaaaacgtttcagtttgctcatggaaaacggtgtaacaagggtgaacacta9540tcccatatcaccagctcaccgtctttcattgccatacg9578<210>146<211>386<212>DNA<213>人工<220><223>从pNB100作为模板用引物NB018和NB019扩增的序列<400>146ccaaaactgagacctgctgcggacgtccaaaggtctcgttttagagctatgctgttttga60atggtcccaaaacttgccgatgataacttgagaaagagggttaataccagcagtcggata120ccttcctattctttctgttaaagcgttttcatgttataataggcaaattttagatgaaga180ttatttcttaataactaaaaatatggtataatactcttaataaatgcagtaatacagggg240cttttcaagactgaagtctagctgagacaaatagtgcgattacgaaattttttagacaaa300aatagtctacgaggttttagagctatgctgttttgaatggtcccaaaactgaagagcgtc360tcggacgcagcgctcttcgttttaga386<210>147<211>744<212>DNA<213>人工<220><223>pNB200中的经修饰的CRISPR阵列<400>147ggtgactgatggccacgtgaactatatgattttccgcagtatattttagatgaagattat60ttcttaataactaaaaatatggtataatactcttaataaatgcagtaatacaggggcttt120tcaagactgaagtctagctgagacaaatagtgcgattacgaaattttttagacaaaaata180gtctacgaggttttagagctatgctattttgaatggtcccaaaactgagacctgctgcgg240acgtccaaaggtctcgttttagagctatgctgttttgaatggtcccaaaacttgccgatg300ataacttgagaaagagggttaataccagcagtcggataccttcctattctttctgttaaa360gcgttttcatgttataataggcaaattttagatgaagattatttcttaataactaaaaat420atggtataatactcttaataaatgcagtaatacaggggcttttcaagactgaagtctagc480tgagacaaatagtgcgattacgaaattttttagacaaaaatagtctacgaggttttagag540ctatgctgttttgaatggtcccaaaactgaagagcgtctcggacgcagcgctcttcgttt600tagagctatgctgttttgaatggtcccaaaacaacattgccgatgataacttgagaaaga660gggttaataccagcagtcggataccttcctattctttctgttaaagcgttttcatgttat720aataggcaaaagaagagtagtgtg744<210>148<211>23<212>DNA<213>人工<220><223>引物序列<400>148ccaactacctccccttgcttaac23<210>149<211>18<212>DNA<213>人工<220><223>引物序列<400>149ggtgactgatggccacgt18<210>150<211>23<212>DNA<213>人工<220><223>引物序列<400>150gggctggcaagccacgtttggtg23<210>151<211>23<212>DNA<213>人工<220><223>引物序列<400>151ccgggagctgcatgtgtcagagg23<210>152<211>43<212>DNA<213>人工<220><223>引物序列<400>152attgaaaaaggaagagtatggaattgcccaatattatgcaccc43<210>153<211>38<212>DNA<213>人工<220><223>引物序列<400>153agtcccgctaggtctcaaccgtcagcgcagcttgtcgg38<210>154<211>42<212>DNA<213>人工<220><223>引物序列<400>154attgaaaaaggaagagtatgcgtgtattagccttatcggctg42<210>155<211>55<212>DNA<213>人工<220><223>引物序列<400>155agtcccgctaggtctcaaccgctagggaataattttttcctgtttgagcacttct55<210>156<211>47<212>DNA<213>人工<220><223>引物序列<400>156attgaaaaaggaagagtatgcgttattttcgcctgtgtattatctcc47<210>157<211>41<212>DNA<213>人工<220><223>引物序列<400>157agtcccgctaggtctcaaccgttagcgttgccagtgctcga41<210>158<211>57<212>DNA<213>人工<220><223>引物序列<400>158attgaaaaaggaagagtatgttaaaagttattagtagtttattggtctacatgaccg57<210>159<211>48<212>DNA<213>人工<220><223>引物序列<400>159agtcccgctaggatgacctggctgaccgctactcggcgactgagcgat48<210>160<211>43<212>DNA<213>人工<220><223>引物序列<400>160attgaaaaaggaagagtatgtcactgtatcgccgtctagttct43<210>161<211>39<212>DNA<213>人工<220><223>KPC-3反向引物<400>161agtcccgctaggtctcaaccgttactgcccgttgacgcc39<210>162<211>60<212>DNA<213>人工<220><223>引物序列<400>162attgaaaaaggaagagtatgagcaagttatctgtattctttatatttttgttttgtagca60<210>163<211>61<212>DNA<213>人工<220><223>引物序列<400>163agtcccgctaggtctcaaccgttagttgcttagttttgatggttttttactttcgtttaa60c61<210>164<211>44<212>DNA<213>人工<220><223>引物序列<400>164attgaaaaaggaagagtatggttaaaaaatcactgcgccagttc44<210>165<211>47<212>DNA<213>人工<220><223>引物序列<400>165agtcccgctaggtctcaaccgttacaaaccgtcggtgacgattttag47<210>166<211>834<212>DNA<213>未知<220><223>NDM-1序列<400>166accgtcagcgcagcttgtcggccatgcgggccgtatgagtgattgcggcgcggctatcgg60gggcggaatggctcatcacgatcatgctggccttggggaacgccgcaccaaacgcgcgcg120ctgacgcggcgtagtgctcagtgtcggcatcaccgagattgccgagcgacttggccttgc180tgtccttgatcaggcagccaccaaaagcgatgtcggtgccgtcgatcccaacggtgatat240tgtcactggtgtggccggggccggggtaaaataccttgagcgggccaaagttgggcgcgg300ttgctggttcgacccagccattggcggcgaaagtcaggctgtgttgcgccgcaaccatcc360cctcttgcggggcaagctggttcgacaacgcattggcataagtcgcaatccccgccgcat420gcagcgcgtccataccgcccatcttgtcctgatgcgcgtgagtcaccaccgccagcgcga480ccggcaggttgatctcctgcttgatccagttgaggatctgggcggtctggtcatcggtcc540aggcggtatcgaccaccagcacgcggccgccatccctgacgatcaaaccgttggaagcga600ctgccccgaaacccggcatgtcgagataggaagtgtgctgccagacattcggtgcgagct660ggcggaaaaccagatcgccaaaccgttggtcgccagtttccatttgctggccaatcgtcg720ggcggatttcaccgggcatgcacccgctcagcatcaatgcagcggctaatgcggtgctca780gcttcgcgaccgggtgcataatattgggcaattccatactcttcctttttcaat834<210>167<211>819<212>DNA<213>未知<220><223>OXA-48序列<400>167accgctagggaataattttttcctgtttgagcacttcttttgtgatggcttggcgcagcc60ctaaaccatccgatgtgggcatatccatattcatcgcaaaaaaccacacattatcatcaa120gttcaacccaaccgacccaccagccaatcttaggttcgattctagtcgagtatccagttt180tagcccgaataatatagtcaccattggcttcggtcagcatggcttgtttgacaatacgct240ggctgcgctccgatacgtgtaacttattgtgatacagctttcttaaaaagctgatttgct300ccgtggccgaaattcgaataccaccgtcgagccagaaactgtctacattgcccgaaatgt360cctcattaccataatcgaaagcatgtagcatcttgctcatacgtgcctcgccaatttggc420gggcaaattcttgataaacaggcacaactgaatatttcatcgcggtgattagattatgat480cgcgattccaagtggcgatatcgcgcgtctgtccatcccacttaaagacttggtgttcat540ccttaaccacgcccaaatcgagggcgatcaagctattgggaattttaaaggtagatgcgg600gtaaaaatgcttggttcgcccgtttaagattattggtaaatccttgctgcttattctcat660tccagagcacaactacgccctgtgatttatgttcagtaaagtgagcattccaacttttgt720tttcttgccattcctttgctaccgcaggcattccgataatcgatgccaccaaaaacacag780ccgataaggctaatacacgcatactcttcctttttcaat819<210>168<211>882<212>DNA<213>未知<220><223>SHV-1序列<400>168accgttagcgttgccagtgctcgatcagcgccgcgccgatcccggcgatttgctgatttc60gctcggccatgctcgccggcgtatcccgcagataaatcaccacaatccgctctgctttgt120tattcgggccaagcagggcgacaatcccgcgcgcaccccgtttggcagctccggtcttat180cggcgataaaccagcccgccggcagcacggagcggatcaacggtccggcgacccgatcgt240ccaccatccactgcagcagctgccgttgcgaacgggcgctcagacgctggctggtcagca300gcttgcgcagggtcgcggccatgctggccggggtagtggtgtcgcgggcgtcgccgggaa360gcgcctcattcagttccgtttcccagcggtcaaggcgggtgacgttgtcgccgatctggc420gcaaaaaggcagtcaatcctgcggggccgccgacggtggccagcagcagattggcggcgc480tgttatcgctcatggtaatggcggcggcacagagttcgccgaccgtcatgccgtcggcaa540ggtgtttttcgctgaccggcgagtagtccaccagatcctgctggcgatagtggatctttc600gctccagctgttcgtcaccggcatccacccgcgccagcactgcgccgcagagcactactt660taaaggtgctcatcatgggaaagcgttcatcggcgcgccaggcggtcagcgtgcggccgc720tggccagatccatttctatcatgcctacgctgcccgacagctggctttcgcttagtttaa780tttgctcaagcggctgcgggctggcgtgtaccgccagcggcagggtggctaacagggaga840taatacacaggcgaaaataacgcatactcttcctttttcaat882<210>169<211>822<212>DNA<213>未知<220><223>VIM-1序列<400>169accgctactcggcgactgagcgatttttgtgtgctttgacaacgttcgctgtgtgctgga60gcaagtctagaccgcccggtagaccgtgcccgggaatgacgacctctgcttccgggtagt120gtttttgaatccgctcaacggaggtgggccattcagccagatcggcatcggccacgttcc180ccgcagacgtgcttgacaactcatgaacggcacaaccaccgtatagcacgttcgctgacg240ggacgtatacaaccagattgtcggtcgaatgcgcagcaccaggatagaagagctctactg300gaccgaagcgcactgcgtccccgctcgatgagagtccttctagagaatgcgtgggaatct360cgttcccctctgcctcggctagccggcgtgtcgacggtgatgcgtacgttgccaccccag420ccgcccgaaggacatcaacgccgccgacgcggtcgtcatgaaagtgcgtggagactgcac480gcgttacgggaagtccaatttgcttttcaatctccgcgagaagtgccgctgtgtttttcg540caccccacgctgtatcaatcaaaagcaactcatcaccatcacggacaatgagaccattgg600acgggtagaccgcgccatcaaacgactgcgttgcgatatgcgaccaaacaccatcggcaa660tctggtaaagtcggacctctccgaccggaatttcgttgactgtcggatactcaccactcg720gctccccggaatgggctaacggacttgcgacagccatgacagacgcggtcatgtagacca780ataaactactaataacttttaacatactcttcctttttcaat822<210>170<211>903<212>DNA<213>未知<220><223>KPC-3序列<400>170accgttactgcccgttgacgcccaatccctcgagcgcgagtctagccgcagcggcgatga60cggcctcgctgtacttgtcatccttgttaggcgcccgggtgtagacggccaacacaatag120gtgcgcgcccagtgggccagacgacggcatagtcatttgccgtgccatacactccgcagg180ttccggttttgtctccgactgcccagtctgccggcaccgccgcgcggatgcggtggttgc240cggtcgtgtttccctttagccaatcaacaaactgctgccgctgcggcgcagccagtgcag300agcccagtgtcagtttttgtaagctttccgtcacggcgcgcggcgatgaggtatcgcgcg360catcgcctgggatggcggagttcagctccagctcccagcggtccagacggaacgtggtat420cgccgatagagcgcatgaaggccgtcagcccggccgggccgcccaactccttcagcaaca480aattggcggcggcgttatcactgtattgcacggcggccgcggacagctccgccaccgtca540tgcctgttgtcagatatttttccgagatgggtgaccacggaaccagcgcatttttgccgt600aacggatgggtgtgtccagcaagccggcctgctgctggctgcgagccagcacagcggcag660caagaaagcccttgaatgagctgcacagtgggaagcgctcctcagcgcggtaacttacag720ttgcgcctgagccggtatccatcgcgtacacaccgatggagccgccaaagtcctgttcga780gtttagcgaatggttccgcgacgaggttggtcagcgcggtggcagaaaagccagccagcg840gccatgagagacaagacagcagaactagacggcgatacagtgacatactcttcctttttc900aat903<210>171<211>762<212>DNA<213>未知<220><223>IMP-4序列<400>171accgttagttgcttagttttgatggttttttactttcgtttaaccctttaaccgcctgct60ctaatgtaagtttcaagagtgatgcgtctccagcttcactgtgacttggaacaaccagtt120ttgccttaccatatttggatattaataatttagcggactttggccaagcttctaaatttg180cgtcacccaaattacctagaccgtacggtttaataaaacaaccaccgaataatattttcc240tttcaggcagccaaactactaggttatctggagtgtgtcctgggcctggataaaaaactt300caattttatttttaactagccaatagttaaccccgccaaatgaatttttagcttgaacct360taccgtcttttttaagcagctcattagttaattcagacgcatacgtggggatggattgag420aattaagccactctattccgcccgtgctgtcactatgaaaatgagaggaaatactgcctt480ttattttatagccacgttccacaaaccaagtgactaacttttcagtatctttagccgtaa540atggagtgtcaattagataagcttcagcatctacaagaacaaccaaaccatgtttaggaa600caacgccccacccgttaacttcttcaaacgaagtatgaacataaacgccttcatcaagtt660tttcaatttttaaatctggcaaagactctgctgcggtagcaatgctacaaaacaaaaata720taaagaatacagataacttgctcatactcttcctttttcaat762<210>172<211>897<212>DNA<213>未知<220><223>CTX-M-15序列<400>172accgttacaaaccgtcggtgacgattttagccgccgacgctaatacatcgcgacggcttt60ctgccttaggttgaggctgggtgaagtaagtgaccagaatcagcggcgcacgatcttttg120gccagatcaccgcgatatcgttggtggtgccatagccaccgctgccggttttatccccca180caacccaggaagcaggcagtccagcctgaatgctcgctgcaccggtggtattgcctttca240tccatgtcaccagctgcgcccgttggctgtcgcccaatgctttacccagcgtcagattcc300gcagagtttgcgccattgcccgaggtgaagtggtatcacgcggatcgcccggaatggcgg360tgtttaacgtcggctcggtacggtcgagacggaacgtttcgtctcccagctgtcgggcga420acgcggtgacgctagccgggccgccaacgtgagcaatcagcttattcatcgccacgttat480cgctgtactgtagcgcggccgcgctaagctcagccagtgacatcgtcccattgacgtgct540tttccgcaatcggattatagttaacaaggtcagattttttgatctcaactcgctgattta600acagattcggttcgctttcacttttcttcagcaccgcggccgcggccatcactttactgg660tgctgcacatcgcaaagcgctcatcagcacgataaagtatttgcgaattatctgctgtgt720taatcaatgccacacccagtctgcctcccgactgccgctctaattcggcaagtttttgct780gtacgtccgccgtttgcgcatacagcggcacacttcctaacaacagcgtgacggttgccg840tcgccatcagcgtgaactggcgcagtgattttttaaccatactcttcctttttcaat897当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1