在种和/或亚种水平鉴定从临床样品分离的菌株的方法与流程
本发明涉及鉴定从临床样品分离的菌株的方法。更具体而言,涉及通过使用基质辅助激光解吸电离飞行时间质谱(MALDI-TOF-MS),在种和/或亚种水平精确并快速鉴定临床分离物的方法。其还涉及用于该鉴定的数据库。
MALDI-TOF-MS是用于检查直接从完整细菌细胞表面检测到的蛋白质的特征谱的技术。
这是一种基于相对分子质量的软电离方法,允许从整个的不同培养微生物中解吸附(desorption)肽和蛋白质。根据其分子质量和电荷来分离并检测离子。通过其质量:电荷比率(m/z)来鉴定种。该方法在数分钟内获得可重复的谱图(spectrum),其由500-20000m/z的一系列峰组成。每个峰对应于在激光解吸附期间从细胞表面释放的分子片段。
MALDI-TOF-MS已经被用于表征细菌。然而,在谱图中鉴定的多种组分中,只有一些出现在一大群的给定基因种的病菌(germ)中,其他则是分离特异性的或根据生长条件(温育的培养基、温度…)变化并且不能用于鉴定细菌以及更通常为病菌的种或亚种。
缺乏鉴定能够区分不同基因种的那些峰的谱图的全局性策略,阻碍了MALDI-TOF-MS在常规临床微生物实验室中用于诊断基因种的应用。实际上,这些峰必须对应于在给定基因种的分离物的大部分中出现的组分,以及大量存在于细胞壁中组分。考虑到基因种中可能的易变性,似乎是基因种的特征不依赖于单个峰的出现而是依赖于一组峰的出现,这组峰或多或少地在基因种间是保守的。
本发明人已经发现可以通过如下方法克服这些问题:(i)通过在MALDI-TOF-MS分析前对待鉴定病菌进行表型分析,和(ii)通过在特定条件下,特别是涉及基质溶液和参照数据库的选择,进行MALDI-TOF-MS分析。
从而本发明的一个目的是提供基于MALDI-TOF-MS分析的新方法,用于在种和/或亚种水平鉴定从临床样品分离的菌株。
本发明的另一个目的是提供由针对于给定病菌的一组菌株而建立的数据库。
因此本发明涉及使用MALDI-TOF-MS分析,在种和/或亚种水平鉴定从临床样品分离的菌株的方法,其包括在进行MALDI-TOF-MS分析前将所述菌株分类的步骤,其中所述分析包括在测试菌株的谱图中保留如下峰的步骤,与具有最高强度并任意设置为1的峰相比,所述峰的相对强度大于0.02,特别地大于0.05,更特别地大于0.1。
本发明特别涉及如下方法,其中所述MALDI-TOF-MS分析包括将待鉴定菌株的谱图特征谱与称为参照数据库相比较,所述参照数据库包含待鉴定菌株所属病菌组的基因种的代表性菌株的特征性MALDI-TOF-MS谱图的,所述数据库对于每个代表性菌株包含这样的峰,与具有最高强度并任意设置为1的峰相比,所述峰的相对强度大于0.02,特别地大于0.05,更特别地大于0.1,直至其出现在至少2种亚培养物,优选5种或甚至10种或更多种亚培养物中。
根据本发明进行的峰的选择使得能够准确鉴定在临床样品中存在的病菌。
分类步骤基于生长条件,和/或菌落的特征,和/或通过显微镜检查的细菌形态,和/或革兰氏染色,和/或简单的表型测试。
在全部完整细胞上或者在裂解后得到的或对应于细胞组分(细胞壁、细胞质等)的级分的蛋白质提取物上进行MALDI-TOF-MS技术。该技术在待测菌株的分离的菌落上可以理想地进行,所述菌株得自在合适的生长培养基上培养并在合适的温度下孵育的临床样品。
在进行MALDI-TOF-MS技术前,将基质培养基添加到分离菌落中。
例如,合适的基质培养基包括苯甲酸衍生物,例如羟基苯甲酸,特别是2,5-二羟基苯甲酸(DHB)。
每个谱图是来自含有待分析菌株的孔的不同区域的至少400个,优选地大于1000个,甚至大于2000个激光发射的总和,在m/z500-50000,更优选地500-2000,更优选地2000-20000的范围内分析所述谱图。
随后使用软件比较特征谱,所述软件通过分析给定基因种或基因亚种(genosubspecies)特异的一种或几种参照菌株,选择受试菌株和所述参照数据库的所选峰之间的最佳匹配。每种菌株代表所研究的一个基因种或一组基因种,从而一组菌株对应于一组病菌的相关基因种或基因亚种。使用MALDI-TOF-MS技术记录每种参照菌株的谱图。如上所述,将具有最高强度并任意设置为1的峰,所有其他峰具有相对于该最高峰的相对强度的数值。保留具有相对强度大于0.02,或更特别地大于0.05或更特别地大于0.1的峰,直至它们出现在至少2种亚培养物,优选5种或甚至10种或更多种亚培养物中。这些峰就是所选的峰。
考虑到一些可能的变体,软件选择受试菌株和数据库之间的最佳匹配,将m/z值得可能误差考虑进去。误差的范围任意设置为+/-20或更优选地+/-10。
峰的存在或缺少被认为是特定分离物的指纹。
所述方法是用于快速鉴定病菌的有力工具,允许将其用于实验室的常规诊断中。昂贵设备的需求被需要最少的操作时间的非常简单的方案所弥补。尤其适合于得到大量的样品的参照实验室或医院特。
根据本发明进行的MALDI-TOF-MS方法的高分辨率使得例如获得给定病菌的每个种或甚至亚种的特征性谱图指纹成为可能。
有利地,所述方法用于在种和/或亚种水平鉴定选自细菌、酵母、霉菌的病菌。
根据特定的实施方式,本发明涉及凝固酶阴性葡萄球菌(Staphyloccocci)(缩写为CNS)的种和/或亚种的鉴定。
在种的水平鉴定CNS不是常规进行的,因为其经常被视为样品污染。另外,对临床上重要的菌株进行的表型方法的结果是令人失望的。商业鉴定试剂盒对于鉴定CNS分离物只有有限的价值。
SodA序列测定尽管是鉴定CNS正确的种的更准确方法,但其费时、昂贵且在技术上有要求。另外,尽管基因型方法明显优于表型鉴定,但基于序列的基因型方法的一个缺点是缺少数据库中的保存序列的质量。
归功于本发明,通过MALDI-TOF-MS相比于SodA方法在亚种水平,例如对于葡萄球菌的亚种的区分更加准确了,因为证明在头状葡萄球菌(S.capitis)、人葡萄球菌(S.hominis)、腐生葡萄球菌(S.saprophyticus)和施氏葡萄球菌(S.schleiferi)亚种中SodA基因没有显著区别。
根据另一个实施方式,本发明涉及鉴定非发酵革兰氏阴性杆菌的种或亚种。
在所述方法中使用的数据库也是本发明的部分。
它们构成了通过MALDI-TOF-MS分析菌株获得的一组所选的峰,所述菌株在属于相同基因种的菌株内至少是部分保守的。
优选地,所述参照峰对应于m/z大于0.02,更特别地大于0.05,更特别地大于0.1的主要的、保守的峰。
通过使用一组菌株,每个菌株代表研究中组的一个基因种,来有利地获得对于每个组的这种数据库,从而该组菌株对应于一组病菌的相关基因种。使用MALDI-TOF-MS技术记录这样的组的每个菌株的谱图。每个菌株代表了该组病菌的基因种,并且被进一步指定为参照菌株。如上所述,将具有最高强度并任意设置为1的峰,所有其他的峰具有对应于该最高峰的相对强度的数值。保留主要的峰,这些峰优选地具有至少0.02,更优选大于0.05,更优选大于0.1的相对强度。随后保留在对于给定参照菌株获得的所有数据组中出现的、具有相对强度大于0.02,更优选大于0.05,更优选大于0.1的峰,直至它们出现在至少2种亚培养物,优选地5种或者甚至10种或更多种亚培养物中。
由此获得的一组峰在给定生长条件下(培养基和持续时间)以及鉴于给定谱仪的特征,是基因种特异的。
对于鉴定1)在种的水平的CNS和2)在囊肿性纤维化中分离的非发酵革兰氏阴性杆菌,本发明的其他特征和优点如下。它们用于示例说明本发明,而不是限制本发明的范围。在本文中图1-7中分别表示
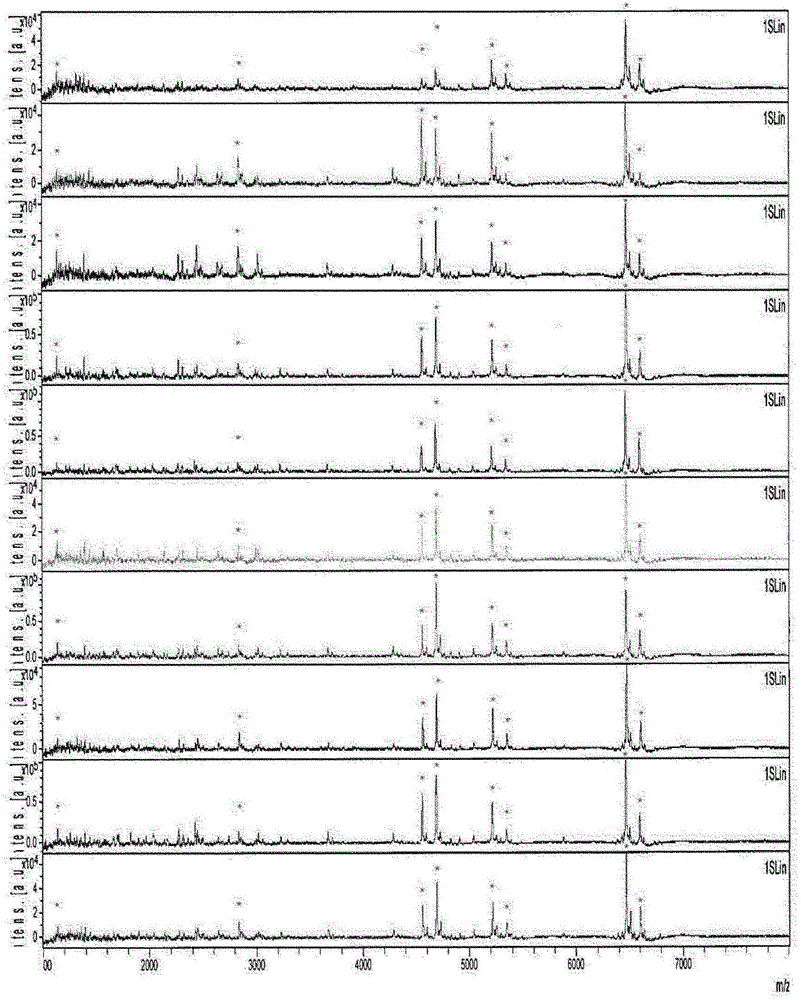
-图1:金黄色葡萄球菌(S.aureus)CIP7625的相同菌株的10个分离物的MALDI-TOF-MS特征谱;
-图2:根据数据库1(Muёller Hinton 24H)和2(Muёller Hinton 48H)的微球菌属(Micrococcacae)物种的MALDI-TOF-MS系统树图,从而指定在该组病菌中待鉴定的基因种;
-图3:对于葡萄球菌属(Staphyloccocci)菌株,具有相对强度大于0.1的峰的m/z值;
-图4和5:在属于相同基因种的菌株中,所选菌株的一组峰的保守性;
-图6:需氧和兼性厌氧细菌的分类;
-图7:6种革兰氏阴性杆菌的MALDI-TOF-MS特征谱。
1.在种的水平鉴定CNS
材料和方法
细菌菌株
通过分析属于微球菌属细菌的51个菌株得到下文给出的结果,这些菌株从巴斯德研究所(Institut Pasteur,Paris,France)的保藏中心获得。
用于建立MALDI-TOF-MS数据库的所选菌株在表1中给出
表1
属于微球菌科的菌株在表2中给出。
表2
*:使用不区分2个亚种的sodA基因的序列鉴定这些临床菌株的基因种
也研究了116个临床凝固酶阴性葡萄球菌(CNS)分离物:74个菌株分离自血液培养物,25个菌株分离自儿童纵隔炎(皮肤、纵隔液、电极),12个菌株分离自骨骼感染(Necker-Enfants malades,Paris;Raymond Poincaré,Garches,France),以及5个菌株分离自尿道感染。另外,分析了从混杂的感染中分离的金黄色葡萄球菌的68个临床菌株。
表型和基因型鉴定
通过包括Slidex乳胶凝集试验(bioMérieux,Marcy I’Etoile,France)和DNA酶试验的常规表型试验区分来自金黄色葡萄球菌的CNS临床菌株。在这两个试验不相符合的情况下,进行试管凝固酶试验。通过对如上所述的SodA基因的内部片段测序来获得CNS的精确鉴定。简言之,使用Quiagene试剂盒(Courtaboeuf,France)从CNS纯培养物中进行基因组DNA的提取。扩增部分SodA基因,并且使用ABI Big Dye Terminator v1.1循环测序读取反应试剂盒(Applied Biosystems,Foster City,CA,USA),如Poyart等人2001,J.Clin.Microbiol.39:4296-4301之前所述的对所得的扩增子测序。将核酸序列发送至GenBank数据库用于物种分配。
使用该策略,鉴定了在表2中给出的116个临床CNS分离物。
MALDI-TOF-MS技术
将菌株培养在Müeller Hinton琼脂或Columbia琼脂+5%马血(bioMérieux)上,在37℃温育24或48小时。对于每次鉴定,将分离的菌落收集在100μl的无菌水中;以3次重复将1μl的该混合物置于靶平板(Bruker Daltonics,Bremen,Germany)上并使之在室温干燥。随后在每个孔中加入1μl无水乙醇,以及1μl基质溶液DHB(2.5二羟基苯甲酸50mg/ml、乙腈30%、三氟乙酸0.1%)。干燥后,加入1μl基质溶液DHB。随后用具有flex控制软件(Bruker Daltonics)的谱仪MALDI-TOF-MS autoflex(Bruker Daltonics)处理样品。以线性模式,20Hz的加速电压提取阳离子。每个谱图是来自相同孔的不同区域的5x200个激光发射的离子的总和,并且在m/z1000-23000的范围内分析每个谱图。使用flex分析软件进行该分析,并用蛋白质校准标准T(蛋白质I,Bruker Daltonics)进行校准。将3次重复中获得的数据加和以最小化随机效应。峰的出现和缺失被视为特定分离物的指纹。使用可在网站http://sourceforge.net/projects/bgp获得的软件BGP-数据库对这些特征谱进行分析和比较。
结果
表1中所列的菌株是作为基因种代表的任意选出的菌株。
如在材料和方法部分中所述,通过MALDI-TOF-MS分析这些所选菌株中每一个的10个分离物。
对于每个谱图,对每个峰给出对应于强度的数值。
将具有最高强度的峰任意设置为1,所有其他峰具有对应于该最高峰的相对强度的数值(图1)。
小峰(相对强度小于0.1)的出现不规律。随后该分析集中在对于给定的菌株所得的所有10组数据中存在的、具有相对强度大于0.1的峰。
为了确定生长条件对细菌鉴定的影响,对培养在Müeller Hinton琼脂或补充5%马血的Columbia琼脂上24或48小时的表1中所列的所有菌株进行上述步骤。
结果在表3中给出。
数据库分别指定为1-4:它们通过使用在Müeller Hinton 24H(数据库1),Müeller Hinton 48H(数据库2),Columbia马血培养基24H(数据库3),Columbia马血培养基24H(数据库4)上的细菌培养物获得。对于给定的菌株,每个保守峰的m/z数值的标准偏差从没有大于7。具有大于0.1的相对强度并且在每种生长条件的所有10组数据中都存在的峰的m/z数值在图3中报道。
表3
*:括号内的数目是具有数据库1的那些的常见峰的数目
**:迟缓微球菌在马血培养基上不生长
表3显示了对于每个所选菌株和每个数据库,已保留的峰的数目。然而,对于某个菌株,无论培养基或者培养时间如何有些峰都是保守的,而另一些峰随改变培养条件而消失或出现。然而,对于给定的培养基和培养时间,一组峰是菌株特异性的,如树状图所示(图2)。
在属于相同基因种的菌株中每个所选菌株的一组峰的保守性
为说明此点,使用在37℃在Müeller Hinton培养基上培养24小时的细菌进行MALDI-TOF MS。所用菌株是表2所列的分离物。
对于每个菌株,保留数值大于0.1的单峰。随后就这些分离物的每一个,将所获得的特征谱与数据库1的那些的特征谱相比较。
为了进行该任务,开发了软件(在http://sourceforge.net/projects/bgp可获得的BGP-数据库)以允许快速鉴定数据库中与受试菌株的数值最接近的一组数值。考虑到已经测量的m/z数值的可能误差,该软件选择受试菌株和数据库的参照菌株之间的最佳匹配。该数值被任意地设置为多至7。
受试菌株经常与属于数据库的相同基因种的菌株具有最佳匹配。所有的数据记录在图4和图5。
在Müeller Hinton培养基上培养24小时的受试菌株的保守峰的平均数目与每个数据库的峰的数目相比较
通过在Müeller Hinton培养基上培养受试细菌24小时期间获得的相同组数据针对数据库2、3和4进行试验。
结果在表4中给出。
表4
*在亚种水平不能区分、不能鉴定的菌株
**鉴定3个腐生葡萄球菌牛亚种
***鉴定6个人葡萄球菌人亚种
****鉴定1个腐生葡萄球菌腐生亚种
*****在人葡萄球菌人亚种和人葡萄球菌novobiosepticus亚种之间不能区分的3个菌株
表4显示了对于每个基因种,在受试菌株和4个数据库的每个之间保守的峰的平均数目。
既使当使用与受试菌株所用的不同培养条件建立用于基因种鉴定的数据库时,峰的数目是保守,也可能在所有情况下在种的水平上进行鉴定。
通过使用数据库2、3和4所观察到的与使用数据库1获得的那些之间唯一的不同是:不同于使用数据库1得到的结果,不可能在亚种水平的鉴定人葡萄球菌和腐生葡萄球菌。
总体来说,所述数据证明通过选择适当的一组菌株并只保留具有大于0.1的m/z的保守峰,可以改造用于基因种鉴定的数据库。另外,这些峰的保守性使得既使待鉴定菌株已经使用不同于用于改造数据库的培养条件培养,也仍然可能进行基因种的鉴定。
2.鉴定非发酵革兰氏阴性杆菌
第一步是针对属于在人体中回收(recover)的一群非发酵革兰氏阴性杆菌的所有种建立完整的数据库革兰氏。随后验证该数据库,以使用MALDI-TOF MS来鉴定在1年期间的囊性纤维化(CF)患者中回收的所有临床非发酵革兰氏阴性杆菌。。
材料和方法
细菌菌株
用于改造MALDI-TOF MS数据库的参照菌株属于可以从患者中回收的非发酵革兰氏阴性杆菌的52个种。这些菌株列于表4中。
表4
从06年1月1日到06年12月31日之间在Necker-Enfants Malades医院(Paris,France)小儿科就诊的CF儿童的痰液中回收非发酵革兰氏阴性杆菌的临床分离物。简言之,将显示绿色或黄绿色着色,氧化酶试验阳性,生长在42℃,生长在溴棕三甲铵琼脂并对结肠霉素易感的分离物被鉴定为铜绿假单胞菌。不显示那些标准的分离物通过API 20NE系统鉴定。使用APILAB PLUS软件包解释说明API 20NE试验的结果。当使用该软件获得的结果不被视为相应的对于铜绿假单胞菌、木糖氧化产碱菌木糖氧化亚种和嗜麦芽糖寡养单胞菌的良好鉴定时,通过测序16S rDNA基因的内部片段来鉴定这些细菌。
在该研究中使用的所有细菌保存在-80℃补充有15%甘油的胰胨豆胨培养液。
MALDI-TOF-MS。将菌株培养在Mueller-Hinton琼脂上并在37℃温育24h。大多数分离物在24h后生长,而将24h后不生长的那些菌株进一步温育48h或72h。将分离的菌落收集在100μl的无菌水中;以3次重复将1μl的该混合物放置在靶平板(Autoflex;Bruker Daltonics,Bremen,Germany)上并使之在室温干燥。随后在每个孔中加入1μl无水乙醇,并使混合物干燥。然后加入1μl基质溶液DHB(2.5二羟基苯甲酸50mg/ml、乙腈30%、三氟乙酸0.1%)。用具有flex控制软件(Bruker Daltonics)的MALDI-TOF-MS谱仪(Bruker Daltonics)中处理样品。以线性模式,20kV的加速电压引出(extract)阳离子。每个谱图是在相同孔的5不同区域的200个激光发射的离子的总和。在m/z 2,000-20,000的范围内分析谱图。使用flex分析软件进行该分析,并用蛋白质校准标准T(蛋白质I,Bruker Daltonics)进行校准。将3次重复中获得的数据加和以最小化随机效应。峰的出现和缺失被视为特定分离物的指纹。使用可在网站http://sourceforge.net/projects/bgp获得的软件BGP-数据库对这些特征谱进行分析和比较。
结果
非发酵革兰氏阴性杆菌数据库的改造
将表4中所列的参照菌株用于确定通过MALDI-TOF-MS获得的m/z数值,其构成每个细菌种的特征性谱图并且随后可用于细菌鉴定。图7显示了使用非发酵革兰氏阴性杆菌的6个种获得的谱图。在Mueller Hinton上培养24H的这些所选菌株中每一个的10个分离物通过在材料和方法部分中描述的MALDI-TOF-MS来进行分析。对于每个谱图,采用了对微球菌使用的相同策略。根据该策略,只保留在对于给定菌株所获得的所有10组数据中一直存在的、具有大于0.1的强度的那些峰。对于每个菌株,每个保守峰的标准偏差的m/z数值(标准化的数据)从没有超过8。
表5显示了关于8个种,为数据库保留的峰的数值。
对每个参照菌株保留的一组峰给出了特征性谱图。
鉴定临床菌株
进行实验以确定上述数据库是否可以用于临床非发酵革兰氏阴性杆菌的鉴定,从而证明每个所选菌株的一组峰至少部分地在相同种的分离物中是保守的。从2006年1月到12月,回收非发酵革兰氏阴性杆菌的811个菌株,并通过表型试验和分子方法进行鉴定:699个铜绿假单胞菌(120个患者),54个木糖氧化产碱菌(12个患者),32个嗜麦芽糖寡养单胞菌(12个患者),9个解甘露醇罗尔斯顿菌(1个患者),14个Bcc(2个患者),1个唐菖蒲伯克氏菌,1个欣氏博德特氏菌和1个I.limosus。在这些其中,在400个铜绿假单胞菌菌株的组和在所有剩余的非铜绿假单胞菌菌株进行MALDI-TOF-MS分析。对于每一个分离物,谱图的所有m/z数值都考虑在内。对于每个菌株,无论其强度如何保留所有的峰,并使用BGP-数据库软件将其与所述数据库的峰进行比较。考虑到m/z数值的可能误差,该软件选择受试菌株和所述数据库之间的最佳匹配。将该数值设置为。将菌株鉴定为属于所述数据库的、给出最佳匹配的菌株的基因种。在如下表6中给出的结果显示了通过BGP软件提供的针对铜绿假单胞菌株获得的匹配。
表6
特征谱名称:铜绿假单胞菌
匹配:13/13
特征谱名称:稻栖黄单胞菌(Flavimonas orzyhabitans)
匹配:7/12
表7显示了对于所有临床菌株所获得的鉴定,与物种鉴定的第二选择相比较
MALDI-TOF-MS正确鉴定了100%的铜绿假单胞菌,100%的木糖氧化产碱菌和100%的嗜麦芽糖寡养单胞菌的菌株。MS在亚种水平鉴定木糖氧化产碱菌临床菌株。MS分析正确鉴定了11个BCC菌株为新洋葱伯克氏菌。对于剩余的3个BCC菌株,在新洋葱伯克氏菌和严格意义上的洋葱伯克氏菌之间没有差别。应当指出除了1个(由MS鉴定为皮氏罗尔斯顿菌)外的9个解甘露醇罗尔斯顿菌被MS正确地鉴定。
结论
这些结果证明根据本发明改造的数据库也适用于非发酵革兰氏阴性杆菌的精确的种鉴定。从而有可能,通过使用MALDI-TOF-MS从非发酵革兰氏阴性杆菌的一个菌落(培养条件、菌落特征)中,在数分钟内获得准确的鉴定,而不需要另外的试验。除了3个新洋葱伯克氏菌和1个解甘露醇罗尔斯顿菌外,在种的水平准确地鉴定了受试的CF临床菌株。有趣地注意到鉴定为唐菖蒲伯克氏菌的菌株也与椰毒唐菖蒲伯克氏菌的参照菌株相匹配,后者是被证明为唐菖蒲伯克氏菌的次异名(junior synonym)的细菌物种。
MALDI-TOF-MS能够非常快速地鉴定细菌,而通过API 20NE的常规生化鉴定通常需要24h-48h以上,并经常有鉴定错误。因此MALDI-TOF-MS菌株鉴定是用于革兰氏阴性杆菌物种鉴定的非常有趣的方法,并且有助于理解它们的临床相关性和分布。
- 还没有人留言评论。精彩留言会获得点赞!