化疗方案选择的制作方法
本发明涉及一种特别用于个人化疗方案选择的试剂盒,包括化疗方案选择的方法和反应系统,以及对化疗方案的疗效预测。
背景技术:
乳腺癌是一类由多种基因突变引起的异质性疾病(癌症基因组图谱网络,2012)。在过去,临床病理特征和预后相关因素,包括肿瘤大小,是否累及淋巴结,病理分级和患者年龄,被医生们广泛地运用在选择化疗方案上,但常导致相当数量的患者过度治疗或治疗不足。现在知道患者即使病理特征相似,也可能对相同的化疗方案反应不同,虽然具体机理目前尚未清楚。最近雌激素受体(ER),上皮生长因子受体2(HER2)和孕激素被证实为预后指标,所以这三者成为评估乳腺癌的常规检查指标。微芯片和第二代基因测序等高通量基因组技术的发展使得基于患者基因组情况的个人化癌症治疗(PCT)成为可能。(Oakman,et al.,2010,Dotan,et al.,2010,Eng-Wong,et al.,2010)由于从这些基因组技术获得的个人基因组信息比起临床常规参数能更准确地预测患者的化疗反应,因此在个人化癌症治疗中,一系列基因标记(genetic markers)将从大量基因信息中仔细选出,结合患者临床信息,建立一个预测治疗反应的模型。化疗中需要做两个决定:是否采用化疗;以及采用哪种化疗。个人化癌症治疗对做这两步的决定都有帮助。目前已发现许多基因标识可用来预测乳腺癌治疗后的生存率和复发率(van de Vijver,et al.,2002,Paik,et al.,2006,Wang,et al.,2005,van’t Veer,et al.,2002,Mook,et al.,2007,Straver,et al.,2010,Buyse,et al.,2006,Foekens,et al.,2006,Look,et al.,2002,Harbeck,et al.,2013),并用于决定是否采用更积极的治疗方案,比如化疗。当前检测21个基因表达的检测试剂盒Oncotype X已广泛用于针对乳腺癌的预后评估(http://www.oncotypedx.com)。针对某一特定治疗方案或者针对某一群患者的综合治疗的预测评估研究也已经有报道(Hatzis,et al.,2011,Graeser,et al.,2010,Shen,et al.,2012,Esserman,et al.,2012,Miyake,et al.,2012,Lips,et al.,2012,Hess,et al.,2006,Takada,et al.,2012,Albain,et al.,2010,Liu,et al.,2012)。但从多个化疗方案中选出并制定个人化癌症治疗方案并无报道。病人接受化疗后,也并无合适的治疗指导来选择 最优方案。过去研究中忽略了一个重要问题,就是当患者决定采用某种化疗方案后,个人化癌症治疗如何帮助患者选择接下来的化疗方案,使治疗效果更为有效。原理上,如果所有患者对目前的化疗方案有一致的疗效,那么个人化癌症治疗并无益处,除非有新的化疗方案介入。此外,考虑到相当数量的患者对至少两种治疗方案有不同反应(他们是个人化癌症治疗的潜在受益者),如何选出这些患者并给予他们更有效的治疗方案成为另外一个挑战。
因此,我们重新分析了以前研究中接受过新辅助化疗的乳腺癌患者的数据。乳腺癌患者可以在癌症不同时期接受化疗:手术前,手术后和转移后。新辅助化疗常常用来消除肿瘤大小以便于手术摘除。乳腺癌中常用的两种细胞毒性化疗试剂是:蒽环类药物及紫杉烷药物。这两类药物的不同组合已经应用在乳腺癌的治疗中,尽管目前尚无具体的治疗导向来帮助患者选择化疗方案(Dotan,et al.,2010)。我们从高通量基因数据库里收集了1111例乳腺癌患者的数据,有包括化疗效果在内的临床信息和基因表达数据等。(Gene Expression Omnibus(Edgar,et al.,2002,Barrett,et al.,2013)),其中化疗效果病理完全缓解用病理完全缓解(pathologic complete response)or残留病灶(residual disease)来表示。病理完全缓解已被证明是病人生存率的一种潜在的替代标记(Kaufmann,et al.,2006,Kuerer,et al.,1999,von Minckwitz,et al.,2012)并用于检测病人对化疗的敏感性(Straver,et al.,2010).在这1111个病例中,大约21.2%的患者对化疗显示病理完全缓解,剩余患者有残留病灶。我们将病理完全缓解和残留病灶的比例(pCR/RD)作为衡量化疗效果的参数,来考察基于基因组变量的个人化化疗方案是否能改进现有的病理完全缓解的比值。将患者分成3个组:只用蒽环类药物治疗的患者(A组),用蒽环类和紫杉醇药物治疗的患者(AT组),用蒽环类和多西紫杉醇治疗的患者(TxA组)。三组数据用随机森林模型进行处理,10折交叉验证用来检测模型的运行。临床变量包括ER,PR和HER2状态和基因变量(基因表达)被用作疾病预测因子。具备临床和基因变量的模型普遍比只有临床变量的模型更有效预测疗效。在A组中发现3个基因;在TA组中有5个基因,在TxA组中发现11个基因能有效预测病理完全缓解。我们还发现基于基因变量的个人化方案选择能使相当数量的患者在选择使用当前化疗方案时受益。基于模型预测的化疗结果,选择那些预计有更高的病理完全缓解的化疗方案用于治疗患者。我们预计采用这个新的PERS(个人化方案选择)治疗方案后,患者的病理完全缓解率为39.1%,相比于之前的21.2%增加了84%。大约有17.28%患者被过度治疗,9.63%患者存在不足治疗的问题。在考察的患者中,接受TxA方案的患者中的病理完全缓解病理完全缓解率为33.1%,比TA组(19.7%)和A组(8.6%)的比例高。如果我们希望患者的病理完全缓解病理完全缓解率最大化,应该基于患者的基因变量和临床 变量来选择化疗方案。如果基于患者的HER2,ER状态,和淋巴结是否累及,将患者进一步分层,也获得与以上类似的疗效。
技术实现要素:
本发明涉及一个用于化疗方案选择的基因诊断试剂盒。这个试剂盒包括:用于检测以下基因群中一个或多个基因的一个或多个试剂成份。这个基因群包括SLC12A7,GZMB,TAF6L,NFIB,METRN,ROPN1B,TTK,CCND1,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,C11orf17,CCL5,XCL1,XCL2,MELK,CTSL2,TPX2,AURKA,CDKN2C,BRP44,PNP,SMC4,NR4A2,C3orf37,MTPAP,CDC25B,ABCF1,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,TSPYL5,SRI,LRP12,CENPF,TUBD1,KIAA1324,DBF4,CCNA2,DLGAP5,FHL1,SIRT3,GTSE1,PCNA,CCNE2,CHD3,CAP1,GPM6B,GUSBP3,GNAI3,LMO4,PSRC1,USP1,STK38,BAT2L1,PMP22,NME5,CENPA,BANK1,和其衍生物大分子。
本发明还包括一种用于预测化疗方案疗效的方法。这个方法包括:
a)确定一个检测样品中至少有一种可用于预测化疗方案疗效的生物分子的表达;
b)对于上步确定有表达的一个或多个生物分子,用模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals)
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测化疗方案疗效的方法。这个方法包括:
a)确定一个样品至少表达有以下基因群中的至少两个生物分子。这个基因群包括:
SLC12A7,GZMB,TAF6L,NFIB,METRN,ROPN1B,TTK,CCND1,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,C11orf17,CCL5,XCL1,XCL2,MELK,CTSL2,TPX2,AURKA,CDKN2C,BRP44,PNP,SMC4,NR4A2,C3orf37,MTPAP,CDC25B,ABCF1,MTAP,SNAPC3,RANBP9, COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,TSPYL5,SRI,LRP12,CENPF,TUBD1,KIAA1324,DBF4,CCNA2,DLGAP5,FHL1,SIRT3,GTSE1,PCNA,CCNE2,CHD3,CAP1,GPM6B,GUSBP3,GNAI3,LMO4,PSRC1,USP1,STK38,BAT2L1,PMP22,NME5,CENPA,BANK1,和其衍生物。
本发明还包括用于预测仅使用蒽环类药物(不包括紫杉醇或多西紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:SLC12A7,GZMB,TAF6L,和其衍生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测同时使用蒽环类药物和紫杉醇药物(不包括多西紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:NFIB,METRN,ROPN1B,TTK,CCND1,和其衍生物大分子。
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测同时使用蒽环类药物和多西紫杉醇(不包括紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,和其衍生物大分子。
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全 缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2阴性癌症患者上只使用蒽环类药物(不包括紫杉醇和多西紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:SLC12A7,GZMB,C11orf17,TAF6L,CCL5,XCL1,XCL2,和其衍生生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2阴性癌症患者上使用蒽环类药物和紫杉醇药物(不包括多西紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:NFIB,ROPN1B,TTK,MELK,CTSL2,METRN和其衍生的生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2阴性癌症患者上使用蒽环类药物和多西紫杉醇药物(不包括紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:TPX2,PTTG1,MCM2,MCM6,AURKA,CDKN2C,BRP44,H2AFZ,PNP,SMC4,DEK,TMEM97,NR4A2,C3orf37,LZTFL1,MTPAP,CDC25B,ABCF1和其衍生的生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2和ER-阴性癌症患者上使用蒽环类药物和紫杉醇药物(不包括多西紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:NFIB,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24和其衍生的生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2和ER-阴性癌症患者上使用蒽环类药物和多西紫杉醇药物(不包括紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:TSPYL5,SRI和其衍生生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2阴性和ER-阳性癌症患者上使用蒽环类药物和紫杉醇药物(不包括多西紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:LRP12,CENPF,TUBD1,KIAA1324,TTK和其衍生生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2阴性和ER-阳性癌症患者上使用蒽环类药物和多西紫杉醇药物(不包括紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:DBF4,DEK,CDC25B,CCNA2,DLGAP5,MCM2,CDKN2C,FHL1,SIRT3,GTSE1,PCNA,CCNE2和其衍生生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2阴性和无淋巴累及的癌症患者上使用蒽环类药物和紫杉醇药物(不包括多西紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:CHD3,CAP1,GPM6B,GUSBP3和其衍生生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
c)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2阴性和无淋巴累及的癌症患者上使用蒽环类药物和多西紫杉醇药物(不包括紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:CDKN2C,GNAI3,LMO4,PSRC1,USP1,STK38和其衍生生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2阴性和有淋巴累及的癌症患者上使用蒽环类药物和紫杉醇药物(不包括多西紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:NFIB,ROPN1B和其衍生生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);
d)对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括用于预测在HER-2阴性和有淋巴累及的癌症患者上使用蒽环类药物和多西紫杉醇药物(不包括紫杉醇药物)的化疗方案疗效的方法。这个方法包括:
a)确定检测的样品中至少表达有以下基因群中的一个生物大分子。这个基因群包括:TPX2,BAT2L1,PMP22,PTTG1,NME5,CENPA,BANK1和其衍生生物大分子;
b)对于上步确定有表达的一个或多个生物分子,代入模型来计算化疗方案的病理完全缓 解的预测概率;
c)将病理完全缓解的预测概率分成一组与化疗方案相关的概率空间(probability intervals);对所述化疗方案的化疗结果的进行定量测量,其中化疗结果的定量测量对患者化疗方案有效性具有预测功能。
本发明还包括一个用于化疗方案选择的方法。这个方法包括:
a)确定用于检测的样品中至少表达有以下基因群中的两个生物大分子。这个基因群包括:SLC12A7,GZMB,TAF6L,NFIB,METRN,ROPN1B,TTK,CCND1,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,C11orf17,CCL5,XCL1,XCL2,MELK,CTSL2,TPX2,AURKA,CDKN2C,BRP44,PNP,SMC4,NR4A2,C3orf37,MTPAP,CDC25B,ABCF1,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,TSPYL5,SRI,LRP12,CENPF,TUBD1,KIAA1324,DBF4,CCNA2,DLGAP5,FHL1,SIRT3,GTSE1,PCNA,CCNE2,CHD3,CAP1,GPM6B,GUSBP3,GNAI3,LMO4,PSRC1,USP1,STK38,BAT2L1,PMP22,NME5,CENPA,BANK1和其衍生生物大分子;
本发明还包括一个用于化疗方案选择的方法。这个方法适用于患者样品中检测到以下包括:
a)确定检测的样品中至少表达有预测首个化疗方案疗效的第一组生物大分子。
b)将检测的第一组生物大分子代入第一个模型里,计算首个化疗方案的病理完全缓解的预测概率;将首个病理完全缓解的预测概率分成第一组与首个化疗方案相关的概率空间(probability intervals);
c)对所述首个化疗方案的化疗结果的进行第一次定量测量,其中首个化疗结果的定量测量对患者首个化疗方案有效性具有预测功能。
本发明还包括一个用于选择可预测化疗方案疗效的生物标记物的方法。这个方法包括:
a)用随机样品筛选法从一系列生物大分子中获得至少一个遗传预测因子;
b)建立一个模型,以至少一个(或多个)遗传预测因子作为变量,模型输出能对模型性能做定量测量;
c)确定以上模型的性能做定量测量;
d)至少重复一遍步骤b)–c),获得其他一个(或多个)模型和相应的模型性能的定量测量;
e)从一组生物大分子中选择一个生物标记物,将这个生物标记物作为变量之一,代入以上建成的模型中,对模型性能做局部最优化定量测量。
本发明还包括一个用于化疗方案疗效的选择系统。这个系统包括:
a)数据采集模块:数据采集模块可从特定患者的某个样品上收集包括诊断标记物概况的数据。诊断标记物中应该包括至少一个可用于对以下化疗方案疗效进行预测的生物大分子。化疗方案包括:(i)仅使用蒽环类药物(不包括紫杉醇药物和多西紫杉醇药物)(ii)使用蒽环类药物和紫杉醇药物(不包括多西紫杉醇药物)及(iii)使用蒽环类药物和多西紫杉醇药物(不包括紫杉醇药物);
b)数据处理模块:数据处理模块采用学习统计分类系统处理数据,对患者的化疗方案疗效生成统计来源的预测数据;
c)显示模块:显示模块可以有来显示统计来源的预测数据。
本发明的一个实施方案是用于受试者选择化疗方案的试剂盒。此试剂盒包括:用来检测以下至少两种生物分子表达的一个或多个试剂成份。生物分子包括:SLC12A7,GZMB,TAF6L,NFIB,METRN,ROPN1B,TTK,CCND1,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,C11orf17,CCL5,XCL1,XCL2,MELK,CTSL2,TPX2,AURKA,CDKN2C,BRP44,PNP,SMC4,NR4A2,C3orf37,MTPAP,CDC25B,ABCF1,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,TSPYL5,SRI,LRP12,CENPF,TUBD1,KIAA1324,DBF4,CCNA2,DLGAP5,FHL1,SIRT3,GTSE1,PCNA,CCNE2,CHD3,CAP1,GPM6B,GUSBP3,GNAI3,LMO4,PSRC1,USP1,STK38,BAT2L1,PMP22,NME5,CENPA,BANK1,和它们的衍生物。
在某些实施方案中,“选择一种化疗方案”和其语法变异的表达方式可指受试者有两个或更多的化疗方案可选择作为潜在的治疗方案。在某些实施方案中,“选择一种化疗方案”和其语法变异的表达方式可指受试者只有一种化疗方案作为潜在的治疗方案。因此,在本发明中,“选择一种化疗方案”的操作可以是医疗专业人员在多个化疗方案中选择最优方案,或是医疗专业人员决定是否对受试者采用化疗方案。
在某些实施方案中,“选择一种化疗方案”和其语法变异的表达方式所指的操作可 在受试者初次化疗给药之前或之后。当在最初化疗给药之前进行选择,所选择的化疗方案将优选在其后给予受试者。当在最初化疗给药后进行,所选择的化疗方案将与实际施用化疗方案进行比较,以确定进一步的疗程。
如本文所用,除非另有说明,“化疗方案”可指任何用在受试者上的化疗药物。本发明中的化疗药物包括,但不限于,DNA损伤剂,抗代谢药物,抗微管剂,和抗菌剂,等。DNA损伤剂包括:烷化剂,铂类药物,插层剂,以及DNA复制抑制剂。非限制性的DNA烷化剂例子包括环磷酰胺,氮芥,尿嘧啶氮芥,美法仑,苯丁酸氮芥,环磷酰胺,卡氮芥,洛莫司汀,链脲菌素,白消安,替莫唑胺,其可药用盐,前体药物,以及它们的组合。非限制性的铂类药物例子包括顺铂,碳化铂,奥沙利铂,奈达铂,沙铂,四硝酸三铂,其可药用盐,前体药物,以及它们的组合。非限制性的嵌入剂的例子包括蒽环类药物如阿霉素,柔红霉素,伊达比星,米托蒽醌,戊柔比星,表阿霉素,其药用盐,前体药物,以及它们的组合。非限制性的DNA复制抑制剂的例子包括伊立替康,拓扑替康,安吖啶,依托泊苷,磷酸依托泊苷,替尼泊甙,其药用盐,前体药物,以及它们的组合。抗代谢物包括叶酸拮抗剂如氨甲喋呤和培美曲塞,嘌呤受体拮抗剂如6-巯基嘌呤,达卡巴嗪,和氟达拉滨,和嘧啶拮抗剂如5-氟尿嘧啶,阿糖胞苷,卡培他滨,吉西他滨,地西他滨,其药用盐,前体药物,以及它们的组合。抗微管剂包括但不限于长春花碱,紫杉醇多西紫杉醇和抗菌剂包括但不限于放线菌素,博莱霉素,普卡霉素,丝裂霉素,其药用盐,前体药物,以及它们的组合。
如本文所用,“受试者”是哺乳动物,最好是人。除了人外,本发明范围中的哺乳动物类别包括:例如农场动物,家养动物和实验动物。农场动物的例子包括:奶牛,猪,马,山羊等。家养动物的例子包括:狗,猫等。实验动物的例子包括灵长类,大鼠,小鼠,兔,豚鼠等。
如本文所用,“受试者”可能表现出于本发明相关的各类基因特征。例如:本发明中的“受试者”包括,但不限于:HER2阳性受试者,HER2阴性受试者,HER2阴性ER-阴性受试者,HER2-阴性ER-阳性受试者,HER2阴性及淋巴结累及阳性受试者,HER2阴性及淋巴结累及阳性受试者。
如本文所用,本发明中用来检测生物分子表达的成份包括,例如,在实验室或临床环境中用于检测给定样品中的生物分子表达产物的任何底物,化合物,组合物,设备,试剂,或检测剂等。本发明中用来检测表达产物的成份包括,但不仅限于核酸,DNA,RNA,一 套引物,多个探针,蛋白,抗体,抗原结合片段,DNA芯片,RNA芯片,寡核苷酸芯片和蛋白质芯片。已知通常这类检测试剂可能采用不同的标记,例如放射性和荧光标记,来帮助检测基因表达。
如本文所用,一种“表达产物”指任何在给定样品中的存在可用来指示该样品中或者是抽样样本来源中有相应生物分子表达的物质;“表达产物”包括但不限于,一个给定的生物分子转录的信息核糖核酸(mRNA)或者是由以上mRNA翻译获得的蛋白。此外,表达产物包括从给定的生物分子转录的mRNA的任何片段,或从以上mRNA的任何片段翻译的蛋白片段。
如本文所用,“表达”指细胞里给定样品的数量的定性和定量测量。“表达”包括本发明中给定生物大分子的“表达水平”。本发明中的表达的适合的形式包括由细胞里给定的物质的绝对量和相对量。在本发明的上下文中,“表达”包括,但不限于,mRNA表达,蛋白质表达,非编码RNA的表达,和miRNA表达。
如本文所用,“生物分子”指存在细胞里或由细胞分泌的任何分子或者分子的任何部分。本发明中“生物分子”包括,但不限于基因和基因片段,转录mRNA和蛋白产物。此外,本发明中的生物分子还包括,但不限于非编码DNA,非编码RNA,例如tRNAs,rRNAs和miRNAs.”
如本文所用,“基因”是一个复杂的概念。为了便于阐述并不产生误解,此发明中“基因”指在一个生命体内所有可被处理并产生有功能的生物物质的核糖核酸序列。例如,基因可转录成mRNA,tRNA,或者核糖酶。然后mRNA可以继续翻译成蛋白产物,例如酶或抗体。本发明中生物分子的衍生物包括:表观遗传修饰的DNA,包括表观遗传修饰的基因或基因片段,转录后修饰RNA(如,但不限于,选择性剪接的RNA),和翻译后修饰的蛋白(如,但不限于,糖基化蛋白或蛋白水解活化蛋白)。
在一些实施方案中,要从有多个生物分子的组合里选出至少两个生物分子。因此,这两个生物分子是指2,3,4,5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,23,24,25,26,27,28,29,30,31,32,33,34,35,36,37,38,39,40,41,42,43,44,45,46,47,48,49,50,51,52,53,54,55,56,57,58,59,60,61,62,63,64,65,66,67,68,69,70,71,72,73,74,75,或更多生物分子.
对应于本发明中的生物分子的核苷酸和多肽序列都可以在公共数据库中查询到。
在一些实施方案中,所述试剂盒包括一个或多个组分,用于检测指定样品组中至少 三个生物分子的表达。
如本文中所用,至少需要检测到表达的三个生物分子指:3,4,5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,23,24,25,26,27,28,29,30,31,32,33,34,35,36,37,38,39,40,41,42,43,44,45,46,47,48,49,50,51,52,53,54,55,56,57,58,59,60,61,62,63,64,65,66,67,68,69,70,71,72,73,74,75,或更多生物分子。
在该实施方案的另一个方面,所述试剂盒包含一个或多个组分,用于检测从生物分子中组1,组2和组3中独立选择的至少两个生物分子的表达
a.组1:SLC12A7,GZMB,TAF6L,以及衍生物;
b.组2:NFIB,METRN,ROPN1B,TTK,CCND1,以及衍生物;
c.组3:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,以及衍生物。
如本文所使用的,“独立选择”是指,一个生物分子的选择的不影响另一生物分子的选择。
在该实施方案的另一方面,试剂盒包含一个或多个组分,可用于检测分别从第4组,第5组和第6组中选出的至少两个生物分子的表达:
a.第4组:SLC12A7,GZMB,C11orf17,TAF6L,CCL5,XCL1,XCL2,及其衍生物;
b.第5组:NFIB,ROPN1B,TTK,MELK,CTSL2,METRN,及其衍生物;
c.第5组:TPX2,PTTG1,MCM2,MCM6,AURKA,CDKN2C,BRP44,H2AFZ,PNP,SMC4,DEK,TMEM97,NR4A2,C3orf37,LZTFL1,MTPAP,CDC25B,ABCF1,及其衍生物。
在该实施方案的另一方面,试剂盒包含一个或多个组分,可用于检测分别从第7组,和第8组中选出的至少两个生物分子的表达:
a.第7组:NFIB,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,及其衍生物;
b.第8组:TSPYL5,SRI,及其衍生物.
在该实施方案的另一方面,试剂盒包含一个或多个组分,可用于检测分别从第9组,和第10组中挑选出的至少两个生物分子的表达:
a.第9组:LRP12,CENPF,TUBD1,KIAA1324,TTK,及其衍生物;
b.第10组:DBF4,DEK,CDC25B,CCNA2,DLGAP5,MCM2,CDKN2C,FHL1,SIRT3,GTSE1,PCNA,CCNE2,及其衍生物。
在该实施方案另一个方面,所述试剂盒包含一个或多个组分,用于检测分别从第11组,和第12组中挑选出的至少两个生物分子的表达:
a.第11组:CHD3,CAP1,GPM6B,GUSBP3,及其衍生物;
b.第12组:CDKN2C,GNAI3,LMO4,PSRC1,USP1,STK38,及其衍生物。
在该实施方案的另一方面,所述试剂盒包含一个或多个组分,用于检测分别从第13组,和第14组中挑选出的至少两个生物分子的表达:
a.第13组:NFIB,ROPN1B,及其衍生物;
b.第14组:TPX2,BAT2L1,PMP22,PTTG1,NME5,CENPA,BANK1,及其衍生物.
该实施方案的优选方面是,至少有两个选中的生物分子是来自至少两个不同组的生物分子。例如,一种生物分子可以选自第1组而另一个生物分子可以选自第2组。
该实施方案的更优选方面是,至少有两个选中的生物分子是两个选自同一组的生物分子。例如,一种生物分子可以选自第1组而另一个生物分子可以也选自第1组。
该实施方案的另一优选方面是,所述试剂盒包括一个或多个组分可检测以上提到的某组里的各个生物分子。例如,可检测第1组里的所有生物分子的组分。
该实施方案的更优选方面是,所述试剂盒包括一个或多个组分可以检测以上所述的至少两组里的各个生物分子。例如,可检测第1组和第2组里的所有生物分子的组分。另外,可检测第1组,第2组和第3组里的所有生物分子的组分。
该实施方案的更优选方面是,所述试剂盒包括一个或多个组分可以检测以上提到的各组的各个生物分子。例如,可检测第1组,第2组和第3组里的所有生物分子的组分。在另一例子中又可检测第13组,和第14组里的所有生物分子的组分。
在该实施方案的优选方面,所述的一个或多个组分可从包括以下分子的组中选择:DNA芯片,RNA芯片,寡核糖核酸片段芯片,蛋白芯片,抗体,抗原结合片段,多个探针,和一组引物。
本发明中的芯片矩阵可包括,例如,结合在芯片上,或者与上述芯片的特殊区域有相互作用的核糖核酸或蛋白。芯片上的特殊区域通常对应一个特定的种类,例如基因,mRNA, cDNA,核糖核酸探针或者蛋白探针。本发明中的芯片矩阵可为任何大小,例如可以是2X 2或者100X 100芯片矩阵。
本发明中的抗原结合片段包括,但不限于,抗体片段例如scFvs或Fabs,和受体,例如细胞表面受体,和受体片段等。
本发明中的引物包括,但不限于,用于多聚酶链式反应的引物和逆转录酶多聚酶链式反应的引物。
本实施方案的另一方面,从由mRNA表达,蛋白表达,非编码RNA表达和miRNA表达构成的组中选取表达数据。
本发明中的试剂盒可进一步包括合适的贮存容器,例如,安瓿,小瓶,试管等用于贮存各个检测剂和其它试剂,例如,在测试样品上检测使用的缓冲剂和平衡盐溶液等。检测试剂和其他试剂可以任何适宜的形式保存于试剂盒里,例如,溶液或粉末。试剂盒可进一步包括包装容器,可选择性分成一个或多个部分以容纳检测试剂和其他自选试剂。
本发明的另一个实施方案是用于预测受试者化疗方案功效的方法。该方法包括:
a.确定受试者的样品中表达了至少一种可对化疗方案功效进行预测的生物分子;
b.将以上所检测到的生物分子表达数据代入模型,计算出该化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定该化疗方案的化疗效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
在一些实施方案中,“化疗方案功效预测”在采用化疗方案前进行,可优化受试者的治疗效果。这类预测可帮助医疗专业人员确定对受试者选用特定的化疗方案。在一些实施方案中,“化疗方案功效预测”在采用化疗方案后进行,以评估化疗中特殊阶段的起始时间是否适宜,并/或决定是否基于变化例如受试者肿瘤遗传因子的改变,来调整化疗方案。
如本文和此后使用的,“受试者”和“化疗方案”的定义如上。
在一些实施方案中,步骤(a),包括,为确定受试者样品中有至少一个可预测化疗方案功效的生物分子的表达,可以使用任何数量的试验方法,包括杂交试验方法,基于扩增的试验方法,免疫试验方法,和免疫组化分析法。
杂交试验方法包括,但不限于,斑点印迹,核糖核酸酶保护,Northern杂交,芯片,荧光原位杂交(FISH)及它们的组合。基于扩增的试验方法包括,但不限于多聚酶链式反应,逆转录酶PCR以它们的组合。免疫实验方法包括,但不限于免疫细胞化学,免疫组织化学,酶联免疫吸附测定,记忆细胞免疫刺激试验(MELISA),克隆酶供体免疫分析法(CEDIA),和它们的组合。免疫组化试验方法包括,但不限于,直接荧光抗体法,间接荧光抗体法(IFA),抗补体免疫荧光法,亲和素-生物素免疫荧光染色法,荧光激活细胞分类法(FACS)。这些试验方法是本领域技术人员所熟知的。
如此处所用的,“样品”是指任何从受试者上获得的生物标本。本发明中的样品包括,但不限于,全血,血浆,血清,唾液,尿液,粪便,痰液,眼泪,任何其他体液,组织样本,如活体组织切片及细胞提取物。
在一些实施方案中,从上述各组的样品中检测至少一个的生物分子的表达,可能涉及到在特定组的样品里至少一个或多个生物分子的表达检测。至少一个生物分子可以指1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,23,24,25,26,27,28,29,30,31,32,33,34,35,36,37,38,39,40,41,42,43,44,45,46,47,48,49,50,51,52,53,54,55,56,57,58,59,60,61,62,63,64,65,66,67,68,69,70,71,72,73,74,75,或更多生物分子。甚至,检测那些未在本发明中公开的其他生物分子的表达,无论它们对本发明中公开的生物分子的表达是否有额外的预测价值,都包括在本发明的范围内。
在一些实施方案中,将上文中公布的生物分子表达“代入模型”的操作可在计算机或者本文公开的系统中进行。本发明中的统计模型,或此处称为“学习统计分类系统”包括,但不限于,随机森林模型,分类和回归树模型,Boosting模型,贝叶斯网络,马尔可夫随机场,线性和广义线性模型,Boosted树模型,神经网络,支持向量机,通用卡方自动交互检测模型,互动树模型,多适应性回归,机器学习分类,以及它们的组合。这些统计模型都是本领域技术人员所熟知的。
本发明的模型可以以,例如,如本文中所用的“模型性能的定量测量”来评价,如准确度,精确度,召回率(recall)和/或f1-值。本发明中的模型优先选用f1-值进行评估。f1-值在本文中定义为2×[(精确度×召回率)/(精确度+召回率)]其中,精确度和召回率是本领域技术人员的熟知的统计技术术语。在本发明的一些实施方案中,多个统计模型是基于若干个变量来开发的,包括临床变量,例如肿瘤大小,年龄,雌激素受体状态,孕激素受体状态,人类表皮生长因子受体状态,淋巴结状态,t_stage,n_stage及遗传变量,如本文公开的任 何生物分子的表达。优选情况下,本发明的统计模型将基于对各个模型评估后的f1-值,新变量并入模型来进行开发的。例如,一个模型只并入临床变量后将返回一定f1-值。另一个新模型结合临床变量和一个单一的基因变量将返回另一个f1-值,并可和上一个f1-值进行比较,较高的f1-值通常表明此模型具更强的预测能力。
在一些实施方案中,优选的统计模型是那些生成局部最大f1-值的模型。更优选的模型是生成第一个局部最大f1-值的统计模型。本文中,“局部最大”是指从第一个统计模型获得的数值,其特征是将代入第一个统计模型中的变量和其他附加变量代入其他统计模型,或者将代入第一个统计模型中的变量的子集代入其他统计模型得到的f1-值都比第一个统计模型更低。局部最大f1-值可是第一个统计模型计算得到的f1-值,其中该统计模型可以是包括除了代入第一个统计模型的变量外,还包括1,2,3,4,5,6,7,8,9,10个,或更多变量的统计模型;和包括1,2,3,4,5,6,7,8,9,10个变量,或者那些代入其他模型都获得比第一个统计模型更小的f1-值的变量组的变量。优化情况下,本发明中的局部最大f1-值可从第一个统计模型中获得,其中,统计模型包括那些除了代入第一个统计模型的变量外还有其它三个变量的模型,或那些比第一个统计模型变量的子集少3个变量且其f1-值比第一个统计变量少的统计模型。当变量代入给定的统计模型,将产生第一个局部最大f1-值。附加变量可提供获得第二个局部最大f1-值的模型,但本发明的优选方案将采用产生的第一个局部最大f1-值的统计模型。
在一些实施方案中,本发明中的统计模型与本发明中的化疗方案相关联。例如使用“蒽环类药物但无紫杉醇或多西紫杉醇药物”的化疗方案可与第一个统计模型相关联,而使用“蒽环类与紫杉醇药物但无多西紫杉醇”的化疗方案可与第二个统计模型关联。
在一些实施方案中,本发明中的统计模型可以计算化疗方案的病理完全缓解的预测概率。病理完全缓解以前就定义过。(Kaufmann,et al.,2006,Kuerer,et al.,1999,von Minckwitz,et al.,2012)。在本发明的上下文中,一个指定的统计模型应该能输出与该统计模型相关联的化疗方案的病理完全缓解的预测概率。同样地,不同的统计模型,可输出与其相关联的不同的化疗方案在同一患者上的病理完全缓解的不同预测概率。因此,患者可以有多个不同的病理完全缓解的预测概率,它们是由与多个化疗方案相关联的多个统计模型计算获得的。
在一些实施方案中,病理完全缓解的预测概率可分类成和化疗方案相关的一组概率区间。在本发明的上下文中,从患者组得到的表达数据可以代入一个统计模型,得到与此统 计模型相关的概率区间。例如,可从已接受第一期化疗方案治疗的患者组上收集表达数据,将其代入统计模型后,可以得到每个患者的病理完全缓解的预测概率。这些病理完全缓解的预测概率可分成任意个概率区间,其中每个概率区间里包含有数目相同的预测概率。(例如.每个概率区间可包含1,2,3,4,5,6,7,8,9,或10或更多个预测概率)。概率区间可定义为在某上限和下限之间,包括上限和下限,的范围。例如,区间[0,1]可以是0到1之间的任何值,包括0和1。优化的分组方式是将预测概率分为5个区间。
在已知一组患者的概率区间时,本实施方案采用的方法是把受试者病理完全缓解的预测概率分类成上述的某一种概率区间。
在一些实施方案中,接受多个化疗方案的受试者可有多个与之相关的病理完全缓解预测概率,可将这些预测概率分类成多个概率区间组,从某一组中选择的一个概率区间与一个特定的化疗方案相关。
在一些实施方案中,要对一个给定的化疗方案的效果进行定量测量。在一些实施方案中,这个化疗方案效果的定量测量是病理完全缓解值(pCR score)。病理完全缓解值与病理完全缓解预测概率不同。当患者的病理反应预测概率被分类在某个特定的概率区间时,病理完全缓解值才定义为该患者对此特定化疗方案的预测概率。在本实施方案的上下文中,在受试者的病理完全缓解预测概率被划分入某个概率区间之前或之后,病理完全缓解值可与概率区间相关。如本文所用,病理完全缓解值是在某个概率区间内有病理完全缓解的患者与此概率区间所有患者的比值。这个比值在此被称为阳性预测值(PPV)。因此当受试者对某个特定化疗的病理完全缓解预测概率被分类到某个概率区间的情况下,和以上提到的概率区间相关的受试者的病理完全缓解值即是该受试者对此给定化疗方案的病理完全缓解值。
在一些实施方案中,接受多个化疗方案的受试者可有多个与之相关的病理完全缓解预测概率,这些预测概率可分类成多个概率区间,每个概率区间可从与特定的化疗方案相关的一组概率区间中选出。同样的,每个概率区间和某个病理完全缓解值相关联,因此受试者与其病理完全缓解预测概率分类后所在区间内的病理完全缓解值相关联。
在一些实施方案中,可就某一化疗方案对受试者的疗效进行预测。根据对化疗结果的定量测试,例如病理完全缓解值,受试者或者医疗专业人员可以预测在受试者上使用与以上所述病理完全缓解值相关的的化疗方案后受试者的病理完全缓解。受试者如果接受多个化疗方案,就有多个病理完全缓解值与其接受的化疗方案相关联,其中与最大病理完全缓解值相关联的化疗方案就是预测为该受试者接受的所有化疗方案中最有效的一个方案。
本发明的另一个实施方案是用于预测化疗方案在受试者上疗效的方法。该方法包括:
a.测定受试者样品中有以下包括SLC12A7,GZMB,TAF6L,NFIB,METRN,ROPN1B,TTK,CCND1,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,C11orf17,CCL5,XCL1,XCL2,MELK,CTSL2,TPX2,AURKA,CDKN2C,BRP44,PNP,SMC4,NR4A2,C3orf37,MTPAP,CDC25B,ABCF1,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,TSPYL5,SRI,LRP12,CENPF,TUBD1,KIAA1324,DBF4,CCNA2,DLGAP5,FHL1,SIRT3,GTSE1,PCNA,CCNE2,CHD3,CAP1,GPM6B,GUSBP3,GNAI3,LMO4,PSRC1,USP1,STK38,,BAT2L1,PMP22,NME5,CENPA,BANK1,及其衍生物在内的至少两个生物分子的表达;
本发明的另一个实施方案是用于预测化疗方案在受试者上疗效的方法,其中所述的化疗方案包括使用蒽环类药物,但不使用紫杉醇类和多西紫杉醇药物的化疗方案。此方法包括:
a.测定受试者样品中有以下包括SLC12A7,GZMB,TAF6L,及其衍生物在内的至少一种生物分子的表达;
b.将生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量可预测化疗方案对受试者的功效。
本发明的另一个实施方案是用于预测化疗方案在受试者上疗效的方法,其中所述的化疗方案包括使用蒽环类和紫杉醇类药物,但不包括使用多西紫杉醇药物的化疗方案。此方法包括:
a.测定受试者样品中表达有以下组中包括NFIB,METRN,ROPN1B,TTK,CCND1,及其衍生物中至少一种生物分子的表达;
b.将生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量可预测化疗方案对受试者的功效。
本发明的另一个实施方案是用于预测化疗方案在受试者上疗效的方法,其中所述的化疗方案包括使用蒽环类和多西紫杉醇类药物,但不包括使用紫杉醇药物的化疗方案。此方法包括:
a.测定受试者样品中表达有以下组中包括PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,及其衍生物中至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
c.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量可预测化疗方案在受试者上的功效。
如本文所用的,“使用蒽环类药物但不涉及紫杉醇或多西紫杉醇药物”的化疗方案包括一个或多个蒽环类药物,与其他任何非紫杉醇及其类似物,及非多西紫杉醇及其类似物的药物的组合。同样,“使用蒽环类和紫杉醇药物,但无西紫杉醇药物”的化疗方案包括一个或多个蒽环类药物,与任何紫杉醇及其类似物药物的组合,但其中不涉及多西紫杉醇及其类似物的药物。与此类似,“使用蒽环类和多西紫杉醇药物,但无西紫杉醇药物”的化疗方案包括一个或多个蒽环类药物,与任何多西紫杉醇及其类似物药物的组合,但其中不涉及紫杉醇及其类似物的药物。
本发明中另一个实施方案是用来预测化疗方案在HER2-阴性受试者上疗效的方法,其中所述的化疗方案使用蒽环类药物,但不使用无紫杉醇和多西紫杉醇药物这个方法包括:
a.测试受试者样品中有以下包括SLC12A7,GZMB,C11orf17,TAF6L,CCL5,XCL1,XCL2,及其衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
c.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量可预测化疗方案在受试者上的功效。
本发明中的另一个实施方案是用于预测化疗方案在HER2-阴性受试者上疗效的方法,其中所述的化疗方案包括使用蒽环类和紫杉醇类药物,但不使用多西紫杉醇药物。该方法包 括:
a.测试受试者样品中有以下包括NFIB,ROPN1B,TTK,MELK,CTSL2,METRN,及其衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
本发明中的另一个实施方案是用于预测化疗方案在HER2-阴性受试者上疗效的方法,其中所述的化疗方案包括使用蒽环类和多西紫杉醇类药物,但不使用紫杉醇药物。该方法包括:
a.测试受试者样品中有以下包括TPX2,PTTG1,MCM2,MCM6,AURKA,CDKN2C,BRP44,H2AFZ,PNP,SMC4,DEK,TMEM97,NR4A2,C3orf37,LZTFL1,MTPAP,CDC25B,ABCF1,及其衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
人体表皮生长因子受体2(HER2)基因表达膜络氨酸激酶,并在特定的肿瘤组织里高表达,使这些组织有特定生物功能,包括对特定化疗的敏感性。癌细胞中HER2的表达水平可以用,例如免疫组化(IHC)和荧光原位杂交(FISH)方法,进行检测。癌症可根据FISH检测到的HER2水平值进行分类。在癌细胞中检测不到HER2染色,其表达水平为0,癌症被分类为HER2阴性。在癌细胞中检测到微弱或者不完整的膜染色,其表达水平为1+,癌症被分类为HER2-阴性。当有至少10%细胞检测到HER2强度不均匀或弱的完整膜染色,或者在30%或更少细胞检测到强烈的HER2膜染色,其表达水平为2+,该癌症被分类为HER2不确定病例(HER2-equivocal)。HER2-阳性癌症表达水平为3+,能在30%侵袭性癌细胞检测到强烈均匀的HER2染色。在本发明的上下文中,用于确定受试者是否为HER2阴性/阳性,ER阴性/阳性,或淋巴结阴性/阳性的标准是本领域共同认可的。此外,用于确定受试者HER2/ER/淋巴结状态的某些标准可能会随着分析技术的提高而随时改变。此处列出的标准仅供说明,并非意在以任何方式限制。
本发明中另外一个实施方案是用于预测化疗方案在HER2-阴性,ER-阴性受试者上疗效的方法,其中所述的化疗方案使用蒽环类和紫杉醇类药物,但不使用多西紫杉醇药物。此方法包括:
a.测试受试者样品中有以下包括NFIB,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,及其衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量可预测化疗方案对受试者的功效。
本发明的另一个实施方案是用于预测化疗方案在HER2-阴性,ER-阴性受试者上效果的方法,其中所述的化疗方案使用蒽环类和多西紫杉醇药物,但无紫杉醇药物。该方法包括:
a.测试受试者样品中有以下包括TSPYL5,SRI,及其衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
本发明的另一个实施方案是用于预测化疗方案在HER2-阴性,ER-阳性受试者上疗效 的方法,其中所述的化疗方案使用蒽环类和紫杉醇药物,但无多西紫杉醇药物。该方法包括:
a.测试受试者样品中有以下包括LRP12,CENPF,TUBD1,KIAA1324,TTK,及其衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
本发明中的另一个实施方案是用于预测化疗方案在HER-阴性,ER-阳性受试者上疗效的方法,其中所述的化疗方案使用蒽环类和多西紫杉醇药物,但无紫杉醇药物。该方法包括:
a.测试受试者样品中有以下包括DBF4,DEK,CDC25B,CCNA2,DLGAP5,MCM2,CDKN2C,FHL1,SIRT3,GTSE1,PCNA,CCNE2,及其衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
本发明中的另一个实施方案是用于预测化疗方案在HER2-阴性,淋巴结阴性受试者上疗效的方法,其中所述的化疗方案使用蒽环类和紫杉醇药物,但无多西紫杉醇药物。该方法包括:
a.测试受试者样品中有以下包括CHD3,CAP1,GPM6B,GUSBP3和其衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
如本文所用,雌激素受体(ER)状态也可以作为受试者对某些化疗反应的表征。类似于HER2,根据癌组织中检测到的ER水平,受试者可分类成ER-阳性或ER-阴性。
本发明中的另一个实施方案是用于预测化疗方案在HER2-阴性,淋巴结阴性受试者上疗效的方法,其中所述的化疗方案使用蒽环类和多西紫杉醇药物,但无紫杉醇药物。该方法包括:
a.测试受试者测试受试者样品中有以下包括CDKN2C,GNAI3,LMO4,PSRC1,USP1,STK38和衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
本发明中的另一个实施方案是用于预测化疗方案在HER2-阴性,淋巴结阳性受试者上疗效的方法,其中所述的化疗方案使用蒽环类和紫杉醇药物,但无多西紫杉醇药物。该方法包括:
a.测试受试者测试受试者样品中有以下包括NFIB,ROPN1B和衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案的化疗效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
本发明中的另一个实施方案是用于预测化疗方案在HER2-阴性,淋巴结阳性受试者上疗效的方法,其中所述的化疗方案使用蒽环类和多西紫杉醇药物,但无紫杉醇药物。该方 法包括:
a.测试受试者测试受试者样品中有以下包括TPX2,BAT2L1,PMP22,PTTG1,NME5,CENPA,BANK1和衍生物在内的至少一个生物分子的表达;
b.将上述检测到的生物分子表达代入模型中,计算此化疗方案的病理完全缓解的预测概率;
c.按病理完全缓解(PCR)的预测概率分成一组与化疗方案相关的概率区间(PIS);
d.确定化疗方案效果的定量测量,其中化疗效果的定量测量对受试者化疗方案的功效具预测性。
癌症,包括乳腺癌,可根据受试者淋巴结中是否检测到癌细胞而分类为淋巴结阳性和淋巴结阴性。淋巴结状态可通过淋巴结活检来检测,而是否存在癌细胞可由,例如病理医生,来进行评估。
在一些实施方案中,在使用本发明中的方法或者检测试剂盒前,可先检测受试者的HER2/ER/淋巴结状态,从而使医护人员知晓本发明中哪个方法或检测试剂盒对受试者最为最有效。
在一些实施方案中,表达可以是mRNA表达,蛋白表达,非编码RNA表达和miRNA表达。
本发明中的另一个实施方案是为受试者选择化疗方案的方法。此方法包括:
a.测试受试者样品中有以下包括SLC12A7,GZMB,TAF6L,NFIB,METRN,ROPN1B,TTK,CCND1,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,C11orf17,CCL5,XCL1,XCL2,MELK,CTSL2,TPX2,AURKA,CDKN2C,BRP44,PNP,SMC4,NR4A2,C3orf37,MTPAP,CDC25B,ABCF1,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,TSPYL5,SRI,LRP12,CENPF,TUBD1,KIAA1324,DBF4,CCNA2,DLGAP5,FHL1,SIRT3,GTSE1,PCNA,CCNE2,CHD3,CAP1,GPM6B,GUSBP3,GNAI3,LMO4,PSRC1,USP1,STK38,,BAT2L1,PMP22,NME5,CENPA,BANK1,及其衍生物在内的至少两个生物分子的表达;
本发明中另一个实施方案是用于为受试者选择化疗方案的方法,此方法包括::
a.确定受试者的样品中表达有用于预测第一个化疗方案效果的第一组生物分子;
b.将上述检测到的第一组生物分子的表达代入第一个模型中,计算第一个 化疗方案的第一个病理完全缓解的的预测概率;
c.按第一个病理完全缓解(PCR)的预测概率分成第一组与第一个化疗方案相关的概率区间(PIS),并且
d.确定第一个化疗方案的化疗效果的定量测量,其中化疗效果的第一次定量测量对受试者接受的第一个化疗方案的功效具预测性。
在此实施方案另一方面,此方法还包括:
a.确定受试者样品中有用于预测第二个化疗方案效果的第二组生物分子的表达;
b.将上述检测到的第二组生物分子的表达代入第二个模型中,计算第二个化疗方案的第二个病理完全缓解的的预测概率;
c.按第二个病理完全缓解(PCR)的预测概率分成第二组与第二个化疗方案相关的概率区间(PIS),并且
d.确定第二个化疗方案的化疗效果的定量测量,其中化疗效果的第二次定量测量对受试者接受的第二个化疗方案的功效具预测性。
在另一实施方案,此方法还包括:
a.确定受试者样品中有用于预测第三个化疗方案效果的第三组生物分子的表达;
b.将上述检测到的第三组生物分子的表达代入第三个模型中,计算出第三个化疗方案的第三个病理完全缓解的的预测概率;
c.按第三个病理完全缓解(PCR)的预测概率分成第三组与第三个化疗方案相关的概率区间(PIS),并且
d.确定第三个化疗方案的化疗效果的定量测量,其中化疗效果的第三次定量测量对受试者接受的第三个化疗方案的功效具预测性。
在本实施方案的另一方面,第一组生物分子(多个分子)是以下1-14组中的至少一组生物分子:
a.组1:SLC12A7,GZMB,TAF6L,及其衍生物;
b.组2:NFIB,METRN,ROPN1B,TTK,CCND1,及其衍生物;
c.组3:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,及其衍生物;
d.组4:SLC12A7,GZMB,C11orf17,TAF6L,CCL5,XCL1,XCL2,及其衍生物;
e.组5:NFIB,ROPN1B,TTK,MELK,CTSL2,METRN,及其衍生物;
f.组6:TPX2,PTTG1,MCM2,MCM6,AURKA,CDKN2C,BRP44,H2AFZ,PNP,SMC4,DEK,TMEM97,NR4A2,C3orf37,LZTFL1,MTPAP,CDC25B,ABCF1,及其衍生物;
g.组7:NFIB,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,及其衍生物;
h.组8:TSPYL5,SRI,及其衍生物;
i.组9:LRP12,CENPF,TUBD1,KIAA1324,TTK,及其衍生物;
j.组10:DBF4,DEK,CDC25B,CCNA2,DLGAP5,MCM2,CDKN2C,FHL1,SIRT3,GTSE1,PCNA,CCNE2,及其衍生物;
k.组11:CHD3,CAP1,GPM6B,GUSBP3,及其衍生物;
l.组12:CDKN2C,GNAI3,LMO4,PSRC1,USP1,STK38,及其衍生物;
m.组13:NFIB,ROPN1B,及其衍生物;
n.组14:TPX2,BAT2L1,PMP22,PTTG1,NME5,CENPA,BANK1,及其衍生物.
在一实施方案,所述的第二组生物分子(多个分子)是以下1-14组中的至少一组生物分子:
a.组1:SLC12A7,GZMB,TAF6L,及其衍生物;
b.组2:NFIB,METRN,ROPN1B,TTK,CCND1,及其衍生物;
c.组3:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,及其衍生物;
d.组4:SLC12A7,GZMB,C11orf17,TAF6L,CCL5,XCL1,XCL2,及其衍生物;
e.组5:NFIB,ROPN1B,TTK,MELK,CTSL2,METRN,及其衍生物;
f.组6:TPX2,PTTG1,MCM2,MCM6,AURKA,CDKN2C,BRP44,H2AFZ,PNP,SMC4,DEK,TMEM97,NR4A2,C3orf37,LZTFL1,MTPAP,CDC25B,ABCF1,及其衍生物;
g.组7:NFIB,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,及其衍生物;
h.组8:TSPYL5,SRI,及其衍生物;
i.组9:LRP12,CENPF,TUBD1,KIAA1324,TTK,及其衍生物;
j.组10:DBF4,DEK,CDC25B,CCNA2,DLGAP5,MCM2,CDKN2C,FHL1, SIRT3,GTSE1,PCNA,CCNE2,及其衍生物;
k.组11:CHD3,CAP1,GPM6B,GUSBP3,及其衍生物;
l.组12:CDKN2C,GNAI3,LMO4,PSRC1,USP1,STK38,及其衍生物;
m.组13:NFIB,ROPN1B,及其衍生物;
n.组14:TPX2,BAT2L1,PMP22,PTTG1,NME5,CENPA,BANK1,及其衍生物.
在另一实施方案,所述的第二组生物分子(多个分子)是以下1-6组中的至少一组生物分子:
a.组1:SLC12A7,GZMB,TAF6L,及其衍生物;
b.组2:NFIB,METRN,ROPN1B,TTK,CCND1,及其衍生物;
c.组3:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,及其衍生物;
d.组4:SLC12A7,GZMB,C11orf17,TAF6L,CCL5,XCL1,XCL2,及其衍生物;
e.组5:NFIB,ROPN1B,TTK,MELK,CTSL2,METRN,及其衍生物;
f.组6:TPX2,PTTG1,MCM2,MCM6,AURKA,CDKN2C,BRP44,H2AFZ,PNP,SMC4,DEK,TMEM97,NR4A2,C3orf37,LZTFL1,MTPAP,CDC25B,ABCF1,及其衍生物.
在一些实施方案中,表达数据可是mRNA表达,蛋白表达,非编码RNA表达,miRNA表达。
在一些实施方案中,用来为受试者选择化疗方案的方法可能会涉及到使用来自一组生物分子中的生物分子。从一组生物分子中选出的生物分子可用做,例如,化疗方案效果的定量测量。而化疗效果的定量测量可帮助医疗人员确定对患者有效的某一个化疗方案。在一些实施方案中,用于受试者选择化疗方案的方法可能会涉及使用从两组或更多组分子(包括三组)中选出的一些生物分子。从多组生物分子选择的生物分子对,例如多个化疗方案效果的定量测量,非常有用,从而可对这些化疗方案进行比较,从而预测哪个化疗方案对患者最有效。
本发明中的另一个实施方案是用来选择预测化疗效果的生物标记物的方法。此方法包括:
a.用随机样品筛选(RSS)流程对一组生物分子进行筛选,确定至少一个基因预测因子(genetic predictor);
b.生成一个模型,此模型应整合入至少一个变量来代表(多个)基因预测因子,模型输出为模型性能的定量测度;
c.确定该模型性能的定量测度;
d.重复步骤(b)-(c)至少一次,生成(多个)附加模型和相应的(多个)模型性能的定量测度;
e.从一组生物分子中挑出生物标记物,以生成的模型中的一个变量来代表被选的生物标记物,代入以上获得的模型中有局部最优定量测度的模型。
在本实施方案的另一个方面,此方法还包括用以确定在使用上述化疗方案后表现出病理完全缓解的第一组受试者和使用同样化疗方案后表现出残留病灶的第二组受试者之间存在表达差异的一组生物分子的初始步骤。
如本文所用,“生物标记物”是任何诊断标记物,例如生化标记物,血清标记物,基因标记物,或其他可用于预测化疗方案效果的临床特征。本发明中的生物标记物包括本发明中的生物分子及其衍生物,例如,但不限于,基因和基因片段,mRNA转录及其蛋白产物。本发明中的生物标记物也包括DNA修饰,如表观遗传修饰和对给定基因和基因片段的拷贝数的修改,转录后修饰的RNA和翻译后修饰的蛋白。
在一些实施方案中,“选择可以预测化疗方案疗效的生物标记物”包括从存在于受试者细胞或分泌的生物分子中选择生物分子,但不意味着限制以其它任何方式获得。
在一些实施方案中,“筛选”指用特定的标准从一组生物分子中选择一个或多个生物分子。
如本文所用,术语“表达差异”和其语法变异指在与其它细胞相关的特定细胞里特定的mRNA和/或蛋白的表达水平的变化。表达差异包括(多个)生物分子的上调和下调。在一些实施方案中,“差异表达”生物分子包括在具病理完全缓解(pCR)的受试者和具残留病灶(RD)受试者上显示出统计性差异表达水平的生物分子。统计显著性可通过使用任何测量统计意义的方法来获得,包括本领域内技术人员熟识的方法。这里,统计显著性优先使用T-test测定,更优则用Welch双样本t-检验来测定。
T-tests得到的P-值可检验统计显著性。P-值小于或者等于0.05表示具统计显著性,但一个合适的P-值可由本领域技术人员轻易获得。最优情况下,本发明中有表达差异的生物分子指P-值为0.05或者更小,包括P-值为0.01或者更小,0.001或更小,甚至0.001或更小。
如本文所用,残留病灶(RD)之前已经定义过。(Kaufmann,et al.,2006,Kuerer,et al.,1999,von Minckwitz,et al.,2012)。
在一些实施方案中,用随机抽样筛选(RSS)程序来筛选一组差异表达的生物分子,确定至少一个基因预测因子。随机抽样筛选的例子,如本文中使用的,包括以下步骤:
a.从探针组S0中随机抽取探针样品C0
b.用C0计算随机森林曲线下面积(area under the curve random forest(AUCRF)或者用另一种机器学习方法由C0得到一组探针R0(x),其中x=1,除非在接下来的步骤里对其另有定义;
c.将R0(x)里的探针加入探针组Sy,其中y=1,除非接下来的步骤对其另有定义;
d.重复步骤(a)-(c),其中在步骤(b)的每次迭代中,X增加1;而且
e.重复步骤(a)–(d),其中用Sy代替S0,在步骤(b)的第一次迭代中,重新设定X为1,并且在步骤(c)的每次迭代中,Y增加1。
如上文中用到的,“探针”等同于上文中描述的“生物分子”。此外S0被定义为上文中讨论的一组有表达差异性的生物分子。
在一些实施方案中,探针样品C0仅受探针组S0的探针数目限制。探针样品C0最好有S0组的最少探针数目的1/4,或者500个。(the sample of probes C0contains the minimum of the number of probes in S0/4,or 500.)
在一些实施方案中,随机样品筛选(RSS)过程中的步骤(d)可以重复数十次。步骤(d)重复上百次更好,最好是重复一千次。
在一些实施方案中,随机样品筛选(RSS)过程中的步骤(e)一直重复,直到Sy里的探针数目与Sy-1里的探针数目相同,或者少于50。由此生成探针组Sy里的探针,“基因预测子”,可以任意选来输入AUCRF,对基因预测子的重要性进行排序。如此处所用的,“基因预测子”是在对一组具表达差异性的生物分子进行随机样品筛选后留下的生物分子,最理想的是基因。
在一些实施方案中,“建立至少一个包括至少一个代表基因预测子的变量的模型,且此模型能输出对其性能的定量测量”的操作将按以上描述来执行。简而言之,可以通过,例如将临床变量代入随机森林模型对f1-score进行初步评估,来建立能代入至少一个代表至少一个基因预测子(例如,一个基因)的变量的统计模型。第一个模型可输出此模型性能的 定量测量,并用以评估。然后,更多的变量,包括至少一个至少代表一个基因预测子的变量,可以代入模型,以建立更多模型,并输出更多的模型性能的定量测量数据。最优情况下,如果用f1-score作模型性能的定量测量,f1-score大的模型比f1-score小的模型更好。
在一些实施方案中,“至少一个变量”代表至少一个基因预测子的变量,例如,一个将基因预测子表达作为输入值的变量。当前发明中的其他变量包括临床变量,例如受试者的年龄和体重。
如此处所用,“确定模型性能的定量测量”是此领域中众所周知的技术,通常涉及到计算某模型质量的数量形式,例如,某给定模型的准确度,精密度,召回,或者它们的组合。
在当前用来选择可预测化疗方案效果的生物标记分子的实施方案中,步骤(b)-(c)可重复至少一次以获得额外的(多个)模型以及与此(多个)模型相对应的性能定量测量数据。可设想,步骤(b)-(c)可重复任何次以获得任意额外的(多个)模型以及与此(多个)模型相对应的性能定量测量数据。优化情况下,当一个模型的性能定量测量获得局部最大值时,可不再重复步骤。更优化的情况下,上述模型性能定量测量的局部最大值是第一个该模型性能定量测量的局部最大值。
在一些实施方案中,“从一组生物分子里选择生物标记分子,被选中的生物标记分子可用一个或多个变量代入以上建成的具有模型性能定量测量局部最大值的模型来表示”意味着,一个能得到模型性能定量测量局部最大值的模型,以代入该模型的任何变量来表示的任何生物标记分子都可被选中。此外,一个具备模型性能定量测量局部最大值的模型如果包括多个变量来表示多个生物分子,那么从以上生物分子中可选出多个生物标记。如本文所用,术语“最优”与模型性能的定量测量相关,根据所用的模型性能的定量测量方法,它可是最大值或最小值。例如,当使用f1-值,大数值比小数值更优。同样的,当f1-值获得最大值时,就是一个模型性能的最优定量测量。
本发明中的一个附加的实施方案是一个用来为受试者选择化疗方案的系统。该系统包括:
a.一个可从受试者的某个样品里生成一个数据集的数据采集模块,这个数据集包括诊断标记物谱,其中所述诊断标记物谱可指出至少一个可预测以下化疗方案疗效的生物分子的表达;其中化疗方案包括(i)使用蒽环类药物,但无紫杉醇或多西紫杉醇的化疗,(ii)使用蒽环类和紫杉醇类药物,但无多西紫杉醇药物的化疗,和(iii)使用蒽环类和多西紫杉醇药 物,但无紫杉醇药物的化疗;
b.一个应用学习统计分类系统处理数据的数据处理模块,可对化疗方案在受试者上的效果进行预测,生成统计学预测数据。和,
c.一个显示统计学预测数据的数据显示模块。
在一些实施方案中,从受试者的样品里生成一个数据集的数据采集模块可是一个计算机系统,例如,一个计算机系统可设定用来专门收集用本发明中检测生物分子表达的某个测量方法获得的数据。又如,一个计算机系统可设置成专门收集那些特别针对本发明中生物分子的检测试剂而获得的基因芯片数据。数据采集模块可进一步转化或以其他方式将上述收集到的数据进行排序,输出一个包括诊断标记物谱的数据集。可以预见的是,本发明中的数据采集模块可以设置为从已建立的数据库中收集数据,并不需要从一个特定的测量方法中获得数据。本发明中的一个诊断标记物谱包括,但不局限于,可预测本发明中的化疗方案在本发明中的样品上的效果的至少一个生物分子的表达数据。在一些实施方案中,诊断标记物谱包括用本发明中的一个测量方法收集到的数据子集。
在一些实施方案中,数据处理模块可设置为一个使用学习统计分类系统处理数据,可对一个化疗方案在受试者上的效果进行预测,生成统计学预测数据的计算机系统。这个计算机系统可以是和数据采集模块同一个的计算机系统,或是分开的计算机系统。
在一些实施方案中,学习统计分类系统是个模型,最优情况是以上提到的统计模型。在本文中,一个统计模型包括各种用于确定独立变量(预测变量)和响应变量之间关系的数学算法。本发明中,变量可以是临床变量,例如受试者年龄或体重,或基因变量,例如受试者样品中可预测化疗方案效果的生物分子表达。本发明中的统计模型不局限于任何特定数目的变量。本发明的统计模型可以包括一个或多个变量。
在一些实施方案中,学习统计分类系统包括机器学习技术,它能调整数据集和基于此数据集的决策过程。一些实施方案使用单个学习统计分类系统。另一些实施方案则使用有多个学习统计分类系统的组合。此处描述的学习统计分类系统可以使用样品或者从不同受试者,例如,健康受试者,化疗后表现病理完全缓解的受试者,化疗后仍有病灶的受试者,收集来的数据来对系统进行训练和测试。本发明中对学习统计分类系统的训练和测试是本此领域中众所周知的技术。
在一些实施方案中,对化疗方案效果的统计学预测可以是对化疗效果的定量测量,例如病理完全缓解值。
在一些实施方案中,数据显示模块可以是一个可显示视觉信息的屏幕。本发明中中首选显示模块包括,但不限于,计算机显示器,电视机,平板显示器和智能手机显示器。
在一些实施方案中,表达数据可选自mRNA表达数据,蛋白表达数据,非编码RNA表达数据和miRNA表达数据。
本发明中的代表性系统可用图3中的流程图来说明。
在一个实施方案中,提供了治疗患有乳腺癌的受试者的方法。方法包括:(i)要求检测受试者样品中是否表达有以下至少一个基因:SLC12A7,GZMB,和TAF6L,从而计算化疗方案的预测概率和病理完全缓解;和(ii)根据化疗结果的定量测量结果对受试者采用使用蒽环类药物的化疗。其中化疗效果的定量测量来自于上述的病理完全缓解预测概率。
在一个实施方案中,提供了治疗患乳腺癌的受试者的方法。方法包括:(i)要求检测受试者样品中是否表达以下至少一个基因:NFIB,METRN,ROPN1B,TTK,和CCND1,从而计算化疗方案的预测概率和病理完全缓解;和(ii)根据化疗结果的定量测量结果对受试者采用使用蒽环类和紫杉醇药物的化疗。其中化疗效果的定量测量来自上述的病理完全缓解预测概率。
在一个实施方案中,提供了治疗患有乳腺癌的受试者的方法。方法包括:(i)要求检测受试者样品中是否表达有以下至少一个基因:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,和LZTFL1,从而计算化疗方案的预测概率和病理完全缓解;和(ii)根据化疗结果的定量测量结果对受试者采用使用蒽环类和多西紫杉醇药物的化疗。其中化疗效果的定量测量来自上述的病理完全缓解预测概率。
在一个实施方案中,提供了治疗患有乳腺癌的受试者的方法。方法包括:(a)要求检测受试者样品中是否(i)表达以下至少一个基因:SLC12A7,GZMB,和TAF6L,从而计算化疗方案的第一预测概率和病理完全缓解,以及(ii)表达以下至少一个基因:NFIB,METRN,ROPN1B,TTK,和CCND1,从而计算该化疗方案的第二预测概率和病理完全缓解;和(b)根据化疗结果的定量测量结果对受试者采用使用蒽环类药物,或者蒽环类和多西紫杉醇药物的化疗。其中化疗效果的定量测量来自上述的病理完全缓解预测概率。
在一个实施方案中,提供了治疗患有乳腺癌的受试者的方法。方法包括:(a)要求检测受试者样品中是否有(i)表达以下至少一个基因:SLC12A7,GZMB,和TAF6L,从而计算化疗方案的第一预测概率和病理完全缓解,以及(ii)表达以下至少一个基因:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,和LZTFL1,从而计算该化疗方案的第二预测概率和病理完全缓解;和(b)根据化疗结果的定量测量结果对受试者采用 使用蒽环类药物,或者蒽环类和多西紫杉醇药物的化疗。其中化疗效果的定量测量来自于上述的病理完全缓解预测概率。
在一个实施方案中,提供了治疗患有乳腺癌的受试者的方法。方法包括:(a)要求检测受试者样品中是否(i)表达有以下至少一个基因:NFIB,METRN,ROPN1B,TTK,和CCND1,从而计算化疗方案的第一预测概率和病理完全缓解,以及(ii)表达有以下至少一个基因:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,和LZTFL1,从而计算该化疗方案的第二预测概率和病理完全缓解;和(b)根据化疗结果的定量测量结果对受试者采用使用蒽环类和紫杉醇药物,或者蒽环类和多西紫杉醇药物的化疗。其中化疗效果的定量测量即是上述化疗方案的病理完全缓解预测概率。
在一个实施方案中,提供了治疗患有乳腺癌的受试者的方法。方法包括:(a)要求检测受试者样品中是否(i)表达有以下至少一个基因:SLC12A7,GZMB,和TAF6L,从而计算化疗方案的第一预测概率和病理完全缓解,以及(ii)表达有以下至少一个基因:NFIB,METRN,ROPN1B,TTK,和CCND1,从而计算该化疗方案的第二预测概率和病理完全缓解;以及(iii)表达有以下至少一个基因:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,和LZTFL1,从而计算该化疗方案的第三预测概率和病理完全缓解和(b)根据上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用化疗方案,其中包括使用蒽环类药物,或蒽环类和紫杉醇药物,或者蒽环类和多西紫杉醇药物的化疗。
在一个实施方案中,提供了治疗HER2-阴性乳腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否表达有以下至少一个基因:SLC12A7,GZMB,C11orf17,TAF6L,CCL5,XCL1,和XCL2,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性乳腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否有至少以下一个基因的表达:NFIB,ROPN1B,TTK,MELK,CTSL2,和METRN,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和紫杉醇药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性乳腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否有至少以下一个基因的表达:TPX2,PTTG1,MCM2,MCM6,AURKA, CDKN2C,BRP44,H2AFZ,PNP,SMC4,DEK,TMEM97,NR4A2,C3orf37,LZTFL1,MTPAP,CDC25B,和ABCF1,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和多西紫杉醇药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性,和ER-阴性乳腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否有表达以下至少一个基因:NFIB,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,和STK24,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和紫杉醇药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性,和ER-阴性乳腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否有以下至少一个基因的表达:TSPYL5和SRI,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和多西紫杉醇药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性,和ER-阳性乳腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否有以下至少一个基因的表达:LRP12,CENPF,TUBD1,KIAA1324,和TTK,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和紫杉醇药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性,和ER-阳性乳腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否有以下至少一个基因的表达:DBF4,DEK,CDC25B,CCNA2,DLGAP5,MCM2,CDKN2C,FHL1,SIRT3,GTSE1,PCNA,和CCNE2,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和多西紫杉醇药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性,和淋巴结阴性乳腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否有以下至少一个基因的表达:CHD3,CAP1,GPM6B,和GUSBP3,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和紫杉醇药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性,和淋巴结阴性腺癌受试者的方法。方法 包括:(i)要求检测受试者样品中是否有以下至少一个基因的表达:CDKN2C,GNAI3,LMO4,PSRC1,USP1,和STK38,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和多西紫杉醇药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性,和淋巴结阳性腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否有以下至少一个基因的表达:NFIB和ROPN1B,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)基于对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和紫杉醇药物的化疗方案。
在一个实施方案中,提供了治疗HER2-阴性,和淋巴结阳性腺癌受试者的方法。方法包括:(i)要求检测受试者样品中是否有以下至少一个基因的表达:TPX2,BAT2L1,PMP22,PTTG1,NME5,CENPA,和BANK1,从而计算该化疗方案的预测概率和病理完全缓解,以及(ii)根据对上述的化疗效果的定量测量,即化疗方案的病理完全缓解预测概率,对受试者采用使用蒽环类和多西紫杉醇药物的化疗方案。
在一个实施方案中,以上方法中的基因表达可以是mRNA表达,蛋白表达,非编码RNA表达,或者是imRNA表达。
在一个实施方案中,基因表达可以通过DNA阵列芯片,RNA阵列芯片,寡核苷酸阵列芯片,蛋白阵列芯片,一个或多个抗体,一个或多个抗原结合片段,多个探针,或一组引物来检测。
在一个实施方案中,提供了一种为患乳腺癌的受试者选择化疗方案的试剂盒。这个试剂盒包括了一个或多个成分用来检测以下至少一个基因表达。这些基因包括:SLC12A7,GZMB,TAF6L,NFIB,METRN,ROPN1B,TTK,CCND1,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,LZTFL1,C11orf17,CCL5,XCL1,XCL2,MELK,CTSL2,TPX2,AURKA,CDKN2C,BRP44,PNP,SMC4,NR4A2,C3orf37,MTPAP,CDC25B,ABCF1,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,STK24,TSPYL5,SRI,LRP12,CENPF,TUBD1,KIAA1324,DBF4,CCNA2,DLGAP5,FHL1,SIRT3,GTSE1,PCNA,CCNE2,CHD3,CAP1,GPM6B,GUSBP3,GNAI3,LMO4,PSRC1,USP1,STK38,BAT2L1,PMP22,NME5,CENPA,和BANK1.
在一个实施方案中,以上试剂盒可检测的基因包括:SLC12A7,GZMB,和TAF6L.
在一个实施方案中,以上试剂盒可检测的基因包括:NFIB,METRN,ROPN1B,TTK,和CCND1.
在一个实施方案中,以上试剂盒可检测的基因包括:PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,and LZTFL1.
在一个实施方案中,以上试剂盒可检测的基因包括:SLC12A7,GZMB,TAF6L,NFIB,METRN,ROPN1B,TTK,和CCND1.
在一个实施方案中,以上试剂盒可检测的基因包括:SLC12A7,GZMB,TAF6L,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,和LZTFL1.
在一个实施方案中,以上试剂盒可检测的基因包括:NFIB,METRN,ROPN1B,TTK,CCND1,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,和LZTFL1.
在一个实施方案中,以上试剂盒可检测的基因包括:SLC12A7,GZMB,TAF6L,NFIB,METRN,ROPN1B,TTK,CCND1,PTTG1,H2AFZ,WDR45L,DEK,MCM2,USP1,CDT1,TMEM97,RER1,MCM6,和LZTFL1.
在一个实施方案中,以上试剂盒可检测的基因包括:SLC12A7,GZMB,C11orf17,TAF6L,CCL5,XCL1,和XCL2.
在一个实施方案中,以上试剂盒可检测的基因包括:NFIB,ROPN1B,TTK,MELK,CTSL2,和METRN.
在一个实施方案中,以上试剂盒可检测的基因包括:TPX2,PTTG1,MCM2,MCM6,AURKA,CDKN2C,BRP44,H2AFZ,PNP,SMC4,DEK,TMEM97,NR4A2,C3orf37,LZTFL1,MTPAP,CDC25B,和ABCF1.
在一个实施方案中,以上试剂盒可检测的基因包括:NFIB,MTAP,SNAPC3,RANBP9,COIL,FAM86B1,ITGA6,S100P,RANBP1,PRSS16,SMARCA2,和STK24.
在一个实施方案中,以上试剂盒可检测的基因包括:TSPYL5和SRI.
在一个实施方案中,以上试剂盒可检测的基因包括:LRP12,CENPF,TUBD1,KIAA1324,和TTK.
在一个实施方案中,以上试剂盒可检测的基因包括:DBF4,DEK,CDC25B,CCNA2,DLGAP5,MCM2,CDKN2C,FHL1,SIRT3,GTSE1,PCNA,和CCNE2.
在一个实施方案中,以上试剂盒可检测的基因包括:CHD3,CAP1,GPM6B,和GUSBP3.
在一个实施方案中,以上试剂盒可检测的基因包括:CDKN2C,GNAI3,LMO4,PSRC1, USP1,和STK38.
在一个实施方案中,以上试剂盒可检测的基因包括:NFIB和ROPN1B.
在一个实施方案中,以上试剂盒可检测的基因包括:TPX2,BAT2L1,PMP22,PTTG1,NME5,CENPA,和BANK1.
在一个实施方案中,以上试剂盒中的一个或多个成分可以是在一个实施方案中,以上试剂盒中的一个或多个成分可以是DNA阵列芯片,RNA阵列芯片,寡核苷酸阵列芯片,蛋白阵列芯片,一个或多个抗体,一个或多个抗原结合片段,多个探针,或一组引物。
附加定义
在本发明一些实施方案中,受试者患有癌症。本发明中的癌症包括,但不限于肾上腺皮质癌,肛门肿瘤/癌,膀胱肿瘤/癌,骨肿瘤/癌症(如骨肉瘤),脑肿瘤,乳腺肿瘤/癌症,类癌,癌,宫颈癌肿瘤/癌,结肠癌肿瘤/癌,子宫内膜肿瘤/癌食管肿瘤/癌,肝外胆管肿瘤/癌,尤因家族肿瘤,颅外生殖细胞肿瘤,眼肿瘤/癌症,胆囊肿瘤/癌症,胃肿瘤/癌,生殖细胞肿瘤,妊娠滋养细胞肿瘤,头颈部肿瘤/癌,下咽癌肿瘤/癌,胰岛细胞癌,肾肿瘤/癌,喉癌肿瘤/癌,白血病,唇和口腔肿瘤/癌,肝癌肿瘤/癌,肺癌肿瘤/癌,淋巴瘤,恶性间皮瘤,Merkel细胞癌,蕈样肉芽肿,骨髓增生异常综合征,骨髓增生性疾病,鼻咽肿瘤/癌症,神经母细胞瘤,口腔肿瘤/癌,口咽肿瘤/癌,骨肉瘤,卵巢上皮性肿瘤/癌,卵巢生殖细胞肿瘤,胰腺肿瘤/癌,鼻窦和鼻腔肿瘤/癌,甲状旁腺肿瘤/癌,阴茎肿瘤/癌,垂体瘤/癌,浆细胞瘤,前列腺肿瘤/癌,横纹肌肉瘤,直肠癌肿瘤/癌,肾细胞瘤/癌,移行细胞瘤/肾盂癌和输尿管,唾液腺肿瘤/癌症,塞扎里综合征,皮肤肿瘤(如皮肤T细胞淋巴瘤,卡波西氏肉瘤,肥大细胞瘤和黑素瘤),小肠肿瘤/癌,软组织肉瘤,胃肿瘤/癌,睾丸肿瘤/癌,胸腺瘤,甲状腺肿瘤/癌,尿道肿瘤/癌,子宫肿瘤/癌,阴道肿瘤/癌,外阴肿瘤/癌,和肾母细胞瘤。具有乳腺癌受试者属于优选实施方案。
在本发明的一些实施方案中,检测试剂包括核酸。“核酸”或者这里使用的“寡核苷酸”或“多核苷酸”包括至少两个由共价键链接的核苷酸。一个核酸可有多个变异体,因此,一个核酸可包括与它相同的核酸以及它的互补序列。
核酸可以是单链或者双链,或同时具有单链双链部分。核酸可以是DNA,基因DNA或者互补DNA,RNA,或者杂交核酸,即该核酸可包含脱氧核糖-和核糖-核苷酸的组合,以及包括尿嘧啶,腺嘌呤,胸腺嘧啶,胞嘧啶,鸟嘌呤,肌苷,黄嘌呤次黄嘌呤,异胞嘧啶和异鸟嘌呤等碱基组合。核酸可以被合成为一个单链分子,或使用合成基因在细胞(体外或 体内)表达。核酸可以通过化学合成方法或者重组方法获得。
核酸可以是RNA,例如mRNA,tRNA,短发夹RNA(shRNA),短干扰RNA(siRNA),双链RNA(dsRNA),转录后基因沉默RNA(ptgsRNA),Piwi相互作用RNA(Piwi-interacting RNA),初级miRNA(pri-miRNA),前miRNA(pre-miRNA),微小miRNA(micro-(miRNA),or反-miRNA(anti-miRNA),正如在美国专利申请号为11/429,720,11/384,049,11/418,870,和11/429,720的专利申请书和公开的国际专利号为WO 2005/116250及WO 2006/126040的申请里所描述的。
短干扰RNA基因靶向(siRNA gene-targeting)可以通过瞬态转移siRNA进入细胞,并通过如脂质介导的转染(如脂质体的封装,与阳离子脂质,胆固醇,和/或缩合聚合物配位,电穿孔,或显微注射)等经典方法来实现。短干扰RNA基因靶向也可用结合抗体的siRNA,或者结合有融合蛋白的siRNA来实现;其中融合蛋白包含了细胞穿透肽,穿透肽连接了可结合siRNA的双链RNA-结合域(DRBD)(可参看美国专利申请号为2009/0093026的专利申请)。
一个短发夹RNA(shRNA)分子有两个序列反向互补区,可以彼此配对形成分子内双链。短发夹RNA基因靶向(shRNA gene-targeting)可以使用载体转移siRNA进入细胞,比如病毒载体(例如慢病毒载体,腺病毒载体,或腺相关病毒载体)。siRNA和shRNA的设计和合成是业界熟知的技术,可以从,例如Gene Link(Hawthorne,NY),Invitrogen Corp.(Carlsbad,CA),Thermo Fisher Scientific,和Dharmacon Products(Lafayette,CO)等商业公司购买。.
核酸可以是核酸适体,intramer,或者是spiegelmer。术语“核酸适体”指的是可以结合特定分子靶标的核酸或者寡核苷酸分子。核酸适体衍生于体外进化过程(例如,SELEX(Systematic Evolution of Ligands by EXponential Enrichment,指数富集的配基系统进化技术),公开在美国专利5,270,163中),此过程可从巨大的组合核酸库里选出靶标特异性的核酸适体。核酸适体可是双链或单链,可包括脱氧核糖核苷酸,核糖核苷酸,核苷酸衍生物或其他核苷酸类似分子。适体的核苷酸组分可是已修饰的糖基(例如在核糖苷酸的2’-羟基可用2’-氟基团或者2’-胺基取代),这样可改进所期望的属性,例如,抗核酸酶的抗性或血液中更长有效期。核酸适体可与其它分子偶联,例如,与高分子载体偶联以减慢适体在循环系统里的降解速度。核酸配体可以与它们的特异性同源配体交联,例如,通过光激活交联剂(Brody,E.N.and L.Gold(2000)J.Biotechnol.74:5-13)交联。
术语“intramer”指在体内表达的一类核酸适体。例如,基于牛痘病毒的RNA表达系统可用来在白细胞胞质里高水平表达特异性的RNA核酸适体。(Blind,M.et al.(1999)Proc.Natl.Acad.Sci.USA 96:3606-3610).
术语“spiegelmer”指包括左旋-DNA,左旋-RNA或其他左旋核苷酸衍生物或核苷酸衍生物的核酸适体。含有左旋核苷酸的适体对天然存在的降解酶有抗性,因为这类降解酶通常以右旋核苷酸作为底物。
核酸一般包括磷酸二酯键,尽管在核酸类似物内可有至少一个不同的连接键,例如在氨基磷酸酯,硫代磷酸酯,二硫代磷酸酯,或O-甲基氨基磷酸酯里的连接键和肽核酸主链和连接键。其他核酸包括正主链(positive backbone),非离子主链,包括在美国专利5,235,033和5,034,506公布的非核糖主链等。核酸还包括那些带有一个或多个非天然或修饰的核苷酸的核酸。例如在核酸5’-末端和/或3’-末端能找到修饰的核苷酸类似物。带有核糖修饰或主链修饰的核酸是核苷酸类似物的代表例子。然而必须要注意的是,碱基修饰的核苷酸,例如在5’-位带有非天然碱基的核苷酸,例如5-(2-氨基)丙基尿苷,5-溴尿苷和腺苷,和在8-位有修饰的鸟苷酸,例如8-溴鸟苷,脱氮核苷酸,例如7-脱氮腺苷,以及O-和N-烷基化的核苷酸,例如甲基腺苷可归入此类。2’-羟基团可以被下列任何基团代替:H,OR,R,卤素基团,SH,SR,NH2,NHR,NR2或CN,其中R是C1-C6烷基,链烯基或炔基,卤素基团是F,Cl,Br或I。修饰的核苷酸包括与胆固醇共轭的核酸,例如在文献Krutzfeldt et al.,Nature(Oct.30,2005),文献Soutschek et al.,Nature 432:173-178(2004)里,和美国专利申请20050107325中所公开的由羟脯氨酸连接的共轭。修饰过的核苷酸和核酸还包括在美国专利申请20020115080中所公开的锁核酸(locked核酸s)。其他修饰的核苷酸和核酸可在美国专利申请20050182005中查询。核糖-磷酸主链可由于不同的原因进行修饰,例如,增加这类分子在生理环境下的稳定性和半衰期,增强此类分子的跨膜扩散,或者是用作生物芯片上的探针。核酸混合物可由混合天然核酸以及类似物获得,或混合不同核酸类似物,或者混合不同天然核酸获得。
在一些实施方案中,检测试剂是蛋白质。术语“肽”,“多肽”和“蛋白质”在本文中可互换使用。在本发明中,该术语指链接的氨基酸序列。氨基酸序列可以是天然的,合成的,或修饰的,或是天然和合成的组合。该术语包括抗体,抗体模拟物,域抗体,脂质运载蛋白,靶向蛋白酶,和多肽模拟物。该术语还包括含有肽或肽片段的疫苗。目的是产生对抗这些肽或肽片段的抗体。
在一些实施方案中,检测试剂是抗体。如本文中所用,“抗体”和“抗原-结合片段”涵盖天然存在的免疫球蛋白(例如,IgM,IgG,IgD,IgA,IgD),和非天然存在的免疫球蛋白,包括,例如,单链抗体,嵌合抗体(如,人源化鼠抗),异源抗体(如,双特异性抗体),和Fab’,F(ab')2,抗原结合片段(Fab),可变区(Fv),和重组免疫球蛋白IgG(rIgG)。另见,皮尔斯公司产品目录和手册,1994-1995(Pierce Chemical Co.,Rockford,IL);Kuby,et al.,1998。如本文所用,“抗原结合片段”指全长抗体上保存有识别抗原的部分,以及包括此部分的各种组合。
非天然的抗体可通过固相多肽合成来构建,也可通过重组生产,或者通过,例如,筛选由可变重链和可变轻链组成的组合库来获得,其中组合库在Huse等人的文章中(Huse et al.,Science 246:1275-1281(1989))有描述,并通过引用结合到本文中。这些和其他,例如获得嵌合抗体,人源化抗体,CDR-移植抗体,单链抗体和双功能抗体的方法,是本领域技术人员熟知的技术(Winter and Harris,Immunol.Today 14:243-246(1993);Ward et al.,Nature 341:544-546(1989);Harlow and Lane,supra,1988;Hilyard et al.,Protein Engineering:A practical approach(IRL Press 1992);Borrabeck,Antibody Engineering,2d ed.(Oxford University Press 1995);以上的各个文献通过引用结合到本文中。
全长抗体可被蛋白酶水解,被消化成若干个离散,保留抗原识别能力的功能性抗体片段。例如,木瓜蛋白酶可把全长的免疫球蛋白裂解成两个抗原结合片段(Fab)和一个结晶片段(Fc)。因此,抗原结合片段通常包括两个可变结构域和来自重链和轻链的两个恒定结构域。可变区片段(Fv)通常被认为是抗原结合区的一个组成部分,通常包括两个可变区,一个来自重链(如本文所用VH,“重链可变区”,)和轻链(如本文所用的VL,“轻链可变区”)。胃蛋白酶裂解抗体铰链区下端,因此产生一个F(ab')2片段和一个pFc'片段。F(ab')2片段是去除恒定区(constant region)被消化的完整抗体。两个Fab’片段则来自被酶进一步消化的F(ab')2片段。抗原结合片段的例子包括,但不限于,可变区(Fv),抗原结合区段(Fab),Fab’,Fab’-SH,F(ab’)2,双抗体,三抗体,单链可变区片段(scFvs),和单域抗体。.
通常,一个全长的抗体至少有一个重链和一个轻链。每个重链包含一个可变结构区(VH),通常还有三个或更多的恒定区(CH1,CH2,CH3,等),而每个轻链有一个可变区(VL)和一个恒定区(CL)。轻链和重链的可变区包含四个被三个高变区(hypervariable regions)亦称为“互补决定区”或是“CDRs”打断的“框架区”。框架区和互补决定区的范围已经被Kabat定义(见文献Kabat et al.,U.S.Dept.of Health and人Services, Sequences of Proteins of Immunological Interest(1983)and Chothia et al.,J.Mol.Biol.196:901-917(1987))同一物种内的不同轻链或重链的框架区序列相对保守。抗体的框架区,即轻链和重链的组合框架区,能在三维空间中定位和对齐互补决定区。
互补决定区主要结合抗原的抗原决定簇。每条多肽链上的互补决定区通常称为互补决定区1,互补决定区2,互补决定区3,从氨基-末端开始依次编号,通常还通过其中特定的互补决定区所在链进行定位。因此,重链可变区的互补决定区3(VH CDR3)位于抗体重链的可变区,一个轻链可变区的互补决定区1在抗体轻链的可变区。
术语“单克隆抗体”,如本文所用,指的是从相同的抗体群中获得的一个抗体,即构成抗体群的各个抗体相同,除了极少数可能的有自然突变的抗体。单克隆抗体高度特异,能特异识别单个抗原决定簇。修饰语“单克隆”表示该抗体来自于相同的抗体群的特性,而不是用以诠释此抗体生产所需的任何特定方法。例如,本发明所使用的单克隆抗体可通过最早由Kohler(Kohler et al.,Nature 256:495(1975))等人描述的杂交瘤方法制备,并用体细胞杂交法对上述方法进行改进;或者通过其他重组DNA方法来获得。(例如,参见美国专利4,816,567).
还有其他抗体可以归入本发明中的单克隆抗体的一部分,它们包括但不限于,嵌合型抗体,人源化抗体,和人抗体。对于用在人上的抗体,通常期望能减少源自其他物种的抗体免疫原性,例如小鼠。这个可通过构建嵌合抗体,或者通过“人性化”的过程,来实现。在这种情况下,“嵌合抗体”可以理解为一个融合了来自一个物种(例如,小鼠)的一个结构域(例如,可变区)和另一不同物种(例如,人类)的一个结构域(例如,恒定区)的抗体。
如本文所用,术语“人源化抗体”指含有非人源(例如,鼠源)序列的抗体以及人抗体。这类抗体是含有来自非人源免疫球蛋白的最小序列的嵌合抗体。通常人源化抗体基本包含全部可变区中的至少一个,通常两个可变区,其中所有或基本上所有的高度可变环(hypervariable loops)与那些非人源免疫球蛋白相对应;其中所有或基本上所有的框架区(FR)是人源免疫球蛋白序列。人源化抗体可选择性地含有免疫球蛋白恒定区的至少一部分(Jones et al.,Nature 321:522-525(1986);Riechmann et al.,Nature 332:323-329(1988);and Presta,Curr.Op.Struct.Biol 2:593-596(1992))。抗体人源化的方法基本上可遵循Winter和他同事等人用的方法(Jones et al.,Nature 321:522-525(1986);Riechmann et al.,Nature 332:323-3'27(1988);Verhoeyen et al.,Science 239:1534-1536(1988),用人类抗体的相应序列取代鼠源抗体的互补决定区序列。
此外,基于人类基因组的序列的抗体构建技术已经开发,例如,噬菌体展示或使用转基因动物(WO 90/05144;D.Marks,H.R.Hoogenboom,T.P.Bonnert,J.McCafferty,A.D.Griffiths and G.Winter(1991)"By-passing immunisation.人antibodies from V-gene libraries displayed on phage."J.Mol.Biol.,222,581-597;Knappik et al.,J.Mol.Biol.296:57-86,2000;Carmen and L.Jermutus,"Concepts in antibody phage display".Briefings in Functional Genomics and Proteomics 2002 1(2):189-203;Lonberg N,Huszar D."人antibodies from transgenic mice".Int Rev Immunol.1995;13(1):65-93.;Bruggemann M,Taussig M J."Production of人antibody repertoires in transgenic mice".Curr Opin Biotechnol.1997August;8(4):455-8.)。在本发明中,这类抗体属于“人源抗体”。
如本文中所用的,“重组”抗体指的是任何在生产过程中涉及在生物体内表达编码所需抗体的非天然DNA序列的一类抗体。本发明中,重组抗体包括:串联scFv(taFv or scFv2),双抗体(diabody),dAb2/VHH2,knob-into-holes衍生物,SEED-IgG,heteroFc-scFv,Fab-scFv,scFv-Jun/Fos,Fab’-Jun/Fos,tribody,DNL-F(ab)3,scFv3-CH1/CL,Fab-scFv2,IgG-scFab,IgG-scFv,scFv-IgG,scFv2-Fc,F(ab')2-scFv2,scDB-Fc,scDb-CH3,Db-Fc,scFv2-H/L,DVD-Ig,tandAb,scFv-dhlx-scFv,dAb2-IgG,dAb-IgG,dAb-Fc-dAb,和它们的组合。
抗体可变区通常分离成单链可变区(scFv)或抗原结合片段。单链可变区片段是由重链可变区(VH)和轻链可变区(VL)组成,两者之间由短的10-15氨基酸链接。一旦分开,单链可变区片段(scFv)可通过柔性肽段链接,例如,一个或多个重复的Ala-Ala-Ala,Gly-Gly-Gly-Gly-Ser肽段。所产生的肽段,即串联的单链可变区片段(taFv or scFv2)可以以不同方式排列,可以是重链可变区-轻链可变区(VH-VL)或是轻链可变区-重链可变区(VL-VH)顺序。(Kontermann,R.E.In:Bispecific Antibodies.Kontermann RE(ed.),Springer Heidelberg Dordrecht London New York,pp.1-28(2011)).
如本文中所用,术语“抗原决定簇”指抗原中被抗体或抗原结合片段识别的部分。单个抗原(例如抗原性多肽)可含有一个以上的抗原决定簇。抗原决定簇可以从结构或功能上来定义。功能性抗原决定簇是结构性抗原决定簇的一个子集,含有直接参与相互作用的氨基酸残基。抗原决定簇也有构象,即,它可由非线性氨基酸构成。.在一些特定实施方案中,抗原决定簇可包含具化学活性表面基团的分子,例如,氨基酸,糖侧链,磷酰基,或黄磺酰基,并且在另一些特定的实施方案中,抗原决定簇可能有特殊的三维结构特征,和/或特定的电荷特征。由连续氨基酸序列形成的抗原决定簇在暴露于变性溶剂中后仍能保留其构象,而由蛋白三级折叠形成的抗原决定簇在由变性溶剂处理后完全丧失其构象。
以下实例进一步说明本发明中的方法。这些例子是说明性的,并不旨在限制本发明的范围。.
附图说明
图1为个人化化疗方案选择过程的流程图。
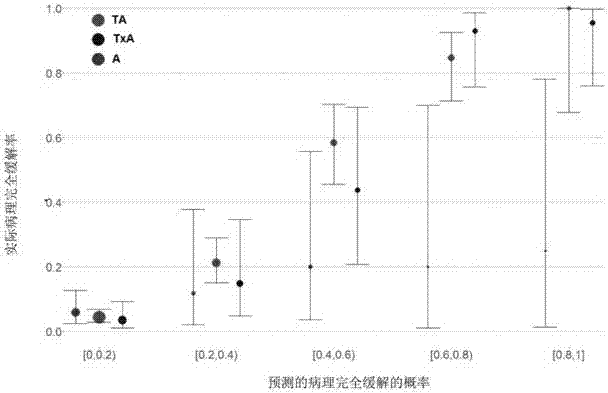
图2显示预测的病理完全缓解率和实际的病理完全缓解率的关系。
图3所示为本发明中为受试者选择化疗方案的系统的流程图。
具体实施方式
实施例1
材料和方法
从GEO数据库(基因表达数据库Gene Expression Omnibus(Takada,et al.,2012,Albain,et al.,2010))中的7个数据序列中收集了1111个乳腺癌样品(表格1)。样品分成三个治疗组:A组(蒽环类药物),TA组(紫杉醇和蒽环类药物),和TxA组(多西紫杉醇和蒽环类药物),用R(Liu,et al.,2012)package Affyio(Edgar,et al.,2002)来读取和规范化 数据。所有样品的反应均以病理完全缓解(pCR)或残留病灶(RD)来编码。
表格1
GEO数据组以及每组中的病人数目
*括号里的数值为具病理完全缓解的患者在其相应治疗组中的百分比。
模型建立和评估
此处所用方法的流程图如图1所示。首先,用Welch双样本t-检验(Welch two-sample t-test)找到在病理完全缓解组和病灶残留组中存在表达差异的探针。设置显著水平为0.05,将具表达显著差异的那组探针选出来,将这组候选探针组设为S0。
用随机抽样筛选(RSS)程序将候选探针组S0进一步缩小,随机抽样筛选的工作原理如下:
1.随机从探针组S0中抽取样品min{(S0/4),500},将抽取的探针组命名为C0;
2.代入探针组C0中的探针,执行AUCRF(Calle,et al.,2011),并记录下由AUCRF选出的探针组,R0(1);
3.重复步骤(1)和(2)一千次,记录所有在R0(1),...,R0(1000)出现的探针,将其设为S1;
4.用S1代替S0,重复步骤(1),(2),和(3);在步骤(3)中,不记录所有在AUCRF出现的探针,而是记录那些出现率(探针出现次数与总共取样次数(1000)的比值)大于50%的探针;
5.重复步骤(4)直到迭代n值满足Sn和Sn-1相同,或者Sn小于50。
Sn是由随机抽样筛选法找出的最后一组候选探针,接下来,代入Sn执行AUCRF,在Sn中的探针将根据它们的重要性进行排序。
鉴于数据组是不平衡的(有残留病灶的病人比有病理完全缓解的病人多),所以f1-score连同正精确率(positive precision)和正召回率(positive recall),而不是准确率(accuracy),被用来测量模型功能。F1-score定义为2×精确率(precision)×召回率(recall)/(正确率+召回率)(precision+recall)。从十折交叉验证计算获得f1-score,将独立使用每个训练数值组进行上述的筛选过程操作,从而获得候选探针组:Sn1,...,Sn10。
为某个模型选择重要的探针并评估模型,可将探针一次一个(从最高排名)地代入仅有临床变量的模型(这是个随机森林模型,仅有临床变量:年龄,ER-状态,HER2-状态,t_stage,和n_stage),并记录过程中的f1-score。此模型的最优探针数目即是得到第一个f1-score局部最大值的探针数目值(此处,局部定义为+/-3探针数)。
除了仅有临床变量的模型(临床模型)和有临床基因变量模型(临床-基因模型)外,使用从临床-基因模型中选出的基因也可在每个治疗组中建立仅有基因变量的模型(基因模型)。由此可显示两种类型变量的相对重要性。
方案选择
一旦三种治疗方案的模型建好,每个患者即可通过模型计算得到他实际接受的治疗方案的病理完全缓解的预测概率。为了避免过度拟合,可用十折交叉验证法计算预测概率,这意味着患者的预测值将通过一个不带患者信息的模型计算获得。将从每个模型计算得到的病理完全缓解的预测概率排序,并分成5个概率区间(probability intervals),这样每个概率区间中的患者人数大致相同(为平衡每个区间覆盖的概率可以对其有微小调整)。每个区间的精度(或阳性预测值)可用在此区间观察到的病理完全缓解患者比例来计算获得。这个比例称为病理完全缓解值,是一个患者使用某个特定治疗方案后其预测概率落在某个特定概率区间的概率。每个治疗方案对应5个概率区间有5个病理完全缓解值。一个手动建立的概率区间将用来和按以上描述方法建立的概率区间做对比。
下一步,在每个模型中对所有患者的病理完全缓解的概率进行预测。再次,对那些用来建模的患者数据,通过十折交叉验证法计算出他们的预测概率。每个患者在由A组,TA组和TxA组化疗方案上建成的三个模型上得到三个相应的病理完全缓解的预测概率。然后每个概率可用来定位每个模型的概率区间。当一个治疗方案的最大病理完全缓解值在该方案的概率区间内,那这个治疗方案为此患者的最优治疗方案。考虑到治疗方案的毒性,如果A组患者的病理完全缓解值和其他两组治疗方案的病理完全缓解值相差在+/-0.02之间,那么这个患者可以使用A组治疗方案。另外,治疗方案的分配也可通过获得最大病理完全缓解值来实现,以获得比预期稍高的病理完全缓解值的比例。在给患者分配治疗方案后,计算所有患者的病理完全缓解值的总和,即是预计出现病理完全缓解的患者数目。
人群分层
合并后数据集中的患者群相当不均匀。可用几种方法对患者群进行分层后来研究分层如何影响研究结果。研究的患者群中有10%的患者为HER2-阳性。目前,针对HER2-阳性患者的有效靶向治疗使用曲妥珠单抗(trastuzumab)。在目前临床条件下,大部分HER2-阳性患者都将接受靶向治疗。值得一提的是,许多HER2-阳性患者除使用曲妥珠单抗(trastuzumab)外,仍接受化疗以增加靶向治疗的疗效。
对HER2-阴性患者的研究采用与整个患者群体相同的研究方案,并得到非常类似的结果。但没有足够的HER2-阳性患者进行此类研究。HER2-阴性患者按其淋巴结或ER状态进一步分层,可分成两层:(1)淋巴结-阳性和淋巴结-阴性,和(2)ER-阳性和ER-阴性。这种分层的所有患者都是HER2-阴性。患者分层中的一个问题是那些被分出去在较小群体的患者,因为群体小将限制有质量的预测模型建立。因此,HER2-阴性患者,无论是接受TA组或者TxA组治疗,都将进一步分层。而接受蒽环类药物治疗的患者(A组)将不包括在这些研究中。
比较紫杉醇(T)和多西紫杉醇(Tx)
一些临床试验已经表明了紫杉烷类药物加入基于蒽环类药物的治疗方案的益处(Gajria,et al.,2010)。紫杉醇和多西紫杉醇都属于抗癌化合物紫杉烷类的家族,它们都有相同的主要结构和作用机制。但是两者在包括解聚抑制活性,毒性等几个方面不同(Verweij,et al.,1994)。对于初次接受蒽环类药物治疗的患者,当单独给药时,紫杉醇和多西紫杉醇功效类似(Chan,et al.,1999,Sledge,et al.,2003)。一些临床试验也显示紫杉醇和多西紫杉醇在改进无病生存率(disease-free survival)和总生存期(overall survival)时功效相似(De Laurentiis,et al.,2008,Sparano,et al.,2008)。在参与这项研究并表现病理完全缓解的患者中,接受多西紫杉醇的患者(33.1%)比接受紫杉醇的患者(19.7%)多。当然,这并不能作为一个说明多西紫杉醇比紫杉醇具有更高疗效的有力证据。依然存在的一个关键问题是:患者对两种药物是否反应相似?是否存在一类患者亚群,他们更适合用其中一种药物,而非另外一种药物。由于紫杉醇和多西紫杉醇是常用的治疗乳腺癌的药物。这是一个具有重要临床意义的问题。在此,可用淋巴结或ER状态对HER-阴性患者的亚群进行分层,如前段所述,并将分层后的患者亚群来进行紫杉醇和多西紫杉醇之间的比较研究。
实施例2
整个患者群的结果
模型性能和基因标签(gene signatures)
对三个不同治疗方案的三个模型的功能在表格2中列出。带有临床和基因变量的模型(clinical-gene models)通常比其他仅有临床变量的模型(clinical models)和仅有基因变量的模型(gene models)性能更好。加入基因变量的模型在处理治疗方案TA和TxA数据时,其性能显著改进,而在处理治疗方案A的数据时,三个模型在性能上并无大异。基因模型和临床-基因模型处理治疗方案TA和TxA数据的性能比临床模型好很多,显示基因变量可能是化疗反应的一个强大预测因子。基于这些比较,在接下来的研究中将使用临床-基因模型。与每个治疗方案中治疗反应相关的特征基因组(gene signatures)在表格3中列出。在治疗方案A,TA和TxA中的特征基因组分别有3,5,和11个基因。我们也比较了预测的病理完全缓解和实际的病理完全缓解的关系。从图2可以看出他们有很好的正相关。
表格2
三种模型的性能:仅有临床变量的临床模型,有临床和基因变量的临床-基因模型,和仅有基因变量的基因模型*
*:A:蒽环类药物,TA:蒽环类和紫杉醇药物,TxA:蒽环类和多西紫杉醇药物粗体数值是每个治疗方案中的最大值
表格3
从三个治疗方案中选出的基因
*pCR status:“+”:基因在病理完全缓解病例中高水平表达;“-”:基因在病理完全缓解病例中低水平表达.
个性化治疗方案选择
如在材料和方法部分中所述,每个模型的预测概率首先排序,然后分成5个相等的区间。由于每个化疗方案中的病理完全缓解率比残留病灶率低,被区间覆盖的概率呈现偏斜趋势。(The probabilities covered by the intervals were skewed due to the fact that pCR rate of each regimen was lower than RD rate.)该分区允许患者在每个区间内均匀分布,从而使大部分区间的预测病理完全缓解率(pCR score)更为可靠。当病理完全缓解的预测值很低时,此模型表现相当好。在三个治疗组中的第一区间的阴性预测值(NPV)为97%或更高。在处理TA和TxA组数据时,在数据值的另一端,即病理完全缓解的预测概率高时,模型也表现很好。在这种情况下,阳性预测值(positive predictive values(PPVs))很高,TA组为0.667而TxA组为0.878。
按照材料和方法部分所描述的方法将最优化疗方案分配给病人。使用预测模型和个性化方案选择方法(personalized regimen selection approach(PERS))得到的病理完全缓解值为435.8(表格4,第一排),与最初选择的方案观察到的病理完全缓解值(236)提高了84%。
表格4中列出来自不同分层的患者上的结果。在HER2-阴性患者中观察到的结果与在所有患者上观察到的结果类似,这个是由于90%总患者中有90%是HER2-阴性。其特征基因组(gene signatures)包括许多类似基因。总体而言,与病人分配的最初化疗方案相比,分层后患者的预计病理完全缓解有显著改善。
可从个性化的治疗方案选择中获益的病人比例
我们也检查了那些可能受益于目前可用的个性化治疗方案的患者。对每个患者,先计算出任何两个化疗方案的病理完全缓解值之间的最大绝对差(MADPS)。对应于三个模型(化疗方案),每个病人有三个病理完全缓解值,即会有三对病理完全缓解值的差异值。这三个差异值的最大绝对值就是这个患者的病理完全缓解之的最大绝对差(MADPS)。MADPS的幅度即表明了个性化治疗方案对此患者的重要程度。该值接近于0则表明此患者对所有的化疗方案反应类似。该值越大,则表明此患者对至少两个化疗方案反应非常不同。总体来说,相当数量的患者可以从个性化方案选择中获益。
结论和讨论
在本研究中,从接受过几种新辅助化疗方案的乳腺癌患者上收集的高通量基因表达数据被用来研究个性化方案选择能否使现已接受化疗的乳腺癌患者受益。将患者分成3组方案组:只接受蒽环类药物化疗的患者(A组),接受蒽环类和紫杉醇类药物化疗的患者(TA组),和接受蒽环类和多西紫杉醇类药物的患者(TxA组)。有相当数量的患者对至少两个方案有不同反应,表明个性化方案对于那些选择这些选项其中一项的患者是非常有益的。另外,变量选择方法可以选择少数基因,这些基因能有效区分出在某些化疗方案中表现更高病理完全缓解的患者。个性化治疗方案选择(A personalized regimen selection strategy,PERS)的策略根据数据组中的患者设计并应用到患者上。病理完全缓解率可从21.2%提高到39.3%,增幅84%。接受TA和TxA化疗方案的患者,有17.28%的患者有可能过度治疗,这意味如果接受A化疗方案,这些患者至少会有相同的病理完全缓解概率。有9.63%接受A化疗方案的患者治疗不足,这意味着如果他们接受TA或TxA化疗方案,他们有更高的病理完全缓解概率。考虑到治疗不足可能是由于费用负担原因,当前化疗方案选择的策略倾向于用在过度治疗的患者上。
在没有化疗方案能提高病理完全缓解概率时,本发明除了可帮助患者选择最佳化疗方案外,病人也可选择不采取这些化疗方案或不参加替代治疗,例如临床试验。个性化治疗方案选择(PERS)把111个患者分到化疗方案A的第二和第三个概率区间;把37个患者分到化疗方案TA的第二和第三个概率区间;把94个患者分到化疗方案TxA的前两个概率区间。在所有概率区间里的负预测值(NPVs)均大于90%。这表明有21.8%患者的病理完全缓解值被预测偏低,而他们实际的病理完全缓解值也很低。尽管是否采用其中的某个化疗方案 是患者的个人决定,但提供这些信息可以指导患者做决策。另一方面,有175名(15.8%)正预测值为0.878的患者被分在化疗方案TxA的第五个概率区间。另有149名(14.3%)正预测值为0.667患者被分在化疗方案TA的第五个概率区间。总而言之,这些预测可帮助一半以上的患者做决策(并且对其余患者的决策也非常有用,因为他们可以知道病理完全缓解的概率。)
将本研究中发现的显著基因(表格3)与之前研究中的显著基因(Hatzis,et al.,2011)相比,发现两套基因组之间并无重叠。Hatzis的数据集里包括470个患者,是本研究人群中的一个子集。鉴于两组数据集之间存在显著重叠,当结果显示两组研究之间并未发现一个共同基因时,确实令人感到意外。我们进一步地考察本研究中使用基于不同患者分层建成的模型鉴定出的基因。出乎我们意外,除了占总患者90%的HER2-阴性患者亚群,大部分模型都生成不同的基因组,且只有小部分的相同基因。因此,我们就不同模型获得的完全病理反应的预测概率的一致性进行研究。
有人可能会注意到在整个研究人群中,大部分患者被分配采用化疗方案TxA。这是有道理的,因为化疗方案TxA的病理完全缓解率是33%,而化疗方案A和TA的病理完全缓解率分别为8.6%和19.7%。用于TxA化疗方案的模型具较高的精度,对TxA的最终疗效结果是有帮助的。本研究中,化疗方案A的模型性能比其它两个模型都差。这可能是由于化疗方案A组中患者总人数和病理完全缓解病例比其它两组低得多造成的。
即使不考虑其中所有HER2-阳性患者的数据,此数据集的患者数据差异非常大。将不同患者数据合并,可帮助找到它们共同的生物标记物。进一步将数据分层,可研究将不同亚群分开研究时是否获得更一致的结果。然而,由于某些数据层的患者数目一定,此研究中并未对数据做彻底比较。对接受TA或者TxA化疗方案的HER2-阴性患者,根据他们的淋巴结或ER状态进行分层,生成了类似的结果。
手动方法能使被覆盖的概率在每个区间分布更平衡,同时使每个区间里有足够的患者数目以保证获得可靠的病理完全缓解预测值,所以可用来研究区间划分如何影响预期的病理完全缓解。
表格5
每组化疗方案中的患者数(重划分的区间)
*初始方案数据是观察数据。(The original group is observed.)
本研究中用到十折交叉验证法。许多以前的研究用不同的训练和测试数据集。原则上,与不同的训练和测试数据集相比,十折交叉验证法不太可能过度拟合,表明目前使用模型比之前所用模型有更高的普遍性。
本研究中使用的方法可以方便地用于开发其他癌症的个人化化疗方案选择,这也将是未来研究的主题。
实施例3
来自HER2-阴性亚群的结果
用于HER2-阴性亚群的三个化疗方案的三种模型的性能列在表格6中。我们看到用新的方案病理完全缓解率可以有很大的提高。
表格6
每个化疗方案中的患者数
*初始方案数据是观察数据。The original group is observed.
实施例4
ER-阳性患者亚群的结果
用在ER-阴性患者亚群的三种化疗方案上的三种模型性能列于表格7。每个模型的预测概率首先分类,然后分成5个等距的区间。用在较低预测概率范围的两个模型的负预测值都很好(两者都大于90%);但是用于较高的预测概率范围的TA模型的正预测值略低(33.3%),可能是由于观察到的病理完全缓解率低导致。新分配方案预测显示病理完全缓解的患者数量可增加超过100%,并且表明至少有四分之一的最初使用化疗方案TA的患者应该用化疗方案TxA。由于用紫杉醇和多西紫杉醇这两种化疗方案的患者数目相当,所以很难解释这两种药物在使用时存在显著偏好。但是有一点很明确,即在最初的化疗方案分配里,有更多的患者使用紫杉醇,但病理完全缓解值更低。
表格7
每个化疗方案的患者数目
*初始方案是观察数据The original group is observed.
实施例5
ER-阴性亚群的结果
ER-阴性亚群的两个模型表现都很好。患者采用TA方案的正预测值为0.824,在高预测概率范围的患者采用TxA方案的正预测值为0.895和0.9;而在低预测概率范围的患者的负预测值为0.9。按五等分划分的区间在这个亚群里均匀分布,重分配后的化疗方案结果表 明虽然方案TxA疗效不如方案TA,但仍有部分患者从TA换到TxA后取得更好的疗效。与初始化疗方案相比,至少有15位患者在换成TxA化疗方案后表现出更佳疗效,患者表现出更高的预期病理完全缓解(160.31>125,有30%增加)也证实了个人化化疗方案能增加患者的病理完全缓解。但是,在此亚群中,紫杉醇疗效更优于多西紫杉醇,因为预测表明更多使用TA化疗方案的患者表现出病理完全缓解。
实施例6
淋巴结-阳性亚群的结果
淋巴结-阳性亚群两个模型的低预测概率范围的负预测值大于90%,而高预测概率范围的正预测值为92%。由于TA化疗方案的最后一个区间覆盖了大于整个范围的65%,有90多名患者在此范围,因此TA化疗方案的区间被重新划分。新区间和它们对应的病理完全缓解值列在表格21中。在此亚群中,重新分配的化疗方案表明多西紫杉醇比紫杉醇更有效,使用个人化化疗方案分配后,预测表现病理完全缓解的患者数目大概增加了50%。
实施例7
淋巴结阴性亚群的结果
淋巴结阴性亚群的两个模型的负预测值(NPV)在低预测概率范围内偏高,而化疗方案TxA的正预测值在高预测概率范围偏高。但是化疗方案TA的最后一个区间覆盖了相对长的范围,而化疗方案TxA的前三个区间并无必要划分。在低预测概率范围内的负预测值仍很高,而两个模型在高预测概率范围内的正预测值也高。重新分配化疗方案结果表明在此亚群中紫杉醇疗效略优,但如果把至少36名患者的紫杉醇化疗方案转成使用多西紫杉醇化疗方案,能优化病理完全缓解的预计值,使预计表现病理完全缓解患者数目提高约85%。
实施例8
单个和成对探针具有预测功能
为了测出鉴定好的探针(基因)组里的每个单个探针(基因)或成对探针(基因)预测能力,每个探针和每对探针都用十折交叉验证随机森林法进行操作。测试部分的f-值(f-score of the test portion)可用来报告预测能力。要显示探针的优越性,可将结果与另一组 随机挑选的含同样数量探针的探针组结果相比较。从每个患者群中每个治疗方案中随机挑出300组探针。计算平均f-值和它们的95%置信区间,用来评估鉴定好的探针性能。
我们研究了以下患者群:使用治疗方案A并用单个探针检测,使用治疗方案A并用成对探针检测,使用治疗方案TA并用单个探针检测,使用治疗方案TA并用成对探针检测,使用治疗方案TxA并用单个探针检测,和使用TxA治疗方案并用成对探针检测。每组患者群按照HER2,和ER,淋巴结进一步分类。我们发现单个探针和成对的探针绝大部分都比随机抽取的探针有更好的预测力。
本申请中引用的所有文献均通过引用整体地结合于本文中。
尽管本文中已经描述了本发明中的说明性实施例,但是应当理解,本发明并不限于所描述的那些实施例,并且本领域技术人员在不脱离本发明范围和实质的情况下,可对其进行不同的变更或修改。
参考文献
ALBAIN,K.S.,et al.Prognostic and predictive value of the 21-gene recurrence score assay in postmenopausal women with node-positive,oestrogen-receptor-positive breast cancer on chemotherapy:a retrospective analysis of a randomised trial.Lancet Oncol 2010;11:55-65.
BARRETT,T.,et al.NCBI GEO:archive for functional genomics data sets--update.核酸s Res 2013;41:D991-5.
BUYSE,M.,et al.Validation and clinical utility of a 70-gene prognostic signature for women with node-negative breast cancer.J Natl Cancer Inst 2006;98:1183-92.
CALLE,M.L.,et al.AUC-RF:a new strategy for genomic profiling with random forest.Hum Hered 2011;72:121-32.
CHAN,S.,et al.Prospective randomized trial of docetaxel versus doxorubicin in patients with metastatic breast cancer.J Clin Oncol 1999;17:2341-54.
DE LAURENTIIS,M.,et al.Taxane-based combinations as adjuvant chemotherapy of early breast cancer:a meta-analysis of randomized trials.J Clin Oncol 2008;26:44-53.
DOTAN,E.,et al.Optimizing chemotherapy regimens for patients with early-stage breast cancer.Clin Breast Cancer 2010;10Suppl 1:E8-15.
EDGAR,R.,et al.Gene Expression Omnibus:NCBI gene expression and hybridization array data repository.核酸s Res 2002;30:207-10.
ENG-WONG,J.,et al.Prediction of benefit from adjuvant treatment in patients with breast cancer.Clin Breast Cancer 2010;10Suppl 1:E32-7.
ESSERMAN,L.J.,et al.Chemotherapy response and recurrence-free survival in neoadjuvant breast cancer depends on biomarker profiles:results from the I-SPY
1TRIAL(CALGB 150007/150012;ACRIN 6657).Breast Cancer Res Treat2012;132:1049-62.
FOEKENS,J.A.,et al.Multicenter validation of a gene expression-based prognostic signature in lymph node-negative primary breast cancer.J Clin Oncol 2006;24:1665-71.
GAJRIA,D.,et al.Adjuvant taxanes:more to the story.Clin Breast Cancer 2010;10Suppl 2:S41-9.
GRAESER,M.,et al.A marker of homologous recombination predicts pathologic complete response to neoadjuvant chemotherapy in primary breast cancer.Clin Cancer Res 2010;16:6159-68.
HARBECK,N.,et al.Ten-year analysis of the prospective multicentre Chemo-N0trial validates American Society of Clinical Oncology(ASCO)-recommended biomarkers uPA and PAI-1for therapy decision making in node-negative breast cancer patients.Eur J Cancer 2013;49:1825-35.
HATZIS,C.,et al.A genomic predictor of response and survival following taxane-anthracycline chemotherapy for invasive breast cancer.JAMA 2011;305:1873-81.
HESS,K.R.,et al.Pharmacogenomic predictor of sensitivity to preoperative chemotherapy with paclitaxel and fluorouracil,doxorubicin,and cyclophosphamide in breast cancer.J Clin Oncol 2006;24:4236-44.
IWAMOTO,T.,et al.Gene pathways associated with prognosis and chemotherapy sensitivity in molecular subtypes of breast cancer.J Natl Cancer Inst 2011;103:264-72.KAUFMANN,M.,et al.Recommendations from an international expert panel on the use of neoadjuvant(primary)systemic treatment of operable breast cancer:an update.J Clin Oncol 2006;24:1940-9.
KUERER,H.M.,et al.Clinical course of breast cancer patients with complete pathologic primary tumor and axillary lymph node response to doxorubicin-based neoadjuvant chemotherapy.J Clin Oncol 1999;17:460-9.
LIPS,E.H.,et al.Neoadjuvant chemotherapy in ER+HER2-breast cancer:response prediction based on immunohistochemical and molecular characteristics.Breast Cancer Res Treat 2012;131:827-36.
LIU,J.C.,et al.Seventeen-gene signature from enriched Her2/Neu mammary tumor-initiating cells predicts clinical outcome for人HER2+:ERalpha-breast cancer.Proc Natl Acad Sci U S A 2012;109:5832-7.
LOOK,M.P.,et al.Pooled analysis of prognostic impact of urokinase-type plasminogen activator and its inhibitor PAI-1in 8377breast cancer patients.J Natl Cancer Inst2002;94:116-28.
MIYAKE,T.,et al.GSTP1expression predicts poor pathological complete response to neoadjuvant chemotherapy in ER-negative breast cancer.Cancer Sci 2012;103:913-20.
MOOK,S.,et al.Individualization of therapy using Mammaprint:from development to the MINDACT Trial.Cancer Genomics Proteomics 2007;4:147-55.
OAKMAN,C.,et al.Breast cancer assessment tools and optimizing adjuvant therapy.Nat Rev Clin Oncol 2010;7:725-32.
PAIK,S.,et al.Gene expression and benefit of chemotherapy in women with node-negative,estrogen receptor-positive breast cancer.J Clin Oncol 2006;24:3726-34.
POPOVICI,V.,et al.Effect of training-sample size and classification difficulty on the accuracy of genomic predictors.Breast Cancer Res 2010;12.
SHEN,K.,et al.Cell line derived multi-gene predictor of pathologic response to neoadjuvant chemotherapy in breast cancer:a validation study on US Oncology 02-103clinical trial.BMC Med Genomics 2012;5:51.
SHEN,K.,et al.Cell line derived multi-gene predictor of pathologic response to neoadjuvant chemotherapy in breast cancer:a validation study on US Oncology 02-103clinical trial.BMC Med Genomics 2012;5.
SLEDGE,G.W.,et al.Phase III trial of doxorubicin,paclitaxel,and the combination of doxorubicin and paclitaxel as front-line chemotherapy for metastatic breast cancer:an intergroup trial(E1193).J Clin Oncol 2003;21:588-92.
SPARANO,J.A.,et al.Weekly paclitaxel in the adjuvant treatment of breast cancer. N Engl J Med 2008;358:1663-71.
STRAVER,M.E.,et al.The 70-gene signature as a response predictor for neoadjuvant chemotherapy in breast cancer.Breast Cancer Res Treat 2010;119:551-8.
TABCHY,A.,et al.Evaluation of a 30-gene paclitaxel,fluorouracil,doxorubicin,and cyclophosphamide chemotherapy response predictor in a multicenter randomized trial in breast cancer.Clinical cancer research:an official journal of the American Association for Cancer Research 2010;16:5351-61.
TAKADA,M.,et al.Predictions of the pathological response to neoadjuvant chemotherapy in patients with primary breast cancer using a data mining technique.Breast Cancer Res Treat 2012;134:661-70.
THE CANCER GENOME ATLAS NETWORK.Comprehensive molecular portraits of人breast tumours.Nature 2012;490:61-70.
VAN DE VIJVER,M.J.,et al.A gene-expression signature as a predictor of survival in breast cancer.N Engl J Med 2002;347:1999-2009.
VAN’T VEER,L.J.,et al.Gene expression profiling predicts clinical outcome of breast cancer.Nature 2002;415:530-6.
VERWEIJ,J.,et al.Paclitaxel(Taxol)and docetaxel(Taxotere):not simply two of a kind.Ann Oncol 1994;5:495-505.
VON MINCKWITZ,G.,et al.Definition and impact of pathologic complete response on prognosis after neoadjuvant chemotherapy in various intrinsic breast cancer subtypes.J Clin Oncol 2012;30:1796-804.
WANG,Y.,et al.Gene-expression profiles to predict distant metastasis of lymph-node-negative primary breast cancer.Lancet 2005;365:671-9.
- 还没有人留言评论。精彩留言会获得点赞!