一种修复酵母染色体结构异常的方法与流程
本发明涉及基因组工程领域,具体涉及一种修复酵母染色体结构异常的方法。
背景技术:
染色体结构异常是指染色体非正常的发生断裂、重组或互换后产生染色体畸变。染色体结构异常的类型可以分为DNA拷贝数变异、大片段插入、大片段复制、大片段翻转、染色体移位等。染色体结构异常广泛发生在绝大多数物种中,很多染色体结构异常与细胞衰老、染色体遗传性疾病、癌症等疾病密切相关。例如,猫叫综合症是人的第五号染色体部分缺失引起的遗传病,果蝇的缺刻翅也是由于一段染色体的缺失造成的;果蝇的棒眼现象是X染色体上的部分片段重复引起的;第九号染色体长臂的倒置会造成女性习惯性流产。染色体结构变异可能来自于遗传性的变异也可能是由于射线、化学试剂、温度等外部环境的诱导或者生物体内代谢失衡、细胞衰老凋亡等引起的病变性质的变异,还可能来自于分子生物学的操作比如基因组编辑和大尺度DNA的合成等。大多数的染色体结构变异对生物体是不利的,会引起细胞生长缺陷,严重的甚至会致死。
酵母是与人类活动关系最密切的微生物之一,比较基因组学的研究表明不同种属的酿酒酵母之间有大量的染色体结构变异,这些染色体的结构差异一方面导致了不同物种的区分,另一方面也使不同类型的酵母拥有不同的生长特性。比如有耐高温耐抑制剂的酿酒酵母,也有高产乙醇的工业酵母菌株。和其他物种相似,酵母的染色体结构变异绝大多数也是不利的,需要对其进行修复。现有的染色体修复技术涉及修复的DNA尺度都比较小,比如利用CRISR-Cas9技术可以较好的实现点突变的修复或者和同源重组技术相结合可以实现数Kb长度的DNA序列的修复,但外源长DNA片段的合成和较低的转化效率使得CRISPR-Cas9技术一般局限于数Kb长度DNA序列的改变,对于数百Kb长度的DNA无法有效修复。目前针对数百Kb长度的DNA修复技术鲜有报道。因此,需要开发一种高效准确的修复酵母染色体结构异常的技术。
技术实现要素:
有鉴于此,本发明的目的在于提供一种修复酵母染色体结构异常的方法,使得所述方法能够更加简便和快速的修复酵母异常染色体,并扩大DNA的修复尺度至数百Kb。
为了实现上述目的,本发明提供如下技术方案:
一种修复酵母染色体结构异常的方法,包括:
步骤1、确认变异酵母的染色体结构异常区域;
步骤2、变异酵母染色体上,在与着丝粒最接近的染色体结构异常区域的近着丝粒端朝着丝粒方向延伸的区域插入筛选标签基因1;正常酵母染色体上,在与筛选标签基因1相对应的位置处朝着丝粒方向延伸的区域插入筛选标记标签2;所述筛选标签基因1与筛选标签基因2不同,所述正常酵母在其与变异酵母的染色体结构异常区域相对应区域上具有正常染色体;
步骤3、将不同交配型的变异酵母和正常酵母交配形成二倍体酵母,然后进行减数分裂获得四分体,对四分体进行拆分获得孢子;
步骤4、根据筛选标签基因1和筛选标签基因2分别设置对应的筛选标签培养基,将所述孢子分别接种到两种筛选标记培养基上,两种筛选培养基上均无法正常生长的孢子为已经修复的酵母。
由于借助CRISR-Cas9技术对异常染色体修复时存在DNA修复尺度较小,且比较繁琐的缺陷,本发明结合酵母高效的同源重组机制,通过筛选标记和减数分裂实现大尺度酵母染色体的高效修复。
为了方便理解本发明的技术思路,在图1所示的染色体修复示意图的基础上对本发明方法的原理进行阐述(在图1中,筛选标签1和2分别选择URA、LEU,简写为U和L,阴影部分表示染色体结构异常区域,交叉区域为同源重组链交换区域,即U和L之间的区域):
本发明利用酵母细胞减数分裂时非姐妹染色单体的同源重组实现结构异常的染色体和结构正常的染色体之间的链交换。在单倍体酵母菌株中出现染色体结构变异的末端位置插入URA基因。在染色体结构正常的单倍体酵母中(与染色体结构变异的单倍体酵母不同的交配型,具有完全正常的染色体),将LEU基因插入到与染色体结构异常区域对应的链交换区域。将上述两个不同交配型的菌株交配形成二倍体酵母,在生孢培养基中培养二倍体菌株使其生孢。使用酵母显微操作仪对生孢的四分体细胞进行拆分,之后将生长的单倍体孢子接种到SC-URA(缺URA培养基)和SC-LEU(却LEU培养基)的平板来筛选在链交换区发生链交换的孢子,只有在设定区间发生了链交换才会引起两个标签基因位置的变换,根据结果可知,形成的四分体经过拆分后会出现四种单倍体孢子形式,其中一种就是不含任何筛选标签的已经完全修复的菌株,既不能在SC-URA平板上生长也不能在SC-LEU平板上生长的单倍体孢子就是所需要的菌株。
对于变异酵母的异常染色体区域的定位方法可按照本领域常规的方法进行,在本发明具体实施中,本发明步骤1为通过脉冲场凝胶电泳和全基因组测序分析确认变异酵母的染色体结构异常区域。
本发明中筛选标签基因1的插入位置是一个区间范围,从与着丝粒最接近的染色体结构异常区域的近着丝粒端开始至着丝粒这一段区域都可以作为筛选标签基因1的插入位置,以图1为例说明,这一区域就是筛选标签U到着丝粒之间的区域,考虑到后续筛选标签基因2的插入,一般不选择极为靠近着丝粒的区域。在本发明具体实施过程中,筛选标签基因1的插入位置通常是距染色体结构异常区域的近着丝粒端100-1000bp长度位置处,即所述与着丝粒最接近的染色体结构异常区域的近着丝粒端朝着丝粒方向延伸的区域为与着丝粒最接近的染色体结构异常区域近着丝粒端朝着丝粒方向延伸100-1000bp的区域。
在本发明方法中,选择的正常酵母具备完全正常的染色体,与变异酵母除了在交配型和染色体结构异常区域的差别外(交配型不同是为了能够形成二倍体),其他位置的染色体基本相同。如此,变异酵母染色体上插入筛选标签基因1的位置,在正常酵母染色体上也会存在相对应的位置(为表述方便,该位置称为筛选标签基因1对应位置),从筛选标签基因1对应位置开始至着丝粒这一个区域为筛选标签基因2的插入位置,以图1为例说明,这一区域就是筛选标签U对应到正常酵母染色体上的位置到着丝粒之间的区域,考虑到后续快捷拆分到正确四分体,筛选标签基因2的插入位置通常是距筛选标签基因1对应位置越远越好,这样预期在链交换区发生链交换的几率则呈正向相关,拆分到正确的四分体也就更加快捷,由于二倍体酵母经过培养,其形成的众多四分体中存在不计其数的各种链交换情形,本发明所预想的链交换情形通常均会发生。
在本发明具体实施过程中,筛选标签基因2的插入位置通常是距筛选标签基因1对应位置10-100kb的区域长度位置处。即所述在与筛选标签基因1相对应的位置处朝着丝粒方向延伸的区域为在与筛选标签基因1相对应的位置处朝着丝粒方向延伸10-100kb的区域。
对于筛选标签基因的插入,可通过组装上游500bp同源臂、筛选标签基因、下游500bp同源臂三个片段构成待整合的外源片段,通过酵母同源重组整合到预期位置。
本发明中所提及的筛选标签基因1和筛选标签基因2可选自微生物领域中经常用于筛选目标微生物所采用的筛选标签基因,如营养缺陷标签基因和抗药标签基因。所述营养缺陷标签一般为氨基酸缺陷标签例如URA、LEU和HIS;所述抗药标签一般选自KanMX、NAT和Hyg。
在根据筛选标签基因1和筛选标签基因2分别设置对应的筛选标签培养基时,本领域技术人员可根据选择的标签轻易设置,如采用URA筛选标签,则对应的筛选标签培养基为缺URA培养基(SC-URA),采用KanMX筛选标签则对应的筛选标签培养基为含有G418的培养基。
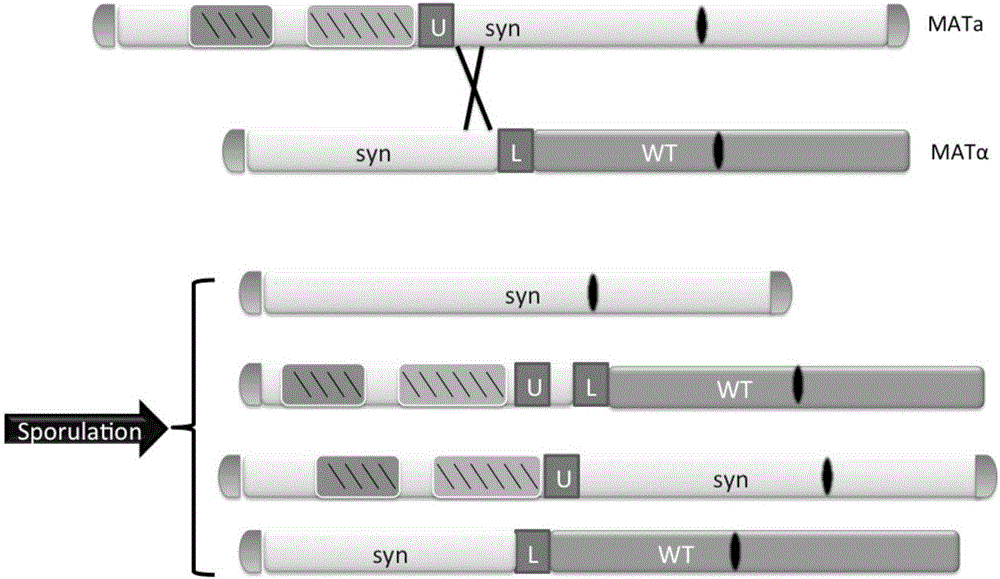
在酵母功能基因组学领域,随着酵母基因组信息的被揭示,合成型染色体的酵母(包含全合成型染色体酵母和半合成型染色体酵母)因其方便性被广泛使用,然而其同样会产生染色体变异,需要进行修复。本发明所述方法同样适用于合成型染色体酵母的染色体结构异常修复,此时本发明所述变异酵母为全合成型染色体或半合成型染色体的酵母,所述正常酵母为半合成型染色体酵母或全合成型染色体酵母;所述正常酵母在变异酵母染色体结构异常区域相对应区域上具有正常的合成型染色体;所述合成型染色体与野生型染色体在每个功能基因上序列存在微小差别(用于识别合成型染色体),高度相同,同时能够表达相同蛋白。具体的染色体修复示意图见图2。
在本发明具体实施方式中,本发明以出现染色体结构异常的全合成型染色体酿酒酵母R-2为对象进行修复,脉冲场凝胶电泳对酿酒酵母R-2菌进行核型分析,可以看到菌株R-2和野生型对照菌株BY4741相比,十号染色体对应的条带明显向上发生了迁移。在全基因组测序分析菌株R-2后,通过测序深度图发现有两处区域有大片段复制重复。一处主要发生在megachunk C区域,涉及序列长度为101kb,为连续发生的三拷贝的复制。第二处主要发生在megachunk E区域,涉及序列长度为147kb,为二拷贝和四拷贝交错排布的复制。综合计算R-2菌株十号染色体的长度比野生型长度长171kb。
然后在单倍体酵母菌株R-2中出现染色体结构变异的末端位置插入URA基因。在染色体结构正常的单倍体半合成型染色体酵母BY4742中(与菌株R-2不同的交配型),将LEU基因插入到与染色体结构异常区域对应的链交换区域。将上述两个不同交配型的菌株R-2和BY4742交配形成二倍体酵母,在生孢培养基中培养二倍体菌株使其生孢。使用酵母显微操作仪对生孢的四分体细胞进行拆分,拆分的单倍体细胞按照四分体的来源整齐排布在YPD平板上培养2天,之后将生长的单倍体孢子平板翻印到SC-URA和SC-LEU的平板来筛选在设定区间发生链交换的孢子。挑取既不能在SC-URA平板上生长也不能在SC-LEU平板上生长的单倍体孢子,只有在设定区间发生了链交换才会引起两个标记基因位置的变换。挑取通过表型验证的单倍体孢子,利用PCR或其他检测方法验证染色体结构异常是否被修复。
由以上技术方案可知,本发明利用酵母高效的同源重组机制以及筛选标签,快速筛选在设计区域发生链交换的酵母染色体,实现染色体结构异常的序列与正常染色体序列的交换,从而达到大尺度高效修复酵母染色体结构变异的目的。
附图说明
图1所示为本发明修复染色体结构异常的示意图;其中,MATa与MATα代表酵母中的两种交配型,阴影部分为染色体结构异常区域,U和L分别代表URA基因和LEU基因,交叉位置为链交换区;
图2所示为本发明修复染色体结构异常的示意图;其中,MATa与MATα代表酵母中的两种交配型,阴影部分为染色体结构异常区域,U和L分别代表URA基因和LEU基因,syn表示合成型染色体,WT表示野生型染色体,sporulation表示生孢,交叉位置为链交换区;
图3所示为脉冲场凝胶电泳;其中,罗马数字表示染色体编号,BY4741表示正常酵母,R-2表示变异酵母,箭头所指处为X号染色体条带;
图4所示为酵母R-2基因组测序深度图;
图5所示为筛选标签培养基筛选染色体结构修复的菌株结果;其中,1-3表示不同四分体下的4个孢子,YPD为正常培养基,-URA为缺URA的筛选培养基,-LEU为缺LEU培养基,图中圆圈选定的孢子为修复后的菌株,即标号1的四分体的第4个孢子;
图6所示为PCR验证染色体结构变异修复正常的凝胶图;其中,BY4741表示正常酵母,R-2表示变异酵母,synX表示修复后的酵母。
具体实施方式
本发明公开了一种修复酵母染色体结构异常的方法,本领域技术人员可以借鉴本文内容,适当改进工艺参数实现。特别需要指出的是,所有类似的替换和改动对本领域技术人员来说是显而易见的,它们都被视为包括在本发明。本发明所述方法已经通过较佳实施例进行了描述,相关人员明显能在不脱离本发明内容、精神和范围内对本文所述的方法进行改动或适当变更与组合,来实现和应用本发明技术。
根据本发明记载的修复方法,其可以概括为如下:
(1)确认染色体结构变异的区域;(2)在染色体结构变异出现的末端位置插入筛选标记基因;(3)在染色体结构正常的单倍体酵母对应位置的链交换区域插入另一个筛选标记基因;(4)染色体变异的酵母和染色体正常的酵母交配形成二倍体;(5)减数分裂生孢;(6)孢子拆分,并进行筛选基因的表型验证。
以下就本发明所提供的一种修复酵母染色体结构异常的方法做进一步说明。
实施例1:修复染色体结构异常的全合成型染色体酿酒酵母R-2
脉冲场凝胶电泳对酿酒酵母R-2菌进行核型分析,观察图3可以看到菌株R-2和野生型对照菌株BY4741相比,十号染色体对应的条带明显向上发生了迁移。在全基因组测序分析菌株R-2后,通过图4测序深度图发现有两处区域有大片段复制重复。一处主要发生在megachunk C区域,涉及序列长度为101kb,为连续发生的三拷贝的复制。第二处主要发生在megachunk E区域,涉及序列长度为147kb,为二拷贝和四拷贝交错排布的复制。综合计算R-2菌株十号染色体的长度比野生型长度长171kb。针对重复区域特异性的连接处位置的序列设计引物,连接处设计扩增大小分别为1kb,500bp两对引物。从结果可以看出只有发生大片段重复的菌株才能扩增出大小正确的条带,而对照BY4741并无条带扩增。将PCR产物送测,可以得到断裂位点的明确序列。根据全基因组测序拼接结果及连接处PCR产物测序结果分析得到染色体结构异常的具体位置信息。
利用酵母细胞减数分裂时非姐妹染色单体的同源重组实现结构异常的染色体和结构正常的染色体之间的链交换(图2所示示意图)。在单倍体酵母菌株R-2中出现染色体结构变异的末端位置插入URA基因。在染色体结构正常的单倍体半合成型染色体酵母BY4742中(与染色体结构变异的单倍体R-2不同的交配型),将LEU基因插入到与染色体结构异常区域对应的链交换区域。将上述两个不同交配型的菌株R-2和BY4742交配形成二倍体酵母,在生孢培养基中培养二倍体菌株4到7天使其生孢。
二倍体酵母细胞液体生孢的流程如下:
1.预生孢。在30℃YPD液体培养基中培养二倍体酵母细胞至OD600值达到4~8。
2.漂洗。2000g室温离心2min将预生孢的二倍体细胞沉淀,移除上清液并用超纯水重悬细胞。重复上述漂洗过程两次。
3.生孢。用超纯水稀释50X生孢液到1X浓度。添加需要补充的氨基酸(依据二倍体酵母细胞的营养缺陷情况)。每毫升的1X生孢液需要额外添加3μl10%的酵母提取物。混合均匀。把漂洗干净的预生孢二倍体酵母加入混合好的生孢培养基内使其OD600值大于1。在25℃下培养3-10天。通过显微观察是否有四分体孢子出现来检验是否生孢成功。
YPD培养基:1%酵母提取物,2%蛋白胨,2%葡萄糖。
50X预生孢培养基:50%醋酸钾,0.25%二水乙酸锌加超纯水溶解。过滤除菌,室温保存。
10%酵母提取物:10%酵母提取物溶解于超纯水。4℃保存。
使用酵母显微操作仪对生孢的四分体细胞进行拆分,拆分步骤如下:
1.吸取50uL生孢液,2000g离心2分钟。
2.去除上清夜,重悬细胞在25uL的0.5mg/mL的裂解酶缓冲液中(溶剂为1M山梨醇)。
3.在37℃条件下孵育10分钟。
4.用1M山梨醇稀释至300uL,置于冰上。
5.利用酵母显微拆分仪,将生孢成功的四分体细胞打散并挑取到对应的平板位置。需要至少拆分十个四分体细胞。
拆分的单倍体细胞按照四分体的来源整齐排布在YPD平板上培养2天,之后将生长的单倍体孢子平板翻印到SC-URA和SC-LEU的平板来筛选在设定区间发生链交换的孢子。挑取既不能在SC-URA平板上生长也不能在SC-LEU平板上生长的单倍体孢子,只有在设定区间发生了链交换才会引起两个标记基因位置的变换。通过图5可知,来自同一个四分体的孢子在SC-URA,SC-LEU平板上都呈现2:2分布,完全符合链交换后的比例分布,其中四分体1的第4个孢子(被圆圈标注)既不能在SC-URA平板上生长也不能在SC-LEU平板上生长,其即为修复成功的菌株,命名为synX。
挑取通过表型验证的单倍体孢子synX,利用PCR验证染色体结构异常是否被修复,从图6 PCR验证胶图结果可以看出,R-2菌株染色体特异性的异常结构不能在修复后的菌株synX中扩增出来,表明染色体结构异常被修复成功。
注:菌株R-2对应的野生型染色体酵母是BY4741,两者交配型相同,区别在于一个是全合成型染色体酵母,一个是野生型染色体;BY4742与BY4741仅为交配型不同,本实施中的BY4742为半合成型染色体酵母,存在的合成型染色体位置对应于R-2中出现染色体结构异常的区域,其他位置染色体均为野生型染色体,与BY4741相同。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!