与小麦抗穗发芽性状相关的KASP分子标记及其应用的制作方法
本发明涉及分子生物学及作物育种技术领域,具体地说,涉及与小麦抗穗发芽性状相关的kasp分子标记及其应用。
背景技术:
小麦是世界第三大粮食作物,仅次于水稻和玉米,在世界范围内广泛种植。小麦穗发芽(pre-harvestsprouting,phs)是指小麦在收获前遇到的连绵阴雨时,未及收获便在穗上发芽的一种现象。穗发芽是一种世界性的灾害,据报告,世界主要小麦栽培国如加拿在、日本、澳大利亚、中国均有严重穗发芽危害。我国有10大小麦主产区,主要以中国中部、东南部麦区如长江中下游麦区、西南麦区穗发芽危害更为严重,随着近年来降雨北移现象,我国北部穗发芽麦区如北部冬麦区也常有穗发芽现象发生。
小麦籽粒在收获前的穗发芽不仅会带来大面积的粮食产量降低,也会导致小麦籽粒中水解等相关的酶活性迅速升高,降低籽粒中储藏物质含量,容重(testingweight)、出粉率和面粉降落值(fallingnumber)下降,造成小麦各种加工食品品质恶化,且会影响小麦籽粒的储存。因此,选育具有穗发芽抗性的小麦资源对小麦育种具有重大的实际意义。
分子标记辅助选择育种技术,是利用与目的性状候选基因连锁的分子标记,通过分子生物学检测候选基因分型,达到对目的性状选择的目的;其优点是简单快速,不受环境及环境互作的影响,能够快速且高效的选育出目标资源材料。单核苷酸的多态性(snp,singlenucleotidepolymorphisms),是指在基因组上单个核苷酸的变异所引起的dna序列多态性,包括转换(transition)、颠换(transversion)、缺失(deletion)和插入(insertion)。snp成为第三代遗传标志,人体许多表型差异、对药物或疾病的易感性等等都可能与snp有关。主要有以下特点:1、数量多,分布广泛;2、适于快速、规模化筛查;3、等位基因频率的容易估计;4、易于基因分型。利用全基因组的snp关联分析方法和候选基因的snp方差分析的方法,可以找到与表型相关的snp变异。
kasp(竞争性等位基因特异性pcr,即kompetitiveallele-specificpcr)技术是lgcgenomics公司的snpline基因分型检测方案。该方案的核心是kasp技术,这项技术是基于引物末端碱基的特异匹配来对snp分型以及检测indels(insertionsanddeletions,插入和缺失)。该项技术优点是:其一,只要合成两个通用荧光探针,两个通用淬灭探针,再加合成多个针对具体位点的snppcr引物,就可以测许多位点;其二,因为荧光探针、和淬灭探针都很贵,kasp方法相比于taqman方法,可以通过通用荧光探针来代替针对位点的荧光探针,大大节约成本。
因此,筛选出与抗穗发芽相关的snp,并将其开发为能准确鉴定其snp位点的kasp标记,对抗穗发芽材料选择,在选育小麦抗穗发芽材料,减小穗发芽对小麦的影响,提高小麦产量和品质等方面均有重要的意义。
中国小麦分布地域辽阔,由于各地自然条件、种植制度、品种类型和生产水平存在着不同程度的差异,一方面形成了明显的种植区域,另一方面极大地丰富了中国地方品种的多样性。利用大规模来自全国各地的地方品种,对其在2012年到2015年不同环境的穗发芽表型鉴定,并进行全基因组的关联分析,鉴定到3个位于小麦第3同源群的与穗发芽表型相关的snp标记。针对这3个分子标记展开进一步研究,并将此开发为可用于分子标记辅助选择育种的kasp标记,能有效提高小麦育种效率和准确性。
技术实现要素:
本发明的目的是提供与小麦抗穗发芽性状相关的kasp分子标记及其应用。
为了实现本发明目的,本发明提供与小麦抗穗发芽性状相关的kasp分子标记,共包含3个分子标记,①分子标记ax-111578083与抗穗发芽基因myb10-a1之间的遗传图距为0.35mb;②分子标记ax-111624595与抗穗发芽区段r-loci之间的遗传图距为3.55mb;③分子标记ax-110772653与抗穗发芽区段r-loci之间的遗传图距为3.73mb。分子标记ax-111578083、ax-111624595和ax-110772653均来自中国地方品种小麦,分别位于中国春survey序列(iwgsc2.28)3a染色体173.81mb、3d染色体112.63mb和112.80mb,它们的核苷酸序列分别如seqidno:1-3所示。
本发明还提供基于kasp技术开发的用于扩增所述分子标记的引物,包括正向引物al1、正向引物al2和反向引物c。
其中,扩增分子标记ax-111578083的引物序列分别如seqidno:4-6所示,扩增分子标记ax-111624595的引物序列分别如seqidno:7-9所示,扩增分子标记ax-110772653的引物序列分别如seqidno:10-12所示。
本发明还提供含有所述引物的用于鉴定与小麦抗穗发芽性状相关的kasp分子标记的检测试剂盒。
本发明还提供所述分子标记、引物或试剂盒在小麦抗穗发芽材料创制中的应用。
本发明还提供所述分子标记、引物或试剂盒在分子标记辅助小麦抗穗发芽选择育种中的应用。
本发明进一步提供所述分子标记、引物或试剂盒在选育具有穗发芽抗性的小麦资源中的应用。包括以下步骤:
1)提取待测小麦样品的dna;
2)取浓度为50-100ng/μl的模板dna1.00μl,引物混合液0.14μl,kaspmastermix5.00μl,50mm的mgcl2溶液0.08μl,h2o3.78μl,进行pcr扩增;引物混合液中正向引物al1和正向引物al2的终浓度均为10μm,反向引物c的终浓度为10μm;
3)采用荧光检测仪分析pcr扩增产物基因型。
步骤2)pcr反应条件为:95℃预变性15分钟;第一步扩增反应:95℃变性20秒,68℃梯度退火并延伸60秒,10个循环,每个循环退火及延伸的温度降低1℃;第二步扩增反应,94℃变性20秒,57℃退火并延伸60秒,30个循环;12℃保存。
步骤3)具体为:利用生物学软件对pcr扩增产物进行分型:等位位点ax-111578083-g为携有抗穗发芽优异等位类型植株,等位位点ax-111624595-c为携有抗穗发芽优异等位类型植株,等位位点ax-110772653-t为携有抗穗发芽优异等位类型植株。
鉴定小麦抗穗发芽的引物以及检测小麦抗穗发芽能力强弱的方法在如下(a)-(e)中的应用也属于本发明的保护范围:
(a)筛选抗穗发芽能力强的小麦品种。
(b)筛选抗穗发芽能力弱的小麦品种。
(c)培育抗穗发芽的小麦新品种。
(d)所述3个kasp分子标记在小麦特异抗穗发芽材料创制中的应用。
(e)所述3个kasp分子标记引物在小麦特异抗穗发芽材料创制中的应用。
本发明小麦抗穗发芽候选位点及其分子标记是通过以下方法获得的:
(1)通过收集来自中国10大麦区的272份地方品种小麦组成自然群体。
(2)自然群体穗发芽多年多点鉴定
于2012年-2015年在温江、雅安、崇州共6个不同的环境下,于蜡熟期收获小麦籽粒,将其悬挂于阴凉通风处自然风干7d后,进行人工脱粒。在有盖塑料培养皿中垫上用3-5ml蒸馏水润湿的滤纸进行发芽试验,试验设计三个重复,每个重复50粒种子。每隔一天挑出已露白和已发霉的种子,并分别计数,种子发芽率以第7天发芽种子数占总种子数百分比表示,计算发芽率。
发芽率(%)=发芽种子数/(种子总数-发霉数)×100%
(3)基因型扫描
a)基因组dna提取:采用ctab法提取272自然群体群体植株dna,检测其浓度,并稀释至50-100ng/ul。
b)wheat660snp扫描
①所有材料送至博奥公司affymetrix平台,并进行genomicdna质量检测,用wheat660snparray进行全基因组扫描。
②wheat660snparray原始数据分析:以上述步骤共获得630517个snp探针,通过目前发表的小麦参考基因组图谱iwgsc2.28,锚定到单一位点的共312,831个snp标记,其中a基因组标记122,338个,b基因组标记137,494个,d基因组标记52,999个。
③自然群体标记质量控制:通过筛选在272份中国地方品种中有差异、缺失率小于20%且最小等位基因频率(maf)大于5%标记共178,803个。
④自然群体分层计算:所选178,803个高质量snp分子标记,利用生物信息学软件structure2.34评估地方品种群体分层,选择最佳群体结果k=5,获得q值。
⑤自然群体亲缘关系评估:所选178,803个高质量snp分子标记,利用生物信息学软件kinship评估地方品种群亲缘关系,获得k值。
⑥穗发芽全基因组关联分析:所选178,803个高质量snp分子标记,以及前面得到的q和k值,加上6个不同环境下的穗发芽表型,利用生物信息学软件tassel5.0进行全基因组关联分析(gwas),鉴定到3个稳定与穗发芽表型强选择相关的分子标记ax-111578083、ax-111624595和ax-110772653。
(4)kasp标记的开发
为了方便对小麦抗穗发芽候选材料进行3个snp标记ax-111578083、ax-111624595和ax-110772653的鉴定和辅助选择,同时为了降低育种成本和工作量,增强育种工作中的可操作性,需要将与目标分子标记探针序列开发为基于常规分子生物学手段可用于鉴定和筛选的kasp标记,以下是相关kasp标记开发过程中的主要步骤:
①探针序列:
为开发基于实验室可用pcr技术的分子标记,方便用于分子标记辅助育种及应用,本发明开展了将ax-111578083、ax-111624595和ax-110772653探针序列转化为常规分子标记的实验,已知的snp标记ax-111578083、ax-111624595和ax-110772653探针序列分别如下:
ax-111578083:
cgtcacatggagcatcaggatcggtagcttccccc[a/g]tgaggattggaccagggatcaccacataagatcca(seqidno:1)
ax-111624595:
tccaccgtagtgggaggcctatgaaccaacgaatt[c/t]gcggaggagccaacaaaaggagaccgcacgacagatctgcccgaagtgacacatgtctccacgactggcgtgta(seqidno:2)
ax-110772653:
gtggtgtggaactgcctcaaccgcgcctcttggag[a/t]tgtgcctcgctggacccgagttcttcccctcatct(seqidno:3)
②引物设计:
根据已知的snp标记ax-111578083、ax-111624595和ax-110772653探针序列设计如下引物:
ax-111578083:
正向引物al1:5’–3’gaaggtgaccaagttcatgctgatcggtagcttccccca(seqidno:4)
正向引物al2:5’–3’gaaggtcggagtcaacggattgatcggtagcttcccccg(seqidno:5)
反向引物c:5’–3’gtgatccctggtccaatc(seqidno:6)
ax-111624595:
正向引物al1:5’–3’gaaggtgaccaagttcatgctggcctatgaaccaacgaattc(seqidno:7)
正向引物al2:5’–3’gaaggtcggagtcaacggattggcctatgaaccaacgaattt(seqidno:8)
反向引物c:5’–3’cagtcgtggagacatgtgtcac(seqidno:9)
ax-110772653:
正向引物al1:5’–3’gaaggtgaccaagttcatgctcaaccgcgcctcttggaga(seqidno:10)
正向引物al2:5’–3’gaaggtcggagtcaacggattcaaccgcgcctcttggagt(seqidno:11)
反向引物c:5’–3’cgagttcttcccctcatct(seqidno:12)
③引物检测:
以22份中国地方品种小麦基因组dna为模板,分别利用以上引物进行pcr扩增,bio-radicyclerthermalcyclerw/iq5opticalmoduleforrt-pcr(bio-radiq5实时荧光定量pcr仪)在37℃进行荧光收集,利用生物信息学软件bioradcfxmanager对pcr产物进行分型。
本发明提供小麦分子育种领域3个用于筛选抗穗发芽基因的分子标记(引物)及方法和应用。分析表明,基于snp标记及其侧翼序列开发出的3个位于小麦第3同源群的kasp标记(竞争性等位基因特异性pcr标记),能准确地对抗穗发芽候选位点进行分型,预测小麦的穗发芽抗性,从而更加方便地在实验室条件下对抗穗发芽材料进行鉴定和筛选。利用分子标记辅助选择育种技术能有效避免环境因素和人为误差对表型鉴定带来的影响。本发明的3个kasp分子标记,能提供强选择位点,提高抗穗发芽育种材料选择的准确性,实现小麦抗穗发芽分子标记辅助选择育种的目标。
附图说明
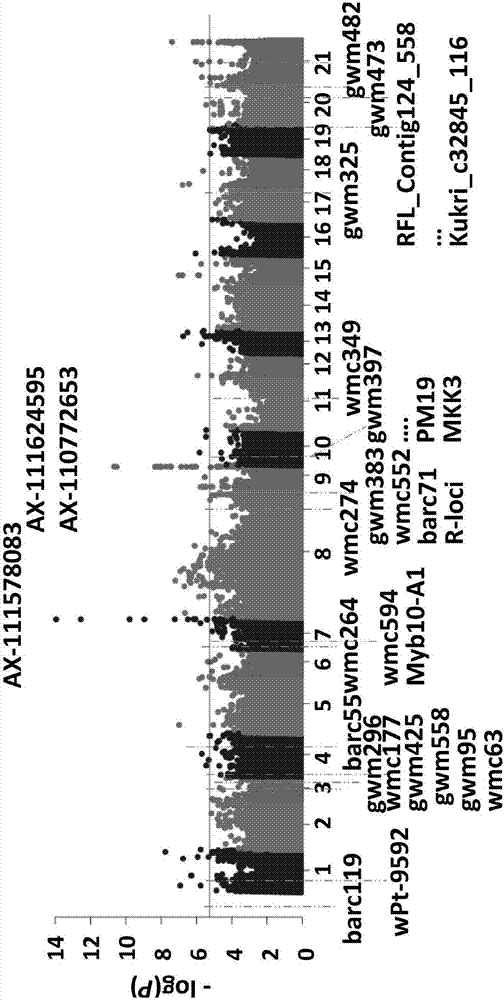
图1为本发明中国地方品种小麦多年多点的表型与基因型关联分析结果(关联分析显著位点ax-111578083、ax-111624595和ax-110772653),图谱信息来自中国春survey序列iwgsc2.28。
图2为本发明snp位点ax-111578083的kasp分子标记在已知基因型的22份中国地方品种中的鉴定结果,其中等位位点ax-111578083-g为携有抗穗发芽优异等位类型植株。
图3为本发明snp位点ax-111624595的kasp分子标记在已知基因型的22份中国地方品种中的鉴定结果,其中等位位点ax-111624595-c为携有抗穗发芽优异等位类型植株。
图4为本发明snp位点ax-110772653的kasp分子标记在已知基因型的22份中国地方品种中的鉴定结果,其中等位位点ax-110772653-t为携有抗穗发芽优异等位类型植株。
图5为本发明3个不同snp位点的kasp标记(ax-111578083、ax-111624595和ax-110772653)在138份中国主推品种中的类型以及对应的穗发芽表型对比结果。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例均按照常规实验条件,如sambrook等分子克隆实验手册(sambrookj&russelldw,molecularcloning:alaboratorymanual,2001),或按照制造厂商说明书建议的条件。
实施例1小麦中国地方品种抗穗发芽候选snp位点的定位及分子标记的获得
1、收集来自中国十大麦区的地方品种小麦,分别于2012年种植于四川农业大学雅安农场,2013年种植于四川农业大学温江实验基地和崇州现代化产业基地,2014年种植于四川农业大学温江实验基地和2015年种植于四川农业大学崇州现代化产业基地。
2、穗发芽鉴定
收获小麦蜡熟期种子,将其悬挂于阴凉通风处自然风干7d后,进行人工脱粒。在有盖塑料培养皿中铺上用3-5ml蒸馏水润湿的滤纸进行发芽试验,试验设计三个重复,每个重复50粒种子。每隔一天挑出已露白和已发霉的种子,并分别计数,种子发芽率以第7天发芽种子数占总种子数百分比表示,计算发芽率。
发芽率(%)=发芽种子数/种子总数-发霉数×100%
3、基因型分析
a)基因组dna提取:采用ctab法提取中国地方品种基因组总dna。检测其浓度,并稀释至50-100ng/ul。
b)wheat660snp扫描
①所有材料送至博奥公司affymetrix平台,并进行genomicdna质量检测,用wheat660snparray进行全基因组扫描。
②wheat660snparray原始数据分析:以上述步骤共获得630517个snp探针,通过目前发表的小麦参考基因组图谱iwgsc2.28,锚定到单一位点的共312,831个snp标记,其中a基因组标记122,338个,b基因组标记137,494个,d基因组标记52,999个。
③自然群体标记质量控制:通过筛选在272份中国地方品种中有差异、缺失率小于20%且最小等位基因频率(maf)大于5%标记共178,803个。
④自然群体分层计算:所选178,803个高质量snp分子标记,利用生物信息学软件structure2.34评估地方品种群体分层,选择最佳群体结果k=5,获得q值。
⑤自然群体亲缘关系评估:所选178,803个高质量snp分子标记,利用生物信息学软件kinship评估地方品种群亲缘关系,获得k值。
⑥穗发芽全基因组关联分析:所选178,803个高质量snp分子标记,以及前面得到的q和k值,加上6个不同环境下的穗发芽表型,利用生物信息学软件tassel5.0进行全基因组关联分析(gwas),鉴定到3个稳定与穗发芽表型强选择相关的分子标记ax-111578083、ax-111624595和ax-110772653(图1)。分别对三个snp标记的不同类型所对应的发芽率进行方差分析,鉴定到等位位点ax-111578083-g,ax-111624595-c,ax-110772653-t为抗穗发芽优异等位位点。
4、kasp标记的开发
为了方便对小麦抗穗发芽候选材料进行3个snp标记ax-111578083、ax-111624595和ax-110772653的鉴定和辅助选择,同时为了降低育种成本和工作量,增强育种工作中的可操作性,需要将与目标分子标记探针序列开发为基于常规分子生物学手段可用于鉴定和筛选的kasp标记。我们分别下载或延长每个snp标记前后至少35bp碱基序列,设计两条正向引物(al1、al2)和一条共用反向引物c。
③引物检测:
以22份中国地方品种小麦基因组dna为模板,利用seqidno:4-12所示的引物对模板进行pcr扩增。
a.pcr扩增体系如下所示:
引物混合液中正向引物al1和正向引物al2的终浓度均为10μm,反向引物c的终浓度为10μm。
b.pcr反应程序如下所示:
c.pcr产物检测使用bio-radicyclerthermalcyclerw/iq5opticalmoduleforrt-pcr(bio-radiq5实时荧光定量pcr仪)在37℃进行荧光收集,利用生物信息学软件bioradcfxmanager对pcr产物进行分型,并与已知基因型进行对照。
实施例2本发明分子标记在选择抗穗发芽候选位点中的应用
1、收集来自中国各麦区主推品种共138份,于2015-2016年种植于四川农业大学崇州现代化农业基地,并在蜡熟期收种进行发芽率鉴定。
2、对收获的主推品种进行kasp标记检测,具体方法为:于三叶期提取叶片基因组总dna;以138份主推品种和16份已知基因型地方品种(对照)的基因组dna为底物,用开发的kasp标记引物分别进行pcr扩增,所述引物为:
ax-111578083:
正向引物al1:5’–3’gaaggtgaccaagttcatgctgatcggtagcttccccca(seqidno:4)
正向引物al2:5’–3’gaaggtcggagtcaacggattgatcggtagcttcccccg(seqidno:5)
反向引物c:5’–3’gtgatccctggtccaatc(seqidno:6)
ax-111624595:
正向引物al1:5’–3’gaaggtgaccaagttcatgctggcctatgaaccaacgaattc(seqidno:7)
正向引物al2:5’–3’gaaggtcggagtcaacggattggcctatgaaccaacgaattt(seqidno:8)
反向引物c:5’–3’cagtcgtggagacatgtgtcac(seqidno:9)
ax-110772653:
正向引物al1:5’–3’gaaggtgaccaagttcatgctcaaccgcgcctcttggaga(seqidno:10)
正向引物al2:5’–3’gaaggtcggagtcaacggattcaaccgcgcctcttggagt(seqidno:11)
反向引物c:5’–3’cgagttcttcccctcatct(seqidno:12)
3、pcr产物检测使用bio-radicyclerthermalcyclerw/iq5opticalmoduleforrt-pcr(bio-radiq5实时荧光定量pcr仪)在37℃进行荧光收集,利用生物信息学软件bioradcfxmanager对pcr产物进行分型,并与已知基因型进行对照。
4、基因分型结果
①用ax-111578083引物进行扩增时,有129份主推品种扩增出了等位位点al1(allelea),仅有7份材料(5.15%)扩增出了优异等位位点al2(alleleg),2份材料缺失。通过方差分析对比两个等位位点的主推品种的发芽率在0.05水平上差异显著(p值=0.0135),优异等位al2(alleleg)所对应主推品种平均发芽率为0.5350,等位位点al1(allelea)所对应主推品种平均发芽率为0.7848(图2)。
②用ax-111624595引物进行扩增时,有127份主推品种扩增出了等位位点al2(allelet),仅有9份材料(6.62%)扩增出了优异等位位点al1(allelec),2份材料缺失。通过方差分析对比两个等位位点的主推品种的发芽率在0.001水平上差异极显著(p值=1.1e-09),优异等位al1(allelec)所对应主推品种平均发芽率为0.2986,等位位点al2(allelet)所对应主推品种平均发芽率为0.8023(图3)。
③用ax-110772653引物进行扩增时,有121份主推品种扩增出了等位位点al1(allelea),仅有15份材料(11.03%)扩增出了优异等位位点al2(allelet),2份材料缺失。通过方差分析对比两个等位位点的主推品种的发芽率在0.001水平上差异极显著(p值=5.3e-08),优异等位al2(allelet)所对应主推品种平均发芽率为0.4518,等位位点al1(allelea)所对应主推品种平均发芽率为0.8108(图4)。
5、利用3个kasp标记(ax-111578083、ax-111624595和ax-110772653)在138份中国主推品种中,对抗穗发芽候选位点进行分型,结果见图5。结果表明:
①从中国地方品种里鉴定到的抗穗发芽优异等位位点,在中国主推品种里同样适用,实验结果与预期一致。说明本发明的抗穗发芽候选标记确实具有抗穗发芽的作用。
②从中国地方品种里鉴定到的抗穗发芽优异等位位点,在中国主推品种里的频率很低(5.15%-11.03%)。说明本发明的抗穗发芽候选位点标记适应用于中国主推栽培品种中。
③本发明的3个kasp分子标记,序列、引物明确,染色体位置清晰,高效、准确,经案例实施,可直接用于分子标记辅助选择育种中。
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之做一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110>四川农业大学
<120>与小麦抗穗发芽性状相关的kasp分子标记及其应用
<130>khp171113057.6
<160>12
<170>patentinversion3.3
<210>1
<211>71
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(36)..(36)
<213>n=a或g
<400>1
cgtcacatggagcatcaggatcggtagcttcccccntgaggattggaccagggatcacca60
cataagatcca71
<210>2
<211>110
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(36)..(36)
<213>n=c或t
<400>2
tccaccgtagtgggaggcctatgaaccaacgaattngcggaggagccaacaaaaggagac60
cgcacgacagatctgcccgaagtgacacatgtctccacgactggcgtgta110
<210>3
<211>71
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(36)..(36)
<213>n=a或t
<400>3
gtggtgtggaactgcctcaaccgcgcctcttggagntgtgcctcgctggacccgagttct60
tcccctcatct71
<210>4
<211>39
<212>dna
<213>人工序列
<400>4
gaaggtgaccaagttcatgctgatcggtagcttccccca39
<210>5
<211>39
<212>dna
<213>人工序列
<400>5
gaaggtcggagtcaacggattgatcggtagcttcccccg39
<210>6
<211>18
<212>dna
<213>人工序列
<400>6
gtgatccctggtccaatc18
<210>7
<211>42
<212>dna
<213>人工序列
<400>7
gaaggtgaccaagttcatgctggcctatgaaccaacgaattc42
<210>8
<211>42
<212>dna
<213>人工序列
<400>8
gaaggtcggagtcaacggattggcctatgaaccaacgaattt42
<210>9
<211>22
<212>dna
<213>人工序列
<400>9
cagtcgtggagacatgtgtcac22
<210>10
<211>40
<212>dna
<213>人工序列
<400>10
gaaggtgaccaagttcatgctcaaccgcgcctcttggaga40
<210>11
<211>40
<212>dna
<213>人工序列
<400>11
gaaggtcggagtcaacggattcaaccgcgcctcttggagt40
<210>12
<211>19
<212>dna
<213>人工序列
<400>12
cgagttcttcccctcatct19
- 还没有人留言评论。精彩留言会获得点赞!