一种基于猪SINE转座子插入多态性研发新型分子标记的方法与流程
本发明涉及分子生物学和生物信息学领域,具体涉及一种基于猪sine转座子插入多态性研发新型分子标记的方法。
背景技术:
dna分子标记技术是一种以基因组dna多态性为基础的新型遗传标记技术,是遗传变异在dna水平上的直接反映。相对于形态学标记、生物化学标记、细胞学标记,dna分子标记具有的优越性有:大多数分子标记为共显性,对隐性的性状的选择十分便利;基因组变异极其丰富,分子标记的数量几乎是无限的;在生物发育的不同阶段,不同组织的dna都可用于标记分析;分子标记揭示来自dna的变异;表现为中性,不影响目标性状的表达,与不良性状无连锁;检测手段简单、迅速。
dna分子标记技术记过历年来的发展,主要有以下几种标记,第一代分子标记技术:限制性片段长度多态性(rflp)标记技术,优点:rflp标记的等位基因具有共显性的特点,结果稳定可靠,重复性好,特别适应于构建遗传连锁图。缺点:在进行rflp分析时,需要该位点的dna片段做探针,用放射性同位素及核酸杂交技术,既不安全又不易自动化。另外,rflp对dna多态性检出的灵敏度不高,rflp连锁图上还有很多大的空间区。随机扩增多态dna标记技术(rapd),优点:与rflp相比,rapd技术简单,dna用量少,试验设备简单,不需dna探针,设计引物简单,而且不需要同位素,安全性好。缺点:rapd技术受许多因素影响,实验的稳定性和重复性差,首先是显性遗传,不能识别杂合子位点,这事的遗传分析相对复杂,在基因定位,做连锁遗传图时,会因显性遮盖作用而使计算位点间遗传距离的准确性下降;其次,rapd对反应条件相当敏感,包括模板浓度,mg2+浓度,所以实验的重复性差。扩增片段长度多态技术(aflp),优点:它兼具rapd与rflp的优点,有较高的稳定性,用少量的选择性引物能在较短时间内监测到大量位点,各染色体上aflp标记的数目与染色体长度呈正相关,通过少量效率高的引物组合,可获得覆盖整个基因组的aflp标记。缺点:aflp对基因组纯度和反应条件较高,容易受dna污染,操作技术难度大。用于遗传作图时,少数的标记与图谱紧密度有出入。第二代分子标记技术,微卫星标记技术ssr,优点:具有很高的多态性,而且一般为共显性。缺点:工作量大,费用也高,实际应用少。第三代分子标记:单核苷酸多态性(snp)优点:具有共显性,能获得的标记数量多,易于实现高通量。是基因组中最简单。最常见的多态性形式,具有很高的遗传稳定性。缺点:芯片成本高,由于dna样品的复杂性,有些snp不能被捡起。表达序列标签(est)优点:成本低,操作简单。有助于人们从分子水平理解物种的多样性,并可利用其中感兴趣的序列信息。缺点:所获基因组信息不全,如不能体现出内含子,调控序列等在基因表达调控中重要的信息。
转座子是一类在基因组上可以自由跳跃(复制或移位)的移动dna序列,广泛存在于生物基因组中。由于转座子能在其寄主的基因组中复制、移动,因此,能在基因组上产生转座子插入多态性(transposoninsertionpolymorphisims,tip),对物种、品种、亚种、品系形成发挥了重要作用。利用转座子插入多态性(tip)建立的新型分子标记技术在遗传进化研究中的应用为我们理解高等生物基因组的进化提供了新视角。
猪基因组学研究目前面临的主要挑战是虽然获得了大量数量性状变异的qtl,但缺乏足够密度的分子标记,难以进行基因精细定位,无法有效开展分子标记辅助选择。而tip具有理想分子标记的诸多优点:如多态性高、共显性、基因组分布广泛、检测手段简单快速、重复性好和开发成本低等。并且sine具有一定长度,检测难度小于snp,微卫星等,并且在猪基因组中分布广泛,通过本专利的方法设计的sine标记,可以简便快捷的检测插入多态性,并且可以确定是纯合还是杂合状态。同时sine转座子插入多态研发的分子标记必将大大推动分子育种的进程和猪功能基因学的研究。
技术实现要素:
本发明是针对现有分子标记的不足,基于猪sine转座子在猪基因组上具有含量丰富,分布广泛,并且在不同的个体间存在插入多态性的特点提供一种研发分子标记的方法。
本发明通过以下技术方案实现:
一种基于猪sine转座子的新型分子标记的获取方式,包括以下步骤:
(1)利用sine的核苷酸序列作为查询序列,在生物学公用数据库中对猪公开基因组进行检索,寻找插入位点;
(2)针对寻找到的sine的每个插入位点,分别向上游和下游延伸300-500个核苷酸序列,然后分别下载每条序列;
(3)将步骤(2)获得的序列去除冗余;
(4)根据步骤(3)获得的序列信息设计检测引物,作为待验证分子标记引物,其中使一侧引物位于sine插入位点的5’端基因组侧翼序列中,另一侧引物位于sine的3’端基因组侧翼序列中;
(5)利用步骤(4)所得的待验证分子标记引物pcr扩增不同品种,或同一品种不同个体基因组样品,选取能清晰扩出条带且有多态性的引物组合,获得分子标记。
其中,sine家族的成员序列有差异,可以用任何一个作为查询序列,获得的位点都属于sine转座子的插入位点。所述sine的核苷酸序列包括但不限于seqidno.1、seqidno.2、seqidno.3、seqidno.4、seqidno.5、seqidno.6、seqidno.7、seqidno.8、seqidno.9、seqidno.10、seqidno.11、seqidno.12、seqidno.13、seqidno.14、seqidno.15、seqidno.16或seqidno.17。
上述方法的一个实例是:
(1)利用sine的核苷酸序列seqidno.1作为查询序列,在ensembl数据和ucsc数据库对猪公开的考基因组进行比对(blat)搜索,寻找sine在基因组上的插入位点;
(2)在各插入位点分别向上游和下游延伸300核苷酸,然后选择比对上的长度大于95%,相似度大于98%的位点,并分别下载每条序列;
(3)将获得的序列利用cd-hit程序按照相似性95%进行合并,去除冗余,最终获得78条序列。
(4)截取由sine5’端基因组侧翼序列、自身序列及3’端基因组侧翼序列组成的序列作为设计引物的模板,根据序列设计检测引物,其中使一侧引物位于sine插入位点的5’端基因组侧翼序列中,另一侧引物位于sine的3’端基因组侧翼序列中,最后获得78对待验证分子标记引物。
(5)利用待验证分子标记引物pcr扩增不同品种,或同一品种不同个体基因组样品,选取能清晰扩出条带且有多态性的引物组合,获得分子标记,共获得44个标记,对应的引物名称,引物组合,引物序列如表1所示。
其中,步骤(5)中pcr扩增的操作可以是包括以下步骤:
1)人工合成上述44对待验证分子标记引物;
2)提取不同品种的池dna以及不同品种的不同个体基因组;
3)以不同品种的池dna以及不同品种的不同个体基因组为模板进行pcr扩增;
4)对pcr扩增结果进行琼脂糖凝胶电泳检测。
本发明主要通过获取sine插入位点序列,设计各位点检测引物,pcr扩增,琼脂糖凝胶电泳等步骤获得转座子插入情况,据此建立基于转座子插入多态性检测方法。为猪的品系鉴定,育种中的标记辅助选择提供有用的分子标记。
附图说明
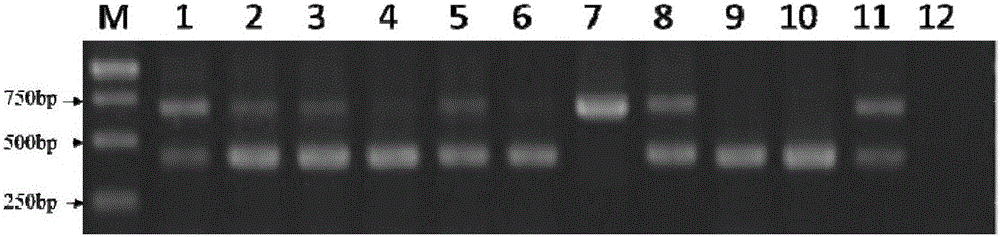
图1标记sine-tip-5在11个不同品种中的扩增结果。marker为宝生物的dl2000,从左到右1-12分别是洛克猪、长白猪、大白猪、梅山猪、姜曲海猪、苏姜猪、藏猪、野猪、巴马香猪、皖南花猪、烟台黑猪混合基因组及空白对照,混合基因组均为5个个体的等质量混合基因组。图中1号箭头所指表示此标记位置上有sine的插入,2号箭头所指表示此标记位置上一条染色体有sine的插入,另一条染色体有sine的插入。3号箭头所指表示此标记位置上没有sine的插入,4号箭头所指表示此标记位置片段缺失,其它图表示意义一致。
图2标记sine-tip-31在11个不同品种中的扩增结果。marker为宝生物的dl2000,从左到右1-12分别是洛克猪、长白猪、大白猪、梅山猪、姜曲海猪、苏姜猪、藏猪、野猪、巴马香猪、皖南花猪、烟台黑猪混合基因组及空白对照,混合基因组均为5个个体的等质量混合基因组。
图3标记sine-tip-6在9个不同品种的不同个体中的检测结果。marker为宝生物的dl15000,从左到右分别是野猪、大白、杜洛克、梅山、二花脸、藏猪、荣昌、巴马香猪、苏姜猪,其中藏猪3头,其余品种均为5头。
图4标记sine-tip-7在9个不同品种的不同个体中的检测结果。marker为宝生物的dl15000,从左到右分别是野猪、大白、杜洛克、梅山、二花脸、藏猪、荣昌、巴马香猪、苏姜猪,其中藏猪3头,其余品种均为5头。
具体实施方式
在进一步描述本发明具体实施方式之前,应理解,本发明的保护范围不局限于下述特定的具体实施方案;还应当理解,本发明实施例中使用的术语是为了描述特定的具体实施方案,而不是为了限制本发明的保护范围。
一、参考基因组中line1插入位点序列获取,具体过程如下:
1、sine转座子在基因组中插入位点检索:利用sine核苷酸序列seqidno.1作为查询序列,在ensembl数据库(http://www.ensembl.org/)和ucsc数据库(http://genome.ucsc.edu/)中利用blat程序对猪的参考基因组(sgcgsscrofa10.2/susscr3)进行比对搜索,运行参数使用数据库中各自的默认设置,寻找line1在基因组上的插入位点。
2、在两个数据库中每个sine插入位点序列获取:在ensembl数据库中检索插入位点后,向比对结果中比对上的长度大于95%,相似度大于98%的每个插入位点的5’端上游和3’下游各延伸300核苷酸,然后下载每条序列,同理在ucsc数据库中进行再次检索,剔除与ensembl数据库中一致的位点,然后向每个插入位点的5’端上游和3’端下游各延伸300核苷酸,下载每条序列,最后合并两个数据库下载的序列。
3、将获得的序列利用cd-hit-est程序
(http://weizhongli-lab.org/cdhit_suite/cgi-bin/index.cgi?cmd=cd-hit-est)将sequenceidentitycut-off参数设置为0.95,其他参数使用默认值进行去冗余。
二、引物的设计
利用获得的序列,使用batchprimer3
(http://batchprimer3.bioinformatics.ucdavis.edu/cgi-bin/batchprimer3/batchprimer3.cgi)在线软件进行引物设计,使上游引物位于sine插入位点的5’端的侧翼序列,下游引物位于sine的3’端侧翼序列中。产物长度设置为500-700bp,其他参数使用软件默认设置。设计好的各引物用olige7进行分析评价,然后由北京六合华大基因科技股份有限公司合成。
三、分子标记在11个不同品种中验证
1、基因组的准备:
(1)用takaraminibestuniversalgenomicdnaextractionkitver.5.0提取杜洛克猪、长白猪、大白猪、梅山猪、姜曲海猪、苏姜猪、藏猪、野猪、巴马香猪、皖南花猪、烟台黑猪各5个个体基因组,主要步骤如下:
a)取2-25mg的耳组织,剪碎,加入180微升的buffergl、20微升的proteinasek和10微升rnasea(10mg/ml),56℃水浴至组织完全裂解。b)向裂解液中加入200微升buffergb和200微升100%乙醇,充分混匀。c)将spincolumn安置于collectiontube上,溶液移至spincolumn中,12000rpm离心2分钟,弃滤液。d)向spincolumn中加入500微升bufferwa,12000rpm离心1分钟,弃滤液。e)将700微升的bufferwb加入至spincolumn中,12000rpm离心1分钟,弃滤液。f)重复操作步骤6。g)将spincolumn安置于collectiontube上,12000rpm离心2分钟。h)将spincolumn安置于新的1.5ml的离心管上,在spincolumn膜的中央处加入50-200微升的灭菌水或elutionbuffer,室温静置5分钟。i)12000rpm离心2分钟洗脱dna.(2)调整各个基因组浓度成为40ng/μl,取每个个体基因组50μl混合为品种池dna,共11种混合基因组,以此作为模板进行pcr扩增。
2、pcr扩增
设计以下pcr反应体系:
(1)在灭菌pcr管中配制20μl反应体系
(2)将pcr管置于pcr仪中,进行如下反应程序
注:1:南京诺唯赞生物科技有限公司
3、琼脂糖凝胶检测
(1)制作1.5%的琼脂糖凝胶:称取1.5g琼脂糖放入三角烧瓶中,并向烧瓶中加入100ml1*tae溶液,放于微波炉中加热使之完全溶解,待凝胶冷却至约60℃时,将琼脂糖胶倒入插有胶梳的模具中,厚度一般在4-5mm,并检查有无气泡存在。室温放置30min待凝胶完全凝固后,小心拔出胶梳,浸没于加有1*tae电泳缓冲液的电泳槽中。
(2)pcr产物电泳:用移液器吸取6μlpcr扩增产物依次加入到凝胶孔中,同时点4μldnamaker作为参照,120v恒压电泳,待指示剂到达胶块的2/3处停止电泳,然后将凝胶放到溴化乙锭溶液中,染色10分钟,将凝胶拿出放到紫外灯下观察并拍照记录。
四、分子标记在9个不同品种中验证
1、基因组的准备:
(1)用takaraminibestuniversalgenomicdnaextractionkitver.5.0提取野猪、大白、杜洛克、梅山、二花脸、藏猪、荣昌、巴马香猪、苏姜猪9个品种的个体基因组,主要步骤如下:
a)取2-25mg的耳组织,剪碎,加入180微升的buffergl、20微升的proteinasek和10微升rnasea(10mg/ml),56℃水浴至组织完全裂解。b)向裂解液中加入200微升buffergb和200微升100%乙醇,充分混匀。c)将spincolumn安置于collectiontube上,溶液移至spincolumn中,12000rpm离心2分钟,弃滤液。d)向spincolumn中加入500微升bufferwa,12000rpm离心1分钟,弃滤液。e)将700微升的bufferwb加入至spincolumn中,12000rpm离心1分钟,弃滤液。f)重复操作步骤6。g)将spincolumn安置于collectiontube上,12000rpm离心2分钟。h)将spincolumn安置于新的1.5ml的离心管上,在spincolumn膜的中央处加入50-200微升的灭菌水或elutionbuffer,室温静置5分钟。i)12000rpm离心2分钟洗脱dna.(2)调整各个基因组浓度成为40ng/μl,以此作为模板进行pcr扩增。
2、pcr扩增
设计以下pcr反应体系:
(1)在灭菌pcr管中配制20μl反应体系
(2)将pcr管置于pcr仪中,进行如下反应程序
注:1:南京诺唯赞生物科技有限公司
3、琼脂糖凝胶检测
(1)制作1.5%的琼脂糖凝胶:称取1.5g琼脂糖放入三角烧瓶中,并向烧瓶中加入100ml1*tae溶液,放于微波炉中加热使之完全溶解,待凝胶冷却至约60℃时,将琼脂糖胶倒入插有胶梳的模具中,厚度一般在4-5mm,并检查有无气泡存在。室温放置30min待凝胶完全凝固后,小心拔出胶梳,浸没于加有1*tae电泳缓冲液的电泳槽中。
(2)pcr产物电泳:用移液器吸取6μlpcr扩增产物依次加入到凝胶孔中,同时点4μldnamaker作为参照,120v恒压电泳,待指示剂到达胶块的2/3处停止电泳,然后将凝胶放到溴化乙锭溶液中,染色10分钟,将凝胶拿出放到紫外灯下观察并拍照记录。
五、结果分析
部分检测结果如图1,图2,图3,图4所示。图1,图2所示基于sine插入多态性的分子标记在不同品种间存在良好的多态性,可以为品种鉴定提供鉴定方法,图3,图4所示基于sine插入多态性的分子标记在不同个体间存在良好的多态,可以作为个体鉴定,分子辅助育种的良好标记。经过两轮实验验证,获得一系列基于sine插入多态性的分子标记。六、结论
经过两轮实验验证,获得表1所示的44个基于sine插入多态性的分子标记,能够在品种内或品种间产生清晰且具有多态的检测结果,可以为品种鉴定提供鉴定方法,可以作为个体鉴定,分子辅助育种的良好标记,具有检测成本低,重复性好,结果清晰等优点;且为共显性,对隐性的性状的选择十分便利;在生物发育的不同阶段,不同组织的dna都可用于标记分析;不影响目标性状的表达,与不良性状无连锁;检测手段简单、迅速。
表1sine转座子插入多态性标记检测引物列表
sequencelisting
<110>扬州大学
<120>一种基于猪sine转座子插入多态性研发新型分子标记的方法
<130>
<160>85
<170>patentinversion3.3
<210>1
<211>263
<212>dna
<213>猪(susscrofa)
<400>1
ggagttcccgtcgtggcgcagtggttaacgaatccgactaggaaccatgaggttgcgggt60
tcggtccctgcccttgctcagtgggttaacgatccggcgttgccgtgagctgtggtgtag120
gttgcagacgcggctcggatcccgcgttgctgtggctctggcgtaggccggcggctacag180
ctccgattcgacccctagcctgggaacctccatatgccgcgggagcggcccaagaaatag240
caaaaagacaaaaaaaaaaaaaa263
<210>2
<211>259
<212>dna
<213>猪(susscrofa)
<400>2
ggagttcccgtcgtggcgcagcggaaacgaatccgactaggaaccatgaggttgcgggtt60
cgatccctggcctcgctcagtgggttaaggatccggcgttgccgtgagctgtggtgtagg120
tcgcagacgcggctcggatccsgcgttgctgtggctgtggcgtaggccggcagctgcagc180
tccgattmgacccctagcctgggaacctccatatgccgcgggtgcggccctaaaaagaca240
aaaaacaaaaaaaaaaaaa259
<210>3
<211>255
<212>dna
<213>猪(susscrofa)
<400>3
ggagttcccgctgtggctcagcgggttaagracccgacgtwgtstccgtgaggatgcggg60
ttcgatccctggcctcgctcagtgggttaaggatccggcgttgccgcaagctgcggcgta120
ggtcgcagatgcggctcggatccggcgttgccgtggctgtggcgtaggccggcagctgca180
gctccgattcgacccctagcccgggaacttccatatgccgcaggtgcggccntaaaaaga240
aaaaaaaaaaaaaaa255
<210>4
<211>254
<212>dna
<213>猪(susscrofa)
<400>4
ggagttcccgtcgtggcgcagtggttaacgaatccgactaggaaccacgaggttgcgggt60
tcgatccctggcctcgctcagtgggttaaggatccggcgttgccgtgagctgtggtgtag120
gtcgcagacgcggctcggatcccgcgttgctgtggctctggcgtaggccggcggctacag180
ctccgattngacccctagcctgggaacctccntatgccncaggngcggccctagaaaagg240
caaaaaaaaaaaaa254
<210>5
<211>216
<212>dna
<213>猪(susscrofa)
<400>5
ggagttcccgtcgtggcgcagcggaaacgaatccgactaggaaccatgaggttgcgggtt60
cgatccctggcctcgctcagtgggttaaggatccggcgttgccgtgagctgtggtgtagg120
ccggcggctgcagctccgattngacccctagcctgggaacctccatatgccgcgggtgcg180
gccctaaaaaaggcaaaaaaaaaaaaaaaaaaaaaa216
<210>6
<211>216
<212>dna
<213>猪(susscrofa)
<400>6
ggagttcccgtcgtggctcagcggttaacgaacccgactagtatccatgaggacgcgggt60
tcgatccctggcctcgctcagtgggttaaggatccggcgttgctgtggctgtggcgtagg120
ccggcagctgcagctccgattcgacccctagcctgggaacttccatatgccgcgggtgcg180
gccctaaaaaaggcaaaaaaanaaaaaaaaaaaaaa216
<210>7
<211>202
<212>dna
<213>猪(susscrofa)
<400>7
ggagttcccgctgtggcgcaacgggatcggcggcgtctcgggagcgccgggacgcaggtt60
cgatccccggcccggcacagtgggttaaggatccggcgttgccgcagctgcggcktaggt120
cgcaactgcggctcggatctgatccctggcccgggaactccatatgccgcggggcggcca180
aaaaagaaaaaaaaaaaaaaaa202
<210>8
<211>225
<212>dna
<213>猪(susscrofa)
<400>8
ggagttcccgctgtggcgcagtgggttaaggatccgactgcagcggctcgggtcgctgcg60
gaggcgcgggttcgatccccggcccggcgcagtgggttaaaggatccggcgttgccgcag120
ctgtggcgtaggtcgcagctgcggctcggattcaatccctggcccgggaacttccatatg180
ccgcgggtgcggccaaaaaaagaaaaaaaanaaaaaaaaaaaaaa225
<210>9
<211>221
<212>dna
<213>猪(susscrofa)
<400>9
gggagttcccgccgtggcgcagtgggttaagaatccgactgcagcggctcgggtcgctgc60
ggaggcgcgggttcgatccccggcccggcgcagtgggttaaaggatccggcgttgctgca120
gctgcggcgtaggtcgcagctgcggctcggattcgatccctggcccgggaacttccatat180
gccgcgggtgcggccgtaaaaagaaaaaaaaaaaaaatcgt221
<210>10
<211>131
<212>dna
<213>猪(susscrofa)
<400>10
gggagttctcttgtggcacagcaggttaaggatccagcgttgtcactgcagtggcttggg60
tcgctgctgtggcacgggttcaatccctggcccaggaacttccacatgccacgggcatgg120
ccaaaaaaaaa131
<210>11
<211>119
<212>dna
<213>猪(susscrofa)
<400>11
ggagttccctggtggctcagtgggttaaggatccggcattgtcactgctgtggctcgggt60
tcgatccctggcccaggaacttctgcatgccgtgggcgcggccaaaaaaaaaaaaaaaa119
<210>12
<211>134
<212>dna
<213>猪(susscrofa)
<400>12
ggagttccctggtggctcagcgggttaaggatccggcgttgtcactgctgtggctctggt60
tacngctgtggcacgggttcaatccctggcccgggaacttccgcatgccacgggcgcggc120
caaaaaaaaaaaaa134
<210>13
<211>127
<212>dna
<213>猪(susscrofa)
<400>13
ggagttcccttgtggcgcagcgggttaaggatccggcgttgtcactgcagcggctcgggt60
cgctgctgtggcgcgggtttgatccctggcccgggaacttccacatgctgtgggcgcggc120
caaaaaa127
<210>14
<211>131
<212>dna
<213>猪(susscrofa)
<400>14
ggagttccctggtggctcagygggttaaggatctagtgttgtcactgctgtggctcrggt60
cactgctgtggtgcaggtttgatccctggcctgggaacttccacatgcygyrggcatggc120
caaaaaaaaaa131
<210>15
<211>123
<212>dna
<213>猪(susscrofa)
<400>15
ggacttcccgtgtggctcacagcataactactcggcgttgcttctgcagtggctgtggct60
cgacccctggcccgggaacttccacatgccgcgggcgtggccaaaaaaaaaaaaaaaaaa120
aaa123
<210>16
<211>137
<212>dna
<213>猪(susscrofa)
<400>16
ggagttccctggtggtctagtggttaggatccggcgctttcactgctgcggcccgggttc60
aatcccctggtctgggaactgggatcccacatcaagccactgcangcgtggccaaaaaca120
aacaaacaaacaaaaaa137
<210>17
<211>155
<212>dna
<213>猪(susscrofa)
<400>17
gggagttctctgatggcctagcgggttgaggctcctgcgttctcaccgctgtggctctgg60
ttgctgctgtgcggcgtaggttcaatccctggcccaggaattcccacatactgcctgtgt120
ggcaaaaaagaaaaaaaaaaaaaatacaaaaaaaa155
<210>18
<211>20
<212>dna
<213>猪(susscrofa)
<400>18
gaaaaatccaggcaaggaca20
<210>19
<211>24
<212>dna
<213>猪(susscrofa)
<400>19
ccacttaggataagaggtcctcaa24
<210>20
<211>20
<212>dna
<213>猪(susscrofa)
<400>20
accagctgggattttgattg20
<210>21
<211>20
<212>dna
<213>猪(susscrofa)
<400>21
gccctgaatgacagacacag20
<210>22
<211>20
<212>dna
<213>猪(susscrofa)
<400>22
tgtgcatgtcaaatgtggtc20
<210>23
<211>20
<212>dna
<213>猪(susscrofa)
<400>23
ctacacaacccccagttggt20
<210>24
<211>20
<212>dna
<213>人工序列
<400>24
caagttccccatgtgctttc20
<210>25
<211>20
<212>dna
<213>人工序列
<400>25
gcccaacaaaagatgaatgg20
<210>26
<211>20
<212>dna
<213>人工序列
<400>26
cattctaacaaccgccaagg20
<210>27
<211>20
<212>dna
<213>人工序列
<400>27
aggtgcagccttaaaaagca20
<210>28
<211>20
<212>dna
<213>人工序列
<400>28
tgagtgtcatccctgcatgt20
<210>29
<211>22
<212>dna
<213>人工序列
<400>29
cagcaacagagaaatcatagcc22
<210>30
<211>21
<212>dna
<213>人工序列
<400>30
gaatgctgttacacaggcaca21
<210>31
<211>21
<212>dna
<213>人工序列
<400>31
ccccacatggtttactttgag21
<210>32
<211>19
<212>dna
<213>人工序列
<400>32
aagcaaggcagagtgagca19
<210>33
<211>20
<212>dna
<213>人工序列
<400>33
gggaaaccatcccatttacc20
<210>34
<211>20
<212>dna
<213>人工序列
<400>34
ccacgctgtggctctaaaat20
<210>35
<211>20
<212>dna
<213>人工序列
<400>35
ctacctctccccaccagtga20
<210>36
<211>20
<212>dna
<213>人工序列
<400>36
cagccacaccatggtttcta20
<210>37
<211>20
<212>dna
<213>人工序列
<400>37
acccacatcaaggaaggtca20
<210>38
<211>20
<212>dna
<213>人工序列
<400>38
ggacacccaattcccctaat20
<210>39
<211>20
<212>dna
<213>人工序列
<400>39
cttttgggggaagggataaa20
<210>40
<211>20
<212>dna
<213>人工序列
<400>40
attaccgctgagccactagg20
<210>41
<211>20
<212>dna
<213>人工序列
<400>41
tttacagcaacacggatgga20
<210>42
<211>19
<212>dna
<213>人工序列
<400>42
ggagctcctgggggtaaat19
<210>43
<211>20
<212>dna
<213>人工序列
<400>43
tgagggaactcaggaaggaa20
<210>44
<211>20
<212>dna
<213>人工序列
<400>44
catggtgtttgtcaggagga20
<210>45
<211>24
<212>dna
<213>人工序列
<400>45
tggtgttcaatgagatgacttagg24
<210>46
<211>20
<212>dna
<213>人工序列
<400>46
tggtgacgtgtccttcaaga20
<210>47
<211>20
<212>dna
<213>人工序列
<400>47
caggaacacagtcctggtca20
<210>48
<211>20
<212>dna
<213>人工序列
<400>48
gggagtcagaaaaaggcaga20
<210>49
<211>20
<212>dna
<213>人工序列
<400>49
ccattctcaaagggcattta20
<210>50
<211>20
<212>dna
<213>人工序列
<400>50
gggtctatggatgcacgttt20
<210>51
<211>20
<212>dna
<213>人工序列
<400>51
tggccagcattatcgtaaca20
<210>52
<211>20
<212>dna
<213>人工序列
<400>52
ccagggaacagtttgctctt20
<210>53
<211>20
<212>dna
<213>人工序列
<400>53
gccgttcttgcatgcttatt20
<210>54
<211>21
<212>dna
<213>人工序列
<400>54
caaaggaaagctgaagccata21
<210>55
<211>20
<212>dna
<213>人工序列
<400>55
tgaacattacgagcggatga20
<210>56
<211>21
<212>dna
<213>人工序列
<400>56
ccgtaggagctcataatgtgg21
<210>57
<211>20
<212>dna
<213>人工序列
<400>57
gaaaacggagagcttgatgg20
<210>58
<211>20
<212>dna
<213>人工序列
<400>58
tgctggagggatcaggatag20
<210>59
<211>21
<212>dna
<213>人工序列
<400>59
cttccatcatgttccatcaca21
<210>60
<211>20
<212>dna
<213>人工序列
<400>60
acggttacgccaatgttttc20
<210>61
<211>20
<212>dna
<213>人工序列
<400>61
ccaagtgcccatcaatgaat20
<210>62
<211>20
<212>dna
<213>人工序列
<400>62
gcccaattttccctctcttc20
<210>63
<211>20
<212>dna
<213>人工序列
<400>63
tttaggccctgctcccttat20
<210>64
<211>20
<212>dna
<213>人工序列
<400>64
gaggcagccacagagaagag20
<210>65
<211>25
<212>dna
<213>人工序列
<400>65
ctgagggacatactttaactcattc25
<210>66
<211>20
<212>dna
<213>人工序列
<400>66
acaaacagacgctgagcaga20
<210>67
<211>20
<212>dna
<213>人工序列
<400>67
cagtcagggagctgattgct20
<210>68
<211>20
<212>dna
<213>人工序列
<400>68
cccctccacctcagttacaa20
<210>69
<211>20
<212>dna
<213>人工序列
<400>69
acacatccacaaggtgacga20
<210>70
<211>20
<212>dna
<213>人工序列
<400>70
tcaaacccacatcttcatgg20
<210>71
<211>20
<212>dna
<213>人工序列
<400>71
ccatggtggttagacccaaa20
<210>72
<211>20
<212>dna
<213>人工序列
<400>72
tggtggtctttggggttatc20
<210>73
<211>20
<212>dna
<213>人工序列
<400>73
tgtggcaaaaatccagaaac20
<210>74
<211>20
<212>dna
<213>人工序列
<400>74
agcacagggaaccatatcc20
<210>75
<211>20
<212>dna
<213>人工序列
<400>75
gatgccttaacccactgcac20
<210>76
<211>20
<212>dna
<213>人工序列
<400>76
gcttaaatggcccacaaaga20
<210>77
<211>20
<212>dna
<213>人工序列
<400>77
gcacaagtttgcagacaacc20
<210>78
<211>21
<212>dna
<213>人工序列
<400>78
ggaaagaaagtgcaatccaga21
<210>79
<211>20
<212>dna
<213>人工序列
<400>79
ggagagagaggaggggagaa20
<210>80
<211>20
<212>dna
<213>人工序列
<400>80
gagagggagagggacacaca20
<210>81
<211>20
<212>dna
<213>人工序列
<400>81
tttcttgatctgtgcctcca20
<210>82
<211>20
<212>dna
<213>人工序列
<400>82
cctcctctggttccttctcc20
<210>83
<211>20
<212>dna
<213>人工序列
<400>83
gaggacccatcctagaccaa20
<210>84
<211>20
<212>dna
<213>人工序列
<400>84
ccactgaacatttgccttca20
<210>85
<211>20
<212>dna
<213>人工序列
<400>85
tgggaactctgctcttggtt20
- 还没有人留言评论。精彩留言会获得点赞!