无细胞生成核糖核酸的制作方法
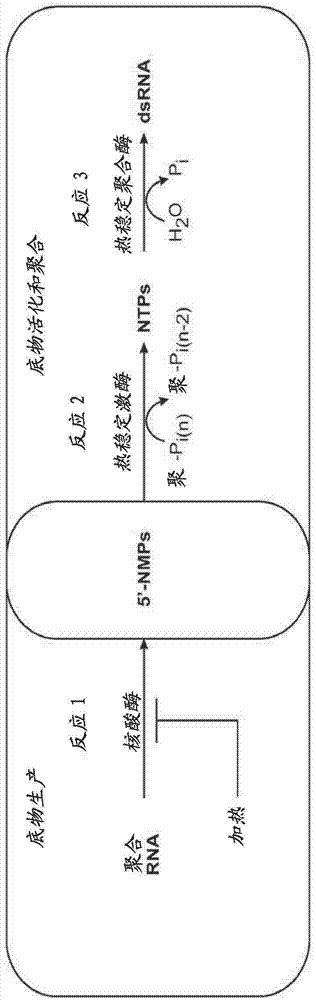
本申请要求2016年4月6日提交的美国临时申请号62/319,220和2017年1月31日提交的美国临时申请号62/452,550的35u.s.c.§119(e)下的权益,其各自通过引用整体并入本文。发明背景核糖核酸(rna)对于生命是普遍存在的。rna充当细胞中信息的关键信使,携带dna的指令用于蛋白质的调节和合成。rna在生物技术中是有意义的,因为合成调节细胞中的mrna水平(通过引入mrna正调节或通过引入sirna或dsrna负调节)在农业作物保护、抗癌治疗剂和疫苗等领域中具有应用。例如,rna干扰(rnai)是指利用基因的dna序列将基因“关闭”(称为“沉默”的过程)的细胞机制。在极其多种生物体(包括动物、植物和真菌)中,rnai由双链rna(dsrna)触发。功能性单链(例如mrna)和双链rna分子已经在活细胞和体外使用纯化的重组酶和纯化的三磷酸核苷酸产生(参见例如欧洲专利号1631675和美国专利申请公开号2014/0271559a1,其中每篇通过引用并入本文)。尽管如此,在能够广泛商业应用的规模上生产rna目前成本过高。发明概述本文提供了用于rna的生产(生物合成)的方法、组合物、细胞、构建体和系统。通常,来自生物量材料的聚合rna酶促解聚成其组成单体,然后这些单体磷酸化为其关联三磷酸化变体(通过一系列激酶),其随后使用相应的核酸(例如dna)模板聚合成聚合rna。在一些实施方案中,本公开的方法、组合物、细胞、构建体和系统用于在无细胞条件下产生rna,例如,使用至少一种细胞裂解物、纯化蛋白质的组合、或细胞裂解物和纯化蛋白质的组合。在一些实施方案中,本公开基于使用细胞裂解物将rna从生物量(例如,内源细胞rna)转化为期望的合成rna(例如,合成的单链或双链rna)。首先,来自生物量的rna(例如,内源rna),例如信使rna(mrna)、转移rna(trna)和/或核糖体rna(rrna)(例如,存在于细胞裂解物中)由一种或多种核酸酶解聚成其单体形式5'-一磷酸核苷(nmp)(图1,反应1)。接下来,这些核酸酶以及天然核酸酶和磷酸酶被失活或部分失活(例如,通过热失活),并且通过一系列热稳定性激酶活性将nmp磷酸化为三磷酸核糖核苷酸(ntp)(图1,反应2)。最后,使用核酸(例如,dna)模板,通过rna聚合酶(例如,热稳定性rna聚合酶)聚合ntp以形成期望的rna(图1,反应3)。期望的合成rna任选地可以从细胞裂解物纯化。因此,本公开的一些方面提供了产生(生物合成)核糖核酸(rna)的无细胞方法,所述方法包括:在导致rna解聚的条件下,温育至少一种包含(i)rna和(ii)至少一种酶活性的细胞裂解物混合物以产生包含一磷酸核苷的细胞裂解物混合物,所述酶活性选自下组:解聚rna的酶活性、热稳定性激酶活性和热稳定性rna聚合酶活性;(b)将步骤(a)中产生的所述细胞裂解物混合物加热至使内源核酸酶和磷酸酶失活或部分失活而不使热稳定性激酶活性和热稳定性rna聚合酶活性完全失活的温度(例如50-80℃),以产生包含热失活的核酸酶和磷酸酶的细胞裂解物混合物;和(c)在能量源(例如,atp再生系统)和脱氧核糖核酸(dna)模板(例如,含有与编码感兴趣rna的核苷酸序列可操作连接的启动子)存在下,在导致三磷酸核苷产生和三磷酸核苷聚合的条件下,温育(b)中产生的所述细胞裂解物混合物以产生包含所述感兴趣rna的细胞裂解物混合物。细胞裂解物混合物可包含从包含rna并表达至少一种酶(包括至少一种融合酶)的细胞获得的单细胞裂解物,所述酶充当核糖核酸酶,充当激酶,和/或充当rna聚合酶。或者,细胞裂解物混合物可包含至少两种(例如,至少3、4、5或6种)细胞裂解物,其中至少一种细胞裂解物获自包含rna的细胞并且至少一种细胞裂解物(例如至少2、3、4或5种)获自表达至少一种充当核酸酶,充当激酶和/或充当rna聚合酶的酶的细胞。若融合酶的酶表现出核酸酶活性(裂解或解聚核酸;例如rna酶r),则认为酶或融合酶“充当核酸酶”。若融合酶的酶表现出激酶活性(催化磷酸基团从一个分子转移到另一个分子;例如多磷酸激酶),则认为酶或融合酶“充当激酶”。若融合酶的酶表现出聚合酶活性(组装核苷酸以产生核酸,例如rna聚合酶),则认为酶或融合酶“充当聚合酶”。在一些实施方案中,步骤(a)的rna是信使rna(mrna)、转移rna(trna)或核糖体rna(rrna)。在一些实施方案中,细胞裂解物混合物包含至少一种核糖核酸酶、至少一种热稳定性激酶和/或至少一种rna聚合酶(例如,热稳定性rna聚合酶)。融合酶的使用也包括在本公开内。例如,细胞裂解物混合物可包含核糖核酸酶和激酶的融合物,或多种激酶的融合物。其他融合酶包括在本公开中。本公开的其他方面提供了工程化细胞、细胞裂解物和细胞裂解物混合物,其包含至少一种一磷酸核苷激酶(例如,热稳定性一磷酸核苷激酶)、至少一种二磷酸核苷激酶(例如,热稳定性二磷酸核苷激酶)和至少一种多磷酸激酶(例如,热稳定性多磷酸激酶)。在一些实施方案中,细胞还可包含至少一种核糖核酸酶和/或至少一种rna聚合酶(例如,热稳定性rna聚合酶)。在一些实施方案中,产生(生物合成)rna的方法包括(a)裂解包含rna(例如,mrna、trna和/或rrna)、rna酶r,热稳定性激酶(例如,pfpyrh、tthadk、tthcmk、pfgmk、aandk、teppk和/或ppk2(例如,参见表6)和热稳定性t7rna聚合酶的培养细胞(例如,工程化细胞),从而产生细胞裂解物,(b)在导致rna解聚成5'-nmp的条件下温育步骤(a)中产生的细胞裂解物,从而产生包含5'-nmp的细胞裂解物,(c)将步骤(b)中产生的细胞裂解物加热至60-80℃以使内源核酸酶和磷酸酶失活而不使热稳定性激酶和热稳定性rna聚合酶完全失活,从而产生包含热失活核酸酶和磷酸酶的细胞裂解物,和(d)在能量源(例如包含多磷酸盐的atp再生系统)和工程化核酸(例如dna)模板(例如,含有与编码感兴趣rna的核苷酸序列可操作地连接的启动子)存在下温育步骤(c)中,在导致三磷酸核苷的产生和三磷酸核苷的聚合的条件下温育步骤(c)中产生的细胞裂解物以产生感兴趣rna。在一些实施方案中,rna、rna酶r、热稳定性激酶和热稳定性t7rna聚合酶包含在培养细胞(例如,工程化细胞)的单一菌株中。在其他实施方案中,裂解含有上述活性/组分子集的培养细胞(例如,工程化细胞),并将裂解物组合以产生细胞裂解物混合物,其包含上述步骤(a)中描述的所有酶活性。在一些实施方案中,将纯化酶形式的酶活性添加到上述步骤(a)中的裂解物中。在一些实施方案中,裂解物和/或纯化的蛋白质在上述步骤(c)中描述的热失活步骤之前组合。在其他实施方案中,裂解物和/或纯化的蛋白质在上述步骤(c)中描述的热失活步骤后组合。感兴趣的rna可以是任何形式的rna,包括单链rna和双链rna。例如,感兴趣的rna可以是信使rna(mrna)、反义rna、微rna、小干扰rna(sirna)或短发夹rna(shrna)。其他rna干扰(rnai)分子包括在本文中。在附图和详细描述中阐述了本发明的若干实施例的细节。根据说明书和权利要求书,本发明的其他特征、目的和优点将显而易见。附图简述图1显示了如本文描述的无细胞rna生产的示意图。将含有来自生物量的rna(例如,内源rna)、核酸酶、热稳定性激酶和/或热稳定性rna聚合酶的细胞裂解(或组合并裂解),并将得到的细胞裂解物在导致rna解聚的条件下温育。然后,加热细胞裂解物以失活核酸酶和任何内源磷酸酶(不使热稳定性激酶和热稳定性rna聚合酶失活)。然后,在导致三磷酸核苷产生和三磷酸核苷聚合的条件下,在编码感兴趣rna的工程化dna模板存在下温育细胞裂解物,从而产生感兴趣rna(例如ssrna或dsrna)。或者,可以在热失活步骤后将个别的纯化途径酶(例如rna聚合酶,例如热稳定性rna聚合酶)添加到细胞裂解物中。因此,在一些情况下,用于产生细胞裂解物的工程化细胞不表达上述一种或多种酶活性,例如核酸酶、热稳定性激酶和/或热稳定性rna聚合酶。图2a显示了用于能量产生的多磷酸盐依赖性激酶途径的示意图。图2b显示了用于本公开的方法和系统的另外的示例性能量转化途径的示意图。ump激酶(例如,从激烈火球菌获得)和多磷酸激酶(例如,从伸长热聚球藻、嗜气暖绳菌、地热异常球菌、红色亚栖热袍菌、meiothermussilvanus、地热异常球菌、嗜热厌氧绳菌、chlorobaculumtepidum、深海洋栖热袍菌、roseiflexuscastenholzii、玫瑰弯菌属种或trueperaradiovctrix获得)可用于将ump转化为udp,并且ndp激酶(例如,由风产液菌ndk基因编码)和多磷酸激酶可用于将udp转化为utp。cmp激酶(例如,从嗜热栖热袍菌获得)和多磷酸激酶可用于将cmp转化为cdp,并且ndp激酶和多磷酸激酶可用于将cdp转化为ctp。gmp激酶(例如,从海洋栖热袍菌获得)和多磷酸激酶可用于将gmp转化为gdp,并且ndp激酶和多磷酸激酶可用于将gdp转化为gtp。amp激酶(例如,从嗜热栖热袍菌获得)和多磷酸激酶可用于将amp转化为adp,并且ndp激酶(例如,由风产液菌ndk基因编码)和多磷酸激酶可用于将adp转化为atp。或者,iii类ppk2酶(参见例如表6)可用于将amp转化为atp。图3a显示了用于双链rna生物合成的dna模板的实例的示意图。编码为质粒一部分的dna模板含有单个编码区,该编码区包括与感兴趣编码区可操作连接的启动子和一个或多个终止子。转录后,rna通过分子内核苷酸碱基配对折叠成发夹结构。dna模板(单独或编码为质粒的一部分)含有由域2分开的两个互补域(1和3)。图3b显示了用于双链rna生物合成的dna模板的另一个实例的示意图。dna模板在互补链上含有会聚的启动子序列。转录后从每个模板链转录的rna序列退火。图3c显示了用于双链rna生物合成的dna模板的另一个实例的示意图。编码为质粒一部分的dna模板含有与互补链上感兴趣的编码区可操作连接的会聚启动子序列,以及一个或多个终止子序列以防止通读转录。图3d显示了用于双链rna生物合成的dna模板的另一个实例的示意图。编码为质粒一部分的dna模板含有独立的盒,每个盒包含与感兴趣编码区可操作连接的启动子和一个或多个终止子,驱动互补序列的转录,所述互补序列在转录后退火。图3e显示了用于双链rna生物合成的dna模板的另一个实例的示意图。dna依赖性rna聚合酶用于产生ssrna模板,并且rna依赖性rna聚合酶用于产生双链rna。图4显示了本公开的无细胞rna生产方法的另一个实例的示意图。该过程从单个发酵容器开始,其中使用标准发酵技术生产工程化细胞。从发酵产生的生物量任选地通过微滤(mf)浓缩,然后通过例如机械均化进行裂解。然后将裂解物泵入第二发酵容器,其中表达的核酸酶将rna转化为其单体成分。加热整个反应以失活任何内源磷酸酶或核酸酶(例如rna酶)活性以及对rna产物稳定性和/或保真度有害的任何其他外源/引入的细胞(例如核酸酶)活性。在热失活后,将多磷酸盐作为高能量磷酸盐源添加到反应中,通过一系列热稳定性激酶将nmp磷酸化为ntp,然后聚合成dsrna。下游加工可用于将纯度增加至按重量计多达99%(例如,50%,60%,70%,80%,90%,95%,98%或99%)dsrna。例如,加工可用于将纯度提高到50-60%,50-70%,50-80%,50-90%,50-95%,70-80%,70-90%或70-95%。示例性下游过程开始于添加蛋白质沉淀剂(例如乙酸铵),然后通过盘式堆叠离心(dsc)或切向流过滤(tff)从产物流中除去蛋白质、脂质和一些dna。然后实施超滤以除去盐并减少体积。向产物流添加到氯化锂导致dsrna产物沉淀,随后使用圆盘堆叠离心从大量液体中分离,产生80%纯度的dsrna产物流。进一步的层析精制产生99%纯的产物(nilsen,tw.coldspringharbprotoc.2012dec1;2012(12))。图5a-5b显示通过消化纯化的大肠杆菌rna来比较核糖核酸酶活性(图5a)。用核酸酶处理释放酸溶性核苷酸(单核苷酸和短寡核苷酸)用benzonase、rna酶a、rna酶r和核酸酶p1是最快速的。(图5b)反应产物的lc-ms分析证明用rna酶r和核酸酶p1处理从rna释放nmp。图6是显示使用外源rna酶r解聚裂解物rna的图,其中通过uplc分析产物特异性鉴定5'-nmp。在没有rna酶r的情况下,裂解物表现出内源rna酶活性,其导致5'-nmp的缓慢积累(实心深灰色线)。外源rna酶r的添加导致快速的5'-nmp释放(深灰色虚线),而不影响2'或3'nmp积累的速率(浅灰色线)。因此,rna酶r的过表达加速聚合rna向5'-nmp转化的速率,降低提取物中存在的磷酸酶/核酸酶活性的有害作用。以50%裂解物的终浓度进行实验。图7a-7c显示在分批阶段培养的1l生物反应器培养物中rna酶r过表达的结果。(图7a)一式两份培养物中蛋白质表达的sds-page分析。空载体培养物含有空蛋白质表达载体(petduet-1)。rna酶r培养物含有克隆到petduet-1中的大肠杆菌rnr。来自诱导培养物(+)的样品表现出rna酶r的强表达(mw92.9kda,具有c-末端六组氨酸标签),如右箭头所示。(图7b)空载体(深灰色)和表达rna酶r的菌株(浅灰色)的生长动力学(包括诱导前和诱导后生长)证明rna酶r过表达对细胞生长无害。虚线表示指数曲线拟合。(图7c)过表达的rna酶r在分批培养的生物量的裂解物中具有活性,释放出酸溶性核苷酸。在空载体菌株(实心深灰色线)中,添加外源rna酶r增加核苷酸释放速率(深灰色虚线)。相反,表达rna酶r的菌株在裂解后表现出快速的核苷酸释放(实心浅灰色线)。添加外源rna酶r不会增加核苷酸释放速率或最终核苷酸产率(浅灰色虚线)。以50%裂解物的终浓度进行实验。图8是描述螯合mg2+对高密度裂解物中解聚速率的影响的图。由含有空载体(深灰色)的生物量制备的裂解物对edta不敏感。具有过表达的rna酶r(浅灰色)的裂解物在除去mg2+的情况下表现出快速的rna解聚,8mmedta提供最大的解聚速率。以90%裂解物的终浓度进行实验。图9a-9d显示的图表明外源同位素标记的“重”nmp(hnmp)在裂解物中的稳定性:(图9a)hamp在裂解物中相对稳定,在37℃温育1小时后剩余90%。(图9b)hcmp在裂解物中降解,约30分钟后剩余70%。添加10mm原钒酸钠(虚线)(几种磷酸酶和激酶的抑制剂)显著改善稳定性。(图9c)hump在裂解物中降解,约20分钟后剩余70%。磷酸钠(150mm)(虚线)和原钒酸钠(虚线)显著改善稳定性。(图9d)hgmp在裂解物中降解,约10分钟后剩余70%。原钒酸钠显著改善稳定性,30分钟后剩余70%hgmp。图10是表明热失活对裂解物中外源ntp稳定性的影响的图。将裂解物在70℃预温育,然后将温度降至37℃并添加到等摩尔的ntp混合物(atp,ctp,utp和gtp)。预温育时间列在右侧的图例中。对照裂解物(不经受热失活)快速消耗ntp(t=0分钟)。增加预温育时间稳定ntp,在70℃持续15分钟消除ntp酶活性(t=15分钟)。图11a-11b是表明热失活对裂解物中nmp和dsrna的稳定性的影响的图。(图11a)热失活使裂解物中的nmp稳定化。在37℃用外源rna酶r处理裂解物(裂解物+rna酶r)(t=0分钟-5分钟)以释放nmp,然后在70℃加热失活(t=5分钟至25分钟)。然后将温度降至37℃并将反应再温育60分钟。热失活后,nmp在裂解物中在很大程度上是稳定的。(图11b)热失活使裂解物中转录反应的反应物和产物稳定化。将裂解物在指定温度预温育15分钟,然后将温度降至37℃并添加转录反应物。在70℃和80℃而非60℃热失活使底物和产物充分稳定化,足以产生类似于阳性对照(无裂解物)的可检测转录产物。图12的图表明来自激烈火球菌的ump激酶(pfpyrh)的温度依赖性活性,通过用于atp消耗的萤光素酶测定法量化。纯化的pfpyrh的比活性对温育温度在很大程度上是不敏感的。图13的图表明来自嗜热栖热袍菌的amp激酶(tthadk)与来自大肠杆菌的adk(ecadk)的温度依赖性活性,其通过萤光素酶测量。纯化的ecadk在低于60℃的温度具有活性。tthadk具有较高的比活性,最高值于70℃。图14的图表明通过萤光素酶测量的来自嗜热栖热袍菌的cmp激酶(tthcmk)的温度依赖性活性。纯化的tthcmk对温度相对不敏感,37-80℃具有高活性。图15的图表明通过萤光素酶测量的来自大肠杆菌(ecgmk)、嗜热栖热袍菌(tthgmk)和海洋栖热袍菌(tmgmk)的gmp激酶的温度依赖性活性。纯化的ecgmk(深灰色)在较低温度更具活性,而tthgmk(浅灰色)和tmgmk(中灰色)在70℃最具活性。图16的数据图表明通过萤光素酶测量的来自风产液菌的纯化ndp激酶(aandk)的活性。使用atp和gdp作为底物,纯化的aandk在37-80℃具有高活性,在50℃具有最佳活性。图17的图表明通过萤光素酶测量的来自大肠杆菌(ecppk)、伸长热聚球藻(teppk)和嗜热栖热袍菌(tthppk)的纯化的多磷酸激酶1(ppk1)酶的活性。ecppk在温度≤60℃最具活性,而teppk在70℃具有最佳活性。tthppk表现出相对较低的活性。图18的图表明商品化t7rna聚合酶在缓冲液中使用其各自制造商推荐的条件下在37℃的活性。在使用双链体dna模板的测试条件下,thermot7和megascript聚合酶表现出比neb聚合酶更高的比活性(例如在图3b中)。图19是比较在具有双链体dna模板的标准化反应条件下在37℃和50℃的稀裂解物中的t7rna聚合酶活性的图。在37℃,thermot7表现出最高的比活性。在50℃,仅thermot7具有可检测的活性,产生超过10g/l/hr的dsrna。图20是表明在缓冲液和高密度热失活裂解物中thermot7活性的活性的图。在热失活后通过离心澄清的裂解物中thermot7活性最高。省略澄清步骤导致活性降低60%,尽管未澄清基质中的聚合酶活性比在仅缓冲液中大。图21是证明thermot7对升高的温度的耐受性的图。在50℃预温育thermot7对在37℃测定的后续聚合酶活性没有影响。在60℃和70℃预温育导致酶功能的快速、不可逆的抑制。图22a是sds-page凝胶的图像,显示嗜热厌氧绳菌ppk2在大肠杆菌菌株gl16-170中的表达和溶解度数据。mw:未染色的蛋白质标准,boardrange(newenglandbiolabs产品目录编号p7704)。-:诱导前培养。+:收获时诱导培养。l:澄清裂解物中的可溶性蛋白质。嗜热厌氧绳菌ppk2:33kda。图22b是显示热失活的裂解物中嗜热厌氧绳菌ppk2的atp产生的图。闭环表示自adp的atp产生。空心圆表示自amp的atp产生。对于这两种底物,嗜热厌氧绳菌ppk2以超过400mm/hr的速率产生atp。图23是琼脂糖凝胶的图像,其证明了热稳定性iii类ppk2用于无细胞dsrna生产中的能量产生的应用。左侧泳道含有阳性对照,证明了来自ntp的dsrna合成。中间泳道含有阳性对照,证明了使用外源atp作为能量源,在表达核苷酸激酶的裂解物中从nmp合成dsrna。右侧泳道含有反应,其表明使用核苷酸激酶和表达嗜气暖绳菌ppk的裂解物自nmp和hmp的dsrna合成。在每种情况下,无细胞rna合成反应均不依赖于mn2+。包括没有聚合酶的反应作为阴性对照,说明每个含裂解物的反应的背景核酸含量。图24是琼脂糖凝胶的图像,其证明了热稳定性iii类ppk2用于无细胞dsrna生产中的能量产生的应用。左侧泳道含有阳性对照,证明了来自ntp的dsrna合成。中间泳道含有阳性对照,证明了使用外源atp作为能量源,在表达核苷酸激酶的裂解物中从nmp合成dsrna。右侧泳道含有反应,其证明了使用核苷酸激酶和表达嗜气暖绳菌ppk的裂解物自nmp和hmp合成dsrna。对于嗜气暖绳菌ppk2,dsrna合成在不存在amp激酶和外源adp的情况下进行。发明详述在一些方面,本文提供了用于核酸(例如rna或dna)的无细胞生产(生物合成)的方法、组合物、细胞、构建体和系统。在一些实施方案中,将单一类型的生物体(例如,细菌细胞群体)工程化以表达至少一种核酸酶、至少一种热稳定性激酶和至少一种热稳定性聚合酶(例如rna或dna聚合酶)。在导致酶表达的条件下培养(培养)工程化细胞。在一些实施方案中,可以将工程化细胞培养至期望的细胞密度,然后可以诱导(激活)某些酶的表达。因此,某些酶的转录可以在诱导型启动子的控制下。然后裂解细胞(例如,工程化的和/或非工程化的细胞)(例如,机械地,化学地或酶促地破碎)以产生细胞裂解物,其包含rna(例如,ssrna或dsrna)的无细胞生产所需的酶活性。在一些实施方案中,在细胞裂解步骤之前,将含有聚合rna(例如,mrna、trna和/或rrna)的细胞与含有途径酶的工程化细胞混合。在其他实施方案中,将从含有聚合rna的细胞获得的细胞裂解物与获自含有途径酶的工程化细胞的细胞裂解物组合(混合)。在其他实施方案中,将一种或多种纯化的途径酶与从工程化细胞获得的细胞裂解物组合(混合)。“途径酶”是生物合成感兴趣rna(例如,从聚合rna开始)所需的酶。为了合成rna,将细胞裂解物(或细胞裂解物混合物)在导致宿主衍生的(内源)rna的核酸酶介导的(例如,rna酶介导的)解聚至期望的5'-一磷酸核苷(nmp,或一磷酸核苷)产率的条件下温育。在一些实施方案中,然后加热细胞裂解物(或细胞裂解物混合物)以失活大多数宿主衍生的酶,包括磷酸酶和核酸酶(例如,rna酶),以及先前添加到细胞裂解物以促进宿主衍生的rna的解聚的任何外源核酸酶。在热失活步骤之后,使用例如热稳定性多磷酸激酶和添加多磷酸盐作为能量源,在导致通过热稳定性激酶(例如,热稳定性一磷酸核苷激酶和二磷酸核苷激酶)将nmp磷酸化为ntp(三磷酸核苷)的条件下温育细胞裂解物。随后通过rna聚合酶(例如,热稳定性rna聚合酶)使用存在于裂解物中的工程化模板(例如,dna模板)(例如,由工程化细胞表达并包括作为细胞裂解物的细胞组分,或后来添加到细胞裂解物)将所得ntp聚合成rna。无细胞生产“无细胞生产”是使用生物方法合成生物分子或化学化合物而不使用活细胞。裂解细胞并将未纯化(粗制)部分或部分纯化的部分(均含有酶)用于生产期望的产物。在一些实施方案中,可以将纯化的酶添加到细胞裂解物。例如,将细胞培养,收获和通过高压均质化或其他细胞裂解方法(例如,化学细胞裂解)裂解。无细胞反应可以分批或补料-分批模式进行。在一些情况下,酶促途径填充反应器的工作体积并且可以比细胞内环境更稀释。然而,提供了基本上所有的细胞催化剂(cellularcatalyst),包括与膜结合的催化剂。在细胞裂解期间将内膜破碎,这些膜的碎片可形成膜囊泡。参见,例如swartz,aichejournal,2012,58(1),5-13,通过引用并入本文。本公开的无细胞方法、组合物和系统利用本文更详细地讨论的细胞裂解物(例如,粗制或部分纯化的细胞裂解物)。例如,通过机械方法(例如,剪切或压碎)制备的细胞裂解物不同于化学透化的细胞。如上所讨论,在一些实施方案中,在细胞裂解(例如,机械细胞裂解)期间,将内细胞膜破碎,使得在细胞裂解物中形成倒置的膜囊泡。此类导致的膜囊泡不是通过化学细胞透化方法产生的。裂解的细胞(例如,至少75%,80%,85%,90%或95%)不再是完整的。因此,认为透化细胞(其是包含穿孔(小孔)的完整细胞)不是裂解细胞。虽然本文提供的方法通常是无细胞的并且使用细胞裂解物,但在一些实施方案中,至少对于该方法的一些步骤,使用透化细胞可能是有利的。因此,本公开不排除在rna生产方法的至少一个步骤中使用透化细胞。应当理解,尽管本文描述的许多实施方案涉及“裂解包含特定酶的培养细胞”,但该短语旨在包括裂解从单一培养物获得的克隆细胞群(例如,包含合成rna所需的所有酶)以及裂解超过一个克隆细胞群,每个从不同的细胞培养物中获得(例如,每个含有合成rna和/或聚合rna底物所需的一种或多种酶)。例如,在一些实施方案中,表达一种热稳定性激酶的细胞群(例如,工程化细胞)可以一起培养并用于产生一种细胞裂解物,并且另一种表达不同热稳定性激酶的细胞群(例如,工程化细胞)可以一起培养并用于产生另一种细胞裂解物。然后可以组合这两种细胞裂解物(每种细胞裂解物包含不同的热稳定性激酶)用于本公开的rna生物合成方法。核糖核酸解聚为一磷酸核苷本公开基于通过涉及一系列酶促反应的无细胞过程使用细胞裂解物将来自生物量的rna(例如,内源细胞rna)转化为期望的合成rna。首先,存在于源自宿主细胞的细胞裂解物中的rna(例如,内源rna)通过核酸酶转化为其组成单体。来自生物量的rna(例如,内源rna)通常包括核糖体rna(rrna)、信使rna(mrna)、转移rna(trna),其他rna或其组合。rna的解聚或降解导致5'-一磷酸核苷(5'-nmp)(也简称为“单体”)合并物的产生。这些单体用作起始下游聚合/合成感兴趣rna的材料,所述单体转化为二磷酸核苷,其转化为三磷酸核苷。在一些实施方案中,感兴趣rna是ssrna(例如,mrna)。在一些实施方案中,感兴趣rna是dsrna。合成感兴趣的rna所需要的rna(例如,内源rna)的量可以变化,这取决于例如感兴趣的rna的期望长度和产率以及相对于细胞(例如,大肠杆菌细胞)的rna(例如,内源rna)的核苷酸组成的rna的核苷酸组成。通常,对于细菌细胞,例如,rna(例如,内源rna)含量范围为总细胞质量的5-50%。起始材料的质量可以例如使用以下方程式计算:(千克(kg)rna/千克干细胞重量)x100%。内源rna可以通过化学或酶促方法解聚或降解成其组成单体。然而,rna的化学水解通常产生2’-和3'-nmp,其不能聚合成rna。因此,如本文提供的方法、组合物和系统主要使用酶来解聚内源rna。“解聚rna的酶”催化rna中两个核苷酸之间磷酸二酯键的水解。因此,“解聚rna的酶”将rna(聚合rna)转化为其单体形式-一磷酸核苷(nmp)。取决于酶,rna的酶促解聚可以产生3'-nmp、5'-nmp或3'-nmp和5'-nmp的组合。由于不可能聚合3'-ntp(从3'-ndp转化而来,3'-ndp转化自3'-nmp),产生5'-nmp(其然后转化为5'-ndp,然后是5'-ntp)的酶(例如rna酶r)是优选的。在一些实施方案中,从工程化细胞的基因组dna中除去产生3'-nmp的酶,以提高rna生产的效率。在一些实施方案中,用于rna解聚的酶是rna酶r。在一些实施方案中,使用的rna酶r的浓度为0.1-1.0mg/ml(例如,0.1、0.2、0.3、0.4、0.5、0.6、0.7、0.8、0.9或1.0mg/ml)。在一些实施方案中,使用的rna酶r的浓度为0.4-0.6mg/ml。在一些实施方案中,使用的rna酶r的浓度为0.5mg/ml。在一些实施方案中,使用的rna酶r的浓度大于1.0mg/ml。解聚rna的酶的实例包括但不限于核酸酶,包括核糖核酸酶(rna酶,例如rna酶r)和磷酸二酯酶。核酸酶催化核酸降解成较小的组分(例如,单体,也称为一磷酸核苷或寡核苷酸)。磷酸二酯酶催化磷酸二酯键的降解。解聚rna的这些酶可以由全长基因或通过基因融合物(例如,包含编码两种不同酶活性的至少两种不同基因(或基因片段)的dna)编码。rna酶在细胞中起作用以调节rna成熟和周转。每种rna酶具有特定的底物偏好-dsrna或ssrna。因此,在一些实施方案中,通常可以使用不同rna酶的组合或不同核酸酶的组合来解聚生物量衍生的聚合rna(例如,内源rna)。例如,1-2、1-3、1-4、1-5、1-6、1-7、1-8、1-9或1-10种不同的核酸酶可以组合使用以解聚rna。在一些实施方案中,可以组合使用至少1种、至少2种、至少3种、至少4种、至少5种、至少6种、至少7种、至少8种、至少9种、或至少10种不同的核酸酶以解聚rna。如本文提供的使用的核酸酶的非限制性实例包括在表1中。在一些实施方案中,使用的核酸酶是rna酶r。表1.解聚核糖核酸的酶解聚rna的酶(例如,rna酶)可以是宿主细胞内源的(宿主衍生的),或者它们可以由外源引入宿主细胞的工程化核酸编码(例如,在附加体载体上或整合到宿主细胞的基因组中)。在一些实施方案中,编码解聚rna的酶的工程化核酸与诱导型启动子可操作地连接。因此,在一些实施方案中,编码解聚rna的酶的工程化核酸的表达在时间上或空间上受到调节。例如,可以改造核酸以编码在宿主细胞的周质中重新定位或隔离的酶(例如,rna酶),使得酶的活性不会干扰细胞生长或其他代谢过程。细胞裂解后,重新定位的酶从周质释放,与内源rna接触,并将rna解聚成单体形式。参见例如2011年11月10日公开的国际公开号wo2011/140516,其通过引用并入本文。“导致rna解聚的条件”是本领域已知的,或者可以由本领域普通技术人员确定,考虑到例如核酸酶(例如,rna酶)活性的最佳条件,包括ph、温度、时间长度和细胞裂解物的盐浓度以及任何外源辅因子。实例包括前面描述的那些(参见,例如wong,c.h.etal.j.am.chem.soc.,105:115-117,1983),ep1587947b1,chengzf,deutschermp.jbiolchem.277:21624–21629,2002)。在一些实施方案中,金属离子(例如,mg2+)从解聚反应中消减。在一些实施方案中,金属离子(例如,mg2+)的浓度为8mm或更低(例如,小于8mm,小于7mm,小于6mm,小于5mm,小于4mm,小于3mm,小于2mm,小于1mm,小于0.5mm)。在一些实施方案中,金属离子(例如,mg2+)的浓度为0.1mm-8mm,0.1mm-7mm或0.1mm-5mm。rna解聚反应期间细胞裂解物的ph可具有3.0至8.0的值。在一些实施方案中,细胞裂解物的ph值为3.0-8.0、4.0-8.0、5.0-8.0、6.0-8.0、7.0-8.0、3.0-7.0、4.0-7.0、5.0-7.0、6.0-7.0、3.0-6.0、4.0-6.0、5.0-6.0、3.0-5.0、3.0-4.0或4.0-5.0。在一些实施方案中,细胞裂解物的ph值为3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5或8.0。在一些实施方案中,细胞裂解物的ph值为7.0、7.1、7.2、7.3、7.4或7.5。根据需要,可以调节细胞裂解物的ph。rna解聚反应期间细胞裂解物的温度可以是15℃至70℃。在一些实施方案中,rna解聚反应期间细胞裂解物的温度为15-60℃、15-50℃、15-40℃、15-30℃、25-70℃、25-60℃、25-50℃、25-40℃、30-70℃、30-60℃或30-50℃。在一些实施方案中,rna解聚反应期间细胞裂解物的温度为37℃。在一些实施方案中,rna解聚反应期间细胞裂解物的温度为15℃、25℃、32℃、37℃、40℃,42℃、45℃、50℃,55℃、56℃、57℃、58℃、59℃、60℃、61℃、62℃、63℃、64℃、65℃、66℃、67℃、68℃、69℃或70℃。rna解聚反应期间的细胞裂解物可以温育5分钟(min)至72小时(hr)。在一些实施方案中,将rna解聚反应期间的细胞裂解物温育5-10分钟,5-15分钟,5-20分钟,5-30分钟或5分钟-48小时。例如,rna解聚反应期间的细胞裂解物可以温育5min、10min、15min、20min、25min、30min、45min、1hr、2hrs、3hrs、4hrs、5hrs、6hrs、7hrs、8hrs、9hrs、10hrs、11hrs、12hrs、18hrs、24hrs、30hrs、36hr,42小时或48小时。在一些实施方案中,rna解聚反应期间的细胞裂解物在37℃的温度温育24小时。在一些实施方案中,rna解聚反应期间的细胞裂解物在37℃的温度温育5-10分钟。在一些实施方案中,rna解聚反应期间的细胞裂解物具有7.0的ph并且在37℃的温度温育15分钟。在一些实施方案中,rna解聚反应期间的细胞裂解物可以在导致rna转化为5'-nmp的转化率大于65%的条件下温育。在一些实施方案中,rna以(或至少)50mm/hr,100mm/hr或200mm/hr的速率转化为5'-nmp。在一些实施方案中,将盐添加到细胞裂解物中,例如,以防止酶聚集。例如,可以将氯化钠、氯化钾、乙酸钠、乙酸钾或其组合添加到细胞裂解物中。rna解聚反应期间细胞裂解物中的盐浓度可以是5mm至1m。在一些实施方案中,rna解聚反应期间细胞裂解物中的盐浓度为5mm、10mm、15mm、20mm、25mm、50mm、100mm、150mm、200mm、250mm、500mm、750mm或1m。在一些实施方案中,细胞裂解物包含含有40-60mm磷酸钾、1-5mmmncl2、和/或10-50mmmgcl2(例如20mmmgcl2)的混合物。在一些实施方案中,将缓冲液添加至细胞裂解物,例如,以实现特定的ph值和/或盐浓度。缓冲剂的实例包括但不限于磷酸盐缓冲液、tris缓冲液、mops缓冲液、hepes缓冲液、柠檬酸盐缓冲液、乙酸盐缓冲液、苹果酸缓冲液、mes缓冲液、组氨酸缓冲液、pipes缓冲液、bis-tris缓冲液和乙醇胺缓冲液。rna的解聚导致5'-nmp的产生,包括5'-amp、5'-ump、5’-cmp和5'-gmp。虽然nmp可以以相对等摩尔量存在于细胞裂解物中,但rna的解聚不会导致任何预定的nmp比率。在一些实施方案中,将裂解后细胞中50-98%的内源rna转化成(解聚成)5'-nmp。例如,将50-95%,50-90%,50-85%,50-80%,75-98%,75-95%,75-90%,75-85%或75-80%的rna转化成(解聚成)5'-nmp。在一些实施方案中,将裂解后细胞中65-70%的内源rna转化成(解聚成)5'-nmp。较低的产率也是可接受的。消除无效循环在通过内源和/或外源核酸酶将来自生物量的rna(例如,内源rna)转化为其单体成分之后,通常在细胞裂解物中保留几种酶,包括核酸酶和磷酸酶,其可以对rna合成具有有害作用。例如,大肠杆菌具有许多磷酸酶,其中许多磷酸酶使ntp、ndp和nmp脱磷酸化。rna解聚后nmp的脱磷酸化导致非磷酸化核苷的积累和可用的nmp底物的损失,从而降低合成rna产率。rna解聚后nmp、ndp或ntp的脱磷酸化导致无效的能量循环(产生低合成rna产率的能量循环),在此期间nmp被磷酸化为ndp和ntp,其继而被脱磷酸化回其nmp或核苷起点。无效循环降低每单位能量输入(例如,多磷酸盐、atp或其他高能量磷酸盐源)的rna产物的产率。在一些实施方案中,通过从宿主基因组中除去来消除酶活性。在一些实施方案中,通过热失活消除酶活性。在一些实施方案中,通过蛋白酶靶向消除酶活性。在一些实施方案中,通过使用化学抑制剂消除酶活性。也可以使用任何前述方法的组合。如本文所提供的对于rna的生物合成有害的酶可以在工程化改造宿主细胞的过程期间从宿主细胞基因组中删除,条件是酶对于宿主细胞(例如,细菌细胞)的存活和/或生长不是必需的。例如,可以通过在宿主细胞基因组中删除或修饰编码必需酶的基因来实现酶或酶活性的删除。若酶是宿主细胞存活所必需的,则酶是“宿主细胞存活必需的”。也就是说,若宿主细胞在没有特定酶的表达和/或活性的情况下不能存活,则认为酶是宿主细胞存活必需的。类似地,若酶是宿主细胞生长所必需的,则酶是“宿主细胞生长必需的”。也就是说,若宿主细胞不能在没有特定酶的表达和/或活性的情况下分裂和/或生长,则认为酶是宿主细胞生长所必需的。若对rna的生物合成有害的酶对宿主细胞存活和/或生长是必需的,则可能无法删除或修饰编码酶的基因。在此类情况下,可以使酶热失活。“热失活”是指将细胞裂解物加热至足以使内源核酸酶和磷酸酶失活(或至少部分失活)的温度的过程。通常,热失活过程涉及有害酶的(解折叠)的变性。内源细胞蛋白变性的温度在生物体间变化。例如,在大肠杆菌中,内源细胞酶通常在高于41℃的温度变性。对于其他生物体,变性温度可高于或低于41℃。如本文提供的细胞裂解物的酶可以在40℃-95℃或更高的温度热失活。在一些实施方案中,细胞裂解物的酶可以在40-90℃、40-80℃、40-70℃、40-60℃、40-50℃、50-80℃、50-70℃、50-60℃、60-80℃、60-70℃或70-80℃的温度热失活。例如,细胞裂解物的酶可以在40℃、42℃、45℃、50℃、55℃、60℃、65℃、70℃、75℃、80℃、85℃、90℃或95℃的温度热失活。在一些实施方案中,细胞裂解物的酶可以在50-80℃的温度热失活。在一些实施方案中,细胞裂解物的酶可以在70℃的温度加热失活。在一些实施方案中,细胞裂解物的酶可以在60℃的温度热失活。也可以引入有害酶的化学抑制剂。此类抑制剂可包括但不限于原钒酸钠(蛋白质磷酸酪氨酸磷酸酶抑制剂)、氟化钠(磷酸丝氨酸磷酸酶和磷酸苏氨酸磷酸酶的抑制剂)、焦磷酸钠(磷酸酶抑制剂)、磷酸钠和/或磷酸钾。细胞裂解物在升高的温度下温育以实现内源酶的热失活的时间段可以变化,这取决于例如细胞裂解物的体积和制备细胞裂解物的生物体。在一些实施方案中,将细胞裂解物在35℃-80℃的温度温育2分钟(min)至48小时(hr)。例如,细胞裂解物可以在35℃-80℃的温度温育2分钟,4分钟,5分钟,10分钟,15分钟,30分钟,45分钟或1小时。在一些实施方案中,将细胞裂解物在35℃-80℃的温度温育2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、24、36、42或48小时。在一些实施方案中,酶在60-80℃的温度热失活10-20分钟。在一些实施方案中,酶在70℃的温度热失活15分钟。在一些实施方案中,解聚内源rna的酶包括使酶对热更敏感的一种或多种修饰(例如突变)。这些酶称为“热敏感性酶”。热敏感性酶在比其野生型对应物的温度更低的温度变性并变得失活,和/或降低热敏感性酶活性所需的时间段短于其野生型对应物的。应当理解,在一些情况下,热失活的酶可以保持一定程度的活性。例如,热失活酶的活性水平可以小于尚未热失活的相同酶的活性水平的50%。在一些实施方案中,热失活酶的活性水平是尚未热失活的相同酶的活性水平的小于40%,小于30%,小于20%,小于10%,小于5%,小于1%或小于0.1%。因此,可以完全消除或减少酶的活性。若酶的变性(热失活)形式不再催化由其天然形式的酶催化的反应,则认为酶是完全无活性的。当热失活酶的活性相对于未加热的酶(例如,在其天然环境中)的活性降低至少50%时,认为热失活的变性酶是“失活的”。在一些实施方案中,相对于未加热的酶的活性,热失活酶的活性降低50-100%。例如,相对于未加热的酶的活性,热失活酶的活性降低50-90%,50-85%,50-80%,50-75%,50-70%,50-65%,50-60%或50-55%。在一些实施方案中,相对于未加热的酶的活性,热失活酶的活性降低25%,30%,35%,40%,45%,50%,55%,60%,65%,70%,75%,80%,85%,90%,95%,98%,99%或100%。可以热失活或从宿主细胞的基因组中删除的酶的实例包括但不限于核酸酶(例如,rna酶iii、rna酶i、rna酶r、pnp酶、rna酶ii和rna酶t)、磷酸酶(例如,核苷一磷酸酶、核苷二磷酸酶、核苷三磷酸酶)和解聚rna或使核苷酸脱磷酸化的其他酶。解聚rna的酶包括能够切割,部分水解或完全水解rna分子的任何酶。表2提供了核酸酶的非限制性实例的列表,所述核酸酶可以是热失活的,或者在一些情况下,从工程化的宿主细胞中删除。表3提供了磷酸酶的非限制性实例的列表,所述磷酸酶可以是热失活的,或者在一些情况下,从工程化的宿主细胞中删除。本公开涵盖了这些和其他核酸酶和磷酸酶的热失活。表2.核酸酶的实例表3.磷酸酶的实例。大肠杆菌rna酶iii优先切割dsrna以及一些单链mrna分子。细胞裂解物中rna酶iii的存在可以限制高浓度合成rna(例如dsrna)的积累,因为合成的rna易于裂解。rna酶iii和编码rna酶iii的基因rnc都不是细胞活力所必需的,因此,在一些实施方案中,rnc在工程化宿主细胞中删除或突变。在其他实施方案中,rna酶iii在内源rna解聚后被热失活。大肠杆菌rna酶i定位于完整细胞中的周质空间并催化极其多种rna分子的解聚,包括rrna、mrna和trna。在生理条件下,该酶的周质定位意味着酶对细胞内的rna稳定性几乎没有影响。然而,细胞裂解物中周质和细胞质的混合允许rna酶i接近细胞rna。细胞裂解物中rna酶i的存在可以通过rna降解降低合成rna的产率。rna酶i和编码rna酶i的基因rna都不是细胞活力所必需的,因此,在一些实施方案中,rna在工程化宿主细胞中删除或突变。在其他实施方案中,rna酶i在内源rna解聚后被热失活。大肠杆菌rna酶r和rna酶t催化dsrna、rrna、trna和mrna以及小的非结构化rna分子的解聚。酶和分别编码酶的基因rnr和rnt对细胞活力不是必需的,因此,在一些实施方案中,rnr和/或rnt在工程化宿主细胞(例如,大肠杆菌宿主细胞)中删除或突变。在其他实施方案中,rna酶r和/或rna酶t在内源rna解聚后被热失活。大肠杆菌rna酶e和pnp酶是降解体的组分,其负责细胞中的mrna周转。认为rna酶e与pnp酶和rna酶ii一起起作用以周转细胞mrna合并物。编码rna酶e的基因rne的破坏在大肠杆菌中是致死的。因此,在一些实施方案中,rna酶e在内源rna解聚后被热失活。pnp酶和编码pnp酶的基因pnp都不是细胞活力所必需的,因此,在一些实施方案中,pnp在工程化宿主细胞(例如大肠杆菌宿主细胞)中删除或突变。在其他实施方案中,pnp酶在内源rna解聚后被热失活。大肠杆菌rna酶ii以3'→5'方向解聚mrna和trna两者。rna酶ii和编码rna酶ii的基因rnb都不是细胞活力所必需的,因此,在一些实施方案中,rnb在工程化宿主细胞中删除或突变。在其他实施方案中,rna酶ii在内源rna解聚后被热失活。虽然pnp和rnbis都不是宿主细胞存活所必需的,但同时破坏两者可以是致死的。因此,在一些实施方案中,pnp酶和rna酶ii两者都被热失活。将一磷酸核苷磷酸化为三磷酸核苷在将内源rna转化为其单体形式后,并且在将内源核酸酶和磷酸酶热失活后,细胞裂解物中得到的一磷酸核苷(nmp)在它们聚合形成期望的合成rna,例如双链rna或单链rna(例如,mrna或反义rna)之前被磷酸化。该过程是高度依赖能量的,因此该过程需要能量源。磷酸盐通常由高能量磷酸盐源,例如磷酸烯醇丙酮酸,atp或多磷酸盐提供。在一些实施方案中,能量源是直接添加至细胞裂解物的atp。在其他实施方案中,使用atp再生系统提供能量源。例如,多磷酸盐和多磷酸激酶可用于产生atp。其他实例包括使用乙酰磷酸和乙酸激酶产生atp;磷酸肌酸和肌酸激酶产生atp;和磷酸烯醇丙酮酸和丙酮酸激酶产生atp。可以使用其他atp(或其他能量)再生系统。在一些实施方案中,将能量源的至少一种组分添加至细胞裂解物或细胞裂解物混合物中。能量源的“组分”包括产生能量(例如atp)所需的底物和酶。这些组分的非限制性实例包括多磷酸盐、多磷酸激酶、乙酰磷酸盐、乙酸激酶、磷酸肌酸、肌酸激酶、磷酸烯醇丙酮酸盐和丙酮酸激酶。激酶是催化磷酸基团从高能量、贡献磷酸盐的分子(例如atp)转移到特定底物/分子的酶。该过程称为磷酸化,其中底物获得磷酸基团并且高能量atp分子贡献磷酸基团。此酯交换产生磷酸化的底物和adp。在一些实施方案中,本发明的激酶将nmp转化为ndp和ndp转化为ntp。在一些实施方案中,激酶是一磷酸核苷激酶,其催化高能量磷酸盐从atp转移至nmp,产生adp和ndp。表4和5中提供了一磷酸核苷激酶的非限制性实例。如下所讨论,表4和5中列出的酶的热稳定性变体包括在本公开中。在一些实施方案中,细胞裂解物包含一种或多种(或全部)以下四种一磷酸核苷激酶:热稳定性尿苷酸激酶、热稳定性胞苷酸激酶、热稳定性鸟苷酸激酶和热稳定性腺苷酸激酶。在一些实施方案中,ump激酶获自激烈热球菌(例如,seqidno:3或包含与seqidno:3所鉴定的氨基酸序列至少70%相同的氨基酸序列的变体)。在一些实施方案中,cmp激酶获自嗜热栖热袍菌(例如,seqidno:4或包含与seqidno:4所鉴定的氨基酸序列至少70%相同的氨基酸序列的变体)。在一些实施方案中,gmp激酶获自海洋栖热袍菌(例如,seqidno:5或包含与seqidno:5所鉴定的氨基酸序列至少70%相同的氨基酸序列的变体)。在一些实施方案中,amp激酶获自嗜热栖热袍菌(例如,seqidno:6或包含与seqidno:6所鉴定的氨基酸序列至少70%相同的氨基酸序列的变体)。因此,在一些实施方案中,nmp激酶具有由seqidno:3-6中任一种的氨基酸序列鉴定的氨基酸序列。在一些实施方案中,nmp激酶具有与seqidno:3-6中任一种的氨基酸序列至少70%相同的氨基酸序列。例如,nmp激酶可具有与seqidno:3-6中任一种所鉴定的氨基酸序列至少75%,至少80%,至少85%,至少90%,至少95%,至少96%,至少97%,至少98%或至少99%相同的氨基酸序列。应当理解,本公开涵盖本文的任何一种或多种酶以及酶的变体(例如,“ppk2变体”)的用途。变体酶可与参考酶共享一定程度的序列同一性。术语“同一性”是指两个或多个多肽或多核苷酸的序列之间的关系,如通过比较序列所确定的。同一性测量两个或更多个序列中较小的序列之间相同匹配的百分比,其中缺口比对(若有的话)由特定数学模型或计算机程序(例如,“算法”)解决。可以通过已知方法容易地计算相关分子的同一性。“百分比(%)同一性”如其应用于氨基酸或核酸序列时定义为在比对序列并且引入缺口(若必要的话)以实现最大百分比同一性之后候选氨基酸或核酸序列中与第二序列的氨基酸序列或核酸序列中的残基相同的残基(氨基酸残基或核酸残基)的百分比。同一性取决于百分比同一性的计算,但由于计算中引入的缺口和罚分,可以在数值上不同。特定序列的变体可以与该特定参考序列具有至少70%,75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%但是小于100%的序列同一性,如通过本文所述和本领域技术人员已知的序列比对程序和参数所确定的。可以使用数学算法完成序列的比较和两个序列之间的同一性百分比的确定。用于确定同一性的技术在公开可用的计算机程序中编码。用于确定两个序列之间同源性的示例性计算机软件包括但不限于gcg程序包(devereux,j.etal.nucleicacidsresearch,12(1):387,1984)、blast套件(altschul,s.f.etal.nucleicacidsres.25:3389,1997)和fasta(altschul,s.f.etal.j.molec.biol.215:403,1990)。其他技术包括:smith-waterman算法(smith,t.f.etal.j.mol.biol.147:195,1981;needleman–wunsch算法(needleman,s.b.etal.j.mol.biol.48:443,1970;和快速最佳全局序列比对算法(fastoptimalglobalsequencealignmentalgorithm)(fogsaa)(chakraborty,a.etal.scirep.3:1746,2013。表4.一磷酸核苷激酶的实例在一些实施方案中,激酶是二磷酸核苷激酶,其将磷酰基转移至ndp,产生ntp。磷酰基的供体可以是但不限于atp、多磷酸盐聚合物或磷酸烯醇丙酮酸盐。将ndp转化为ntp的激酶的非限制性实例包括二磷酸核苷激酶、多磷酸激酶和丙酮酸激酶。如下所讨论,前述酶的热稳定性变体包括在本公开中。在一些实施方案中,ndp激酶从风产液菌获得(例如,seqidno:9或包含与由seqidno:9鉴定的氨基酸序列至少70%相同的氨基酸序列的变体)。在一些实施方案中,ndp激酶具有与seqidno:9鉴定的氨基酸序列至少70%相同的氨基酸序列。例如,ndp激酶可具有与seqidno:9所鉴定的氨基酸序列至少75%,至少80%,至少85%,至少90%,至少95%,至少96%,至少97%,至少98%或至少99%相同的氨基酸序列。在一些实施方案中,通过多磷酸盐依赖性激酶途径发生nmp向ntp的磷酸化(图2a和2b),其中高能量磷酸盐通过多磷酸激酶(ppk)从多磷酸盐转移至adp。在一些实施方案中。多磷酸激酶属于多磷酸激酶1(ppk1)家族,它将高能量磷酸盐从多磷酸盐转移到adp形成atp。该atp随后被nmp激酶(例如,amp激酶、ump激酶、gmp激酶和cmp激酶)用于将nmp转化为其关联的核糖核苷酸二磷酸(ndp)。此外,atp随后被核苷酸二磷酸激酶用于将ndp转化为ntp。关于示例性酶,参见例如表5和6。在一些实施方案中,多磷酸激酶属于多磷酸激酶2(ppk2)家族。在一些实施方案中,多磷酸激酶属于i类ppk2家族,其将高能量磷酸盐从多磷酸盐转移至ndp以形成ntp。该系统产生的atp用作高能量磷酸盐供体,将nmp转化为ndp。在一些实施方案中,多磷酸激酶属于iii类ppk2家族,其将高能量磷酸盐从多磷酸盐转移至nmp和ndp以形成ntp。在一些实施方案中,单独使用iii类ppk2以从nmp产生ntp。在其他实施方案中,iii类ppk2与其他激酶组合使用。iii类ppk2从adp、amp和多磷酸盐产生atp,其随后被nmp和ndp激酶用于将nmp转化为ntp。如本文提供的使用的ppk2酶的非限制性实例列于表6中(seqidno:8-18)。因此,在一些实施方案中,ppk2酶是热稳定性。例如,ppk2酶可以是热稳定性iii类ppk2酶,其相对于多磷酸盐聚合有利于atp合成,并且将adp和amp两者转化为atp。在一些实施方案中,ppk2酶用于以速率将多磷酸盐(例如六偏磷酸盐)转化为atp,所述速率的范围为例如每小时10至800mm(例如,每小时10、15、20、25、50、75、100、150、200、250、300、350、400、450、500、550、600、650、700、750或800mm)。在一些实施方案中,本公开的rna生物合成方法利用ppk2酶,其包含与seqidno:8-18中任一种所鉴定的氨基酸序列相同的氨基酸序列。在一些实施方案中,ppk2酶包含与seqidno:8-18中任一种所鉴定的氨基酸序列至少70%相同的氨基酸序列。例如,ppk2酶可以包含与seqidno:8-18中任一种所鉴定的氨基酸序列至少75%,至少80%,至少85%,至少90%,至少95%,至少96%,至少97%,至少98%或至少99%相同的氨基酸序列。本公开还包括融合酶。融合酶可以表现出多种活性,每种活性对应于不同酶的活性。例如,可以使用具有一磷酸核苷激酶活性和二磷酸核苷激酶活性两者的融合酶(或任何其他酶),而非使用独立的一磷酸核苷激酶和独立的二磷酸核苷激酶。表5.途径酶的实例enz.#酶名称ec#生物体uniprot#序列标识号1rna酶r3.1.13.-大肠杆菌p21499seqidno:12ppk12.7.4.1伸长热聚球藻q8dma8seqidno:23ump激酶2.7.4.22激烈火球菌q8u122seqidno:34cmp激酶2.7.4.25嗜热栖热袍菌q5sl35seqidno:45gmp激酶2.7.4.8海洋栖热袍菌q9x215seqidno:56amp激酶2.7.4.3嗜热栖热袍菌q72i25seqidno:67ndp激酶2.7.4.6风产液菌o67528seqidno:78rna聚合酶表6.ppk2酶的实例生物体登录号序列标识号红色亚栖热袍菌dsm1279add29239.1seqidno:8meiothermussilvanusdsm9946wp_013159015.1seqidno:9地热异常球菌dsm11300wp_011531362.1seqidno:10伸长热聚球藻bp-1np_682498.1seqidno:11嗜热厌氧绳菌uni-1wp_013558940seqidno:12嗜气暖绳菌dsm14535wp_014433181seqidno:13chlorobaculumtepidumtlsnp_661973.1seqidno:14深海洋栖热袍菌dsm14977wp_013458618seqidno:15roseiflexuscastenholziidsm13941wp_012120763seqidno:16玫瑰弯菌属种rs-1wp_011956376seqidno:17trueperaradiovictrixdsm17093wp_013178933seqidno:18三磷酸核苷聚合成核糖核酸感兴趣的rna的生物合成中的最终步骤是使用例如dna依赖性rna聚合酶将ntp聚合成rna(例如,dsrna或ssrna)终产物。在该方法的该步骤中,设计用于编码感兴趣rna的dna充当合成感兴趣rna的模板。在一些情况下,dna模板可以工程化改造成具有选择性地驱动感兴趣rna转录的转录启动子。在图3a中显示示例性dna模板。dna模板编码三个rna域:有义域(域1)、柔性铰链域(域2)和与有义域互补的域(反义域3)。在dna模板的转录后,反义域结合(杂交)到有义域以形成双链rna发夹茎域和相邻的发夹环域。在图3b-3e中显示dna模板的其他实例。图3b中的dna模板包含互补链上的会聚启动子序列。从每个模板链转录的rna序列在转录后退火。编码为质粒一部分的图3c中的dna模板含有互补链上的会聚启动子序列,以及一个或多个终止子序列以最小化通读转录。编码为质粒一部分的图3d中的dna模板含有驱动互补序列转录的独立启动子-终止子盒,所述互补序列在转录后退火。图3e中的dna模板编码单个rna域。dna依赖性rna聚合酶和rna依赖性rna聚合酶两者的使用产生双链rna终产物。rna的聚合需要ntp、包含转录启动子的dna模板和对转录启动子特异的聚合酶(rna聚合酶)。通常,如本文提供的使用的聚合酶是单亚基聚合酶,对其关联的转录启动子具有高选择性,具有高保真度并且是高效的。聚合酶的实例包括但不限于t7rna聚合酶、t3rna聚合酶和sp6rna聚合酶。噬菌体t7rna聚合酶是dna依赖性rna聚合酶,其对t7噬菌体启动子具有高度特异性。99kd酶在t7启动子控制下从克隆的dna序列催化体外rna合成。噬菌体t3rna聚合酶是dna依赖性rna聚合酶,其对t3噬菌体启动子具有高度特异性。99kd酶在t3启动子下从克隆的dna序列催化体外rna合成。噬菌体sp6rna聚合酶是一种dna依赖性rna聚合酶,其对sp6噬菌体启动子具有高度特异性。98.5kd聚合酶在sp6启动子下从克隆的dna模板催化体外rna合成。t7、t3和sp6聚合酶中的每种在37-40℃是最佳活性的。在一些实施方案中,使用t7、t3和sp6聚合酶的热稳定性变体。热稳定性变体聚合酶通常在高于40℃(或约50-60℃)的温度具有最佳活性。“导致三磷酸核苷产生和三磷酸核苷聚合的条件”,也称为“rna生物合成的条件”,可以由本领域普通技术人员确定,考虑到例如聚合酶活性的最佳条件,包括ph、温度、时间长度和细胞裂解物的盐浓度以及任何外源辅因子。rna生物合成期间细胞裂解物的ph可以具有3.0至8.0的数值。在一些实施方案中,细胞裂解物的ph值为3.0-8.0、4.0-8.0、5.0-8.0、6.0-8.0、7.0-8.0、3.0-7.0、4.0-7.0、5.0-7.0、6.0-7.0、3.0-6.0、4.0-6.0、5.0-6.0、3.0-5.0、3.0-4.0或4.0-5.0。在一些实施方案中,细胞裂解物的ph值为3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5或8.0。在一些实施方案中,rna生物合成期间细胞裂解物的ph值为7.0。rna生物合成期间细胞裂解物的温度可以是15℃至70℃。在一些实施方案中,rna生物合成期间细胞裂解物的温度为15-60℃、15-50℃、15-40℃、15-30℃、25-70℃、25-60℃、25-50℃、25-40℃、30-70℃、30-60℃、30-50℃、40-70℃、40-60℃、40-50℃、50-70℃或50-60℃。在一些实施方案中,rna生物合成期间细胞裂解物的温度为15℃、25℃、32℃、37℃、42℃、45℃、55℃、56℃、57℃、58℃、59℃、60℃、61℃、62℃、63℃、64℃、65℃、66℃、67℃、68℃、69℃或70℃。或在一些实施方案中,rna生物合成期间细胞裂解物的温度为50℃。rna生物合成期间的细胞裂解物可以温育15分钟(min)至72小时(hrs)。在一些实施方案中,rna生物合成期间的细胞裂解物温育30分钟-48小时。例如,rna生物合成期间的细胞裂解物可以温育30min、45min、1hr、2hrs、3hrs、4hrs、5hrs、6hrs、7hrs、8hrs、9hrs、10hrs、11hrs、12hrs、18hrs、24hrs、30hrs、36hrs,42小时或48小时。在一些实施方案中,rna生物合成期间的细胞裂解物温育3小时。在一些实施方案中,rna生物合成期间的细胞裂解物在37℃的温度温育24小时。在一些实施方案中,rna生物合成期间的细胞裂解物在ph7.0在50℃的温度温育2-4小时。一些聚合酶活性可以需要金属离子的存在。因此,在一些实施方案中,将金属离子添加到细胞裂解物中。金属离子的非限制性实例包括mg2+、li+、na+、k+、ni2+、ca2+、cu2+和mn2+。可以使用其他金属离子。在一些实施方案中,可以使用超过一种金属离子。细胞裂解物中金属离子的浓度可以是0.1mm至100mm,或10mm至50mm。在一些实施方案中,细胞裂解物中金属离子的浓度为0.1、0.2、0.5、1.0、1.5、2.0、2.5、3.0、3.5、4.0、4.5、5.0、5.5.、6.0、6.5、7.0、7.5、8.0、8.5、9.0、9.5、10.0、20.0、25.0、30.0、35.0、40.0、45.0、50.0、60.0、70.0、80.0、90.0或100.0mm。在一些实施方案中,将盐添加到细胞裂解物,例如以防止酶聚集。例如,可以将氯化钠、氯化钾、乙酸钠、乙酸钾或其组合添加到细胞裂解物。rna解聚反应期间细胞裂解物中的盐浓度可以是5mm至1m。在一些实施方案中,rna解聚反应期间细胞裂解物中的盐浓度为5mm,10mm,15mm,20mm,25mm,50mm,100mm,150mm,200mm,250mm,500mm,750mm或1m。热稳定性酶本公开的无细胞rna-生物合成方法的一个优点是例如将内源rna转化为合成双链rna所需的所有酶可以(但不需要)在单一工程化细胞中表达。例如,将工程化细胞的克隆群体培养至期望的细胞密度,裂解细胞,在导致内源rna解聚成其单体形式的条件(例如,在30-37℃的温度)下温育,经受足以使内源核酸酶和磷酸酶失活的温度(例如,40-90℃),并在导致rna(例如,dsrna或ssrna)聚合的条件(例如,30-50℃)下温育。为了进行到终产物合成rna,将nmp转化为ndp(例如,一磷酸核苷激酶和/或多磷酸激酶),从ndp到ntp(例如,二磷酸核苷激酶和/或多磷酸激酶),以及从ntp到rna(例如聚合酶)所需的酶应当是热稳定的,以避免在内源核酸酶(和/或外源核酸酶)和磷酸酶的热失活期间的变性。热稳定性是指在相对较高温度抗变性的酶的质量。例如,若酶在42℃的温度变性(失活),则具有相似活性(例如,激酶活性)的酶当其在42℃不变性时认为是“热稳定性的”。若酶(a)在暂时暴露于使其它天然酶变性的高温后保持活性或(b)在暂时暴露于天然酶以低速率发挥功能的中等至高温度后以高速率发挥功能,则酶(例如,激酶或聚合酶)被认为是热稳定的。在一些实施方案中,热稳定性酶在暂时暴露于相对高的温度后保持大于50%的活性(例如,对于从大肠杆菌获得的激酶高于41℃,对于许多rna聚合酶高于37℃),所述相对高的温度在其它情况下会使类似的(非热稳定性)天然酶变性。在一些实施方案中,热稳定性酶在暂时暴露于相对高的温度后保留50-100%的活性,所述相对高的温度在其它情况下会使类似的(非热稳定性)天然酶变性。例如,热稳定性酶在暂时暴露于相对高的温度后可以保留50-90%,50-85%,50-80%,50-75%,50-70%,50-65%,50-60%或50-55%的活性,所述相对高的温度在其它情况下会使类似的(非热稳定性)天然酶变性。在一些实施方案中,热稳定性酶在暂时暴露于相对高的温度后保留25%,30%,35%,40%,45%,50%,55%,60%,65%,70%,75%,80%,85%,90%,95%,98%,99%或100%活性,所述相对高的温度在其它情况下会使类似的(非热稳定性)天然酶变性。在一些实施方案中,在暂时暴露于中等至高的温度(例如,42-80℃)后热稳定性酶的活性大于相似(非热稳定)天然酶的活性(例如,大25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%)。例如,热稳定性激酶的活性可以通过激酶能够磷酸化的nmp或ndp的量来测量。因此,在一些实施方案中,热稳定性激酶,在相对高的温度(例如,42℃)以37℃完成类似转化所需的相同时间量将大于50%的nmp转化为ndp,或大于50%的ndp转化为ntp。在一些实施方案中,热稳定性激酶,在相对高的温度(例如,42℃)以37℃完成类似转化所需的相同时间量将大于60%的nmp转化为ndp,或大于60%的ndp转化为ntp。在一些实施方案中,热稳定性激酶,在相对高的温度(例如,42℃)以37℃完成类似转化所需的相同时间量将大于70%的nmp转化为ndp,或大于70%的ndp转化为ntp。在一些实施方案中,热稳定性激酶,在相对高的温度(例如,42℃)以37℃完成类似转化所需的相同时间量将大于80%的nmp转化为ndp,或大于80%的ndp转化为ntp。在一些实施方案中,热稳定性激酶,在相对高的温度(例如,42℃)以37℃完成类似转化所需的相同时间量将大于90%的nmp转化为ndp,或大于90%的ndp转化为ntp。例如,基于保真度和聚合动力学(例如,聚合速率)评估热稳定性聚合酶的活性。因此,例如,一个单位的热稳定性t7聚合酶可以在高于37℃的温度(例如,50℃)在30分钟内将10纳摩尔的ntp掺入酸不溶性材料中。热稳定性酶(例如,激酶或聚合酶)可以在42℃至80℃或更高的温度保持活性(能够催化反应)。在一些实施方案中,热稳定性酶在42-80℃、42-70℃、42-60℃、42-50℃、50-80℃、50-70℃、50-60℃、60-80℃、60-70℃或70-80℃的温度保持活性。例如,热稳定性酶可在42℃、43℃、44℃、45℃,46℃、47℃、48℃、49℃、50℃、51℃、52℃、53℃、54℃、55℃、55℃、56℃、57℃、58℃、59℃、60℃、61℃、62℃、63℃、64℃、65℃、66℃、67℃、68℃、69℃、70℃、71℃、72℃、73℃、74℃、75℃、76℃、77℃、78℃、79℃、或80℃的温度保持活性。热稳定性酶可在相对高的温度下保持活性15分钟至48小时或更长时间。例如,热稳定性酶可以在相对高的温度下保持活性1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、24、36、42或48小时。热稳定性nmp激酶的非限制性实例列于表5和7中。其他热稳定性激酶包括热稳定性二磷酸核苷激酶、热稳定性丙酮酸激酶和热稳定性多磷酸激酶(参见例如表6)。其他热稳定性激酶包含在本公开中。表7.热稳定性一磷酸核苷激酶的实例。表8中列出了rna聚合酶的非限制性实例。本公开涵盖其它rna聚合酶,包括热稳定性rna聚合酶。表8.rna聚合酶的实例可以通过修饰野生型酶来制备热稳定性rna聚合酶。这些修饰(例如突变)是已知的。例如,变体热稳定性t7rna聚合酶可包括一个或多个下列点突变:v426l、a702v、v795i、s430p、f849i、s633i、f880y、c510r和s767g(ep2377928和ep1261696a1,其各自通过引用并入本文)。在一些实施方案中,变体热稳定性t7rna聚合酶包括v426l,a702v和v795i突变。在一些实施方案中,变体热稳定性t7rna聚合酶包括s430p,f849i,s633i和f880y突变。在一些实施方案中,变体热稳定性t7rna聚合酶包括f880y,s430p,f849i,s633i,c510r和s767g突变。在一些实施方案中,变体热稳定性t7rna聚合酶包括y639v,h784g,e593g和v685a突变。在一些实施方案中,变体热稳定性t7rna聚合酶包括s430p,n433t,s633p,f849i和f880y突变。本公开涵盖其他变体和重组热稳定性聚合酶。在一些实施方案中,使用热稳定性t7聚合酶来产生感兴趣rna。例如,具有1-2%总蛋白浓度的热稳定性t7聚合酶(例如,在40-60℃的温度温育)可用于以大于2g/l/hr(或例如,2g/l/hr-10g/l/hr)的速率合成感兴趣的rna。作为另一个实例,具有3-5%总蛋白浓度的热稳定性t7聚合酶(例如,在40-60℃的温度温育)可用于以大于10g/l/hr(或例如,10g/l/hr-20g/l/hr)的速率合成感兴趣rna。应当理解,虽然本公开的许多实施方案描述了热稳定性聚合酶/酶的使用,但是可以使用其他酶/聚合酶。在一些实施方案中,聚合酶可以外源添加到热失活的细胞裂解物,例如,以补偿热稳定性酶的任何活性降低或丧失。感兴趣的rna本公开的方法用于生物合成感兴趣rna。rna可以是单链或双链的。在一些实施方案中,rna是双链rna干扰分子。例如,感兴趣的rna可以是sirna或发夹rna干扰分子。如上所讨论,感兴趣的rna由dna模板编码,其实例显示在图3a-3e中。使用图3a的模板产生的rna包括有义域(域1)、柔性铰链域(域2)和与有义域互补的域(反义域3)。转录dna模板后,反义域与有义域结合(杂交),以形成双链rna发夹茎域和相邻的发夹环(铰链)域。通过两个互补核酸域(例如,离散的核苷酸序列)彼此结合形成双链发夹茎域。若核酸域(通过watson-crick相互作用碱基对,杂交)彼此结合形成双链核酸,则它们是“互补的”。编码感兴趣rna的dna模板的互补域可以变化,这取决于例如期望的最终产物。互补域可具有例如4至1000个核苷酸或更长的长度。例如,互补域可以具有4至10、4至20、4至30、4至50、4至60、4至70、4至80、4至90、4至100、4至200、4至300、4至400、或4至500、或4至1000个核苷酸的长度。在一些实施方案中,互补域具有15、16、17、18、19、20、21、22、23、24或25个核苷酸的长度。在一些实施方案中,互补域具有4、5、6、7、8、9、10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95或100个核苷酸的长度。还通过两个互补核酸域的结合形成发夹环域。发夹环域是两个互补域之间的居间序列。通常,发夹环域是非特异性的,意味着它不被设计成分子内结合或与另一种核酸结合。发夹环域在互补域结合后形成环样结构,以形成双链发夹茎域。在一些实施方案中,发夹环域具有4至500个核苷酸或更长的长度。例如,发夹环域可以具有4至10、4至20、4至30、4至50、4至60、4至70、4至80、4至90、4至100、4至200、4至300、4至400或4至500个核苷酸的长度。在一些实施方案中,发夹环域具有4、5、6、7、8、9、10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95或100个核苷酸的长度。本公开的“双链rna”包括完整的双链分子,其不包含单链区域(例如,环或突出端),以及部分双链的分子,其包含双链区和单链区(例如,环或突出端)。认为图3a底部描绘的dsrna产物是部分双链分子,而认为图3b底部描绘的dsrna产物是完全双链分子。感兴趣的“单链rna”的实例包括信使rna(mrna)和反义rna。因此,本文提供了合成mrna和其他单链rna分子的方法。这些方法可包括(a)裂解包含rna、解聚rna的酶、热稳定性激酶、热稳定性rna聚合酶的培养工程化细胞,从而产生细胞裂解物,(b)在导致rna解聚的条件下,温育步骤(a)中产生的细胞裂解物,从而产生包含一磷酸核苷的细胞裂解物,(c)将步骤(b)中产生的细胞裂解物加热至使内源核酸酶和磷酸酶失活而不使热稳定性激酶和热稳定性rna聚合酶失活的温度,从而产生包含热失活的核酸酶和磷酸酶的细胞裂解物,和(d)在导致三磷酸核苷产生和三磷酸核苷聚合的条件下在能量源和含有与编码感兴趣rna的核苷酸序列可操作连接的启动子的工程化dna模板的情况下温育(c)中产生的细胞裂解物,从而产生包含感兴趣mrna的细胞裂解物。或者,此类方法可以包括(a)组合从包含内源聚合rna、解聚rna的酶、热稳定性一磷酸核苷(nmp)激酶、热稳定性二磷酸核苷(ndp)激酶、热稳定性ppk2激酶和/或多磷酸盐的工程化细胞获得的细胞裂解物,以产生细胞裂解物混合物,(b)在导致rna解聚的条件下温育步骤(a)中产生的细胞裂解物混合物,从而产生包含一磷酸核苷的细胞裂解物,(c)将步骤(b)中产生的细胞裂解物加热至使磷酸酶和rna酶(以及可能对rna稳定性或聚合保真度有害的任何其他活性,例如天然rna聚合酶,nmp还原酶和/或核苷酶)失活而不会使热稳定性激酶和热稳定性rna聚合酶失活的温度,从而产生包含热失活磷酸酶和rna酶(及其他有害的细胞活性)的细胞裂解物,并且(d)在导致三磷酸核苷的产生和三磷酸核苷的聚合的条件下,在能量源和工程化dna模板的存在下温育步骤(c)中产生的细胞裂解物,从而产生包含mrna的细胞裂解物,所述工程化dna模板含有与编码感兴趣rna的核苷酸序列可操作地连接的启动子。在一些实施方案中,使用dna依赖性rna聚合酶(例如t7rna聚合酶)转录编码含有单个靶域的rna的dna模板,并且得到的rna转录物充当rna依赖性rna聚合酶,例如噬菌体rd6rdrp的模板,以合成互补rna分子,产生dsrna。参见例如图3b。噬菌体φ6是一种双链rna病毒,其感染假单胞菌属的成员。该噬菌体编码rdrp,其能够使用rna模板合成rna,产生dsrna分子。φ6rdrp能够在没有引物分子的情况下聚合rna,因此聚合酶仅需要模板rna(wright,s.etal,2012.journalofvirology.mar;86(5):2837-49;vandijk,aa.,etal,2004.jgenvirol.may;85(pt5),其通过引入并入本文)。其他rna依赖性rna聚合酶(rdrp)包括在本公开中。在一些实施方案中,工程化细胞包含编码感兴趣rna的dna模板。编码rna的dna模板可以掺入工程化细胞的基因组dna中,或者可以将dna模板在质粒上引入工程化细胞中。在其他实施方案中,在感兴趣的rna的生物合成期间(例如,在热失活步骤之后)将dna模板添加至细胞裂解物。在一些实施方案中,细胞裂解物中dna模板的浓度为0.05-1μg/μl。在一些实施方案中,细胞裂解物中dna模板的浓度为0.05μg/μl、0.1μg/μl、0.5μg/μl、1.0μg/μl。如上所讨论,感兴趣的rna终产物的其他实例包括信使rna(mrna)和短/小干扰rna(sirna)(设计用于特异性靶向特定mrna用于降解的合成rna双链体)。在一些实施方案中,rna终产物(生物合成的感兴趣rna)的浓度为至少1g/l至50g/l的细胞裂解物。例如,rna终产物的浓度可以是1,5,10,15,20,25,30,35,40,45或50g/l或更多。在一些实施方案中,设计感兴趣rna以结合感兴趣的靶核酸,并且例如用作治疗剂、预防剂或诊断剂。蛋白酶靶向本公开的工程化细胞可以表达(例如,内源表达)对细胞健康所必需的酶,所述酶可能对核酸(例如rna)的产生具有负面影响。此类酶在本文中称为“靶酶”。例如,由工程化细胞表达的靶酶可以与增加提供给rna生物合成途径的前体的速率的酶竞争底物或辅因子。作为另一个实例,由工程化细胞表达的靶酶可以与作为rna生物合成途径的关键途径进入酶的酶竞争底物或辅因子。作为又一个实例,由工程化细胞表达的靶酶可以与提供rna生物合成途径的底物或辅因子的酶竞争底物或辅因子。为了消除或减少此种负面影响,可以修饰靶酶以在其蛋白质序列中包括位点特异性蛋白酶识别序列,使得靶酶可以被“靶向”和切割以在rna产生期间失活(参见例如2012年3月1日公布的美国公开号2012/0052547a1;和2015年2月12日公开的国际公开号wo2015/021058a2,其各自通过引用并入本文。含有位点特异性蛋白酶识别序列的靶酶的切割源自与关联位点特异性蛋白酶的接触,所述关联位点特异性蛋白酶在细胞生长期期间(例如在培养工程化细胞时)在细胞周质中被隔离(与靶酶分开),并且在rna生产期期间(例如在细胞裂解以生成细胞裂解物后)与靶酶接触。因此,在一些实施方案中,本公开的工程化细胞包含(i)编码靶酶的工程化核酸,所述靶酶负面影响rna产生速率并且包括靶酶的蛋白质序列中的位点特异性蛋白酶识别序列,和(ii)编码位点特异性蛋白酶的工程化核酸,所述位点特异性蛋白酶切割靶酶的位点特异性蛋白酶识别序列,并包括周质靶向序列。该周质靶向序列负责将位点特异性蛋白酶隔离到细胞的周质空间,直至细胞裂解。下文提供了周质靶向序列的实例。可根据本发明使用的蛋白酶的实例包括但不限于丙氨酸羧肽酶、从蜜环菌(armillariamellea)获得的蛋白酶、虾红素、细菌亮氨酰氨肽酶、癌促凝血剂、组织蛋白酶b、梭菌蛋白酶,胞质溶胶丙氨酰氨肽酶、弹性蛋白酶、内蛋白酶brg-c、肠激酶、胃亚蛋白酶(gastricsin)、明胶酶,gly-x羧肽酶、甘氨酰内肽酶,人鼻病毒3c蛋白酶、hypoderminc、iga特异性丝氨酸内肽酶、亮氨酰氨肽酶、亮氨酰内肽酶、lysc、溶酶体pro-x羧肽酶、赖氨酰氨肽酶、甲硫氨酰氨肽酶、粘球菌(myxobacter)、nardilysin、胰内肽酶e、picornain2b、picornain3c、内肽酶原(proendopeptidase)、脯氨酰氨肽酶、前蛋白转化酶i、前蛋白转化酶ii、russellysin、糖胃蛋白酶(saccharopepsin)、semenogelase,t-纤溶酶原激活物、凝血酶、组织激肽释放酶、从烟草蚀刻病毒(tev)获得的蛋白酶、togavirin、色氨酰氨肽酶、u-纤溶酶原激活物、v8,venombinb、venombinbb和xaa-pro氨肽酶。周质靶向核酸(例如,rna)生物合成途径的酶可包括至少一种对细胞的健康(例如,存活力)具有负面影响的酶。为了消除或减少此种负面影响,可以修饰酶以包括重新定位序列,使得酶重新定位到细胞或细胞外区室,在那里它不是天然定位的,并且酶不对细胞的健康产生负面影响(参见例如公开号us-2011-0275116-a1,2011年11月10日公开,通过引用并入本文)。例如,生物合成途径的酶可以重新定位到细胞的周质空间。因此,在一些实施方案中,本公开的工程化细胞包含与周质靶向序列连接的核酸(例如rna)生物合成途径的至少一种酶。“周质靶向序列”是将与其连接的蛋白质靶向到细胞周质的氨基酸序列。与周质靶向序列连接的蛋白质将被隔离在表达蛋白质的细胞的周质中。例如,周质靶向序列可以源自细菌分泌蛋白的n-末端。序列长度从约15至约70个氨基酸变化。周质靶向序列的一级氨基酸序列可以变化,但通常具有共同的结构,包括以下组分:(i)n-末端部分具有可变长度并且通常带有净正电荷;(ii)后面是约6至约15个氨基酸的中心疏水核心;和(iii)最终组分包括4-6个氨基酸,其定义信号肽酶的切割位点。在一些实施方案中,本公开的周质靶向序列可以源自革兰氏阴性细菌中分泌的蛋白质。分泌性蛋白质可以由细菌编码,或由感染细菌的噬菌体编码。分泌性蛋白的革兰氏阴性细菌来源的实例包括但不限于埃希氏菌属、假单胞菌属(pseudomonas)、克雷伯氏菌属(klebsiella)、沙门氏菌属(salmonella)、柄杆菌属(caulobacter)、甲基单胞菌属(methylomonas)、醋杆菌属(acetobacter)、无色杆菌属(achromobacter)、不动杆菌属(acinetobacter)、气单胞菌属(aeromonas)、土壤杆菌属(agrobacterium)、产碱杆菌属(alcaligenes)、固氮菌属(azotobacter)、伯霍尔德杆菌属(burkholderia)、柠檬酸杆菌属(citrobacter)、丛毛平胞菌属(comamonas)、肠杆菌属(enterobacter)、欧文氏菌属(erwinia)、根瘤菌属(rhizobium)、弧菌属(vibrio)和黄单胞菌属(xanthomonas)。根据本公开使用的周质靶向序列的实例包括但不限于选自下组的序列:mkiktgarilalsalttmmfsasala(seqidno:19);mkqstialallpllftpvtka(seqidno:20);mmitlrklplavavaagvmsaqama(seqidno:21);mnkkvltlsavmasmlfgaaaha(seqidno:22);mkyllptaaagllllaaqpama(seqidno:23);mkkiwlalaglvlafsasa(seqidno:24);mmtkikllmliifyliisasaha(seqidno:25);mkqalrvafgflilwasvlha(seqidno:26);mrvllflllslfmlpafs(seqidno:27);和manndlfqasrrrflaqlggltvagmlgpslltprrata(seqidno:28)。工程化细胞本公开的工程化细胞通常包含生物合成rna所需的至少一种、大多数或全部酶活性。“工程化细胞”是包含至少一种工程化(例如,重组或合成)核酸的细胞,或者以其他方式被修饰使得它们在结构上和/或功能上与其天然存在的对应物不同的细胞。因此,认为含有工程化核酸的细胞是“工程化细胞”。在一些实施方案中,本公开的工程化细胞包含rna、解聚rna的酶、热稳定性激酶和/或热稳定性聚合酶。在一些实施方案中,工程化细胞还包含dna模板,其含有与编码感兴趣rna的核苷酸序列可操作地连接的启动子。在一些实施方案中,工程化细胞表达选择标志物。选择标志物通常用于选择在转染细胞后(或者在用于将外源核酸引入细胞中的其他程序之后)吸收并表达工程化核酸的工程化细胞。因此,编码产物的核酸也可编码选择标志物。选择标志物的实例包括但不限于编码增加或降低对抗生素的抗性或敏感性的蛋白质的基因(例如,氨苄青霉素抗性基因、卡那霉素抗性基因、新霉素抗性基因、四环素抗性基因和氯霉素抗性基因)或其他化合物。选择标志物的其他实例包括但不限于编码蛋白质的基因,所述蛋白质使得细胞能够在缺乏其他情况下必需的营养物的培养基中生长(营养缺陷型标志物)。可以根据本公开使用其他选择标志物。若在细胞中产生由核酸(例如,工程化核酸)编码的产物,则工程化细胞“表达”产物。本领域已知基因表达是指使用核酸形式的遗传指令合成产物,如蛋白质(例如酶)的过程。工程化细胞可以是原核细胞或真核细胞。在一些实施方案中,工程化细胞是细菌细胞、酵母细胞、昆虫细胞、哺乳动物细胞或其他类型的细胞。本公开的工程化细菌细胞包括但不限于工程化的埃希氏菌属种(escherichiaspp.)、链霉菌属种(streptomycesspp.)、zymonas属种、醋杆菌属种(acetobacterspp.)、柠檬酸杆菌属种(citrobacterspp.)、集胞藻属种(synechocystisspp.)、根瘤菌属种(rhizobiumspp.)、梭菌属种(clostridiumspp.)、棒状杆菌属种(corynebacteriumspp.)、链球菌属种(streptococcusspp.),黄单胞菌属种(xanthomonasspp.)、乳杆菌属种(lactobacillusspp.)、乳杆菌属种(lactococcusspp.)、芽孢杆菌属种(bacillusspp.)、产碱杆菌属种(alcaligenesspp.)、假单胞菌属种(pseudomonasspp.)、气单胞菌属种(aeromonasspp.)、固氮菌属种(azotobacterspp.)、丛毛平胞菌属种(comamonasspp.)、分枝杆菌属种(mycobacteriumspp.)、红球菌属种(rhodococcusspp.)、葡糖杆菌属种(gluconobacterspp.)、罗尔斯通氏菌属种(ralstoniaspp.)、酸硫杆状菌属种(acidithiobacillusspp.)、小月菌属种(microlunatusspp.),地杆菌属种(geobacterspp.)、土芽孢杆菌属种(geobacillusspp.)、节杆菌属种(arthrobacterspp.)、黄杆菌属种(flavobacteriumspp.)、沙雷菌属种(serratiaspp.)、糖多孢菌属种(saccharopolysporaspp.)、栖热菌属种(thermusspp.)、狭长平胞属种(stenotrophomonasspp.)、色杆菌属种(chromobacteriumspp.)、大豆根瘤菌属种(sinorhizobiumspp.)、糖多孢菌属种(saccharopolysporaspp.)、土壤杆菌属种(agrobacteriumspp.)和泛菌属种(pantoeaspp.)。本公开的工程化酵母细胞包括但不限于工程化酵母属种(saccharomycesspp.)、裂殖酵母属(schizosaccharomyces)、汉逊酵母属(hansenula)、假丝酵母属(candida)、克鲁维酵母属(kluyveromyces)、耶氏酵母(yarrowia)和毕赤酵母属(pichia)。在一些实施方案中,本公开的工程化细胞是工程化的大肠杆菌细胞、枯草芽孢杆菌(bacillussubtilis)细胞、恶臭假单胞菌(pseudomonasputida)细胞、酿酒酵母(saccharomycescerevisae)细胞或短乳杆菌(lactobacillusbrevis)细胞。在一些实施方案中,本公开的工程化细胞是工程化的大肠杆菌细胞。工程化核酸。术语“核酸”指共价连接在一起的至少两个核苷酸,并且在一些情况下可以含有磷酸二酯键(例如,磷酸二酯“主链”)。核酸(例如,核酸的组分或部分)可以是天然存在的或工程化的。“天然存在的”核酸存在于自然界中在没有人为干预的情况下存在的细胞中。“工程化核酸”包括重组核酸和合成核酸。“重组核酸”指通过连接核酸分子(例如,来自相同物种或来自不同物种)构建并且通常可以在活细胞中复制的分子。“合成核酸”指以生物合成,以化学方式或通过其它手段合成或扩增的分子。合成核酸包括以化学方式或以其它方式修饰但可与天然存在的核酸分子碱基配对的那些分子。重组和合成核酸还包括由前述任一种的复制产生的那些分子。应当理解,工程化核酸可以含有天然存在的核酸的部分,但是作为整体,工程化核酸不天然存在并且需要人为干预。在一些实施方案中,编码本公开的产物的核酸是重组核酸或合成核酸。在其它实施方案中,编码产物的核酸是天然存在的。如本文提供的,编码rna的工程化核酸可以与“启动子”可操作地连接,所述“启动子”是核酸的控制区,在该控制区处控制核酸的剩余部分的转录起始和速率。启动子驱动其调节的核酸的表达或驱动该核酸的转录。启动子可以是与基因或序列天然相关的启动子,其可以通过分离位于给定基因或序列的编码区段上游的5'非编码序列而获得。此类启动子可称为“内源的”。在一些实施方案中,编码核酸序列可以位于重组或异源启动子的控制下,所述重组或异源启动子是指在其天然环境中通常不与编码序列相关的启动子。此类启动子可包括其他基因的启动子;从任何其他细胞分离的启动子;和非“天然存在”的合成启动子或增强子,诸如例如含有不同转录调节区的不同元件和/或通过本领域已知的基因工程化方法得到的改变表达的突变的那些。除了合成产生启动子和增强子的核酸序列之外,可以使用重组克隆和/或核酸扩增技术,包括聚合酶链式反应(pcr)产生序列。当启动子相对于其调节的核酸处于正确功能位置和取向以控制(“驱动”)该核酸的转录起始和/或表达时,认为启动子是“可操作地连接的”。本公开的工程化核酸可含有组成型启动子或诱导型启动子。“组成型启动子”是指在细胞中始终有活性的启动子。“诱导型启动子”是指当在诱导剂或诱导试剂的存在下,受到诱导剂或诱导试剂影响或由诱导剂或诱导试剂接触,或在不存在引起抑制的因子的情况下活化时起始或增强转录活性的启动子。根据本公开使用的诱导型启动子包括本文所述或本领域普通技术人员已知的任何诱导型启动子。诱导型启动子的实例包括但不限于化学/生物化学调节和物理调节的启动子,例如醇调节的启动子、四环素调节的启动子、类固醇调节的启动子、金属调节的启动子、发病机制调节的启动子、温度/热诱导型、磷酸盐调节的(例如,phoa)和光调节的启动子。诱导剂或诱导试剂可以是内源的或通常外源的条件(例如光)、化合物(例如化学或非化学化合物)或蛋白质,其以使得在调节自诱导型启动子的转录活性中有活性的方式接触诱导型启动子。因此,“调节核酸转录的信号”是指作用于诱导型启动子的诱导剂信号。根据所使用的调节系统,调节转录的信号可以激活或失活转录。转录的激活可以涉及直接作用于启动子以驱动转录或通过使阻止启动子驱动转录的阻遏物失活而间接作用于启动子。相反,转录的失活可以涉及直接作用于启动子以防止转录或通过激活阻遏物,所述阻遏物然后作用于启动子而间接作用于启动子。可以使用本领域已知的任何手段将工程化核酸导入宿主细胞中,包括但不限于转化、转染(例如,化学(例如,磷酸钙、阳离子聚合物或脂质体)或非化学(例如电穿孔、声穿孔、impalefection、光转染、水动力学))和转导(例如,病毒转导)。由天然存在的细胞内核酸编码的酶或其他蛋白质可称为“内源酶”或“内源蛋白质”。细胞培养物和细胞裂解物通常,培养工程化细胞。“培养”是指细胞在受控条件下,通常在其自然环境之外培养的过程。例如,工程化细胞如工程化细菌细胞可以作为液体营养肉汤,也称为液体“培养基”中的细胞悬浮液培养。常用的细菌大肠杆菌生长培养基的实例包括但不限于lb(lysogenybroth)miller肉汤(broth)(1%nacl):1%蛋白胨、0.5%酵母提取物和1%nacl;lb(lysogenybroth)lennox肉汤(0.5%nacl):1%蛋白胨、0.5%酵母提取物和0.5%nacl;sob培养基(superoptimalbroth):2%蛋白胨、0.5%酵母提取物、10mmnacl、2.5mmkcl、10mmmgcl2、10mmmgso4;soc培养基(具有分解代谢阻遏物的superoptimalbroth):sob+20mm葡萄糖;2xyt肉汤(2x酵母提取物和胰蛋白胨):1.6%蛋白胨、1%酵母提取物和0.5%nacl;tb(terrificbroth)培养基:1.2%蛋白胨、2.4%酵母提取物、72mmk2hpo4、17mmkh2po4和0.4%甘油;和sb(superbroth)培养基:3.2%蛋白胨、2%酵母提取物和0.5%nacl和/或korz培养基(korz,dj等人,1995)。高密度细菌大肠杆菌生长培养基的实例包括但不限于dnagrotm培养基、progrotm培养基、autoxtm培养基、detoxtm培养基、induxtm培养基、和secprotm培养基。在一些实施方案中,在导致酶或核酸表达的条件下培养工程化细胞。此种培养条件可取决于所表达的特定产物和期望的产物量。在一些实施方案中,工程化细胞在30℃至40℃的温度培养。例如,工程化细胞可以在30℃、31℃、32℃、33℃、34℃、35℃、36℃、37℃、38℃、39℃或40℃的温度下培养。通常,工程化细胞如工程化的大肠杆菌在37℃的温度培养。在一些实施方案中,将工程化细胞培养12小时至72小时或更长的时间段。例如,可以将工程化细胞培养12、18、24、30、36、42、48、54、60、66或72小时的时间段。通常,将工程化细胞如工程化细菌细胞培养12至24小时的时间段。在一些实施方案中,将工程化细胞在37℃的温度培养12至24小时。在一些实施方案中,将工程化细胞培养(例如,在液体细胞培养基中)至5至200的光密度,如在600nm波长测量(od600)。在一些实施方案中,将工程化细胞培养至5、10、15、20、25、50、75、100、150或200的od600。在一些实施方案中,将工程化细胞培养至1x108(od<1)至2x1011(od~200)个活细胞/ml细胞培养基的密度。在一些实施方案中,将工程化细胞培养至1x108、2x108、3x108、4x108、5x108、6x108、7x108、8x108、9x108、1x109、2x109、3x109、4x109、5x109、6x109、7x109、8x109、9x109、1x1010、2x1010、3x1010、4x1010、5x1010、6x1010、7x1010、8x1010、9x1010、1x1011、或2x1011个活细胞/ml的密度(转换因子:od1=8x108个细胞/ml)。在一些实施方案中,工程化细胞在生物反应器中培养。生物反应器简单地指培养细胞的容器,例如培养瓶/皿或可以是一次使用(一次性)、可高压灭菌或可灭菌的袋。生物反应器可以由玻璃制成,或者它可以是基于聚合物的,或者它可以由其他材料制成。生物反应器的实例包括但不限于搅拌罐(例如,良好混合的)生物反应器和管状(例如,活塞流)生物反应器、空气提升生物反应器、膜搅拌罐、旋转过滤搅拌槽、振动混合器(vibromixer)、流化床反应器和膜生物反应器。操作生物反应器的方式可以是分批或连续过程,并且将取决于所培养的工程化细胞。当连续补料并且从系统取出进料和产物流时,生物反应器是连续的。分批生物反应器可以具有连续的再循环流动,但是无营养物或产物收获物的连续补料。对于间歇-收获和补料-分批(或分批补料)培养物,将细胞以较低的活细胞密度在培养基中接种,所述培养基在组成上类似于分批培养基。允许细胞在基本上没有外部操作的情况下指数生长,直到营养物略微消减并且细胞接近稳定的生长期。在此点时,为了间歇-收获分批-补料过程,可以收获细胞和产物的部分,并用新鲜培养基补充取出的培养基。可以将此过程重复几次。为了生成重组蛋白和抗体,可以使用补料-分批方法。虽然细胞以指数生长,但营养物在消减,连续或间歇地添加浓缩的补料培养基(例如,10-15倍浓缩的基础培养基)以提供额外的营养物,允许细胞浓度和生产阶段长度的进一步增加。可以在不取出培养基(肉汤)的情况下与细胞浓度成比例添加新鲜培养基。为了容纳培养基添加,以比生物反应器的全容量(例如,最大体积的约40%至50%)低得多的体积开始补料分批培养。本公开的一些方法涉及rna(例如ssrna或dsrna)的大规模生产。对于大规模生产方法,工程化细胞可以在5升(l)至250,000l或更多体积的液体培养基中培养。在一些实施方案中,工程化细胞可以在大于(或等于)10l、100l、1000l、10000l或100000l的体积的液体培养基中培养。在一些实施方案中,工程化细胞在5l、10l、15l、20l、25l、30l、35l、40l、45l、50l、100l、500l、1000l、5000l、10000l、100000l、150000l、200000l、250000l或更多体积的液体培养基中培养。在一些实施方案中,工程化细胞可以在5l至10l,5l至15l,5l至20l,5l至25l,5l至30l,5l至35l,5l至40l,5l至45l,10l至15l,10l至20l,10l至25l,20l至30l,10l至35l,10l至40l,10l至45l,10l至50l,15l至20l,15l至25l,15l至30l,15l至35l,15l至40l,15l至45l的体积的液体培养基中培养。在一些实施方案中,工程化细胞可以在100l至300000l,100l至200000l,或100l至100000l的体积的液体培养基中培养。通常,培养工程化细胞之后裂解细胞。“裂解”是指细胞被破坏的过程,例如,通过病毒,酶促,机械或渗透机制。“细胞裂解物”是指含有裂解细胞(例如裂解的工程化细胞)内容物的流体,包括例如细胞器,膜脂,蛋白质,核酸和倒置膜囊泡。如本文提供的,可以通过裂解任何工程化细胞群来产生本公开的细胞裂解物。细胞裂解的方法,称为“裂解”,是本领域已知的,其中任何一种都可以根据本发明使用。此类细胞裂解方法包括但不限于物理裂解,例如均质化。细胞裂解可以干扰仔细控制的细胞环境,导致蛋白质降解和通过不受调节的内源蛋白酶和磷酸酶的修饰。因此,在一些实施方案中,蛋白酶抑制剂和/或磷酸酶抑制剂可以在裂解前添加到细胞裂解物或细胞,或者可以通过热失活、基因失活或蛋白酶靶向除去这些活性。在一些实施方案中,细胞裂解物可以与至少一种营养物组合。例如,细胞裂解物可以与na2hpo4、kh2po4、nh4cl、nacl、mgso4、cacl2组合。其他营养素的实例包括但不限于硫酸镁、氯化镁、乳清酸镁、柠檬酸镁、磷酸二氢钾、磷酸氢二钾、磷酸三钾、磷酸二氢钠、磷酸氢二钠、磷酸三钠、磷酸二氢铵、磷酸氢二铵、硫酸铵、氯化铵和氢氧化铵。在一些实施方案中,细胞裂解物可以与至少一种辅因子组合。例如,细胞裂解物可以与二磷酸腺苷(adp)、三磷酸腺苷(atp)、烟酰胺腺嘌呤二核苷酸(nad+)或酶活性所需的其他非蛋白质化合物(例如无机离子和辅酶)组合。在一些实施方案中,细胞裂解物在导致rna解聚的条件下温育。在一些实施方案中,细胞裂解物在导致ssrna或dsrna产生的条件下温育。用于单一反应的细胞裂解物的体积可以变化。在一些实施方案中,细胞裂解物的体积为0.001至250m3。例如,细胞裂解物的体积可以是0.001m3、0.01m3、0.1m3、1m3、5m3、10m3、15m3、20m3、25m3、30m3、35m3、40m3、45m3、50m3、55m3、60m3、65m3、70m3、75m3、80m3、85m3、90m3、95m3、100m3、105m3、110m3、115m3、120m3、125m3、130m3、135m3、140m3、145m3、150m3、155m3、160m3、165m3、170m3、175m3、180m3、185m3、190m3、195m3、200m3、205m3、210m3、215m3、220m3、225m3、230m3、235m3、240m3、245m3或250m3。在一些实施方案中,细胞裂解物的体积为25m3至250m3、50m3至250m3、或100m3至250m3。下游加工在一些实施方案中,本文提供的方法和系统产生浓度为1-50g/l(例如,30、35、40、45或50g/l)的rna(例如,dsrna,ssrna)产物。下游加工将纯度增加至按重量计多达99%(例如,75、80、85、90、95、96、97、98或99%)dsrna。图4中显示了下游加工的实例,开始于添加蛋白质沉淀剂(例如乙酸铵),然后进行盘式堆叠离心(dsc)以从产物流中除去蛋白质、脂质和一些dna。然后实施超滤以除去盐和体积。对产物流添加氯化锂导致rna产物沉淀,其随后使用盘式堆叠离心分离从大量液体中分离,例如,产生约80%纯度的rna产物流。进一步的层析精制产生约99%纯的产物。其它实施方案以下编号的段落1-46涵盖本公开的另外的实施方案:1.生物合成核糖核酸(rna)的无细胞方法,该方法包括:(a)裂解包含rna、解聚rna的酶、热稳定性激酶、热稳定性rna聚合酶的培养的工程化细胞,从而产生细胞裂解物;(b)在导致rna解聚的条件下温育步骤(a)中产生的细胞裂解物,从而产生包含一磷酸核苷的细胞裂解物;(c)将步骤(b)中产生的细胞裂解物加热至使内源核酸酶和磷酸酶失活而不使热稳定性激酶和热稳定性rna聚合酶失活的温度,从而产生包含热失活核酸酶和磷酸酶的细胞裂解物;和(d)在导致三磷酸核苷产生和三磷酸核苷聚合的条件下,在能量源和工程化dna模板存在下温育(c)中产生的细胞裂解物,从而产生包含感兴趣rna的细胞裂解物,所述工程化dna模板含有与编码感兴趣rna的核苷酸序列可操作地连接的启动子。2.段落1的方法,其中能量源是多磷酸盐,多磷酸激酶,或多磷酸盐和多磷酸激酶。3.段落1或2的方法,其中所述培养的工程化细胞包含工程化dna模板。4.段落1或2的方法,其中将工程化dna模板添加到步骤(d)的细胞裂解物中。5.段落1-4中任一段的方法,其中将atp再生系统添加到步骤(d)的细胞裂解物中。6.段落1的方法,其中所述培养的工程化细胞还包含热稳定性多磷酸激酶。7.段落1-6中任一段的方法,其中步骤(a)的工程化细胞的rna是内源rna。8.段落1-7中任一段的方法,其中所述rna包含核糖体rna、信使rna、转移rna或其组合。9.段落1-8中任一段的方法,其中所述培养的工程化细胞包含至少两种解聚rna的酶。10.段落1-9中任一段的方法,其中解聚rna的酶选自:s1核酸酶、核酸酶p1、rna酶ii、rna酶iii、rna酶r、rna酶ji、nuca、pnp酶、rna酶t、rna酶e、rna酶g及其组合。11.段落10的方法,其中解聚rna的酶是核酸酶p1。12.段落1-11中任一段的方法,其中步骤(b)的细胞裂解物包含mg2+螯合剂。13.段落12的方法,其中所述mg2+螯合剂是乙二胺四乙酸(edta)。14.段落13的方法,其中edta的浓度为0.1mm至25mm。15.段落14的方法,其中edta的浓度为8mm。16.段落1-15中任一段的方法,其中所述热稳定性激酶包含热稳定性一磷酸核苷激酶。17.段落16的方法,其中所述热稳定性一磷酸核苷激酶选自下组:热稳定性尿苷酸激酶、热稳定性胞苷酸激酶、热稳定性鸟苷酸激酶和热稳定性腺苷酸激酶。18.段落17的方法,其中稳定的一磷酸核苷激酶选自下组:由pyrh基因(pfpyrh)编码的热稳定性激烈火球菌尿苷酸激酶、由adk基因(tthadk)编码的热稳定性嗜热栖热袍菌腺苷酸激酶、由cmk基因(tthcmk)编码的热稳定性嗜热栖热袍菌胞苷酸激酶和由gmk基因(pfgmk)编码的热稳定性海洋栖热袍菌鸟苷酸激酶。19.段落1-18中任一段的方法,其中所述热稳定性激酶包含热稳定性二磷酸核苷激酶。20.段落19的方法,其中所述热稳定性二磷酸核苷激酶选自下组:热稳定性核苷磷酸激酶、热稳定性丙酮酸激酶和热稳定性多磷酸激酶。21.段落20的方法,其中所述热稳定性二磷酸核苷激酶中的至少一种是由ndk基因编码的热稳定性风产液菌酶。22.段落1-21中任一段的方法,其中所述细胞包含热稳定性一磷酸核苷激酶和热稳定性二磷酸核苷激酶。23.段落1-22中任一段的方法,其中所述培养的工程化细胞包含热稳定性尿苷酸激酶、热稳定性胞苷酸激酶、热稳定性鸟苷酸激酶、热稳定性腺苷酸激酶和热稳定性多磷酸激酶。24.段1-23中任一项的方法,其中所述热稳定性rna聚合酶是热稳定性dna依赖性rna聚合酶。25.段落24的方法,其中所述dna依赖性rna聚合酶选自下组:热稳定性t7rna聚合酶、热稳定性sp6rna聚合酶和热稳定性t3rna聚合酶。26.段落25的方法,其中所述dna依赖性rna聚合酶是热稳定性t7rna聚合酶。27.段落1-26中任一段的方法,其中步骤(c)中的温度为至少50℃。28.段落27的方法,其中步骤(c)中的温度为50℃-80℃。29.段落1-28任一项的方法,其中步骤(c)包括加热细胞裂解物达至少15分钟。30.段落1-29中任一段的方法,其中步骤(c)包括将所述细胞裂解物加热至至少65℃的温度达15分钟。31.段落1-30中任一段的方法,其中步骤(d)中的三磷酸核苷以15-30mm/小时的速率产生。32.段落1-31中任一段的方法,其中步骤(d)中产生的感兴趣rna是双链rna。33.段落1-32中任一段的方法,其中步骤(d)中产生的感兴趣rna是rna干扰分子。34.段落1-33中任一段的方法,其中步骤(d)中产生的感兴趣rna是含有通过铰接域连接的互补域的mrna。35.段落1-34中任一段的方法,其中步骤(d)中产生的感兴趣rna以至少4g/l、至少6g/l、至少6g/l或至少10g/l的浓度产生。36.段落35的方法,其进一步包括纯化双链rna。37.段落36的方法,其中所述纯化步骤包括将步骤(d)的细胞裂解物与蛋白质沉淀剂组合并除去沉淀的蛋白质、脂质和dna。38.通过段落1-37中任一段的方法产生的细胞裂解物。39.包含rna、解聚rna的酶、热稳定性激酶和热稳定性rna聚合酶的工程化细胞。40.段落39的工程化细胞,所述工程化细胞进一步包含工程化dna模板,所述工程化dna模板含有与编码感兴趣rna的核苷酸序列可操作地连接的启动子。41.段落39或40的工程化细胞的群体。42.方法,其包括:在细胞培养基中维持段落39的工程化细胞。43.段落42的方法,其进一步包括裂解培养的工程化细胞以产生细胞裂解物。44.段落43的方法,其进一步包括在导致rna解聚的条件下温育所述细胞裂解物以产生包含一磷酸核苷的细胞裂解物。45.段落44的方法,其进一步包括将所述细胞裂解物加热至使内源核酸酶和磷酸酶失活而不使热稳定性激酶和热稳定性rna聚合酶失活的温度,以产生包含热失活核酸酶和磷酸酶的细胞裂解物。46.段落45的方法,其进一步包括在导致三磷酸核苷产生和三磷酸核苷聚合的条件下,在能量源和工程化dna模板存在下温育包含热失活的核酸酶和磷酸酶的细胞裂解物,从而产生包含感兴趣rna的细胞裂解物,所述工程化dna模板含有与编码感兴趣rna的核苷酸序列可操作地连接的启动子。实施例实施例1核酸酶向下选择(downselection)为了鉴定用于消化裂解物rna的最佳核酸酶,使用商品化酶进行一系列筛选实验,所述商品化酶基于其产生5'-nmp或寡核苷酸的能力选择。首先使用纯化的大肠杆菌rna和由制造商推荐的反应条件测定这些酶的活性,其中通过酸溶性核苷酸的释放监测rna解聚。在这些条件下,四种核酸酶表明高于背景的解聚活性。充当阳性对照的内切核酸酶benzonase和rna酶a产生rna至酸溶性核苷酸的立即转化(图5a)。用外切核酸酶p1和rna酶r处理rna产生rna至酸溶性核苷酸的时间依赖性转化,其中rna酶r在2小时内达到接近100%的解聚。剩余的核酸酶(terminator外切核酸酶、rna酶iii和rna酶t)在此测定法中不产生可检测的解聚。通过lc-ms的后续分析揭示在用rna酶r和核酸酶p1,而非benzonase或rna酶a处理的样品中的nmp释放(图5b)。这些结果表明rna酶r和核酸酶p1可能适合于将裂解物rna解聚成5'-nmp。出于几个原因,选择rna酶r进行进一步研究,包括其缺乏dna酶活性、其降解dsrna和结构化rna的能力和其持续性3'→5'外切核酸酶活性。裂解物中的rna解聚然后,对rna酶r测试其在细菌裂解物中解聚内源rna的能力。在这些实验中,将纯化的rna酶r(0.5mg/ml终浓度)添加到裂解物(50%终浓度),并通过uplc定量游离核苷酸。图6中显示了代表性实验。对裂解物添加纯化的rna酶r导致从裂解物rna快速释放5'-nmp,在5-10分钟后释放出最大的nmp。在快速解聚的此初始阶段后,nmp浓度稳定,然后开始缓慢下降。内源rna酶活性也导致5'-nmp释放,尽管以低得多的速率。重要的是,rna酶r添加不增加从rna的2'或3'nmp释放的速率,这与其已知的作用机制一致。在多个独立实验中,对裂解物添加rna酶r导致在5-10分钟内以超过200mm/hr的速率将68%的裂解物rna转化为5'-nmp。为了评估rna酶r表达的毒性,构建两种细菌菌株。一种菌株包括用空蛋白质表达载体转化的基础菌株(gl16-170),而另一种菌株包括用编码rna酶r的相同蛋白质表达载体转化的gl16-170。这两种菌株在分批条件下在1l生物反应器中培养,于od600=20诱导,并在葡萄糖耗尽前收获。诱导产生rna酶r的强表达(图7a),生长速率没有可检测的变化(图7b)。在裂解和50%稀释后,表达rna酶r的菌株表现出rna快速解聚成酸溶性核苷酸(图7c),指示过表达的rna酶r是有功能的。值得注意的是,通过对反应添加纯化酶提供的额外rna酶r活性不增加解聚速率或酸溶性核苷酸的产率,这表明过表达的rna酶r在裂解和稀释后是完全有活性的。接下来,在高密度裂解物中评估过表达的rna酶r的活性。已知稳定核糖体结构并针对核酸酶保护rrna的mg2+也是rna酶r活性需要的(以少量)。因此,在不同浓度的edta存在下测量解聚速率(图8)。来自空载体菌株的裂解物表现出对edta不敏感的相对缓慢的解聚速率,而具有过表达的rna酶r的裂解物随着edta浓度的增加表现出更高的解聚速率,于8mmedta具有最大速率。高于8mm,解聚速率降低,可能是由于mg2+依赖性rna酶r的失活。总之,这些结果表明过表达的rna酶r是无毒的并且可以在裂解后活化。nmp在裂解物中的稳定性在rna解聚后,在聚合成dsrna之前将所得的nmp合并物逐渐磷酸化为ntp。有害的酶活性(如nmp降解成核苷并随后水解成糖和碱基)负面影响dsrna产率。因此,在裂解物中评估个别的nmp的稳定性。通过对裂解物添加同位素标记的“重”nmp(hamp、hcmp、hump和hgmp)并且使用lc-ms随时间量化丰度来进行稳定性评估(图9a-9d,实线)。与相对稳定的hamp相反,hcmp、hump和hgmp分别以1小时、30分钟和20分钟的近似半衰期(t1/2)被裂解物主动降解。nmp代谢的一种途径是脱磷酸化成核苷。为了评估脱磷酸化是否有助于nmp降解,通过添加便宜的磷酸酶抑制剂重复进行稳定性评估。在添加hnmp之前将增加浓度的磷酸盐(po4,150mm)以及结构模拟物原钒酸盐(vo4,10mm)与裂解物预温育。增加磷酸盐浓度稳定hump(t1/2≈60分钟),同时最小程度地影响hcmp和hgmp(图9a-9d,虚线)。相反,原钒酸盐稳定hcmp(t1/2>>60分钟)、hump(t1/2≈60分钟)和hgmp(t1/2≈45分钟)(图9a-9d,虚线)。总之,这些稳定性评估定义裂解物中的13mm/hr总体nmp降解速率,15分钟后存在70%外源添加的nmp。即使在没有磷酸酶抑制剂的情况下,与rna酶r依赖性解聚速率(>200mm/hr)相比,相对较低的hnmp消耗速率(13mm/hr)表明从rna释放的nmp应当可用于聚合成dsrna。热失活的开发为了稳定nmp以及ndp、ntp和dsrna,开发热失活方案。目的是鉴定最低温度和最短的温育时间,其将消除裂解物中的核苷酸和rna降解活性。为了评估热失活的功效,通过lc-ms比较热失活裂解物中的ntp消耗速率(在37℃),其中热失活的时间和温度是变化的。在热失活之前,裂解物以约120mm/hr消耗ntp(图10)。低于70℃的温度不影响ntp酶活性,而在70℃温育产生ntp酶活性的时间依赖性降低。在70℃温育15分钟后发生ntp酶活性的完全抑制(图10)。接下来,对这些条件评估其稳定裂解物中的nmp和dsrna的能力。为了评估热失活对nmp降解的影响,用外源rna酶r处理裂解物以释放rna,然后在70℃进行热失活。失活后,将温度降至37℃,并将反应再温育60分钟。如图11a中所示,用rna酶r处理5分钟快速解聚rna。在热失活和37℃温育后,nmp浓度未改变,表明所选择的热失活条件使nmp变得稳定。最后,对这些条件评估其稳定裂解物中体外转录反应的反应物和产物(包括ntp和dsrna)的能力。首先,将裂解物在升高的温度预温育15分钟。然后,将温度降至37℃并添加转录反应物(包括外源ntp、dna模板和纯化的t7rna聚合酶)。如图11b所示,在≥70℃热失活的裂解物中的转录反应产生与阳性对照反应(在缓冲液中进行)在定性上相似的rna产物。在60℃进行的反应中,无rna产物是明显的。总之,这些结果表明70℃温育15分钟足以在无细胞反应中稳定nmp、ntp和dsrna。热稳定性激酶的选择和评估在热失活后,需要一系列激酶活性以将从rna释放的5'-nmp顺序磷酸化为可以聚合以形成dsrna的ntp。这些激酶(其使用来自atp的高能量磷酸基团来磷酸化nmp和ndp)必须具有足够的热稳定性以在高温温育后保持活性,并且具有足够的活性以高速率(21mm/hrntp,对于1gdsrna/l/hr)产生ntp。基于在大肠杆菌中成功表达的文献报道和重组酶的生物化学表征选择来自嗜热生物体的酶用于评估(表9)。表9.针对每类活性测试的热稳定性激酶的来源。为了评估这些酶对dsrna的无细胞生产的适合性,将酶克隆到具有n-末端六组氨酸标签的大肠杆菌蛋白质表达载体中,过表达,并使用固定化金属亲和层析(imac)纯化。首先使用萤光素酶偶联测定法定量纯化的酶的活性,其中atp的消耗充当nmp和ndp磷酸化的代表。在温育温度范围下进行测定以确定每种酶的最佳反应温度。来自嗜热栖热袍菌和大肠杆菌的ump激酶(由pyrh基因编码)的表达在测试的诱导和纯化条件下产生不溶性蛋白质。来自激烈火球菌的纯化pyrh表现出约2μmol/min/mg蛋白质的比活性,其在很大程度上是不依赖于温度的(图12)。基于此比活性,在过量atp存在下,以1%总蛋白质表达pfpyrh的高密度细胞裂解物(90gdcw/l)将以超过50mm/hr的速率磷酸化ump(表10)。由于以1g/l/hr产生dsrna需要约5.25mm/hr的ump激酶活性,因此选择pfpyrh用于进一步评估。表10显示高密度(90gdcw/l)裂解物中的预测的pyrh速率,假设pfpyrh占总裂解物蛋白质的1%。表10.预测的pyrh反应速率来自大肠杆菌和嗜热栖热袍菌的amp激酶(由adk基因编码)的表达产生可溶性重组蛋白,而热聚球藻酶在测试的表达和纯化条件下是不可溶的。纯化的大肠杆菌酶在37℃和50℃具有活性,但在较高温度下没有表现出可检测的活性(图13)。在所有测试温度下,嗜热栖热袍菌酶表现出比大肠杆菌酶更高的比活性,在70℃下每mg酶的最佳活性接近1mm/min。当酶在高密度裂解物中以总蛋白质的0.01%表达时,该活性转化为裂解物中超过250mm/hramp磷酸化的预期速率(表11)。由于合成1g/l/hrdsrna需要约5.25mm/hr的amp激酶活性,因此选择tthadk用于进一步研究。表11显示了在高密度(90gdcw/l)裂解物中预测的adk反应速率,假设tthadk占总裂解物蛋白质的0.01%。表11.预测的adk反应速率温度(℃)预测速率(mm/hr)37n.d.5086.460199.570253.880106.1来自大肠杆菌和激烈火球菌的cmp激酶(由cmk基因编码)的表达产生不溶性蛋白质,而嗜热栖热袍菌酶的表达在测试条件下产生可溶性蛋白质。嗜热栖热袍菌cmp激酶表现出很大程度上不依赖于温度的活性,尽管在高于60℃的温度下酶活性略有下降(图14)。基于这些结果,以总蛋白质的0.02%在高密度裂解物中表达tthcmk将产生30-45mm/hr的cmp激酶活性(取决于温度,表12),完全超过5.25mm/hr目标。因此,选择tthcmk用于进一步评估。表12显示了在高密度(90gdcw/l)裂解物中预测的cmk反应速率,假设tthcmk占总裂解物蛋白的0.02%。表12.预测的cmk反应速率温度(℃)预测的速率(mm/hr)3744.85044.06040.87030.28032.2与测试的cmp激酶相反,来自大肠杆菌、嗜热栖热袍菌和海洋栖热袍菌的gmp激酶的表达产生可溶性重组蛋白。大肠杆菌酶在37℃具有最高的测试比活性,但在较高温度下活性较低(图14)。虽然嗜热栖热袍菌和海洋栖热袍菌酶在较高温度下都表现出最佳活性,但海洋栖热袍菌酶在所有测试温度下更具活性。基于测量的tmgmk的比活性,与目标速率5.25mm/hr相比,在高密度细胞裂解物中以0.1%的总蛋白质表达将在存在过量atp的情况下在70℃产生超过60mm/hr的预期速率(表13)。因此,选择tmgmk用于进一步评估。表13显示了高密度(90gdcw/l)裂解物中的预测gmk反应速率,假设tmgmk占总裂解物蛋白质的0.1%。表13.预测的gmk反应速率温度(℃)预测速率(mm/hr)3741.85042.86055.27062.08052.4与nmp激酶(其在很大程度上对单一底物是特异的)不同,ndp激酶使adp、cdp、udp和gdp磷酸化。为了比较ndp激酶,克隆来自适温性嗜热栖热袍菌和风产液菌的酶并在大肠杆菌中表达。虽然嗜热栖热袍菌酶在测试条件下是不溶的,但是风产液菌酶的表达产生可溶性蛋白质。使用atp和gdp作为底物的萤光素酶测定中的活性测量揭示aandk在宽温度范围间具有高活性,温度最佳值为50℃(图16)。底物间的比较揭示每种核苷酸在50℃的超过100μmol/min/mg的比活性,证实aandk可使多种ndp底物磷酸化(表14)。基于这些测量,aandk以0.01%总蛋白质在高密度裂解物中的表达转化为完全超过60mm/hr的udp激酶速率。鉴于需要21mm/hr的ndp激酶活性来支持1g/l/hrdsrna合成,选择aandk进行进一步评估。表14显示在50℃,aandk对cdp、udp和gdp底物具有高活性。表14.通过底物得到的aandk比活性底物比活性(μmol/min/mg)cdp259.6udp110.4gdp217多磷酸激酶(ppk)在聚合磷酸盐链和腺苷核苷酸之间可逆转移高能量磷酸基团。评估属于i型多磷酸激酶家族的多种ppk酶的活性,包括来自大肠杆菌以及嗜热生物体嗜热栖热袍菌和伸长热聚球藻的酶。选择这些酶用于测试,因为它们属于充分表征的i型家族或之前已经显示具有活性。ppk酶的表达和纯化产生可溶性蛋白质,然后使用adp和六偏磷酸钠作为底物测试萤光素酶测定系统中的活性。如图17中所示,所有测试的ppk酶的比活性相对于其他激酶活性较低。大肠杆菌ppk在较低温度(高达60℃)具有最高的比活性,而热聚球藻酶在70℃具有最高的活性。另外,在热失活条件(70℃,15分钟)下温育大肠杆菌酶导致不可逆的失活(未显示)。基于热聚球藻酶的比活性,在高密度裂解物中以总蛋白质的2%表达将导致在70℃预期的42mm/hr的速率(表15),与支持1g/l/hrdsrna合成所需的atp生产速率匹配。然而,在较低温度(例如,50℃)下无细胞dsrna合成将需要更高的teppk表达(超过4%总蛋白质)。表15显示高密度(90gdcw/l)裂解物中预测的ppk反应速率,假设teppk占总裂解物蛋白质的2%。表15.预测的ppk1反应速率温度(℃)预测速率(mm/hr)376.55018.96038.97042.1800在评估纯化系统中的每种酶活性后,测试酶在热失活的裂解物中的活性。将表达个别激酶的裂解物在70℃预温育15分钟,然后添加到底物。如在纯化的反应中,使用萤光素酶测定试剂盒定量atp消耗(对于nmp和ndp激酶)或atp产生(对于多磷酸激酶)。如下表16所示,nmp和ndp磷酸化的速率完全超过目标。表16:热失活裂解物中的激酶活性在确认个别激酶在裂解物中具有足够活性(除ppk之外)后,在多酶系统中评估激酶将nmp转化为ntp的能力。在这些研究中,将等体积的表达个别激酶的裂解物(5)合并,热失活,然后通过lc-ms测定来自等摩尔nmp混合物的ntp的atp依赖性产生。如表17所示,对于utp、ctp和gtp,总体ntp生产速率超过24mm/hr,表明裂解物的简单混合物(没有任何反应条件的优化)可以在存在足够的atp的情况下以足够的速率提供1g/l/hrdsrna的合成。表17.使用等摩尔nmp混合物作为底物和atp作为高能量磷酸盐供体,在表达个别激酶活性的裂解物的热失活的1:1混合物中的ntp生产途径速率(mm/hr)ump→utp3.24cmp→ctp9.24gmp→gtp11.8amp→atpn.d.nmps→ntps>24.3rna聚合酶向下选择在将rna解聚成nmp并将nmp磷酸化为其相应的ntp后,需要rna聚合酶将ntp转化为dsrna产物。出于几个原因,来自噬菌体t7的rna聚合酶是用于重组系统的有吸引力的酶。t7rna聚合酶包括单个亚基(与来自细菌和真核生物(eukarya)的许多rna聚合酶不同)并且已经通过生物化学和分子生物学研究广泛地表征。另外,已经描述了多种t7rna聚合酶突变体,其赋予改善的热稳定性(参见表18)。表18.评估活性和热稳定性的t7rna聚合酶的列表首先,使用双链体dna模板(例如图3b)和37℃反应温度评估商品化t7rna聚合酶的活性。使用quant-itrna试剂盒(broadrange)(thermofisherscientific)随时间定量rna的产生。在每个制造商推荐的条件下,thermot7和megascript酶具有高活性,而neb酶显示出显著较低的活性(图18)。接下来,克隆具有n-末端六组氨酸标签的t7rna聚合酶的lvi突变体,在大肠杆菌中表达,并使用imac纯化。接下来,在标准化反应条件下,在稀的热失活裂解物(35%最终裂解物浓度)中,与lvi突变体聚合酶一起测试megascript和thermot7聚合酶的活性(图19)。与纯化系统中一样,thermot7在37℃展现出比megascript酶更高的比活性(3.1nmolrna/min/mg蛋白质)。lvi突变体具有所测试的三种酶的最低比活性(0.33nmol/min/mg)。当在其他方面相同的条件下在50℃测试时,megascript酶和lvi突变体都没有表现出任何可检测的活性。相比之下,thermot7的活性比在37℃高。在测定中使用约4%的总蛋白作为thermot7聚合酶,在50℃使用双链体dna模板和thermot7聚合酶,rna合成速率超过11g/l/hr(图19)。然后选择thermot7用于进一步表征。在50℃在稀释的热失活的裂解物中确认thermot7的活性后,在代表大规模无细胞dsrna产生的条件下研究thermot7的活性。除了增加的裂解物浓度之外,大规模反应可包括由热失活过程产生的沉淀的裂解物组分(例如蛋白质)。为了评估thermot7在这些条件下的性能,在热失活的高密度裂解物(68%最终裂解物浓度)中,在有或没有热失活后除去沉淀蛋白质的澄清的情况下定量rna聚合(图20)。rna合成速率在基质中显著高于在缓冲液中,最高速率发生在已经通过离心澄清的热失活基质中。在未澄清的反应中,总rna合成速率(在2小时反应内)超过2g/l/hr,测定中1.4%的总蛋白作为thermot7聚合酶。最后,在较高温度测试thermot7的热稳定性,以评估与该程序中早先建立的热失活条件的相容性。在这些研究中,将thermot7酶在升高的温度(50-70℃)预温育不同的时间长度(0-15分钟),并且剩余的活性在37℃定量。如图21所示,50℃的温育是酶充分耐受的,但是较高的温度导致聚合酶活性的快速的不可逆的失活。因此,在这些实验中,该特定的thermot7与50-70℃的热失活不相容。因此,在一些情况下,可以在热失活后将纯化的thermot7酶添加至无细胞反应,或者可以使用替代的t7rna聚合酶突变体,例如在70℃具有足够的半衰期。材料和方法核苷酸分析通过液相层析与质谱术联用(lc-ms)进行一磷酸核苷酸(amp、cmp、ump和gmp)的分析。在室温使用配备有zic-chilic柱(2.1x20mm,3μmi.d.)(merck)的agilent1100系列hplc以流速0.5ml/min和注射体积2μl分离样品。流动相由20mm乙酸铵(a)和90%乙腈中的20mm乙酸铵(b)组成。分离方法由15-50%(b)的梯度3.5分钟,然后50%(b)1.5分钟,然后15%b3分钟组成。在多反应监测(mrm)模式下,使用电喷雾电离(毛细管电压:-3000v,温度:600℃,去溶剂化气体:20psi)在absciexapi3200质谱仪上进行定量。一磷酸、二磷酸和三磷酸核苷酸物质(nmp、ndp和ntp)的分析使用具有以下分离梯度的上述方法:15%b至50%b3.5分钟,然后50%b2.5分钟,然后15%b4分钟。将峰面积与由纯化的化合物(sigma-aldrich)组成的标准曲线进行比较。为了分析裂解物中的样品,在裂解物背景中制备标准曲线,所述裂解物背景已经经过酸淬灭,澄清,ph中和,并过滤,如在下文描述的样品制备步骤中。在40℃以流速0.5ml/min和注射体积0.5μl使用配备有acquitycsh氟苯基柱(2.1x150mm,1.7μmi.d.)(waters)的acquityh-classuplc(waters)通过液相层析进行2’-、3’-和5’-nmp的分析。流动相由在0.2%甲酸中的10mm乙酸铵(a)和95%乙腈,0.2%甲酸中的10mm乙酸铵(b)组成。分离方法由1%b2.8分钟,然后是1%-30%b的梯度2.2分钟,接着100%b7分钟,然后是1%b3分钟组成。使用acquityuplcpda(waters)在260、254和210nm进行定量。将峰面积与由纯化的化合物(购自sigma-aldrich,除购自biologlifescienceinstitute的2'和3'cmp、ump和gmp以外)组成的标准曲线比较。为了分析裂解物中的样品,在裂解物背景中制备标准曲线,所述裂解物背景已经经过酸淬灭,澄清,ph中和,并过滤,如在下文描述的样品制备步骤中。大肠杆菌rna的提取和纯化根据已建立的方案(mohanty,b.k.,giladi,h.,maples,v.f.,&kushner,s.r.(2008).analysisofrnadecay,processing,andpolyadenylationinescherichiacoliandotherprokaryotes.methodsinenzymology,447,3-29)从高密度大肠杆菌裂解物(蛋白质浓度:40-50mg/ml)中提取和纯化rna。对于每400μl冷冻的大肠杆菌裂解物,添加67μl的20mm乙酸以降低rna酶活性。将样品在37℃的珠浴中解冻。解冻后立即添加400μl10%(w/v)三甲基(十四烷基)溴化铵(trimethyl(tetradecyl)ammoniumbromide)(sigma-aldrich)溶液。然后通过在4℃的微量离心机中以10,000xg离心使所得悬浮液澄清,并除去上清液。将团粒重悬于35%乙醇中的1ml2m氯化锂(sigma-aldrich)溶液中。将悬浮液在室温温育5分钟,然后通过在4℃以15,000xg离心6分钟澄清,并除去上清液。然后将团粒重悬于1ml2m氯化锂水溶液中,并在室温温育2分钟,然后以15,000xg澄清6分钟。然后除去上清液,通过重悬于70%乙醇中清洗剩余的团粒,并在4℃以最大速度(21,000xg)离心5分钟。然后除去上清液,将团粒在室温风干15分钟。然后将团粒重悬于200μl无核酸酶的水中,并在4℃温育过夜以溶解rna。通过离心(最高速度在4℃5分钟)澄清rna溶液,并将含有可溶性rna的上清液转移到无菌无rna酶的管中并在-20℃储存。核酸酶向下选择如下从商业来源获得核酸酶:benzonase和核酸酶p1获自sigma-aldrich,rna酶r、terminator外切核酸酶和rna酶iii获自epicenter,rna酶a获自thermofisher,外切核酸酶t获自newenglandbiolabs。对于筛选测定法,将1μl每种酶溶液添加到100μl2x测定缓冲液(100mm磷酸钾ph7.4、10mm氯化镁、1mm氯化锌)中,然后在时间t=0时与等体积的1mmrna溶液(约340ng/μl)组合并充分混合。将反应在37℃温育,并通过在冰上将20μl转移至酸淬灭溶液(180μl的0.2m硫酸)中定期取样。在完成时间过程后,通过在4℃以3,000xg离心5分钟澄清淬灭的样品。然后将来自每个样品的170μl上清液转移到uv-透明的96孔半区域板(corning)中,并使用微板阅读器和10665m-1cm-1消光系数在260nm处通过吸光度定量酸溶性核苷酸,通过对每个单核苷酸的个别消光系数取平均值来估计。对于通过lc-ms的后续分析,将45μl澄清的上清液用5μl的2.5m氢氧化钾进行ph中和。通过rna的碱性水解测定总核苷酸合并物(即100%解聚):将rna与等体积的0.2m氢氧化钾合并,然后加热至99℃达20分钟。然后淬灭碱性水解的样品并如上所述进行分析。蛋白质表达和纯化将重组蛋白质从编码相关基因以及六组氨酸标签的合成dna克隆到petduet-1(novagen)中。将质粒转化到大肠杆菌t7express(newenglandbiolabs)中,然后使用补充有50μg/ml羧苄青霉素的zym-505培养基(studier,f.w.(2005).proteinproductionbyauto-inductioninhigh-densityshakingcultures.proteinexpressionandpurification,41(1),207-234)在1l培养物中培养。在a600=0.6时诱导表达。对于rna酶r和激酶,用0.1mmiptg诱导表达,将温度降至16℃,并且将培养物在16℃培养24小时。对于t7rna聚合酶,用0.8mmiptg诱导表达,并将培养物在37℃培养3小时。通过离心收获生物量,倾倒上清液,然后将细胞团粒储存在-80℃。将细胞团粒解冻并通过重悬浮于4个体积的补充有benzonase(0.04μl/ml)的b-percomplete(thermofisherscientific)中并在室温在温和搅拌的情况下温育15分钟来裂解。然后通过在4℃以16,000xg离心1小时澄清裂解物。使用连接至aktaprimeplusfplc系统(gehealthcare)的hisgravitrap柱(gehealthcare)或histraphp柱,通过固定化金属亲和层析纯化蛋白质。对于这两种纯化方法,将柱在平衡/清洗缓冲液(50mm磷酸盐缓冲液ph7.4、500mmnacl、20mm咪唑)中平衡,加载裂解物,然后用30倍柱体积的平衡/清洗缓冲液清洗。用洗脱缓冲液(50mm磷酸盐缓冲液ph7.4、500mmnacl、500mm咪唑)洗脱蛋白质。为了纯化激酶,平衡/清洗和洗脱缓冲液使用50mmtris-hclph7.5代替磷酸盐缓冲液。通过sds-page分析洗脱级分,并通过bca(thermofisherscientific)定量蛋白质含量。然后将级分合并,并且通过透析将缓冲液交换到1000体积的2x储存缓冲液中。对于rna酶r,2x存储缓冲液由补充有额外的500mmnacl的2xpbs组成。对于t7rna聚合酶,2x存储缓冲液由2xpbs组成。对于激酶,2x存储缓冲液由100mmtris-hclph7.0和100mmnacl组成。透析后,将蛋白质与等体积的100%甘油(50%终浓度)混合并储存在-20℃。细胞裂解物制备将大肠杆菌菌株gl16-170(bl21(de3).t526pgi.δedd.δtktb.δtolc_wt-7-e1.δmgsa*-f3.δappa*.δamn*-f1.nagd(keio)::zeor-1.δphoa*.t352baa1644.δusha*-c4.rna::tolc-b04)和gl14-322(bl21(de3).t526pgi.δedd.δtktb.δtolc_wt-7-e1.δmgsa*-f3.δappa*.δamn*-f1.nagd(keio)::zeor-1.δphoa*.t352baa1644.δusha::tolc-a01)在korz培养基中在10l生物反应器中培养直至分批阶段结束,然后通过离心收获并在-80℃冷冻。在58.8mm磷酸氢二钾中将团粒重悬至10%细胞干重,并使用2次通过冷却至4℃的pandaplus匀浆器(geanirosoavi)在15,000psi进行裂解。通过在4℃以16,000xg离心1小时澄清裂解物,并在-80℃储存之前通过bca测定(thermofisher)分析蛋白质含量。用外源rna酶r解聚裂解物rna在2℃预平衡gl16-170裂解物(蛋白质含量34.5mg/ml)和rna酶r溶液(1mg/ml,在300mm磷酸钾缓冲液ph7.4、200mmkcl、2mmmgcl2中),然后启动反应。在时间t=0时,将50μl大肠杆菌裂解物和50μlrna酶r溶液混合,并通过转移至预热的37℃块开始反应。如上所述组装包含脱氧胆酸盐的反应,只是将裂解物与0.2个体积的5x脱氧胆酸钠水溶液预混合,并在启动前在2℃温育15分钟。启动后,将反应物在37℃温育,并通过在冰上将10μl转移至酸淬灭溶液(90μl的0.2m硫酸)中定期取样。在完成时间过程后,通过在2℃以3,200xg离心10分钟澄清淬灭的样品。首先通过酸溶性核苷酸的吸光度定量解聚:在uv-透明的96孔半区域板(corning)中将10μl淬灭和澄清的反应添加到160μl的0.2m硫酸中。使用微板阅读器(参见上文)通过260nm处的吸光度定量酸溶性核苷酸。也通过对5'、2'和3'nmp的uplc分析定量解聚:通过添加10μl的1mkoh将30μl的每种酸淬灭的样品进行ph中和,然后通过0.2μm滤器,之后进行uplc分析。通过裂解物rna的碱性水解测定总核苷酸库(即100%解聚):将50μl裂解物与150μl的0.2m氢氧化钾合并,然后加热至99℃达20分钟。然后淬灭碱性水解的样品并进行分析,如上所述。用过表达的rna酶解聚裂解物中的rna用编码具有c-末端六组氨酸标签的大肠杆菌rnr基因的petduet-1转化大肠杆菌菌株gl16-170。将该菌株与用空petduet-1转化的gl16-170一起在补充有50mg/l羧苄青霉素的korz培养基中分批培养。在a600=20用0.8mmiptg诱导培养物,并在诱导时补充额外的10g/l葡萄糖。诱导后1小时,通过离心收获生物量并冷冻。如上所述从冷冻生物量制备裂解物(对于具有空petduet-1的gl16-170生物量,蛋白质浓度:36.6mg/ml;对于具有携带克隆的rna酶r的petduet-1的gl16-170为53.2mg/ml)。如上所述用反应中50%最终裂解物浓度评估稀裂解物中的解聚。通过在2℃将9体积裂解物与1体积10xedta溶液预温育5分钟来评估浓缩裂解物中的解聚。然后,如上所述,通过转移到预热的37℃块并取样来启动反应。nmp稳定性评估在冰上将4体积的gl14-322裂解物(蛋白质浓度:50.5mg/ml)与1体积的磷酸酶抑制剂溶液(终浓度50mm磷酸钾ph7.4、150mm磷酸钾ph7.4或10mm原钒酸钠)组合。在水中制备同位素标记的nmp的等摩尔溶液(腺苷-13c10,15n55'-一磷酸,胞苷-15n3-5′-一磷酸,尿苷-15n2-5′-一磷酸和鸟嘌呤-15n5-5′-一磷酸[sigma-aldrich],每种25mm)。在启动反应之前,将裂解物和nmp平衡至37℃持续10分钟。为了启动反应,将90μl裂解物溶液添加到10μlnmp溶液,并将反应充分混合。通过在指定时间点取样监测反应。在取样期间,将12μl反应混合物在冰上转移至108μl的0.2m硫酸。然后通过离心澄清淬灭的反应,进行ph中和,并过滤用于lc-ms分析,如上所述。热失活的开发将gl14-322裂解物在冰上等分取样到5个微量离心管中,然后转移到期望的热失活温度平衡的加热块。在指定的时间,将管在冰上冷却,然后通过离心(21,000xg,5分钟)澄清,并且收获上清液。将来自热失活的裂解物的上清液与ntp的等摩尔混合物(sigma-aldrich,各25mm)一起在37℃平衡10分钟。在时间t=0时,将9个体积的热失活的裂解物上清液与1个体积的ntp溶液组合,并通过取样到酸淬灭溶液中监测反应,进行ph中和,过滤,并通过lc-ms分析,如上所述。对于裂解物中的转录反应,使用megascriptt7转录试剂盒(thermofisher)按照试剂盒用法说明进行10μl反应,除了减少的酶混合物量(最终反应体积的5%),并包括热失活的裂解物上清液(最终反应体积的40%)以外。在单独的megascript反应缓冲液中进行阳性对照反应,而阴性对照反应包括裂解物但省略酶混合物。通过用1kbdna梯(newenglandbiolabs)旁边的sybrsafe(invitrogen)染色的琼脂糖凝胶电泳分析反应。核苷酸激酶活性测定在由50mmtris-hclph7.0、4mmmgso4、4mmatp和4mm相应的nmp或ndp组成的缓冲液中,在不同温度(37℃,50℃,60℃,70℃和80℃)测定核苷酸激酶。在启动反应之前将反应缓冲液(1.2x浓缩物)和酶溶液(0.5mg/ml)在反应温度预平衡1分钟。在时间t=0,通过将80μl反应缓冲液与20μl酶混合来启动反应。通过在指定时间点取样监测反应。在取样期间,将15μl反应混合物在冰上转移至135μl的0.2m硫酸。在完成反应后,如上所述用1mkoh对样品进行ph中和,然后在冰冷的水中以1:10稀释。使用atp生物发光测定试剂盒(sigma-aldrich)按照试剂盒用法说明在每个样品中定量atp。对于裂解物中的反应,如下修改上述方案:将裂解物等分取样到个别的反应管中,然后通过在70℃温育15分钟进行热失活。将反应缓冲液(2x浓缩物)和热失活的裂解物在反应温度预平衡,并通过组合等体积的裂解物和反应缓冲液来启动反应。通过用9体积酸淬灭溶液淬灭个别的反应管对反应进行取样,然后如上所述进行分析。为了测定裂解物中激酶的组合活性(即从nmp到ntp),将个别表达每种激酶的裂解物以1:1比率混合,分成10μl等分试样,然后如上所述进行热失活。在由50mmtris-hclph7.0、16mmmgso4、2mm每种一磷酸核苷酸(amp、cmp、ump和gmp)和16mmatp组成的缓冲液中分析激酶活性。将在反应温度预平衡的反应缓冲液(2x浓缩物)与等体积的裂解物合并以启动反应。在70℃进行反应,并通过用9体积酸淬灭溶液淬灭个别反应管进行取样,然后如上所述进行分析。多磷酸激酶活性测定在由50mmtris-hclph7.0、4mmmgso4、25mm(nh4)2so4、1mmadp和1mm六偏磷酸钠组成的缓冲液中在不同温度(37℃,50℃,60℃,70℃和80℃)下测定多磷酸激酶。在反应启动之前,将反应缓冲液(1.2x浓缩物)和酶溶液(0.25mg/ml)在反应温度预平衡1分钟。在时间t=0,通过将80μl反应缓冲液与20μl酶混合来启动反应。通过在指定时间点取样监测反应。在取样期间,将15μl反应混合物在冰上转移至135μl的0.2m硫酸。在完成反应后,如上所述用1mkoh对样品进行ph中和,然后在冰冷的水中以1:10稀释。使用atp生物发光测定试剂盒(sigma-aldrich)按照试剂盒用法说明在每个样品中定量atp。对于裂解物中的反应,如下修改上述方案:将裂解物等分取样到个别的反应管中,然后通过在70℃温育15分钟进行热失活。将反应缓冲液(2x浓缩物)和热失活的裂解物在反应温度预平衡,并通过组合等体积的裂解物和反应缓冲液来启动反应。通过用9体积酸淬灭溶液淬灭个别的反应管对反应进行取样,然后如上所述进行分析。通过扣除来自相同的反应条件下对照裂解物(没有过表达的多磷酸激酶)的信号来计算裂解物中的反应速率。生成转录模板通过合成gblockdna(integrateddnatechnologies)的pcr扩增制备双链体dna模板。纯化反应物并通过异丙醇沉淀浓缩。rna聚合酶向下选择使用由每个制造商推荐的条件比较商品化rna聚合酶。每个50μl反应由10x反应缓冲液(由制造商提供)、ntp、dna模板(0.5μg)和酶组成。对于nebt7rna聚合酶,反应包括0.5mm的每种ntp、5mmdtt和100u酶。除了省略dtt外,与thermot7聚合酶的反应是相同的。与megascriptt7的反应包括7.5mm每种ntp和5μl酶混合物。通过bca测定(thermofisher)测定酶浓度。通过在指定时间点取样监测反应。在取样期间,将10μl反应混合物转移至90μlrna淬灭溶液(10mmtris-hclph8.0,5mmedta)中并储存在冰上。使用quant-itrnabroadrangeassaykit(thermofisher)按照试剂盒用法说明定量淬灭溶液中的rna样品。使用megascript试剂盒制备并按照试剂盒用法说明纯化的纯化dsrna的连续稀释液用于构建用于定量的标准曲线。通过琼脂糖凝胶电泳定性分析反应。裂解物中的rna聚合酶评估如前所述,将gl14-322裂解物加热失活并通过离心澄清。每个20μl反应由澄清的裂解物(7μl)、10x辅因子溶液(300mmmgcl2,20mm亚精胺)、ntp(各7.5mm,由ph中和的储备溶液制备)、dna模板(0.6μg)和酶组成。(1μl)。将反应在37℃或50℃温育1小时,然后通过添加9体积rna淬灭溶液淬灭。将淬灭反应在淬灭溶液中进一步稀释10倍(最终稀释度:100倍)。然后使用quant-it试剂盒(见上文)定量稀释的反应。通过扣除对照反应(省略rna聚合酶)中定量的rna来计算由反应产生的rna。如上所述进行高密度裂解物中的rna聚合酶测定,具有以下修改。每个100μl反应由裂解物(67.5μl)、10x辅因子溶液(300mmmgcl2,20mm亚精胺)、ntp(各7.5mm,由ph中和的储备溶液制备)、dna模板(3μg)和酶(10μl)组成。对于在未澄清的反应基质中进行的反应,将gl14-322裂解物(67.5μl)等分取样到个别的反应管中,然后如前所述进行热失活。将另外的反应物添加到到热失活的基质中,然后通过涡旋振荡充分混合直至均质。对于在缓冲液中进行的反应,10x辅因子溶液由300mmmgcl2、20mm亚精胺和400mmdtt组成。此外,在仅缓冲反应中包括50mm磷酸钾ph7.4。将所有反应在50℃温育2小时。通过添加9体积rna淬灭溶液淬灭来自每个反应的样品,然后通过以最大速度(21,000xg)离心1分钟澄清。将来自这些反应的上清液在淬灭溶液中进一步稀释40倍(最终稀释:400倍),然后使用quant-it试剂盒(参见上文)进行定量。实施例2对热稳定性ppk2酶进行密码子优化以在大肠杆菌中表达,合成并克隆到petduet-1(novagen)中。然后,将质粒转化到gl16-170中。为了产生对照菌株,将空的petduet-1质粒转化到gl16-170中。在5mllysogenybroth(lb)中预培养过夜后,将菌株在1llb中于37℃培养直至细胞密度达到od600约0.5。然后将培养物在冰上短暂冷却,并通过添加异丙基硫代半乳糖吡喃糖苷(isopropylthiogalactopyranoside)(iptg)至终浓度0.25mm诱导ppk2表达。诱导后,将培养物在20℃培养约16小时。通过离心收获培养物,并将细胞团粒储存在-80℃。通过解冻冷冻的团粒产生裂解物,重悬于2个团粒体积的150mmmops-naohph7中,并使用4次通过emulsiflexc3匀浆器(avestin)在15,000psi均质化。然后通过在4℃以15,000xg离心1小时澄清裂解物。将裂解物的等分试样在使用前在-80℃冷冻。然后在热失活的裂解物中测量热稳定性ppk2活性。首先将表达ppk2酶的解冻裂解物以1:100稀释到从对照菌株制备的裂解物中,除了以1:10稀释的地热异常球菌ppk2裂解物以外。将预冷的氯化锰(mncl2)和六偏磷酸钠(hmp)溶液分别添加至终浓度10mm和1mm。然后,通过在预热的70℃热循环仪中温育15分钟来热失活裂解物。然后通过将热失活的裂解物与等体积的2x反应缓冲液混合来启动反应,所述反应缓冲液由10mmmncl2、2mm二磷酸腺苷(adp)或腺苷一磷酸(amp)和9mmhmp组成。将反应物在70℃温育,并通过取出反应混合物的等分试样并在冰上用9份淬灭溶液(200mmh2so4)稀释来获得时间点。通过将裂解物与淬灭溶液直接混合,将淬灭的裂解物在冰上储存15分钟,然后添加2x反应缓冲液来获得初始时间点(t0)。在测定结束时,通过在3,200xg离心10分钟澄清淬灭的时间点溶液。然后通过将3份骤冷的反应溶液与1份中和溶液(1mkoh)混合,将来自淬灭反应的上清液进行ph中和。然后将淬灭且中和的样品用水以1:10稀释,然后使用腺苷5'-三磷酸(atp)生物发光测定试剂盒(sigma-aldrich产品目录编号:flaa)按照试剂盒用法说明进行定量。基于含ppk2的反应中atp的累积(扣除来自对照裂解物的atp浓度)计算初始反应速率。5种iii类ppk2酶在70℃在裂解物中表现出可溶性表达、热稳定性和高反应速率。代表性的表达和活性数据显示在图22a-22b中。表17中显示了每种测试酶的表达和动力学数据的总结。使用hmp作为底物,嗜气暖绳菌、玫瑰弯菌、嗜热栖热袍菌和r.castenholzii酶将amp和adp快速转化为atp。对于amp和adp底物两者,地热异常球菌酶比其他测试的ppk2表现出低约20倍的转化速率。表17.裂解物中热稳定性iii类ppk2酶的表达和速率数据的总结。实施例3然后使用热稳定性嗜气暖绳菌ppk2来提供atp,用于从nmp、adp和hmp无细胞产生dsrna。用个别过表达表18中详述的激酶的6种大肠杆菌裂解物的混合物进行无细胞dsrna合成反应。表18.用于从nmp和hmp产生dsrna的激酶。首先,将表18中详述的裂解物在冰上以等体积混合。将预冷的氯化锰(mncl2)、硫酸镁(mgso4)和六偏磷酸钠(hmp)溶液分别添加至终浓度0-2.5mm、30mm和1mm。然后通过在预热的70℃热循环仪中温育15分钟来热失活裂解物混合物。为了启动反应,将热失活的裂解物与以下组分组合:一磷酸核苷酸(腺苷5'-一磷酸、胞苷5'-一磷酸、尿苷5'-一磷酸和鸟苷5'-一磷酸,各2mm)的等摩尔混合物、50mmtrisph7.0、30mmmgso4、0-2.5mmmncl2、1mm腺苷5'-二磷酸、2mm亚精胺、1.5μg质粒dna模板和3μg热稳定性t7rna聚合酶(s430p、f849i、f880y),总体积为20μl。作为对照,从2mmntp的等摩尔混合物合成dsrna(省略裂解物、adp和hmp)。作为另外的对照,dsrna由2mmnmp的等摩尔混合物合成(省略表达ppk2的裂解物、adp和hmp,但包括8mmatp作为能量源)。作为阴性对照,省略聚合酶进行一式两份反应。将所有反应在50℃温育2小时,然后通过添加到9体积te+缓冲液(10mmtris-hclph8.0,5mmedta)终止反应。将样品与等体积的2xrna上样染料(newenglandbiolabs)混合并加热至70℃10分钟,然后进行琼脂糖/tae凝胶电泳。如图23所示,在缓冲液中使用ntp(左侧泳道)、在表达核苷酸激酶的裂解物自nmp和atp(中间泳道)、以及在表达核苷酸激酶和多磷酸激酶的裂解物中自nmp和hmp(右侧泳道)产生期望的dsrna产物。在任何反应中都不需要氯化锰,证明嗜气暖绳菌酶可以利用mg2+作为辅助因子以及mn2+。因此,对于含有嗜气暖绳菌ppk2的无细胞反应,先验地不需要mn2+。如图24所示,在缓冲液中使用ntp(左侧泳道)、在表达核苷酸激酶的裂解物自nmp和atp(中间泳道)、以及在表达核苷酸激酶和多磷酸激酶的裂解物中自nmp和hmp(右侧泳道)产生dsrna产物。自nmp和hmp的dsrna产生不需要外源adp或嗜热栖热袍菌amp激酶。因此,嗜气暖绳菌酶ppk2可以用作5-激酶系统的一部分,以在无细胞反应中从nmp和hmp产生dsrna。其他实施方案在权利要求书中,诸如“一个”、“一种”和“所述/该”的冠词可以表示一个/种或超过一个/种,除非相反地指出或者另外从上下文中显而易见。若组成员之一、超过一个或全部在给定产品或过程中存在,使用或以其他方式与给定产品或过程相关,则认为满足包括组中一个或多个成员之间的“或”的权利要求或描述,除非相反指出或另外从上下文中显而易见。本发明包括组的恰好一个成员在给定产品或过程中存在,使用或以其他方式与给定产品或过程相关的实施方案。本发明包括组成员的超过一个或全部在给定产品或过程中存在,使用或以其他方式与给定产品或过程相关的实施方案。此外,本发明包括所有变型、组合和置换,其中将一个或多个所列权利要求的一个或多个限制、要素、条款和描述性术语引入另一个权利要求中。例如,可以修改从属于另一个权利要求的任何权利要求以包括在从属于相同基本权利要求的任何其他权利要求中找到的一个或多个限制。在要素被呈现为列表的情况下,例如,以马库什组格式,要素的每个子组也被公开,并且可以从组中移除任何要素。应当理解,通常,在将本发明或本发明的方面称为包括特定元件和/或特征的情况下,本发明的某些实施方案或本发明的方面包括此类要素和/或特征或基本上由此类要素和/或特征组成。出于简化的目的,那些实施方案尚未在本文中具体阐述。还应注意,术语“包含”和“含有”旨在是开放的并且允许包含额外的元件或步骤。在给出范围的情况下,包括端点。此外,除非另外指出或另外从上下文和本领域普通技术人员的理解中显而易见,否则表示为范围的值可以在本发明的不同实施方案中的所述范围内呈现任何特定值或子范围,至范围下限单位的十分之一,除非上下文另有明确规定。本申请涉及各种公告的专利、公开的专利申请、期刊文章和其他出版物,所有这些都通过引用并入本文。若任何并入的参考文献与本说明书之间存在冲突,则应当以本说明书为准。另外,落入现有技术内的本发明的任何特定实施方案可以明确地从任何一个或多个权利要求中排除。因为认为这些实施方案是本领域普通技术人员已知的,所以即使在此未明确阐述排除,也可以排除它们。无论是否与现有技术的存在相关,本发明的任何特定实施方案可以出于任何原因而从任何权利要求排除。本领域技术人员将认识到或仅使用常规实验能够确定本文所述的具体实施方案的许多等同方案。本文描述的本实施方案的范围不旨在限于以上描述,而是在所附权利要求书中阐述。本领域普通技术人员将理解,在不脱离如所附权利要求书所限定的本发明的精神或范围的情况下,可以对本说明书进行各种改变和修改。参考文献1.maekewak.,tsunasawas.,dibog.,sakiyamaf.1991."primarystructureofnucleasep1frompenicilliumcitrinum."eur.j.biochem.200:651-661.2.volbedaa.,lahma.,sakiyamaf.,suckd.1991.crystalstructureofpenicilliumcitrinump1nucleaseat2.8-aresolution.emboj.10:1607-1618(1991)3.romierc.,dominguezr.,lahma.,dahlo.,suckd.1998.recognitionofsingle-strandeddnabynucleasep1:highresolutioncrystalstructuresofcomplexeswithsubstrateanalogs.proteins32:414-4244.chengz.f.,deutscherm.p.2002.purificationandcharacterizationoftheescherichiacoliexoribonucleasernaser.comparisonwithrnaseii.j.biol.chem.277:21624-21629.5.zilhaor.,camelol.,arraianoc.m.1993.dnasequencingandexpressionofthegenernbencodingescherichiacoliribonucleaseii.mol.microbiol.8:43-516.marchp.e.,ahnnj.,inouyem.1985.thednasequenceofthegene(rnc)encodingribonucleaseiiiofescherichiacoli.nucleicacidsres.13:4677-46857.chens.m.,takiffh.e.,barbera.m.,duboisg.c.,bardwellj.c.,courtd.l.1990.expressionandcharacterizationofrnaseiiianderaproteins.productsoftherncoperonofescherichiacoli.j.biol.chem.265:2888-28958.robertsonh.d.,websterr.e.,zindern.d.1968.purificationandpropertiesofribonucleaseiiifromescherichiacoli.j.biol.chem.243:82-91.9.molinal.,bernalp.,udaondoz.,seguraa.,ramosj.l.2013.completegenomesequenceofapseudomonasputidaclinicalisolate,strainh8234.genomeannounc.1:e00496-13;andcheng,z.f.andm.p.deutscher.2002.purificationandcharacterizationoftheescherichiacoliexoribonucleasernaser.comparisonwithrnaseii.jbiolchem.277(24).10.evens.,pellegrinio.,zigl.,labasv.,vinhj.,brechemmier-baeyd.,putzerh.2005.ribonucleasesj1andj2:twonovelendoribonucleasesinb.subtiliswithfunctionalhomologytoe.colirnasee.nucleicacidsres.33:2141-2152.11.lidelasierra-gallayi.,zigl.,jamallia.,putzerh.2008.structuralinsightsintothedualactivityofrnasej.nat.struct.mol.biol.15:206-212.12.ballt.k.,sauruggerp.n.,benedickm.j.1987.theextracellularnucleasegeneofserratiamarcescensanditssecretionfromescherichiacoli.gene57:183-192.13.biedermannk.,jepsenp.k.,riisee.,svendseni.1989.purificationandcharacterizationofaserratiamarcescensnucleaseproducedbyescherichiacoli.carlsbergres.commun.54:17-27.14.shlyapnikovs.v.,luninv.v.,perbandtm.,polyakovk.m.,luninv.y.,levdikovv.m.,betzelc.,mikhailova.m.2000.atomicstructureoftheserratiamarcescensendonucleaseat1.1aresolutionandtheenzymereactionmechanism.actacrystallogr.d56:567-572.15.zuoy.,deutscherm.p.2002.mechanismofactionofrnaset.i.identificationofresiduesrequiredforcatalysis,substratebinding,anddimerization.j.biol.chem.277:50155-50159.16.zuoy.,zhengh.,wangy.,chruszczm.,cymborowskim.,skarinat.,savchenkoa.,malhotraa.,minorw.2007.crystalstructureofrnaset,anexoribonucleaseinvolvedintrnamaturationandendturnover.structure15:417-428.17.huangs.,deutscherm.p.1992.sequenceandtranscriptionalanalysisoftheescherichiacolirntgeneencodingrnaset.j.biol.chem.267:25609-25613.18.chauhana.k.,miczaka.,tarasevicienel.,apiriond.1991.sequencingandexpressionofthernegeneofescherichiacoli.nucleicacidsres.19:125-129.19.cormackr.s.,genereauxj.l.,mackieg.a.1993.rnaseeactivityisconferredbyasinglepolypeptide:overexpression,purification,andpropertiesoftheams/rne/hmp1geneproduct.proc.natl.acad.sci.u.s.a.90:9006-9010.20.meadorj.iii,kennelld.1990.cloningandsequencingthegeneencodingescherichiacoliribonucleasei:exactphysicalmappingusingthegenomelibrary.gene95:1-7.21.awanon.,rajagopalv.,arbingm.,patels.,huntj.,inouyem.,phadtares.2010.escherichiacolirnaserhasdualactivities,helicaseandrnase.j.bacteriol.192:1344-1352.22.regnierp.,grunberg-managom.,portierc.1987.nucleotidesequenceofthepnpgeneofescherichiacoliencodingpolynucleotidephosphorylase.homologyoftheprimarystructureoftheproteinwiththerna-bindingdomainofribosomalproteins1.j.biol.chem.262:63-68.23.kimhiy.,littaueru.z.1968.purificationandpropertiesofpolynucleotidephosphorylasefromescherichiacoli.j.biol.chem.243:231-240.24.shiz.,yangw.z.,lin-chaos.,chakk.f.,yuanh.s.2008.crystalstructureofescherichiacolipnpase:centralchannelresiduesareinvolvedinprocessivernadegradation.rna14:2361-2371.25.thallerm.c.,schippas.,boncia.,crestis.,rossolinig.m.1997.identificationofthegene(apha)encodingtheclassbacidphosphatase/phosphotransferaseofescherichiacolimg1655andcharacterizationofitsproduct.femsmicrobiol.lett.146:191-198.26.forleoc.,benvenutim.,calderonev.,schippas.,docquierj.d.,thallerm.c.,rossolinig.m.,manganis.2003.expression,purification,crystallizationandpreliminaryx-raycharacterizationoftheclassbacidphosphatase(apha)fromescherichiacoli.actacrystallogr.d59:1058-1060.27.shuttleworthh.,taylorj.,mintonn.1986.sequenceofthegeneforalkalinephosphatasefromescherichiacolijm83.nucleicacidsres.14:8689-8689.28.bradshawr.a.,canceddaf.,ericssonl.h.,neumannp.a.,piccolis.p.,schlesingerm.j.,shrieferk.,walshk.a.1981.aminoacidsequenceofescherichiacolialkalinephosphatase.proc.natl.acad.sci.u.s.a.78:3473-3477.29.lic.,ichikawaj.k.,ravettoj.j.,kuoh.-c.,fuj.c.,clarkes.1994.anewgeneinvolvedinstationary-phasesurvivallocatedat59minutesontheescherichiacolichromosome.j.bacteriol.176:6015-6022.30.kuznetsovae.,proudfootm.,gonzalezc.f.,browng.,omelchenkom.v.,borozani.,carmell.,wolfy.i.,morih.,savchenkoa.v.,arrowsmithc.h.,koonine.v.,edwardsa.m.,yakunina.f.2006.genome-wideanalysisofsubstratespecificitiesoftheescherichiacolihaloaciddehalogenase-likephosphatasefamily.j.biol.chem.281:36149-36161.31.burnsd.m.,beachami.r.1986.nucleotidesequenceandtranscriptionalanalysisofthee.coliushagene,encodingperiplasmicudp-sugarhydrolase(5'-nucleotidase):regulationoftheushagene,andthesignalsequenceofitsencodedproteinproduct.nucleicacidsres.14:4325-4342.32.knoefelt.,straetern.1999.x-raystructureoftheescherichiacoliperiplasmic5'-nucleotidasecontainingadimetalcatalyticsite.nat.struct.biol.6:448-453.33.tremblayl.w.,dunaway-marianod.,allenk.n.2006.structureandactivityanalysesofescherichiacolik-12nagdprovideinsightintotheevolutionofbiochemicalfunctioninthehaloalkanoicaciddehalogenasesuperfamily.biochemistry45:1183-1193.34.golovans.,wangg.,zhangj.,forsbergc.w.2000.characterizationandoverproductionoftheescherichiacoliappaencodedbifunctionalenzymethatexhibitsbothphytaseandacidphosphataseactivities.can.j.microbiol.46:59-71.35.greinerr.,janyk.-d.1991.characterizationofaphytasefromescherichiacoli.biol.chem.hoppe-seyler372:664-665.36.elbakkourim,gutschei,kanjeeu,zhaob,yum,goretg,schoehng,burmeisterwp,hourywa.2010.structureofravamoxraaa+proteinrevealsthedesignprinciplesofamolecularcagemodulatingtheinduciblelysinedecarboxylaseactivity.procnatlacadsciusa107(52);22499-504.pmid:2114842037.tchigvintseva,tchigvintsevd,flickr,popovica,donga,xux,browng,luw,wuh,cuih,dombrowskil,joojc,beloglazovan,minj,savchenkoa,caudyaa,rabinowitzjd,murzinag,yakuninaf.2013.biochemicalandstructuralstudiesofconservedmafproteinsrevealednucleotidepyrophosphataseswithapreferenceformodifiednucleotides.chembiol20(11);1386-98.pmid:2421021938.zhangj.,inouyem.2002.mazg,anucleosidetriphosphatepyrophosphohydrolase,interactswithera,anessentialgtpaseinescherichiacoli.j.bacteriol.184:5323-5329.39.smallshawj.e.,kellnr.a.1992.cloning,nucleotidesequenceandexpressionoftheescherichiacolik-12pyrhgeneencodingumpkinase.lifesci.adv.(genet.)11:59-65.40.briozzop.,evrinc.,meyerp.,assairil.,jolyn.,barzuo.,gillesa.-m.2005.structureofescherichiacoliumpkinasediffersfromthatofothernucleosidemonophosphatekinasesandshedsnewlightonenzymeregulation.j.biol.chem.280:25533-25540.41.masuir.,kurokawak.,nakagawan.,tokunagaf.,koyamay.,shibatat.,oshimat.,yokoyamas.,yasunagat.,kuramitsus.completegenomesequenceofthermusthermophilushb8.submitted(nov-2004)totheembl/genbank/ddbjdatabases.42.marco-marinc.,escamilla-honrubiaj.m.,rubiov.2005.first-timecrystallizationandpreliminaryx-raycrystallographicanalysisofabacterial-archaealtypeumpkinase,akeyenzymeinmicrobialpyrimidinebiosynthesis.biochim.biophys.acta1747:271-275.43.marco-marinc.,escamilla-honrubiaj.m.,rubiov.2005.first-timecrystallizationandpreliminaryx-raycrystallographicanalysisofabacterial-archaealtypeumpkinase,akeyenzymeinmicrobialpyrimidinebiosynthesis.biochim.biophys.acta1747:271-275.44.frickej.,neuhardj.,kellnr.a.,pedersens.1995.thecmkgeneencodingcytidinemonophosphatekinaseislocatedintherpsaoperonandisrequiredfornormalreplicationrateinescherichiacoli.j.bacteriol.177:517-523.45.briozzop.,golinelli-pimpaneaub.,gillesa.m.,gaucherj.f.,burlacu-mirons.,sakamotoh.,janinj.,barzuo.1998.structuresofescherichiacolicmpkinasealoneandincomplexwithcdp:anewfoldofthenucleosidemonophosphatebindingdomainandinsightsintocytosinenucleotidespecificity.structure6:1517-1527.46.maederd.l.,weissr.b.,dunnd.m.,cherryj.l.,gonzalezj.m.,diruggieroj.,robbf.t.1999.divergenceofthehyperthermophilicarchaeapyrococcusfuriosusandp.horikoshiiinferredfromcompletegenomicsequences.genetics152:1299-1305.47.gentryd,bengrac,ikeharak,cashelm.1993.guanylatekinaseofescherichiacolik-12."jbiolchem1993;268(19);14316-21.pmid:8390989.48.hibleg,daalovap,gillesam,cherfilsj.2006.crystalstructuresofgmpkinaseincomplexwithganciclovirmonophosphateandap5g."biochimie88(9);1157-64.pmid:1669019749.nelsonk.e.,claytonr.a.,gills.r.,gwinnm.l.,dodsonr.j.,haftd.h.,hickeye.k.,petersonj.d.,nelsonw.c.,ketchumk.a.,mcdonaldl.a.,utterbackt.r.,malekj.a.,linherk.d.,garrettm.m.,stewarta.m.,cottonm.d.,prattm.s.fraserc.m.1999.evidenceforlateralgenetransferbetweenarchaeaandbacteriafromgenomesequenceofthermotogamaritima.nature399:323-329.50.brunem.,schumannr.,wittinghoferf.1985.cloningandsequencingoftheadenylatekinasegene(adk)ofescherichiacoli.nucleicacidsres.13:7139-7151.51.berrym.b.,baee.,bilderbackt.r.,glaserm.,phillipsg.n.jr.2006.crystalstructureofadp/ampcomplexofescherichiacoliadenylatekinase.proteins62:555-556.52.hennea.,brueggemannh.,raaschc.,wiezera.,hartscht.,liesegangh.,johanna.,lienardt.,gohlo.,martinez-ariasr.,jacobic.,starkuvienev.,schlenczecks.,denckers.,huberr.,klenkh.-p.,kramerw.,merklr.,gottschalkg.,fritzh.-j.2004.thegenomesequenceoftheextremethermophilethermusthermophilus.nat.biotechnol.22:547-553.53.tanzw,liuj,zhangxf,mengfg,zhangyz.nanfangyikedaxuexuebao.2010.expression,purificationandenzymaticcharacterizationofadenylatekinaseofthermusthermophilushb27inescherichiacoli..jan;30(1):1-654.moffattb.a.,dunnj.j.,studierf.w.1984.nucleotidesequenceofthegeneforbacteriophaget7rnapolymerase.j.mol.biol.173:265-269.55.sousar.,chungy.j.,rosej.p.,wangb.-c.1993.crystalstructureofbacteriophaget7rnapolymeraseat3.3-aresolution.nature364:593-599.56.mindichl.,nemhauseri.,gottliebp.,romantschukm.,cartonj.,fruchts.,strassmanj.,bamfordd.h.,kalkkinenn.1988.nucleotidesequenceofthelargedouble-strandedrnasegmentofbacteriophagephi6:genesspecifyingtheviralreplicaseandtranscriptase.j.virol.62:1180-1185.57.mcgrawn.j.,baileyj.n.,cleavesg.r.,dembinskid.r.,gockec.r.,joliffel.k.,macwrightr.s.,mcallisterw.t.1985.sequenceandanalysisofthegeneforbacteriophaget3rnapolymerase.nucleicacidsres.13:6753-6766.58.kotanih.,ishizakiy.,hiraokan.,obayashia.1987.nucleotidesequenceandexpressionoftheclonedgeneofbacteriophagesp6rnapolymerase.nucleicacidsres.15:2653-2664.序列大肠杆菌rna酶rmsqdpfqereaekyanpipsrefilehltkrekpasrdelavelhiegeeqleglrrrlramerdgqlvftrrqcyalperldlvkgtvighrdgygflrvegrkddlylsseqmktcihgdqvlaqplgadrkgrrearivrvlvpktsqivgryfteagvgfvvpddsrlsfdilippdqimgarmgfvvvveltqrptrrtkavgkivevlgdnmgtgmavdialrtheipyiwpqaveqqvaglkeevpeeakagrvdlrdlplvtidgedardfddavycekkrgggwrlwvaiadvsyyvrpstpldrearnrgtsvyfpsqvipmlpevlsnglcslnpqvdrlcmvcemtvsskgrltgykfyeavmssharltytkvwhilqgdqdlreqyaplvkhleelhnlykvldkareerggisfeseeakfifnaerrierieqtqrndahklieecmilanisaarfvekakepalfrihdkpsteaitsfrsvlaelglelpggnkpeprdyaellesvadrpdaemlqtmllrsmkqaiydpenrghfglalqsyahftspirrypdltlhraikyllakeqghqgnttetggyhysmeemlqlgqhcsmaerradeatrdvadwlkcdfmldqvgnvfkgvissvtgfgffvrlddlfidglvhvssldndyyrfdqvgqrlmgessgqtyrlgdrvevrveavnmderkidfslisseraprnvgktarekakkgdagkkggkrrqvgkkvnfepdsafrgekktkpkaakkdarkakkpsaktqkiaaatkakraakkkvae(seqidno:1)伸长热聚球藻ppk(ppk1)mpsaksprrkapepidldnpqyyfnrslswlefnkrvlheaydprtpllerlkfmaifssnldeffmvrvaglkqqvesgilqvgadgmppaeqlqavrqyllpivteqhryfdqelrallakesifltrfneltpeqqaylndyfqaqvfpvltplavdpahpfpyisslslnlavlirdpesgqerlarvkvpnqfprfvalpqhlhspqgvhwlgvpleeiiahnlsalfpgmeieayfafritrsadleletdkaddlliaieqeirkrrfgsvvrlevqrgippllrqtlmeemdleeidvyelegllclndlfafmglplpqfkdpewqpqvppsfqrveeresmfdtsseittlgtdyweavanelfsliregdiivhhpyhsfaatvqrfitlaahdpqvlaikitlyrtsgdspivsalikaaengkqvavlvelkarfdeennilwarklekvgvhvvygvpglkthtktvlvvrqeagqirryvhigtgnynpktaslyedlglfscreelgadlselfnvltgyarqrdyrkllvapvtmrdrtlqliyreieharngqpariiakmnaitdtqviralyeasqagvdidliirgmcclrpgvpgvsdrirvisiigrflehsrifyfgnngdpeyyigsadwrsrnldrrveaitpiedpaiqlelkerleimladnrqawelqpdgtyrqrqpapgeaergthsvlmartlkdvqgsh(seqidno:2)激烈火球菌umkmrivfdiggsvlvpenpdidfikeiayqltkvsedhevavvvgggklarkyievaekfnssetfkdfigiqitranamlliaalrekaypvvvedfweawkavqlkkipvmggthpghttdavaallaeflkadllvvitnvdgvytadpkkdptakkikkmkpeelleivgkgiekagsssvidplaakiiarsgiktivigkedakdlfrvikgdhngttiep(seqidno:3)嗜热栖热袍菌cmkmrgivtidgpsasgkssvarrvaaalgvpylssgllyraaaflalragvdpgdeegllalleglgvrllaqaegnrvladgedltsflhtpevdrvvsavarlpgvrawvnrrlkevpppfvaegrdmgtavfpeaahkfyltaspevrawrrarerpqayeevlrdllrrderdkaqsapapdalvldtggmtldevvawvlahirr(seqidno:4)海洋栖热袍菌gmkmkgqlfvicgpsgagktsiikevlkrldnvvfsvscttrpkrpheedgkdyffiteeeflkrvergeflewarvhghlygtlrsfveshinegkdvvldidvqgalsvkkkysntvfiyvappsyadlrerilkrgtekeadvlvrlenakwelmfmdefdyivvnenledavemvvsivrserakvtrnqdkierfkmevkgwkkl(seqidno:5)嗜热栖热袍菌adkmdvgqaviflgppgagkgtqasrlaqelgfkklstgdilrdhvargtplgervrpimergdlvpddlilelireelaervifdgfprtlaqaealdrllsetgtrllgvvlvevpeeelvrrilrraelegrsddneetvrrrlevyrekteplvgyyeargvlkrvdglgtpdevyariraalgi(seqidno:6)风产液菌ndkmavertliivkpdamekgalgkildrfiqegfqikalkmfrftpekagefyyvhrerpffqelvefmssgpvvaavlegedaikrvreiigptdseearkvapnsiraqfgtdkgknaihasdspesaqyeicfifsgleiv(seqidno:7)红色亚栖热袍菌dm1279ppk2mgfcsieflmgaqmkkyrvqpdgrfelkrfdpddtsafeggkqaalealavlnrrleklqellyaegqhkvlvvlqamdaggkdgtirvvfdgvnpsgvrvasfgvpteqelardylwrvhqqvprkgelvifnrshyedvlvvrvknlvpqqvwqkryrhirefermladegttilkfflhiskdeqrqrlqerldnpekrwkfrmgdledrrlwdryqeayeaairetsteyapwyvipanknwyrnwlvshilvetleglamqypqpetasekivie(seqidno:8)meiothermussilvanusdsm9946ppk2maktigatlnlqdidprstpgfngdkekalallekltarldelqeqlyaehqhrvlvilqgmdtsgkdgtirhvfknvdplgvrvvafkaptppelerdylwrvhqhvpangelvifnrshyedvlvarvhnlvppaiwsrrydhinafekmlvdegttvlkfflhiskeeqkkrllerlveadkhwkfdpqdlvergywedymeayqdvldkthtqyapwhvipadrkwyrnlqvsrllvealeglrmkyprpklniprlkselekm(seqidno:9)地热异常球菌dsm11300ppk2mqldryrvppgqrvrlsnwptdddgglskaegeallpdlqqrlanlqerlyaesqqallivlqardaggkdgtvkhvigafnpsgvqvsnfkvpteeerahdflwrihrqtprlgmigvfnrsqyedvlvtrvhhliddqtaqrrlkhicafeslltdsgtrivkfylhispeeqkkrlearladpskhwkfnpgdlqerahwdaytavyedvlttstpaapwyvvpadrkwfrnllvsqilvqtleemnpqfpapafnaadlriv(seqidno:10)伸长热聚球藻bp-1ppk2mipqdfldeinpdryivpaggnfhwkdydpgdtaglkskveaqellaagikklaayqdvlyaqniyglliifqamdaagkdstikhvmsglnpqacrvysfkapsaeeldhdflwranralpergcigifnrsyyeevlvvrvhpdllnrqqlppetktkhiwkerfedinhyeryltrngililkfflhiskaeqkkrflerisrpeknwkfsiedvrdrahwddyqqayadvfrhtstkwapwhiipanhkwfarlmvahfiyqklaslnlhypmlseahreqlleakallenepded(seqidno:11)嗜热厌氧绳菌uni-1ppk2mgeameryfikpgekvrlkdwspdppkdfegdkestraavaelnrklevlqerlyaerkhkvlvilqgmdtsgkdgvirsvfegvnpqgvkvanfkvptqeeldhdylwrvhkvvpgkgeivifnrshyedvlvvrvhnlvppevwkkryeqinqferllhetgttilkfflfisreeqkqrllerladpakhwkfnpgdlkeralweeyekayedvlsrtsteyapwilvpadkkwyrdwvisrvlvetlegleiqlpppladaetyrrqlleedapesr(seqidno:12)嗜气暖绳菌dsm14535ppk2mdvdryrvppgstihlsqwppddrslyegdkkqgkqdlsalnrrletlqellyaegkhkvliilqgmdtsgkdgvirhvfngvnpqgvkvasfkvptavelahdflwrihrqtpgsgeivifnrshyedvlvvrvhglvppevwarryehinafekllvdegttilkfflhiskeeqrqrllerlempekrwkfsvgdlaerkrwdeymaayeavlsktsteyapwyivpsdrkwyrnlvishviinaleglnmrypqpediafdtivie(seqidno:13)chlorobaculumtepidumtlsppk2mkldldafriqpgkkpnlakrptridpvyrskgeyhellanhvaelsklqnvlyadnryaillifqamdaagkdsaikhvmsgvnpqgcqvysfkhpsatelehdflwrtncvlpergrigifnrsyyeevlvvrvhpeilemqniphnlahngkvwdhryrsivsheqhlhcngtrivkfylhlskeeqrkrfleriddpnknwkfstadleerkfwdqymeayesclqetstkdspwfavpaddkknarlivsrivldtleslnlkypepsperrkelldirkrlenpengk(seqidno:14)深海洋栖热袍菌dsm14977ppk2mdvsryrvppgsgfdpeawptredddfaggkkeakkelarlavrlgelqarlyaegrqallivlqgmdtagkdgtirhvfravnpqgvrvtsfkkptalelahdylwrvhrhapargeigifnrshyedvlvvrvhelvppevwgrrydhinaferlladegtrivkfflhiskdeqkrrlearlenprkhwkfnpadlserarwgdyaaayaealsrtssdrapwyavpadrkwqrnrivaqvlvdaleamdprfprvdfdpasvrve(seqidno:15)roseiflexuscastenholziidsm13941ppk2myaqrvvpgmrvrlhdidpdangglnkdegrarfaelnaeldvmqeelyaagihalllilqgmdtagkdgairnvmlnlnpqgcrvesfkvpteeelahdflwrvhrvvprkgmvgvfnrshyedvlvvrvhslvpesvwrarydqinaferlladtgtiivkcflhiskeeqeqrllarerdvskawklsagdwrerafwddymaayeealtrcstdyapwyiipanrkwyrdlaisealvetlrpyrddwrraldamsrarraeleafraeqhamegrpqgaggvsrr(seqidno:16)玫瑰弯菌rs-1ppk2mhyahtvipgtqvrlrdidpdasggltkdegrerfasfnatldamqeelyaagvhalllilqgmdtagkdgairnvmhnlnpqgcrvesfkvpteeelahdflwrvhkvvprkgmvgvfnrshyedvlvvrvhslvpehvwrarydqinaferlltdtgtiivkcflhiskdeqekrllareqdvtkawklsagdwrererwdeymaayeealtrcsteyapwyiipanrkwyrdlaisevlvetlrpyrddwqraldamsqarlaelkafrhqqtagatrl(seqidno:17)trueperaradiovictrixdsm17093ppk2msqgsakglgkldkkvyarelallqlelvklqgwikaqglkvvvlfegrdaagkgstitritqplnprvcrvvalgapterertqwyfqryvhhlpaagemvlfdrswynragvervmgfcteaeyreflhacptferllldagiilikywfsvsaaeqerrmrrrnenpakrwklspmdleararwvayskakdamfyhtdtkaspwyvvnaedkrrahlsciahllslipyedltppplempprdlagadegyerpdkahqtwvpdyvpptr(seqidno:18)序列表<110>绿光生物科技股份有限公司<120>无细胞生成核糖核酸<130>g0830.70020wo00<140>尚未分配<141>2017-04-06<150>us62/452,550<151>2017-01-31<150>us62/319,220<151>2016-04-06<160>28<170>patentinversion3.5<210>1<211>813<212>prt<213>大肠杆菌<400>1metserglnasppropheglngluargglualaglulystyralaasn151015proileproserargglupheileleugluhisleuthrlysargglu202530lysproalaserargaspgluleualavalgluleuhisileglugly354045glugluglnleugluglyleuargargargleuargalametgluarg505560aspglyglnleuvalphethrargargglncystyralaleuproglu65707580argleuaspleuvallysglythrvalileglyhisargaspglytyr859095glypheleuargvalgluglyarglysaspaspleutyrleuserser100105110gluglnmetlysthrcysilehisglyaspglnvalleualaglnpro115120125leuglyalaasparglysglyargargglualaargilevalargval130135140leuvalprolysthrserglnilevalglyargtyrphethrgluala145150155160glyvalglyphevalvalproaspaspserargleuserpheaspile165170175leuileproproaspglnilemetglyalaargmetglyphevalval180185190valvalgluleuthrglnargprothrargargthrlysalavalgly195200205lysilevalgluvalleuglyaspasnmetglythrglymetalaval210215220aspilealaleuargthrhisgluileprotyriletrpproglnala225230235240valgluglnglnvalalaglyleulysglugluvalproglugluala245250255lysalaglyargvalaspleuargaspleuproleuvalthrileasp260265270glygluaspalaargasppheaspaspalavaltyrcysglulyslys275280285argglyglyglytrpargleutrpvalalailealaaspvalsertyr290295300tyrvalargproserthrproleuaspargglualaargasnarggly305310315320thrservaltyrpheproserglnvalileprometleuprogluval325330335leuserasnglyleucysserleuasnproglnvalaspargleucys340345350metvalcysglumetthrvalserserlysglyargleuthrglytyr355360365lysphetyrglualavalmetserserhisalaargleuthrtyrthr370375380lysvaltrphisileleuglnglyaspglnaspleuarggluglntyr385390395400alaproleuvallyshisleuglugluleuhisasnleutyrlysval405410415leuasplysalaargglugluargglyglyileserphegluserglu420425430glualalyspheilepheasnalagluargargilegluargileglu435440445glnthrglnargasnaspalahislysleuilegluglucysmetile450455460leualaasnileseralaalaargphevalglulysalalysglupro465470475480alaleupheargilehisasplysproserthrglualailethrser485490495pheargservalleualagluleuglyleugluleuproglyglyasn500505510lysprogluproargasptyralagluleuleugluservalalaasp515520525argproaspalaglumetleuglnthrmetleuleuargsermetlys530535540glnalailetyraspprogluasnargglyhispheglyleualaleu545550555560glnsertyralahisphethrserproileargargtyrproaspleu565570575thrleuhisargalailelystyrleuleualalysgluglnglyhis580585590glnglyasnthrthrgluthrglyglytyrhistyrsermetgluglu595600605metleuglnleuglyglnhiscyssermetalagluargargalaasp610615620glualathrargaspvalalaasptrpleulyscysaspphemetleu625630635640aspglnvalglyasnvalphelysglyvalileserservalthrgly645650655pheglyphephevalargleuaspaspleupheileaspglyleuval660665670hisvalserserleuaspasnasptyrtyrargpheaspglnvalgly675680685glnargleumetglygluserserglyglnthrtyrargleuglyasp690695700argvalgluvalargvalglualavalasnmetaspgluarglysile705710715720asppheserleuilesersergluargalaproargasnvalglylys725730735thralaargglulysalalyslysglyaspalaglylyslysglygly740745750lysargargglnvalglylyslysvalasnphegluproaspserala755760765pheargglyglulyslysthrlysprolysalaalalyslysaspala770775780arglysalalyslysproseralalysthrglnlysilealaalaala785790795800thrlysalalysargalaalalyslyslysvalalaglu805810<210>2<211>732<212>prt<213>伸长热聚球藻<400>2metproseralalysserproargarglysalaprogluproileasp151015leuaspasnproglntyrtyrpheasnargserleusertrpleuglu202530pheasnlysargvalleuhisglualatyraspproargthrproleu354045leugluargleulysphemetalailepheserserasnleuaspglu505560phephemetvalargvalalaglyleulysglnglnvalglusergly65707580ileleuglnvalglyalaaspglymetproproalagluglnleugln859095alavalargglntyrleuleuproilevalthrgluglnhisargtyr100105110pheaspglngluleuargalaleuleualalysgluserilepheleu115120125thrargpheasngluleuthrprogluglnglnalatyrleuasnasp130135140tyrpheglnalaglnvalpheprovalleuthrproleualavalasp145150155160proalahispropheprotyrileserserleuserleuasnleuala165170175valleuileargaspprogluserglyglngluargleualaargval180185190lysvalproasnglnpheproargphevalalaleuproglnhisleu195200205hisserproglnglyvalhistrpleuglyvalproleuglugluile210215220ilealahisasnleuseralaleupheproglymetgluilegluala225230235240tyrphealapheargilethrargseralaaspleugluleugluthr245250255asplysalaaspaspleuleuilealailegluglngluilearglys260265270argargpheglyservalvalargleugluvalglnargglyilepro275280285proleuleuargglnthrleumetgluglumetaspleuglugluile290295300aspvaltyrgluleugluglyleuleucysleuasnaspleupheala305310315320phemetglyleuproleuproglnphelysaspproglutrpglnpro325330335glnvalproproserpheglnargvalglugluargglusermetphe340345350aspthrsersergluilethrthrleuglythrasptyrtrpgluala355360365valalaasngluleupheserleuilearggluglyaspileileval370375380hishisprotyrhisserphealaalathrvalglnargpheilethr385390395400leualaalahisaspproglnvalleualailelysilethrleutyr405410415argthrserglyaspserproilevalseralaleuilelysalaala420425430gluasnglylysglnvalalavalleuvalgluleulysalaargphe435440445aspglugluasnasnileleutrpalaarglysleuglulysvalgly450455460valhisvalvaltyrglyvalproglyleulysthrhisthrlysthr465470475480valleuvalvalargglnglualaglyglnileargargtyrvalhis485490495ileglythrglyasntyrasnprolysthralaserleutyrgluasp500505510leuglyleuphesercysargglugluleuglyalaaspleuserglu515520525leupheasnvalleuthrglytyralaargglnargasptyrarglys530535540leuleuvalalaprovalthrmetargaspargthrleuglnleuile545550555560tyrarggluilegluhisalaargasnglyglnproalaargileile565570575alalysmetasnalailethraspthrglnvalileargalaleutyr580585590glualaserglnalaglyvalaspileaspleuileileargglymet595600605cyscysleuargproglyvalproglyvalseraspargileargval610615620ileserileileglyargpheleugluhisserargilephetyrphe625630635640glyasnasnglyaspproglutyrtyrileglyseralaasptrparg645650655serargasnleuaspargargvalglualailethrproilegluasp660665670proalaileglnleugluleulysgluargleugluilemetleuala675680685aspasnargglnalatrpgluleuglnproaspglythrtyrarggln690695700argglnproalaproglyglualagluargglythrhisservalleu705710715720metalaargthrleulysaspvalglnglyserhis725730<210>3<211>225<212>prt<213>激烈火球菌<400>3metargilevalpheaspileglyglyservalleuvalprogluasn151015proaspileasppheilelysgluilealatyrglnleuthrlysval202530sergluasphisgluvalalavalvalvalglyglyglylysleuala354045arglystyrilegluvalalaglulyspheasnsersergluthrphe505560lysasppheileglyileglnilethrargalaasnalametleuleu65707580ilealaalaleuargglulysalatyrprovalvalvalgluaspphe859095trpglualatrplysalavalglnleulyslysileprovalmetgly100105110glythrhisproglyhisthrthraspalavalalaalaleuleuala115120125glupheleulysalaaspleuleuvalvalilethrasnvalaspgly130135140valtyrthralaaspprolyslysaspprothralalyslysilelys145150155160lysmetlysproglugluleuleugluilevalglylysglyileglu165170175lysalaglyserserservalileaspproleualaalalysileile180185190alaargserglyilelysthrilevalileglylysgluaspalalys195200205aspleupheargvalilelysglyasphisasnglythrthrileglu210215220pro225<210>4<211>208<212>prt<213>嗜热栖热袍菌<400>4metargglyilevalthrileaspglyproseralaserglylysser151015servalalaargargvalalaalaalaleuglyvalprotyrleuser202530serglyleuleutyrargalaalaalapheleualaleuargalagly354045valaspproglyaspglugluglyleuleualaleuleugluglyleu505560glyvalargleuleualaglnalagluglyasnargvalleualaasp65707580glygluaspleuthrserpheleuhisthrprogluvalaspargval859095valseralavalalaargleuproglyvalargalatrpvalasnarg100105110argleulysgluvalproproprophevalalagluglyargaspmet115120125glythralavalpheproglualaalahislysphetyrleuthrala130135140serprogluvalargalatrpargargalaarggluargproglnala145150155160tyrglugluvalleuargaspleuleuargargaspgluargasplys165170175alaglnseralaproalaproaspalaleuvalleuaspthrglygly180185190metthrleuaspgluvalvalalatrpvalleualahisileargarg195200205<210>5<211>207<212>prt<213>海洋栖热袍菌<400>5metlysglyglnleuphevalilecysglyproserglyalaglylys151015thrserileilelysgluvalleulysargleuaspasnvalvalphe202530servalsercysthrthrargprolysargprohisglugluaspgly354045lysasptyrphepheilethrglugluglupheleulysargvalglu505560argglyglupheleuglutrpalaargvalhisglyhisleutyrgly65707580thrleuargserphevalgluserhisileasngluglylysaspval859095valleuaspileaspvalglnglyalaleuservallyslyslystyr100105110serasnthrvalpheiletyrvalalaproprosertyralaaspleu115120125arggluargileleulysargglythrglulysglualaaspvalleu130135140valargleugluasnalalystrpgluleumetphemetaspgluphe145150155160asptyrilevalvalasngluasnleugluaspalavalglumetval165170175valserilevalargsergluargalalysvalthrargasnglnasp180185190lysilegluargphelysmetgluvallysglytrplyslysleu195200205<210>6<211>186<212>prt<213>嗜热栖热袍菌<400>6metaspvalglyglnalavalilepheleuglyproproglyalagly151015lysglythrglnalaserargleualaglngluleuglyphelyslys202530leuserthrglyaspileleuargasphisvalalaargglythrpro354045leuglygluargvalargproilemetgluargglyaspleuvalpro505560aspaspleuileleugluleuileargglugluleualagluargval65707580ilepheaspglypheproargthrleualaglnalaglualaleuasp859095argleuleusergluthrglythrargleuleuglyvalvalleuval100105110gluvalprogluglugluleuvalargargileleuargargalaglu115120125leugluglyargseraspaspasnglugluthrvalargargargleu130135140gluvaltyrargglulysthrgluproleuvalglytyrtyrgluala145150155160argglyvalleulysargvalaspglyleuglythrproaspgluval165170175tyralaargileargalaalaleuglyile180185<210>7<211>142<212>prt<213>风产液菌<400>7metalavalgluargthrleuileilevallysproaspalametglu151015lysglyalaleuglylysileleuaspargpheileglngluglyphe202530glnilelysalaleulysmetpheargphethrproglulysalagly354045gluphetyrtyrvalhisarggluargprophepheglngluleuval505560gluphemetserserglyprovalvalalaalavalleugluglyglu65707580aspalailelysargvalarggluileileglyprothraspserglu859095glualaarglysvalalaproasnserileargalaglnpheglythr100105110asplysglylysasnalailehisalaseraspserprogluserala115120125glntyrgluilecyspheilepheserglyleugluileval130135140<210>8<211>280<212>prt<213>红色亚栖热袍菌<400>8metglyphecysserileglupheleumetglyalaglnmetlyslys151015tyrargvalglnproaspglyargphegluleulysargpheasppro202530aspaspthrseralaphegluglyglylysglnalaalaleugluala354045leualavalleuasnargargleuglulysleuglngluleuleutyr505560alagluglyglnhislysvalleuvalvalleuglnalametaspala65707580glyglylysaspglythrileargvalvalpheaspglyvalasnpro859095serglyvalargvalalaserpheglyvalprothrgluglngluleu100105110alaargasptyrleutrpargvalhisglnglnvalproarglysgly115120125gluleuvalilepheasnargserhistyrgluaspvalleuvalval130135140argvallysasnleuvalproglnglnvaltrpglnlysargtyrarg145150155160hisilearggluphegluargmetleualaaspgluglythrthrile165170175leulysphepheleuhisileserlysaspgluglnargglnargleu180185190glngluargleuaspasnproglulysargtrplyspheargmetgly195200205aspleugluaspargargleutrpaspargtyrglnglualatyrglu210215220alaalailearggluthrserthrglutyralaprotrptyrvalile225230235240proalaasnlysasntrptyrargasntrpleuvalserhisileleu245250255valgluthrleugluglyleualametglntyrproglnprogluthr260265270alaserglulysilevalileglu275280<210>9<211>268<212>prt<213>meiothermussilvanus<400>9metalalysthrileglyalathrleuasnleuglnaspileasppro151015argserthrproglypheasnglyasplysglulysalaleualaleu202530leuglulysleuthralaargleuaspgluleuglngluglnleutyr354045alagluhisglnhisargvalleuvalileleuglnglymetaspthr505560serglylysaspglythrilearghisvalphelysasnvalasppro65707580leuglyvalargvalvalalaphelysalaprothrproprogluleu859095gluargasptyrleutrpargvalhisglnhisvalproalaasngly100105110gluleuvalilepheasnargserhistyrgluaspvalleuvalala115120125argvalhisasnleuvalproproalailetrpserargargtyrasp130135140hisileasnalapheglulysmetleuvalaspgluglythrthrval145150155160leulysphepheleuhisileserlysglugluglnlyslysargleu165170175leugluargleuvalglualaasplyshistrplyspheaspprogln180185190aspleuvalgluargglytyrtrpgluasptyrmetglualatyrgln195200205aspvalleuasplysthrhisthrglntyralaprotrphisvalile210215220proalaasparglystrptyrargasnleuglnvalserargleuleu225230235240valglualaleugluglyleuargmetlystyrproargprolysleu245250255asnileproargleulyssergluleuglulysmet260265<210>10<211>266<212>prt<213>地热异常球菌<400>10metglnleuaspargtyrargvalproproglyglnargvalargleu151015serasntrpprothraspaspaspglyglyleuserlysalaglugly202530glualaleuleuproaspleuglnglnargleualaasnleuglnglu354045argleutyralagluserglnglnalaleuleuilevalleuglnala505560argaspalaglyglylysaspglythrvallyshisvalileglyala65707580pheasnproserglyvalglnvalserasnphelysvalprothrglu859095glugluargalahisasppheleutrpargilehisargglnthrpro100105110argleuglymetileglyvalpheasnargserglntyrgluaspval115120125leuvalthrargvalhishisleuileaspaspglnthralaglnarg130135140argleulyshisilecysalaphegluserleuleuthraspsergly145150155160thrargilevallysphetyrleuhisileserproglugluglnlys165170175lysargleuglualaargleualaaspproserlyshistrplysphe180185190asnproglyaspleuglngluargalahistrpaspalatyrthrala195200205valtyrgluaspvalleuthrthrserthrproalaalaprotrptyr210215220valvalproalaasparglystrppheargasnleuleuvalsergln225230235240ileleuvalglnthrleugluglumetasnproglnpheproalapro245250255alapheasnalaalaaspleuargileval260265<210>11<211>300<212>prt<213>伸长热聚球藻<400>11metileproglnasppheleuaspgluileasnproaspargtyrile151015valproalaglyglyasnphehistrplysasptyraspproglyasp202530thralaglyleulysserlysvalglualaglngluleuleualaala354045glyilelyslysleualaalatyrglnaspvalleutyralaglnasn505560iletyrglyleuleuileilepheglnalametaspalaalaglylys65707580aspserthrilelyshisvalmetserglyleuasnproglnalacys859095argvaltyrserphelysalaproseralaglugluleuasphisasp100105110pheleutrpargalaasnargalaleuprogluargglycysilegly115120125ilepheasnargsertyrtyrglugluvalleuvalvalargvalhis130135140proaspleuleuasnargglnglnleuproprogluthrlysthrlys145150155160hisiletrplysgluargphegluaspileasnhistyrgluargtyr165170175leuthrargasnglyileleuileleulysphepheleuhisileser180185190lysalagluglnlyslysargpheleugluargileserargproglu195200205lysasntrplyspheserilegluaspvalargaspargalahistrp210215220aspasptyrglnglnalatyralaaspvalphearghisthrserthr225230235240lystrpalaprotrphisileileproalaasnhislystrppheala245250255argleumetvalalahispheiletyrglnlysleualaserleuasn260265270leuhistyrprometleuserglualahisarggluglnleuleuglu275280285alalysalaleuleugluasngluproaspgluasp290295300<210>12<211>281<212>prt<213>嗜热厌氧绳菌<400>12metglyglualametgluargtyrpheilelysproglyglulysval151015argleulysasptrpserproaspproprolysaspphegluglyasp202530lysgluserthrargalaalavalalagluleuasnarglysleuglu354045valleuglngluargleutyralagluarglyshislysvalleuval505560ileleuglnglymetaspthrserglylysaspglyvalileargser65707580valphegluglyvalasnproglnglyvallysvalalaasnphelys859095valprothrglnglugluleuasphisasptyrleutrpargvalhis100105110lysvalvalproglylysglygluilevalilepheasnargserhis115120125tyrgluaspvalleuvalvalargvalhisasnleuvalproproglu130135140valtrplyslysargtyrgluglnileasnglnphegluargleuleu145150155160hisgluthrglythrthrileleulysphepheleupheileserarg165170175glugluglnlysglnargleuleugluargleualaaspproalalys180185190histrplyspheasnproglyaspleulysgluargalaleutrpglu195200205glutyrglulysalatyrgluaspvalleuserargthrserthrglu210215220tyralaprotrpileleuvalproalaasplyslystrptyrargasp225230235240trpvalileserargvalleuvalgluthrleugluglyleugluile245250255glnleuproproproleualaaspalagluthrtyrargargglnleu260265270leuglugluaspalaprogluserarg275280<210>13<211>270<212>prt<213>嗜气暖绳菌<400>13metaspvalaspargtyrargvalproproglyserthrilehisleu151015serglntrpproproaspaspargserleutyrgluglyasplyslys202530glnglylysglnaspleuseralaleuasnargargleugluthrleu354045glngluleuleutyralagluglylyshislysvalleuileileleu505560glnglymetaspthrserglylysaspglyvalilearghisvalphe65707580asnglyvalasnproglnglyvallysvalalaserphelysvalpro859095thralavalgluleualahisasppheleutrpargilehisarggln100105110thrproglyserglygluilevalilepheasnargserhistyrglu115120125aspvalleuvalvalargvalhisglyleuvalproprogluvaltrp130135140alaargargtyrgluhisileasnalapheglulysleuleuvalasp145150155160gluglythrthrileleulysphepheleuhisileserlysgluglu165170175glnargglnargleuleugluargleuglumetproglulysargtrp180185190lyspheservalglyaspleualagluarglysargtrpaspglutyr195200205metalaalatyrglualavalleuserlysthrserthrglutyrala210215220protrptyrilevalproserasparglystrptyrargasnleuval225230235240ileserhisvalileileasnalaleugluglyleuasnmetargtyr245250255proglnprogluaspilealapheaspthrilevalileglu260265270<210>14<211>294<212>prt<213>chlorobaculumtepidum<400>14metlysleuaspleuaspalapheargileglnproglylyslyspro151015asnleualalysargprothrargileaspprovaltyrargserlys202530glyglutyrhisgluleuleualaasnhisvalalagluleuserlys354045leuglnasnvalleutyralaaspasnargtyralaileleuleuile505560pheglnalametaspalaalaglylysaspseralailelyshisval65707580metserglyvalasnproglnglycysglnvaltyrserphelyshis859095proseralathrgluleugluhisasppheleutrpargthrasncys100105110valleuprogluargglyargileglyilepheasnargsertyrtyr115120125glugluvalleuvalvalargvalhisprogluileleuglumetgln130135140asnileprohisasnleualahisasnglylysvaltrpasphisarg145150155160tyrargserilevalserhisgluglnhisleuhiscysasnglythr165170175argilevallysphetyrleuhisleuserlysglugluglnarglys180185190argpheleugluargileaspaspproasnlysasntrplyspheser195200205thralaaspleuglugluarglysphetrpaspglntyrmetgluala210215220tyrglusercysleuglngluthrserthrlysaspserprotrpphe225230235240alavalproalaaspasplyslysasnalaargleuilevalserarg245250255ilevalleuaspthrleugluserleuasnleulystyrproglupro260265270serprogluargarglysgluleuleuaspilearglysargleuglu275280285asnprogluasnglylys290<210>15<211>269<212>prt<213>深海洋栖热袍菌<400>15metaspvalserargtyrargvalproproglyserglypheasppro151015glualatrpprothrarggluaspaspaspphealaglyglylyslys202530glualalyslysgluleualaargleualavalargleuglygluleu354045glnalaargleutyralagluglyargglnalaleuleuilevalleu505560glnglymetaspthralaglylysaspglythrilearghisvalphe65707580argalavalasnproglnglyvalargvalthrserphelyslyspro859095thralaleugluleualahisasptyrleutrpargvalhisarghis100105110alaproalaargglygluileglyilepheasnargserhistyrglu115120125aspvalleuvalvalargvalhisgluleuvalproprogluvaltrp130135140glyargargtyrasphisileasnalaphegluargleuleualaasp145150155160gluglythrargilevallysphepheleuhisileserlysaspglu165170175glnlysargargleuglualaargleugluasnproarglyshistrp180185190lyspheasnproalaaspleusergluargalaargtrpglyasptyr195200205alaalaalatyralaglualaleuserargthrserseraspargala210215220protrptyralavalproalaasparglystrpglnargasnargile225230235240valalaglnvalleuvalaspalaleuglualametaspproargphe245250255proargvalasppheaspproalaservalargvalglu260265<210>16<211>290<212>prt<213>roseiflexuscastenholzii<400>16mettyralaglnargvalvalproglymetargvalargleuhisasp151015ileaspproaspalaasnglyglyleuasnlysaspgluglyargala202530argphealagluleuasnalagluleuaspvalmetglnglugluleu354045tyralaalaglyilehisalaleuleuleuileleuglnglymetasp505560thralaglylysaspglyalaileargasnvalmetleuasnleuasn65707580proglnglycysargvalgluserphelysvalprothrglugluglu859095leualahisasppheleutrpargvalhisargvalvalproarglys100105110glymetvalglyvalpheasnargserhistyrgluaspvalleuval115120125valargvalhisserleuvalprogluservaltrpargalaargtyr130135140aspglnileasnalaphegluargleuleualaaspthrglythrile145150155160ilevallyscyspheleuhisileserlysglugluglngluglnarg165170175leuleualaarggluargaspvalserlysalatrplysleuserala180185190glyasptrparggluargalaphetrpaspasptyrmetalaalatyr195200205gluglualaleuthrargcysserthrasptyralaprotrptyrile210215220ileproalaasnarglystrptyrargaspleualailesergluala225230235240leuvalgluthrleuargprotyrargaspasptrpargargalaleu245250255aspalametserargalaargargalagluleuglualapheargala260265270gluglnhisalametgluglyargproglnglyalaglyglyvalser275280285argarg290<210>17<211>282<212>prt<213>玫瑰弯菌属种<400>17methistyralahisthrvalileproglythrglnvalargleuarg151015aspileaspproaspalaserglyglyleuthrlysaspgluglyarg202530gluargphealaserpheasnalathrleuaspalametglngluglu354045leutyralaalaglyvalhisalaleuleuleuileleuglnglymet505560aspthralaglylysaspglyalaileargasnvalmethisasnleu65707580asnproglnglycysargvalgluserphelysvalprothrgluglu859095gluleualahisasppheleutrpargvalhislysvalvalproarg100105110lysglymetvalglyvalpheasnargserhistyrgluaspvalleu115120125valvalargvalhisserleuvalprogluhisvaltrpargalaarg130135140tyraspglnileasnalaphegluargleuleuthraspthrglythr145150155160ileilevallyscyspheleuhisileserlysaspgluglnglulys165170175argleuleualaarggluglnaspvalthrlysalatrplysleuser180185190alaglyasptrparggluarggluargtrpaspglutyrmetalaala195200205tyrgluglualaleuthrargcysserthrglutyralaprotrptyr210215220ileileproalaasnarglystrptyrargaspleualaileserglu225230235240valleuvalgluthrleuargprotyrargaspasptrpglnargala245250255leuaspalametserglnalaargleualagluleulysalaphearg260265270hisglnglnthralaglyalathrargleu275280<210>18<211>274<212>prt<213>trueperaradiovictrix<400>18metserglnglyseralalysglyleuglylysleuasplyslysval151015tyralaarggluleualaleuleuglnleugluleuvallysleugln202530glytrpilelysalaglnglyleulysvalvalvalleupheglugly354045argaspalaalaglylysglyserthrilethrargilethrglnpro505560leuasnproargvalcysargvalvalalaleuglyalaprothrglu65707580arggluargthrglntrptyrpheglnargtyrvalhishisleupro859095alaalaglyglumetvalleupheaspargsertrptyrasnargala100105110glyvalgluargvalmetglyphecysthrglualaglutyrargglu115120125pheleuhisalacysprothrphegluargleuleuleuaspalagly130135140ileileleuilelystyrtrppheservalseralaalagluglnglu145150155160argargmetargargargasngluasnproalalysargtrplysleu165170175serprometaspleuglualaargalaargtrpvalalatyrserlys180185190alalysaspalametphetyrhisthraspthrlysalaserprotrp195200205tyrvalvalasnalagluasplysargargalahisleusercysile210215220alahisleuleuserleuileprotyrgluaspleuthrpropropro225230235240leuglumetproproargaspleualaglyalaaspgluglytyrglu245250255argproasplysalahisglnthrtrpvalproasptyrvalpropro260265270thrarg<210>19<211>26<212>prt<213>人工序列<220><223>合成多肽<400>19metlysilelysthrglyalaargileleualaleuseralaleuthr151015thrmetmetpheseralaseralaleuala2025<210>20<211>21<212>prt<213>人工序列<220><223>合成多肽<400>20metlysglnserthrilealaleualaleuleuproleuleuphethr151015provalthrlysala20<210>21<211>25<212>prt<213>人工序列<220><223>合成多肽<400>21metmetilethrleuarglysleuproleualavalalavalalaala151015glyvalmetseralaglnalametala2025<210>22<211>23<212>prt<213>人工序列<220><223>合成多肽<400>22metasnlyslysvalleuthrleuseralavalmetalasermetleu151015pheglyalaalaalahisala20<210>23<211>22<212>prt<213>人工序列<220><223>合成多肽<400>23metlystyrleuleuprothralaalaalaglyleuleuleuleuala151015alaglnproalametala20<210>24<211>19<212>prt<213>人工序列<220><223>合成多肽<400>24metlyslysiletrpleualaleualaglyleuvalleualapheser151015alaserala<210>25<211>23<212>prt<213>人工序列<220><223>合成多肽<400>25metmetthrlysilelysleuleumetleuileilephetyrleuile151015ileseralaseralahisala20<210>26<211>21<212>prt<213>人工序列<220><223>合成多肽<400>26metlysglnalaleuargvalalapheglypheleuileleutrpala151015servalleuhisala20<210>27<211>18<212>prt<213>人工序列<220><223>合成多肽<400>27metargvalleuleupheleuleuleuserleuphemetleuproala151015pheser<210>28<211>39<212>prt<213>人工序列<220><223>合成多肽<400>28metalaasnasnaspleupheglnalaserargargargpheleuala151015glnleuglyglyleuthrvalalaglymetleuglyproserleuleu202530thrproargargalathrala35当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1