结直肠癌标志物及其应用的制作方法
本发明涉及基因检测领域,具体涉及一种结直肠癌标志物及其应用,尤其涉及一种结直肠癌标志物、引物对、核酸探针、试剂盒以及对待测者进行结直肠癌检测的系统。
背景技术:
据统计,结直肠癌(colorectalcancer,crc)在各类癌症中发病率排名第三,致死率排名第四,近55%的发病案例发生在较发达国家。在年龄小于40岁的人中发生率较低,但随着年龄增加发生率亦逐渐增高。通常认为饮食习惯的西方化会增加crc发病率,但目前美国及其他高收入国家发病率却保持稳定甚至下降,推测与乙状结肠镜检查及结肠镜下的息肉切除术的增加有关。结直肠癌也是目前最可预防的肿瘤之一,医学界认为如果及早发现,肠癌是最易治愈的癌症。传统的结直肠癌筛选方法主要是结肠镜检查,另外还有活体组织检查和脱落细胞学检查,这几种检查方法都是侵入式的,尤其是脱落细胞学检查取材繁琐,不易获得满意的标本,临床应用少。所以如果没有相关症状,很多人并不愿意去做结直肠癌的早期筛查。
因此,针对结直肠癌的早期诊断和筛查,还需要进一步改进。
技术实现要素:
本发明旨在至少在一定程度上解决相关技术中的技术问题之一。为此,本发明的一个目的在于提出一种结直肠癌肿瘤标志物、引物对、试剂盒以及对待测者进行结直肠癌检测的系统。本发明提供的结直肠癌肿瘤标志物能够用于结直肠癌病人的早期筛查和诊断。
本发明是基于发明人的如下发现所获得的:
人体肠道微生物组包含了数量巨大的微生物,它们参与食物药物代谢、人体免疫调节,在保持人体健康中起着极其重要的作用。有大量文献报道消化链球菌与结直肠癌有很强的相关性,研究疾病的不同阶段中微生物组成分的改变能够作为一种新的用于癌症诊断及预后效果测试的手段:因此本研究想通过测定肠道中消化链球菌的特定基因,来对结直肠癌的发生与发展进行早期检测和及时干预治疗。
在有case-control的人群中对消化链球菌的特异性基因进行定量pcr(qpcr)检测,确定基因的量,分析并验证这些基因预测结直肠癌的概率。以开发结直肠癌患者的肠道微生物基因检测试剂盒,为广大人群提供快速简便的结肠癌早期筛查、诊断的有效措施。为实现结直肠癌的早期预警、诊断和干预提供依据。
具体而言,本发明提供了如下技术方案:
根据本发明的第一方面,本发明提供了一种结直肠癌标志物,所述结直肠癌标志物包括选自下列中的至少一种:seqidno:1;或者来源于消化链球菌且与所述seqidno:1具有95%以上同源性的核酸序列。研究表明消化链球菌与结直肠癌具有很强的相关性,本发明通过利用宏基因组测序研究,筛选了消化链球菌中与结直肠癌相关性强的特定核酸序列,作为结直肠癌检测的标志物,能够用于结直肠癌的早期诊断和筛查。其中,结直肠癌标志物可以为seqidno:1,也可以是来源于消化链球菌且与seqidno:1具有95%以上同源性的核酸序列,优选为具有98%以上同源性的核酸序列,更优选为具有99%以上同源性的核酸序列。通过这些标志物可以用来反映结直肠癌的情况。
在本发明的一些实施例中,所述来源于消化链球菌且与所述seqidno:1具有同源性的核酸序列可以表现为:与seqidno:1所示核酸序列存在密码子简并的核酸序列。即将seqidno:1所示核酸序列进行同义替换所得到的核酸序列,也可以作为结直肠癌标志物。
根据本发明的第二方面,本发明提供了一组引物对,所述引物对能够特异性扩增本发明第一方面所述的结直肠癌标志物中的至少之一,所述引物对选自seqidno:6~seqidno:13中的任意一对,其中seqidno:6~seqidno:13中每两个相邻编号的核酸序列为一对。通过这些引物对,能够特异性扩增seqidno:1或其同源序列,从而可以用作结直肠癌的早期诊断和检测。
根据本发明的第三方面,本发明提供了一种核酸探针,所述核酸探针能特异性捕获包括本发明第一方面所述的结直肠癌标志物。设计核酸探针捕获本发明所用到的结直肠癌标志物,能够实现结直肠癌的快速检测。
根据本发明的第四方面,本发明提供了一种构建体,所述构建体包括根据本发明第一方面所述的结直肠癌标志物。通过将结直肠癌标志物构建到载体上,形成构建体,可以用作阳性标准品,用来表征结直肠癌的诊断情况。
在本发明的一些实施例中,所述构建体为含有本发明第一方面所述的结直肠癌标志物的重组载体。
在本发明的一些实施例中,所述载体为质粒。例如利用puc57质粒,将本发明第一方面所述的结直肠癌标志物构建到质粒中,形成重组质粒,从而可以作为阳性标准品,用来表征结直肠癌的诊断情况。
根据本发明的第五方面,本发明提供了一种试剂盒,所述试剂盒包括用于检测本发明第一方面所述的结直肠癌标志物的试剂。
在本发明的一些实施例中,所述试剂盒包括以下至少之一:本发明第二方面所述的引物对;本发明第三方面所述的核酸探针;和/或本发明第四方面所述的构建体。
根据本发明的第六方面,本发明提供了一种对待测者进行结直肠癌检测的系统,包括:定量装置,所述定量装置用来确定所述待测者的粪便dna中的所述靶标核酸的含量,所述靶标核酸为本发明第一方面所述的标志物;判断装置,所述判断装置与所述定量装置相连,所述判断装置基于所述靶标核酸的含量,确定所述待测者是否为结直肠癌患者。本发明所述“对待测者进行结直肠癌检测的系统”也可以根据需要表述为“对待测者进行结直肠癌检测的设备”,指的是能够用于结直肠癌检测的一种产品或者装置。
在本发明的一些实施例中,以上对待测者进行结直肠癌检测的系统可以进一步附加如下技术特征:
在本发明的一些实施例中,所述定量装置能够按照包括如下步骤的方法确定所述待测者的粪便dna中的所述靶标核酸的含量:对所述靶标核酸进行pcr扩增,确定所述待测者的粪便dna中的所述靶标核酸的含量。
在本发明的一些实施例中,所述pcr为荧光定量pcr。
在本发明的一些实施例中,所述判断装置基于如下标准来确定所述待测者是否为结直肠癌患者:所述靶标核酸的含量大于等于预定阈值,则所述待测者为结直肠癌患者;所述靶标核酸的含量小于预定阈值,则所述待测者为非结直肠癌患者。
在本发明的一些实施例中,所述预定阈值是通过下列方法确定的:以由结直肠癌患者和非结直肠癌患者组成的群体为研究对象,对所述群体中每个个体的粪便dna中的所述靶标核酸分别进行荧光定量pcr扩增,以便获得检测结果;基于所述检测结果绘制roc曲线,并从所述roc曲线中找出灵敏度和特异性最佳的点;基于所述灵敏度和特异性最佳的点,对应所述荧光定量pcr,得到所述靶标核酸的拷贝数(绝对数值)即为所述预定阈值。根据本发明的实施例,在利用检测结果绘制roc曲线的时候,依照结直肠癌患者和非结直肠癌患者分组的方式绘制roc曲线。
其中,所述靶标核酸的拷贝数(绝对数值)是采用标准曲线法获得的,用的是已知拷贝数的标准品做的标准曲线。根据本发明的实施例,所述预定阈值为73.09943。
附图说明
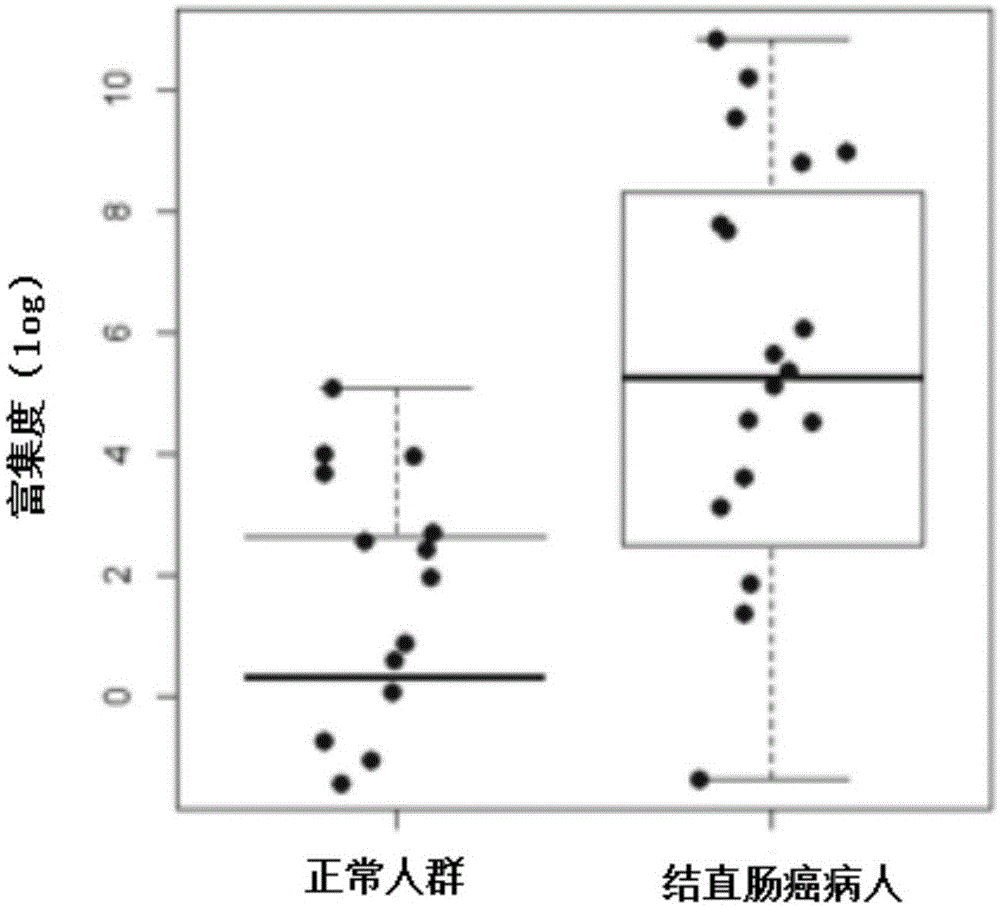
图1是根据本发明的一个实施例提供的箱形图(boxplot)结果。
图2是根据本发明的一个实施例提供的接收者操作特征曲线图(receiveroperatingcharacteristiccurve,roc曲线)。
图3是根据本发明的一些实施例提供的对待测者进行结直肠癌检测的系统的示意图。
具体实施方式
下面详细描述本发明的实施例,所述实施例的示例在附图中示出,其中自始至终相同或类似的标号表示相同或类似的元件或具有相同或类似功能的元件。下面通过参考附图描述的实施例是示例性的,旨在用于解释本发明,而不能理解为对本发明的限制。
本研究主要是建立一种通过肠道微生物中消化链球菌的特异基因序列来筛查结直肠癌的方法。本发明通过筛选获得新的结直肠癌标志物,利用这些标志物通过对待测样本的粪便dna进行检测,实现无创低成本的早期筛查或诊断技术。利用可以通过高通量测序技术对待测者的粪便dna中的核酸进行测序,来获得这些标志物的含量信息,从而表征待测者的患病情况。再比如可以借助于荧光定量pcr技术通过对待测者粪便dna中的核酸进行表征,来获得关于这些标志物的含量信息,且借助于荧光定量pcr技术进行检测,可以方便快速获得检测结果,有利于对于结直肠癌检测的推广和产业化。
为此,根据本发明的一个方面,本发明提供了一种结直肠癌标志物,所述标志物选自seqidno:1。其中seqidno:1来自于消化链球菌。本发明通过全基因组关联分析(metabolomegenome-wideassocitaionstudy,mgwas)方法对消化链球菌的特异性序列进行筛选,获得seqidno:1来表征结直肠癌的患病情况。
本文中,消化链球菌指的是消化链球菌属中所有菌群,包括厌氧消化链球菌(p.anaerobius)、大消化链球菌(p.magnus)、微小消化链球菌(p.micros)、不解糖消化链球菌(p.asaccharolyticus)、普氏消化链球菌(p.prevotii)、四链消化链球菌(p.tetradius)及延展消化链球菌(p.productus)等。
具体来说,通过mgwas方法寻找结直肠癌肠道菌群标志物,可以采用本领域文献中记载的方法。例如具体可以包括:将reads比对到消化链球菌的基因上,得到基因相对丰度数据;根据基因丰度挑选检验差异最显著的几个基因(即特异性序列);然后利用群体样本进行验证,例如可以利用小样本和大样本分别进行验证,来评价不同特异性序列对于结直肠癌的表征效果。
在本发明的另一个方面,本发明提供了一种对待测者进行结直肠癌检测的方法,包括:(1)基于所述待测者的粪便,确定所述待测者的粪便dna中的所述靶标核酸的含量,所述靶标核酸为seqidno:1;(2)基于所述靶标核酸的含量,确定所述待测者是否为结直肠癌患者。
在本发明的一些实施例中,以上所述对待测者进行结直肠癌检测的方法可以进一步附加如下技术特征:
在本发明的一些实施例中,步骤(1)中,基于对所述待测者的粪便dna中的靶标核酸进行pcr扩增,确定所述待测者的粪便dna中的所述靶标核酸的含量。
在本发明的一些实施例中,所述pcr为荧光定量pcr。
在本发明的一些实施例中,所述靶标核酸的含量大于等于预定阈值,则所述待测者为结直肠癌患者;所述靶标核酸的含量小于预定阈值,则所述待测者不为结直肠癌患者。
在本发明的一些实施例中,所述预定阈值是通过下列方法确定的:以由结直肠癌患者和非结直肠癌患者组成的群体为研究对象,对所述群体中每个个体的粪便dna中的所述靶标核酸分别进行荧光定量pcr扩增,以便获得检测结果;基于所述检测结果,按照结直肠癌患者和非结直肠癌患者分组的方式绘制roc曲线,并从所述roc曲线中找出灵敏度和特异性最佳的点;基于所述灵敏度和特异性最佳的点,对应所述荧光定量pcr,得到所述靶标核酸的拷贝数(绝对数值)即为所述预定阈值。
根据本发明的另一方面,本发明提供了一种对待测者进行结直肠癌检测的系统,如图3所示,包括:定量装置和判断装置,所述定量装置和所述判断装置相连,所述定量装置通过对所述待测者的粪便dna中的靶标核酸进行pcr扩增,确定所述待测者的粪便dna中的所述靶标核酸的含量,所述靶标核酸为seqidno:1;所述判断装置基于所述靶标核酸的含量,确定所述待测者是否为结直肠癌患者。根据本发明的实施例,所述判断装置基于如下标准来确定所述待测者是否为结直肠癌患者:所述靶标核酸的含量大于等于预定阈值,则所述待测者为结直肠癌患者;所述靶标核酸的含量小于预定阈值,则所述待测者为非结直肠癌患者。
下面将结合实施例对本发明的方案进行解释。本领域技术人员将会理解,下面的实施例仅用于说明本发明,而不应视为限定本发明的范围。实施例中未注明具体技术或条件的,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购获得的常规产品。
实施例1
消化链球菌特异序列筛选:具体通过mgwas方法寻找结直肠癌肠道菌群标志物(该方法是本领域公知的,文献中有详细描述,可以根据需要将文献junyu,qiangfengeal.,.metagenomicanalysisoffaecalmicrobiomeasatooltowardstargetednon-invasivebiomarkersforcolorectalcancer.gut2015;0:1–9.doi:10.1136/gutjnl-2015-309800全部或者部分引入本申请中);通过mwas分析可以得到与疾病相关的marker,如下为其大概过程:将reads比对到消化链球菌的基因上,得到基因相对丰度数据;然后根据基因丰度挑选检验差异最显著的几个基因。
本实施例主要针对消化链球菌(peptostreptococcusstomatis菌)筛选的特异基因序列,具体序列如下所示:
表1特异基因序列
实施例2
采用oligo7软件对筛选得到的特异菌群的特异序列进行引物设计,尽可能满足重要原则的基础上,要求上下引物扩增产物在70bp~200bp,每个基因各设计3~5套引物。
针对实施例1筛选获得的特异性序列,设计了如下所示的引物序列,用于各特异性序列的扩增,所设计的引物序列如下表所示:
表2引物序列
对于每种特异性序列所设计的特异性引物进行引物测试:即通过普通pcr扩增,琼脂糖凝胶电泳检测其条带,若目的条带大小与预期一致且条带单一,说明引物特异性好。经过检测发现,利用各引物进行扩增,目的条带大小与预期一致且条带单一,说明利用本发明实施例2提供的引物,扩增的特异性好。具体而言,在对标志物seqidno:1进行扩增的时候,用到的引物序列为seqidno:6和seqidno:7表现出最佳的扩增效果;在对标志物seqidno:2进行扩增的时候,用到的引物序列为seqidno:14和seqidno:15表现出最佳的扩增效果;在对标志物seqidno:3进行扩增的时候,用到的引物序列为seqidno:20和seqidno:21表现出最佳的扩增效果;在对标志物seqidno:4进行扩增的时候,用到的引物序列为seqidno:28和seqidno:29表现出最佳的扩增效果;在对标志物seqidno:5进行扩增的时候,用到的引物序列为seqidno:32和seqidno:33表现出最佳的扩增效果。
实施例3
本实施例主要基于qpcr技术对结直肠癌患者和正常人粪便dna进行测试,所示出的结果是按照实施例2提供的最佳引物所提供的结果。首先使用20例结直肠癌病人粪便dna和20例正常人粪便dna进行qpcr检测(所使用的试剂为:sybrpremixextaq(tm)ii(perfectrealtime)货号:drr081a)。其中,sybrpremixextaq(tm)ii为采用sybrgreeni嵌合法进行realtimepcr(实时荧光定量pcr)的专用试剂。通过pcr反应生成双链dna,sybrgreeni能够与双链dna结合后发出荧光,通过检测pcr反应液中的荧光信号强度,可以对目的基因进行准确定量。具体而言,在pcr反应体系中,加入过量sybr荧光染料,sybr荧光染料特异性地掺入双链dna后,发射荧光信号,而不掺入链中的sybr染料分子不会发射任何荧光信号,从而能够保证荧光信号的增加与pcr产物的增加完全同步。
本方法使用的是标准曲线法,本发明采用的标准品为分别含有seqidno:1、seqidno:2、seqidno:3、seqidno:4、seqidno:5对应序列,阳性标准品即为将这些序列克隆到载体上的质粒;该部分标准品为商业公司帮忙合成(南京金斯瑞公司,标准载体puc57)。商业公司寄过来的质粒标准品为干粉,将干粉离心后稀释,测定其浓度后换算为对应的拷贝数,然后进行梯度稀释,稀释5个梯度作为标准曲线(标准曲线的横坐标为拷贝数,纵坐标为ct值)。通过利用一系列已知浓度的标准品制作标准曲线,在相同的条件下目的基因所测得的荧光信号量同标准曲线进行比较,从而得到目的基因的量,从而实现目的基因拷贝数的绝对定量。
实验具体操作步骤如下:
配制反应液:将引物、dna模板、roxreferencedye、sybrpremixextaqii配制成10μl反应液,加入到96孔反应板上,具体如下:
2)qpcr扩增:将96孔板放在qpcr仪上,进行扩增检测;
3)分析数据:使用qpcr软件导出结果,根据qpcr的定量结果使用r语言对数据进行分析作图,按照结直肠癌病人、正常人画箱形图(boxplot)和接收者操作特征曲线图(roc曲线);箱形图是利用数据中的几个统计量,包括:最小值、下四分位数,中位数,上四分位数以及最大值等来描述数据的一种方法,利用箱线图可以看出数据是否有对称性、分布的离散程度等信息。
以seqidno:1作为标志物,以seqidno:6和seqidno:7作为引物序列为例,所得到的结果如图1和图2所示,其中图1是一种箱形图,显示结肠癌病人和正常人两组数据的分散情况;图中横坐标代表正常人和结肠癌两类人群,纵坐标代表富集度。其中每个箱的最下面一条线代表下四分位数;中间那条线代表中位数;最上面那条线代表上四分位数;两个人群的中位数差距越大,代表两个人群区分的越开,该肿瘤标志物的检测效率越高。图中示出的每个点代表qpcr检测结果经过进一步计算对应的值。
图2是接收者操作特征曲线(roc曲线),图中横坐标代表特异性,纵坐标代表敏感性;中间那条对角线代表auc=0.5的情况,图中得到的auc为0.818,说明该肿瘤标志物检测效果较好。图中圆圈圈起来的那个点即为灵敏度和特异性最佳的点,分别为65%和95%,对应的qpcr的阈值(cutoff值)为73.09943,利用该阈值可以对结直肠癌的患病情况进行判断,qpcr得到的质量值(quantity值)大于等于该值即可判断为结直肠癌病人,小于该值可判断为正常,此处qpcr所得的quantity值,即为标准曲线得出的待测样本中靶标基因的拷贝数绝对值。
具体数据见下表3(利用seqidno:6和seqidno:7作为引物序列对seqidno:1进行荧光定量pcr检测的结果)。
表320例正常人和20例病人的qpcr检测结果
注:表中“-”代表检测结果低于cutoff值,判断结果为正常;“+”代表检测结果高于cutoff值,判断结果为结直肠癌肿瘤患者。
从表3可以看出,利用seqidno:6和seqidno:7对seqidno:1进行荧光定量pcr检测,在对正常样本进行测定的过程中,在20例样本中仅有1例样本测定的质量值大于阈值,在对结直肠癌患者样本进行测定的过程中,在20例样本中有7例样本测定的质量值小于阈值;说明利用seqidno:1结直肠癌标志物可准确高效的用于结直肠癌的检测。
同样地,以seqidno:2作为标志物对这20例正常人和20例结直肠癌病人进行检测,以seqidno:3作为标志物对这20例正常人和20例结直肠癌病人进行检测,以seqidno:4作为标志物对这20例正常人和20例结直肠癌病人进行检测,以seqidno:5作为标志物对这20例正常人和20例结直肠癌病人进行检测,检测结果表明,以seqidno:2~seqidno:5所示序列作为标志物进行检测,并不能获得准确高效的检测效果。
结果表明,同样来源于消化链球菌的特定序列,只有seqidno:1所示序列作为标志物可以准确高效的用于结直肠癌的检测。
同样地,分别以seqidno:1作为标志物,以实施例2中给出的最佳引物按照上述方法对大样本(100例结直肠癌病人和100例正常样本)进行验证,结果表明这些标志物可以准确高效的用于结直肠癌的检测。
在本发明中,除非另有明确的规定和限定,术语“安装”、“相连”、“连接”、“固定”等术语应做广义理解,例如,可以是固定连接,也可以是可拆卸连接,或成一体;可以是机械连接,也可以是电连接或彼此可通讯;可以是直接相连,也可以通过中间媒介间接相连,可以是两个元件内部的连通或两个元件的相互作用关系,除非另有明确的限定。对于本领域的普通技术人员而言,可以根据具体情况理解上述术语在本发明中的具体含义。
此外,术语“第一”、“第二”仅用于描述目的,而不能理解为指示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”的特征可以明示或者隐含地包括至少一个该特征。在本发明的描述中,“多个”的含义是至少两个,例如两个,三个等,除非另有明确具体的限定。
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不必针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任一个或多个实施例或示例中以合适的方式结合。此外,在不相互矛盾的情况下,本领域的技术人员可以将本说明书中描述的不同实施例或示例以及不同实施例或示例的特征进行结合和组合。
尽管上面已经示出和描述了本发明的实施例,可以理解的是,上述实施例是示例性的,不能理解为对本发明的限制,本领域的普通技术人员在本发明的范围内可以对上述实施例进行变化、修改、替换和变型。
sequencelisting
<110>深圳华大生命科学研究院
<120>结直肠癌标志物及其应用
<130>pidc3184868
<160>35
<170>patentinversion3.5
<210>1
<211>495
<212>dna
<213>人工序列
<220>
<223>特异序列
<400>1
cctgacaggggccagatagatataggtgggcaggccctctaccttatgcaggatgcagac60
tttcaattatttggtgcctcctgtcttaaggaactagagattacccaaaaagatgaaaag120
attaacctagatgcccttgaagatttgggcataggaagactaaaagacgagcatccacag180
agtctaagtggtggtgaaaagcaaaggctacaaatggccattgccagggttagcagcaac240
aaggtaataatcttggacgaacctaccagcgggcttgataaggactcaatggagcgggta300
tgctcaatgataggagatttgaaaaaagataggaccataatagtaatttcacatgattat360
gaatttatccggagggtgagtgattctattgcctatctttcagatggggttatcaaggaa420
aaattctatctaacggacagcaatatagaaaagctgaatagtatttataaagaaatggag480
gtttattatgaatag495
<210>2
<211>1200
<212>dna
<213>人工序列
<220>
<223>特异序列
<400>2
atgtttaagaaaacatcatctgtggccttatctttggcaattttattgtcaacaggttca60
gtagcccttgccaatcaaaatcttcaaggattagatttaaatgtggtcgaaaaaattaac120
acgaaaaatattaagggagcttctaatcaagaggtaacggatttacaaaatgaattaaaa180
aaattaattgaccaggcagaagacacagatacattggtattaaaaacagatgaaagtatc240
aacaagctaaaaatttttgtaaaaactgcaaagacatatttaaatagtgaagatactaat300
aaacttaagtctactattgaggaattagatcaaaatttaattcaaattgggggtgatgaa360
aacaatatcaaggatatttattcattgaaaacaaagcctcaattggaaaagggagtctat420
aatgttcccatagttttgaggaattttgagttaacagaacaaaattcaatgggaaataat480
gctatcatgagtaaggccaaactaattgtagatgaaagtggaaaaataagactaaggttt540
aacctaagaggtctgtactttatgggagcatatggtcatttgacgaatttgtggtattat600
aataatactaaggaagaaaatgttgataaaagtcacttggagtacctgggggaattgacc660
agagtaaaggtattaaaaactaagaatgaactagatgcagagggtaatttaagagatttt720
ctaaaaagtgttgaaataaatatagacaagcatgtagataatcccaaaagatatcttaaa780
gtaaaggttgatgcgatggatatgcttaatggtataaatccatatgaaaatttggatgaa840
aattctacgtcacctaatgccattcttacccttgactataggtttattgaaaaagatcct900
acaaacaatgaaaatccgtatagccaaggtgacgagttagaacaagctaaaaaagaagcc960
aaaataagtattgataatttgtcaaaattagactcaaatagtaaggctaaatacaaggca1020
gatatagacaatgccgagtcaaaagaaaaaatcaacgaggtactaaaagaagcaaagtat1080
gccaatgataaaattgaaaaggatattgataaatcgaaaaaagatgcaaaatctgaaata1140
gacagattaacagacttatcaagtgatgaaaaaactaattttaaaaataagattgattca1200
<210>3
<211>1413
<212>dna
<213>人工序列
<220>
<223>特异序列
<400>3
ctaatggataaaagcatacatatggattatagaacctacataatttcctttgttatttcg60
attataccttttgcaattgttttcttatatgaattttatgtgataaataatttaagtgta120
gtcgatttaaccaactataatcatagaagaaaagaaatcaatagaatcttactaagacca180
actacaaaaatacataagaaactattattaataaatatacaaaaaaatgctatatatttg240
atggtaccggttattatgttgagcttatcactgtctagatatgtaattaatattaataga300
gattattttgataagaatgttgattcttcagtaaatttttttatgggaagagaatataac360
cttacaaactcaagtaatatttatagaccaaaatccaacttgaatgatagaaatataggt420
aggtttagagatgataatagaattaataatattgttactaaatctagtaaaggggcctat480
tttatcagtaatggaaagatgttcaatgaatactttttaaacaatagagatttaaatggt540
agagaaatagaaacagttatcacaggagtagatgatgacttcctaataagaaatggattt600
ataacaaataataaatttaaatcgactttagaatacccggtagcctatcttagatcgaag660
gtctataataaattagaaacgaagatgataccaactatgaaaaatataaaagttggagat720
gtaataaagatcaaagttgcccaatactccaatggaagagaagcctacaaaacgaaaaaa780
attattgttggtggatttattgatgattcaaaatggtcaagaaaagtagtttcacagttg840
ctacttcctgaaataatcactgatacagataattttaagaaattagtaggatatggaggt900
gctgatgaaataaatttcaattataatggcaatagtaaaaatctaagagcatatatggat960
aaagtgttaggaaagaataactacaaaattatatccgaaaccgaatttaagcatattgat1020
caatatgagcatagattattaatgaaaaaaattattgtaaatattatttatatttcagtg1080
ctgtcaataataacaatatatacaagcttaaatatgatttttgaacaaagaaaaagagaa1140
aatactataatgaggcatattggtctatcgaaaaagaatatgatacttatgtatatcaag1200
gagagtgtattctgggcgatattttcaggggcaataatgtttggaatagtatcgtataaa1260
tcaattcatgactacttagttaatataaaatatcttggtggcggatataaaatgatattt1320
agttttgagacgatatcattagcaacattatatgtaagtgtattgtttgttttggttttt1380
attgtggttttaaaaaagaggattgaagagtag1413
<210>4
<211>314
<212>dna
<213>人工序列
<220>
<223>特异序列
<400>4
aaatcactagaaaaatcactaccagtagtggcagaaagattaagtggtaaaacaagatac60
gaaacagctatagaaatagcatccaaaggattaaaaacttcaaacaaagcatttgttgca120
aatggagaacattggatggacgcactagtaataggaccagtaggcggaatgttaaatatg180
ccaatcctattaacaggagcaaacagcgcaccaaaatcactaagagaccatatcgctaaa240
tcaaaaatcgataagataactgcaattggtggtacaagtatggtaagtgataaagttatt300
aatgaactaagtaa314
<210>5
<211>193
<212>dna
<213>人工序列
<220>
<223>特异序列
<400>5
ctaaattcgataaagatggcgtagaaagaatttctggaaaagatagatatgaaacatcag60
cacaagttgcaagaagattagcaaaaataactggaaaaactgaccatgcagtgatagcat120
caggtgaagttttcgcagatgcattagcagttggtccatacgcagcaagagaaggctatc180
caatactattagt193
<210>6
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>6
agattaacctagatgccctt20
<210>7
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>7
aggcaatagaatcactcacc20
<210>8
<211>25
<212>dna
<213>人工序列
<220>
<223>引物
<400>8
caacaaggtaataatcttggacgaa25
<210>9
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>9
tttctatattgctgtccgtt20
<210>10
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>10
ggccctctaccttatgcag19
<210>11
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>11
tcttcaagggcatctaggtt20
<210>12
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>12
ggccctctaccttatgcag19
<210>13
<211>18
<212>dna
<213>人工序列
<220>
<223>引物
<400>13
agtcttcctatgcccaaa18
<210>14
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>14
aatgaaaatccgtatagcc19
<210>15
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>15
cttcttttagtacctcgttg20
<210>16
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>16
tacaaacaatgaaaatccgta21
<210>17
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>17
tatctgccttgtatttagcc20
<210>18
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>18
atagtaaggctaaatacaagg21
<210>19
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>19
tttcaattttatcattggcat21
<210>20
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>20
tataatgaggcatattggtct21
<210>21
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>21
atcattttatatccgccac19
<210>22
<211>22
<212>dna
<213>人工序列
<220>
<223>引物
<400>22
aataaatttcaattataatggc22
<210>23
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>23
ttcattaataatctatgctca21
<210>24
<211>22
<212>dna
<213>人工序列
<220>
<223>引物
<400>24
tgtaataaagatcaaagttgcc22
<210>25
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>25
gaatcatcaataaatccacc20
<210>26
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>26
tgtagtcgatttaaccaac19
<210>27
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>27
aagctcaacataataaccg19
<210>28
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>28
gacgcactagtaataggacca21
<210>29
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>29
tacttgtaccaccaattgcag21
<210>30
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>30
gcggaatgttaaatatgccaa21
<210>31
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>31
tacttgtaccaccaattgcag21
<210>32
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>32
aaattcgataaagatggcgta21
<210>33
<211>19
<212>dna
<213>人工序列
<220>
<223>引物
<400>33
catctgcgaaaacttcacc19
<210>34
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<400>34
aaattcgataaagatggcgta21
<210>35
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>35
tcttgctgcgtatggaccaa20
- 还没有人留言评论。精彩留言会获得点赞!