只产一种支链氨基酸的重组菌及其应用的制作方法
本发明涉及只产一种支链氨基酸的重组菌及其应用,属于基因工程和酶工程领域。
背景技术:
支链氨基酸包括l-亮氨酸、l-异亮氨酸和l-缬氨酸,属于必需氨基酸,高等生物体不能自身合成,需要从外界摄取。支链氨基酸在生理功能和代谢中具有重要作用,可用于膳食产品,药品和化妆品和饲料行业。另外,支链氨基酸还具有非常好的抗分解作用,有助于预防蛋白分解和肌肉丢失。由于微生物发酵法具有生产成低、反应条件温和,污染小等优点,而成为了当今工业生产支链氨基酸的主要方法。
支链氨基酸的合成途径是一个平行途径并且相互关联,在生物合成途径中,存在着多种共用酶。l-缬氨酸和l-异亮氨酸是从磷酸烯醇式丙酮酸开始分支,由乙酰羟酸合成酶(ahas)、乙酰羟酸异构体还原酶(ahair)、二羟酸脱水酶(dhad)、支链氨基酸转氨酶(bcat)四种共用酶催化反应;由l-缬氨酸合成途径中的α-酮基异戊酸分支一条途径,在α-异丙基苹果酸合成酶(ipms)、α-异丙基苹果酸异构酶(ipmi)、α-异丙基苹果酸脱氢酶(ipmd)和支链氨基酸转氨酶(bcat)酶的催化下合成l-亮氨酸。支链氨基酸转氨酶是三种氨基酸共用的最后一个酶,是催化合成氨基酸的最后一步。
由ilve基因编码的支链氨基酸转氨酶(bcat)是5’-磷酸吡哆醛(plp)依赖型酶,属于iv折叠型,涉及到三种氨基酸和酮酸之间的转氨转移。由于此酶是三种氨基酸合成中的共用酶,若过表达ilve基因,则三种支链氨基酸会同时增加,无法得到唯一的一种氨基酸。
技术实现要素:
基于上述问题,本发明将谷氨酸棒杆菌cornebacteriumglutamiucm支链氨基酸合成途径中的参与最后一步反应的共用酶bcat基因敲除,获得一株支链氨基酸缺陷型的基因工程菌株c.glutamiucm△ilve,首次将来源于大肠杆菌(e.coli)的芳香族氨基酸转氨酶(tyrb)和来自谷氨酸棒杆菌(c.glutamicum)的天冬氨酸转氨酶(aspb),引入到该菌株中,重组菌株经发酵只产生l-亮氨酸,解决了c.glutamiucm发酵过程中同时生产三种支链氨基酸的缺点,并且得到了只产一种支链氨基酸的菌株。
本发明的第一个目的是提供只产一种支链氨基酸的谷氨酸棒杆菌重组菌,所述重组菌是在谷氨酸棒杆菌的基础上敲除了支链氨基酸转氨酶基因,并且过表达了芳香族氨基酸转氨酶或者天冬氨酸转氨酶。
在本发明的一种实施方式中,所述芳香族氨基酸转氨酶来源于大肠杆菌。
在本发明的一种实施方式中,所述芳香族氨基酸转氨酶基因是来源于大肠杆菌w3110的tyrb。
在本发明的一种实施方式中,芳香族氨基酸转氨酶氨基酸序列如seqidno:1所示,核苷酸序列如seqidno:2所示。
在本发明的一种实施方式中,所述天冬氨酸转氨酶来源于谷氨酸棒杆菌。
在本发明的一种实施方式中,所述天冬氨酸转氨酶基因是来源于谷氨酸棒杆菌atcc13032中的aspb基因。
在本发明的一种实施方式中,天冬氨酸转氨酶氨基酸序列如seqidno:3所示,核苷酸序列如seqidno:4所示。
在本发明的一种实施方式中,所述重组菌是以谷氨酸棒杆菌c.glutamicumfa-1为宿主构建得到的。所述c.glutamicumfa-1为异亮氨酸缺陷型。
在本发明的一种实施方式中,所述谷氨酸棒杆菌宿主,还可以是c.glutamiucmatcc13032,在2009年4月公开的《l-亮氨酸高产菌tgl8207的定向选育及其发酵过程研究》文献中记载并公开了的谷氨酸棒杆菌tgl8207等。
在本发明的一种实施方式中,所述过表达是以pec-xk99e作为表达载体。
在本发明的一种实施方式中,所述支链氨基酸转氨酶基因序列与c.glutamiucmatcc13032来源的ilve基因序列一致(氨基酸序列如seqidno:5所示,核苷酸序列如seqidno:6所示)。
本发明的第二个目的是提供一种所述重组菌的构建方法。
在本发明的一种实施方式中,构建方法中,支链氨基酸转氨酶基因的敲除具体是:
(1)自杀性质粒pk18mobsacb-△ilve的构建
根据genebank中c.glutamiucmatcc13032基因组中的ilve基因序列设计ilve基因的上下游以及中间引物(表1)。以c.glutamiucmfa-1或c.glutamiucmatcc13032基因组为模板,分别以ilve-l-f/ilve-l-r和ilve-r-f和ilve-r-r为引物进行pcr反应,获得分别在5’端和3’端有相同限制性内切酶酶切位点的pcr产物。将以上纯化后的pcr产物和线性化的整合型质粒pk18mobsacb通过酶连构建重组质粒pk18mobsacb-△ilve。
(2)重组菌株c.glutamiucmfa-1△ilve或c.glutamiucmatcc13032△ilve的构建
将验证正确的重组质粒pk18mobsacb-△ilve电击转化c.glutamicumfa-1或c.glutamiucmatcc13032,经含有25mg/l卡那霉素的lbhis固体培养基筛选,获得第一次同源重组转化子;再将目标转化子在含有100g/l蔗糖的lbg固体培养集中进行胁迫二次重选筛选,最终在lbg平板上进行分离并挑取多个转化子以ilve-f和ilve-r为引物进行pcr验证,将经过pcr验证正确的转化子接入lbg液体培养基,提取基因组pcr验证及测序鉴定,获得基因工程菌c.glutamicumfa-1△ilve或c.glutamiucmatcc13032△ilve。
本发明的第三个目的是提供一种发酵生产l-亮氨酸的方法,所述方法利用本发明的重组菌。
在本发明的一种实施方式中,所述方法是将所述重组菌单菌落接种至液体种子培养基,培养得到的种子液按照3%-12%的接种量转接至发酵培养基进行发酵培养。
在本发明的一种实施方式中,所述种子液是在28-32℃、80-200r/min培养8-16h得到的。
在本发明的一种实施方式中,所述种子液是在30℃,100r/min培养12h得到的。
在本发明的一种实施方式中,所述发酵培养,是在28-32℃、80-200r/min培养56-96h。
在本发明的一种实施方式中,所述种子液是按照5%的接种量转接至发酵培养基,30℃,100r/min培养72h。
在本发明的一种实施方式中,所述种子培养基(g/l):葡萄糖30,玉米浆,酵母膏5,(nh4)2so45,kh2po42,mgso4·7h2o0.5,mnso4·4h2o0.05,柠檬酸钠5,尿素2,l-蛋氨酸0.5,硫胺素300μg,生物素200μg,caco320。
在本发明的一种实施方式中,所述发酵培养基(g/l):葡萄糖130,玉米浆25,(nh4)2so415,ch3coonh415,kh2po4·3h2o1.3,mgso4·7h2o0.4,mnso4·h2o0.01,l-蛋氨酸0.7,l-异亮氨酸0.06,l-谷氨酸0.5,l-缬氨酸0.6,硫胺素160μg,生物素50μg,柠檬酸钠2,尿素2,caco330。
本发明的第四个目的是提供所述重组菌在饲料工业、医药工业或食品工业中的应用。
本发明的有益效果是:本发明通过在敲除c.glutamiucmfa-1中编码bcat基因ilve的菌株中,过表达来自e.coliw3110的tyrb和c.glutamiucmatcc13032的aspb基因,获得对l-亮氨酸有专一性的重组菌株c.glutamiucmfa-1△ilve/pec-xk99e-tyrb和c.glutamiucmfa-1△ilve/pec-xk99e-aspb,解决了c.glutamiucmfa-1发酵同时生产l-亮氨酸和l-缬氨酸的缺点,从而获得了只产一种支链氨基酸的菌株。
附图说明
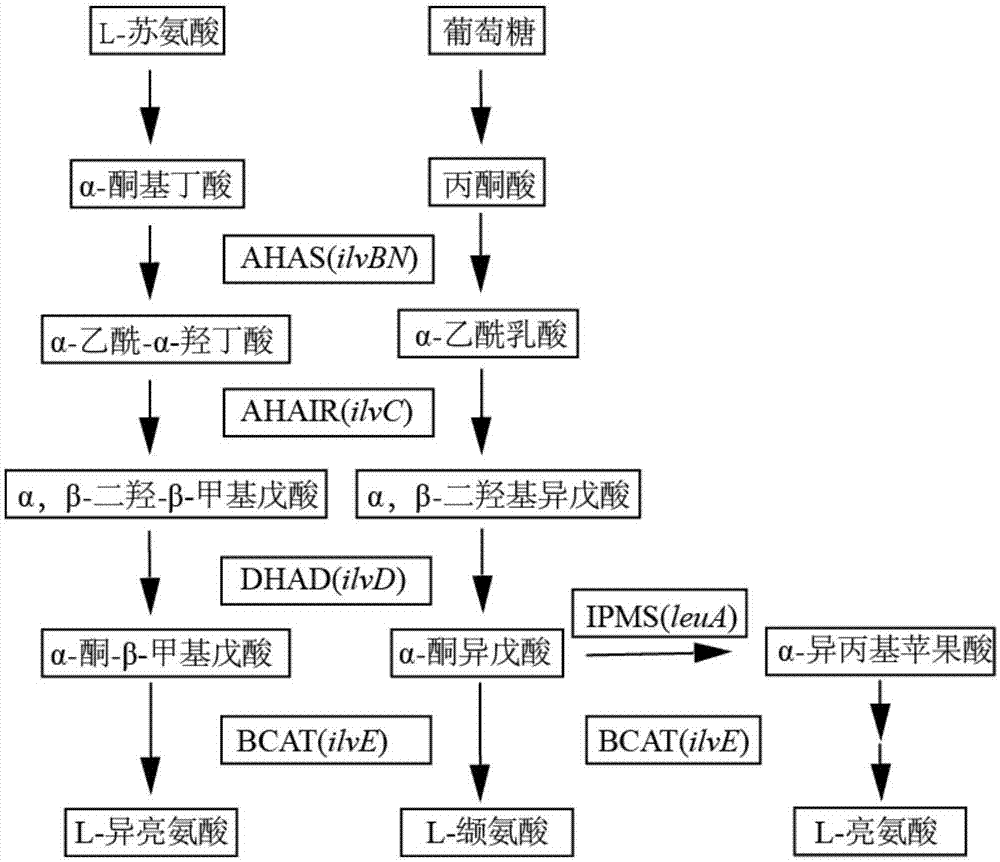
图1:支链氨基酸的合成;
缩写说明:ahas,乙酰羟酸合酶;ahair,乙酰羟酸异构还原酶;dhad,二羟酸脱水酶;bcat,支链氨基酸转氨酶;ipms,异丙基苹果酸合成酶。
图2:tyrb和aspb在e.colibl21中的表达;
泳道说明:m泳道为蛋白质分子量标准marker;1号泳道为e.colibl21/pec-xk99e上清液加iptg诱导;2号道为e.colibl21/pec-xk99e-aspb上清液加iptg诱导;3号道为e.colibl21/pec-xk99e上清液加iptg诱导;4号道为e.colibl21/pec-xk99e-tyrb上清液加iptg诱导;5号道为e.colibl21/pec-xk99e上清液诱导。
图3:不同菌株转氨酶比酶活力测定;
图4:不同菌株发酵生产支链氨基酸过程曲线;
编号说明:a-菌体生长;b-葡萄糖变化曲线;c-支链氨基酸生产曲线;
图5:不同菌株发酵液副产物测定结果;
图6:大肠杆菌中的天冬氨酸转氨酶aspc和枯草芽孢杆菌的天冬氨酸转氨酶aspb对支链氨基酸的影响。
具体实施方式
实施例1:c.glutamiucmfa-1中bcat编码基因ilve基因的敲除
以c.glutamiucmatcc13032基因组为模板,先以ilve-l-f/ilve-l-r(表1)为引物进行pcr扩增反应,获得ilve-l片段。将pcr产物ilve-l胶回收后与pk18mobsacb同时用smai和xbai双酶切,酶切之后纯化的产物ilve-l和质粒pk18mobsacb通过酶连,获得重组质粒pk18mobsacb-ilve-l;再以c.glutamiucmatcc13032基因组为模板,以ilve-r-f/ilve-r-r为引物扩增ilve-r片段,将得到的ilve-r片段和质粒pk18mobsacb-ilve-l同时用xbai和sali双酶切,之后将纯化后的ilve-r片段和质粒pk18mobsacb-ilve-l进行酶连,获得重组质粒pk18mobsacb-△ilve。
将验证正确的质粒pk18mobsacb-△ilve电击转化c.glutamiucmfa-1或c.glutamiucmatcc13032,经lbhis+km固体培养基筛选,获得第一次同源重组转化子。再将转化子在含有10%蔗糖的培养基中进行胁迫发生第二次同源重组,最终在lbg平板上进行划线分离并挑取多个转化子,以ilve基因的上下游引物对其进行pcr鉴定,鉴定正确的转化子提取基因组进一步pcr鉴定,并将pcr产物进行测序鉴定。鉴定正确的转化子,命名为c.glutamiucmfa-1△ilve或c.glutamiucmatcc13032△ilve。
表1pcr扩增所需引物序列(下划线为酶切位点)
实施例2:重组菌株c.glutamicumfa-1△ilve/pec-xk99e-tyrb和c.glutamicumfa-1△ilve/pec-xk99e-aspb的构建
(1)tyrb和aspb的获得
片段tyrb和aspb的获得,是分别以e.coliw3110和c.glutamicumatcc13032基因组为模板,以tyrb-f和tyrb-r、aspb-f和aspb-r为引物,通过pcr反应扩增1251bp的tyrb片段和1281bp的aspb片段,在tyrb片段和aspb片段5’和3’端分别引入ecori和bamhi、kpni和xbai限制性酶切位点。
(2)重组质粒pec-xk99e-tyrb和pec-xk99e-aspb的构建
将经过ecori和bamhi双酶切纯化后的tyrb片段和经过kpni和xbai双酶切纯化后的aspb片段分别与质粒pec-xk99e连接,反应体系如下:质粒pec-xk99e:tyrb/aspb基因=1:3/1:4,t4dna连接酶1μl,t4dnaligationbuffer1μl,加ddh2o至10μl,混合反应液,将其置于16℃金属浴中连接10h以上,转化e.colijm109感受态细胞中,并涂布于50μg·ml-1卡那霉素抗性平板中,从相应的抗性平板中挑取转化子进行pcr验证,将pcr验证正确的转化子分别提取质粒单、双酶切验证并测序鉴定,获得重组质粒pec-xk99e-tyrb和pec-xk99e-aspb。
(3)重组菌株c.glutamicumfa-1△ilve/pec-xk99e-tyrb和c.glutamicumfa-1△ilve/pec-xk99e-aspb的构建
将验证正确的重组质粒pec-xk99e-tyrb和pec-xk99e-aspb电击转化c.glutamicumfa-1△ilve,经含有25μg/ml卡那霉素的lbhis固体培养基筛选,再分别将转化子在含有50μg/ml卡那霉素的lbg固体培养基再次筛选,最终分别挑取转化子进行pcr验证,将验证正确的转化子分别接入lbg液体培养基,提取质粒验证,获得重组菌株c.glutamicumfa-1△ilve/pec-xk99e-tyrb和c.glutamicumfa-1△ilve/pec-xk99e-aspb。
基于类似的方法,构建得到了重组菌株c.glutamicumatcc13032△ilve/pec-xk99e-tyrb和c.glutamicumatcc13032△ilve/pec-xk99e-aspb。
实施例3:tyrb或aspb基因在e.colibl21中的表达
分别将菌株e.colibl21/pec-xk99e-tyrb、e.colibl21/pec-xk99e-aspb和e.colibl21/pec-xk99e接种至液体培养基lb,iptg诱导后收集菌体经超声波破碎后,上清液跑sds-page,检测到分子量约44kda和47kda的特异性条带(图2),与报道的目标蛋白大小一致,说明tyrb和aspb基因可以在大肠杆菌中正确表达。
实施例4:不同菌株转氨酶酶活力的测定
转氨酶酶活力的测定:将c.glutamiucmfa-1△ilve/pec-xk99e-tyrb和c.glutamicumfa-1△ilve/pec-xk99e-aspb分别接种于含有50μg/ml卡那霉素的lbg液体培养基中,培养至od600≈0.6时,添加终浓度为1mmiptg于30℃振荡诱导培养8h。c.glutamiucmfa-1和c.glutamiucmfa-1△ilve接种于lbg液体培养基中,培养结束后,于4℃,12,000r/min冷冻离心20min收集菌体,随后将菌体悬浮于200mmol/ltris-hcl(ph8.0)缓冲液中再次离心收集菌体,添加终浓度为20mg/ml的溶菌酶,37℃处理1h,菌悬液经超声破碎法制备粗酶液。加入30μl50g/l高氯酸+38%(v/v)乙醇混合液来终止反应。反应体系:100mmol/ltris-hcl(ph8.0),0.25mmol/lplp,5mmol/lα-酮异己酸或α-酮异戊酸,10mmol/l谷氨酸钠;反应温度:30℃,反应时间20min。加入30μl50g/l高氯酸终止反应。一个酶活力单位(u)定义为在测定条件下每分钟催化生成1μmoll-亮氨酸所需的酶量。通过加入20mmol/lk2co3离心样品以除去蛋白质和盐(12,000r/min,10min,4℃)。随后,通过高压液相色谱(hplc)测定l-亮氨酸和l-缬氨酸。结果如图3所示,从图中可以看出菌株c.glutamiucmfa-1△ilve/pec-xk99e-tyrb和c.glutamicumfa-1△ilve/pec-xk99e-aspb具有亮氨酸酶活没有缬氨酸酶活,说明了基因tyrb和aspb在c.glutamicumfa-1△ilve中成功表达并具有亮氨酸活性。
实施例5:发酵生产支链氨基酸
菌株c.glutamiucmfa-1、c.glutamiucmfa-1△ilve、c.glutamiucmfa-1△ilve/pec-xk99e-tyrb和c.glutamiucmfa-1△ilve/pec-xk99e-aspb发酵生产支链氨基酸。
培养基:①种子培养基(g/l):葡萄糖30,玉米浆25,(nh4)2so45,kh2po4·3h2o1.3,mgso4·7h2o0.4,mnso4·h2o0.01,l-蛋氨酸0.4,vb1300μg,vh200μg,柠檬酸钠10,尿素2,酵母膏10,caco320,ph7.0,121℃灭菌20min;②发酵培养基(g/l):葡萄糖135,玉米浆25,(nh4)2so415,ch3coonh4,kh2po4·3h2o1.3,mgso4·7h2o0.4,mnso4·h2o0.01,l-蛋氨酸0.7,l-异亮氨酸0.06,l-谷氨酸0.5,vb1160μg,vh50μg,柠檬酸钠2,尿素2,caco330,ph7.0,115℃灭菌10min。
所述发酵培养基中添加0.6g/ll-缬氨酸和0.6g/ll-亮氨酸培养c.glutamiucmfa-1△ilve、c.glutamiucmfa-1△ilve/pec-xk99e-tyrb和c.glutamiucmfa-1△ilve/pec-xk99e-aspb。
将上述经验证的重组菌c.glutamiucmfa-1△ilve/pec-xk99e-tyrb、c.glutamiucmfa-1△ilve/pec-xk99e-aspb及c.glutamiucmfa-1和c.glutamiucmfa-1△ilve分别进行摇瓶发酵实验,从新鲜活化的lbg液体培养基中以1%接种量于种子培养基中(50ml/500ml),30℃,100r/min往复式摇床培养12-18h;种子培养基以5%接种量,接种于发酵培养基中,30-32℃,100r/min往复式摇床培养72h,分时间区段测定支链氨基酸、葡萄糖及生物量,结果与对照菌株c.glutamiucmfa-1相比,结果如图4所示。从图4中可以看出菌株c.glutamiucmfa-1△ilve的葡萄糖消耗能力下降,由于葡萄糖到氨基酸的通路受到阻碍,发酵液中残留较多的葡萄糖。菌株c.glutamiucmfa-1△ilve/pec-xk99e-tyrb和c.glutamiucmfa-1△ilve/pec-xk99e-aspb发酵结束后,均有亮氨酸产量并且残留的葡萄糖较少,l-亮氨酸产量分别达18.55±0.45和20.81±0.02g/l,高于敲除菌株c.glutamiucmfa-1△ilve的3.63±0.72g/l;另外这两株重组菌株的l-缬氨酸产量为分别为0.92±0.04和0.81±0.02g/l,与敲除菌株相比没有什么变化,说明了基因tyrb和aspb的参与亮氨酸的合成,对缬氨酸没有催化活性。
此外,分别采用高效液相色谱仪和氨基酸分析仪测定重组菌c.glutamiucmfa-1△ilve/pec-xk99e-tyrb和c.glutamiucmfa-1△ilve/pec-xk99e-aspb及菌株c.glutamiucmfa-1和c.glutamiucmfa-1△ilve中副产物积累情况(包括有机酸和氨基酸),结果如图5所示,原始菌株c.glutamiucmfa-1发酵液中杂酸较少,由于ilve基因的敲除导致杂酸含量有不同程度的增加,两株重组菌株c.glutamiucmfa-1△ilve/pec-xk99e-tyrb和c.glutamiucmfa-1△ilve/pec-xk99e-aspb发酵液中也含有较多的丙氨酸,说明在c.glutamiucmfa-1△ilve中过表达转氨酶基因,不同菌株的葡萄糖代谢能力得到强化,亮氨酸产量有所提高,但是会影响副产物的合成。
采用相同的方法,分析了重组菌株c.glutamicumatcc13032△ilve/pec-xk99e-tyrb和c.glutamicumatcc13032△ilve/pec-xk99e-aspb发酵生产支链氨基酸的情况。结果显示:菌株c.glutamiucmatcc13032△ilve/pec-xk99e-tyrb和c.glutamiucmatcc13032△ilve/pec-xk99e-aspb发酵结束后,均有亮氨酸产量并且残留的葡萄糖较少,l-亮氨酸产量分别达12.22±0.23和13.56±0.04g/l,高于敲除菌株c.glutamiucmatcc13032△ilve的2.99±0.48g/l。
对比例1:大肠杆菌中的天冬氨酸转氨酶aspc对支链氨基酸的影响
以e.coliw3110基因组为模板,以aspc-f/aspc-r为引物进行pcr扩增反应,获得aspc片段。将pcr产物aspc胶回收后与pec-xk99e同时用ecori和bamhi双酶切,酶切之后纯化的产物aspc和质粒pec-xk99e通过酶连,获得重组质粒pec-xk99e-aspc。将构建成功的质粒电转入c.glutamiucmfa-1△ilve,验证正确的重组菌株c.glutamiucmfa-1△ilve/pec-xk99e-aspc分别进行摇瓶发酵实验,分时间区段测定支链氨基酸、葡萄糖及生物量,结果发现在c.glutamiucmfa-1△ilve菌株中过表达来自大肠杆菌的天冬氨酸转氨酶,发酵结束后,亮氨酸和缬氨酸的产量相对于c.glutamiucmfa-1△ilve没有变化,说明该酶对亮氨酸和缬氨酸没有催化作用(图6)。
上述所使用的大肠杆菌的天冬氨酸转氨酶序列如seqidno:7所示。
对比例2:枯草芽孢杆菌的天冬氨酸转氨酶aspb对支链氨基酸的影响
以b.subtilis168基因组为模板,以aspb-f/aspb-r为引物进行pcr扩增反应,获得aspb片段。将pcr产物aspb胶回收后与pec-xk99e同时用kpni和xbai双酶切,酶切之后纯化的产物aspb和质粒pec-xk99e通过酶连,获得重组质粒pec-xk99e-aspb。将构建成功的质粒电转入c.glutamiucmfa-1△ilve,验证正确的重组菌株c.glutamiucmfa-1△ilve/pec-xk99e-aspb分别进行摇瓶发酵实验,分时间区段测定支链氨基酸、葡萄糖及生物量,结果与对照菌株c.glutamiucmfa-1△ilve相比,结果如图6所示,在c.glutamiucmfa-1△ilve菌株中过表达来自枯草芽孢杆菌的天冬氨酸转氨酶,亮氨酸和缬氨酸的产量相对于c.glutamiucmfa-1△ilve没有变化,说明该酶对亮氨酸和缬氨酸没有催化活性。
上述所使用的枯草芽孢杆菌的天冬氨酸转氨酶序列如seqidno:8所示。
以上所述实施例仅是为充分说明本发明而所举的较佳的实施例,本发明的保护范围不限于此。本技术领域的技术人员在本发明基础上所作的等同替代或变换,均在本发明的保护范围之内。本发明的保护范围以权利要求书为准。
sequencelisting
<110>江南大学
<120>只产一种支链氨基酸的重组菌及其应用
<160>18
<170>patentinversion3.3
<210>1
<211>397
<212>prt
<213>tyrb
<400>1
valpheglnlysvalaspalatyralaglyaspproileleuthrleu
151015
metgluargphelysgluaspproargserasplysvalasnleuser
202530
ileglyleutyrtyrasngluaspglyileileproglnleuglnala
354045
valalaglualaglualaargleuasnalaglnprohisglyalaser
505560
leutyrleuprometgluglyleuasncystyrarghisalaileala
65707580
proleuleupheglyalaasphisprovalleulysglnglnargval
859095
alathrileglnthrleuglyglyserglyalaleulysvalglyala
100105110
asppheleulysargtyrpheprogluserglyvaltrpvalserasp
115120125
prothrtrpgluasnhisvalalailephealaglyalaglypheglu
130135140
valserthrtyrprotrptyraspglualathrasnglyvalargphe
145150155160
asnaspleuleualathrleulysthrleuproalaargserileval
165170175
leuleuhisprocyscyshisasnprothrglyalaaspleuthrasn
180185190
aspglntrpaspalavalilegluileleulysalaarggluleuile
195200205
propheleuaspilealatyrglnglypheglyalaglymetgluglu
210215220
aspalatyralaileargalailealaseralaglyleuproalaleu
225230235240
valserasnserpheserlysilepheserleutyrglygluargval
245250255
glyglyleuservalmetcysgluaspalaglualaalaglyargval
260265270
leuglyglnleulysalathrvalargargasntyrserserpropro
275280285
asnpheglyalaglnvalvalalaalavalleuasnaspglualaleu
290295300
lysalasertrpleualagluvalgluglumetargthrargileleu
305310315320
alametargglngluleuvallysvalleuserthrglumetproglu
325330335
argasnpheasptyrleuleuasnglnargglymetphesertyrthr
340345350
glyleuseralaalaglnvalaspargleuarggluglupheglyval
355360365
tyrleuilealaserglyargmetcysvalalaglyleuasnthrala
370375380
asnvalglnargvalalalysalaphealaalavalmet
385390395
<210>2
<211>1194
<212>dna
<213>tyrb
<400>2
gtgtttcaaaaagttgacgcctacgctggcgacccgattcttacgcttatggagcgtttt60
aaagaagaccctcgcagcgacaaagtgaatttaagtatcggtctgtactacaacgaagac120
ggaattattccacaactgcaagccgtggcggaggcggaagcgcgcctgaatgcgcagcct180
catggcgcttcgctttatttaccgatggaagggcttaactgctatcgccatgccattgcg240
ccgctgctgtttggtgcggaccatccggtactgaaacaacagcgcgtagcaaccattcaa300
acccttggcggctccggggcattgaaagtgggcgcggatttcctgaaacgctacttcccg360
gaatcaggcgtctgggtcagcgatcctacctgggaaaaccacgtagcaatattcgccggg420
gctggattcgaagtgagtacttacccctggtatgacgaagcgactaacggcgtgcgcttt480
aatgacctgttggcgacgctgaaaacattacctgcccgcagtattgtgttgctgcatcca540
tgttgccacaacccaacgggtgccgatctcactaatgatcagtgggatgcggtgattgaa600
attctcaaagcccgcgagcttattccattcctcgatattgcctatcaaggatttggtgcc660
ggtatggaagaggatgcctacgctattcgcgccattgccagcgctggattacccgctctg720
gtgagcaattcgttctcgaaaattttctccctttacggcgagcgcgtcggcggactttct780
gttatgtgtgaagatgccgaagccgctggccgcgtactggggcaattgaaagcaacagtt840
cgccgcaactactccagcccgccgaattttggtgcgcaggtggtggctgcagtgctgaat900
gacgaggcattgaaagccagctggctggcggaagtagaagagatgcgtactcgcattctg960
gcaatgcgtcaggaattggtgaaggtattaagcacagagatgccagaacgcaatttcgat1020
tatctgcttaatcagcgcggcatgttcagttataccggtttaagtgccgctcaggttgac1080
cgactacgtgaagaatttggtgtctatctcatcgccagcggtcgcatgtgtgtcgccggg1140
ttaaatacggcaaatgtacaacgtgtggcaaaggcgtttgctgcggtgatgtaa1194
<210>3
<211>426
<212>prt
<213>aspb
<400>3
metserservalserleuglnasppheaspalagluargileglyleu
151015
phehisgluaspilelysarglyspheaspgluleulysserlysasn
202530
leulysleuaspleuthrargglylysprosersergluglnleuasp
354045
phealaaspgluleuleualaleuproglylysglyaspphelysala
505560
alaaspglythraspvalargasntyrglyglyleuaspglyileval
65707580
aspileargglniletrpalaaspleuleuglyvalprovalglugln
859095
valleualaglyaspalaserserleuasnilemetpheaspvalile
100105110
sertrpsertyrilepheglyasnasnaspservalglnprotrpser
115120125
lysglugluthrvallystrpilecysprovalproglytyrasparg
130135140
hispheserilethrgluargpheglypheglumetileservalpro
145150155160
metasngluaspglyproaspmetaspalavalglugluleuvallys
165170175
asnproglnvallysglymettrpvalvalprovalpheserasnpro
180185190
thrglyphethrvalthrgluaspvalalalysargleuseralamet
195200205
gluthralaalaproasppheargvalvaltrpaspasnalatyrala
210215220
valhisthrleuthraspglupheprogluvalileaspilevalgly
225230235240
leuglyglualaalaglyasnproasnargphetrpalaphethrser
245250255
thrserlysilethrleualaglyalaglyvalserphepheleuthr
260265270
seralagluasnarglystrptyrthrglyhisalaglyilearggly
275280285
ileglyproasnlysvalasnglnleualahisalaargtyrphegly
290295300
aspalagluglyvalargalavalmetarglyshisalaalaserleu
305310315320
alaprolyspheasnlysvalleugluileleuaspserargleuala
325330335
glutyrglyvalalaglntrpthrvalproalaglyglytyrpheile
340345350
serleuaspvalvalproglythralaserargvalalagluleuala
355360365
lysglualaglyilealaleuthrglyalaglysersertyrproleu
370375380
argglnaspprogluasnlysasnleuargleualaproserleupro
385390395400
provalglugluleugluvalalametaspglyvalalathrcysval
405410415
leuleualaalaalagluhistyralaasn
420425
<210>4
<211>1281
<212>dna
<213>aspb
<400>4
atgagttcagtttcgctgcaggattttgatgcagagcgaattggtttgttccacgaggac60
attaagcgcaagtttgatgagctcaagtcaaaaaatctgaagctggatcttactcgcggt120
aagccttcgtcggagcagttggatttcgctgatgagttgttggcgttgcctggtaagggt180
gatttcaaggctgcggatggtactgatgtccgtaactatggcgggctggatggcatcgtt240
gatattcgccagatttgggcggatttgctgggtgttcctgtggagcaggtcttggcgggg300
gatgcttcgagcttgaacatcatgtttgatgtgatcagctggtcgtacattttcggtaac360
aatgattcggttcagccttggtcgaaggaagaaaccgttaagtggatttgccctgttccg420
ggctatgatcgccatttctccatcacggagcgtttcggctttgagatgatttctgtgcca480
atgaatgaagacggccctgatatggatgctgttgaggaattggtgaagaatccgcaggtt540
aagggcatgtgggttgttccggtgttttctaacccgactggtttcacggtgacagaagac600
gtcgcaaagcgtctaagcgcaatggaaaccgcagctccggacttccgcgttgtgtgggat660
aatgcctacgccgttcatacgctgaccgatgaattccctgaggttatcgatatcgtcggg720
cttggtgaggccgctggcaacccgaaccgtttctgggcgttcacttctacttcgaagatc780
actctcgcgggtgcgggcgtgtcgttcttcctcacctctgcggagaaccgcaagtggtac840
accggccatgcgggtatccgtggcattggccctaacaaggtcaatcagttggctcatgcg900
cgttactttggcgatgctgagggagtgcgcgcggtgatgcgtaagcatgctgcgtcgttg960
gctccgaagttcaacaaggttctggagattctggattctcgccttgctgagtacggtgtc1020
gcgcagtggactgtccctgcgggcggttacttcatttcccttgatgtggttcctggtacg1080
gcgtctcgcgtggctgagttggctaaggaagccggcatcgcgttgacgggtgcgggttct1140
tcttacccgctgcgtcaggatccggagaacaaaaatctccgtttggcaccgtcgctgcct1200
ccagttgaggaacttgaggttgccatggatggcgtggctacctgtgtgctgttggcagca1260
gcggagcattacgctaactaa1281
<210>5
<211>367
<212>prt
<213>ilve
<400>5
metthrserleugluphethrvalthrargthrgluasnprothrser
151015
proaspargleulysgluileleualaalaprolyspheglylysphe
202530
phethrasphismetvalthrileasptrpasnglusergluglytrp
354045
hisasnalaglnleuvalprotyralaproileprometaspproala
505560
thrthrvalphehistyrglyglnalailephegluglyilelysala
65707580
tyrarghisseraspgluthrilelysthrpheargproaspgluasn
859095
alagluargmetglnargseralaalaargmetalametproglnleu
100105110
prothrgluasppheilelysalaleugluleuleuvalaspalaasp
115120125
glnasptrpvalproglutyrglyglyglualaserleutyrleuarg
130135140
prophemetileserthrgluileglyleuglyvalserproalaasp
145150155160
alatyrlyspheleuvalilealaserprovalglyalatyrphethr
165170175
glyglyilelysprovalservaltrpleusergluasptyrvalarg
180185190
alaalaproglyglythrglyaspalalysphealaglyasntyrala
195200205
alaserleuleualaglnserglnalaalaglulysglycysaspgln
210215220
valvaltrpleuaspalailegluhislystyrilegluglumetgly
225230235240
glymetasnleuglypheiletyrargasnglyaspglnvallysleu
245250255
valthrprogluleuserglyserleuleuproglyilethrarglys
260265270
serleuleuglnvalalaargaspleuglytyrgluvalglugluarg
275280285
lysilethrthrthrglutrpglugluaspalalysserglyalamet
290295300
thrglualaphealacysglythralaalavalilethrprovalgly
305310315320
thrvallysseralahisglythrphegluvalasnasnasngluval
325330335
glygluilethrmetlysleuarggluthrleuthrglyileglngln
340345350
glyasnvalgluaspglnasnglytrpleutyrproleuvalgly
355360365
<210>6
<211>1104
<212>dna
<213>ilve
<400>6
atgacgtcattagagttcacagtaacccgtaccgaaaatccgacgtcacccgatcgtctg60
aaggaaattcttgccgcaccgaagttcggtaagttcttcaccgaccacatggtgaccatt120
gactggaacgagtcggaaggctggcacaacgcccaattagtgccatacgcgccgattcct180
atggatcctgccaccaccgtattccactacggacaggcaatttttgagggaattaaggcc240
taccgccattcggacgaaaccatcaagactttccgtcctgatgaaaacgccgagcgtatg300
cagcgttcagcagctcgaatggcaatgccacagttgccaaccgaggactttattaaagca360
cttgaactgctggtagacgcggatcaggattgggttcctgagtacggcggagaagcttcc420
ctctacctgcgcccattcatgatctccaccgaaattggcttgggtgtcagcccagctgat480
gcctacaagttcctggtcatcgcatccccagtcggcgcttacttcaccggtggaatcaag540
cctgtttccgtctggctgagcgaagattacgtccgcgctgcacccggcggaactggtgac600
gccaaatttgctggcaactacgcggcttctttgcttgcccagtcccaggctgcggaaaag660
ggctgtgaccaggtcgtatggttggatgccatcgagcacaagtacatcgaagaaatgggt720
ggcatgaaccttgggttcatctaccgcaacggcgaccaagtcaagctagtcacccctgaa780
ctttccggctcactacttccaggcatcacccgcaagtcacttctacaagtagcacgcgac840
ttgggatacgaagtagaagagcgaaagatcaccaccaccgagtgggaagaagacgcaaag900
tctggcgccatgaccgaggcatttgcttgcggtactgcagctgttatcacccctgttggc960
accgtgaaatcagctcacggcaccttcgaagtgaacaacaatgaagtcggagaaatcacg1020
atgaagcttcgtgaaaccctcaccggaattcagcaaggaaacgttgaagaccaaaacgga1080
tggctttacccactggttggctaa1104
<210>7
<211>1191
<212>dna
<213>aspc
<400>7
atgtttgagaacattaccgccgctcctgccgacccgattctgggcctggccgatctgttt60
cgtgccgatgaacgtcccggcaaaattaacctcgggattggtgtctataaagatgagacg120
ggcaaaaccccggtactgaccagcgtgaaaaaggctgaacagtatctgctcgaaaatgaa180
accaccaaaaattacctcggcattgacggcatccctgaatttggtcgctgcactcaggaa240
ctgctgtttggtaaaggtagcgccctgatcaatgacaaacgtgctcgcacggcacagact300
ccggggggcactggcgcactacgcgtggctgccgatttcctggcaaaaaataccagcgtt360
aagcgtgtgtgggtgagcaacccaagctggccgaaccataagagcgtctttaactctgca420
ggtctggaagttcgtgaatacgcttattatgatgcggaaaatcacactcttgacttcgat480
gcactgattaacagcctgaatgaagctcaggctggcgacgtagtgctgttccatggctgc540
tgccataacccaaccggtatcgaccctacgctggaacaatggcaaacactggcacaactc600
tccgttgagaaaggctggttaccgctgtttgacttcgcttaccagggttttgcccgtggt660
ctggaagaagatgctgaaggactgcgcgctttcgcggctatgcataaagagctgattgtt720
gccagttcctactctaaaaactttggcctgtacaacgagcgtgttggcgcttgtactctg780
gttgctgccgacagtgaaaccgttgatcgcgcattcagccaaatgaaagcggcgattcgc840
gctaactactctaacccaccagcacacggcgcttctgttgttgccaccatcctgagcaac900
gatgcgttacgtgcgatttgggaacaagagctgactgatatgcgccagcgtattcagcgt960
atgcgtcagttgttcgtcaatacgctgcaggaaaaaggcgcaaaccgcgacttcagcttt1020
atcatcaaacagaacggcatgttctccttcagtggcctgacaaaagaacaagtgctgcgt1080
ctgcgcgaagagtttggcgtatatgcggttgcttctggtcgcgtaaatgtggccgggatg1140
acaccagataacatggctccgctgtgcgaagcgattgtggcagtgctgtaa1191
<210>8
<211>1182
<212>dna
<213>aspb
<400>8
ttgaaactggcaaaaagagtatccgcattaacaccatcaaccacactggcaatcacagcg60
aaagcgaaagaactgaaagccgcaggccatgatgtcatcggcttaggagcaggcgagcct120
gacttcaatacaccgcagcatattattgatgccgctgtgcgttcgatgaacgagggccat180
acgaaatacacgccttccggtggtttggctgaactgaaaaacagcattgcagagaaattt240
aaacgcgaccagaatattgaatacaaaccgtctcaaattattgtctgcactggtgccaaa300
catgctctgtatacccttttccaagtgattttggatgaagaagatgaagttattattcca360
acgccttactgggtaagctacccggagcaggtgaaactcgctggcggaaagcctgtttat420
gtagaagggcttgaggaaaatcacttcaaaatttcgcctgagcagctgaaaaatgcaatt480
acggaaaaaacaaaagcaatcgttattaactctccaagcaacccgactggtgtcatgtat540
acggaagaagaactatctgcgctcggtgaagtgtgccttgaacatgatatcttgattgtg600
tcagatgaaatttatgaaaaacttacatacggcggaaagaaacatgtttccattgcacag660
ctgtcagacagactgaaagagcaaacggtcattataaacggcgtatcgaagtcccacagc720
atgacaggctggagaatcggttatgcggcaggctctgaagacattattaaagcgatgacg780
aaccttgcaagccacagcacgtcaaacccgacatcaatcgcacaatacggagcgattgct840
gcttataacgggccttctgagccattggaagaaatgagagaagcatttgaacatagatta900
aatacgatctatgcaaagctcatagaaatcccagggttcagctgcgtgaagccggaaggt960
gctttttacttgttcccaaatgcaaaagaggcagctcaatcttgcggtttcaaagatgta1020
gatgaatttgttaaagcgcttctcgaggaggaaaaagttgcgattgttccaggatccgga1080
tttggctctcctgagaatgtccgtctttcttatgcaacatcgttagatcttcttgaagaa1140
gccattgaaagaatcaagcgttttgtagaaaaacatagctaa1182
<210>9
<211>27
<212>dna
<213>人工合成
<400>9
tcccccgggcaagcctagccattcctc27
<210>10
<211>27
<212>dna
<213>人工合成
<400>10
gctctagacgtctaccagcagttcaag27
<210>11
<211>29
<212>dna
<213>人工合成
<400>11
gctctagatgggatacgaagtagaagagc29
<210>12
<211>27
<212>dna
<213>人工合成
<400>12
acgcgtcgactttccaaccgtcagctg27
<210>13
<211>19
<212>dna
<213>人工合成
<400>13
atgacgtcattagagttca19
<210>14
<211>19
<212>dna
<213>人工合成
<400>14
ggtcttaaaaccggttgat19
<210>15
<211>28
<212>dna
<213>人工合成
<400>15
ggaattctggagaaccatcgcatgtttc28
<210>16
<211>28
<212>dna
<213>人工合成
<400>16
cgggatcctaatttcactgcaggctggg28
<210>17
<211>27
<212>dna
<213>人工合成
<400>17
ggggtaccatgagttcagtttcgctgc27
<210>18
<211>30
<212>dna
<213>人工合成
<400>18
gctctagatctccgctgtattcacttttag30
- 还没有人留言评论。精彩留言会获得点赞!