一种水稻叶绿体避光运动调控基因CRD1及其应用的制作方法
本发明涉及分子生物学技术领域,具体涉及一种水稻叶绿体避光运动调控基因crd1及其应用。
背景技术:
水稻(oryzasativa)作为一种重要的粮食作物,确保其高产量是解决我国以及世界许多其他国家粮食问题的关键,是我国可持续发展的坚强保障。植物普遍具有的向光性一直以来都是人们关注的热点,光作为地球上绝大部分生物能量的来源,对人类社会的农业生产有着无可比拟的作用。
位于植物叶肉细胞中的叶绿体,是植物进行光合作用的器官,对干物质积累有重要影响。植物为了获取合适的光合效率,自身有着一套精密的调控机制,在弱光的情况下,叶绿体垂直于入射光,平铺在细胞内进壁侧,使光合效率最大化;在强光的情况下,叶绿体平行于入射光,排布在细胞侧壁上,以减少强光直射造成的光伤害。叶绿体运动发生在叶肉细胞,持续时间短,是植物的自我保护反应,已经在多个物种中发现。
随着分子生物学和基因组学近年来的开展,越来越多的与植物叶绿体光运动有关的基因被发现,在模式植物拟南芥中,向光素atphot1和atphot2主要参与调控植物下胚轴的向光弯曲、叶绿体运动、气孔开闭以及叶片的伸展,都具有光氧电压结构域1(lov1)和光氧电压结构域2(lov1),以及一个ser/thr蛋白激酶区域。lov2是参与叶绿体正常避光反应的结构域,据文献报道,phot2lov2拟南芥突变体具有正常的趋光反应,但叶绿体运动不同于正常野生型。大量的下游信号因子随之被一一发现,包括web1、pmi2、jac1等等,这些基因一旦突变,植株在叶绿体趋光或避光运动就会出现问题,同时还有向光素互作蛋白nrl,其与rpt2冗余地调控叶绿体的趋光运动。
在整个叶绿体运动信号传导过程中,其存在的机制尚不明确,叶绿体的运动关键在于叶绿体肌动蛋白丝,有研究指出,cp-actin本身的聚合反应产生的拉力使叶绿体运动。chup1是参与其聚合的重要蛋白,在强光的情况下,chup1通过n端结合到叶绿体的外膜,聚合f-actin,thrumin1则作为连接f-actin和固定他们的锚点,从而牵引叶绿体运动,使用微丝抑制剂或者肌动蛋白聚合抑制剂可以阻止叶绿体的运动。
在强光下,叶绿体的避光运动是一种自我保护机制,如果抑制或停止该生理反应,长时间的强光照射会破坏叶绿体结构和活性,造成叶片死亡,极大地影响植株的生长繁殖。尽管拟南芥中已有众多基因被克隆,但对于作为粮食作物的水稻而言,研究甚少,因此发掘水稻叶绿体避光运动突变体,克隆其调控基因对揭示叶绿体避光运动信号传导机理以及水稻高产育种实践具有重要的意义。
技术实现要素:
本发明的目的在于提供一种调控叶绿体避光运动的基因及编码的蛋白,以及利用该基因调控植物叶绿体避光运动的应用,为揭示叶绿体避光运动信号传导机理以及水稻高产育种实践提供理论研究依据。
为实现上述目的,本发明采用如下技术方案:
本发明通过对籼稻品种蜀恢527进行ems诱变,进而分离得到了一个叶绿体避光运动缺陷突变体,该突变体在强光情况下丧失叶绿体避光反应,通过与野生型水稻的正交实验,证明该突变体受隐性单基因控制。运用图位克隆技术,首次克隆了crd1(chloroplastrelocationdefective1)基因,该基因位于水稻第七号染色体长臂末端74.3kb的物理范围内,通过对该区段进行测序,确定候选基因crd1。在此基础上,综合利用了遗传学、分子生物学、生物化学等手段,深入研究了crd1基因的生物学功能及其调控水稻叶绿体避光运动的分子基础。
因此,本发明提供了一种水稻叶绿体避光运动调控基因crd1,其核苷酸序列如seqidno.1所示。
该基因编码一个含774个氨基酸的formin蛋白,其中包含1个信号肽、1个跨膜区域、fh1结构域和fh2结构域,且其与玉米和短柄草的蛋白序列同源性较高。
本发明提供了由所述的水稻叶绿体避光运动调控基因crd1编码的crd1蛋白,其氨基酸序列如seqidno.2所示。本发明还包括与seqidno.2所示序列至少具有60%的同源性,包括在seqidno.2序列中进行氨基酸的替换、插入或缺失氨基酸所获得的功能类似物。
本发明还提供了一种重组质粒,包括原始载体和插入原始载体中的目标片段,所述目标片段的核苷酸序列如seqidno.1所示。该重组质粒可以表达由上述核酸序列编码的多肽或同源类似物。
作为优选,所述原始载体为pmdc83。
本发明还提供了一种包含所述的重组质粒的重组转化子。
本发明还提供了所述的水稻叶绿体避光运动调控基因crd1在调控水稻叶绿体避光运动中的应用。
本发明通过转基因的方法,验证了crd1的功能,通过crispr/cas9技术创造的突变体crd1-2,由于翻译提前终止而完全丧失该蛋白功能,研究发现该突变体叶绿体丧失避光运动;通过构建过表达载体35s::crd1,将此质粒转入crd1突变体中,其避光运动恢复正常。另外,通过yeasttwo-hybridassay、lci和pull-down实验证实crd1与osphot2存在互作,两者在细胞中的定位同时受光照的调控,当黑暗时,两者都位于细胞质膜,在光照30秒时,两者都从质膜向叶绿体迁移,60秒时,几乎全部都位于叶绿体上。在osphot21突变体背景下,crd1的迁移情况正常;但在crd1突变体背景下,osphot2无法从质膜迁移至叶绿体,导致叶绿体避光反应丧失。
所述应用包括:将所述的水稻叶绿体避光运动调控基因crd1连入植物表达载体中,构建得到重组表达载体,再将所述的重组表达载体转化受体植物材料中,培育获得转基因植株。
作为优选,所述植物表达载体为pmdc83。
所述应用包括:利用rnai或crispr/cas9技术降低水稻叶绿体避光运动调控基因crd1编码蛋白的表达或活性。通过该技术适当减少植物在稍高光照的生长环境发生的叶绿体避光运动,从而适当增加植株叶片的光合效率,有利于植物在稍高光照的生长环境中的生长。
本发明具备的有益效果:
(1)本发明通过图位克隆技术获得水稻叶绿体避光运动调控基因crd1,并通过转基因功能互补实验验证了所述基因的功能,crd1是水稻叶绿体正常避光反应所必需的,crd1与osphot2的亚细胞定位共同受光强的调控,crd1与osphot2存在互作,crd1基因的突变导致osphot2的定位异常,从而引起叶绿体避光反应的丧失。利用本发明的蛋白和编码基因可以进一步阐释水稻叶绿体运动的分子机制。
(2)本发明提供了水稻叶绿体避光运动调控基因crd1在调控水稻叶绿体避光运动中的应用,如应用于作物遗传改良,对创制高光效育种具有重要意义。
附图说明
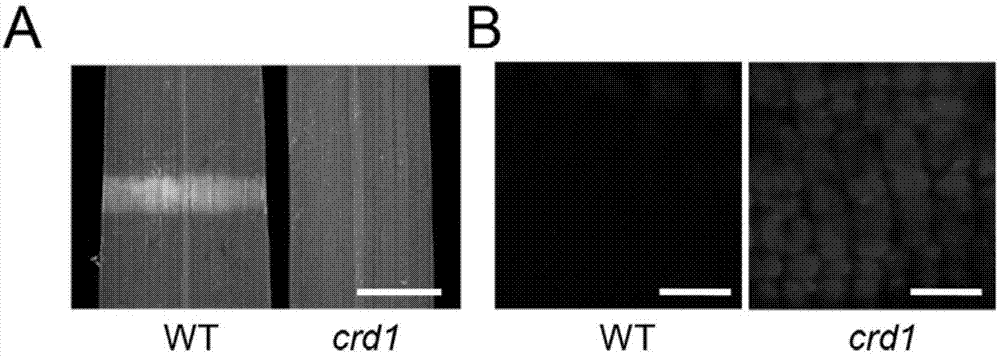
图1为野生型(wt)和突变体(crd1)的表型,其中a为叶片强光照处理后的表型;b为激光共聚焦显微镜下观察强光处理部位的叶绿体分布情况。
图2为crd1基因的图位克隆。
图3为pmdc83-crd1载体图谱。
图4为转基因功能互补验证,其中a为实时定量检测野生型、突变体和过表达转基因苗crd1的表达量;b为westernblot检测野生型、突变体和过表达转基因苗crd1的蛋白水平;c为野生型、突变体和过表达转基因苗的叶绿体避光反应;d为野生型(左)、突变体(中)和过表达转基因苗(右)的表型。
图5为crd1在不同组织部位的定量分析,其中a为qrtpcr检测crd1在不同组织中的表达;b为western检测crd1在不同组织中的表达;c-h为crd1在转基因水稻分别在根尖、胚芽鞘、维管束、叶片、叶肉细胞和韧皮部细胞中的gus表达。
图6为crd1的亚细胞定位分析。a为基因crd1的三种亚细胞定位模式;b为光照处理后crd1的迁移。
图7为酵母双杂交、lci方法和pull-down方法分析crd1的互作蛋白;a,b为酵母双杂交分析crd1和osphot2的相互作用;c,d,e为农杆菌侵染烟草,利用lci检测crd1基因与osphot2基因的相互作用;f为pull-down方法分析crd1和osphot2的相互作用。
图8为osphot2突变体的相关信息,其中a为osphot2基因结构图,突变体osphot2在基因cds的521位插入碱基“a”;b为荧光定量pcr方法检测日本晴与突变体osphot2中osphot2的表达情况;c为突变体osphot2的测序结果;d为突变体osphot2的生长表型;f为日本晴与突变体osphot2中叶绿素a、叶绿素b和总叶绿素的含量。
图9为野生型和突变体背景下,crd1和osphot2的亚细胞定位和叶片强光照处理后的表型,其中a为在野生型、osphot2突变体和crd1突变体中蛋白crd1随光照时间而发生的细胞内定位变化情况;b为在野生型、osphot2突变体和crd1突变体中蛋白osphot2随光照时间而发生的细胞内定位变化情况;c为野生型(wt)、crd1突变体、osphot2突变体和crd1/osphot2双突变体叶片强光照处理后的表型;d为野生型(wt)、crd1突变体、osphot2突变体和crd1/osphot2双突变体在激光共聚焦显微镜下观察强光处理部位的叶绿体分布情况。
具体实施方式
下面结合具体实施例对本发明进行进一步描述,但本发明的保护范围并不仅限于此。
实施例1:
1、水稻材料
水稻(oryzasativa)crd1突变体植株是由籼稻蜀恢527品种(购自中国农科院作物品种资源库)经过ems诱变处理得到。
crd1突变体植株在强光照处理后,叶绿体避光运动消失。与野生型(籼稻蜀恢527)差异显著;利用激光共聚焦显微镜观察叶绿体自发荧光,发现突变体中叶绿体依旧排列致密,不同于野生型,由于叶绿体向细胞侧壁移动,而叶绿体荧光信号极大地减弱,如图1所示。
2、分析和定位群体
f2定位群体是通过纯合的crd1突变体和粳稻品种“日本晴”(oryzasativasubsp.japonicacv.nipponbare)杂交产生f1代,f1代自交得到f2群体。从f2群体中筛选出叶绿体避光反应消失的单株用于图位克隆。
3、通过ssr,sts标记定位crd1基因
基因组dna的提取:tbs消化:取水稻叶片2~3cm(约50~100mg)放入2.0ml的离心管中,幼苗:加入400μltbs溶液,于全自动样品快速研磨仪中震荡,频率为45赫兹,时间为2分钟;老叶:将2.0ml离心管加入液氮约一分钟,用普通镊子取出于全自动样品快速研磨仪中震荡,频率为35赫兹,时间为1分钟,再加入400μltbs溶液,随后在75℃消化20分钟,每5min振荡混匀一次;12000rpm离心10分钟,取上清至新1.5ml离心管;加入等体积预冷的异丙醇,轻轻混匀,稍放置,12000rpm离心10分钟,去上清;加入75%的乙醇溶液200μl洗涤dna沉淀,7500rpm离心5min;弃上清,倒扣离心管晾干,加入200-300μlddh2o,震荡混匀溶解,于-20℃保存备用。
crd1基因的初定位:随机选取20株的等量dna制备混池,利用本实验室200多对具有多态性的分子筛选标记对基因进行初步定位,通过pcr扩增,琼脂糖凝胶电泳后,筛选出与目的基因连锁的分子标记,结果显示,在第7号染色体的长臂末端的两对ssr标记(p1,p2)具有较好连锁性,随后通过将剩余f2代突变体单株进行扩增分析,将目标基因初步定位于分子标记p5和p6之间。pcr反应体系(20μl):dna模板2μl,2×specifictmtaqmastermix10μl,上、下游引物各1μl,超纯水补足至20μl。pcr反应条件为94℃预变性3min;94℃变性30s,55℃退火30s,72℃延伸30s,32循环;72℃延伸5min。
crd1基因的精细定位:在rgp(ricegenomeresearchprogram,http://rgp.dna.affrc.go.jp/e/index.html)网站中下载突变位点附近的bac序列,用ssrhunter软件搜索bac中潜在的ssr序列,然后在该ssr序列两侧设计引物,利用tm值计算软件对引物进行评估,所扩增片段大小应为150~250bp。通过对两水稻品种差异碱基分析,设计snp引物。随后用该引物将两侧重组单株进行筛选,最后将目标基因定位于s2和s3之间的74.3kb区域中利用已知的水稻基因组bac序列。图位克隆过程所用的引物序列见表1。
表1.crd1图位克隆所用的引物
4、基因预测和序列分析
查看rgap网站的预测,在74.3kb区域中,共有13个开放阅读框,我们设计基因的测序引物通过用pcr方法(反应体系和条件同上)从野生型和突变型植株扩增出候选基因并进行测序分析。测序结果发现loc_os07g39920基因在野生型和突变体中上存在单个碱基差异,其编码区第610位碱基由胞嘧啶(c)变为胸腺嘧啶(t),而蜀恢527和日本晴序列在这一位点的碱基并不存在差异,突变体中这一单碱基改变导致其编码的第204位氨基酸脯氨酸(pro)被丝氨酸(ser)替代。在该基因的其他序列中没有突变位点,因此,我们推测这一突变位点很可能是crd1的突变位点,并把该orf确定为控制水稻叶绿体避光运动的相关基因crd1的候选基因,如图2所示,该基因全长2325bp,核苷酸序列为seqidno.1所示,该基因编码蛋白序列如seqidno.2所示。
表2.候选基因测序引物
实施例2
crd1的功能验证:
根据目的基因(loc_os07g39920)设计引物:
pmdc83-drt1-f:5’-caccatgaggaggagagttgcac-3’;
pmdc83-drt1-r:5’-agatgacgagctgtcctcat-3’。
利用neb公司q5高保真酶体系进行pcr扩增,反应体系为(20μl):dna模板2μl,2×mastermix10μl,上、下游引物各1μl,超纯水补足至20μl。pcr反应条件为98℃预变性30s;98℃变性10s,65℃退火30s,72℃延伸2min30s,30循环;72℃延伸10min。
pcr产物经电泳后,回收目标片段并接入petnr/d-topo载体,片段全长2322bp,利用gateway重组体系,将测序正确的片段重组入pmdc83,构建过表达载体,如图3所示,并通过农杆菌介导的方法将其转入crd1突变体中。实时定量pcr检测crd1基因表达情况,获得了过量表达株系oe-10;通过western的方法检测crd1蛋白水平,其表达量也高于野生型植株。该过量表达株系在强光处理时,叶绿体避光运动恢复至野生型水平,如图4所示。
实施例3
1、crd1组织表达分析
分别提取日本晴植株的根、茎、叶、叶鞘、孕穗、穗和花药的总rna,采用toyobo反转录试剂盒(revertra
反转录获取cdna后,定量pcr检测crd1基因在不同组织和器官中的表达量,发现crd1在叶片和叶鞘中表达量较高。
随后,以pcambia1301为骨架载体,根据启动子序列,设计引物pgus-f(yp2736)(5’-aagcttagccgtgctcgcggtta-3’)和pgus-r(yp2737)(5’-ccatggttcgaactcctccggta-3’),将crd1的启动子插入载体,构建pcrd1::gus。通过农杆菌介导的方法将其转入日本晴中,获取到转基因植株后,对其不同时期不同部位组织进行gus染色,结果发现crd1在胚芽鞘、叶片和根尖表达量较高,如图5所示。
2、crd1蛋白的亚细胞定位
我们构建了crd1全长的亚细胞定位载体,探究crd1在水稻中的亚细胞定位情况。质粒构建方法同实施例2中所述:引物为pmdc83-drt1-f(5’-caccatgaggaggagagttgcac-3’)和pmdc83-drt1-r(5’-agatgacgagctgtcctcat-3’),片段包含所述基因cds序列全长,利用neb公司q5高保真酶体系进行pcr扩增,反应体系和条件同实施例2。回收目标片段并接入petnr/d-topo载体,片段全长2322bp,利用gateway重组体系,将测序正确的片段重组入pgwb405,获得了用于转化的质粒pgwb405-35s::crd1-gfp。我们用peg转化法将亚细胞定位载体转到新鲜制备的水稻原生质体(粳稻品种日本晴)中,在激光共聚焦显微镜下观察水稻原生质体,可以明显的观察到融合蛋白gfp信号出现在细胞质膜、叶绿体或者二者都有,如图6所示。当用持续的蓝光照射细胞时,发现原来在细胞膜上的信号逐渐迁移至叶绿体,而叶绿体上的信号在持续的蓝光照下不发生变化,如图6所示。
3、crd1互作蛋白osphot2鉴定及功能验证
为了深入分析crd1影响叶绿体避光运动的分子机理,我们通过酵母双杂交方法验证crd1的互作蛋白。质粒构建采用实施例2中所述方法,首先通过pcr扩增crd1基因和osphot2的cds全长(所用引物见表3),测序验证后crd1基因片段克隆至pgbkt7载体,osphot2基因则分别克隆至pgadt7载体。酵母双杂交的实验采用matchmakergoldyeasttwo-hybridsystem(购自clontech),参照说明书方法操作。
结果如图7所示,crd1基因与osphot2基因共转化的酵母细胞能在sd/-his/-leu/-trp的培养基上生长,阴性对照均不能生长,说明crd1与osphot2在体外具有相互作用。同时,我们将具有突变位点的crd1序列扩增,连接入pgbkt7载体,其与osphot2基因共转化的酵母细胞不能在sd/-his/-leu/-trp的培养基上生长,说明该突变位点对于二者的互作具有重要作用。
此外,通过双分子荧光互补(lci,fireflyluciferasecomplementationimagingassay;方法可参照chen,h等,plantp,2008,146:368-376),实验分析进一步验证了在体内crd1与osphot2也发生相互作用,基因突变后,相互作用消失。
最后,我们利用pull-down实验进一步验证crd1与osphot2的互作关系。通过引物设计(见表3所示),pcr扩增野生型crd1基因和携带突变位点的crd1基因的fh1fh2结构域片段(1914bp)、fh1片段(546bp)和fh2片段(1369bp),用q5high-fidelity2×mastermix进行扩增,具体方法实施例2中所述。将目的片段构建入pgex-6p-1载体,为诱导表达crd1蛋白,我们将构建完成的3个质粒分别转化入大肠杆菌bl21(de3)中,转化方法同dh5α。在大肠杆菌bl21(de3)中诱导表达crd1fh1fh2-gst蛋白、crd1fh1-gst蛋白、crd1fh2-gst蛋白和crd1fh1fh2-gst(mt)蛋白,通过超声波破碎细胞后用还原型谷胱甘肽-琼脂糖亲和介质(beads)纯化目的蛋白,操作步骤如下:挑取单菌落于5mllb液体培养基(含50mg/l氨苄青霉素)中,37℃200rpm培养过夜;将菌液加至500ml相同培养基中,37℃,180rpm摇床培养至od600=0.6~0.8,室温静置1小时,加入10μl1m的iptg溶液,22℃,150rpm摇床诱导过夜;集菌体于-20℃冷冻过夜;在50ml离心管中加入30mlpbs(ph=7.2,含30μl蛋白酶抑制剂),振荡悬浮细胞后,用超声波细胞破碎仪破碎细胞(35赫兹),于4℃10000rpm离心60分钟,取上清于新离心管,再次离心;同时,取500μlbeads,用pbs清洗两次。将第二次离心所得上清与beads混合,4℃旋转结合过夜;将离心管于冰上静置,待beads沉淀后,弃上清,并用25ml预冷的pbs清洗beads两次,弃上清,将所得beads转至1.5ml离心管,4℃,500g离心除尽上清;加500μl10mm的还原型谷胱甘肽溶液,4℃洗脱过夜,离心收集上清;最后通过sds-聚丙烯酰胺凝胶电泳及考马斯亮蓝染色鉴定诱导蛋白。
利用相同的方法诱导纯化osphot2lov1-his、osphot2lov2-his和osphot2stk-his(将osphot2按结构域分为三个分段蛋白)。将gst、crd1fh1fh2-gst、crd1fh1-gst、crd1fh2-gst和crd1fh1fh2-gst(mt)分别与gst-sefinoseresin孵育1小时后,分别加入osphot2lov1-his、osphot2lov2-his和osphot2stk-his,4℃孵育2小时后westernblot检测,结果证实crd1与osphot2存在相互作用。
表3.蛋白相互作用载体构建引物
4、利用crispr/cas9技术创造osphot2突变体及其相关性状分析
鉴于osphot2是一个光受体,且证实与crd1存在互作关系,为进一步研究二者的互作对于植物叶绿体避光运动产生的作用,随后我们利用crispr/cas9技术创造了osphot2突变体,通过测序(测序引物为yp5393(5’-catcatctacgccagcgagg-3’)和yp5394(5’-ggcgacgaacgactagtgct-3’),由杭州擎科梓熙生物技术有限公司完成)发现,由于在第一个外显子末端一个“a”碱基的插入,使蛋白翻译提前终止,导致蛋白功能完全丧失。通过定量pcr检测,引物为osphot2-qpcr-f(yp6699)(5’-atgcctccacttgccacaat-3’)和osphot2-qpcr-r(yp6700)(5’-cacgaacaggatgaatccac-3’),osphot2基因的表达量极显著地降低。但该蛋白功能的缺失,对于植株的正常生长不产生影响,但其成株期叶片的叶绿素含量较野生型有明显下降,如图8所示。
5、crd1和osphot2的亚细胞定位及其对叶绿体避光运动的影响研究
有研究发现osphot2在细胞中的定位与crd1相似,在黑暗的情况下,其定位于细胞质膜,而当细胞感受到光照时,则从细胞质膜迁移至叶绿体上,这与我们所发现的crd1的定位情况极其相似,鉴于以上情况,我们更进一步的研究了二者相互之间对于彼此的亚细胞定位是否存在影响。通过构建crd1-gfp和osphot2-gfp质粒(方法见实施例2),通过peg介导分别转化入野生型、osphot2和crd1突变体中,激光共聚焦观察发现,crd1-gfp转入野生型、osphot2和crd1突变体三类原生质体后,黑暗时定位于细胞质膜,随着光照时间的持续,逐渐迁移至叶绿体上,基因突变对其定位与迁移不产生影响;osphot2-gfp转入野生型和osphot2突变体原生质体时,其定位与迁移也不受影响,但当转入crd1突变体后,黑暗情况下,定位于细胞质膜,随着光照的持续,osphot2-gfp无法迁移至叶绿体,由此可见,crd1对于osphot2在光照情况下的定位有重要作用。同时,我们对野生型、osphot2突变体、crd1突变体以及杂交获得的crd1/osphot2双突变体进行了叶绿体避光反应实验,结果显示,osphot2突变体、crd1突变体以及杂交获得的crd1/osphot2双突变体都丧失了叶绿体在强光下的避光反应。利用激光共聚焦显微镜观察叶绿体自发荧光也证实了这一结果,如图9所示。
序列表
<110>杭州师范大学
<120>一种水稻叶绿体避光运动调控基因crd1及其应用
<160>2
<170>siposequencelisting1.0
<210>1
<211>2325
<212>dna
<213>水稻(oryzasativa)
<400>1
atgaggaggagagttgcactgagcacggcgattgcgttgttagtgggtgctcaattatgt60
gtggcggcggaggtggaggtcgccggcgccggcggtggggttgtgcggcggcggtcgctc120
caccagccgttcttcccaatcgagtggtcgccgccgccgccgatgtccgggagcgaggcc180
gtcccgccgccgccgcctgcggccgcggcgtcggccaccaccggcggggggcggtcgacg240
accaccgtcatgaacaccgtcgccatcgcgctctcggcgggcctcgtggcgctcgcggtg300
gcgtcctactcctgctgcctcctgctgcggcgacgacgacgcgaggaggaggacgacggc360
gaccgcgccgcgaagcgggcggtgggggcggcggcggcggtggcggcgagagtccctagc420
gatgtcgggagcagctcccgccagcaccggtcgccgccgccgagctcgacggcgtcggac480
gcgatatacctcgacccgctcacgacgctggtggaggtgcgccagcacgaaaagagcccg540
gacctccgcccgctcccgctgctgaagcagccgagccctgacctccggccgttgccgccg600
ctgaagcggccggaatcgcaacccccgcctccgccaccgtccactccgccgttgaccacc660
acggggtattccaccgacgaggaggaccaggccacgtactacaccgcgccgaagaccgcc720
atgtcgtcgttcagccggagcacgagccagcacagcacgctggaacaaacagccatgccg780
cccatggccgcgcctgcaccgccgcaaaccaatcccccacgacctgttcgtccgccaccg840
ccgccgcccccgccgaggcagaggctgctccggccgctcccggcagaatcgccgccgccc900
gccgcgctggccaatctggaactaaccggttcccccgtcaaacccgctgttgaagacagg960
ggaggcgagaattccggcgccgcgcggccgcccaagccgccgcacctgaagccgctgcac1020
tgggacaagctccgggcaatctccggccgcaccaccgtgtgggatcaggtcaagaactcc1080
gacacattccgcgtcgacgaggaggcgatggagagcttgttcctgaacagcggcggcggc1140
ggcgcggggagctcggacccggcggcgaggagggggggatccgggaagcaggagaggcgg1200
ttgcttgacccgaagcggctgcagaacgtcgcgatcatgctcaaatcgctcaacgtggcc1260
gccgacgaagtgattggtgcactcgtgcgtggaaatcctgaagatttaggatctgagttc1320
tatgaaacactagctaagatggcaccaacaaaagaagaagaattgaaactaaaaggttat1380
agtggcgatttgtcaaaaattgatccagcagagcgctttctgaaagatgtacttggagtt1440
ccttttgccttcgagagagtggatgcgatgctgtacagagcaaattttgacaatgaagtg1500
aattatttaaggaaatcttttggaacattggaggcagcctgtgaagagttaagaagcagc1560
aaactttttttgaagctgctagatgcagttctaaaaaccgggaaccgcatgaatgatgga1620
accaatcgaggcgaggcgagggctttcaagcttgatactctcctaaagcttgcagacatc1680
aagtcaacagatggtagaacgacattgcttcacttcgtggttaaagagatcatccgatca1740
gaaggcttcgattctgatcagagcgcggttaatcctggcagcggcagcaaggaacagttc1800
aaaagggacggcttgaaattacttgcagggctcagcagtgagctctccaacgtgaagaga1860
gcggccacactggagatggacacactgagcggcaacatcctgaggctcgaagccgatctc1920
gagaaggtgaagcttgtcctacagctcaaggagacttgctcagatcaaggcgcgagcgag1980
aacttcttccaggcaatggttgtcttcctcagaagagcagaggcagagatcaagaacatg2040
aaaaccgcagaggagaacgcgttgcgcctcgtgaaggagacgacggagtacttccatggc2100
gacgccaccaaggaggagcctcaccctctgaggatattcgtggtggtggatgaattcctc2160
ctgatactggaccgtgtttgcagggacgtcggcaggacgccggagagggtgatgatgggc2220
tccggtaagtcgttccgcgtccctgcaggtacctcattgccgcctcatcggaatgagaac2280
cggcgagttcttagctcctccgatgaggacagctcgtcatcttag2325
<210>2
<211>774
<212>prt
<213>水稻(oryzasativa)
<400>2
metargargargvalalaleuserthralailealaleuleuvalgly
151015
alaglnleucysvalalaalagluvalgluvalalaglyalaglygly
202530
glyvalvalargargargserleuhisglnprophepheproileglu
354045
trpserproproproprometserglyserglualavalpropropro
505560
proproalaalaalaalaseralathrthrglyglyglyargserthr
65707580
thrthrvalmetasnthrvalalailealaleuseralaglyleuval
859095
alaleualavalalasertyrsercyscysleuleuleuargargarg
100105110
argarggluglugluaspaspglyaspargalaalalysargalaval
115120125
glyalaalaalaalavalalaalaargvalproseraspvalglyser
130135140
serserargglnhisargserproproproserserthralaserasp
145150155160
alailetyrleuaspproleuthrthrleuvalgluvalargglnhis
165170175
glulysserproaspleuargproleuproleuleulysglnproser
180185190
proaspleuargproleuproproleulysargprogluserglnpro
195200205
proproproproproserthrproproleuthrthrthrglytyrser
210215220
thraspglugluaspglnalathrtyrtyrthralaprolysthrala
225230235240
metserserpheserargserthrserglnhisserthrleuglugln
245250255
thralametproprometalaalaproalaproproglnthrasnpro
260265270
proargprovalargproproproproproproproproargglnarg
275280285
leuleuargproleuproalagluserproproproalaalaleuala
290295300
asnleugluleuthrglyserprovallysproalavalgluasparg
305310315320
glyglygluasnserglyalaalaargproprolysproprohisleu
325330335
lysproleuhistrpasplysleuargalaileserglyargthrthr
340345350
valtrpaspglnvallysasnseraspthrpheargvalaspgluglu
355360365
alametgluserleupheleuasnserglyglyglyglyalaglyser
370375380
seraspproalaalaargargglyglyserglylysglngluargarg
385390395400
leuleuaspprolysargleuglnasnvalalailemetleulysser
405410415
leuasnvalalaalaaspgluvalileglyalaleuvalargglyasn
420425430
progluaspleuglysergluphetyrgluthrleualalysmetala
435440445
prothrlysgluglugluleulysleulysglytyrserglyaspleu
450455460
serlysileaspproalagluargpheleulysaspvalleuglyval
465470475480
prophealaphegluargvalaspalametleutyrargalaasnphe
485490495
aspasngluvalasntyrleuarglysserpheglythrleugluala
500505510
alacysglugluleuargserserlysleupheleulysleuleuasp
515520525
alavalleulysthrglyasnargmetasnaspglythrasnarggly
530535540
glualaargalaphelysleuaspthrleuleulysleualaaspile
545550555560
lysserthraspglyargthrthrleuleuhisphevalvallysglu
565570575
ileileargsergluglypheaspseraspglnseralavalasnpro
580585590
glyserglyserlysgluglnphelysargaspglyleulysleuleu
595600605
alaglyleusersergluleuserasnvallysargalaalathrleu
610615620
glumetaspthrleuserglyasnileleuargleuglualaaspleu
625630635640
glulysvallysleuvalleuglnleulysgluthrcysseraspgln
645650655
glyalasergluasnphepheglnalametvalvalpheleuargarg
660665670
alaglualagluilelysasnmetlysthralaglugluasnalaleu
675680685
argleuvallysgluthrthrglutyrphehisglyaspalathrlys
690695700
glugluprohisproleuargilephevalvalvalaspglupheleu
705710715720
leuileleuaspargvalcysargaspvalglyargthrprogluarg
725730735
valmetmetglyserglylysserpheargvalproalaglythrser
740745750
leuproprohisargasngluasnargargvalleuserserserasp
755760765
gluaspserserserser
770
- 还没有人留言评论。精彩留言会获得点赞!