一种淀粉蔗糖酶突变体及其制备方法与应用与流程
本发明涉及一种淀粉蔗糖酶突变体及其制备方法与应用,属于基因工程和酶工程领域。
背景技术:
淀粉蔗糖酶(amylosucrase,as)是一种葡萄糖基转移酶(glucosyltransferase,e.c.2.4.1.4),隶属于糖苷水解酶(glycosidehydrolase,gh)13家族。它的主要功能是催化合成不可溶性的多糖,在葡聚糖存在的情况下,淀粉蔗糖酶能够以普通蔗糖作为唯一的能量来源和底物来催化合成直链淀粉,是一种工业上生产多糖的应用价值极高的酶。淀粉蔗糖酶可以以廉价的蔗糖为底物生产多聚糖,而不需要例如udp之类的昂贵的前体物质,使得工业上具有广泛的应用前景。淀粉蔗糖酶含有5个结构域(a、b、b'、c和n),其中a、b和b'-结构域构成了淀粉蔗糖酶的催化核心。
现有的淀粉蔗糖酶大多转苷能力很强,水解能力相对比较弱。研究淀粉蔗糖酶的水解和转苷作用的决定机制一直是一个热门的话题。目前,已经有许多关于水解和转苷方面的报道,但是大多集中在供体和受体位点,能显著改变水解和转苷平衡的的方法报道的还很少。因此,本发明通过单点突变显著提高了淀粉蔗糖酶的水解能力,说明此位点对淀粉蔗糖酶的水解和转苷作用很重要。进而该位点可以为其他淀粉蔗糖酶的水解和转苷的改造提供参考和借鉴。
技术实现要素:
本发明所要解决的一个技术问题是提供一种淀粉蔗糖酶的突变体,对deinococcusgeothermalis来源的淀粉蔗糖酶的第285位丙氨基酸进行突变;
或,对alteromonasmacleodii来源的淀粉蔗糖酶第287位丙氨基酸进行突变;
或,对neisseriapolysaccharea来源的淀粉蔗糖酶第295位丙氨基酸进行突变
所述突变位点与淀粉蔗糖酶的转苷作用和水解作用相关。
在本发明的一种实施方式中,所述来源于deinococcusgeothermalis的淀粉蔗糖酶的氨基酸序列如seqidno.1所示,所述来源于alteromonasmacleodii的淀粉蔗糖酶的氨基酸序列如seqidno.2所示,所述来源于neisseriapolysaccharea的淀粉蔗糖酶的氨基酸序列如seqidno.3所示。
所述发生突变的氨基酸位点是淀粉蔗糖酶突变体第285位或第287或第295的丙氨酸残基残基。
在本发明的一种实施方式中,所述淀粉蔗糖酶突变是将氨基酸序列如seqidno.1所示的第285位丙氨酸残基变为丝氨酸残基,突变体命名为a285s;
或,所述淀粉蔗糖酶突变是将氨基酸序列如seqidno.2所示的第287丙氨酸残基变为丝氨酸残基,突变体命名为a287s;
或,所述淀粉蔗糖酶突变是将氨基酸序列如seqidno.3所示的第295丙氨酸残基变为丝氨酸残基,突变体命名为a295s。
编码所述淀粉蔗糖酶突变体的基因。
携带所述淀粉蔗糖酶突变体的基因的载体。
携带权所述淀粉蔗糖酶突变体的基因的重组细胞。
本发明所要解决的另一个技术问题是提供一种淀粉蔗糖酶突变体的制备方法,包括如下步骤:
(1)粉蔗糖酶酶氨基酸序列的基础上确定突变位点;设计定点突变的突变引物,以携带淀粉蔗糖酶基因的载体为模板进行定点突变;构建含突变体的质粒载体;
(2)将突变体质粒转化进宿主细胞;
(3)挑选阳性克隆进行发酵培养,并分别纯化淀粉蔗糖酶酶突变体a285s、a287s,a295s。
所述质粒载体为puc系列,pet系列,或pgex中的任意一种。
所述宿主细胞为细菌和真菌细胞,其也为本发明的保护范围。
所述的细菌为革兰氏阴性菌或革兰氏阳性菌。
所述淀粉蔗糖酶突变体在生产多聚糖的应用
有益效果:
本发明分别对来源于deinococcusgeothermalis的淀粉蔗糖酶第285位丙氨酸残基进行定点突变,对来源于alteromonasmacleodii的淀粉蔗糖酶第287位丙氨酸残基进行定点突变,对来源于neisseriapolysaccharea的淀粉蔗糖酶第295位丙氨酸残基进行定点突变。获得的单突变体酶的水解率较野生型淀粉蔗糖酶均得到提高。在最优的酶转化条件下,a285s突变体的水解率是野生酶水解率的8倍,转苷率降低至野生型的32%左右;a295s突变体的水解率是野生酶水解率的6.6倍,转苷率降低至野生型的23%左右;a287s突变体的水解率是野生酶水解率的5.5倍,转苷率降低至野生型的22%左右。因此,本发明提供的淀粉蔗糖酶突变体a285s、a287s和a295s可以应用于糖苷水解酶工业生产多聚糖。
附图说明
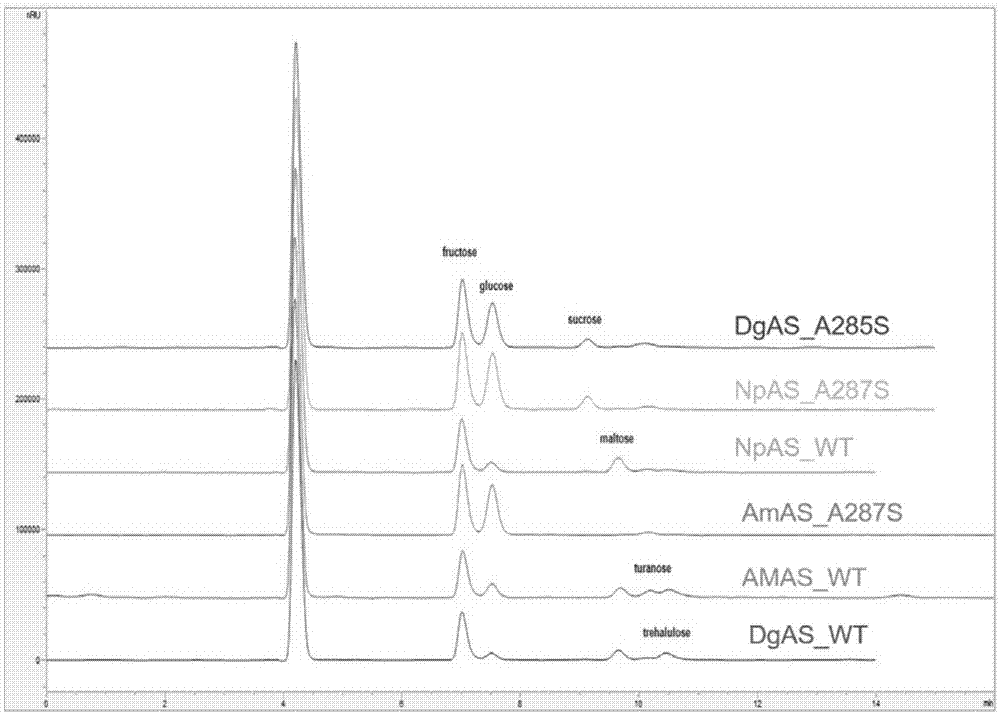
图1野生酶以及突变体的水解率、异构率、聚合率和转苷率的hplc检测结果。
具体实施方式
下述实施例中所涉及的培养基和计算方法如下:
lb固体培养基:5g/l酵母粉,10g/l蛋白胨,5g/lnacl,2%琼脂粉。
lb液体培养基:5g/l酵母粉,10g/l蛋白胨,5g/lnacl。
水解率={(生成的葡萄糖含量)/(消耗的蔗糖含量-生成的果糖含量)}*100%
异构率={(异构产物含量)/(消耗的蔗糖含量-生成的果糖含量)}*100%
聚合率={(聚合产物含量)/(消耗的蔗糖含量-生成的果糖含量)}*100%
转苷率=异构率+聚合率
实施例1:重组菌构建
根据ncbi上登录号为abf44874.1的淀粉蔗糖酶基因序列采用化学合成法合成含有淀粉蔗糖酶的dgas基因,登录号为q9zeu2.1的淀粉蔗糖酶基因序列采用化学合成法合成含有淀粉蔗糖酶的npas基因,登录号为bag82876.1的淀粉蔗糖酶基因序列采用化学合成法合成含有淀粉蔗糖酶的amas基因。将dgas基因、npas基因、amas基因分别与pet-24a(+)质粒用ndei和hindiii双酶切,酶切产物割胶回收后,再用t4连接酶连接,连接产物转化e.colijm109感受态细胞,得到重组细胞。将重组细胞37℃培养8h,挑取转化子在lb液体培养基(含30mg/l卡纳霉素)中震荡培养,提取质粒,酶切验证后分别得到表达质粒dgas/pet-24a(+)、npas/pet-24a(+)、amas/pet-24a(+)。
将质粒dgas/pet-24a(+)、npas/pet-24a(+)、amas/pet-24a(+)分别转化e.colibl21(de3)宿主菌,涂布lb平板(含30mg/l卡纳霉素),37℃培养8h,挑单菌落到lb液体培养基(含30mg/l卡纳霉素)中,37℃培养过夜,保存于甘油管中。
分别挑取重组菌e.colijbl21(de3)/dgas/pet-24a(+)、e.colijbl21(de3)/amas/pet-24a(+)、e.colibl21(de3)/npas/pet-24a(+),于lb液体培养基(含30μg/ml卡纳霉素)生长8~10h,按5%接种量将种子发酵液接到tb培养基(含30μg/ml卡纳霉素)中,当光密度在600nm下达到0.6时,加当600nm处的光密度达到0.6时,加入0.4mm异丙基β-d-1-硫代吡喃半乳糖苷(iptg)进行诱导,在25℃摇床中培养24h后,将发酵液于4℃、8000rpm离心20min除菌体,收集离心上清液得到野生菌粗酶液。
实施例2:淀粉蔗糖酶突变体的制备
(1)淀粉蔗糖酶单突变的制备
根据淀粉蔗糖酶的dgas基因序列,设计并合成引入a285s突变的引物,利用快速pcr技术,以携带编码野生型淀粉蔗糖酶的基因的质粒dgas/pet-24a(+)为模板,对淀粉蔗糖酶dgas基因序列进行定点突变,测定dna编码序列,鉴别出第285位ala密码子变成ser密码子的基因,得到淀粉蔗糖酶单突变a285s。
根据淀粉蔗糖酶的amas基因序列,设计并合成引入a287s突变的引物,利用快速pcr技术,以携带编码野生型淀粉蔗糖酶的基因的质粒amas/pet-24a(+)为模板,对淀粉蔗糖酶amas基因序列进行定点突变,测定dna编码序列,鉴别出第287位ala密码子变成ser密码子的基因,得到淀粉蔗糖酶单突变a287s。
根据淀粉蔗糖酶的npas基因序列,设计并合成引入a295s突变的引物,利用快速pcr技术,以携带编码野生型淀粉蔗糖酶的基因的质粒npas/pet-24a(+)为模板,对淀粉蔗糖酶npas基因序列进行定点突变,测定dna编码序列,鉴别出第295位ala密码子变成ser密码子的基因,得到淀粉蔗糖酶单突变a295s。
引入a285s突变的定点突变引物为:
核苷酸序列为seqidno.4的正向引物:
5’-gtgtttcgcctggatagcattgcatttatttgg-3’(下划线为突变碱基)
核苷酸序列为seqidno.5的反向引物:
5’-ccaaataaatgcaatgctatccaggcgaaacac-3’(下划线为突变碱基)
引入a287s突变的定点突变引物为:
核苷酸序列为seqidno.6的正向引物:
5’-cgccctgcgcttagatagcctggcctttatttgg-3’(下划线为突变碱基)
核苷酸序列为seqidno.7的反向引物:
5’-ccaaataaaggccaggctatctaagcgcagggcg-3’(下划线为突变碱基)
引入a295s突变的定点突变引物为:
核苷酸序列为seqidno.8的正向引物:
5’-atattacgcatggatagcgttgcgtttatttgg-3’(下划线为突变碱基)
核苷酸序列为seqidno.9的反向引物:
5’-ccaaataaacgcaacgctatccatgcgtaatat-3’(下划线为突变碱基)
pcr反应体系均为:5×psbuffer10μl,dntpsmix(2.5mm)4μl,正向引物(10μm)1μl,反向引物(10μm)1μl,模板dna1μl,primerstarhs(5u/μl)0.5μl,加入双蒸水至50μl。
pcr扩增条件为:94℃预变性4min;随后30个循环(98℃10s,55℃5s,72℃8min);72℃继续延伸10min。
pcr产物经dpnⅰ消化,转化大肠杆菌jm109感受态,感受态细胞在lb固体培养基(含30μg/ml卡纳霉素)培养过夜后,挑克隆于lb液体培养基(含30μg/ml卡纳霉素)中培养后提取质粒,所有突变质粒均测序正确,得到的重组菌命名为e.colijm109/dgas/pet-24a(+)(a285s)、e.colijm109/amas/pet-24a(+)(a287s)、e.colijm109/npas/pet-24a(+)(a295s)。
测序正确的突变体,从甘油管接种至lb培养基,过夜培养,提取质粒,将质粒转化表达宿主大肠杆菌bl21(de3)感受态细胞,得到的重组菌命名为e.colijbl21(de3)/dgas/pet-24a(+)(a285s)、e.colijbl21(de3)/amas/pet-24a(+)(a287s)、e.colibl21(de3)/npas/pet-24a(+)(a295s)。
(2)突变体酶的发酵与纯化
分别挑取重组菌e.colijbl21(de3)/dgas/pet-24a(+)(a285s)、e.colijbl21(de3)/amas/pet-24a(+)(a287s)、e.colibl21(de3)/npas/pet-24a(+)(a295s)。于lb液体培养基(含30μg/ml卡纳霉素)生长8~10h,按5%接种量将种子发酵液接到tb培养基(含30μg/ml卡纳霉素)中,当光密度在600nm下达到0.6时,当600nm处的光密度达到0.6时,加入0.4mm异丙基β-d-1-硫代吡喃半乳糖苷(iptg)进行诱导,在25℃摇床中培养24h后,将发酵液于4℃、8000rpm离心20min除菌体,收集离心上清液得到粗酶液。
实施例3:粗酶液的浓缩
将实施例1和2中获得的粗酶液边搅拌边缓慢加入浓度为相对于酶液质量分数20%的硫酸铵,搅拌至硫酸铵溶解,在4℃条件下静置8~10小时沉淀蛋白。混合物经离心(8000rpm,10min)收集沉淀,再用最小体积的50mmkh2po4-na2hpo4缓冲液(ph7.0)复溶,复溶后经过再次离心除去固形物,收集上清透析后获得浓缩酶液。
实施例4:hplc检测水解和转苷产物的产量
在反应器中加入100mm蔗糖作为底物,加入3u/ml酶活的实施例3中获得的突变体浓缩酶液,和野生型的加酶量保持一致。在30℃,150rpm的水浴摇床中反应24小时后取样,终止反应后,过膜过滤并进行hplc分析。色谱条件如下:agilent1200hplc色谱仪,agilent自动进样器,agilent氨基柱5mm,(4.6mm×250mm)示差折光检测器;流动相为80%乙腈,20%超纯水,流速0.8mlmin-1;柱温35℃。hplc检测结果见图1,其中水解率表示生成的葡萄糖含量,异构率表示生产的异构产物(异构产物为松二糖和海藻酮糖)的含量,聚合率表示生产的聚合产物(聚合产物为麦芽寡糖),转苷率包括异构率加聚合率。
结果数值见表1,淀粉蔗糖酶水解率大幅度提升,其中a285s突变体的水解率提升幅度最大,是野生酶水解率的8倍,转苷率降低至野生型的32%左右;a287s突变体的水解率是野生酶水解率的5.5倍,转苷率降低至野生型的22%左右;a295s突变体的水解率是野生酶水解率的6.6倍,转苷率降低至野生型的23%左右。这说明突变位点是淀粉蔗糖酶糖苷和水解功能的关键位点,对于糖苷水解酶的糖苷和水解性质的研究具有重要意义。
表1野生酶以及突变体的水解率、异构率、聚合率和转苷率
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
sequencelisting
<110>江南大学
<120>一种淀粉蔗糖酶突变体及其制备方法与应用
<160>9
<170>patentinversion3.3
<210>1
<211>650
<212>prt
<213>deinococcusgeothermalis
<400>1
metleulysaspvalleuthrsergluleualaalaglnvalargasp
151015
alapheaspaspaspargaspalagluthrpheleuleuargleuglu
202530
argtyrglygluaspleutrpgluserleuargalavaltyrglyasp
354045
glnvalargalaleuproglyargleuleugluvalmetleuhisala
505560
tyrhisalaargproalagluleuargargleuaspglualaargleu
65707580
leuargproasptrpleuglnargproglumetvalglytyrvalala
859095
tyrthraspargphealaglythrleulysglyvalglugluargleu
100105110
asptyrleugluglyleuglyvallystyrleuhisleumetproleu
115120125
leuargproarggluglygluasnaspglyglytyralavalglnasp
130135140
tyrargalavalargproaspleuglythrmetaspaspleuserala
145150155160
leualaargalaleuargglyargglyileserleuvalleuaspleu
165170175
valleuasnhisvalalaarggluhisalatrpalaglnlysalaarg
180185190
alaglyaspprolystyrargalatyrphehisleupheproasparg
195200205
argglyproaspalapheglualathrleuprogluilepheproasp
210215220
phealaproglyasnphesertrpaspglugluileglygluglyglu
225230235240
glyglytrpvaltrpthrthrpheasnsertyrglntrpaspleuasn
245250255
trpalaasnproaspvalpheleugluphevalaspileileleutyr
260265270
leualaasnargglyvalgluvalpheargleuaspalailealaphe
275280285
iletrplysargleuglythraspcysglnasnglnprogluvalhis
290295300
hisleuthrargalaleuargalaalaalaargilevalalaproala
305310315320
valalaphelysalaglualailevalalaproalaaspleuilehis
325330335
tyrleuglythrargalahishisglylysvalseraspmetalatyr
340345350
hisasnserleumetvalglnleutrpserserleualaserargasn
355360365
thrargleuphegluglualaleuargalapheproprolysprothr
370375380
serthrthrtrpglyleutyrvalargcyshisaspaspileglytrp
385390395400
alaileseraspgluaspalaalaargalaglyleuasnglyalaala
405410415
hisarghispheleuseraspphetyrserglyglnpheproglyser
420425430
phealaargglyleuvalpheglntyrasnprovalasnglyasparg
435440445
argileserglyseralaalaserleualaglyleuglualaalaleu
450455460
gluthrglyaspproglyargilegluaspalavalargargleuleu
465470475480
leuleuhisthrvalileleuglypheglyglyvalproleuleutyr
485490495
metglyaspgluleualaleuleuasnasptyralaphegluaspval
500505510
progluhisalaproaspasnargtrpvalhisargproglnmetasp
515520525
trpalaleualagluargvalargglngluproserserproalagly
530535540
argvalasnthrglyleuarghisleuleuargvalargargaspthr
545550555560
proglnleuhisalaserilegluserglnvalleuproserproasp
565570575
serargalaleuleuleuargargasphisproleuglyglymetval
580585590
glnvaltyrasnpheserglugluthrvalmetleuproserhisval
595600605
leuargaspvalleuglyasphisvalglnaspargleuserglyser
610615620
alapheargleuaspargprothrvalargleugluglytyrargala
625630635640
leutrpleuthralaglyglualaproala
645650
<210>2
<211>649
<212>prt
<213>alteromonasmacleodii
<400>2
metsertyralaalaaspserglyvalcysleuaspargileleuala
151015
serthraspleuserglyleuserlyslysaspalathrthrphethr
202530
argargleulysgluhispheproargleupheasplystyrthrtyr
354045
valtyrglyhisleutyraspcystyrphehisleuglnlysleuval
505560
gluileleuargaspglyleulysasnarglysproalaleulyslys
65707580
leuaspasngluargleulysasnproleutrptyrargaspglugln
859095
glnvalglymetalacystyrvalaspleumetglyprothrleuasn
100105110
aspleuhisasplysileprotyrphelysserleuglyvalthrtyr
115120125
valhisleumetproleutyraspalaprolysglyaspseraspgly
130135140
glytyralavalserasptyrarglysvalasnprothrleuglythr
145150155160
vallysaspleuglulysleualaseralaleuglnlysgluglyile
165170175
asnleuvalleuaspphevalpheasnhisthrseraspgluhisala
180185190
trpalalysalaalaleualaglyglulysglnpheglnasptyrtyr
195200205
tyrleupheaspaspargthrileproaspglntyrgluhisthrleu
210215220
arggluilepheproglnvalargargglyserphethrpheasnglu
225230235240
glnileglulystrpvaltrpthrthrpheasnserpheglntrpasp
245250255
leuasntyrserasnprogluvalpheasnalailethrserglumet
260265270
leupheleualaasnileglyseralaalaleuargleuaspalaleu
275280285
alapheiletrplysgluleuglythrasncysgluasnglnglulys
290295300
alahisvalleuileglnalapheasncyscysleuglnilevalala
305310315320
proalavalvalphelysserglualailevalhisproaspgluval
325330335
leulystyrileasplysaspglucysglnleusertyrasnproleu
340345350
leumetalaleuleutrpasnserleualathrarglysthrargleu
355360365
leuthrargsermetlyslysserpheserilesergluasncysthr
370375380
trpvalasntyrvalargcyshisaspaspileglytrpthrpheasp
385390395400
aspalavalalaasngluleuglyileasnalapheasphisarghis
405410415
pheleuasnglnphetyrthrglyargphegluglyserphealaasn
420425430
glyvalprophealagluasnproserasnglyaspcysargvalcys
435440445
glyserleualaserleucysglyleugluglyalaleulysserasn
450455460
aspserlysglyileaspaspalavallysargmetmetleuleutyr
465470475480
glyilethrpheserileglyglyileproleuleutyrserserasp
485490495
gluvalglylysleuasnasptyrsertyrargpheaspaspthrlys
500505510
lyshisaspaspargtrpvalasnargilealavalthrargalaasp
515520525
thrgluleualaleuglyasnthrserproalaasnprothrgluthr
530535540
alaserleuargvalpheasnglyleuarglysmetilealailearg
545550555560
lysglnasnalailepheglyglnserserthrglnileleuaspala
565570575
aspasnhishiscysphealapheleuarggluasnthrglnglyhis
580585590
lysleuleuvalilecysasnphesergluhisproglnthrilegln
595600605
argserleuleualathrleulysglyalaserserargaspleuleu
610615620
thraspgluthrvalglumetasnalaserproleuleuleuglupro
625630635640
pheglnglnmettrpleuleualaasp
645
<210>3
<211>636
<212>prt
<213>neisseriapolysaccharea
<400>3
metleuthrprothrglnglnvalglyleuileleuglntyrleulys
151015
thrargileleuaspiletyrthrprogluglnargalaglyileglu
202530
lyssergluasptrpargglnpheserargargmetaspthrhisphe
354045
prolysleumetasngluleuaspservaltyrglyasnasngluala
505560
leuleuprometleuglumetleuleualaglnalatrpglnsertyr
65707580
serglnargasnserserleulysaspileaspilealaarggluasn
859095
asnproasptrpileleuserasnlysglnvalglyglyvalcystyr
100105110
valaspleuphealaglyaspleulysglyleulysasplysilepro
115120125
tyrpheglngluleuglyleuthrtyrleuhisleumetproleuphe
130135140
lyscysprogluglylysseraspglyglytyralavalsersertyr
145150155160
argaspvalasnproalaleuglythrileglyaspleuarggluval
165170175
ilealaalaleuhisglualaglyileseralavalvalasppheile
180185190
pheasnhisthrserasngluhisglutrpalaglnargcysalaala
195200205
glyaspproleupheaspasnphetyrtyrilepheproaspargarg
210215220
metproaspglntyraspargthrleuarggluilepheproaspgln
225230235240
hisproglyglypheserglnleugluaspglyargtrpvaltrpthr
245250255
thrpheasnserpheglntrpaspleuasntyrserasnprotrpval
260265270
pheargalametalaglyglumetleupheleualaasnleuglyval
275280285
aspileleuargmetaspalavalalapheiletrplysglnmetgly
290295300
thrsercysgluasnleuproglnalahisalaleuileargalaphe
305310315320
asnalavalmetargilealaalaproalavalphephelysserglu
325330335
alailevalhisproaspglnvalvalglntyrileglyglnaspglu
340345350
cysglnileglytyrasnproleuglnmetalaleuleutrpasnthr
355360365
leualathrarggluvalasnleuleuhisglnalaleuthrtyrarg
370375380
hisasnleuprogluhisthralatrpvalasntyrvalargserhis
385390395400
aspaspileglytrpthrphealaaspgluaspalaalatyrleugly
405410415
ileserglytyrasphisargglnpheleuasnargphephevalasn
420425430
argpheaspglyserphealaargglyvalpropheglntyrasnpro
435440445
serthrglyaspcysargvalserglythralaalaalaleuvalgly
450455460
leualaglnaspaspprohisalavalaspargilelysleuleutyr
465470475480
serilealaleuserthrglyglyleuproleuiletyrleuglyasp
485490495
gluvalglythrleuasnaspaspasptrpserglnaspserasnlys
500505510
seraspaspserargtrpalahisargproargtyrasnglualaleu
515520525
tyralaglnargasnaspproserthralaalaglyglniletyrgln
530535540
glyleuarghismetilealavalargglnserasnproargpheasp
545550555560
glyglyargleuvalthrpheasnthrasnasnlyshisileilegly
565570575
tyrileargasnasnalaleuleualapheglyasnpheserglutyr
580585590
proglnthrvalthralahisthrleuglnalametprophelysala
595600605
hisaspleuileglyglylysthrvalserleuasnglnaspleuthr
610615620
leuglnprotyrglnvalmettrpleugluileala
625630635
<210>4
<211>33
<212>dna
<213>人工合成
<400>4
gtgtttcgcctggatagcattgcatttatttgg33
<210>5
<211>33
<212>dna
<213>人工合成
<400>5
ccaaataaatgcaatgctatccaggcgaaacac33
<210>6
<211>34
<212>dna
<213>人工合成
<400>6
cgccctgcgcttagatagcctggcctttatttgg34
<210>7
<211>34
<212>dna
<213>人工合成
<400>7
ccaaataaaggccaggctatctaagcgcagggcg34
<210>8
<211>33
<212>dna
<213>人工合成
<400>8
atattacgcatggatagcgttgcgtttatttgg33
<210>9
<211>33
<212>dna
<213>人工合成
<400>9
ccaaataaacgcaacgctatccatgcgtaatat33
- 还没有人留言评论。精彩留言会获得点赞!