一种提高细胞分辨率染色质可及性的方法与流程
本发明属于细胞生物学实验
技术领域:
,具体涉及一种提高细胞分辨率染色质可及性的方法。
背景技术:
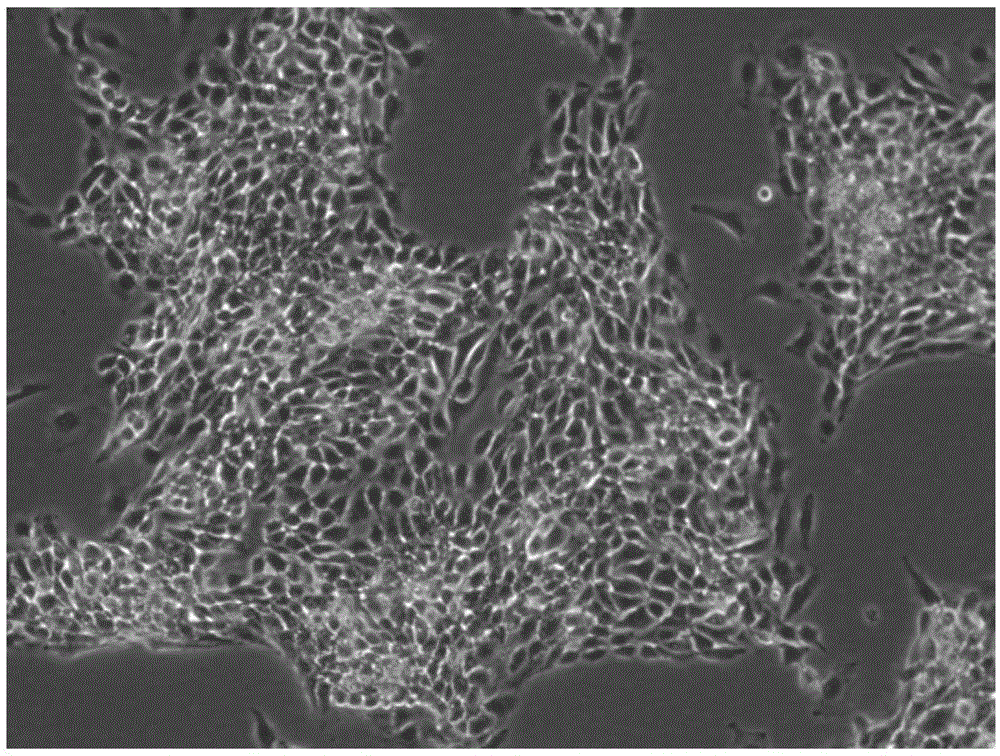
:目前有关3d基因组的研究越来越火热,其中染色质可及性(chromatinaccessibility)的研究主题是热点之一,其对基因表达起着至关重要的调控作用(buenrostro,giresietal.2013)。在真核生物中,染色质被紧密地包装成一连串的核小体,每个核小体由组蛋白八聚体和dna组成,大多数基因组中的染色质都紧紧盘绕在细胞核内,但也有一些区域经染色质重塑后呈现出松散的状态,这种状态或者基因结构被称为染色质的可及性。目前研究染色质可及性的方法有很多种,如:mnase-seq,dnase-seq,faire-seq,atac-seq(assayfortransposase-accessiblechromatinwithhigh-throughputsequencing),除了mnase外,其他方法均直接分离染色质开放区域(mariatsompana1andmichaeljbuck12014,lu,liuetal.2016)。atac-seq方法相比dnase-seq,mnase-seq,faire-seq步骤更为简单,需要的细胞也更少只需500到50000个新鲜细胞,操作时间缩短至几个小时,但是由于使用了tn5转座酶导致成本较高,得到的数据和dnase-seq,mnase-seq不能完全的匹配,需要结合chip-seq,rna-seq进行整合分析,并且数据的分辨率较低(buenrostro,wuetal.2015)。所以开展一种获得高分辨率可及性的方法是很关键的,这对以后在组织上开展染色质可及性至关重要。技术实现要素:本发明的目的在于客服现有技术的缺陷,提供一种提高细胞分辨率染色质可及性的方法。本发明基于新的nebnextfs片段化酶以及片段选择策略,针对性的进行了系统优化和配置,使染色质切割效率和精度最大化,最终获得高分辨率的染色质开放区域信息,与atac-seq等方法相比较极大的降低了实验成本,为细胞染色质开放区域的研究提供了新的思路和可行方法。本发明通过以下技术方案实现:一种细胞高分辨率染色质可及性的方法,包括以下步骤:(1)用10%澳洲胎牛血清培养基培养猪肺泡巨噬细胞,培养2-3天,用500μl胰酶消化,再用1ml澳洲胎牛血清培养基重悬细胞,用细胞计数板计算细胞量,收集500-50000个细胞,在500g,4℃下离心5min;(2)弃上清,取沉淀,用50μl冷磷酸盐缓冲液(商购)重悬细胞团,用移液枪上下吹匀,进行清洗,然后在500g,4℃下离心5min;将所得的沉淀再次用50μl冷磷酸盐缓冲液(商购)重悬,吹匀,然后在500g,4℃下离心5min,最后弃上清;(3)加50μl冷裂解液(商购)重悬细胞团,用移液枪上下吹匀,然后在rocker10上4℃旋转8min,然后在500g,4℃下离心5min,最后弃上清;(4)用24μlte缓冲液(商购)重悬细胞团,向其中加入nebnextultraiifs反应缓冲液(商购)7μl;nebnextultraiifs酶混合液(商购)4μl,最后放置在热循环仪上孵育,孵育条件为:37℃,14min;65℃,30min;4℃,保持;(5)使用qiagenpcr产物纯化试剂盒(商购)回收片段化后的染色质,用35μlqiagen洗脱缓冲液(商购)洗脱dna;(6)直接向步骤(5)中35μl缓冲液(商购)洗脱dna中加入nebnextultraii连接混合液(商购)30μl;nebnext连接体系1μl;nebnext适配器2.5μl;至总体积68.5μl,用枪吹匀,并放置在热循环仪上孵育,孵育条件为:20℃,15min,pcr盖子温度关闭;(7)向步骤(6)连接后的体系中加3μl尿嘧啶-特异性切除酶,用枪吹匀,短暂离心;将离心后的产物放置在热循环仪上孵育,孵育条件为:37℃,15min,热循环仪盖子温度应大于等于47℃;(8)用体积比为1:1.8的128.7μlampure磁珠纯化回收连接后的产物,用17μl0.1xte缓冲液洗脱;(9)使用nebnextultraiifsdna文库制备试剂盒(商购)进行pcr扩增;(10)用体积比为1:150μlampure磁珠回收扩增后的产物,用33μl0.1xte缓冲液(商购)洗脱dna;(11)使用高敏感dna芯片在agilent2100检测扩增后文库的片段大小的分布。(12)使用sagesciencebluepippin仪器对文库进行片段选择,选择280-350bp的片段进行测序;其中步骤(1)中的澳洲胎牛血清培养基成分按质量计为:澳洲胎牛血清10%,非必需氨基酸1%,补充rpmi1640培养基178ml;步骤(3)中的裂解液按质量计为:tris·hclph7.510mm;氯化钠10mm;氯化镁3mm;聚乙氧基乙醇辛基苯基-聚乙烯二醇0.1%;补充双蒸水9.75ml。操作方法中其他培养基或试剂或溶液成分及其制备方法见《具体实施方式》。本发明的难点在于如何得到裂解合适的细胞核,不同的细胞裂解时间需要把控,并且该技术关键的步骤是使用nebnextultraiifs文库制备试剂盒中的fs酶替换tn5转座酶,通过fs酶的作用得到合适大小的片段,进行建库和扩增,核心是对建库后的dna进行片段选择,得到具有高分辨率的染色质开放区域。本发明操作的方法是专门化针对传代细胞进行了优化,使用裂解液进行裂解,所述的裂解液主要包括10mmtrisph7.5,10mmnacl,3mmmgcl2,0.1%igepalca-630等成分。本发明中使用fs酶替代tn5转座酶,转换必须依赖nextera建库到依赖nebnextultraii建库,大大节约成本,从fs剪切的片段大小看优于tn5。对比之前atac的实验结果,发现后期对文库片段大小的筛选对于得到高分辨率的结果至关重要(wu,xuetal.2018)。本发明中的实验方法操作简便(详见《具体实施方式》),更重要的是利用此方法获得的片段大小可以得到更好的数据质量,与greenleaf等人报道的atac结果对比,使用fs所需要的成本较低,上机测序比较便捷。利用fs得到的结果并不比greenleaf等人报道的atac-seq结果差,而且更是产生了高分辨率的结果。本发明使得研究染色质可及性的方法得到了创新,不在依赖于tn5转座酶,大大降低了成本,并对未来在细胞和组织上开展染色质可及性研究奠定了基础。本发明的应用使得研究染色质开放区域不仅仅在一个或者几个核小体的范围内,甚至可以到一个核小体或是一个核小体的亚结构,这使得对染色质开放区有了更深一步的了解,为染色质可及性研究拓展了新的思路。附图说明图1:本发明涉及的3d4/21细胞结果。附图标记说明:10%fbsin1640细胞培养基,100x显微镜下细胞的生长图。图2:本发明中fs酶切片段大小在agilent2100上展示的结果图。附图标记说明:通过agilent2100仪器分析建库后的atac的片段大小分布,发现经过fs酶切以后的片段大小符合预期。图3:本发明测序后插入片段大小展示出高分辨率的分布图。附图标记说明:对比其他atac-seq实验结果(图3中的c图),hica-seq插入片段大小集中在180bp(图3中的a图,图3中的b图),表明本发明选择的片段具有高分辨率结果。图4:基因转录起始位点(tss)富集分析的展示图。附图标记说明:经过快速傅里叶变换(fastfouriertransformation,fft)后的read分布,自由区域的reads在tss周围分布数目极少(图4中的a图),而跨核小体的reads则在tss周围有富集分布(图4中的b图)。图5:本发明所得数据展现的结果图。附图标记说明:在3d4细胞系上实验数据展示及h3k27ac,h3k27me3,的chip数据呈现很好的峰形,且基因内部的亚结构得到了细致的呈现,具有典型高分辨特征。具体实施方式1.主要试剂及其配制方法(1)主要试剂:水(nuclease-freewater)(货号#am9932,ambion,usa);tris·hclph7.5(货号#am9856,ambion,usa);氯化钠(货号#am9760g,ambion,usa);氯化镁(cat#am9530g,ambion,usa);igepalca-630(货号#i8896,sigma,usa);磷酸盐缓冲液(pbs)(货号#344198,millipore,usa);qiagenminelutepcr产物纯化试剂盒(货号#28004,qiagen,usa);聚乙氧基乙醇辛基苯基-聚乙烯二醇(igepalca-630)(货号#13021,sigma,usa);rpmi1640培养基(货号#11875-093,gibco,usa);澳洲胎牛血清(fetalbovineserum,qualified,australiaorigin)(货号#10099-141,gibco,usa);非必需氨基酸(non-essentialaminoacid)(货号#11140050,life,usa)(2)试剂的配制:1)lysisbuffer裂解液的配制见表1(配置后4℃下储存一周):配方:按质量计:tris·hclph7.510mm;氯化钠10mm;氯化镁3mm;聚乙氧基乙醇辛基苯基-聚乙烯二醇(igepalca-630)0.1%;补充双蒸水9.75ml。表1lysisbuffer裂解液配方试剂储存浓度工作浓度10mltris·hclph7.51m10mm100ul氯化钠5m10mm20ul氯化镁1m3mm30uligepalca-63010%0.1%100ul水9.75ml2)澳洲胎牛血清培养基的配制澳洲培养基配方及制备方法见表2。配方:按质量计:10%澳洲胎牛血清(fetalbovineserum)10%,非必需氨基酸(non-essentialaminoacid)1%,补充(商购)rpmi1640培养基178ml。表210%澳洲胎牛血清培养基配方试剂储存浓度工作浓度200ml澳洲胎牛血清100%10%20ml非必需氨基酸100%1%2mlrpmi1640培养基178ml2.具体步骤:(1)用(10%)澳洲胎牛血清培养基培养猪肺泡巨噬细胞,培养2-3天,用500μl胰酶消化,再用1ml澳洲胎牛血清培养基(澳洲胎牛血清10%,非必需氨基酸1%,补充商购生物rpmi1640培养基178ml)重悬细胞,用细胞计数板计算细胞量,收集500-50000个细胞,在500g,4℃下离心5min。(2)弃上清,取沉淀,用50μl冷磷酸盐缓冲液重悬细胞团,用移液枪上下吹匀,进行清洗,然后在500g,4℃下离心5min;将所得的沉淀再次用50μl冷磷酸盐缓冲液重悬,吹匀,然后在500g,4℃下离心5min,最后弃上清。(3)加50μl冷裂解液重悬细胞团,用移液枪上下吹匀,然后在rocker10上4℃旋转8min。然后在500g,4℃下离心5min,最后弃上清。(4)用24μlte缓冲液重悬细胞团,向其中加入nebnextultraiifs反应缓冲液7μl;nebnextultraiifs酶混合液4μl,之后放置在热循环仪上孵育(37℃,14min;65℃,30min;4℃,保持)。(5)使用qiagenpcr产物纯化试剂盒回收片段化后的染色质,35μlqiagen洗脱缓冲液洗脱dna。(6)直接向步骤(5)中35μl缓冲液洗脱dna中加入nebnextultraii连接混合液30μl;nebnext连接体系1μl;nebnext适配器2.5μl;至总体积68.5μl,用枪吹匀,并放置在热循环仪上孵育(20℃,15min,pcr盖子温度关闭)。(7)向步骤(6)连接后的体系中加3μl尿嘧啶-特异性切除酶(user),用枪吹匀,短暂离心。并放置在热循环仪上孵育(37℃,15min,热循环仪盖子温度应大于等于47℃)。(8)用128.7μlampure磁珠(体积比为1:1.8)纯化回收连接后的产物,17μl0.1xte缓冲液洗脱。(9)使用nebnextultraiifsdna文库制备试剂盒进行pcr扩增。(10)用50μlampure磁珠回收(体积比为1:1)扩增后的产物,用33μl0.1xte缓冲液洗脱dna。(11)使用高敏感dna芯片在agilent2100检测扩增后文库的片段大小的分布。(12)使用sagesciencebluepippin仪器对文库进行片段选择,选择280-350bp的片段进行测序。其中步骤(1)中的澳洲胎牛血清培养基成分按质量计为:澳洲胎牛血清10%,非必需氨基酸1%,补充rpmi1640培养基178ml;步骤(3)中的裂解液按质量计为:tris·hclph7.510mm;氯化钠10mm;氯化镁3mm;聚乙氧基乙醇辛基苯基-聚乙烯二醇0.1%;补充双蒸水9.75ml。3.试验结果与数据分析采用esatac(wei,zhangetal.2018)报道的方法与软件对数据进行检测与分析,分析结果如下所述:(1)分析插入片段大小展示出高分辨率的分布图hica-seq插入片段大小集中在180bp,说明经过合适的片段选择会获得高分辨率的数据结果,结果见图3。(2)基因转录起始位点(tss)富集分析dna缠绕在核小体上,每个核小体占据dna的180~247bp长度,连接两个核小体之间的自由dna长度小于100bp。由于活性基因的tss区域处于开放状态,即染色质可及性,因此该区域附近reads最多,即可富集(enrichment)。本发明的实施结果表明,说明本发明对于开放染色质可及性的获得具有高度特异性,对跨核小体dna可进行有效的富集,结果见图4。(3)hica-seq数据的结果呈现对得到的hica-seq数据进行分析,每个hica-seq与chip-seq的峰形匹配,且具有良好的生物学意义上的对应关系,结果见图5。参考文献:1.buenrostro,j.d.,p.g.giresi,l.c.zaba,h.y.changandw.j.greenleaf(2013)."transpositionofnativechromatinforfastandsensitiveepigenomicprofilingofopenchromatin,dna-bindingproteinsandnucleosomeposition."natmethods10(12):1213-1218.2.mariatsompana1andmichaeljbuck1(2014)."<1chromatinaccessibility,awindowintothegenome.pdf>."epigenetics&chromatin.3.lu,f.,y.liu,a.inoue,t.suzuki,k.zhaoandy.zhang(2016)."establishingchromatinregulatorylandscapeduringmousepreimplantationdevelopment."cell165(6):1375-1388.4.buenrostro,j.d.,b.wu,h.y.changandw.j.greenleaf(2015)."atac-seq:amethodforassayingchromatinaccessibilitygenome-wide."currprotocmolbiol109:212921-29.5.wu,j.,j.xu,b.liu,g.yao,p.wang,z.lin,b.huang,x.wang,t.li,s.shi,n.zhang,f.duan,j.ming,x.zhang,w.niu,w.song,h.jin,y.guo,s.dai,l.hu,l.fang,q.wang,y.li,w.li,j.na,w.xieandy.sun(2018)."chromatinanalysisinhumanearlydevelopmentrevealsepigenetictransitionduringzga."nature557(7704):256-260.6.wei,z.,w.zhang,h.fang,y.liandx.wang(2018)."esatac:aneasy-to-usesystematicpipelineforatac-seqdataanalysis."bioinformatics34(15):2664-2665。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1