一种经优化的高温酸性海藻糖酶TreMT1及其编码基因与应用的制作方法
本发明涉及基因工程领域,具体涉及一种经优化的高温酸性海藻糖酶tremt1及其编码基因与应用。
背景技术:
海藻糖trehalose(α-d-吡喃葡萄糖基-α-d-吡喃葡萄糖苷)是由两个葡萄糖分子以1,1-糖苷键构成的非还原性二糖,广泛存在于包括细菌、真菌、原生动物、植物、哺乳动物在内的有机体中。
海藻糖酶(trehalase)是一种葡萄糖苷水解酶,在酶分类上归属于水解酶,分类编号为ec3.2.1.28,可以特异性将一分子的海藻糖水解生成两分子的葡萄糖,广泛存在于细菌、霉菌、植物和动物中。根据海藻糖酶发挥酶活性的ph环境不同,可以将海藻糖酶分为酸性海藻糖酶和中性海藻糖酶,酸性海藻糖酶的最适ph大约是4.5-5.5,一般分泌到胞外发挥作用,将海藻糖水解为葡萄糖供细胞利用,中性海藻糖酶的最适ph大约是7.0,一般分布在胞内,主要是分解胞内的海藻糖供生命所需。现在研究得最多的酸性海藻糖酶来源于酵母和丝状真菌,酵母中的酸性海藻糖酶定位于液泡,在丝状真菌中则定位于细胞壁。
目前关于海藻糖酶的表达研究较少,研究主要集中在重组蛋白表达和酶学性质的测定方面,已报道的海藻糖酶重组表达宿主主要为大肠杆菌和毕赤酵母,而且重组表达的海藻糖酶活力和蛋白产量都很低。yingchunliu等人利用毕赤酵母作为表达宿主,表达来源于绿僵菌的一个海藻糖酶,发酵上清液酶活力为5.35u/mg蛋白,从1l发酵上清液中仅可以回收到0.2mg海藻糖酶(małgorzatadmitryjuk,krystynazółtowska.purificationandcharacterizationofacidtrehalasefrommuscleofascarissuum(nematoda)[j].comparativebiochemistry&physiologypartb,2003,136(1):61-69.);jin-halee等人在毕赤酵母中表达来源于蜜蜂的海藻糖酶,发酵液的海藻糖酶活力仅为5.1u/ml(leejh,saitos,morih,etal.molecularcloningofcdnafortrehalasefromtheeuropeanhoneybee,apismelliferal.anditsheterologousexpressioninpichiapastoris[j].journaloftheagriculturalchemicalsocietyofjapan,2007,71(9):10.);fabianam.dealmeida等人在大肠杆菌里表达中性海藻糖酶,破碎后上清液酶活力为0.15u/ml(almeidafmd,boninibm,betond,etal.heterologousexpressioninescherichiacoliofneurosporacrassaneutraltrehalaseasanactiveenzyme.[j].proteinexpression&purification,2009,65(2):185-189.)。酶学性质方面,鉴定表达出来的海藻糖酶分子量范围较广,在60kda-170kda,主要为中性海藻糖酶,最适ph为7.0左右,酸性海藻糖酶研究较少,最适的作用温度在30-40℃。
现如今的酒精发酵主要是以淀粉质原料为主,在玉米酒精工厂中未发酵残糖中二糖的主要成分通常是海藻糖,如果在发酵前用海藻糖酶将海藻糖水解为葡萄糖,就可以有更多的葡萄糖用于生产更多的乙醇。在欧洲,玉米酒精工厂的平均产能为2.2亿升(约合17.6万吨乙醇),通过更好地转化降低残糖比如海藻糖,可为酒精工厂,仅在欧洲,可增产价值高至85万欧元的乙醇。
同时也有文献报道,在谷氨酸发酵过程中,在发酵的后期添加海藻糖酶,可以提高糖源的转化效率以及提高最后的谷氨酸产量(韩隽,姬慧军.海藻糖酶在谷氨酸发酵中的应用[j].广州化工,2017,45(7):73-74.)。
因此,海藻糖酶可以作为工业上淀粉质原料发酵生产过程的一个关键添加酶,可以与传统的糖化酶形成一个复配酶,提高淀粉质原料的利用效率,提高能源利用率,从而达到降低生产成本的目的。而目前的文献报道中,关于海藻糖酶的研究,主要集中在酶学性质的测定方面,而且重组海藻糖酶的表达水平都很低,未见有专门的海藻糖酶高效表达文献报道。
工业上的淀粉液化主要在高温条件下进行,液化结束后冷却到一定温度后才能进行下一步的操作,所以工业上迫切需要筛选出在高温条件下能发挥酶活性,将液化后产生的大量二糖水解成单糖的海藻糖酶,从而降低淀粉液化后的冷却耗能和提高酵母酒精发酵过程中淀粉质原料的利用率,减少资源浪费,提高生物能源的利用效率。故筛选出适合于工业生产条件的高温海藻糖酶,而且实现海藻糖酶的高效表达甚至工业化规模表达具有一定的现实意义和应用价值。
技术实现要素:
为了克服现有技术的不足,本发明的目的是提供一种经优化的高温酸性海藻糖酶tremt1及其编码基因与应用。
本发明提供一种经优化的高温酸性海藻糖酶氨基酸序列、经优化的编码上述高温酸性海藻糖酶的基因、包含上述基因的重组表达载体、包含上述基因的重组菌株以及制备上述高温酸性海藻糖酶的基因工程方法。
本发明的目的通过如下技术方案实现。
本发明的高温酸性海藻糖酶原始基因来源于嗜热毁丝菌(myceliophthorathermophila)。
本发明提供的一种经优化的高温酸性海藻糖酶tremt1,氨基酸序列(trehalase-prt(经优化))如seqidno.1所示。
其中,经优化的高温酸性海藻糖酶包括692个氨基酸,n端含信号肽序列。
本发明的经优化的海藻糖酶tremt1的最适ph为5.5,在ph4.0-6.0稳定性好,在ph4.0-6.0范围内反应,酶活力为最高酶活力的80%以上;最适温度为60℃,在55-65℃范围内反应,仍保持最高酶活力的80%以上。
本发明所述的一种经优化的高温酸性海藻糖酶tremt1,编码基因的核苷酸序列(trehalase-dna(经优化))如seqidno.2所示,该编码基因可以在黑曲霉中高效表达。
本发明中的经优化的高温酸性海藻糖酶tremt1结构基因全长为2076bp,n端含信号肽编码序列。
本发明提供的一种重组表达载体,插入了权利要求2中如seqidno.2所示的核苷酸序列。
进一步地,所述的重组表达载体包含有pmd20-t载体、黑曲霉中性淀粉酶启动子pamya、黑曲霉tef终止子、构巢曲霉尿嘧啶(u)生物合成基因pyrg、黑曲霉中性淀粉酶编码基因最后1000bp序列(作为同源臂)(如seqidno.5所示)以及所述经优化的高温酸性海藻糖酶tremt1的编码基因,使所述的高温酸性海藻糖酶编码基因核苷酸序列位于诱导型启动子pamya的下游并受其调控,而且能通过同源重组的方式整合至黑曲霉的中性淀粉酶位点。该重组载体为pmd20-pamya-tremt1-ttef-pyrg-amya,将本发明的海藻糖酶基因优化序列插入到表达载体合适的限制性酶切位点之间,使其核苷酸序列与其它表达元件相连接,得到重组表达载体pmd20-pamya-tremt1-ttef-pyrg-amya。
本发明提供的一种转基因细胞系,含有上述的重组表达载体。
进一步地,所述的转基因细胞系,宿主细胞为黑曲霉菌;所述转基因细胞系为黑曲霉dmt-17(aspergillusnigerdmt-17),于2018年12月17日保藏于中国典型培养物保藏中心,保藏编号为cctccno:m2018902。
进一步地,所述经优化的高温酸性海藻糖酶tremt1的编码基因、所述重组表达载体,在制备所述经优化的高温酸性海藻糖酶tremt1的应用。
进一步地,所述制备所述经优化的高温酸性海藻糖酶tremt1的应用,包括以下步骤:
(1)构建重组表达载体;
(2)用步骤(1)构建的重组表达载体转化宿主细胞,得到重组菌株即转基因细胞系;
(3)培养重组菌株,用发酵培养基诱导所述经优化的高温酸性海藻糖酶tremt1的表达;
(4)回收并纯化所述经优化的高温酸性海藻糖酶tremt1。
所述转基因细胞系,在制备权利要求1所述经优化的高温酸性海藻糖酶tremt1的应用。
进一步地,本发明所用的表达宿主为黑曲霉(aspergillusniger)。本发明构建得到的重组海藻糖酶基因的黑曲霉表达菌株(黑曲霉dmt-17)已送达中国典型培养物保藏中心(cctcc)做专利菌种保藏,菌种保藏号为:cctccno:m2018902;保藏的日期为2018年12月17日;保藏地址:中国武汉武汉大学。
本发明构建的通用表达载体含有黑曲霉中性淀粉酶的同源臂,将海藻糖酶基因插入到一个构建好的通用表达载体中,同时使用黑曲霉中性淀粉酶启动子,使其可以通过同源重组的方式整合至黑曲霉宿主的中性淀粉酶位点,得到重组菌株aspergillusniger-tremt1。
进一步地,步骤(2)中所述的重组菌株为转化了所述经优化的高温酸性海藻糖酶tremt1编码基因的菌株。
本发明所述的经优化的高温酸性海藻糖酶tremt1可应用于现有玉米淀粉发酵酒精和谷氨酸发酵原料的预处理。
与现有技术相比,本发明有以下优点和有益效果:
(1)本发明得到的重组菌株aspergillusniger-tremt1在100ml发酵培养基中进行摇瓶发酵,第七天的发酵上清液中海藻糖酶活力达到15647.69u/ml,经过凝胶过滤层析纯化,从1ml发酵上清液中可以纯化得到3mg以上的重组海藻糖酶,仅从100ml的摇瓶发酵液就可以回收300mg以上重组海藻糖酶,纯化的重组海藻糖酶比酶活达到5156.28u/mg,远高于现有文献报道的重组海藻糖酶表达纯化水平;
(2)本发明提供的海藻糖酶稳定性好,在高温酸性条件下也具有活性,能将二糖水解成单糖;
(3)本发明提供的海藻糖酶可对现有玉米淀粉发酵酒精和谷氨酸发酵原料的预处理问题提供了一个具有经济价值和减少资源浪费的解决方案,玉米淀粉发酵酒精的淀粉预处理主要包括高温液化,但是现有的高温液化和酶法液化结束后,会产生很多二糖,而这些二糖中的主要成分是海藻糖,酵母发酵过程中也会产生许多海藻糖,而此海藻糖不能为酵母所利用,如果添加海藻糖酶,将产生的海藻糖水解成单分子的葡萄糖,为酵母所利用,可以提高最后乙醇的产量,提高了玉米淀粉的利用率,降低淀粉液化后的冷却耗能和提高淀粉质原料的利用率,减少资源浪费,提高生物能源的利用效率。对实现海藻糖酶的高效表达甚至工业化规模表达具有一定的现实意义和应用价值。
附图说明
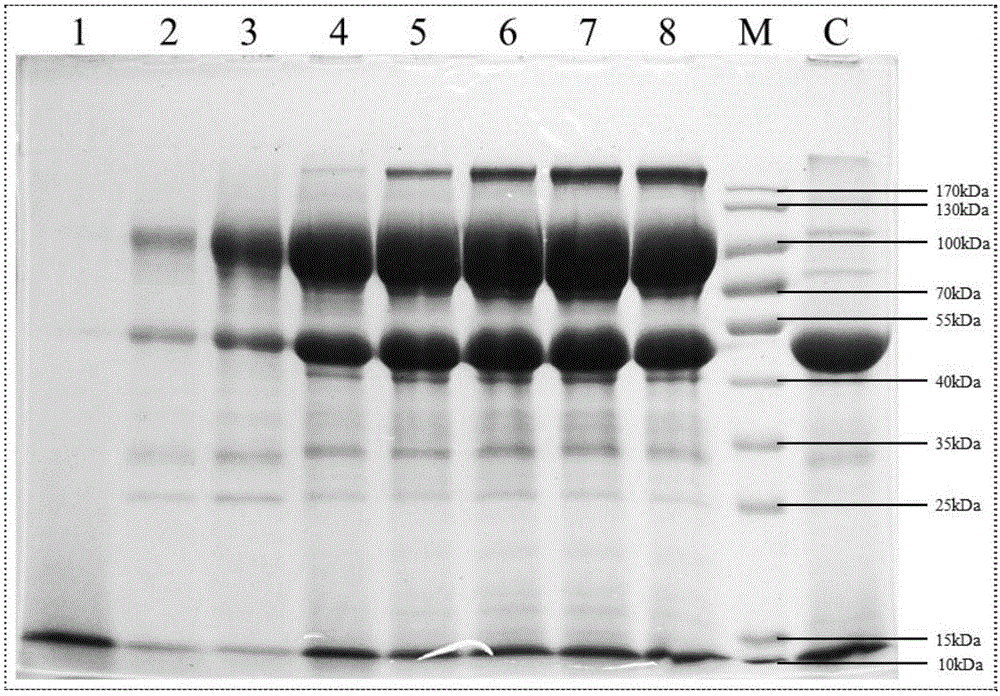
图1是发酵上清液的经优化的高温酸性海藻糖酶sds-page结果图,其中泳道1为第一天发酵上清液,泳道2为第二天发酵上清液,泳道3为第三天发酵上清液,泳道4为第四天发酵上清液,泳道5为第五天发酵上清液,泳道6为第六天发酵上清液,泳道7为第七天发酵上清液,泳道8为第八天发酵上清液,泳道m为thermoscientificproteinladdersno.26616,泳道c为宿主第六天发酵液上清液;
图2为发酵上清液的海藻糖酶tremt1活力测定结果图;
图3为经优化的高温酸性海藻糖酶tremt1的最适温度;
图4为经优化的高温酸性海藻糖酶tremt1的最适ph;
图5为经优化的高温酸性海藻糖酶tremt1凝胶过滤层析纯化洗脱峰图;
图6为经优化的高温酸性海藻糖酶tremt1凝胶过滤层析纯化洗脱峰sds-page结果图,其中泳道1为第七天发酵上清液,泳道2为第第一洗脱峰洗脱液,泳道m为thermoscientificproteinladdersno.26616。
具体实施方式
下面结合实施例以及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
已报道的海藻糖酶表达基因有很大部分来源于酵母、里氏木霉和其它细菌,因为我们的表达宿主为黑曲霉,所以我们希望能从丝状真菌属里面筛选出一个合适的海藻糖酶基因,故我们将里氏木霉的海藻糖酶基因通过数据库比对的方法寻找同源性高的序列。由于淀粉液化都是在较高温度下进行的,液化结束冷却后才能进行下一步的处理,于是希望能从嗜热菌里面筛选出耐高温的或者在高温条件下还能发挥酶活性的海藻糖酶基因,最后从嗜热毁丝菌(myceliophthorathermophila)中筛选出一个海藻糖酶基因,该基因通过序列比对的方式,发现该基因的氨基酸序列与目前被验证的海藻糖酶氨基酸序列具有相似的结构序列,预测该基因的产物具有海藻糖酶活性,但是未有实验证明该基因的产物具有海藻糖酶活性。
根据数据库中的目的基因以及氨基酸序列,对目的基因已经氨基酸序列进行优化,得到的优化后氨基酸序列如seqidno.1所示。根据优化后的氨基酸序列,对其核苷酸序列进行优化,使其适合于在黑曲霉(aspergillusniger)中高效表达,体外合成优化的核苷酸序列。设计引物以合成的目的基因核苷酸序列为模板扩增目的基因片段。将扩增到的目的基因片段、黑曲霉amya启动子片段和线性化的通用表达载体进行连接,构建包含有目的基因片段的表达载体。转化黑曲霉原生质体,并对转化子进行pcr上下游鉴定和表达框扩增,挑选阳性克隆,接种液体cd培养,最后接种至发酵培养基中发酵,每天取样测定上清液中的海藻糖酶活力,sds-page检测发酵上清液中重组海藻糖酶的表达量,从而确定该基因的产物具有海藻糖酶活性,该基因确实是一个海藻糖酶编码基因,而且优化后的海藻糖酶在黑曲霉中可以高效地表达。
需指出的是,以下若有未特别详细说明之过程或参数,均是本领域技术人员可参照现有技术理解或实现的。
实施例1、通用表达载体的构建
以黑曲霉基因组为模板,做pcr扩增(primestarpremixhs(购自takara公司)),使用引物ttef-fw(如seqidno.8所示)和引物ttef-rev(如seqidno.9所示)扩增tef终止子(如seqidno.3所示),使用引物amya-fw(如seqidno.12所示)和引物amya-rev(如seqidno.13所示)扩增中性淀粉酶amya编码基因的最后1000bp(如seqidno.5所示),以构巢曲霉基因组为模板使用引物pyrg-fw(如seqidno.10所示)和引物pyrg-rev(如seqidno.11所示)扩增pyrg标记(如seqidno.4所示),得到的pcr产物分别命名为ttef、amya、pyrg(分别如seqidno.3、如seqidno.5、如seqidno.4所示),使用nebuilderhifidnaassemblycloningkit试剂盒,将ttef、pyrg、amya、t载体pmd20-t(如seqidno.6所示)四个片段连接成为一个环状质粒pmd20-ttef-pyrg-amya(具体参考试剂盒说明书)。连接产物转化大肠杆菌match1t1(购自takara公司)感受态,37℃培养12h后挑取转化子于液体lb+amp(终浓度100μg/ml)培养基,37℃条件下、摇床转速为200rpm培养12h,做菌液电泳,初步筛选出阳性转化子。菌液电泳筛选到的阳性转化子提质粒、酶切验证,挑选最后质粒大小正确而且酶切验证正确的阳性转化子3个送去测序公司测序。拿到测序结果后与模板序列做比对,挑选测序完全正确的转化子于液体lb+amp(终浓度100μg/ml)培养基,37℃条件下、摇床转速为200rpm培养12h,提质粒,用限制性内切酶ecorv酶切4h,同时用去磷酸化酶fastap做去磷酸化处理,防止载体自连,最后得到线性化的通用表达载体。
实施例2、含目的基因表达载体构建
分别以合成的tremt1基因优化核苷酸序列、黑曲霉基因组为模板,以引物tre-fw(如seqidno.16所示)和引物tre-rev(如seqidno.17所示)扩增得到tremt1基因序列(如seqidno.2所示),以引物pamya-fw(如seqidno.14所示)和引物pamya-rev(如seqidno.15所示)扩增得到黑曲霉中性淀粉酶启动子pamya序列(如seqidno.7所示)。将两个pcr扩增得到的pcr片段与线性化通用表达载体做fusionpcr连接。连接产物转化大肠杆菌match1t1(购自takara公司)感受态,37℃培养12h以后挑取转化子于液体lb+amp(终浓度100μg/ml)培养基,37℃条件下、摇床转速为200rpm培养12h,做菌液电泳,初步筛选出阳性转化子。菌液电泳筛选到的阳性转化子提质粒、酶切验证,挑选最后质粒大小正确而且酶切验证正确的阳性转化子3个送去测序公司测序。拿到测序结果后与模板序列做比对,挑选测序完全正确的转化子,接种100ml液体lb+amp(终浓度100μg/ml)培养基培养,提质粒,得到表达载体pmd20-pamya-tremt1-ttef-pyrg-amya。
实施例3、表达载体pmd20-pamya-tremt1-ttef-pyrg-amya质粒在黑曲霉中的转化
按照(gomik,iimuray,haras.integrativetransformationofaspergillusoryzaewithaplasmidcontainingtheaspergillusnidulansargbgene[j].agriculturalandbiologicalchemistry,1987,51(9):2549-2555.)中提供的步骤制备宿主菌黑曲霉(δpyrg)的原生质体,将上述得到的得到表达载体pmd20-pamya-tremt1-ttef-pyrg-amya质粒转化入原生质体中,涂布高渗cd培养基(包含1m蔗糖,0.3%(w/v)nano3,0.2%(w/v)kcl,0.05%(w/v)mgso4.7h2o,0.1%(w/v)k2hpo4.3h2o,0.001%(w/v)feso4.7h2o,2%(w/v)琼脂粉,ph5.5,w/v的单位为g/ml),放入30℃培养箱,5d后观察转化子生长情况。
实施例4、黑曲霉转化子目的基因pcr鉴定
待上述转化子在高渗cd板上长出后,挑至新的普通cd固态板(包含2%(w/v)葡萄糖,0.3%(w/v)nano3,0.2%(w/v)kcl,0.05%(w/v)mgso4.7h2o,0.1%(w/v)k2hpo4.3h2o,0.001%(w/v)feso4.7h2o,2%(w/v)琼脂粉,ph5.5,w/v的单位为g/ml),放入30℃培养箱培养5d至菌落长大,待菌落长大后,挑菌落的一小部分至淀粉固态板上,放入30℃培养箱中培养,待淀粉板上的菌落长大后,将菌体刮下至1.5mlep管中,加入溶菌缓冲液,用组织研磨器研磨提取基因组。
设计扩增目的基因片段的引物,表达框鉴定引物要求能扩出目的基因的表达框,所以表达框鉴定引物的正向引物tremt1-fw(如seqidno.22所示)设在pamya(如seqidno.7所示)上,反向引物tremt1-rev(如seqidno.23所示)设在ttef上;上游鉴定引物的正向引物up-fw(如seqidno.18所示)设在pamya上,反向引物up-rev(如seqidno.19所示)设在目的基因片段上,下游鉴定引物的正向引物down-fw(如seqidno.20所示)设在目的基因片段上,反向引物down-rev(如seqidno.21所示)设在标记pyrg上。
以转化子的基因组为模板,用引物up-fw和up-rev检测目的基因表达框的上游片段,用引物down-fw和down-rev检测目的基因表达框的下游片段,用引物tremt1-fw和引物tremt1-rev检测目的基因的表达框,同时以构建的表达载体质粒为模板作为阳性对照,以宿主基因组为模板作为阴性对照,筛选上游、下游和表达框鉴定都正确的转化子。
实施例5、黑曲霉阳性转化子接种液体cd和发酵培养基
pcr上游鉴定、下游鉴定和表达框鉴定都正确的转化子从固态cd板上刮下,加无菌水用低速组织研磨器研磨后,接种至液体cd(包含2%(w/v)葡萄糖,0.3%(w/v)nano3,0.2%(w/v)kcl,0.05%(w/v)mgso4.7h2o,0.1%(w/v)k2hpo4.3h2o,0.001%(w/v)feso4.7h2o,0.05%(w/v)琼脂粉,ph5.5,w/v的单位为g/ml)中,30℃培养箱中静置培养5天后,接种至发酵培养基(包含5%淀粉,3%玉米浆,2%豆粕粉,此处百分号表示为质量百分含量),30℃条件,摇床转速为250rpm条件发酵,每隔24h取一次样,测定每天发酵上清液的海藻糖酶活力,通过sds-page检测上清液中的重组海藻糖酶含量。
实施例6、发酵上清液海藻糖酶活性测定
海藻糖酶可以将一分子的非还原性二糖海藻糖水解生成两分子
的还原性单糖葡萄糖,通过dns法检测最后产生的还原性单糖葡萄糖的量,来测定发酵上清液中的海藻糖酶活力。
发酵上清液经凝胶过滤层析分离纯化所述经优化的高温酸性海藻糖酶tremt1。
发酵上清液经0.22μm滤膜过滤后,上gehealthcarehiprep16/60sephacryls-200highresolution凝胶过滤层析柱,用50mmpbs(含0.15mnacl)洗脱,流速为0.5ml/min,收集各个洗脱峰的洗脱液,dns法测定各个洗脱峰的海藻糖酶活力,sds-page检测各个洗脱峰溶液的蛋白条带。
从图1发酵上清液sds-page结果图和图2发酵上清液的海藻糖酶活力测定结果图可以看出,重组菌株aspergillusniger-tremt1在100ml发酵培养基中进行摇瓶发酵,第七天的发酵上清液中海藻糖酶活力达到15647.69u/ml,目的蛋白条带大小为90kda左右,在第五天之后蛋白条带就很粗,目的蛋白浓度高。
结合图5的发酵上清液凝胶过滤层析纯化洗脱峰图和图6凝胶过滤层析纯化洗脱峰sds-page结果图可以看出,目的重组海藻糖酶为凝胶过滤层析的第一洗脱峰,经过纯化的重组海藻糖酶基本去除了其它的杂蛋白,经蛋白浓度测定和洗脱峰海藻糖酶活力测定,纯化的重组海藻糖酶比酶活达到5156.28u/mg,从1ml发酵上清液中可以纯化得到3mg以上目的重组海藻糖酶,远高于现有文献报道的重组海藻糖酶表达纯化水平。
从图3的经优化的高温酸性海藻糖酶tremt1的最适温度结果图、图4的经优化的高温酸性海藻糖酶tremt1的最适ph结果可以看出,本发明的经优化的海藻糖酶tremt1的最适ph为5.5,在ph4.0-6.0稳定性好,在ph4.0-6.0范围内反应,酶活力为最高酶活力的80%以上;最适温度为60℃,在55-65℃范围内反应,仍保持最高酶活力的80%以上。
以上实施例仅为本发明较优的实施方式,仅用于解释本发明,而非限制本发明,本领域技术人员在未脱离本发明精神实质下所作的改变、替换、修饰等均应属于本发明的保护范围。
序列表
<110>华南理工大学
<120>一种经优化的高温酸性海藻糖酶tremt1及其编码基因与应用
<160>23
<170>siposequencelisting1.0
<210>1
<211>692
<212>prt
<213>嗜热毁丝菌(myceliophthora_thermophila)
<400>1
metserpheargserleuleualaleuserglyleuvalcysthrgly
151015
leualaleutyrileasnglyservalthralaprocysaspserpro
202530
iletyrcyshisglygluleuleulysglyvalgluleualahispro
354045
phevalaspserlysthrphevalaspmetprothrleulysproval
505560
aspgluvalleualaalapheserlysleuargglnproleuserasn
65707580
asnsergluleuasnasnpheleualaglutyrphealaproalagly
859095
hisgluleuglugluvalprolysglygluleuglnileaspprolys
100105110
pheleuasnlysleugluaspargthrilelysgluphevalserlys
115120125
valileaspiletrpproaspleuthrargargtyralaglyprogly
130135140
aspcysserglycysalaasnserpheileprovalasnargthrphe
145150155160
valvalalaglyglyargphearggluprotyrtyrtrpaspsertyr
165170175
trpileleugluglyleuleuargthrglyglyalaphethrglnile
180185190
serlysasnileilegluasnpheleuasppheileaspthrilegly
195200205
pheileproasnglyalaargiletyrtyrleuasnargserglnpro
210215220
proleuleuthrargmetvallyssertyrvalasptyrthrasnasp
225230235240
thrserileleugluargalaleuproleuleuilelysgluhisasp
245250255
phephethrasnasnargservalservalthralaserasnglylys
260265270
thrtyrthrleuhisargtyrhisvalgluasnasnglnproargpro
275280285
glusertyrarggluasptyrilethralaasnasnglysertyrtyr
290295300
alaalaserglyileiletyrprovallysthrproleuasngluthr
305310315320
glulysalavalleutyrserasnleualaserglyalaglusergly
325330335
trpasptyrthralaargtrpleuargvalproaspaspalaalaarg
340345350
aspvaltyrpheproleuargserleuasnvalargglumetvalpro
355360365
valaspleuasnserileleutyrgluasngluvalileilealaglu
370375380
tyrleuglulysalaglyasnserserglualalysargphealaser
385390395400
alaalalysglnargserglualamettyrasnleumettrpasnala
405410415
thrhistrpsertyrpheasptyrasnleuthrserasnalaglnasn
420425430
ilephevalproalaaspgluaspthralaserpheaspargtyrala
435440445
alaproproglyglnglnvalleuphehisvalalaglnleutyrpro
450455460
phetrpthrglyalaalaproalahisleulysserasnproleuala
465470475480
valglnlysalatyralaargvalserargargleuaspthrlyslys
485490495
glyalailealaalathrasntyrargthrglyglnglntrpaspgln
500505510
proasnvaltrpproproleuglnhisvalleumetglnglyleuleu
515520525
asnthrproalathrpheglygluseraspproalatyrglnglyval
530535540
glnlysleualaleuargleualaglnargtyrleuaspserthrphe
545550555560
cysthrtrptyralathrglyglyserthrserasppheproglnleu
565570575
glnglyvalserproaspalathrglyilemetpheglulystyrala
580585590
aspseralathrasnvalalaglyglyglyglyglutyrgluvalval
595600605
gluglypheglytrpthrasnglyvalleuiletrpalaalaaspval
610615620
pheglyasnlysleulysargproaspcysglyasnilethralaala
625630635640
histhrhisserglualalysargserleuglyaspglyglyleuala
645650655
argargalavalgluleuaspprotrpaspalaalatrpthrlysmet
660665670
pheglyargserlysleuargargargglualagluaspvalarglys
675680685
argtrpserser
690
<210>2
<211>2079
<212>dna
<213>黑曲霉(aspergillusniger)
<400>2
atgtcgttccgatctctactcgccctgagcggcctcgtctgcacagggttggcactgtac60
atcaacggtagcgtcaccgctccttgcgactcccccatctactgccacggcgagctgctc120
aagggtgtggaactcgcccatcctttcgtcgacagcaagaccttcgtggatatgcctacc180
ctcaaaccggtcgatgaggtgctggctgccttctccaagctccgtcagccgctgtccaac240
aacagcgagctcaacaacttcctggccgaatacttcgctcccgccggccatgagctcgag300
gaagtccctaaaggtgaactgcaaatcgacccgaagttcctcaacaaactggaggatcgg360
accatcaaggaattcgtcagcaaagtgatcgacatctggcctgatctcacccgtcgttac420
gctggccccggtgactgcagcggttgcgccaactccttcatccccgtgaaccggaccttc480
gtcgtggctggtggtcgtttccgtgagccttactactgggatagctactggatcctggaa540
ggtctgctccgtaccggtggtgctttcacccagatctccaagaacatcatcgagaacttc600
ctcgacttcatcgataccatcggcttcatccccaacggtgcccgcatctactacctgaac660
cgttcccaaccccctctgctcacccgcatggtcaaaagctacgtggactacaccaacgat720
acctccatcctcgagcgtgctctgcctctgctcatcaaggaacacgacttcttcaccaac780
aaccgttccgtcagcgtgaccgctagcaacggcaaaacctacaccctgcaccgctaccat840
gtcgaaaacaaccagccccggcctgagtcctaccgcgaagactacatcaccgccaacaac900
ggcagctactacgctgcctccggtatcatctaccccgtcaagacccctctcaacgagacc960
gaaaaagctgtgctctactccaacctggctagcggtgctgagtccggttgggactacacc1020
gctcgttggctgcgtgtccctgacgatgctgctcgtgatgtgtacttcccgctccgtagc1080
ctgaacgtccgggaaatggtcccggtggacctcaactccatcctgtacgagaacgaagtg1140
atcatcgctgagtacctcgaaaaagccggcaactccagcgaggctaagcggttcgccagc1200
gctgccaaacagcgctccgaagctatgtacaacctcatgtggaacgccacccactggagc1260
tacttcgactacaacctgacctccaacgctcaaaacatcttcgtccccgctgacgaggat1320
accgccagcttcgatcgttacgctgctcctcctggtcagcaagtcctcttccacgtggcc1380
cagctgtaccctttctggaccggtgctgctcctgctcatctcaagagcaaccctctggcc1440
gtccaaaaagcttacgctcgtgtgtcccgtcgtctggacaccaagaaaggcgccatcgct1500
gccaccaactaccgtaccggtcagcaatgggatcagcccaacgtctggcctccgctccag1560
catgtgctgatgcaaggcctgctcaacacccccgctaccttcggcgaaagcgaccctgcc1620
taccagggtgtccaaaagctggctctccgcctggcccaacgttacctcgattccaccttc1680
tgcacctggtacgctaccggtggttccaccagcgacttccctcagctgcaaggtgtcagc1740
cctgatgctaccggtatcatgttcgagaaatacgctgactccgctaccaacgtggctggt1800
ggtggtggcgagtacgaagtcgtggaaggcttcggttggaccaacggcgtcctcatctgg1860
gctgccgacgtgttcggcaacaagctgaaacgtcctgattgcggtaacatcaccgctgcc1920
cacacccatagcgaggctaagcgttccctcggtgacggtggtctggctcgtcgtgctgtc1980
gaactcgacccttgggatgctgcctggaccaagatgttcggtcgtagcaaactgcgtcgt2040
cgtgaggctgaagatgtgcgtaaacgctggtccagctag2079
<210>3
<211>489
<212>dna
<213>黑曲霉(aspergillusniger)
<400>3
gcggacattcgatttatgccgttatgacttccttaaaaaagcctttacgaatgaaagaaa60
tggaattagacttgttatgtagttgattctacaatggattatgattcctgaacttcaaat120
ccgctgttcattattaatctcagctcttcccgtaaagccaatgttgaaactattcgtaaa180
tgtacctcgttttgcgtgtaccttgcttatcacgtgatattacatgacctggacagagtt240
ctgcgcgaaagtcataacgtaaatcccgggcggtaggtgcgtcccgggcggaaggtagtt300
ttctcgtccaccccaacgcgtttatcaacctcaactttcaacaaccatcatgccaccaaa360
agcgcgtaaaacaaagcgagatttgattgagcaagagggcaggatccaatgcgcgattca420
agacattaaaaatggaaaatttcaaaaaattgcgcccgcagcgcgtgcatacaaaattca480
tcccaatac489
<210>4
<211>1398
<212>dna
<213>构巢曲霉(aspergillusnidulans)
<400>4
gcaacttcctcgagaacgcgccgcagacaatgctctctatcctggtggcaggcgtcaagt60
acccagaggcagcagcgggcttaggagcggcctgggttgttctccgcaccctctacatgc120
tgggctatatttatagcgacaagccgaacggcaccggcaggtacaatggttcgctgtact180
tgcttgcgcaagcgggtctttggggattgagcgcatttggtgttgcaaaggatttgatgt240
aaatgtagtcgacatcttagcacagaggggagagttgataaaatgtggtctgtttgaatg300
atagtcgggttcgtgacctatattcgtgatagtggagataggtctgcgcctatcttatcg360
ggccggagcaaaaattccaccgcagcggggtgagttttcgttatacagccatcccacttc420
cagcttcaaattgtcagtttaatccagcccaattcaatcattggagaaccgccatcatgt480
cttcgaagtcccacctcccctacgcaattcgcgcaaccaaccatcccaaccctttaacat540
ctaaactcttctccatcgccgaggagaagaaaaccaacgtcaccgtctccgcagacgtta600
ctacttccgccgagctcctcgatcttgctgaccgtacatcctgcaccaatgcccctccag660
gataacaaatagctgatgcgtagtgagtacaggcctaggcccctatatcgcagttctgaa720
aacccacatcgacatcctcaccgatctcaccccgtcgaccctttcctcgctccaatccct780
cgcgacaaagcacaacttcctcatctttgaggaccgcaagttcatcgacatcggcaacac840
cgtgcaaaagcagtaccacggtggcgctctccgcatctccgaatgggcacacatcatcaa900
ctgcgccatcctgccgggcgaagggatcgtcgaggccctcgcacagacaaccaagtctcc960
tgactttaaagacgcgaatcaacgaggtctcctgattcttgccgagatgacgagtaaggg1020
atctcttgcgacaggggagtacacggcacgctcggttgagtacgcgcggaagtataaggg1080
gtttgtgatgggattcgtgagtacaagggcgttgagtgaggtgctgcccgaacagaaaga1140
ggagagcgaggattttgtcgtctttacgactggggtgaatctgtcggataagggggataa1200
gctggggcagcagtatcagacacctgggtcggcggttgggcgaggtgcggactttatcat1260
tgcgggtaggggcatctataaggcggacgatccagtcgaggcggttcagaggtaccggga1320
ggaaggctggaaagcttacgagaaaagagttggactttgagtgtgagtggaaatgtgtaa1380
cggtattgactaaaaggg1398
<210>5
<211>1000
<212>dna
<213>黑曲霉(aspergillusniger)
<400>5
acaaagccgcaggcgtgtactgtatcggcgaggtgctcgacggtgatccggcctacactt60
gtccctaccagaacgtcatggacggcgtactgaactatcccatgtatggttcctccaacc120
atgagccttcttgcaagtctcatctcctaacgaaacggctaaaaccagttactatccact180
cctcaacgccttcaagtcaacctccggcagcatggacgacctctacaacatgatcaacac240
cgtcaaatccgactgtccagactcaacactcctgggcacattcgtcgagaaccacgacaa300
cccacggttcgcttcgtaagtcttcccttttattttccgttcccaatttccacacagaac360
cccacctaacaagagcaaagttacaccaacgacatagccctcgccaagaacgtcgcagca420
ttcatcatcctcaacgacggaatccccatcatctacgccggccaagaacagcactacgcc480
ggcggaaacgaccccgcgaaccgcgaagcaacctggctctcgggctacccgaccgacagc540
gagctgtacaagttaattgcctccgcgaacgcaatccggaactatgccattagcaaagat600
acaggattcgtgacctacaaggtaagcacaacctctaagcataccctaatggcctatctt660
cagagtatctgacacaagagactaatcactggcaatacagaactggcccatctacaaaga720
cgacacaacgatcgccatgcgcaagggcacagatgggtcgcagatcgtgactatcttgtc780
caacaagggtgcttcgggtgattcgtataccctctccttgagtggtgcgggttacacagc840
cggccagcaattgacggaggtcattggctgcacgaccgtgacggttggttcggatggaaa900
tgtgcctgttcctatggcaggtgggctacctagggtattgtatccgactgagaagttggc960
aggtagcaagatctgtagtagctcgtgaagggtggagagt1000
<210>6
<211>2737
<212>dna
<213>未知(人工序列)
<400>6
atcggatccccgggtaccgagctcgaattcactggccgtcgttttacaacgtcgtgactg60
ggaaaaccctggcgttacccaacttaatcgccttgcagcacatccccctttcgccagctg120
gcgtaatagcgaagaggcccgcaccgatcgcccttcccaacagttgcgcagcctgaatgg180
cgaatggccctgatgcggtattttctccttacgcatctgtgcggtatttcacaccgcata240
tagtgcactctcagtacaatctgctctgatgccgcatagttaagccagccccgacacccg300
ccaacacccgctgacgcgccctgacgggcttgtctgctcccggcatccgcttacagacaa360
gctgtgaccgtctccgggagctgcatgtgtcagaggttttcaccgtcatcaccgaaacgc420
gcgagacgaaagggcctcgtgatacgcctatttttataggttaatgtcatgataataatg480
gtttcttagacgtcaggtggcacttttcggggaaatgtgcgcggaacccctatttgttta540
tttttctaaatacattcaaatatgtatccgctcatgagacaataaccctgataaatgctt600
caataatattgaaaaaggaagagtatgagtattcaacatttccgtgtcgcccttattccc660
ttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaagtaaaa720
gatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatctcaacagcggt780
aagatccttgagagttttcgccccgaagaacgttttccaatgatgagcacttttaaagtt840
ctgctatgtggcgcggtattatcccgtattgacgccgggcaagagcaactcggtcgccgc900
atacactattctcagaatgacttggttgagtactcaccagtcacagaaaagcatcttacg960
gatggcatgacagtaagagaattatgcagtgctgccataaccatgagtgataacactgcg1020
gccaacttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttgcacaac1080
atgggggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagccatacca1140
aacgacgagcgtgacaccacgatgcctgtagcaatggcaacaacgttgcgcaaactatta1200
actggcgaactacttactctagcttcccggcaacaattaatagactggatggaggcggat1260
aaagttgcaggaccacttctgcgctcggcccttccggctggctggtttattgctgataaa1320
tctggagccggtgagcgtgggtctcgcggtatcattgcagcactggggccagatggtaag1380
ccctcccgtatcgtagttatctacacgacggggagtcaggcaactatggatgaacgaaat1440
agacagatcgctgagataggtgcctcactgattaagcattggtaactgtcagaccaagtt1500
tactcatatatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtg1560
aagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactga1620
gcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgta1680
atctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaa1740
gagctaccaactctttttccgaaggtaactggcttcagcagagcgcagataccaaatact1800
gttcttctagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgcctaca1860
tacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtctt1920
accgggttggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacgggg1980
ggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacag2040
cgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggta2100
agcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtat2160
ctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcg2220
tcaggggggcggagcctatggaaaaacgccagcaacgcggcctttttacggttcctggcc2280
ttttgctggccttttgctcacatgttctttcctgcgttatcccctgattctgtggataac2340
cgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacgaccgagcgcagc2400
gagtcagtgagcgaggaagcggaagagcgcccaatacgcaaaccgcctctccccgcgcgt2460
tggccgattcattaatgcagctggcacgacaggtttcccgactggaaagcgggcagtgag2520
cgcaacgcaattaatgtgagttagctcactcattaggcaccccaggctttacactttatg2580
cttccggctcgtatgttgtgtggaattgtgagcggataacaatttcacacaggaaacagc2640
tatgaccatgattacgccaagctatttaggtgacactataggggaaagcttgcatgcctg2700
caggtcgactctagaggatctactagtcatatggatt2737
<210>7
<211>616
<212>dna
<213>黑曲霉(asperguillusniger)
<400>7
aattcatggtgttttgatcattttaaatttttatatggcgggtggtgggcaactcgcttg60
cgcgggcaactcgcttaccgattacgttagggctgatatttacgtaaaaatcgtcaaggg120
atgcaagaccaaaccgttaaatttccggagtcaacagcatccaagcccaagtccttcacg180
gagaaaccccagcgtccacatcacgagcgaaggaccacctctaggcatcggacgcaccat240
ccaattagaagcagcaaagcgaaacagcccaagaaaaaggtcggcccgtcggccttttct300
gcaacgctgatcacgggcagcgatccaaccaacaccctccagagtgactaggggcggaaa360
tttatcgggattaatttccactcaaccacaaatcacagtcgtccccggtattgtcctgca420
gacggcaatttaacggcttctgcgaatcgcttggattccccgcccctggccgtagagctt480
aaagtatgtcccttgtcgatgcgatgtatcacaacatataaatactggcaagggatgcca540
tgcttggagtttccaactcaatttacctctatccacacttctcttccttcctcaatcctc600
tatatacacaactggg616
<210>8
<211>55
<212>dna
<213>未知(人工序列)
<400>8
atctactagtcatatggattgggcccgatatcgcggacattcgatttatgccgtt55
<210>9
<211>45
<212>dna
<213>未知(人工序列)
<400>9
cgcgttctcgaggaagttgcgtattgggatgaattttgtatgcac45
<210>10
<211>21
<212>dna
<213>未知(人工序列)
<400>10
gcaacttcctcgagaacgcgc21
<210>11
<211>24
<212>dna
<213>未知(人工序列)
<400>11
cccttttagtcaataccgttacac24
<210>12
<211>42
<212>dna
<213>未知(人工序列)
<400>12
aacggtattgactaaaagggacaaagccgcaggcgtgtactg42
<210>13
<211>54
<212>dna
<213>未知(人工序列)
<400>13
tcggtacccggggatccgattctagagggcccactctccacccttcacgagcta54
<210>14
<211>60
<212>dna
<213>未知(人工序列)
<400>14
atctactagtcatatggattgggcccgatatcaattcatggtgttttgatcattttaaat60
<210>15
<211>44
<212>dna
<213>未知(人工序列)
<400>15
agtagagatcggaacgacatcccagttgtgtatatagaggattg44
<210>16
<211>44
<212>dna
<213>未知(人工序列)
<400>16
atcctctatatacacaactgggatgtcgttccgatctctactcg44
<210>17
<211>41
<212>dna
<213>未知(人工序列)
<400>17
ggcataaatcgaatgtccgcgctggaccagcgtttacgcac41
<210>18
<211>20
<212>dna
<213>未知(人工序列)
<400>18
ctcaaccacaaatcacagtc20
<210>19
<211>19
<212>dna
<213>未知(人工序列)
<400>19
ggttggagtagagcacagc19
<210>20
<211>18
<212>dna
<213>未知(人工序列)
<400>20
accagcgacttccctcag18
<210>21
<211>19
<212>dna
<213>未知(人工序列)
<400>21
ataggtcacgaacccgact19
<210>22
<211>20
<212>dna
<213>未知(人工序列)
<400>22
tcaaccacaaatcacagtcg20
<210>23
<211>21
<212>dna
<213>未知(人工序列)
<400>23
taatgaacagcggatttgaag21
- 还没有人留言评论。精彩留言会获得点赞!