一种水稻抗白叶枯病蛋白及其编码基因与应用的制作方法
本发明属于生物技术领域,特别一种水稻抗白叶枯病蛋白及其编码基因与应用。
背景技术:
白叶枯病(xanthomonasoryzaepv.oryzae(xoo))是我国以及世界稻米主产区东南亚水稻的主要病害之一,严重威胁稻米安全生产。利用寄主抗性是控制该病害的有效措施。然而,由于病菌致病性变异,常造成品种抗性丧失,品种抗性的持效性及其机制成为当今抗病性研究的一个关键点,深入了解持久抗病性分子机制,对水稻品种获得持久抗性,持续有效地控制病害有着重要意义[伍尚忠,1982,《水稻白叶枯病及其防治》,上海科技出版社;mew,1987,currentstatusandfutureprospectsofresearchonbacterialblightofrice.ann.rev.phytopathol.,25:359-382.]。在已鉴定的白叶枯病抗性基因中,xa7被认为是一个持久抗病基因,能有效抵抗不同的致病型病原菌,在世界众多国家表现出优异而稳定的抗性[onaetal.,1998,epidemicdevelopmentofbacterialblightonricecarryingresistancegenesxa-4,xa-7,andxa-10.plantdis.,82:1337-1340.;adhikarietal.,1999,virulenceofxanthomonasoryzaepv.oryzaeonricelinescontainingsingleresistancegenesandgenecombinations.plantdis.,83:46-50;veraetal.,2000,predictingdurabilityofadiseaseresistancegenebasedonanassessmentofthefitnesslossandepidemiologicalconsequencesofavirulencegenemutation.proc.natl.acad.sci.,97:13500-13505.;曾列先等,2006,水稻抗白叶枯病近等基因系对华南菌系的抗性研究.植物病理学报,36:177-180]。
xa7基因最初是由国际水稻所(irri)在水稻品种dv85上鉴定出来的[sidhuetal.,1978geneticanalysisofbacterialblightresistanceinseventy-fourcultivarsofrice,oryzasatival.theorapplgenet,53:105-111.]。ogawa等通过dv85与ir24重复回交,把基因xa7导入近等基因系irbb7中[ogawaetal.,1991,breedingofnear-isogeniclinesofricewithsinglegenesforresistancetobacterialblightpathogenxanthomonascampestrispv.oryzae.jpnjbreed,41:523-529.]。hopkins等研究表明xa7是与一个无毒基因家族直接相对应的显性抗性基因[hopkinsetal.,1992,identificationofafamilyofavirulencegenesfromxanthomonasoryzaepv.oryzae.molplantmicrobeinteract,5:451-459.],kajiandogawa将该基因标记定位于第6染色体rgp图谱107.5cm位置,与g1091标记的重组率为8%[kajirandogawat.identificationofthelocatedchromosomeoftheresistancegene,xa-7,tobacterialleafblightinrice.breed.sci.,1995,45(suppl.1):79.],porter等对xa7进行了精细定位,但由于当时水稻基因组测序尚为完善,无法对目标基因区域的候选基因进行分析及预测[porteretal.,2003,developmentandmappingofmarkerslinkedtothericebacterialblightresistancegenexa7.cropscience,43:1484-1493.]。本课题组通过大群体分析,将xa7基因更精细地界定于分子标记gdssr02和rm20593之间,同样由于水稻基因组已测序品种代表性的局限,该基因定位的区域存在较大间隙(gap),仍然未能克隆目的基因[chenetal.,high-resolutionmappingandgenepredictionofxanthomonasoryzaepv.oryzaeresistancegenexa7.molecularbreeding,2008,3:433-441.]。
技术实现要素:
本发明的首要目的在于克服现有技术的缺点与不足,提供一种水稻抗白叶枯病蛋白。
本发明的另一目的在于提供编码上述水稻抗白叶枯病蛋白的基因。
本发明的再一目的在于提供上述基因的启动子区病原诱导调控元件。
本发明的再一目的还在于提供上述蛋白、基因以及启动子区病原诱导调控元件的应用。
本发明的目的通过下述技术方案实现:一种水稻抗白叶枯病蛋白,名称为xa7蛋白,其氨基酸序列如下所示:
maaadhpdrmpvavaglrhhyafpanlrpaarlltvnsgvflistagaivlvhtagnppaidndpayalvafvlfllgiwlmsialvadqfpraagvavaiaralqdyliggn。
编码上述水稻抗白叶枯病蛋白的基因,名称为xa7基因,其核苷酸序列如下所示:
atggcggccgctgatcatcctgatcgtatgcccgttgcagttgcaggcttgcgccaccattacgccttccctgcaaaccttcgccccgccgctcgactgctgaccgtcaactccggcgtcttcctcatctccaccgccggggccatcgtcctcgtccacaccgccggtaacccacccgccatcgacaacgatccagcctacgccttggtcgcattcgtgctcttcctcctcggaatctggctcatgtctattgccctcgtcgccgaccagttcccgcgcgccgctggggtcgccgtggccattgccagggcgctgcaggattacctcatcggtggcaattaa。
编码上述水稻抗白叶枯病蛋白的基因的启动子区病原诱导调控元件,其核苷酸序列如下所示:tataacccccccccccccagataacca。
所述的水稻抗白叶枯病蛋白可通过化学合成法合成得到;或是通过将编码所述的水稻抗白叶枯病蛋白的基因克隆入表达载体中,得到的重组表达载体转化宿主细胞,表达后纯化得到。
所述的编码水稻抗白叶枯病蛋白的基因的制备,可通过以下方式实现:通过化学合成方式获得;或者设计引物,以dv85、irbb7或其他携带xa7基因的水稻品种基因组dna为模板,pcr扩增获得;或者从携带xa7基因的质粒中酶切、筛选获得。
所述的编码水稻抗白叶枯病蛋白的基因的应用,可用于研究水稻抗白叶枯病的机制,也可用于培育对水稻白叶枯病具有抗病性的水稻品种或者其它抗病性作物,或是将其作为分子标记,用于选育对水稻白叶枯病具有抗病性的水稻品种。
所述的用于培育对水稻白叶枯病具有抗病性的水稻品种或者其它抗病性作物的步骤优选如下:将上述编码水稻抗白叶枯病蛋白的基因和上述启动子区病原诱导调控元件导入易感病的水稻或其它作物中,得到具有抗病性的水稻或抗病性作物;或者将组成型表达启动子或其它病原诱导型启动子与上述基因的编码序列串联,导入易感病的水稻或其它作物中,得到具有抗病性的水稻或抗病性作物。
所述的用于选育对水稻白叶枯病具有抗病性的水稻品种的步骤优选如下:将携带上述基因的水稻品种作为供体亲本,与易感白叶枯病的水稻品种进行花粉杂交,得到的一系列后代利用xa7作为分子标记进行筛选,鉴定得到抗白叶枯病水稻品种。
所述的供体亲本优选为dv85或irbb7。
编码上述水稻抗白叶枯病蛋白的基因的启动子区病原诱导调控元件的应用,可用于研究水稻抗白叶枯病的机制,也可用于培育对水稻白叶枯病具有抗病性的水稻品种或者其它抗病性作物。
所述的用于培育对水稻白叶枯病具有抗病性的水稻品种或者其它抗病性作物的步骤优选如下:将上述编码水稻抗白叶枯病蛋白的基因和上述启动子区病原诱导调控元件导入易感病的水稻或其它作物中,得到具有抗病性的水稻或抗病性作物;或者将上述启动子区病原诱导调控元件与其它抗病基因编码序列串联,导入易感病的水稻或其它作物中,得到具有抗病性的水稻或抗病性作物。
本发明相对于现有技术具有如下的优点及效果:
本发明是在前期研究基础上,通过构建水稻品种irbb7的基因组bac文库,文库筛选、候选插入片段测序、目标插入片段序列对avrxa7识别位点预测、以及一系列的转基因功能互补试验、基因敲除试验,最终完成了xa7功能基因的克隆。本发明首次提供了xa7功能基因的序列。
附图说明
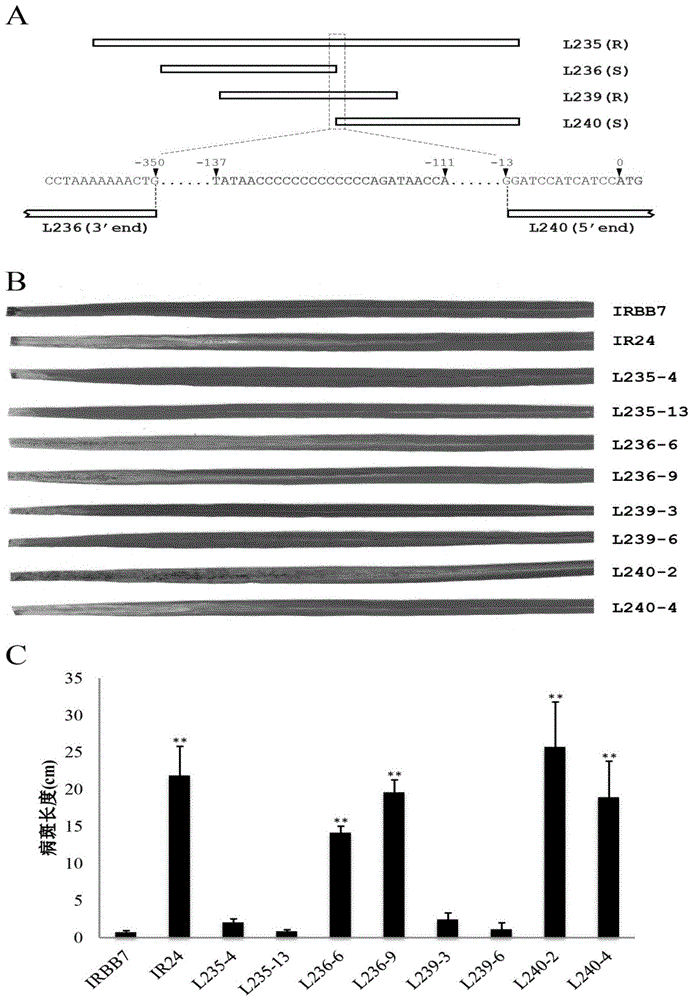
图1是亚克隆片段位置示意图及其转基因株系抗病表型图;其中,图a为用于转基因功能互补实验的亚克隆片段重叠区位置与序列示意图,图b为亚克隆转基因水稻株系对白叶枯病菌pxo86的抗性表型照片图;图c为亚克隆转基因水稻株系对白叶枯病菌pxo86的病斑长度统计结果图。
图2是xa7基因结构特征与抗性表达模式图;其中,图a为xa7基因序列结构特征示意图,图b为xa7基因对白叶枯病菌pxo86的抗性表达模式图。
图3是xa7基因启动子区病原诱导元件敲除功能验证结果图;其中,图a为xa7基因启动子区病原诱导元件进行基因编辑后的突变纯合株系序列,图b为各突变株系对白叶枯病菌pxo86的抗性表达模式图,图c为各突变株系对白叶枯病菌pxo86的抗病表型照片图。
图4是xa7基因编码区敲除功能验证结果图;其中,图a为xa7基因编码区进行基因编辑后的突变纯合株系序列,图4为各突变株系对白叶枯病菌pxo86的抗性表达模式,图c为各突变株系对白叶枯病菌pxo86的抗病表型。
具体实施方式
以下结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式并不限于此。若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。在本发明的实施例部分,阐述了xa7基因的分离克隆、功能特征及其功能验证。
实施例1xa7基因的分离克隆
在本发明前期研究基础上,发现xa7基因位点附近的基因组序列与常见的水稻参考基因组日本晴、9311、明恢63和珍汕97等均存在很大差异。明确该区域基因组序列是实现该基因克隆的前提基础。本发明通过构建xa7抗源品种irbb7基因组bac文库,用xa7两侧紧密连锁分子标记筛选文库,钓取阳性克隆,并对阳性克隆插入片段进行测序,从而获得该区域完整、精确的基因组序列。
水稻品种irbb7已在文献“ogawaetal.,1991,breedingofnear-isogeniclinesofricewithsinglegenesforresistancetobacterialblightpathogenxanthomonascampestrispv.oryzae.japanjbreed,41:523-529.”)中公开。
构建基因组bac文库的植物材料为含有xa7基因的近等基因系irbb7,载体为epicentre公司的copycontroltmpcc1bactm,亚克隆与转基因表达载体为pyltac747h/sacb(已在文献“xuetal.,2008,constructionandcharacterizationofthetransformation-competentartificialchromosome(tac)librariesofleymusmulticaulis.scienceinchina(seriesc:lifesciences),(07):604-613.”中公开)。
bac文库构建:按照文献“liuetal.,2002,developmentofnewtransformation-competentartificialchromosomevectorsandricegenomiclibrariesforefficientgenecloning.gene,282(1):247-255.”报道的实验步骤操作。于苗期提取irbb7基因组dna,用hindⅲ限制性内切酶部分酶切,用脉冲场电泳分离120-140kb长度的dna片段,纯化后与bac载体pcc1bactm进行连接。75ngbac载体分别与五组基因组dna酶切产物混合(a:30ng、b:60ng、c:120ng、d:160ng、e:350ng),按
bac阳性克隆筛选:用碱裂解法提取各个“bac克隆混合池”质粒dna,以混合池质粒为模板,扩增xa7两侧紧密连锁分子标记u05-indel和poz-indel,其中,扩增分子标记u05-indel的引物对为u05-indelfw和u05-indelrv,扩增分子标记poz-indel的引物对为poz-indelfw和poz-indelrv。检测到阳性的混合池后,将阳性池稀释40000倍,按每孔5×倍率分装到384孔板,培养过夜后取菌液为模板,再次用对应的分子标记引物进行pcr扩增筛选,直至筛选到阳性单克隆。经过多轮筛选,鉴定分离到三个阳性克隆:p1-10g、p3-12f和p2-9d。经酶切和电泳检测,这三个bac克隆插入片段分别为150kb、125kb和107kb。
u05-indelfw:5’-cagacaagtgttgttcatgttcg-3’;
u05-indelrv:5’-gaagtccgagctggggacgatgtac-3’;
poz-indelfw:5’-ccaagaaaggtccaactcgcttag-3’;
poz-indelrv:5’-gaacagtcctcagaattcgaccac-3’。
阳性克隆bac质粒测序及序列分析:用omega公司的bac/pacdnamaxikit提取p1-10g、p3-12f和p2-9d克隆的质粒,构建350bp小片段文库,采用hiseqpe150测序平台进行二代测序,每个克隆的测序数据量为1gb。测序数据进行denovo无参组装,并剔除载体pcc1bactm的骨架序列,分别获得这三个阳性质粒的插入片段序列。这三个插入片段序列经拼接组装后得到相互间首尾有重叠交错的307.5kb片段。与国际通用的水稻基因组序列比对,发现该片段对应于粳稻品种日本晴基因组增加了101.1kb,对应于籼稻品种明恢63基因组增加了80kb。利用softberry的fgenesh工具(http://www.softberry.com/berry.phtml?topic=fgenesh&group=programs&subgroup=gfind)对307.5kb的拼接序列进行预测,得到82个开放阅读框(orf)。为进一步缩小目标范围,通过galaxy的talgetter工具(http://galaxy2.informatik.uni-halle.de:8976)在拼接序列中预测avrxa7的识别结合位点(avrxa7ebe)。结果检索到四个识别结合位点的p-value小于1.0e-6,其中仅有一个avrxa7ebe位于orf上游的启动子区。这个orf编号为52,该候选基因命名为cg52,其序列如seqidno.4所示。
bac亚克隆文库构建与功能互补实验:将携带有cg52基因的bac质粒p1-10g和p3-12f的插入片段分别用bamhⅰ和sau3aⅰ不完全酶切,连接到亚克隆载体pyltac747h/sacb上。分别用cg52启动子区和cds区扩增引物筛选文库,阳性亚克隆再分别提取质粒进行末端测序,确定插入片段的序列。根据序列信息挑选四个亚克隆bac质粒通过农杆菌eha105(购于北京华越洋生物科技有限公司,质粒转化农杆菌步骤按文献“hoodetal.,1993,newagrobacteriumhelperplasmidsforgenetransfertoplants.transgenicres,2,208-218”操作)介导,转化籼稻感病品种ir24(水稻品种ir24在文献“ogawaetal.1991.breedingofnear-isogeniclinesofricewithsinglegenesforresistancetobacterialblightpathogenxanthomonascampestrispv.oryzae..japanjbreed.41:523-529.”中公开),籼稻品种的遗传转化按照文献“linandzhang,2005,optimisingthetissuecultureconditionsforhighefficiencytransformationofindicarice.plantcellrep,23:540-547.”报道的方法操作。转基因水稻孕穗期以“剪叶法”(按文献“伍尚忠等,1985,华南及菲律宾稻白叶枯病病原菌株致病性比较研究,植物病理学报,15-2:65-72.”操作)接种水稻白叶枯菌pxo86(菌株已在文献“mewtwetal.1982,pathotypesofxanthomonascompestrispv.oryzaeinasia.irriresearchpaperseries,no75.”中公开),于接菌后21天进行抗病表型调查,参照国际水稻所inger的方法调查评价抗性(按文献“ingergeneticresourcescenter,1996,standardevaluationsystemforrice(4thedition)[m].philippines:internationalriceresearchinstitutepress,p20-21.”操作)。
用作功能互补遗传转化的四个亚克隆的插入片段位置信息如图1a所示。l235和l239克隆覆盖率cg52基因全长,这两个克隆的转基因水稻株系均表现抗病;l236克隆的序列与l235片段的5’端重合,其3’端只包含了cg52基因的部分启动子区,在avrxa7ebe上游213bp位置,其转基因水稻株系表现感病;而l240克隆3’端与l235克隆重合,其5’端仅包含cg52的cds区和13bp的utr序列,缺失了avrxa7ebe序列,其转基因水稻株系表现感病。由此可知,cg52基因就是抗病功能基因,而且启动子区avrxa7ebe和完整的cds序列都是该基因功能不可或缺的部分。(图1b和图1c)。
实施例2xa7基因的序列结构与表达特征
xa7基因的序列结构分析:用携带xa7基因的近等基因系irbb7提取总rna,通过invitrogentm的generacertmkit分别扩增xa7的5’端和3’端全长。其中,5’race扩增特异引物为5’-tgccaccgatgaggtaatcctgc-3’,3’race扩增特异引物为5’-cctcctcggaatctggctcatgtc-3’。race产物通过
xa7基因的表达模式分析:irbb7和ir24在孕穗期以“剪叶法”接种白叶枯菌(pxo86)后分别于0、1、3、5天后取样,提取总rna。用takaratm的primescripttmrtreagentkitwithgdnaeraser反转录成cdna,通过
目的基因xa7的扩增引物对如下:
xa7fw:5’-gatcgtatgcccgttgcagttgc-3’;
xa7rv:5’-ggagttgacggtcagcagtcgag-3’。
内参基因tf2的扩增引物对如下:
tf2fw:5’-gcctgaagtgtactgtaccaccac-3’;
tf2rv:5’-caaagggttcagaaatgaggaagg-3’。
结果如图2b所示,xa7基因在接菌前表达水平很低,接菌1天后表达量开始上调,3天后达到峰值,5天后表达量开始回落。因此,xa7基因的表达模式是病原菌诱导激活型的。
实施例3由crispr/cas9介导的基因敲除验证xa7基因的关键功能位点
为进一步验证xa7启动子区avrxa7ebe和cds区序列的功能,本发明还利用crispr/cas9系统构建这两个功能区域的基因敲除转基因株系。基因敲除所用的载体是华南农业大学刘耀光实验室提供的双元表达载体pylcrispr/cas9pubi-h(已在文献“maetal.2015,arobustcrispr/cas9systemforconvenienthigh-efficiencymutiplexgenomeeditinginmonocotanddiocotplants.mol.plant.8,1274-1284.”中公开),中间载体pylsgrna-osu6al(已在文献“maetal.2015,arobustcrispr/cas9systemforconvenienthigh-efficiencymutiplexgenomeeditinginmonocotanddiocotplants.mol.plant.8,1274-1284.”中公开),pylsgrna-osu3al(已在文献“maetal.2015,arobustcrispr/cas9systemforconvenienthigh-efficiencymutiplexgenomeeditinginmonocotanddiocotplants.mol.plant.8,1274-1284.”中公开)和pylsgrna-osu6c(已在文献“maetal.2015,arobustcrispr/cas9systemforconvenienthigh-efficiencymutiplexgenomeeditinginmonocotanddiocotplants.mol.plant.8,1274-1284.”中公开)。编辑位点sgrna靶向序列的选择和设计是采用在线工具crispr-p(http://crispr.hzau.edu.cn/crispr/)辅助完成。
首先通过crispr-p在xa7启动子区avrxa7ebe和cds区检索pam(protospaceradjacentmotif)序列,选择编辑的靶位点,根据靶位点序列设计sgrna靶标接头。相应靶标接头序列如下:
target1(靶向avrxa7ebe,靶序列位于图3a的-126~-107位置):
osu6at1f:5’-gccgtatgtggttatctggggggg-3’;
osu6at1r:5’-aaaccccccccagataaccacata-3’;
target2(靶向avrxa7ebe,靶序列位于图3a的-121~-102位置):
osu6at2f:5’-gccgttcgtatgtggttatctgg-3’;
osu6at2r:5’-aaacccagataaccacatacgaa-3’;
target3(靶向cds区,靶序列位于图4a的+22~+41位置):
osu3t3f:5’-ggcactgcaacgggcatacgatc-3’;
osu3t3r:5’-aaacgatcgtatgcccgttgcag-3’;
target4(靶向cds区,靶序列位于图4a的+94~+113位置):
osu6ct4f:5’-tcagcgactgctgaccgtcaactc-3’;
osu6ct4r:5’-aaacgagttgacggtcagcagtcg-3’。
按照文献“maetal.2015,arobustcrispr/cas9systemforconvenienthigh-efficiencymutiplexgenomeeditinginmonocotanddiocotplants.mol.plant.8,1274-1284.”报道的实验步骤,分别将靶标接头连接到相应的bsaⅰ酶切sgrna中间载体,经过两轮pcr反应及产物退火、巢式pcr扩增得到特异的sgrna表达盒模板。表达盒再通过连接反应组装进双元表达载体。其中,target1和target2分别独立构建双元表达载体,target3和target4一起构建到同一个双元表达载体。这三个基因编辑表达载体分别通过eha105农杆菌介导,转入irbb7品种中。
各基因敲除转基因家系用引物(qcfw:5’-gaactgctctgctcaagtgcctc-3’;qcrv:5’-tgccaccgatgaggtaatcctgc-3’)pcr扩增靶向位点后测序,在t0代、t1代筛选编辑成功的纯合株系。
如图3a所示,target1编辑转基因获得的w6-4和w7-4纯合株系,target2编辑转基因获得的w8-7和w9-6纯合株系,都分别在xa7启动子区arvxa7ebe元件产生了碱基缺失。这些转基因株系的基因表达分析表明(图3b),arvxa7ebe元件的缺失会造成xa7受病原诱导表达的功能丧失,从而对病原菌表现感病(图3c)。
target3和target4编辑转基因获得的w12-1、w12-6、w13-4和w15-3纯合株系,分别在xa7基因cds区产生了不同类型的碱基缺失、插入和替换突变(图4a),导致xa7突变后的编码蛋白发生提前终止、移码、替换等突变。尽管基因cds区突变后,其转录仍受病原菌激活表达(图4b),但这些突变纯合株系对病原菌的抗性仍旧丧失了(图4c)。
本实施例一方面反向验证了xa7的功能基因,另一方面阐明了xa7基因的抗病功能由两个必要因素构成:一是其启动子区avrxa7ebe元件在病原诱导下的转录激活功能,另一个是完整的xa7基因编码蛋白执行抗病反应。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
序列表
<110>广东省农业科学院植物保护研究所
<120>一种水稻抗白叶枯病蛋白及其编码基因与应用
<130>1
<160>22
<170>siposequencelisting1.0
<210>1
<211>113
<212>prt
<213>水稻(oryzasativa)
<220>
<223>xa7蛋白
<400>1
metalaalaalaasphisproaspargmetprovalalavalalagly
151015
leuarghishistyralapheproalaasnleuargproalaalaarg
202530
leuleuthrvalasnserglyvalpheleuileserthralaglyala
354045
ilevalleuvalhisthralaglyasnproproalaileaspasnasp
505560
proalatyralaleuvalalaphevalleupheleuleuglyiletrp
65707580
leumetserilealaleuvalalaaspglnpheproargalaalagly
859095
valalavalalailealaargalaleuglnasptyrleuileglygly
100105110
asn
<210>2
<211>342
<212>dna
<213>水稻(oryzasativa)
<220>
<223>xa7基因
<400>2
atggcggccgctgatcatcctgatcgtatgcccgttgcagttgcaggcttgcgccaccat60
tacgccttccctgcaaaccttcgccccgccgctcgactgctgaccgtcaactccggcgtc120
ttcctcatctccaccgccggggccatcgtcctcgtccacaccgccggtaacccacccgcc180
atcgacaacgatccagcctacgccttggtcgcattcgtgctcttcctcctcggaatctgg240
ctcatgtctattgccctcgtcgccgaccagttcccgcgcgccgctggggtcgccgtggcc300
attgccagggcgctgcaggattacctcatcggtggcaattaa342
<210>3
<211>27
<212>dna
<213>水稻(oryzasativa)
<220>
<223>xa7基因启动子区病原诱导调控元件
<400>3
tataacccccccccccccagataacca27
<210>4
<211>2337
<212>dna
<213>水稻(oryzasativa)
<220>
<223>xa7基因基因组核苷酸序列
<220>
<222>(337)..(363)
<223>xa7基因启动子区病原诱导元件核苷酸序列
<220>
<222>(369)..(472)
<223>5'utr
<220>
<222>(473)..(814)
<223>外显子
<220>
<222>(815)..(1057)
<223>3'utr
<400>4
aataaaaaaaattctcagttctaccggggttcagttcggctgcggcctgcagtggctggc60
aaaaatctcgaactgctctgctcaagtgcctcaactggcagagataaatcctaaaaaaac120
tgaaaaataggccggtatcccagctcttctgctgaaaaaaactgaaaaactgggtgtaag180
attgatcgcaaaagtacttctggcacttgtcattttcgccacttctttggtctcttcgtc240
ctatttttgacatttctcttcgtccttttttcttttctctttcaaacgtgcagtcgccgt300
cgaggacggtgaaagccctgactgctaaaaccaatatataacccccccccccccagataa360
ccacatacgaacgaaggctttgaagcatcgcacacttgaagagcccccttcccaaccaca420
gccagcggtttccaaactccacgcctcgctcaacctgggggatccatcatccatggcggc480
cgctgatcatcctgatcgtatgcccgttgcagttgcaggcttgcgccaccattacgcctt540
ccctgcaaaccttcgccccgccgctcgactgctgaccgtcaactccggcgtcttcctcat600
ctccaccgccggggccatcgtcctcgtccacaccgccggtaacccacccgccatcgacaa660
cgatccagcctacgccttggtcgcattcgtgctcttcctcctcggaatctggctcatgtc720
tattgccctcgtcgccgaccagttcccgcgcgccgctggggtcgccgtggccattgccag780
ggcgctgcaggattacctcatcggtggcaattaactagaagcttcgaccatggctctgca840
cattcctctgctccagttgttcccggcttcccgtacgtgtgcctgatgattgtctttctc900
tgtttatttggctagtattttaggcttggaagttgaaaaactgtaaatctgcttcttttt960
cccctctgtactactactagactttcttttttaagctgacgtcatacacacaccccagat1020
tcataacgtgtcgtatagtaaatgtattcgaggcttgtaataaaaaggcacccgtaggtg1080
tatgctctgttcagtctgatgttctaaataatcaagatactattagtcgtattttctgct1140
tcatggcggcagtggtggtaatatttcaattccatacgaagggtcctcgctgactcactg1200
tgtggtgtgcgtgctcatttgctgacatgctaaagtcggactatagccaggtgtggccgt1260
gagggcattcggtgtactgaaaatagtgcaaaaacatccgccgcggtcgcgccaagtttg1320
tcgagaatacgcacaccgaaccggcctcacaactgatttcgtcggttttcagttgaaact1380
tcgctctttatttttggcagttcttgttcgtattcgttttctttctttcttttttaagaa1440
ttggaatcgaaataaaaaactccagatacaaaaaaaatgtaatcgaatattgtcgaaacc1500
ggaaacggagcgagtacgaattgacgtggatacggtaacgaaaatttatcagaatgaaaa1560
acccctcaaactgagttcaaactttatcaaatacatcacaaaacacaatcaattaagatg1620
ctaacttaaattggggtacgctattattatggtatatggagttatggaactccagaaaat1680
tccaagaaactgttaggtaagttttcgacggattccacgttccgactgaaactacaccta1740
tcgtatccgttttcgtttctgaaaaaaaaaaaaagaaatctccgaattggtttttgtttc1800
taaaaataggtttggaatccaaaaggctttcctatcgttttcatcctagttgtaaacctg1860
aacataccagtaaagcctattagaacaccccagcagatgcaccatccaatccaccacctg1920
agaaatagtgcactggaggggaaaaccggccttcagggaaatgggaaggaatgagacttt1980
tagatcttcctcgcaaaacagggagttaaatggacttacgaaggaatcattaattttatt2040
aaagaaaaaaattaagaagttaaaatctaaattaagaaggcaatgtatgaaacattgaaa2100
ataaagcgaaaggaaatgggaaggaattttcgtttatgacaagtcaacttgtaagagtca2160
attatctcaccaattctggaagcacagtgggacgtgggtccccctataggcctctttgat2220
gagcccattggaaagcccatttatacatggaataagttcatctgaggtctcttaacttgt2280
caacgaatccgattttcatccctcaaccgaaaaaccagacacaacgagtctctcaac2337
<210>5
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物u05-indelfw
<400>5
cagacaagtgttgttcatgttcg23
<210>6
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物u05-indelrv
<400>6
gaagtccgagctggggacgatgtac25
<210>7
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物poz-indelfw
<400>7
ccaagaaaggtccaactcgcttag24
<210>8
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物poz-indelrv
<400>8
gaacagtcctcagaattcgaccac24
<210>9
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物xa7fw
<400>9
gatcgtatgcccgttgcagttgc23
<210>10
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物xa7rv
<400>10
ggagttgacggtcagcagtcgag23
<210>11
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物tf2fw
<400>11
gcctgaagtgtactgtaccaccac24
<210>12
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物tf2rv
<400>12
caaagggttcagaaatgaggaagg24
<210>13
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物osu6at1f
<400>13
gccgtatgtggttatctggggggg24
<210>14
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物osu6at1r
<400>14
aaaccccccccagataaccacata24
<210>15
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物osu6at2f
<400>15
gccgttcgtatgtggttatctgg23
<210>16
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物osu6at2r
<400>16
aaacccagataaccacatacgaa23
<210>17
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物osu3t3f
<400>17
ggcactgcaacgggcatacgatc23
<210>18
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物osu3t3r
<400>18
aaacgatcgtatgcccgttgcag23
<210>19
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物osu6ct4f
<400>19
tcagcgactgctgaccgtcaactc24
<210>20
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物osu6ct4r
<400>20
aaacgagttgacggtcagcagtcg24
<210>21
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物qcfw
<400>21
gaactgctctgctcaagtgcctc23
<210>22
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物qcrv
<400>22
tgccaccgatgaggtaatcctgc23
- 还没有人留言评论。精彩留言会获得点赞!