一种适用于植物ATAC-seq测序技术的文库构建方法与流程
本发明涉及分子生物学技术领域,更具体地,涉及一种适用于植物atac-seq测序技术的文库构建方法。
背景技术:
核小体是真核细胞染色质的基本结构单位,由dna和组蛋白构成。每个核小体由146bp的dna缠绕在八聚体的组蛋白上形成,两个核小体之间通过一段连接dna相连,dna与组蛋白的结合可以发生动态变化。缠绕在组蛋白上的dna不易与其它蛋白结合,这些dna通常处于表达抑制状态。没有核小体结合的开放的dna区域易于与调控蛋白相结合,则可以使其下游的基因处于活跃表达的状态。细胞通过改变dna与组蛋白的结合位置,从而调控基因的表达。因此,获得处于开放状态的dna序列可用于研究基因表达的调控机制,是表观遗传学研究的热点,而atac-seq则是用于这类研究的最佳利器。
atac-seq技术是一种表观遗传学研究技术,通过转座酶对某种特定时空下开放的核染色质区域进行切割,获得在该时空下基因组中所有活跃转录的调控序列。然而,现有技术中绝大多数atac-seq实验存在3个方面的问题:
1、atac-seq实验都是首先通过流式细胞分选技术收集50,000个活细胞或者使用流式细胞核分选筛选50,000个细胞核进行实验,此项技术所需的工作量较大,成本太高,受众很窄,不适用于普通实验室大量进行atac-seq实验。
2、针对起始量的问题,几乎所有atac-seq实验要求的起始量都是50,000个细胞,但是不同生物50,000个细胞所含的dna数量完全不一样,视该生物的基因组大小而定,且所有的参考文献都是使用dapi对细胞核进行染色,使用荧光显微镜进行计数。首先荧光显微镜价格很高,很多实验室没有该仪器,再就是dapi为致癌的试剂,另外使用显微镜计数需要很高的人力,不适合进行大通量的操作。
3、植物atac-seq相比较于动物或者细胞系,有两大难点,首先是叶绿体的污染,其次是多糖多酚类物质对dna质量的影响。
技术实现要素:
针对现有技术中存在的技术问题,本发明提出了一种适用于植物组织atac-seq测序技术的文库构建方法,该方法操作简单,适用性广,尤其适用于物种或组织稀少、保存状态较差植物组织进行文库构建。
为实现上述目的,本发明是通过以下技术方案实现的:
一种适用于植物atac-seq测序技术的文库构建方法,所述方法包括以下步骤:
(1)制备植物单细胞悬液;
(2)nucleiextractionbuffer裂解细胞膜,获得细胞核并纯化;所述纯化方法为以下处理:依次经细胞核提纯缓冲液提纯细胞核、经细胞核分离缓冲液进行密度梯度离心分离细胞核;
(3)收集细胞核进行定量和细胞核完整性检测;
(4)完整细胞核中加入转座酶进行转座反应并纯化;
(5)pcr扩增,构建二代文库并纯化;
(6)进行高通量测序,得到基因数据。
进一步地,所述nucleiextractionbuffer裂解液包括0.25msucrose、10mmtris-hcl、1vol%tritonx-100、1×rochecompleteproteaseinhibitor。
进一步地,所述细胞核提纯缓冲液包括20mmmops、40mmnacl、90mmkcl、2mmedta、0.5mmegta、0.5mmspermidine、0.2mmspermine和1×rochecompleteproteaseinhibitor,ph=7.0。
进一步地,所述细胞核分离缓冲液包括10mmmes-kohph5.4、10mmnacl、10mmkcl、2.5mmedta、250mmsucrose、0.1mmspermine、1mmdtt、1×pvp、0.5mmspermidine、0.5vol%tritonx-100、1×rochecompleteproteaseinhibitor。
进一步地,所述步骤(3)中细胞核定量和细胞核完整性检测的方法为:取rsb重悬细胞核,吹打混匀后,分别取出50ul于两个离心管中,其中一管加入5ul的proteinasek,另一管不加proteinasek,56℃反应10分钟后检测dna含量,并通过比较两个离心管中dna含量来判断细胞核的完整性。
更进一步地,步骤(4)中转座反应为:取500ngdna对应的完整的核小体悬液,立即入转座酶及td转座缓冲液进行转座反应,轻轻涡旋后短暂离心,37℃水浴反应30min;转座结束后用qiagenminelutekit进行纯化,10ul洗脱液洗脱,纯化后的dna于-20度保存。
更进一步地,所述转座酶为tn5转座酶。
与现有技术相比,本发明的有益效果在于:
(1)本发明提出的适用于植物atac-seq测序技术的文库构建方法,基于转座酶对dna含量敏感的特征,直接通过dna含量进行定量操作,适用性广,尤其适用于物种或组织稀少、保存状态较差组织进行文库构建。
本发明的方法操作简单,前期处理不需要对细胞精确或者对组织做精确称量,直接使用大体系的裂解液处理,回收到细胞核悬液后,取少量混合液加入proteinasek反应10min,细胞核内的核小体解聚,dna释放,通过qubit定量变可估算原混合液中核小体含量;此时直接取出大约500ngdna对应的核小体悬液进行酶转座反应即可满足转座酶通常作用于50ngdna的要求。该方法尤其适用于物种或组织较为稀少,或者保存状态较差的样本,该方法针对不同物种和不同情况,从100ng-1000ngdna对应的细胞核作为起始量进行实验,均得到了合理的结果。
(2)本发明的技术方案中针对植物叶绿体的污染问题,采用tritonx-100裂解,再通过percoll进行梯度筛选,可以对叶绿体进行有效的去除;通过更换不同ph的缓冲液,同时使用pvp,有效的与糖类和酚类物质结合去除干净,在去除杂质时为了保证细胞核的稳定性,我们在三种缓冲液中分别使用了tris-hclph=8.0、mopsph=7.0、以及mes-kohph=5.4,此三种物质配合精胺和亚精胺,都能起到稳定dna的效果。
(3)本发明的方法在细胞核定量和细胞核完整性检测时,增加了一份不加proteinasek作为对照,如果核小体完整,不加proteinasek处理,核小体和dna完整的缠绕在一起,此时上清液中无法或者只能检测到少量的dna。使用proteinasek处理后dna的含量为不使用proteinasek处理的10倍以上,则视为核小体完整性良好。
(4)现有技术中atac-seq技术均使用illumina的转座酶,而本发明通过前期的优化使得回收到的细胞核可以用于国产vazyme公司开发的tn5转座酶并得到了良好的实验结果,使得atac-seq技术不再受转座酶种类的限制。
(5)本发明针对质量较差的组织样,采取了percoll密度梯度离心法,根据细胞核悬液不同物质的特性,使用不同密度梯度进行筛选,回收可用的细胞核。这样使得atac-seq技术的使用范围更加广泛而不是仅局限于新鲜组织。
附图说明
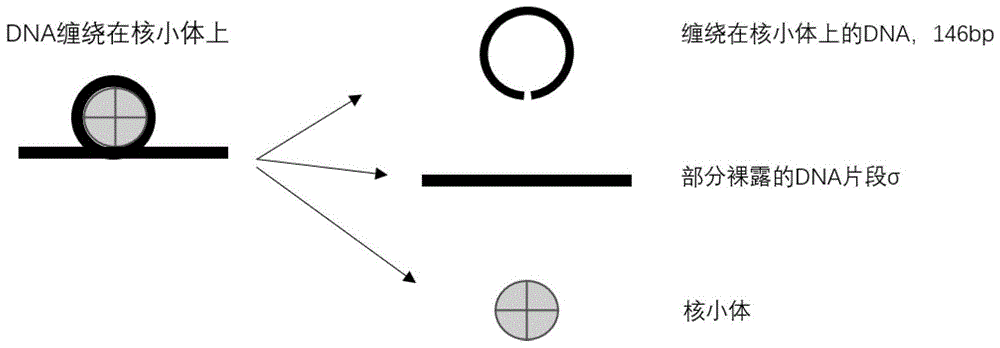
图1为缠绕核小体的dna结构示意图;
图2为通过本发明的方法获得dna文库的2100质检图。
图3为通过本发明的方法测序获得的reads数大小的分布图。
图4为通过本发明的方法测序获得的peaks的富集情况示意图。
具体实施方式
展示一下实例来具体说明本发明的某些实施例,且不应解释为限制本发明的范围。对本发明公开的内容可以同时从材料、方法和反应条件进行改进,所有这些改进,均应落入本发明的的精神和范围之内。若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
实施例1:
本发明针对植物组织的特殊性,提出了一种适用于植物atac-seq测序技术的文库构建方法,所述方法包括以下步骤:
1.制备植物单细胞悬液
取0.1g植物组织,液氮下充分研磨。
2.裂解细胞膜,获得细胞核并纯化
2.1溶液配置
(1)配置npbi缓冲液,其中包含20mmmops,40mmnacl,90mmkcl,2mmedta,0.5mmegta,使用2mkoh调整ph值到7.0,过滤除菌,在真空条件下脱气10min,储存在4℃。
(2)配置裂解液neb,其中包含0.25msucrose,10mmtris-hcl,1%tritonx-100,1×rochecompleteproteaseinhibitor,现配现用。
(3)配置npb细胞核提纯缓冲液:在配置好的npbi中加入终浓度为0.5mmspermidine,0.2mmspermine和1×proteaseinhibitor,现配现用。
(4)配置nib细胞核分离缓冲液:其中包含10mmmes-kohph5.4,10mmnacl,10mmkcl,2.5mmedta,250mmsucrose,0.1mmspermine,1mmdtt,1×pvp,0.5mmspermidine,0.5%tritonx-100,1×proteaseinhibitor现配现用。
2.2裂解细胞膜
向植物单细胞悬液中加入配置好的neb裂解液,冰上孵育5min,1200rcf4℃离心10min,小心去上清;
2.3纯化
2.3.1使用npb重悬,过40um细胞筛,1200rcf4℃离心10min,去上清;
2.3.2下层物质使用nib细胞核分离缓冲液重悬细胞核,进行密度梯度离心,方法如下:
1)取15ml离心管,加入2ml2.5m蔗糖;
2)沿着管壁缓慢加入5ml60%percoll,会在2ml的位置形成明显的蔗糖/percoll分层;
3)将上步的细胞核悬浮液延管壁缓慢加入,会在7ml的位置形成明显的percoll/细胞核悬液分层;
4)4℃1800g离心30min进行梯度离心,要求使用活动角度的离心机,固定角度的离心机会在开始和停止时离心力方向的改变破坏分离效果;
5)植物体中含有较多的糖类物质(如淀粉),与pvp结合后会形成大块颗粒,穿透percoll层进入蔗糖层或停留在2ml分界线上;而解聚的细胞核包括被triton裂解的叶绿体,由于蛋白质和dna的缠绕松开了,导致分子密度降低,且蛋白质和dna本身带有电荷,因此无法穿透不带电荷的percoll层,反而更容易留在上清缓冲液中,使得最上层为绿色,而percoll层收集到的为完整的细胞核;
6)取出percoll层于一个新的15ml离心管中。由于在1800g离心时,细胞核悬浮于30%percoll层中,而加大离心力对细胞核完整性不利,因此为了收集完整的细胞核,将percoll稀释,减小阻力,从而使用1800g离心使得将悬浮的细胞核沉淀下来。
2.3.3使用npb清洗细胞核沉淀,1200rcf4℃离心10min,小心去上清。
3.收集细胞核进行定量和细胞核完整性检测
采用dna定量,取2份50ul细胞核悬液,一份加蛋白酶k一份不加,56℃处理10min,使用qubit进行定量,若不加蛋白酶k的dna浓度比加了蛋白酶k浓度少10倍以上则认为核小体完整性良好。
本实施例中同时增加一份不加蛋白酶k(non_pk)作为对照,如果核小体完整,不加蛋白酶k处理,核小体和dna完整的缠绕在一起,此时上清液中无法或者只能检测到少量的dna。通常使用蛋白酶k处理后dna的含量为不使用蛋白酶k处理的10倍以上,则视为核小体完整性良好。
4.完整细胞核中加入tn5转座酶进行转座反应并纯化
4.1取出500ngdna对应的细胞核,500g4℃离心5min收集细胞核后,用35ulddh2o重悬,用于转座酶切,37℃处理30min。
该实施例中直接取500ngdna对应的细胞核进行转座反应的理由为:1个细胞的dna大约为10pg,50,000个细胞所含的dna大约就是500ng,而细胞开放区域及转座酶可以工作的区域大约为10%,也就是说500ngdna对应的细胞核中大约有50ng的区域可被转座酶工作,这也和转座酶通常作用于50ngdna的要求吻合。针对有些物种或组织较为稀少、或者保存状态较差的情况,能回收到的细胞核量较少,所以我们针对不同物种和不同情况,从100ng-1000ngdna对应的细胞核作为起始量进行了尝试,都得到了合理的结果。
4.2转座反应完成合,立即使用磁珠进行纯化,防止过度酶切。
5.pcr扩增,构建二代文库并纯化(pcr条件参照vazymetd501试剂盒中的步骤,所用试剂名称为vazymetd501和td202试剂盒中的试剂)
5.1配制pcr体系
26ul上述转座酶切产物,10ul5×tab,5ulppm,5uln5xx,5uln7xx,1ultae(n5xx、n7xx为两端的index,n5xx有n501-n508一共8种,n7xx有n701-n712一共12种)。
5.2pcr反应体系
105℃热盖,72℃3min(转座产物扩增特有的步骤,链置换反应,补平转座酶切产生的dna缺口);98℃30sec;以以下参数扩增12-18个循环,循环数视酶切产物dna含量而定:98℃15sec、60℃30sec、72℃3min;最后72℃5min。
5.3使用qiagen凝胶回收试剂盒去除引物二聚体和1kb以上的大片段;使用凝胶电泳和agilent2100进行质量检测。
6.进行高通量测序,得到基因数据:将所述扩增产物进行高通量测序得到atac-seq数据。
实施例2:
1.文库质检
如图1所示,缠绕核小体的dna可以看成两个部分,缠绕核小体的部分,长度为146bp,还有一段部分裸露的片段σ,长度不定。
根据实施例1的atac-seq实验,得到核小体之间的片段(大约60bp)加上136bp的接头共200bp左右的片段为第一个峰;和缠绕一个核小体、两个核小体的片段。dna缠绕核小体片段大小约为146bp,中间加上一些裸露的片段,最后加上136bp的接头,大约在300-400bp左右形成第二个峰;两个核小体缠绕的dna加上接头形成第三个峰,呈现出atac-seq实验特有的现象。结果如图2所示,其中峰1为核小体之间的片段,约60bp,加上136bp的接头大约是200bp,峰2为缠绕一个核小体的片段146bp加上136bp的接头再加上边缘的部分裸露的片段σ,大约350bp附近皆为正常;峰3为缠绕两个核小体的片段,两个核小体146bp×2,加上两个核小体之间60bp加上接头136bp再加上边缘的部分裸露的片段σ,大约550-600bp左右。因为σ的长度不定,所以导致每个峰的位置会出现一定范围的波动。
2.插入片段分布情况
图3为测序后的reads数大小的分布图,去掉了136bp的接头后形成了和图2一样的片段分布。和图2相比,不只有对应的每个缠绕核小体片段的reads,还有转座酶作用产生的特有的锯齿状。因为转座酶不是平滑的切除各个长度的dna片段,而是具有一定的跳跃性,因此得到的reads长度并不是平滑分布,而是特定的长度会有更多的片段,从而在图中显示出锯齿状。图3中146bp处为空白,即表示转座酶无法将缠绕核小体以外的dna全部切除,因转座酶体积较大,产生了空间位阻,所以每次都会留下一部分裸露的片段σ,因此在146bp处没有或者存在极少的reads,而旁边的146bp+σ处reads分布却特别高。同时根据二代测序的特异性,更加趋向于测小片段,因此相比较于图2,越小的片段测得的数据越多。
3.peaks在tss附近的富集情况
atac-seq得到的开放区域多为转录活性区域,因此peaks会在转录起始位点附近得到富集,图4显示了peaks的富集情况,在离tss越近的区域颜色越深,表明富集效果越好。该结果进一步证明了本发明的文库构建方法的有效性。
实施例3:
本实施例基于实施例1的建库方法,得到了棉花的atac-seq数据情况,如表1所示:
表1棉花的atac-seq数据情况
从表1中数据可以看出,采用本发明建库方法得到的棉花的atac-seq数据中完全不存在细胞器污染。
以上所述,仅为本发明较佳的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,根据本发明的技术方案及其发明构思加以等同替换或改变,都应涵盖在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!