一种用于罗氏沼虾遗传选育的分子标记的制作方法
本发明属于罗氏沼虾育种技术领域,具体涉及一种用于罗氏沼虾遗传选育的分子标记。
背景技术:
微卫星(microsatellites)又称简单重复序列(simplesequencerepeats,ssr),为第二代分子标记,具有多态性高、信息含量丰富、共显性遗传等有点,被广泛应用于水产动物的种质鉴定、遗传结构分析、遗传连锁图谱构建、qtl定位等方面。有关罗氏沼虾ssr相关的研究工作已有开展,但大多集中在群体遗传结构评估方面,目前尚未见运用ssr标记对罗氏沼虾生产性状进行关联分析的研究。
罗氏沼虾(macrobrachiumrosenbergii)由于食性广、病害少、生长快、营养价值高,因此具有重要的经济价值,它是世界上许多地区内陆水产养殖中重要的经济甲壳动物之一。中国于1976年从日本引进该虾,经40多年的发展,中国罗氏沼虾产业处于世界第一,据不完全统计,至2018年,中国苗种年生产量达到300亿尾,养殖面积超过3万公顷,总产量超过15万吨。产业的稳定快速发展得益于中国罗氏沼虾育种项目的开展及选育良种的产业化推广,浙江省淡水水产研究所自2006年开始运用blup育种技术,经过连续4年的家系选育,2009年,获得了第一个经中国水产原种和良种审定委员会审定通过的罗氏沼虾选育新品种“南太湖2号”(品种登记号:gs-01-001-2009),经遗传改良后,罗氏沼虾“南太湖2号”的生长速度和成活率显著提高,与商业苗种相比生长速度提高36.87%,成活率提高7.76%。
然而,传统的选择育种是基于性状表型值进行选育,育种周期长、效率低,随着现代分子生物学技术的发展,分子标记辅助育种成为热点,该技术有效地克服了常规育种过程中所采用的形态学标记存在数目少、受环境影响较大、不易直接选择等缺点,从而可加速育种进程,提高育种效率,缩短育种年限。
技术实现要素:
本发明提供一种用于罗氏沼虾遗传选育的分子标记,所提供的分子标记是通过对转录组中的ssr位点进行了多态性筛选,然后后进行ssr标记与体质量关联分析获得的,从而弥补现有技术的不足。
本发明所提供的分子标记,为ssr分子标记,其中标记mr28的核心序列为(attt)n,检测核心序列多态性的引物对,其上游引物序列为5′-tggcacaatgttttattggc-3′(seqidno:1),下游引物序列为5′-aaaaggggtttttgctggat-3′(seqidno:2);
标记mr32的核心序列为(catt)n,检测核心序列多态性的引物对中上游引物序列为5′-cgttgtcatcaccgtttcac-3′(seqidno:3),下游引物序列为5′-gttcctgagggctgtgctaa-3′(seqidno:4)。
本发明所提供的分子标记可用于生长性状关联分析、遗传图谱构建,qtl定位的分析中;
本发明再一个方面是提供一种筛选高体质量罗氏沼虾的方法,是通过检测待筛选个体中mr28标记的基因型是否为277/285;mr32标记的基因型是否为266/266或266/270来实现的。
所述的方法,是通过pcr引物进行扩增后,再对扩增产物进行测序后,对基因型进行分析完成的。
其中检测mr28标记的引物,其上下游引物的序列信息如下:
f:5′-tggcacaatgttttattggc-3′、
r:5′-aaaaggggtttttgctggat-3′;
其中检测mr32标记的引物,其上下游引物的序列信息如下:
f:5′-cgttgtcatcaccgtttcac-3′、
r:5′-gttcctgagggctgtgctaa-3′。
本发明所提供的筛选高体质量罗氏沼虾的方法,其一种具体操作如下:
1)提取待检测个体的基因组dna,
2)pcr扩增与荧光检测:
在正向引物5′端标记荧光素进行pcr反应,pcr产物进行自动荧光检测;
3)数据分析
用genemapper4.0软件生成位点的图谱文件,测出pcr扩增产物长度与荧光强度峰值,获得每个位点的基因型;根据基因型确定是否为高体质量罗氏沼虾。
本发明筛选获得的罗氏沼虾体质量相关ssr标记是从转录组数据中筛选得到的多态性位点,与功能基因相关,可为后续与生长性状关联分析、遗传图谱构建,qtl定位等提供有力支持。
附图说明
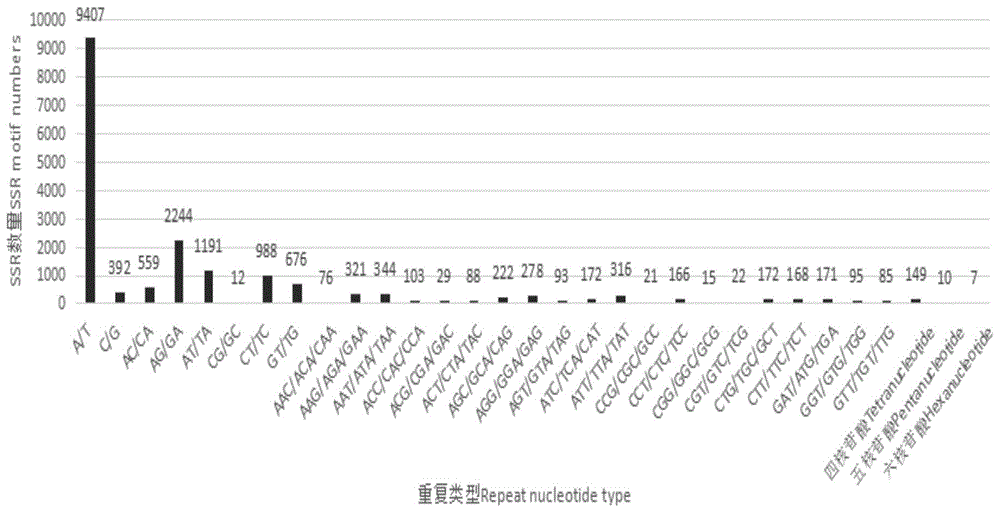
图1:罗氏沼虾转录组中unigenes的ssr分析结果图;
图2:mr28位点基因型分型结果图;
图3:mr32位点基因型分型结果图。
具体实施方式
下面结合实施例对本发明进行详细的描述。
实施例1:筛选ssr标记
1、获得实验虾
2017年4月,从浙江国家级罗氏沼虾遗传育种中心内通过群体随机交配,构建群体(mjl)1个,该群体为2014年从孟加拉国天然河流中引进。于5月份繁殖出苗,设置养殖密度2万/亩,养殖4个月,随机起捕199尾虾,测定每尾虾的体质量。剪取肌肉组织,固定于95%的乙醇中用于dna提取。
2、多态性ssr引物筛选
采用illuminahiseqtm4000高通量测序平台对罗氏沼虾卵巢组织进行转录组测序,原始数据经经质控、组装后,拼接获得95379个unigenes,采用misa软件对unigenes进行ssr检测。选取重复次数在5次以上的三碱基、四碱基或五碱基重复序列,使用primerpremier5.0软件进行引物设计,随机选择10个个体进行多态性ssr位点筛选,引物由生工生物工程(上海)股份有限公司合成。
3、基因组dna提取
采用takara基因组抽提试剂盒对两个群体的样品进行基因组dna抽提,具体操作方法参照说明书,1%的琼脂糖凝胶电泳和分光光度计检测dna质量和浓度,dna样品保存于-20℃备用。
4、pcr扩增与荧光检测
pcr反应总体积为25μl,模板为基因组dna50ng,其它组分根据taq酶(takara)说明书要求,在正向引物5’端标记荧光素fam(蓝色)。pcr反应条件为:94℃2min;94℃30s,60℃45s,72℃2min,30个循环;72℃延伸10min;4℃保存。用abi3730xl(美国应用生物系统公司)dna分析仪对pcr产物进行自动荧光检测。
5、数据分析
用genemapper4.0软件生成每个位点的图谱文件,测出pcr扩增产物长度与荧光强度峰值,获得每个位点的基因型。popgene32计算等位基因数(numbersofthealleles,na)、有效等位基因数(numbersoftheeffectivealleles,ne)、期望杂合度(expectedheterozygosity,he)、观测杂合度(observedheterozygosity,ho)及hard-weinberg平衡检验(p值)。参照botstein等的方法计算多态信息含量(polymorphisminformationcontent,pic)。
利用spss15.0软件进行统计分析,首先对表型数据正态性检验,应用一般线性模型(generallinearmodel,glm)进行微卫星位点与体质量关联分析,并对同一位点不同基因型进行多重比较(lsd)。由于一些位点中某些基因型频率太少,不具统计分析意义,因此实际统计分析中,每种基因型的样本数出现4次以上观察值时才被考虑。
共查找到18592个由一至六个核苷酸重复序列组成的ssr位点,占unigenes总数的比例为19.49%。单核苷酸重复类型比例最高,达52.71%,随后依次为二、三、四、五、六核苷酸重复类型,占比分别为32.61%、15.90%、0.80%、0.05%、0.04%。在单、二、三核苷酸重复类型中,数量最多的重复基元分别为a/t(9407个)、ag/ga(2244个)、aat/ata/taa(344个),数量最少的重复基元分别为c/g(392个)、cg/gc(12个)、cgg/ggc/gcg(15个)。随机选取了63个位点进行了多态性筛选,结果31个位点表现出多态,32个位点没有多态性,多态位点比例占49.2%(图1)。
群体遗传多样性分析
在31个多态位点中,选取19个位点进行遗传多样性分析。结果显示,共检测到65个等位基因,平均等位基因数为3.4211,有效等位基因数为1.0901-4.2309,平均值为2.0941,观测杂合度为0.0854-0.6734,平均值为0.4105,期望杂合度为0.0826-0.7656,平均值为0.4496,多态信息含量为0.0805-0.7267,平均值为0.3882,6个位点pic值大于0.5,4个位点pic值小于0.25,9个位点的pic值在0.25至0.5之间,19个位点中,mr08、mr10、mr17、mr22、mr27、mr30、mr34、mr46与mr65位点符合hard-weinberg平衡,其余10个位点显著(p<0.05)或极显著(p<0.01)偏离hard-weinberg平衡(表1)。
表1:群体遗传多样性分析
实施例2:与体质量关联的ssr位点
利用glm模型进行ssr位点与罗氏沼虾体质量关联分析,结果显示,mr28、mr32两个ssr位点与体质量显著相关(p<0.05)(表2)。对具有显著差异的ssr位点进行不同基因型与体质量多重比较,其中mr28位点277/285基因型个体体质量均值极显著高于277/289、285/285基因型个体(p<0.01),显著高于285/289、273/289、270/273、285/293基因型个体(p<0.05);mr32位点266/266、266/270基因型个体体质量均值显著高于270/270基因型个体(p<0.05)(表3)。
表2:mr28、mr32位点与体质量性状的关联性表
注:表中数值为微卫星位点与体质量关联分析的概率值,上表“*”代表体质量性状与标记呈显著相关(p<0.05),没有上标标注的代表性状与标记不相关(p>0.05)。“—”代表该位点没有多态性。
表3:基因型与体质量多重比较表
其中检测mr28标记的引物,其上下游引物的序列信息如下:
f:5′-tggcacaatgttttattggc-3′、
r:5′-aaaaggggtttttgctggat-3′;
其中检测mr32标记标记的引物,其上下游引物的序列信息如下:
f:5′-cgttgtcatcaccgtttcac-3′、
r:5′-gttcctgagggctgtgctaa-3′。
两个位点扩增的基因型如下表4
表4:mr28和mr32的基因型信息表
本发明中所筛选到的与体质量相关标记可以快速准确筛选生长性状优良亲本用于罗氏沼虾育种,其一种方法步骤如下:
1、takara基因组抽提试剂盒对两个群体的样品进行基因组dna抽提,具体操作方法参照说明书,1%的琼脂糖凝胶电泳和分光光度计检测dna质量和浓度,dna样品保存于-20℃备用。
2、pcr扩增与荧光检测
pcr反应总体积为25μl,模板为基因组dna50ng,其它组分根据taq酶(takara)说明书要求,在正向引物5’端标记荧光素fam(蓝色)。pcr反应条件为:94℃2min;94℃30s,60℃45s,72℃2min,30个循环;72℃延伸10min;4℃保存。用abi3730xl(美国应用生物系统公司)dna分析仪对pcr产物进行自动荧光检测。
3、数据分析
用genemapper4.0软件生成每个位点的图谱文件,测出pcr扩增产物长度与荧光强度峰值,获得每个位点的基因型。
初步研究结果表明用本发明两个标记筛选的罗氏沼虾个体作为亲本繁育的子代显示出更好的生长特性,能用于罗氏沼虾的遗传选育。
序列表
<110>宁波大学
<120>一种用于罗氏沼虾遗传选育的分子标记
<160>4
<170>siposequencelisting1.0
<210>1
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>1
tggcacaatgttttattggc20
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
aaaaggggtttttgctggat20
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
cgttgtcatcaccgtttcac20
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
gttcctgagggctgtgctaa20
- 还没有人留言评论。精彩留言会获得点赞!