草鱼补体C5基因、用于克隆草鱼补体C5基因的引物及克隆方法与流程
本发明涉及生物技术领域,具体涉及一种草鱼补体c5基因,同时还涉及用于克隆该基因的引物和克隆方法。
背景技术:
补体c5是补体系统中一种重要组分,是含硫酯键的α-2巨球蛋白超家族,其基因序列与补体c3、补体c4具有高度的同源性,但在功能上,补体c5参与所有补体激活途径,活化产生过敏毒素小片段c5a和c5b,c5b随后形成膜攻击复合体c5b-9,穿透细胞膜,c5a则能介导白细胞呼吸暴发,但这种介导功能具有种的特异性。目前有关补体c5基因在人类、小鼠、虹鳟、鲤鱼、牛这几种生物钟都有报道,但没有关于草鱼补体c5基因cdna的克隆和序列分析方面的研究报道。
技术实现要素:
本发明针对上述现有技术的不足,提供一种草鱼补体c5基因、用于克隆草鱼补体c5基因的引物及克隆方法,通过race法,从草鱼肝脏中克隆得到草鱼补体c5基因cdna全长序列,并利用软件对其进行氨基酸理化性质分析、信号肽预测、三级结构预测和同源性分析,为进一步研究该基因的结构和功能奠定基础,同时也为草鱼补体其他基因的研究提供可靠的参考。
本发明所采用的技术方案是:草鱼补体c5基因,该基因的核苷酸序列如seqidno.1所示。
本发明同时提供草鱼补体c5基因编码的氨基酸,序列如seqidno.2所示。
本发明还提供用于克隆草鱼补体c5基因的引物,其中,该引物分别为c5-orf-f/r、c5-5'-外、c5-3'-外,序列分别如下:
c5-orf-f:aattctgcagtcgacggtaccatggcacgtttatttttgttctgt,
c5-orf-r:atggtggcgaccggtggatccgcatcacagcccgataataagaaatct;
c5-5'-外:gattacgccaagcttagcaattccatctccccgactcgtcact,
c5-3'-外:5’-aatgtcaattgacattcatgacagcgt-3’。
本发明还提供一种克隆草鱼补体c5基因的方法,其中,该方法步骤如下:
(1)从草鱼肝脏中提取总rna;
(2)设计扩增草鱼补体c5基因的保守结构域核苷酸片段的引物c5-orf-f和c5-orf-r,将提取的草鱼rna反转录成cdna第一链;并以合成的cdna第一链为模板,pcr扩增得cdna保守结构域核苷酸片段;
(3)利用试剂盒合成草鱼补体c5基因的5′-race-readycdna和3′-race-readycdna;
(4)根据步骤(2)扩增得到的cdna保守结构域片段设计5’-race下游内外巢引物和3’-race上游内外巢引物,并分别以步骤(3)合成的5′-race-readycdna和3′-race-readycdna为模板,扩增得到草鱼补体c5基因的cdna5′端和cdna3′端片段;
(5)将cdna保守结构域核苷酸片段、cdna5′端和cdna3′端片段进行拼接得到草鱼补体c5基因的全长cdna序列。
上述引物c5-orf-f和c5-orf-r是根据ncbi中草鱼补体c5基因序列设计的。
本发明首次用该发明的针对草鱼补体c5基因的克隆race方法获得草鱼补体c5基因的cdna全长序列。该草鱼补体c5基因cdna序列全长5365bp,编码1693个氨基酸,同源性分析表明,草鱼补体c5基因和鲤鱼在进化上亲缘关系最近,同源性为79.33%;并在草鱼补体c5蛋白中发现有a2m-n-2、anato、a2m、a2m–recep和c345c五个共同结构域,从生物信息学方面确定了草鱼补体c5的存在。由于草鱼补体c5序列较长,对于每一步实验条件的要求都比较高,因此本发明通过不断优化实验条件,探索出克隆草鱼补体c5基因的最佳实验体系。其中最大的特点是通过对引物的修饰,将引物tm控制在理想范围之内,设计出了高度有效的5’和3’端的引物。并将最终结果通过ncbiblast比对,确证草鱼补体c5cdna序列的完整性。
附图说明
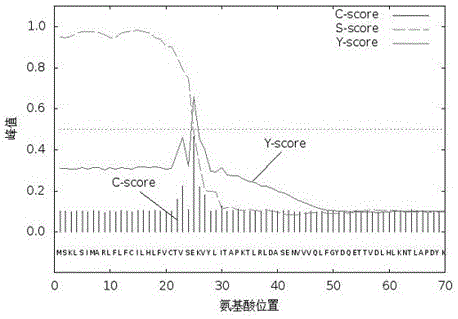
图1是草鱼补体c5的cdna基因氨基酸序列中的信号肽切割位点图。
其中,c-score:用来区分是否为剪切位点,最高峰值为剪切位点后的第一个氨基酸(即成熟蛋白的第一个氨基酸残基);s-score:用来区分相应位置是否为信号肽区域;y-score:c-score和s-score的几何平均数,用于避免多个高分c-score值对结果的影响。
图2是草鱼补体c5的蛋白质跨膜结构示意图。
其中,左侧的圈代表第一个跨膜结构,位于4-25aa;右侧的圈代表第二个跨膜结构,位于904-925aa。
图3是草鱼补体c5的系统发育树。
图4是swiss-model预测草鱼补体c5蛋白同源三级结构模型。
具体实施方式
为了更好的解释本发明,以下结合具体的实施例进一步阐明本发明的主要内容,但本发明的内容不仅仅局限于以下实施例。
实施例1草鱼补体c5蛋白基因cdna全长序列结构与分析
实验动物与样品采集:1龄健康草鱼采自湖南浏阳市乌龙渔场渔场。丁香麻醉后解剖采集草鱼肝脏,剪成100mg左右大小分装样品管中,液氮速冻,-70℃保存备用。
1.草鱼肝脏总rna抽提
将用于研磨组织的研钵于烤箱中180℃烘烤4小时,待研钵冷却至室温后,注入液氮预冷,使研钵的温度与-70℃保存的草鱼肝脏组织样本基本一致。取-70℃保存的肝脏冰冻组织约100mg置于已预冷的研钵中,迅速加入液氮把肝脏组织反复研磨成粉末。在已经研磨成粉末的草鱼肝脏组织中加入总rna提取液,迅速匀浆;把研钵中的匀浆液转移到已经depc处理的一无rnase的离心管中;12000r/min、4℃离心10min,留取上清,室温静置5分钟,加入1ml氯仿,上下颠倒混匀15秒,又室温静置8分钟,12000r/min、4℃离心15分钟;取最上层上清液至另一干燥的无rnase离心管,加与上清液等体积异丙醇,上下颠倒混匀20秒,室温放置10分钟,再12000r/min、4℃离心10分钟;离心,小心吸除上清,在留取的下层液中加入depc水配制的75%乙醇1ml,悬浮沉淀,7500r/min、4℃离心5分钟,后再重复7500r/min、4℃离心5分钟,小心彻底吸除乙醇,留取沉淀,室温干燥30分钟,加30μl无rnase的灭菌去离子水,置于55℃水浴锅中溶解沉淀;检测提取的草鱼肝脏总rna浓度和纯度。样品的浓度计算公式:[rna]=od260×d×40ng/μl,其中,d为样品的稀释倍数,根据od260/od280比值判断rna的纯度。
2.草鱼补体c5基因的5′-race-readycdna和3′-race-readycdna的合成
使用clontech公司smartracecdnaamplificationkit试剂盒分别合成草鱼补体c5基因的5′-race-readycdna和3′-race-readycdna,用tebuffer稀释10倍-20℃保存备用。
3.草鱼补体c5基因的cdna保守结构域核苷酸片段序列克隆
根据ncbi中草鱼补体c5基因序号auw36852.1序列设计扩增草鱼补体c5基因保守结构域核苷酸片段的引物:
c5-orf-f:aattctgcagtcgacggtaccatggcacgtttatttttgttctgt,seqidno.3,
c5-orf-r:atggtggcgaccggtggatccgcatcacagcccgataataagaaatct,seqidno.4。
并采用fermentasrevertaidtmfirststrandcdnasynthesiskit反转录试剂盒将提取的rna反转录成草鱼补体c5基因保守结构域cdna第一链。以该cdna第一链为模板,c5-orf-f、c5-orf-r为引物,进行pcr扩增,获得草鱼补体c5基因的cdna保守结构域核苷酸片段。用taqdna聚合酶(takara)进行pcr扩增,扩增条件为:95℃预变性5min、94℃变性30s、68℃退火30s、72℃延伸1.5min、35个循环,72℃延伸7min。
4.草鱼补体c5基因的cdna5’race的获得
根据上述克隆得到的草鱼补体c5基因的cdna保守结构域核苷酸片段设计5’-race下游内外巢引物,引物序列如下:
c5-5'-外:gattacgccaagcttagcaattccatctccccgactcgtcact,seqidno.5;
c5-5'-内:gattacgccaagcttattcgcgccgttgagcttcagggact,seqidno.6。
采用c5-5'-外引物和试剂盒自带的通用引物10*universalprimeramix(upm)对上述合成的5′-race-readycdna进行扩增。其中,touchdownpcr反应条件为:2×seqampbuffer25μl、5′-race-readycdna或3′-racereadycdna2.5μl、upm5μl、5′或3′引物1μl、seqampdnapolymerase1μl及pcr-gradewater15.5μl,总体积50μl。反应程序为:95℃预变性5min;94℃变性30s、72℃延伸1.5min、5个循环;94℃变性30s、70℃退火30s、72℃延伸1.5min、5个循环;94℃变性30s、68℃退火30s、72℃延伸1.5min,25个循环;72℃延伸7min。获得草鱼补体c5基因cdna5′端片段。
5.草鱼补体c5基因cdna3’race的获得
根据上述已克隆得到的草鱼补体c5基因cdna保守结构域核苷酸片段序列设计3’-race上游内外巢引物,引物序列如下:
c5-3'-外:5’-aatgtcaattgacattcatgacagcgt-3’,seqidno.7;
c5-3'-内:5’-taatgataaattgcaatgtcaatagaaga-3’,seqidno.8。
采用c5-3'-外引物和试剂盒提供的通用引物10*universalprimeramix(upm)对上述合成的3′-race-readycdna进行pcr扩增,反应体系和反应条件同5’-race的pcr。从而获得基因cdna3′端片段。
6.结果
6.1草鱼补体c5基因序列结构与分析
通过rt-pcr和race法,从草鱼肝脏中克隆获得了草鱼补体c5基因cdna保守结构域核苷酸片段、5’和3’端cdna片段,经克隆测序后,测序所得的这三段片段通过bioedit软件拼接得到完整的cdna全长。比对所得有效序列运用dnastar软件,expasy程序(http://www.expasy.ch/tools/)推算其开放阅读框和编码氨基酸序列情况。结果表明草鱼补体c5基因全长cdna序列全长为5365bp(见seqidno.1),包括60bp的5'非编码区(5'-utr),226bp的3'非编码区(3'-utr)与5079bp的开放阅读框(orf),共编码1693个氨基酸(见seqidno.2),翻译起始密码子为atg,序列终止密码子为taa,以“aataaa”为特征加尾信号。
6.2草鱼补体c5基因信号肽预测
用smart(http://smart.emblheidelberg.de/)进行蛋白质序列的信号肽分析,信号肽预测草鱼补体c5为分泌信号肽,信号肽区域为1-27aa,结合参见图1,说明草鱼补体c5基因具有信号肽,且信号肽的剪切位点位于第24和25号氨基酸之间。
6.3草鱼补体c5蛋白的理化性质
草鱼补体c5基因cdna共编码1693氨基酸,相对分子质量191342.83,理论pi值7.26,正电荷残基数209,负电荷残基数209,不稳定系数35.56,蛋白稳定。脂肪系数86.18,总平均亲水性-0.274,为亲水性氨基酸。蛋白质疏水作用是蛋白质折叠的主要驱动力,分析蛋白质氨基酸亲疏水性是了解蛋白质折叠的第一步,氨基酸疏水分析为蛋白质二级结构预测提供佐证,是分析蛋白质跨膜区的重要一步。草鱼补体c5蛋白有2个跨膜区,分别在4-25aa、904-925aa,参见图2。
6.4草鱼c5基因氨基酸同源性分析和系统进化树
ncbi(http://www.ncbi.nlm.nih.gov/)上进行同源基因搜索比对,mega4.0构建系统发育树,系统进化树的分析结果显示,草鱼的c5序列先与鲤鱼c5聚为一支,再与斑马鱼聚为一支,然后与虹鳟、大马哈鱼等聚合,最后与罗非鱼、黄鳝等鱼类聚合,草鱼c5基因的进化地位和物种的进化地位一致(图3)。氨基酸同源性比对,草鱼c5基因与鲤鱼的同源性为79.33%,与斑马鱼的同源性为73.86%,而与非鲤科鱼类的同源性较低。
6.5草鱼补体c5蛋白的结构域分析
结构域分析是反映基因保守性和基因鉴定的重要方法,ncbi网站structure工具检索cdd数据库分析蛋白质序列的保守结构域,对草鱼c5结构域分析表明,草鱼c5预测蛋白包含26个α螺旋,72个β折叠,包含a2m-n-2、anato、a2m、a2m–recep和c345c五个结构域,都属于α-2巨球蛋白家族。anato是过敏毒素因子的同源结构域,c345c是膜攻击复合体结构域,这些保守结构区和超基因家族都是补体因子所特有的,结合参见图4。
本发明首次用该发明的针对草鱼补体c5基因的克隆race方法获得草鱼补体c5基因的cdna全长序列。结果表明,草鱼补体c5基因cdna序列全长5365bp,编码1693个氨基酸,同源性分析表明,草鱼补体c5基因和鲤鱼在进化上亲缘关系最近,同源性为79.33%。补体c5基因本身有高度保守性,但鱼类的补体存在种群特异性,因此各种类鱼之间补体c5的同源性都不高,模式动物斑马鱼补体c5与鮸鱼补体c5同源性最低为50.9%,与罗非鱼的同源性最高也只有75.4%。补体c3、c4、c5源于同一祖先,拥有共同的结构域a2m-n、a2m-n-2、anato、a2m、a2m–comp、a2m–recep和c345c。本发明也在草鱼补体c5蛋白中发现有a2m-n-2、anato、a2m、a2m–recep和c345c五个共同结构域,因此,从生物信息学方面确定了草鱼补体c5的存在。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受实施例的限制,其它任何未背离本发明的精神实质与原理下所做的改变、修饰、组合、替代、简化均应为等效替换方式,都包含在本发明的保护范围之内。
序列表
<110>湖南农业大学
<120>草鱼补体c5基因、用于克隆草鱼补体c5基因的引物及克隆方法
<130>005
<160>8
<170>siposequencelisting1.0
<210>1
<211>5365
<212>dna
<213>合成()
<400>1
ctaatacgactcactatagggcaagcagtggtatcaacgcagagtacatgggaccacacc60
atgagcaaactgtctataatggcacgtttatttttgttctgtatacttcatctcttcgtc120
tgcgytactgtttctgaaaaagtatacttaatcactgcacctaaaaccctgcgtctcgat180
gcttcagaaaatgttgtggtgcagttgttcggttatgaccaggaaactacagtagatctt240
cacctgaagaacacactggcaccagattataaagaatatgcatctcagtccctgaagctc300
aacggcgcgaataactatcaagcttcggctactttacggatcatgcctgtagatttcaca360
aaagaggacaaatatgtcttccttcaggcgatttcttctgctttcactgcatataaaaaa420
atcccagtcactactgtcaatggttttctctttattcagactgacaagccactatataca480
cccgaacaagaggtacaagtgcgtgtttactccctcaacgaggagctgaggaaatctaga540
cggaccgtcacccttacattcatggaccctaacggtatcaaggtagagattattgatatg600
gcagacatcaatggagcaaaaccacttcttccaccgttcaaaatacccttgaagccggct660
tatggaatatggaaaattgaggccacctatgccgacacttttaaaacaactgccactgct720
gagtttgaagtcaaagaatatgtgcttcccagtatttcaatccacattgagcctgaaacc780
aactatatcagcgaagagaactttgagtctttccagctgaagatttcagccaagtatgtc840
catggaactccagttagttcagcggatattttcttgatgtttggatacagttctccagaa900
gaaaccgttatgattccagcaacatatagcaaatatatgctgataaatggaaaggaggaa960
ataaacttaaacatcaggtcagctctgtcaaccaaacctgatgggcctcaacgcctgagt1020
gacatgaaagaaaacacctttcttcgtgtgacagttttgctccaagagacaactggtggt1080
atctcccaggaggctgtgctctcaaatgtgaaatttgtagaaactccatttaccctgagt1140
ctgactgcaacccctcctttcatcaaaccgggtctaccatacaatatgagagtcttggtg1200
aaggatcctttgggtaaaccagtgaaaggtgtgtcagtcaagacaatggcaacaataatt1260
acagctaacaaagaacgagagagcctcagatttttagggcaccatgatacaatgacagtg1320
acgagtcggggagatggaattgcttacttcacctgtaatatacctgataatgtaaaagaa1380
gctgaattcacttttgagacagaagatcctcgttggagtcaggctccacttaaactaaag1440
gctgaatcatatgtgtctgtcaaccatcgttacctgtacattgatttacatcctgaatcc1500
tcagtatttgaagttggacggtacattaacatcaaggttttctttcattatcgtgactac1560
ctttctctggagaccttcagctaccagatcatttccagaggaaaaattgtgcagtttgcc1620
acagttaagcgtgtcagtgaaaagagtcagagccttaacattaagatcacccctgacatg1680
gtgccgtctgctcggctactggtgtactatgtcctgtatggggaggaaaaagcagagctg1740
gtggcagactctgcatggattgatgtgaaggctaaatgtgttcaaaatttaaatatggac1800
atatcaactctcaactcgcagtataaacccaaagataaactcgaattaagagtgtcaact1860
aaaactaaagaagagtcactggtggctttttctgctgtagatacggccctgtacaaccta1920
aaatctaatgacaaagaccctctgaaaaaggtgttacatcatgttgagcgaagtgacttg1980
ggttgtggaagaggaggaggcaaaaataatgctgatgtgtttgaccgtgctggccttatg2040
ttcattacaaacgccatggcctcaagtgcagaggggacctgctcagacctggttaggttg2100
aagcgttcacctgacctggggcaaaagtttgaatcaaaagccaaaatgtacgggccgtcc2160
aggaattgctgcttatctgggacacgcagtatcccgacgcttgaaacctgtggggatcgt2220
gctaagagactgcctttttctgaaaagactactgattactggaagaaaaggtgccagcgt2280
gccttcttggagtgctgtgaatttgctataaagctccgtaaagacagtgtggataagatc2340
attctaagccgtggagcgatcgactttttgctggatgcgatgccatctcagataaggagc2400
tacttccctgagagctggttatgggaggaacacaccagcaagtctgggtcagttagcatt2460
accaaaaatcttccagattcactgactacatgggagctaaaagcagttggggtgttcagt2520
gaggggatctgtgtgtcagaagaaaaggttcaggtgtcccaggacattagtgtggatgtt2580
cccttgccatattccatggttcgtggagagcagattgagctcaggggttcagtctacaat2640
cagcgtttttcaaaaacttggttccgtgtgaccctaacggcaacagatggagtgtgtgtg2700
tttcatggaactcaacaggggaaacatggaaagccaaatgaaatcaaggggcaaatagac2760
ggtcgttcagtggctttggtcacattctacatcatggctttggaagcgggcactcataca2820
ctcactttcactttaacaactaagtgggggggtgagactgtggtcaagaaattgaaagtg2880
gtgccagagggaattagaaaggaaatacaaagtggtggaagaatagatccaaatggagtc2940
tttggcacttccatgcgaaaacttgagttaaaacatgccattccaccaaacattgcaccc3000
aagtcaactgtggatcgtattctaacaattaatggtgaagtactgggagagcttctgtca3060
ataataaataacccagaaggtttaaaacagttgactaacctgccacggggaaatgcagag3120
atggagctaatgggtctcctgccgatctactatgtctaccactacctagaaaagacagag3180
aaatgggacattctgggaaaagagtcagctgcaagtgaaagggaattaaagaggaagatg3240
caagctggcatcaccagcatcatgtcatataagctgaggaatgaatatgctttcagcatg3300
tggtcaaacaaggacaaatcagccagtacctgggtgacggctcttatagccaagacttta3360
gctgacatgcatgaatatgtctctttggagtctgaagtgctgaccaacaccatctattgg3420
ctgattaaaaatcgtcagaatgatgatggctcgttctatgagaaatcacaaaccaatcca3480
attaaactcatgggtgccggagcagatgtcactgaaaagacagtattcctcacatctttt3540
gtcgtgattggaattaaaaatgctctgaaggtcccaaacattaacatacaggcctttaaa3600
gatgctttagatcacgccacgcagtacctctgctctaagtcaaagaagattgaaagttta3660
tatgttagagccatcgcatcctatgctctgacactggtagacacccatagcatgtcagca3720
gttgatctttatgaaaaaatcaaacagcaggctcaagttaaaggaaacccagctacaatt3780
cgtttctgggaagattctaaacctaagaaggattcattaaaacccagtgatgtgacagca3840
aagactgtggaaacaacagtttacatcctgcttaacactttagtcagaggtgattccacc3900
tatgcaaagcccatcctgaactggttaacacaagaccagagatacggagggggctttcat3960
tcaacacaggacagcattcttactttggaggcccttaccaagtacagcatcttagccagt4020
caagccaccttagacatggtggttgacattgaatacaagactaaaggtgatatcagcaga4080
atcaaactaactcagcaaaagcctgtggctaaaccaatagaggtgaccaaaaatgatgat4140
ataataatcaaaactgcaatgagctctggtatttcatttgccaaattgagaacagtctac4200
tatgagatgacagagagcaatgaaaattgccaatttgaaatgtcaattgacattcatgac4260
agcgtcccattttcagatgatcctatgcttttgtcacagaggattgtggcctgtgcaaaa4320
tacaagccacatgaaaatacatttgaaacggagtcaggactcactgtgatggaaatttat4380
ttgccaaccggtgtgatcccagtccaggaggatctagatatgtaccaaaatggacttgag4440
tctcagatttccaactatgaaataatgggtgaccaagtcatccttcaaatagattcgata4500
ccatcaaaagatttctactgtgtgggtttccgtattcaagaggaatttgaaactggcctg4560
accagagcctcggtattcagagtttatgaattttatgatcaagaaaataagtgtatgaag4620
ttttaccacaaacaaacccgcaaactattgagactctgtgagggagaccagtgtcagtgc4680
atggcagcggaatgctgcaactttaaatccaccattgataccacaatcactgctgaacag4740
cgcagaaattatttgtgtaaaaaaagcatgaaatatgcatttaaagtgttaattgaatcc4800
tctgaggcagtaggagattttgtgacctacaaagcaaaagttaaaacagtgctcaaaaaa4860
gaacaaacggacgacttaaaaacagattcagagatagattttgtgaagaaggcaacttgc4920
actgaaacaaattttgaagttggcaagcagtatctgatcatgacttctgagagtataaag4980
ataaaagtggatcatgcttataagtataagtttcctttggactcgcatgcttgggtggac5040
tggtggccacttgaatctgactgcaggaatgatgcctgcaaacaatacaccggtgttttg5100
ggtgattttggactagatttcttattatcgggctgtgattaatgataaattgcaatgtca5160
atagaagagatcattcttatgcatgttcttattctataacaacatcctctgaataagaat5220
aaacagaaatgttcaataaattaatcaataatcaattttagaaattttaaatatatgtga5280
ttattttataaacttagtcatttattttgtattaaccacaaacatatacaacattaaaca5340
aaaaaaaaaaaaaaaaaaaaaaaaa5365
<210>2
<211>1693
<212>prt
<213>合成()
<400>2
metserlysleuserilemetalaargleupheleuphecysileleu
151015
hisleuphevalcysxaathrvalserglulysvaltyrleuilethr
202530
alaprolysthrleuargleuaspalasergluasnvalvalvalgln
354045
leupheglytyraspglngluthrthrvalaspleuhisleulysasn
505560
thrleualaproasptyrlysglutyralaserglnserleulysleu
65707580
asnglyalaasnasntyrglnalaseralathrleuargilemetpro
859095
valaspphethrlysgluasplystyrvalpheleuglnalaileser
100105110
seralaphethralatyrlyslysileprovalthrthrvalasngly
115120125
pheleupheileglnthrasplysproleutyrthrprogluglnglu
130135140
valglnvalargvaltyrserleuasnglugluleuarglysserarg
145150155160
argthrvalthrleuthrphemetaspproasnglyilelysvalglu
165170175
ileileaspmetalaaspileasnglyalalysproleuleupropro
180185190
phelysileproleulysproalatyrglyiletrplysilegluala
195200205
thrtyralaaspthrphelysthrthralathralagluphegluval
210215220
lysglutyrvalleuproserileserilehisilegluprogluthr
225230235240
asntyrileserglugluasnphegluserpheglnleulysileser
245250255
alalystyrvalhisglythrprovalserseralaaspilepheleu
260265270
metpheglytyrserserproglugluthrvalmetileproalathr
275280285
tyrserlystyrmetleuileasnglylysglugluileasnleuasn
290295300
ileargseralaleuserthrlysproaspglyproglnargleuser
305310315320
aspmetlysgluasnthrpheleuargvalthrvalleuleuglnglu
325330335
thrthrglyglyileserglnglualavalleuserasnvallysphe
340345350
valgluthrprophethrleuserleuthralathrpropropheile
355360365
lysproglyleuprotyrasnmetargvalleuvallysaspproleu
370375380
glylysprovallysglyvalservallysthrmetalathrileile
385390395400
thralaasnlysgluarggluserleuargpheleuglyhishisasp
405410415
thrmetthrvalthrserargglyaspglyilealatyrphethrcys
420425430
asnileproaspasnvallysglualagluphethrphegluthrglu
435440445
aspproargtrpserglnalaproleulysleulysalaglusertyr
450455460
valservalasnhisargtyrleutyrileaspleuhisprogluser
465470475480
servalphegluvalglyargtyrileasnilelysvalphephehis
485490495
tyrargasptyrleuserleugluthrphesertyrglnileileser
500505510
argglylysilevalglnphealathrvallysargvalserglulys
515520525
serglnserleuasnilelysilethrproaspmetvalproserala
530535540
argleuleuvaltyrtyrvalleutyrglygluglulysalagluleu
545550555560
valalaaspseralatrpileaspvallysalalyscysvalglnasn
565570575
leuasnmetaspileserthrleuasnserglntyrlysprolysasp
580585590
lysleugluleuargvalserthrlysthrlysglugluserleuval
595600605
alapheseralavalaspthralaleutyrasnleulysserasnasp
610615620
lysaspproleulyslysvalleuhishisvalgluargseraspleu
625630635640
glycysglyargglyglyglylysasnasnalaaspvalpheasparg
645650655
alaglyleumetpheilethrasnalametalaserseralaglugly
660665670
thrcysseraspleuvalargleulysargserproaspleuglygln
675680685
lysphegluserlysalalysmettyrglyproserargasncyscys
690695700
leuserglythrargserileprothrleugluthrcysglyasparg
705710715720
alalysargleupropheserglulysthrthrasptyrtrplyslys
725730735
argcysglnargalapheleuglucyscysgluphealailelysleu
740745750
arglysaspservalasplysileileleuserargglyalaileasp
755760765
pheleuleuaspalametproserglnileargsertyrpheproglu
770775780
sertrpleutrpglugluhisthrserlysserglyservalserile
785790795800
thrlysasnleuproaspserleuthrthrtrpgluleulysalaval
805810815
glyvalphesergluglyilecysvalsergluglulysvalglnval
820825830
serglnaspileservalaspvalproleuprotyrsermetvalarg
835840845
glygluglnilegluleuargglyservaltyrasnglnargpheser
850855860
lysthrtrppheargvalthrleuthralathraspglyvalcysval
865870875880
phehisglythrglnglnglylyshisglylysproasngluilelys
885890895
glyglnileaspglyargservalalaleuvalthrphetyrilemet
900905910
alaleuglualaglythrhisthrleuthrphethrleuthrthrlys
915920925
trpglyglygluthrvalvallyslysleulysvalvalproglugly
930935940
ilearglysgluileglnserglyglyargileaspproasnglyval
945950955960
pheglythrsermetarglysleugluleulyshisalailepropro
965970975
asnilealaprolysserthrvalaspargileleuthrileasngly
980985990
gluvalleuglygluleuleuserileileasnasnprogluglyleu
99510001005
lysglnleuthrasnleuproargglyasnalaglumetgluleumet
101010151020
glyleuleuproiletyrtyrvaltyrhistyrleuglulysthrglu
1025103010351040
lystrpaspileleuglylysgluseralaalasergluarggluleu
104510501055
lysarglysmetglnalaglyilethrserilemetsertyrlysleu
106010651070
argasnglutyralaphesermettrpserasnlysasplysserala
107510801085
serthrtrpvalthralaleuilealalysthrleualaaspmethis
109010951100
glutyrvalserleuglusergluvalleuthrasnthriletyrtrp
1105111011151120
leuilelysasnargglnasnaspaspglyserphetyrglulysser
112511301135
glnthrasnproilelysleumetglyalaglyalaaspvalthrglu
114011451150
lysthrvalpheleuthrserphevalvalileglyilelysasnala
115511601165
leulysvalproasnileasnileglnalaphelysaspalaleuasp
117011751180
hisalathrglntyrleucysserlysserlyslysilegluserleu
1185119011951200
tyrvalargalailealasertyralaleuthrleuvalaspthrhis
120512101215
sermetseralavalaspleutyrglulysilelysglnglnalagln
122012251230
vallysglyasnproalathrileargphetrpgluaspserlyspro
123512401245
lyslysaspserleulysproseraspvalthralalysthrvalglu
125012551260
thrthrvaltyrileleuleuasnthrleuvalargglyaspserthr
1265127012751280
tyralalysproileleuasntrpleuthrglnaspglnargtyrgly
128512901295
glyglyphehisserthrglnaspserileleuthrleuglualaleu
130013051310
thrlystyrserileleualaserglnalathrleuaspmetvalval
131513201325
aspileglutyrlysthrlysglyaspileserargilelysleuthr
133013351340
glnglnlysprovalalalysproilegluvalthrlysasnaspasp
1345135013551360
ileileilelysthralametserserglyileserphealalysleu
136513701375
argthrvaltyrtyrglumetthrgluserasngluasncysglnphe
138013851390
glumetserileaspilehisaspservalpropheseraspasppro
139514001405
metleuleuserglnargilevalalacysalalystyrlysprohis
141014151420
gluasnthrphegluthrgluserglyleuthrvalmetgluiletyr
1425143014351440
leuprothrglyvalileprovalglngluaspleuaspmettyrgln
144514501455
asnglyleugluserglnileserasntyrgluilemetglyaspgln
146014651470
valileleuglnileaspserileproserlysaspphetyrcysval
147514801485
glypheargileglnglugluphegluthrglyleuthrargalaser
149014951500
valpheargvaltyrgluphetyraspglngluasnlyscysmetlys
1505151015151520
phetyrhislysglnthrarglysleuleuargleucysgluglyasp
152515301535
glncysglncysmetalaalaglucyscysasnphelysserthrile
154015451550
aspthrthrilethralagluglnargargasntyrleucyslyslys
155515601565
sermetlystyralaphelysvalleuilegluserserglualaval
157015751580
glyaspphevalthrtyrlysalalysvallysthrvalleulyslys
1585159015951600
gluglnthraspaspleulysthraspsergluileaspphevallys
160516101615
lysalathrcysthrgluthrasnphegluvalglylysglntyrleu
162016251630
ilemetthrsergluserilelysilelysvalasphisalatyrlys
163516401645
tyrlyspheproleuaspserhisalatrpvalasptrptrpproleu
165016551660
gluseraspcysargasnaspalacyslysglntyrthrglyvalleu
1665167016751680
glyasppheglyleuasppheleuleuserglycysasp
16851690
<210>3
<211>45
<212>dna
<213>合成()
<400>3
aattctgcagtcgacggtaccatggcacgtttatttttgttctgt45
<210>4
<211>48
<212>dna
<213>合成()
<400>4
atggtggcgaccggtggatccgcatcacagcccgataataagaaatct48
<210>5
<211>43
<212>dna
<213>合成()
<400>5
gattacgccaagcttagcaattccatctccccgactcgtcact43
<210>6
<211>41
<212>dna
<213>合成()
<400>6
gattacgccaagcttattcgcgccgttgagcttcagggact41
<210>7
<211>27
<212>dna
<213>合成()
<400>7
aatgtcaattgacattcatgacagcgt27
<210>8
<211>29
<212>dna
<213>合成()
<400>8
taatgataaattgcaatgtcaatagaaga29
- 还没有人留言评论。精彩留言会获得点赞!