一种带有分子标签的单链建库方法和接头组合、试剂盒与流程
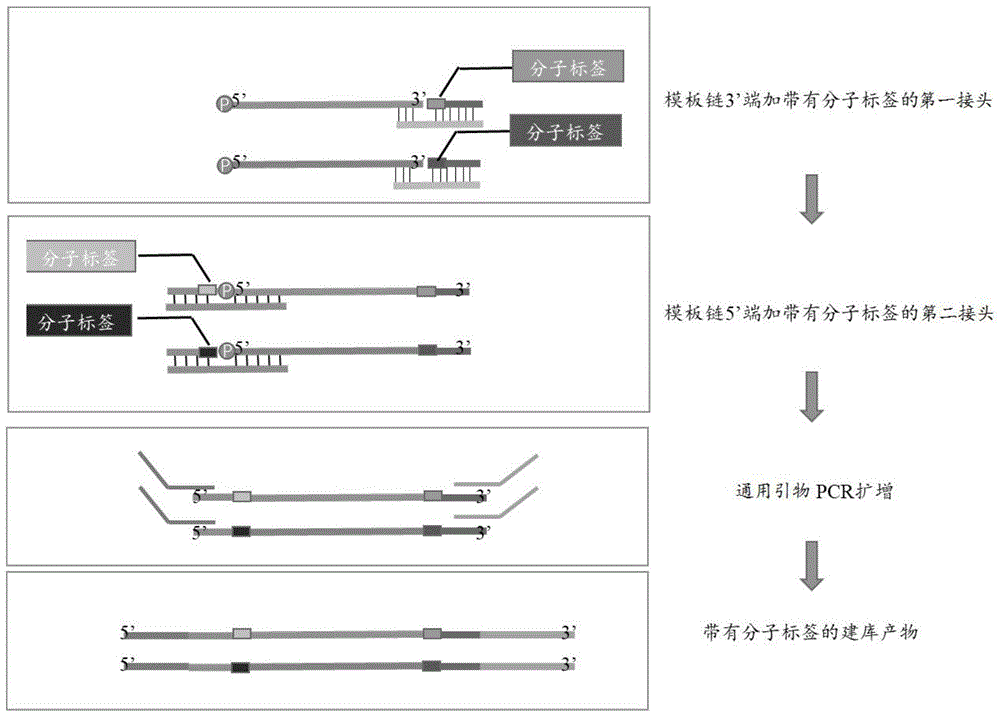
本发明属于基因测序文库
技术领域:
,尤其涉及一种带有分子标签的单链建库方法和接头组合、试剂盒。
背景技术:
:血浆游离dna又称cfdna(cellfreedna),是指外周中游离于细胞外的dna,cfdna的来源既包括正常细胞,也有异常细胞(如肿瘤细胞)或者外源的微生物(如病毒dna),具体包括自身正常细胞代谢的凋亡小体,肿瘤细胞碎片,外泌体等。循环肿瘤dna(circulatingtumordna)ctdna是指从原发肿瘤甚至是转移形成的新肿瘤细胞破裂掉落下来到循环外周血中的dna。ctdna在临床医学中具有重要意义,通过检测ctdna在肿瘤的早期检测,肿瘤的靶向治疗和后期的疾病监控等方面具有重要的临床价值。ctdna检测难点有主要是由于是含量低,人群在10ng-25ng。另外发现有部分ctdna呈单链形式,双链建库往往会忽略这类单链dna,造成检测有遗漏。目前采用ngs方法的主流检测过程均会涉及到pcr扩增和测序,这两个过程都会出现一些系统错误,如illuminahiseq平台的测序错误率在0.1%,在普通大量样本模板检测中问题并不大。但是,在肿瘤早期筛查中,由于本身模板类就是在万分之一到千分之一的范围之内,这些测序错误跟我们的检测下线在一个数量级,就会对结果造成非常严重的干扰,其次,由于pcr引入的错误也无法进行有效识别。常用双链建库无法针对cfdna中的单链dna进行建库。对于甲基化检测,常规的双链建库主要存在的问题如下:1.样本起始量100ng起,无法针对低起始量的dna建库;2.成本较高,需要对接头序列碱基进行修饰;3.检测灵敏度低,无法检测低频的甲基化位点;4.冗余高,由于采用的是先建库后转化的策略,导致建好的文库发生断裂导致效率低下进一步导致冗余度上升。单链建库可以有效避免冗余度高的弊端。这种方案的流程是先对dna进行转化,虽然会有片段化以及单链dna产生,但在随后的单链建库过程中可以有效地被建库保留进入下一个实验环节。目前市场上推出的主要单链建库方法有swift公司的accel-ngsmethyl-seq,qiagen的qiaseqmethyllibkit方案。以上两种建库方案都能进行单链建库,但主要存在的问题是都不带有分子标签,无法进行去冗余,因此灵敏度较低。技术实现要素:本发明的目的在于提供一种带有分子标签的单链建库方法和接头组合、试剂盒,旨在解决现有单链建库无法引入分子标签的技术问题。为实现上述发明目的,本发明采用的技术方案如下:一方面,一种带有分子标签的单链建库方法,包括如下步骤:提供样本dna,将所述样本dna进行磷酸化处理,得到单链dna模板;将所述单链dna模板的3’端连上第一接头,得到第一连接产物;将所述第一连接产物的5’端连上第二接头,得到第二连接产物;利用通用引物对所述第二连接产物进行pcr扩增,得到文库;其中,所述第一接头和/或第二接头含有分子标签,且所述第一接头和第二接头为双链接头。另一方面,一组用于单链建库的接头组合,包括用于连接单链dna模板的3’端的第一接头,用于连接单链dna模板的5’端的第二接头;其中,所述第一接头和/或第二接头含有分子标签,且所述第一接头和第二接头为双链接头。以及一种用于单链建库的试剂盒,包括本发明的上述接头组合,以及通用引物、聚核苷酸激酶pnk和t4dna连接酶。本发明提供一种带有分子标签的单链建库方法,在单链dna模板的3’端连上第一接头,在单链dna模板的5’端连上第二接头,而第一接头和/或第二接头含有分子标签,通过分子标签的引入,可以对低起始量dna进行有效建库,而且可以排除pcr和测序引入的系统错误,有效提高测序灵敏度,保证测序准确度。本发明提供的接头组合和试剂盒,包括用于连接单链dna模板的3’端的含的第一接头,用于连接单链dna模板的5’端的第二接头;而第一接头和/或第二接头含有分子标签,通过分子标签的引入,可以对低起始量dna进行有效建库,而且可以排除pcr和测序引入的系统错误,有效提高测序灵敏度,保证测序准确度。附图说明图1是本发明实施例中带加分子标签的单链建库方法的流程示意图;图2是本发明实施例中添加分子标签的单链建库方法与不添加分子标签的质控结果对比图;图3是本发明实施例中添加分子标签的单链建库方法的分子标签分布结果图。具体实施方式为了使本发明的目的、技术方案及优点更加清楚明白,以下结合附图及实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。需要理解的是,术语“第一”、“第二”仅用于描述目的,而不能理解为指示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”的特征可以明示或者隐含地包括一个或者更多个该特征本发明实施例一方面提供一种带有分子标签的单链建库方法,包括如下步骤:s01:提供样本dna,将所述样本dna进行磷酸化处理,得到单链dna模板;s02:将所述单链dna模板的3’端连上第一接头,得到第一连接产物;s03:将所述第一连接产物的5’端连上第二接头,得到第二连接产物;s04:利用通用引物对所述第二连接产物进行pcr扩增,得到文库;其中,所述第一接头和/或第二接头含有分子标签,且所述第一接头和第二接头为双链接头。本发明实施例提供一种带有分子标签的单链建库方法,在单链dna模板的3’端连上第一接头,在单链dna模板的5’端连上第二接头,而第一接头和/或第二接头含有分子标签,通过分子标签的引入,可以对低起始量dna进行有效建库,而且可以排除pcr和测序引入的系统错误,有效提高测序灵敏度,保证测序准确度。单链建库技术应用广泛,特别是针对低启示量dna测序,甲基化dna测序以及游离dna(cfdna)中的单链dna测序技术特别有效。本发明实施例提供一种带有分子标签的,通过该方法可以对单链dna进行建库,以符合二代测序甚至三代测序平台要求。在一个实施例中,所述样本dna包括cfdna,无需打断处理;或者是基因组dna(gdna),则经打断至100-400bp。若需要甲基化测序则经重亚硫酸盐处理,在一实施例中,所述单链建库方法得到的文库用于甲基化测序,在将所述样本dna进行磷酸化处理的步骤之前,先用重亚硫酸盐处理。本发明实施例不仅可以用于甲基化建库方案,还可以用于常规dna建库,用于常规dna建库方法时,可以有效避免常规建库方案用到的末端补平以及加a这一步骤,同时针对超低量的cfdna(0.5ng)也有良好的建库效果,满足多方面的需求。在一个实施例中,所述磷酸化处理包括多聚核苷酸激酶pnk处理,将所述单链dna模板的3’端连上第一接头的步骤中使用的是t4dna连接酶;将所述第一连接产物的5’端连上第二接头的步骤中使用的是t4dna连接酶。本发明实施例开发了一种带有分子标签的单链建库方案,该方案不仅可以用于对甲基化dna进行建库(需要先进行重亚硫酸盐处理)也能对低起始量的dna进行建库。本发明实施例中,不仅可以双端带分子标签也能单端带分子标签,即可以是第一接头(如下a3badaptor)上含有分子标签而第二接头(如下a5badaptor)不含分子标签,也可以是第二接头(如下a5badaptor)含分子标签而第一接头(如下a3badaptor)不含分子标签,也可以使第一接头和第二接头同时含分子标签。本发明实施例中,不仅可以使用固定碱基序列作为分子标签,也能使用随机碱基序列作为分子标签后混合使用,还能使用半固定半随机的碱基序列作为分子标签。通过随机碱基序列作为分子标签可以更进一步有效地降低冗余,也可以进一步提高检测灵敏度保证测序准确度。在一个实施例中,以illumina平台为例,所述第一接头由互补配对的seqidno.1和seqidno.2组成,所述第二接头由互补配对的seqidno.3和seqidno.4组成;其中,seqidno.1中的na和seqidno.2中的nb为互补配对的分子标签,seqidno.3中的nc和seqidno.4中的nd为互补配对的分子标签;所述通用引物为seqidno.5和seqidno.6;具体建库步骤如下:合成带有分子标签的碱基序列,如表1所示。表1表1中,nnnnnn是随机序列,x6/y6分别是i5端i7端index。na-nd作为分子标签,可以是3-9bp(即na、nb、nc、nd的长度可以为3-9bp),优选的选择3bp。可以使用固定碱基充当分子标签,但需要合成多条含不同序列的固定分子标签的a3-1b,a3-2b,a5-1b以及a5-2b退火后再混合,并且a3-1b和a3-2b(a5-1b和a5-2b)的分子标签需要相互配对,即na和nb(nc和nd)互补配对。也可以使用随机碱基充当分子标签,只需要合成一条a3-1b,a3-2b,a5-1b以及a5-2b。并且a3-1b和a3-2b(a5-1b和a5-2b)的分子标签需要相互配对。以上a3-1b与a3-2b退火配对,形成接头a3badaptor。a5-1b与a5-2b退火配对,形成接头a5badaptor。两种接头浓度均为20μm-100μm,存储于-20℃冰箱。dna经过多聚核苷酸激酶pnk(终浓度:0.5-1u/μl)37℃处理15分钟,再加热到95℃反应5分钟,反应完成的dna混合物立刻插入冰中孵育5分钟。进行3’端接头连接,在同一反应管的dna混合物中加入t4dna连接酶(终浓度1u/μl~60u/μl)和a3b接头(终浓度:1-10μm),并加入连接缓冲液(反应终浓度:50-66mmtris-hcl,10mmmgcl2,1mmdtt,1-2mmatp,6-8%peg6000),加水补足后于20-30℃(根据连接酶反应条件设定)反应10-60分钟(根据连接酶浓度设定)。随后在同一反应管中按照1:2(体积比)比例加入ampurexp(beckmancoulter)磁珠或同等纯化磁珠进行纯化。在80%乙醇洗涤之后晾干直接加入去离子水,可以去除磁珠抑或保留磁珠,95℃反应5分钟。反应完成的含磁珠的dna混合物立刻插入冰中孵育5分钟。进行5’端接头连接,在同一反应管的dna混合物中加入t4dna连接酶(终浓度4u/μl~60u/μl)和a5b接头(终浓度:1-10μm),并加入快速连接缓冲液(反应终浓度:66mmtris-hcl,10mmmgcl2,1mmdtt,1-2mmatp,6-8%peg6000),加水补足后反应20-30℃(根据连接酶反应条件设定),10-60分钟(根据连接酶浓度设定)。再次进行纯化,在同一反应管中按照1:2(体积比)比例加入ampurexp(beckmancoulter)磁珠或同等纯化磁珠进行纯化。在80%乙醇洗涤之后晾干直接加入去离子水。反应完成的含磁珠的dna混合物可直接进行pcr扩增或进一步纯化去除磁珠后进行pcr扩增。按照pcr反应体系进行扩增,本环节中会引入illuminaindex。按照pcr反应说明进行扩增,扩增完成的产物按照1:1-1:1.2(体积比)比例加入加入ampurexp(beckmancoulter)磁珠或同等纯化磁珠进行纯化。在80%乙醇洗涤之后晾干直接加入去离子水,经进一步纯化后去除磁珠,产物可以进入下一步测序或捕获阶段。建库步骤如图1所示。本发明实施例也适用于mgi平台,可以使用mgiadaptor,在一实施例中,所述第一接头由互补配对的seqidno.7和seqidno.8组成,所述第二接头由互补配对的seqidno.9和seqidno.10组成,其中,seqidno.7中的ne和seqidno.8中的nf为互补配对的分子标签,seqidno.9中的ng和seqidno.10中的nh为互补配对的分子标签;所述通用引物为seqidno.11和seqidno.12,y6为index,序列如表2所示。表2另一方面,本发明实施例提供一组用于单链建库的接头组合,包括用于连接单链dna模板的3’端的第一接头,用于连接单链dna模板的5’端的第二接头;其中,所述第一接头和/或第二接头含有分子标签,且所述第一接头和第二接头为双链接头。具体第一接头和第二接头的选择上文已经详细阐述。最后,本发明实施例还提供一种用于单链建库的试剂盒,包括本发明实施例的所述的接头组合,以及通用引物、聚核苷酸激酶pnk和t4dna连接酶。本发明实施例先后进行过多次试验,现举一部分试验结果作为参考对发明进行进一步详细描述,下面结合具体实施例进行详细说明。实施例1本实施例对10ng打断的gdna进行建库测试,使用随机碱基,a3badaptor及a5badaptor各含3bp随机碱基作为分子标签,详细步骤如下:(1)lambdagdna经covariss220打断至100-400bp,亚硫酸盐处理后,取10nggdna于pcr反应管中建库,经pnk处理反应15min后加热到95℃,立即插入到冰上,静置3-5min,得到单链模板。(2)进行3’端接头连接:在同一反应管中加入30u的t4dnaligase,加入t4dnaligase缓冲液,加入a3b接头(终浓度:10μm)补足h2o,20℃反应60min。(3)随后在同一反应管中按照1:2比例加入ampurexp(beckmancoulter)磁珠或同等纯化磁珠进行纯化。在80%乙醇洗涤之后晾干加入13μl去离子水纯化取12μldna,95℃反应5分钟。反应完成的的dna立刻插入冰中孵育5分钟。(4)进行5’端接头连接:在dna中加入20ut4dna连接酶和a5b接头(终浓度:10μm),并加入连接缓冲液,20℃反应60分钟。(5)再次进行纯化,在同一反应管中按照1:2比例加入ampurexp(beckmancoulter)磁珠或同等纯化磁珠进行纯化。在80%乙醇洗涤之后晾干直接加入去离子水。反应完成的含磁珠的dna混合物直接进行pcr扩增。(6)按照以下条件进行扩增,本环节中会引入illuminaindex,引物序列如下:5’aatgatacggcgaccaccgagatctacacx6acactctttccctacacgacgctcttccgatct;5’caagcagaagacggcatacgagaty6gtgactggagttcagacgtgtgctcttccgatct;反应体系如下表3,反应条件如下表4:。表3dna23μlkapahifiuracil+readymix25primeri5(25μm)1primeri7(25μm)1total50表4(7)扩增完成的产物按照1:1.2比例加入加入ampurexp(beckmancoulter)磁珠或同等纯化磁珠进行纯化。在80%乙醇洗涤之后晾干直接加入去离子水,经进一步纯化后去除磁珠,产物进入探针捕获实验。(8)生物信息处理数据,原始测序下机文件会先去除低质量的reads和被adapter污染的片段,然后处理的文件直接用bwa软件将reads与参考基因对比,得到有reads位置信息的sam文件。进行snv和indel分析,并利用分子标签进行降噪处理。通过mpileup文件得到snv和indel信息,然后链接数据库对突变信息进行注释。所有输出被集合到report文档保存。对质控数据以及分子标签分布进行分析。图2为添加分子标签(图中的2列withbc)与不添加分子标(图中的1列nobc)签质控结果,图3为分子标签分布结果。从结果可知:通过随机碱基序列作为分子标签可以有效地降低冗余,提高回帖率,并可以有效地分析甲基化水平。以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明的保护范围之内。序列表<110>深圳易倍科华生物科技有限公司<120>一种带有分子标签的单链建库方法和接头组合、试剂盒<160>12<170>siposequencelisting1.0<210>1<211>27<212>dna<213>人工序列(artificialsequence)<400>1gacgtgtgctcttccgatctnnnnnnn27<210>2<211>21<212>dna<213>人工序列(artificialsequence)<400>2nagatcggaagagcacacgtc21<210>3<211>21<212>dna<213>人工序列(artificialsequence)<400>3acacgacgctcttccgatctn21<210>4<211>27<212>dna<213>人工序列(artificialsequence)<400>4nnnnnnnagatcggaagagcgtcgtgt27<210>5<211>63<212>dna<213>人工序列(artificialsequence)<400>5aatgatacggcgaccaccgagatctacacnacactctttccctacacgacgctcttccga60tct63<210>6<211>59<212>dna<213>人工序列(artificialsequence)<400>6caagcagaagacggcatacgagatygtgactggagttcagacgtgtgctcttccgatct59<210>7<211>27<212>dna<213>人工序列(artificialsequence)<400>7gaccgcttggcctccgacttnnnnnnn27<210>8<211>21<212>dna<213>人工序列(artificialsequence)<400>8naagtcggaggccaagcggtc21<210>9<211>21<212>dna<213>人工序列(artificialsequence)<400>9acatggctacgatccgacttn21<210>10<211>28<212>dna<213>人工序列(artificialsequence)<400>10nnnnnnnaagtcggatcgtagccatgtc28<210>11<211>24<212>dna<213>人工序列(artificialsequence)<400>11gaacgacatggctacgatccgact24<210>12<211>50<212>dna<213>人工序列(artificialsequence)<400>12tgtgagccaaggagttgytgtattgtcttcctaagaccgcttggcctccg50当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1