在植物中高效生成不携带转基因元件的突变体的CRISPR/Cas9系统和方法与流程
本发明属于植物基因编辑领域。具体来说,本发明涉及一种用于在植物中进行crispr/cas9基因编辑的基于自切割肽的构建体,以及利用该构建体高效获得经过编辑且不携带转基因元件的突变体的系统和方法。本发明还进一步提供用于在植物中进行crispr/cas9多重基因编辑的构建体,以及利用该构建体高效获得经过多重编辑且不携带转基因元件的突变体的系统和方法。
背景技术:
源自ii型原核生物适应性免疫系统的规律成簇间隔短回文重复序列(clusteredregularlyinterspacedshortpalindromicrepeats;crispr)/crispr相关的(cas)基因组编辑系统是一种精确的基因编辑体系,近年来已经被广泛用于动植物等功能基因组的研究。常规的crispr/cas9系统含有两个组分,即用于识别和匹配靶dna的单导向rna(single-guiderna,sgrna)和用于切割靶dna的cas9核酸内切酶。
在使用crispr/cas9技术对植物进行基因编辑之后,保留在植物基因组中的crispr/cas9表达元件会使经过编辑的植物落入转基因植物的范畴。实践中通常希望经过编辑的生物体最终不携带任何用于基因编辑的外源元件,换言之希望获得非转基因生物体。无论在农业上还是商业上,非转基因植物都更受青睐,更容易通过审批,更广泛得到应用,实现更高的价值。然而与动物不同,获得不含crispr/cas9表达元件的植物更为困难。在动物中,可以通过同时向受精卵中注射多个sgrna和cas9的转录本实现快速高效地获得多个位点编辑的非转基因动物。在植物中编辑通常是针对植物组织如愈伤组织或生殖细胞等进行的,例如通过农杆菌侵染的方法将crispr/cas9表达元件先导入植物中。因此,想要获得经过基因编辑但又不含crispr/cas9表达元件的植株需要待crispr/cas9将目的基因编辑后,再利用孟德尔分离定律将crispr/cas9元件分离并采用基因型鉴定来进行,这样的方法耗时耗力。
除了对于获得非转基因植物的期望,去除经编辑植物中的crispr/cas9元件在技术上也有其必要性。如果不去掉crispr/cas9元件,任其残留在植物体内,一方面难以区分变异的来源,不易获得稳定的、可遗传的突变;另一方面增加了脱靶的风险。具体而言,在通过crispr/cas9系统生成的突变体植物中仍然存在cas9和sgrna的情况下,将难以区分某个突变是遗传自上一代还是在当前世代中新产生的,而明确这一点又非常重要,因为在当前世代中新产生的突变可能是无法遗传的体细胞突变。另外,crispr系统中使用的编辑元件包括cas9和sgrna以组成型表达的方式存在,若存在于t2代或者甚至是后续世代中,可能借由sgrna错配对已经突变的靶序列进行再次编辑或对基因组其它的相似序列进行编辑生成脱靶突变。此外,若crispr/cas9元件残留在植物体内,将无法进行基因功能的互补验证,因为新转入的基因序列会被crispr/cas9元件编辑。同时,含有crispr/cas9元件残留在植物通常被认为是转基因植物,很难被批准在商业上生长应用。
因此,开发一种能够简便地筛选不携带crispr/cas9转基因元件的编辑植物特定基因的方法对于植物功能基因研究和农业生产都至关重要。
最近报道了一种基于mcherry荧光的无cas9体系,其中mcherry荧光在at2s3启动子调控下特异性表达于拟南芥的种子中,由此能够通过目测检视荧光的存在来选择t2代中不含cas9转基因的种子(gaoetal.,plantphysiology171,1794-1800,2016;yu,h.,andzhao,y.d.,methodsinmolecularbiology1917,147-154,2019)。该系统使得无cas9植物的选择变得十分便利。然而在该系统中,cas9和mcherry仍然存在于两个单独的表达盒中,由不同的启动子所驱动,因此发出的荧光强度与cas9的表达水平并不相关,无法用于预测基因编辑的效率。
platt等人(plattetal.,cell159,440-455,2014)在其关于小鼠模型的文章中提到了一种用于小鼠的cas9-p2a-egfp构建体,其通过增强绿色荧光蛋白使表达cas9的细胞可视化,并且利用了2a肽的自切割性质,但是该项研究并不涉及任何在植物中的应用。
petersen等人(petersenetal.,bmcbiotechnol19,36,2019)使用了农杆菌(agrobacterium)介导的递送系统来递送病毒复制子以表达用gfp标记的rrna/cas9,并与荧光激活细胞分选(facs)联用以提高编辑效率。其中提到了在烟草中使用以camv35s启动子驱动的cas9-2a-gfp表达盒,但是并没有明确使用的是何种来源的2a肽。由于该文章着眼于gfp和facs分选的联用,其局限于对植物的部分组织的转化和基因编辑,并没有在整个植物植株的层面讨论编辑效率和对编辑元件的移除效果,也没有研究编辑对t2代植物的影响。
因此,本领域尚需要一种适用于植物基因编辑的通用的crispr/cas9系统,以供高效、简便地生成并筛选经过有效编辑但又不携带转基因元件的突变体植物。
技术实现要素:
为了解决上述问题,本发明的发明人开发了一种用于植物基因编辑的基于自切割肽的构建体,具体来说所述构建体包含结构为“cas9-p2a-报告基因”的cas9表达盒,其中p2a代表猪捷申病毒2a肽。发明人发现p2a肽在与cas9蛋白和报告分子gfp偶联并用于植物基因编辑时,自断裂效率高达95%,显著优于其它来源的2a肽,保证了偶联的报告分子不影响cas9蛋白在基因编辑中的切割活性;同时由于cas9蛋白和报告分子处于同一表达盒中,在同一启动子调控下被一同转录,因而cas9蛋白和报告分子总是等化学计量地表达,由此报告分子gfp的荧光强度可以指示cas9蛋白水平,而cas9蛋白水平与基因编辑效率呈现一定的正相关性,因而可以通过报告分子来简便地判断第一代植物中的基因编辑效率;还可通过报告分子的表达与否来筛选第二代植物中不携带转基因元件者,以便获得不携带转基因元件的突变体。发明人通过实验验证了本发明的编辑系统作用于不同靶基因时的效力,其中包含本发明的cas9表达盒的构建体在用于植物中的crispr/cas9基因编辑时,能够高效产生并选择出经过基因编辑的t1代植物,也有助于选择出经过基因编辑且不携带转基因元件的t2代植物。在此基础上,发明人还将该构建体与基于同尾酶构建多重sgrna的策略联用,实现了高效的多重基因编辑,并证明了该构建体同样适用于同时编辑多个基因的应用。基于上述发现,完成了本发明。
因此,在第一方面,本发明涉及一种用于植物的crispr/cas9基因编辑系统的构建体,其包含cas9表达盒,所述cas9表达盒从5’到3’包含:(a)用于cas9表达盒的启动子,(b)核酸酶编码序列,(c)自切割肽编码序列,和(d)报告基因序列,其中(a)和(b)-(d)可操作地连接使得所述核酸酶编码序列和报告基因序列在所述启动子的调控下,所述核酸酶编码序列编码cas9或其具有dna切割活性的片段或变体,并且所述自切割肽编码序列编码源自猪捷申病毒的2a肽。
在一个实施方案中,所述构建体进一步包含sgrna模块,所述sgrna模块包含一个或多个sgrna表达盒,其中每个sgrna表达盒从5’到3’包含:(e)用于sgrna表达盒的启动子;和(f)针对待编辑的靶dna序列的sgrna。在所述sgrna模块包含多个sgrna表达盒时,其用于多重基因编辑。
在第二个方面,本发明涉及一种制备用于在植物中进行crispr/cas9基因编辑的构建体的方法,包括向载体中引入cas9表达盒,所述cas9表达盒从5’到3’包含:(a)用于cas9表达盒的启动子,(b)核酸酶编码序列,(c)自切割肽编码序列,和(d)报告基因序列,其中(a)和(b)-(d)可操作地连接使得所述核酸酶编码序列和报告基因序列在所述启动子的调控下,所述核酸酶编码序列编码cas9或其具有dna切割活性的片段或变体,并且所述自切割肽编码序列编码源自猪捷申病毒的2a肽。
在优选的实施方案中,本发明的构建体制备方法进一步包括向载体中引入包含一个或多个sgrna表达盒的sgrna模块,其中每个sgrna表达盒从5’到3’包含:(e)用于sgrna表达盒的启动子;和(f)针对带标记的靶dna序列的sgrna。
在优选的实施方案中,本发明的构建体制备方法中使用的包含多个sgrna表达盒的sgrna模块通过将多个sgrna表达盒组装在一起获得,所述组装包括在每个sgrna表达盒的上游和下游分别添加上游限制酶和下游限制酶,所述上游限制酶和下游限制酶为不同的限制酶且互为同尾酶,其中为所有sgrna表达盒添加的上游限制酶均相同,且为所有sgrna表达盒添加的下游限制酶均相同。
在具体的实施方案中,所述同尾酶为spei和nhei。
在第三方面,本发明涉及通过第二方面的方法获得的构建体。
在第四方面,本发明涉及在植物中进行基因编辑的方法,包括使用本发明第一方面或第三方面的构建体。
在第五方面,本发明涉及在植物中获得不携带转基因元件的突变体的方法,包括:(i)使用权利要求6或10的构建体转化植物;(ii)选择报告基因表达阳性的第一代植物,作为转化体;(iii)传代(ii)中的第一代植物获得第二代植物;和(iv)选择报告基因表达阴性的第二代植物,作为经过基因编辑且不携带转基因元件的突变体。
在第五方面优选的实施方案中,在步骤(ii)和(iv)中通过报告基因表达选择出植物之后,鉴定选出植物的靶基因序列以确认其经过编辑。在具体的实施方案中,所述鉴定通过测序进行,例如通过sanger测序进行。
在本发明各个方面的优选实施方案中,所述自切割肽编码序列是如seqidno:5所示的核苷酸序列或其编码相同多肽的突变体。在可选的实施方案中,所述自切割太编码序列是与seqidno:5的核苷酸序列具有至少75%、至少80%、至少85%、至少90%、至少95%、至少97%、至少98%或至少99%同源性,并且编码与seqidno:5编码的氨基酸序列相同的氨基酸序列的核苷酸序列。在另一个实施方案中,所述自切割肽编码序列是在严紧条件下,优选中-高严紧条件下,更优选高严紧条件下,能够与seqidno:5杂交的核苷酸序列。
在本发明各个方面的优选实施方案中,本发明的cas9是源自酿脓链球菌的cas9(spcas9)。
在本发明各个方面的优选实施方案中,本发明的cas9表达盒中包含的报告基因编码选自下组的蛋白质:萤光素酶、绿色荧光蛋白、红色荧光蛋白、蓝色荧光蛋白和黄色荧光蛋白。
在本发明各个方面的优选实施方案中,本发明的cas9表达盒中包含的启动子为在植物中组成型表达的启动子。优选地,所述启动子为拟南芥泛素10基因(atubq10)启动子或花椰菜花叶病毒(camv)35s启动子。最优先地,所述启动子为atubq10启动子。
在本发明各个方面的优选实施方案中,本发明的cas9表达盒在核酸酶编码序列的两个侧翼均包含核定位信号(nls)编码序列。
在本发明各个方面的优选实施方案中,所述构建体是基于常用于植物的载体构建的。优选地,所述载体是双元载体。
本发明的编辑系统的优势在于能够简便、高效地生成并筛选经过编辑但又不携带转基因元件的突变体植物;能够通过将本发明的表达盒与同尾酶联用实现对多基因的编辑,并获得不携带转基因元件的多重突变体植物。另一方面,通过本发明生成的不携带crispr/cas9转基因元件的植物使得互补实验(compensationexperiment)的开展成为了可能,因为如果不去掉经过编辑的植物中的crispr/cas9转基因元件,用于互补实验的野生型基因也可能被编辑,从而无法通过互补实验确立基因-表型的关联性。
附图说明
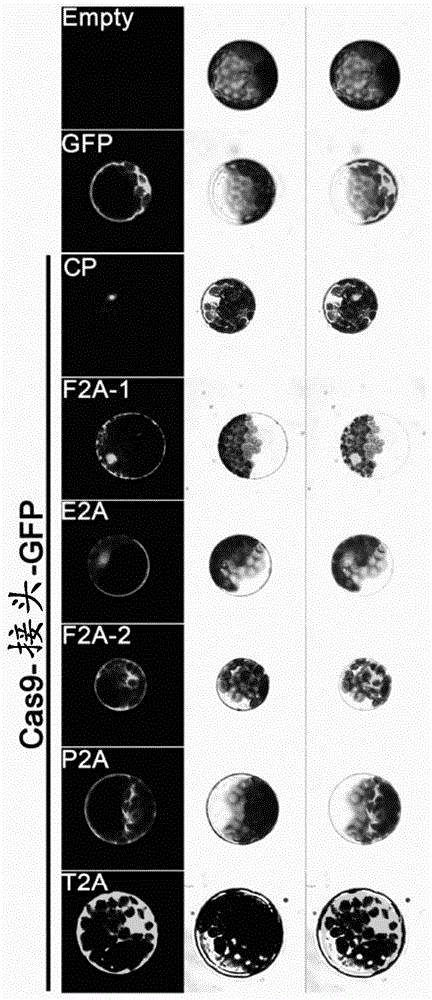
图1a-e涉及在拟南芥中高效生成无转基因元件且经过多重基因编辑的突变体的过程。图1a-c展现了实施例1中使用5种2a肽获得的实验结果。图1a是原生质体的照片,显示了构建体“cas9-接头-gfp”在拟南芥叶肉原生质体中表达时产生的荧光的分布(“接头”为对照肽或不同的2a肽)。从左到右的三列分别为:gfp通道、明场和合并的图像。由上至下分别为:空质粒(empty)、gfp对照(gfp)、对照肽(cp),以及5种不同来源的2a肽,即f2a-1、e2a、f2a-2、p2a和t2a。图1b是从图1a中所示的拟南芥叶肉原生质体中提取总蛋白,并使用gfp抗体进行蛋白质印迹分析的免疫印迹分析结果,其展示了不同2a肽的裂解效率。图1c是使用imagej作图软件量化图1b中所示的裂解效率的结果,结果来自五次独立实验,显示的数据是平均值±sem。图1d是使用同尾酶构建包含多个sgrna表达盒的sgrna模块的示意图。图1e示范了本发明的crispr/cas9体系中载体的实例。其中由atubq10启动子或双重camv35s启动子驱动cas9-p2a-gfp表达盒,并以rbcs-e9作为终止子。组装好的包含多个sgrna表达盒的多重sgrna模块借由限制酶kpni和sali被插入到双元载体中。hygr,潮霉素抗性基因;gmr,硫酸庆大霉素抗性基因;kanr,卡那霉素抗性基因;smr,壮观霉素抗性基因。hygr和kanr属于pcambia1300-cas9-p2a-gfp双元载体构建体,而gmr和smr属于pjim19-cas9-p2a-gfp双元载体构建体。
图2a-d图示了选择不携带转基因元件的植物的流程,如实施例3中所示。图2a是本发明的编辑系统的示例性工作流程图。其中通过农杆菌介导的转化将具有靶sgrna表达盒和cas9-p2a-gfp的构建体递送到拟南芥中,将来自转化植物的种子播种在具有抗生素(图中为潮霉素或硫酸庆大霉素,本领域技术人员应该理解,应该基于选用的载体在选择培养基中使用相应的抗生素)的ms平板上。挑出具有高gfp表达的t1转基因植物,而选择没有gfp表达的t2转基因植物(不携带转基因元件的植物)。进行基因型鉴定以确定t1植物中靶基因的存在和t2中不携带转基因元件者。图2b显示了具有或不具有gfp信号的t2转基因植物的分离。使用图1d中所述的方法组装了四个sgrna,两个靶向bzr1,另外两个靶向bes1,并克隆到双元载体p35s:cas9-p2a-gfp(gent)中。将获得的构建体转入野生型中以编辑bzr1和bes1。使用共聚焦显微镜拍摄3天大的光照生长幼苗。每个小图右下角的数字是观察到的植物数量。图2c是针对cas9-p2a-gfp表达盒的琼脂糖凝胶分析。从图2b的植物中随机选择三株没有gfp信号的t2植物和九株具有gfp信号的t2植物,使用cas9的引物扩增pcr产物,并通过琼脂糖凝胶进行分析,以确认基于荧光的选择的准确性。图2d是在没有gfp表达的代表性t2转基因植物中,bzr1和bes1靶位点的dna序列(编号10-19)。bzr1基因座1和bzr1基因座2是两个包含bzr1sgrna的基因组位点,而bes1基因座1和bes1基因座2是两个包含bes1sgrna的基因组位点。sgrna序列带有下划线。加号表示插入,减号表示缺失。
图3a-d涉及使用cas9-p2a-gfp编辑基因组的过程,如实施例2中所示。图3a和3c为植物照片,显示了转基因植物的荧光强度与编辑bri1导致的表型相关,其中最左侧一列是4天大的t1植物幼苗照片,第二列是4天大的t1植物幼苗的荧光照片(使用leica荧光立体镜拍摄),剩余的右侧部分是在土壤中生长数周后的植株照片。cas9-p2a-gfp由双重camv35s启动子(a)或atubq10启动子(c)驱动。图3b和3d是图3a和3c中代表性的t1转基因品系的sanger测序结果。用下划线标出bri1sgrna序列,其中下划线之后方框中的三个核苷酸agg为原始间隔子相邻基序(pam)序列。
图4图示了用于基因组多重编辑和选择不携带转基因元件的经多重编辑植物的质粒的构建流程。将sgrna的引物褪火并使用bbsi插入atu6-26和支架之间。使用spei/sali和nhei/sali组装多个sgrna表达盒。用kpni和sali消化串联的sgrna表达盒,然后克隆到带有cas9-p2a-gfp表达盒的双元载体中。cas9-p2a-gfp由双重35s启动子或atubq10启动子驱动,并由rbcs-e9终止子终止。
图5展示了用于sgrna的载体patu6-26-m的序列。第一段斜体加粗的序列为拟南芥atu6-26启动子,第二段斜体加粗的序列为sgrna支架。各个限制酶位点带有下划线并标注了相应的限制酶。同时还显示了相应的sgrna引物序列。
图6a-d展示了在具有高gfp表达的t1转基因植物中有效地编辑了多个目标位点。图6a显示了通过本发明的系统进行多重基因组编辑之后产生的t1转基因植物中的gfp信号。该多重编辑使用了四个sgrna表达模块,两个用于靶向bzr1,另外两个用于靶向bes1,由同尾酶组装在一起并插入到p35s:cas9-p2a-gfp(hyg)中。将最终的双元载体转移到农杆菌中,并通过浸花递送到col-0植株中。通过lsm710共聚焦显微镜从5天大的光照生长幼苗中收集荧光信号。显示了两个具有较强gfp荧光的代表性幼苗(#1和#10)。图6b-d是图6a中的t1转基因品系的dna测序图。基因座1和基因座2是bzr1sgrna所针对的基因组基因座,而基因座3和基因座4是bes1sgrna所针对的基因组基因座。sgrna是带有起始位点g的20bp序列,以下划线显示。
图7a-b展示了在t2植物中检测cas9-p2a-gfp。图7a显示了p2a肽在用于编辑拟南芥bzr1和bes1基因时的自切割效率。具有gfp荧光的t2转基因植物来自图2b和2c中所述的t1品系(#1和#10)。从5天大的光照生长幼苗中提取总蛋白,并使用gfp抗体通过免疫印迹进行分析。星号表示一个非特异性的条带;rpn6为上样对照。图7b是在没有gfp荧光的植物中对cas9-p2a-gfpdna表达盒进行的pcr和琼脂糖凝胶分析。提取没有gfp荧光的t2植物的dna并使用针对cas9的特异性引物扩增。“c”表示阳性对照。
发明详述
除非另有说明,否则本文公开的一些方法的实践采用植物学、生物化学、化学、分子生物学、细胞生物学、遗传学和重组dna的常规技术,这些技术在本领域的技术范围内。参见例如sambrook和green,molecularcloning:alaboratorymanual,4thedition(2012)等。
术语“约”或“近似”意指在本领域普通技术人员确定的特定值的可接受误差范围内,这将部分取决于如何测量或确定该值,即,测量系统的局限性。例如,根据本领域的实践,“约”可以表示在1或大于1的标准偏差内。或者,“约”可表示给定值的最多20%,最多10%,最多5%或最多1%的范围。或者,特别是对于生物系统或过程,该术语可以表示数值的一个数量级即10倍,优选地在5倍内,更优选地在2倍内。在申请和权利要求中描述特定值的情况下,除非另有说明,否则应当将术语“约”理解为在该语境下可接受的误差范围内。
如本文所用的术语“基因”是指核酸(例如dna,例如基因组dna和cdna)及其相应的编码rna转录物的核苷酸序列。如本文所用,关于基因组dna的术语包括插入的非编码区以及调节区,并且可包括5'和3'末端。在一些用途中,该术语包括转录序列,包括5'和3'非翻译区(5'-utr和3'-utr),外显子和内含子。在一些基因中,转录区域将包含编码多肽的“开放阅读框”。在该术语的一些用途中,“基因”仅包含编码多肽所必需的编码序列(例如,“开放阅读框”或“编码区”)。在一些情况下,基因不编码多肽,例如核糖体rna基因(rrna)和转移rna(trna)基因。在一些情况下,术语“基因”不仅包括转录序列,而且还包括非转录区域,包括上游和下游调节区,增强子和启动子。基因可以指生物基因组中其天然位置中的“内源基因”或天然基因。基因可以指“外源基因”或非天然基因。非天然基因可以指通常不在宿主生物体中发现但通过基因转移引入宿主生物体的基因。非天然基因也可以指不在生物体基因组中的天然位置的基因。非天然基因还可以指天然存在的核酸或多肽序列,其包含突变,插入和/或缺失(例如,非天然序列)。
如本文所用的术语“核苷酸”通常是指碱-糖-磷酸盐组合。核苷酸可包含合成核苷酸。核苷酸可包含合成的核苷酸类似物。核苷酸可以是核酸序列的单体单元(例如脱氧核糖核酸(dna)和核糖核酸(rna))。术语核苷酸可包括核糖核苷三磷酸腺苷三磷酸(atp),尿苷三磷酸(utp),三磷酸胞嘧啶(ctp),三磷酸鸟苷(gtp)和脱氧核糖核苷三磷酸如datp、dctp、ditp、dutp、dgtp、dttp或其衍生物。这些衍生物可包括,例如,[αs]datp,7-脱氮-dgtp和7-脱氮-datp,以及赋予含有它们的核酸分子核酸酶抗性的核苷酸衍生物。本文使用的术语核苷酸可以指双脱氧核糖核苷三磷酸(ddntp)及其衍生物。双脱氧核糖核苷三磷酸的说明性实例可包括但不限于ddatp,ddctp,ddgtp,dditp和ddttp。核苷酸可以通过众所周知的技术进行未标记或可检测标记。标记也可以用量子点进行。可检测标记可包括例如放射性同位素,荧光标记,化学发光标记,生物发光标记和酶标记。
术语“多核苷酸”,“寡核苷酸”和“核酸”可互换使用,指任何长度的聚合形式的核苷酸,脱氧核糖核苷酸或核糖核苷酸,或其类似物,可以是单链、双链或多链形式。多核苷酸对细胞可以是外源的或内源的。多核苷酸可以存在于无细胞环境中。多核苷酸可以是其基因或片段。多核苷酸可以是dna。多核苷酸可以是rna。多核苷酸可以具有任何三维结构,并且可以执行已知或未知的任何功能。多核苷酸可包含一种或多种类似物(例如改变的主链,糖或核碱基)。
术语“同源性”是指两个或多个核苷酸序列之间的序列相似性的百分比。本领域技术人员知晓可以使用工具性计算机算法如blast以其默认参数来比对和确定核苷酸序列之间的同源性。
术语“严紧条件”在本文中指杂交实验进行的条件,其可以为低严紧条件、中严紧条件或高严紧条件,优选为高严紧条件。举例而言,“低严紧条件”可指在30℃下使用5xssc、5xdenhardt溶液、0.5%sds和52%甲酰胺的条件;“中严紧条件”可指在40℃下使用5xssc、5xdenhardt溶液、0.5%sds和52%甲酰胺的条件;“高严紧条件”可指在50℃下使用5xssc、5xdenhardt溶液、0.5%sds和52%甲酰胺的条件。本领域技术人员可以理解的是在更高的温度下能够相互杂交的核苷酸序列之间的同源性也更高。
“crisp-cas9技术”是一种基因定点编辑技术,被广泛地用于植物基因编辑。该系统主要包含切割dna的cas9蛋白和一段引导rna。引导rna与cas9结合,并通过其上游大约20个核苷酸与目的dna序列进行识别与配对,将cas9带到待切割区域并切割双链dna。cas9蛋白可以来源于多种不同的物种。可以是源自选自下组的微生物的cas9蛋白:双歧杆菌属菌种(bifidobacteriumspp.)、坎德里亚属菌种(kandleriaspp.)、乳杆菌属菌种(lactobacillusspp.)、明串珠菌属菌种(leuconostocspp.)、酒球菌属菌种(oenococcusspp.)、欧陆森氏菌属菌种(olsenellaspp.)、片球菌属菌种(pediococcusspp.)、葡萄球菌属菌种(staphylococcusspp.)如金黄色葡萄球菌(s.aureus)、链球菌属菌种(streptococcusspp.)如酿脓链球菌(s.pyogenes)和魏斯氏菌属菌种(weissellaspp.)。在优选的实施方案中,所述cas9蛋白是源自酿脓链球菌的cas9即spcas9或源自金黄色葡萄球菌的cas9即sacas9。在本文中还可以使用cas9蛋白的具有cas9的dna切割活性的变体或片段。本发明的“cas9蛋白的编码序列”指编码cas9蛋白或其具有cas9的dna切割活性的变体或片段的核苷酸序列。在进一步的实施方案中,所述cas9蛋白的编码序列可以经过密码子优化以在待编辑的植物中更好的表达。在具体的实施方案中,所述cas9序列来自本领域已知的或可商购的载体,如feng等人(fengetal.,cellresearch23,1229-1232,2013)的文献中使用的hspcas9。
“转基因元件”是指使受体生物产生可预期的、定向的遗传改变的相关外源的dna片段。在本发明中主要包含cas9蛋白的启动子、cas9-p2a-gfp基因编码区、转录终止元件、sgrna的启动子、转录区以及来源于载体的其它相关序列。在本发明的上下文中,“转基因元件”有时与“基因编辑元件”可互换使用。
“sgrna”是指用于基因编辑的一段引导rna,包括成熟的crrna:tracrrna,sgrna通过与cas9集合并将其运送至目的dna处,通过sgrna与目的dna互补配对从而使得cas9蛋白切割dna双链。本领域技术人员知晓根据待编辑的靶dna设计sgrna的原理和方法。本发明中针对示例性的靶基因给出了sgrna的实例,但不应将此理解为对本发明的限制。
“自切割肽”,具体而言“2a肽”是能够自我剪切的肽。2a序列通常编码18-22个氨基酸的短肽,其介导自其3’末端附近的甘氨酰-脯氨酰肽键开始的翻译“跳跃”(donnellyetal.,journalofgeneralvirology82,1013-1025,2001;defelipeetal.,biotechnologyjournal5,213-223,2010)。2a肽能够高效自剪切并且其侧翼蛋白能够进行化学计量表达,因此已经被广泛用于动物细胞中。然而,各种2a肽在植物细胞中的切割效力尚不明确。kim等人(kimetal.,plosone6,e18556,2011)的文章中提到,尽管2a肽可用于构建多顺反子或双顺反子载体,且相对于常用于该目的的ires(内部核糖体插入位点)而言更小、切割更高效,但是其并没有得到广泛使用,其中一个阻碍在于迄今为止报道过的多种来源的2a肽在不同的背景下切割效率各异。kim等人在人细胞系、斑马鱼和小鼠中比较了四种不同来源的2a肽的效力,但是并未在植物中开展相关的研究,也没有涉及将2a肽与crispr-cas9系统联用或是与cas9融合。
“报告基因”是指一类在特定情况下得到表达,由此使得它们产生易于检测且实验材料原本不会产生的性状的基因。报告基因通常可用于指示与之可操作地连接的其他表达元件的表达。在本发明的实施方案中,由于报告基因和核酸酶编码基因如cas9编码基因在同一启动子的调控下,因此报告基因产生的可检测信号可用于判断cas9的表达。在一些实施方案中,报告基因编码报告蛋白,如某种酶,包括但不限于半乳糖苷酶、萤光素酶或碱性磷酸酶。在一些实施方案中,用于指示核酸酶表达的报告基因编码荧光蛋白,如绿色荧光蛋白(gfp)、红色荧光蛋白(rfp)、蓝色荧光蛋白(bfp)和黄色荧光蛋白(yfp)。优选地,在本发明的构建体中使用编码荧光蛋白的报告基因,特别是使用编码gfp的报告基因。
“表达盒”在本发明的上下文中指一段dna,通常是某个载体或构建体的一部分或者将会被引入载体或构建体中,其通常包含一个或多个待表达的编码序列和调控所述编码序列表达的控制序列如启动子。构建体中可以包含一个或多个表达盒,例如本发明的构建体可以包含一个用于表达cas9的表达盒,以及一个或多个sgrna表达盒。在本发明的上下文中,“cas9表达盒”通常指包含启动子和cas9的表达盒,而“sgrna表达盒”通常指包含启动子和sgrna的表达盒。
“sgrna模块”在本发明的上下文中指可以包含一个或多个sgrna表达盒的序列段。
“载体”指在基因工程重组dna技术中将dna片段(目的基因)转移至受体细胞的一种能自我复制的dna分子。三种最常用的载体是细菌质粒、噬菌体和动植物病毒。在本发明中优选使用双元载体。可用于本发明的载体包括但不限于pjim系列载体如pjim19等、pcambia系列载体如pcambia1301等。
“构建体”或“表达构建体”在本发明的上下文中通常指以载体为基础,通过引入外源序列片段而用于特定功能或目的的一段dna,其通常为环状,也可为线性。
“启动子”启动子是rna聚合酶识别、结合和开始转录的一段dna序列,它含有rna聚合酶特异性结合和转录起始所需的保守序列,多数位于结构基因转录起始点的上游,其本身通常不被转录。已经知晓多种适合于在植物中使用的启动子,包括组成型启动子、诱导型启动子、特异性启动子如组织特异性或发育阶段特异性启动子。在本发明的用于crispr/cas9的构建体中通常会包含多个启动子,例如用于驱动cas9表达盒的启动子,和用于驱动sgrna表达盒的启动子。
在本发明的实施方案中,优选使用“组成型启动子”,即能够在全部组织中发挥功能并启动持续表达的启动子。常用于植物的组成型启动子包括,例如,泛素启动子如玉米泛素启动子、拟南芥泛素启动子、肌动蛋白启动子、花椰菜花叶病毒启动子(camv35s)等。然而,本领域技术人员也知晓,尽管多种常用的组成型启动子是本领域已知的,但是对于不同的植物、不同的组织、不同的被驱动基因和不同的应用中,启动子的表现会有所差异,并且在不通过大量工作的情况下各种启动子的适用性通常是难以预见的。在本发明优选的实施方案中,启动子是拟南芥泛素10基因启动子(atubq10)或花椰菜花叶病毒35s启动子(camv35s),更优选拟南芥泛素启动子。相应地,在本发明的上下文中,“atubq10”或“ubq10”均指拟南芥泛素10基因启动子,除非另行说明。
“可操作地连接”指两个或多个元件的在功能上相关的一种排列方式,这种排列方式使得各个元件都可以正常发挥功能,且其中至少一个元件(如转录调节元件,例如启动子、终止子、增强子)可以调节其余元件中的至少一个(如编码序列、开放式阅读框)的功能。例如,当启动子与编码序列可操作地连接时,说明该启动子能够调控该编码序列的转录水平。“可操作地连接”并不必然指两个或多个所述元件直接相邻。
本发明的构建体、系统和方法尤其适用于植物中的基因编辑。在本发明的上下文中,“植物”包括单子叶植物和双子叶植物,其中单子叶植物如油棕、甘蔗、香蕉、玉米、小麦、黑麦、大麦、燕麦、水稻、小米或高粱,所述双子叶植物如拟南芥、大豆、土豆、油菜、花生或向日葵。本发明的植物可以是经济作物、模式植物、花卉、树木。
本发明的构建体可以通过多种常规的转基因技术引入到植物中,例如可以通过粒子轰击,可以通过电穿孔和显微注射进行的原生质体转化,也可以通过农杆菌介导的转化。
本发明的有益效果包括:能够简洁、便利地挑选出编辑效率较高的植株,能够简洁、便利地获得并筛选出不携带转基因元件的植株,还可以用于植物中的多重基因编辑。
在本发明进一步的实施方案中,本发明涉及用于进行多重基因编辑的构建体、系统和方法。具体来说,将本发明第一方面的构建体与一对同尾酶进行联用,所述同尾酶用于多个sgrna的组装。
“同尾酶”、“同尾限制酶”和“同尾限制性内切酶”在本发明的上下文中可以互换使用,指一类限制性核酸内切酶,它们识别并切割不同的靶序列,但切割之后产生序列相同的粘性末端。因此,由互为同尾酶的两种或多种酶消化产生的dna片段能够通过其粘性末端之间的互补作用彼此连接起来。在将用两个互为同尾酶的不同限制酶切割出的dna片段通过对应的粘末端连接之后,由于粘末端序列所形成双链序列的5'和3'的核苷酸分别来自不同的限制酶识别位点,因此连接后的片段的相应位置不再构成原先进行切割的两个限制酶中任何一个的识别位点,不能被原来的限制性内切酶所识别。以下示例性地列出了一些同尾酶组,每组中包括两种或更多种限制酶,同在一组的限制酶之间两两互为同尾酶。
表1.同尾酶的实例
在本发明中,利用同尾酶来连接多个sgrna的表达盒,从而形成可用于多重基因编辑的构建体。可用于本发明的同尾酶可选自包括但不限于选自如下任意一组中的:ecori和mfei;psti和sbfi;xbai、spei、nhei中的任意两个;bamhi和bglii;以及xhoi和sali。在本发明优选的实施方案中,使用spei和nhei。
实施例
为了更全面地理解和应用本发明,下文将参考实施例和附图详细描述本发明,所述实施例仅是意图举例说明本发明,而不是意图限制本发明的范围。本发明的范围由后附的权利要求具体限定。
实施例1.不同来源2a肽的效力
为了获得一种在植物中能够通过视觉检视测定功能性cas9丰度的crispr体系,本发明的发明人探索了多种适用于植物的自切割肽,用于连接cas9和报告蛋白。源自小核糖核酸病毒的2a肽被证明在超过100种动物细胞和组织中展现出自切割性质(kimetal.,plosone6,e18556,2011),为了测试2a肽是否在植物中也能够起到类似作用,以及何种2a肽在植物中具有最好的效果,测试了如下五种不同的2a肽:源自口蹄疫病毒的两种2a肽(f2a-1和f2a-2,编码核苷酸序列分别为seqidno:2和seqidno:4),源自马甲型鼻炎病毒的2a肽(e2a,编码核苷酸序列为seqidno:3),源自东亚细亚病毒(thoseaasignavirus)的2a肽(t2a,编码核苷酸序列为seqidno:6)和源自猪捷申病毒的2a肽(p2a,编码核苷酸序列为seqidno:5)。另外还使用了核苷酸序列如seqidno:1所示的肽作为对照肽(cp),该对照肽没有2a肽在体内发生自切割的能力,长度与上述2a肽类似。将上述五种2a肽和对照肽(cp)分别用来连接cas9和gfp,然后将各种cas9-2a-gfp或cas9-cp-gfp表达盒连接至组成型表达载体中。其中将两个核定位信号(nls)肽分别融合至cas9的c末端和n末端。通过共聚焦显微镜和蛋白质印迹分析在原生质体中评估这些2a肽的切割效率。下文中将详细描述实验过程。
植物材料和生长条件
在本实施例中使用了野生型拟南芥哥伦比亚-0型(arabidopsisthalianacolumbia-0)。将种子用15%naclo灭菌10分钟,然后用灭菌的ddh2o清洗5次。使灭菌的t1代种子在ms平板(4.4g/lmurashigeskoog粉末、1%蔗糖、8g/l琼脂,ph5.8)上生长,所述平板含有50mg/l潮霉素或200g/l硫酸庆大霉素。
将t2代种子播种在不含抗生素的ms平板上。将铺有种子的平板置于4℃层析处理4-5天之后,将平板放置于22℃光照培养箱中在80μmol·m-2·s-1持续白光下培养。将七日龄幼苗转移至土壤中,在长日照条件下生长(60-80μmol·m-2·s-1白光,16h光照22℃/8h黑暗18℃循环)。
质粒构建
如下制备含有以2a肽融合的cas9和gfp的构建体用于原生质体转化。
gfp序列的克隆
首先,将gfp从pjim19(bar)-gfp(sunetal.,proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica113,6071-6076,2016)扩增出来,利用spei和saci将其克隆至pjim19(gent)中以生成pjim19(gent)-gfp。从phee401e(wangetal.,genomebiology16,144,2015)克隆rbcs-e9终止子并插入pjim19(gent)-gfp的saci和ecori位点之间以取代nos终止子,生成pjim19(gent)-gfp-rbcs-e9t。
2a肽的克隆
合成前述五种2a肽和对照肽(cp)的核苷酸序列以及kpni和spei限制酶位点,褪火并克隆至用kpni和spei消化的pjim19(gent)-gfp-rbcs-e9t以生成pjim19(gent)-接头-gfp-rbcs-e9t。其中“接头”即为cp、f2a-1、e2a、f2a-2、p2a或t2a的核苷酸序列中的任意一种。
cas9的克隆
然后,从p35s-cas9-sk(fengetal.,2013,同上)扩增了不含终止密码子的cas9,并插入pbs载体中以生成pbs-cas9(不含终止密码子)。
cas9-2a-gfp表达盒的组装
将“接头-gfp-rbcs-e9t”片段从pjim19(gent)-接头-gfp-rbcs-e9t载体扩增出来,并克隆至pbs-cas9(不含终止密码子)中以生成pbs-cas9-接头-gfp-rbcs-e9t。
最后,通过将双重camv35s启动子上游的一个xhoi位点缺失并在hindiii和sali之间加入nhei位点来修饰p35s-cas9-sk。
将修饰的p35s-cas9-sk和pbs-cas9-接头-gfp-rbcs-e9t二者使用xhoi和ecori消化以生成最终的质粒p35s-cas9-接头-gfp-rbcs-e9t。全部的引物序列参见表7。
sgrna表达模块构建
为了生成多个sgrna的表达模块,将patu6-26-sk载体(feng等,2013,同上)通过缺失sgrna支架(scaffold)下游的spei位点并在xhoi和sali之间加入nhei位点来修饰,得到patu6-26-m(图5和seqidno:7显示了该载体关键部分的结构,即用于构建多个sgrna串联体的atu6-26-scaffold模块)。将多个sgrna分别插入patu6-26-m的bbsi位点,得到如seqidno:39的序列。使用sali和一对同尾限制酶spei/nhei组装起多个sgrna表达盒,如图1d所示。
基因组编辑质粒的构建
为了产生最终的基因组编辑质粒,使用kpni和sali消化由同尾限制酶组装的多个sgrna表达模块,然后如上所述将其插入含有cas9-p2a-gfp的双元载体中。
全部的引物序列参见实施例之后的表7。
叶肉原生质体的分离和转染
将三至四周龄的拟南芥通过以下所述的程序用于原生质体分离(yoo等,natureprotocol2,1565-1572,2007)。将叶片切成0.5-1mm的条状叶片,并立即在15ml的酶溶液(1.87%纤维素酶r10、0.32%离析酶r10、0.4m甘露醇、20mmmes,ph5.7、20mmkcl、10mmcacl2、5mmβ-巯基乙醇和0.1%bsa)中消化3小时。将消化的产物用15ml预冷的w5溶液(154mmnacl、125mmcacl2、5mmkcl和2mmmes,ph5.7)稀释,并通过70μm尼龙网过滤。通过以1400rpm离心2分钟使原生质体沉淀,并用10mlw5溶液洗涤,然后再次离心。使用10ml预冷的w5缓冲液重悬原生质体,在冰上温育30分钟,然后弃去上清液。将mmg缓冲液(0.4m甘露醇、15mmmgcl2和4mmmes,ph5.7)添加到原生质体中,使原生质体的浓度为105-106细胞/ml。使用2ml圆底微离心管进行dna转染。将200μl原生质体与20μg质粒和220μlpeg溶液(40%peg4000、0.2m甘露醇和0.1mcacl2)充分混合。在室温下温育5分钟后,通过添加1ml预冷的w5缓冲液淬灭转化反应,并通过在1200rpm下离心2分钟收集转染的原生质体。接下来,将转染的原生质体用100μlw5溶液重悬,并转移到六孔培养板中,该培养板预先涂有1mlwi溶液(0.5m甘露醇、20mmkcl、5%胎牛血清和4mmmes,ph5.7)。将该培养板用卫生纸覆盖并放置在培养箱中16小时,然后在显微镜下观察或提取总蛋白。
蛋白质提取和蛋白质印迹
对于原生质体,通过以100g离心1分钟收集培养的原生质体。加入20μl变性缓冲液(8m尿素、100mmnah2po4、100mmtris-hcl,ph8.0、1mmpmsf和1×蛋白酶抑制剂)和5μl5×sds上样缓冲液(50%甘油、10%sds、0.1%溴酚蓝、10mmdtt,5%β-巯基乙醇和250mmtris-hcl,ph6.8)提取总蛋白。将经过充分混合的原生质体和缓冲液在100℃下煮沸10分钟。
对于植物,收获7日龄的光照生长(light-grown)幼苗,并在液氮中研磨成粉末。使用变性缓冲液提取总蛋白。以13,000rpm离心10分钟后,将含有总蛋白质的上清液与5×sds上样缓冲液混合,然后在100℃煮沸10分钟。
蛋白样品在8%sds-page凝胶上分离,并转移到pvdf膜上。用5%牛奶封闭后,将膜与抗gfp、抗rpn6或抗rpt5抗体一起温育。之后,将膜洗涤3次并与相应的二抗一起温育,再洗涤3次,然后用ecl引物(gehealthcare)检测。
结果
从图1a中可以看出,与用cas9-cp-gfp转化时荧光均定位于细胞核中的情况不同,用cas9-2a-gfp转化的原生质体中的某些gfp荧光明显位于细胞质中,表明gfp蛋白已经成功从cas9上裂解下来。此外,在所有这些2a肽中,p2a表现出最高的切割效率,其在原生质体中显示出大约95%的自切割效率(参见图1b和1c)。由于cas9和gfp的表达水平相关,并且通过p2a的高效自我切割,gfp荧光强度可以很好地反映cas9的丰度,同时gfp又不会中断cas9的功能。因此,选择p2a来融合cas9和gfp进行后续实验。
实施例2.不同启动子的效力
启动子是crispr系统中可以影响编辑效率的关键因素之一。有一些先前的研究显示出35s启动子调控的cas9系统效率不高,并且引发的体细胞突变无法遗传。
为了选择驱动cas9-p2a-gfp的有效启动子并测试gfp强度是否与基因编辑效率相关,将拟南芥bri1(brassinosteroid-insensitive1)基因选为靶基因研究了不同的启动子,包括双重camv35s启动子和拟南芥泛素10(ubq10)基因启动子,并测试了不同的载体,包括pjim19和pcambia1300。
bri1基因编码一种具有激酶活性的受体蛋白,其通过与油菜素甾醇结合调节生长发育的信号级联反应。如果对bri1基因进行编辑使其无法正常发挥功能,那么经过基因编辑的植株会产生严重矮化的表型,并导致不育。
为了测试两种启动子的效力,使用两种载体共生成了四种不同的构建体,具体构建方式如下。
使用hindiii、stui、spei、kpni和ecori的序列修饰双元载体pjim19(gent)(seqidno:40)以取代camv35s启动子和nos终止子,从而生成pjim19(gent)-m载体。使用ubq10nheif和ubq10xhoir引物从拟南芥基因组dna克隆ubq10启动子。用nhei和xhoi消化ubq10启动子和p35s:cas9-p2a-gfp-rbcs-e9t载体,并进行组装从而得到pubq10-cas9-p2a-gfp-rbcs-e9t载体。将ubq10-cas9-p2a-gfp-rbcs-e9t克隆到ecori和kpni消化的pjim19(gent)-m中,生成双元载体pjim19-proubq10:cas9-p2a-gfp-rbcs-e9t,缩写为pubq10:cas9-p2a-gfp(gent)。
以类似的方式从p35s-cas9-p2a-gfp-rbcs-e9t中消化2×35s-cas9-p2a-gfp-rbcs-e9t模块,并将其克隆到pjim19(gent)-m中以获得双元载体pjim19-2×35s-cas9-p2a-gfp-rbcs-e9t,缩写为p35s:cas9-p2a-gfp(gent)。
另外,将cas9-p2a-gfp表达盒插入具有潮霉素抗性的pcambia1300的修饰版本pcambia1300-m(seqidno:41)中,以产生另外两个双元载体pubq10:cas9-p2a-gfp(hyg)和p35s:cas9-p2a-gfp(hyg)。
由于经常使用双重camv35s启动子来调控cas9的表达,因此首先将bri1sgrna表达盒克隆到由双重camv35s启动子驱动的携带cas9-p2a-gfp的双元载体p35s:cas9-p2a-gfp(gent)中,然后将其通过农杆菌(agrobacterium)递送到野生型中。在60株t1转基因植物中,5株具有强gfp荧光的幼苗均显示出生长的严重缺陷和不育表型,其中三株如图3a所示。相比之下,在没有明显gfp荧光的t1植物中,只有3株表现出半矮表型(参见表2)。通过sanger测序,进一步证实了所有5个具有高gfp表达的品系都编辑了靶基因座并导致了嵌合突变(如图3b和表2)。
然后,将bri1sgrna表达盒插入由拟南芥泛素10(atubq10)基因启动子驱动的带有cas9-p2a-gfp的双元载体pubq10:cas9-p2a-gfp(gent)中。在47个t1转基因幼苗中,有10株植物表现出较强的gfp荧光,并且它们在土壤中生长时全部表现出类似于bri1突变体的严重矮化表型(参见图3c和表2)。测序数据显示10株幼苗中有3株是纯合突变体,即(图3d和表2)。出乎意料的是,没有明显的gfp荧光的12株t1幼苗也表现出严重矮化的表型,其中两株是纯合突变体。这些结果表明,对于camv35s启动子和ubq10这两种启动子而言,具有高gfp荧光的t1转基因植物的编辑效率都比没有明显荧光的t1转基因植物高(表2)。结果还表明ubq10在驱动cas9-p2a-gfp编辑基因组方面优于35s启动子。
表2.使用双重camv35s启动子(2x35s)或泛素10启动子(ubq10)以pjim19载体为基础进行编辑的t1代植物的表型和基因型汇总
实施例3.使用同尾酶进行多重编辑
为了探索本发明的体系是否适用于多重基因编辑,将本发明的构建体与一种基于同尾限制性内切酶的组装方法联用。由于本实施例中的实验是在实施例2的实验之前进行的,因此使用的仍然是35s启动子。
为了生成多个sgrna的表达盒,将patu6-26-sk载体(feng等,2013,同上)通过缺失sgrna支架(scaffold)下游的spei位点并在xhoi和sali之间加入nhei位点来修饰,得到patu6-26-m(图5)。
将多个sgrna插入patu6-26-m的bbsi位点。使用sali和一对同尾限制酶spei/nhei组装起多个sgrna表达盒,如图1d所示。
为了产生最终的基因组编辑质粒,使用kpni和sali消化由同尾限制酶组装的多个sgrna表达盒,然后如上所述将其插入含有cas9-p2a-gfp的双元载体中。
使sgrna的引物褪火,并通过bbsi位点插入atu6-26和支架之间(图4和图5)。如图4所示,本实施例的方法利用了spei和nhei这一对同尾酶,spei和nhei在切割双链dna过后能够产生相同的粘末端ctag。通过使用spei/sali和nhei/sali消化并连接后,将多个sgrna表达盒连续组装在一起(图1d)。然后,使用kpni和sali消化包含多个串联sgrna的表达盒并将其插入如实施例1和2中构建的包含由双重camv35s启动子或拟南芥泛素10基因(atubq10)启动子驱动的cas9-p2a-gfp的双元载体中(图1e;图4和2)。接下来,使用这些构建体来转化植物,再将转化的种子在含抗生素的ms平板上生长,然后通过荧光立体镜或显微镜挑选出高gfp表达的幼苗。具有强gfp荧光的t1转基因幼苗被转移到土壤中以产生后代。最后,在t2代转基因植物中选择不表达gfp的个体,并对其进行基因型鉴定,最终获得了不携带转基因元件并且由多个基因得到编辑的突变体(参见图3a)。使用本发明的这种多重编辑构建体,通过检查转基因植物中的荧光强度轻松检测了cas9的蛋白质水平,同时实现了对多个基因的编辑。通过选择具有高gfp荧光的t1植物和没有荧光的t2植物,即可简便地获得不含cas9的多重突变体。具体结果如实施例4中所述。
实施例4.基于简单的荧光检测来选择不携带转基因元件的多重突变体
本发明的体系可以利用cas9-p2a-gfp构建体来生成经过多重基因编辑且不含转基因元件的植物。
首先,使用同尾限制性内切酶组装多个sgrna表达元件,并使用kpni和sali将组装好的多个sgrna表达盒插入包含cas9-p2a-gfp的双元载体中(如实施例3中所述,参见图4)。在t2转基因植物中,选择没有gfp表达的植物并进行基因分型以获得不携带转基因元件且经过多重基因编辑的突变体(流程如图2a所示)。
为了测试本发明的crispr系统的效率,选择了bzr1和bes1作为靶标用于产生不携带转基因元件的突变体。设计了四个sgrna(如图6b所示,引物参见表7),两个用于bzr1,两个用于bes1,并将它们插入如实施例2所构建的p35s:cas9-p2a-gfp(hyg)双元载体。对野生型(col-0)植物进行了转化。从野生型背景下的62株t1植物中选出了4个高gfp表达的幼苗,图6a中显示了其中2个代表性幼苗的荧光,下表3中总结了t1代植物中带有荧光的植物数,和这些荧光植物中包含的突变数量。
表3.t1植物中的荧光和基因编辑效率
在没有明显gfp表达的代表性幼苗中,所有四个靶位点均未被编辑(如图6b),而在具有高gfp表达的植物中被有效编辑(图6c和6d)。在这两个具有高gfp表达的代表性t1植物的t2子代中,gfp和非gfp植物的分离比接近3:1,表明为单个转基因插入(图2b和表7)。
表4.t2植物中的荧光和基因编辑效率
带有荧光的t2植物中的大部分cas9-p2a-gfp蛋白得到了自切割(图7a),表明p2a在拟南芥植物中效果很好,这与在拟南芥原生质体中的结果一致(图1a至1c)。
使用cas9特异性引物对t2植物进行进一步的基因分型,这证实了gfp荧光与含有cas9dna片段之间的完美对应(图2c;图7b)。sanger测序表明,所有四个sgrna基因座均得到了有效编辑(参见下文表5)。
表5.不携带转基因元件的t2代植物中四种sgrna的编辑效率
发明人惊讶地发现,#10品系的8/40(20%)t2植物在所有四个基因座处都具有突变,同时3/40(7.5%)t2植物在所有这四个基因座上都是纯合的(表6)。代表性纯合品系的测序结果显示在图2d中。
表6.同时编辑了四个位点的不携带转基因元件的t2代植物
*带下划线表明全部四个靶位点均为纯合突变
在另一项研究中,发明人使用了图1d中所示的方法共同构建了含12个sgrna表达盒的模块以靶向8个saur基因,并且发现至少有10个sgrna正常发挥作用。
这些结果表明,本发明使用cas9-p2a-gfp的新型crispr系统方便且有效地生成了不携带转基因元件的多重突变体。
以上结果也证明了cas9的高表达对于有效编辑的重要性,特别是在使用多个sgrna进行多重编辑的情况下,因为多个sgrna可能会竞争cas9蛋白。这些结果也证明了本发明使用cas9-p2a-gfp的crispr系统能够让高表达cas9的植物的筛选变得简单易行。之前的方法无论是不对t1代植物进行筛选,还是利用基因型鉴定进行筛选,都没有本发明的方法高效。使用本发明的构建体和方法,通过强荧光这个标志物选出的t1代植株由于具有较高的cas9表达,其中会有更高比例的植株能够获得不携带crispr/cas9转基因元件的t2代植株,进而提高生成理想的、不携带crispr/cas9转基因元件的多重编辑突变体的效率,并大幅减少工作量。
上述实施例证明了本发明的系统在模式植物拟南芥中的效力,同时也说明其也可在其他植物中使用。
表7.实施例中使用的引物
序列表
<110>北京大学
<120>在植物中高效生成不携带转基因元件的突变体的crispr/cas9系统和方法
<130>pq2748pk33cn
<160>41
<170>siposequencelisting1.0
<210>1
<211>54
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>cp编码序列
<400>1
acaaccaccaccaccgcaccctccaccgccttcttctattccttcggaactacc54
<210>2
<211>60
<212>dna
<213>口蹄疫病毒(foot-and-mouthdiseasevirus)
<220>
<223>f2a-1编码序列
<400>2
cagctgttgaattttgaccttcttaaactggcgggagacgtcgagtccaaccctgggccc60
<210>3
<211>69
<212>dna
<213>马甲型鼻炎病毒(equinerhinitisavirus)
<220>
<223>e2a肽编码序列
<400>3
ggaagcggacagtgtactaattatgctctcttgaaattggctggagatgttgagagcaac60
cctggacct69
<210>4
<211>75
<212>dna
<213>口蹄疫病毒(foot-and-mouthdiseasevirus)
<220>
<223>f2a-2编码序列
<400>4
ggaagcggagtgaaacagactttgaattttgaccttctcaagttggcgggagacgtggag60
tccaaccctggacct75
<210>5
<211>66
<212>dna
<213>猪捷申病毒(porcineteschovirus)
<220>
<223>p2a编码序列
<400>5
ggaagcggagctactaacttcagcctgctgaagcaggctggagacgtggaggagaaccct60
ggacct66
<210>6
<211>63
<212>dna
<213>东亚细亚病毒(thoseaasignavirus)
<220>
<223>t2a编码序列
<400>6
ggaagcggagagggcagaggaagtctgctaacatgcggtgacgtcgaggagaatcctgga60
cct63
<210>7
<211>693
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>atu6-26-scaffold
<400>7
ggtaccgagctcggatccactagtaacggccgccagtgtgctggaattgcccttaagctt60
cgttgaacaacggaaactcgacttgccttccgcacaatacatcatttcttcttagctttt120
tttcttcttcttcgttcatacagtttttttttgtttatcagcttacattttcttgaaccg180
tagctttcgttttcttctttttaactttccattcggagtttttgtatcttgtttcatagt240
ttgtcccaggattagaatgattaggcatcgaaccttcaagaatttgattgaataaaacat300
cttcattcttaagatatgaagataatcttcaaaaggcccctgggaatctgaaagaagaga360
agcaggcccatttatatgggaaagaacaatagtatttcttatataggcccatttaagttg420
aaaacaatcttcaaaagtcccacatcgcttagataagaaaacgaagctgagtttatatac480
agctagagtcgaagtagtgattgggtcttcgagaagacctgttttagagctagaaatagc540
aagttaaaataaggctagccgttatcaacttgaaaaagtggcaccgagtcggtgcttttt600
ttgtcccttcgaagggcctttctcagatatccatcacactggcggccgctcgaggctagc660
gatatcgtcgacggtatcgataagcttgatatc693
<210>8
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bri1sgrnaf
<400>8
gattgttgtctggatacataccga24
<210>9
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bri1sgrnar
<400>9
aaactcggtatgtatccagacaac24
<210>10
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bri1-cas9testf
<400>10
agaaagagtgtcatggagctggt23
<210>11
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bri1-cas9testr
<400>11
tcccatcgagcttattgcttg21
<210>12
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bzr1sgrna1f
<400>12
gattgagaaagggagaataatcgg24
<210>13
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bzr1sgrna1r
<400>13
aaacccgattattctccctttctc24
<210>14
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bzr1sgrna2f
<400>14
gattggtgcagaaaccgcatagaa24
<210>15
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bzr1sgrna2r
<400>15
aaacttctatgcggtttctgcacc24
<210>16
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bes1sgrna1f
<400>16
gattgcggcggagaagagctgttg24
<210>17
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bes1sgrna1r
<400>17
aaaccaacagctcttctccgccgc24
<210>18
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bes1sgrna2f
<400>18
gattgaagttggggatgacactgg24
<210>19
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bes1sgrna2r
<400>19
aaacccagtgtcatccccaacttc24
<210>20
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bzr1-cas9testf1
<400>20
cgtgagcactaacttctcactcctc25
<210>21
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bzr1-cas9testr1
<400>21
ccctcaaattctcagattcaaacc24
<210>22
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bzr1-cas9testf2
<400>22
gttgtccagttaccccaccggtc23
<210>23
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bzr1-cas9testr2
<400>23
gaaggcagcagtatttggagacatc25
<210>24
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bes1-cas9testf1
<400>24
gagagttgaaggaagaagatgacg24
<210>25
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bes1-cas9testr1
<400>25
atgcgtttgatactgaccttgcg23
<210>26
<211>26
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bes1-cas9testf2
<400>26
cccataaccaaagtcctctttcttcc26
<210>27
<211>26
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>bes1-cas9testr2
<400>27
ctcttggattgctgctgtgtttggag26
<210>28
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>cas9t-dnatestf
<400>28
gtggaccatatcgtgcctcagag23
<210>29
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>cas9t-dnatestr
<400>29
ctgcacctcggtctttttcacg22
<210>30
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>pcambia1300modifiedf
<400>30
aattccggggtaccgctctagat23
<210>31
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>pcambia1300modifiedr
<400>31
agctatctagagcggtaccccgg23
<210>32
<211>59
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>ubq10nheif
<400>32
ctagctagcgaccgttagaaattgtggttgtcgaggagtcagtaataaacggcgtcaaa59
<210>33
<211>38
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>ubq10xhoir
<400>33
ccgctcgagctgttaatcagaaaaactcagattaatcg38
<210>34
<211>18
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>atu6deletspeif
<400>34
aattcctgcagcccgggt18
<210>35
<211>18
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>atu6deletspeir
<400>35
ctagacccgggctgcagg18
<210>36
<211>18
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>atu6addnheif
<400>36
tcgaggctagcgatatcg18
<210>37
<211>18
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>atu6addnheir
<400>37
tcgacgatatcgctagcc18
<210>38
<211>656
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>ubq10promoter
<400>38
gaccgttagaaattgtggttgtcgaggagtcagtaataaacggcgtcaaagtggttgcag60
ccggcacacacgagtcgtgtttatcaactcaaagcacaaatacttttcctcaacctaaaa120
ataaggcaattagccaaaaacaactttgcgtgtaaacaacgctcaatacacgtgtcattt180
tattattagctattgcttcaccgccttagctttctcgtgacctagtcgtcctcgtctttt240
cttcttcttcttctataaaacaatacccaaagagctcttcttcttcacaattcagatttc300
aatttctcaaaatcttaaaaactttctctcaattctctctaccgtgatcaaggtaaattt360
ctgtgttccttattctctcaaaatcttcgattttgttttcgttcgatcccaatttcgtat420
atgttctttggtttagattctgttaatcttagatcgaagacgattttctgggtttgatcg480
ttagatatcatcttaattctcgattagggtttcatagatatcatccgatttgttcaaata540
atttgagttttgtcgaataattactcttcgatttgtgatttctatctagatctggtgtta600
gtttctagtttgtgcgatcgaatttgtcgattaatctgagtttttctgattaacag656
<210>39
<211>3592
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>patu6-26-m-sgrna构建体
<220>
<222>(707)..(1255)
<223>atu6-sgrna
<400>39
cacctgacgcgccctgtagcggcgcattaagcgcggcgggtgtggtggttacgcgcagcg60
tgaccgctacacttgccagcgccctagcgcccgctcctttcgctttcttcccttcctttc120
tcgccacgttcgccggctttccccgtcaagctctaaatcgggggctccctttagggttcc180
gatttagtgctttacggcacctcgaccccaaaaaacttgattagggtgatggttcacgta240
gtgggccatcgccctgatagacggtttttcgccctttgacgttggagtccacgttcttta300
atagtggactcttgttccaaactggaacaacactcaaccctatctcggtctattcttttg360
atttataagggattttgccgatttcggcctattggttaaaaaatgagctgatttaacaaa420
aatttaacgcgaattttaacaaaatattaacgcttacaatttccattcgccattcaggct480
gcgcaactgttgggaagggcgatcggtgcgggcctcttcgctattacgccagctggcgaa540
agggggatgtgctgcaaggcgattaagttgggtaacgccagggttttcccagtcacgacg600
ttgtaaaacgacggccagtgaattgtaatacgactcactatagggcgaattgggtaccga660
gctcggatccactagtaacggccgccagtgtgctggaattgcccttaagcttcgttgaac720
aacggaaactcgacttgccttccgcacaatacatcatttcttcttagctttttttcttct780
tcttcgttcatacagtttttttttgtttatcagcttacattttcttgaaccgtagctttc840
gttttcttctttttaactttccattcggagtttttgtatcttgtttcatagtttgtccca900
ggattagaatgattaggcatcgaaccttcaagaatttgattgaataaaacatcttcattc960
ttaagatatgaagataatcttcaaaaggcccctgggaatctgaaagaagagaagcaggcc1020
catttatatgggaaagaacaatagtatttcttatataggcccatttaagttgaaaacaat1080
cttcaaaagtcccacatcgcttagataagaaaacgaagctgagtttatatacagctagag1140
tcgaagtagtgattgggtcttcgagaagacctgttttagagctagaaatagcaagttaaa1200
ataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgctttttttgtccc1260
ttcgaagggcctttctcagatatccatcacactggcggccgctcgaggctagcgatatcg1320
tcgacggtatcgataagcttgatatcgaattcctgcagcccgggtctagagcggccgcca1380
ccgcggtggagctccagcttttgttccctttagtgagggttaatttcgagcttggcgtaa1440
tcatggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacata1500
cgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagctaactcacatta1560
attgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgcattaa1620
tgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttcctcg1680
ctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaag1740
gcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatgtgagcaaaa1800
ggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccataggctc1860
cgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgaca1920
ggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccg1980
accctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttct2040
catagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggctgt2100
gtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttgag2160
tccaacccggtaagacacgacttatcgccactggcagcagccactggtaacaggattagc2220
agagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggctac2280
actagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaaga2340
gttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgc2400
aagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacg2460
gggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatgagattatca2520
aaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaagt2580
atatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatctca2640
gcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataactacg2700
atacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgctca2760
ccggctccagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtggt2820
cctgcaactttatccgcctccatccagtctattaattgttgccgggaagctagagtaagt2880
agttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggtgtca2940
cgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaaggcgagttaca3000
tgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcaga3060
agtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctcttact3120
gtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctga3180
gaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgcg3240
ccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaactc3300
tcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaactga3360
tcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaat3420
gccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatactcttccttttt3480
caatattattgaagcatttatcagggttattgtctcatgagcggatacatatttgaatgt3540
atttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgc3592
<210>40
<211>8388
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>pjim19(gent)-m
<220>
<222>(5023)..(5043)
<223>pjim19gentamseqr
<220>
<222>(5132)..(5161)
<223>限制酶识别位点
<220>
<222>(5280)..(5301)
<223>pjim19gentamseqf
<400>40
gcggaaagcagaaagacgacctggtagaaacctgcattcggttaaacaccacgcacgttg60
ccatgcagcgtacgaagaaggccaagaacggccgcctggtgacggtatccgagggtgaag120
ccttgattagccgctacaagatcgtaaagagcgaaaccgggcggccggagtacatcgaga180
tcgagctagctgattggatgtaccgcgagatcacagaaggcaagaacccggacgtgctga240
cggttcaccccgattactttttgatcgatcccggcatcggccgttttctctaccgcctgg300
cacgccgcgccgcaggcaaggcagaagccagatggttgttcaagacgatctacgaacgca360
gtggcagcgccggagagttcaagaagttctgtttcaccgtgcgcaagctgatcgggtcaa420
atgacctgccggagtacgatttgaaggaggaggcggggcaggctggcccgatcctagtca480
tgcgctaccgcaacctgatcgagggcgaagcatccgccggttcctaatgtacggagcaga540
tgctagggcaaattgccctagcaggggaaaaaggtcgaaaaggtctctttcctgtggata600
gcacgtacattgggaacccaaagccgtacattgggaaccggaacccgtacattgggaacc660
caaagccgtacattgggaaccggtcacacatgtaagtgactgatataaaagagaaaaaag720
gcgatttttccgcctaaaactctttaaaacttattaaaactcttaaaacccgcctggcct780
gtgcataactgtctggccagcgcacagccgaagagctgcaaaaagcgcctacccttcggt840
cgctgcgctccctacgccccgccgcttcgcgtcggcctatcgcggccgctggccgctcaa900
aaatggctggcctacggccaggcaatctaccagggcgcggacaagccgcgccgtcgccac960
tcgaccgccggcgcccacatcaaggcaccctgcctcgcgcgtttcggtgatgacggtgaa1020
aacctctgacacatgcagctcccggagacggtcacagcttgtctgtaagcggatgccggg1080
agcagacaagcccgtcagggcgcgtcagcgggtgttggcgggtgtcggggcgcagccatg1140
acccagtcacgtagcgatagcggagtgtatactggcttaactatgcggcatcagagcaga1200
ttgtactgagagtgcaccatatgcggtgtgaaataccgcacagatgcgtaaggagaaaat1260
accgcatcaggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcggc1320
tgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcagggg1380
ataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaagg1440
ccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaatcgac1500
gctcaagtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccccctg1560
gaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtccgcct1620
ttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctcagttcgg1680
tgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccgct1740
gcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccac1800
tggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacagagt1860
tcttgaagtggtggcctaactacggctacactagaaggacagtatttggtatctgcgctc1920
tgctgaagccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaaacca1980
ccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaaggat2040
ctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaactcac2100
gttaagggattttggtcatgcatgatatatctcccaatttgtgtagggcttattatgcac2160
gcttaaaaataataaaagcagacttgacctgatagtttggctgtgagcaattatgtgctt2220
agtgcatctaacgcttgagttaagccgcgccgcgaagcggcgtcggcttgaacgaatttc2280
tagctagacattatttgccgactaccttggtgatctcgcctttcacgtagtggacaaatt2340
cttccaactgatctgcgcgcgaggccaagcgatcttcttcttgtccaagataagcctgtc2400
tagcttcaagtatgacgggctgatactgggccggcaggcgctccattgcccagtcggcag2460
cgacatccttcggcgcgattttgccggttactgcgctgtaccaaatgcgggacaacgtaa2520
gcactacatttcgctcatcgccagcccagtcgggcggcgagttccatagcgttaaggttt2580
catttagcgcctcaaatagatcctgttcaggaaccggatcaaagagttcctccgccgctg2640
gacctaccaaggcaacgctatgttctcttgcttttgtcagcaagatagccagatcaatgt2700
cgatcgtggctggctcgaagatacctgcaagaatgtcattgcgctgccattctccaaatt2760
gcagttcgcgcttagctggataacgccacggaatgatgtcgtcgtgcacaacaatggtga2820
cttctacagcgcggagaatctcgctctctccaggggaagccgaagtttccaaaaggtcgt2880
tgatcaaagctcgccgcgttgtttcatcaagccttacggtcaccgtaaccagcaaatcaa2940
tatcactgtgtggcttcaggccgccatccactgcggagccgtacaaatgtacggccagca3000
acgtcggttcgagatggcgctcgatgacgccaactacctctgatagttgagtcgatactt3060
cggcgatcaccgcttcccccatgatgtttaactttgttttagggcgactgccctgctgcg3120
taacatcgttgctgctccataacatcaaacatcgacccacggcgtaacgcgcttgctgct3180
tggatgcccgaggcatagactgtaccccaaaaaaacagtcataacaagccatgaaaaccg3240
ccactgcgccgttaccaccgctgcgttcggtcaaggttctggaccagttgcgtgagcgca3300
tacgctacttgcattacagcttacgaaccgaacaggcttatgtccactgggttcgtgccc3360
gaattgatcacaggcagcaacgctctgtcatcgttacaatcaacatgctaccctccgcga3420
gatcatccgtgtttcaaacccggcagcttagttgccgttcttccgaatagcatcggtaac3480
atgagcaaagtctgccgccttacaacggctctcccgctgacgccgtcccggactgatggg3540
ctgcctgtatcgagtggtgattttgtgccgagctgccggtcggggagctgttggctggct3600
ggtggcaggatatattgtggtgtaaacaaattgacgcttagacaacttaataacacattg3660
cggacgtttttaatgtactgaattaacgccgaattaattcgggggatctggattttagta3720
ctggattttggttttaggaattagaaattttattgatagaagtattttacaaatacaaat3780
acatactaagggtttcttatatgctcaacacatgagcgaaaccctataggaaccctaatt3840
cccttatctgggaactactcacacattattatggagaaactcgagcttgtcgatcgactc3900
taggccgggaagccgatctcggcttgaacgaattgttaggtggcggtacttgggtcgata3960
tcaaagtgcatcacttcttcccgtatgcccaactttgtatagagagccactgcgggatcg4020
tcaccgtaatctgcttgcacgtagatcacataagcaccaagcgcgttggcctcatgcttg4080
aggagattgatgagcgcggtggcaatgccctgcctccggtgctcgccggagactgcgaga4140
tcatagatatagatctcactacgcggctgctcaaacttgggcagaacgtaagccgcgaga4200
gcgccaacaaccgcttcttggtcgaaggcagcaagcgcgatgaatgtcttactacggagc4260
aagttcccgaggtaatcggagtccggctgatgttgggagtaggtggctacgtctccgaac4320
tcacgaccgaaaagatcaagagcagcccgcatggatttgacttggtcagggccgagccta4380
catgtgcgaatgatgcccatacttgagccacctaactttgttttagggcgactgccctgc4440
tgcgtaacatcgttgctgctgcgtaccatggtcgatcgacagatctgcgaaagctcgaga4500
gagatagatttgtagagagagactggtgatttcagcgtgtcctctccaaatgaaatgaac4560
ttccttatatagaggaagggtcttgcgaaggatagtgggattgtgcgtcatcccttacgt4620
cagtggagatatcacatcaatccacttgctttgaagacgtggttggaacgtcttcttttt4680
ccacgatgctcctcgtgggtgggggtccatctttgggaccactgtcggcagaggcatctt4740
gaacgatagcctttcctttatcgcaatgatggcatttgtaggtgccaccttccttttcta4800
ctgtccttttgatgaagtgacagatagctgggcaatggaatccgaggaggtttcccgata4860
ttaccctttgttgaaaagtctcaatagccctttggtcttctgagactgtatctttgatat4920
tcttggagtagacgagagtgtcgtgctccaccatgtttgcaagctgctctagcattcgcc4980
attcaggctgcgcaactgttgggaagggcgatcggtgcgggcctcttcgctattacgcca5040
gctggcgaaagggggatgtgctgcaaggcgattaagttgggtaacgccagggttttccca5100
gtcacgacgttgtaaaacgacggccagtgccaagcttaggcctactagtggtaccgaatt5160
cgtaatcatgtcatagctgtttcctgtgtgaaattgttatccgctcacaattccacacaa5220
catacgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagctaactcac5280
attaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgca5340
ttaatgaatcggccaacgcgcggggagaggcggtttgcgtattggctagagcgctctaga5400
gcagcttgagcttggatcagattgtcgtttcccgccttcagtttaaactatcagtgtttg5460
acaggatatattggcgggtaaacctaagagaaaagagcgtttattagaataatcggatat5520
ttaaaagggcgtgaaaaggtttatccgttcgtccatttgtatgtgcatgccaaccacagg5580
gttcccctcgggatcaaagtactttgatccaacccctccgctgctatagtgcagtcggct5640
tctgacgttcagtgcagccgtcttctgaaaacgacatgtcgcacaagtcctaagttacgc5700
gacaggctgccgccctgcccttttcctggcgttttcttgtcgcgtgttttagtcgcataa5760
agtagaatacttgcgactagaaccggagacattacgccatgaacaagagcgccgccgctg5820
gcctgctgggctatgcccgcgtcagcaccgacgaccaggacttgaccaaccaacgggccg5880
aactgcacgcggccggctgcaccaagctgttttccgagaagatcaccggcaccaggcgcg5940
accgcccggagctggccaggatgcttgaccacctacgccctggcgacgttgtgacagtga6000
ccaggctagaccgcctggcccgcagcacccgcgacctactggacattgccgagcgcatcc6060
aggaggccggcgcgggcctgcgtagcctggcagagccgtgggccgacaccaccacgccgg6120
ccggccgcatggtgttgaccgtgttcgccggcattgccgagttcgagcgttccctaatca6180
tcgaccgcacccggagcgggcgcgaggccgccaaggcccgaggcgtgaagtttggccccc6240
gccctaccctcaccccggcacagatcgcgcacgcccgcgagctgatcgaccaggaaggcc6300
gcaccgtgaaagaggcggctgcactgcttggcgtgcatcgctcgaccctgtaccgcgcac6360
ttgagcgcagcgaggaagtgacgcccaccgaggccaggcggcgcggtgccttccgtgagg6420
acgcattgaccgaggccgacgccctggcggccgccgagaatgaacgccaagaggaacaag6480
catgaaaccgcaccaggacggccaggacgaaccgtttttcattaccgaagagatcgaggc6540
ggagatgatcgcggccgggtacgtgttcgagccgcccgcgcacgtctcaaccgtgcggct6600
gcatgaaatcctggccggtttgtctgatgccaagctggcggcctggccggccagcttggc6660
cgctgaagaaaccgagcgccgccgtctaaaaaggtgatgtgtatttgagtaaaacagctt6720
gcgtcatgcggtcgctgcgtatatgatgcgatgagtaaataaacaaatacgcaaggggaa6780
cgcatgaaggttatcgctgtacttaaccagaaaggcgggtcaggcaagacgaccatcgca6840
acccatctagcccgcgccctgcaactcgccggggccgatgttctgttagtcgattccgat6900
ccccagggcagtgcccgcgattgggcggccgtgcgggaagatcaaccgctaaccgttgtc6960
ggcatcgaccgcccgacgattgaccgcgacgtgaaggccatcggccggcgcgacttcgta7020
gtgatcgacggagcgccccaggcggcggacttggctgtgtccgcgatcaaggcagccgac7080
ttcgtgctgattccggtgcagccaagcccttacgacatatgggccaccgccgacctggtg7140
gagctggttaagcagcgcattgaggtcacggatggaaggctacaagcggcctttgtcgtg7200
tcgcgggcgatcaaaggcacgcgcatcggcggtgaggttgccgaggcgctggccgggtac7260
gagctgcccattcttgagtcccgtatcacgcagcgcgtgagctacccaggcactgccgcc7320
gccggcacaaccgttcttgaatcagaacccgagggcgacgctgcccgcgaggtccaggcg7380
ctggccgctgaaattaaatcaaaactcatttgagttaatgaggtaaagagaaaatgagca7440
aaagcacaaacacgctaagtgccggccgtccgagcgcacgcagcagcaaggctgcaacgt7500
tggccagcctggcagacacgccagccatgaagcgggtcaactttcagttgccggcggagg7560
atcacaccaagctgaagatgtacgcggtacgccaaggcaagaccattaccgagctgctat7620
ctgaatacatcgcgcagctaccagagtaaatgagcaaatgaataaatgagtagatgaatt7680
ttagcggctaaaggaggcggcatggaaaatcaagaacaaccaggcaccgacgccgtggaa7740
tgccccatgtgtggaggaacgggcggttggccaggcgtaagcggctgggttgtctgccgg7800
ccctgcaatggcactggaacccccaagcccgaggaatcggcgtgagcggtcgcaaaccat7860
ccggcccggtacaaatcggcgcggcgctgggtgatgacctggtggagaagttgaaggccg7920
cgcaggccgcccagcggcaacgcatcgaggcagaagcacgccccggtgaatcgtggcaag7980
cggccgctgatcgaatccgcaaagaatcccggcaaccgccggcagccggtgcgccgtcga8040
ttaggaagccgcccaagggcgacgagcaaccagattttttcgttccgatgctctatgacg8100
tgggcacccgcgatagtcgcagcatcatggacgtggccgttttccgtctgtcgaagcgtg8160
accgacgagctggcgaggtgatccgctacgagcttccagacgggcacgtagaggtttccg8220
cagggccggccggcatggccagtgtgtgggattacgacctggtactgatggcggtttccc8280
atctaaccgaatccatgaaccgataccgggaagggaagggagacaagcccggccgcgtgt8340
tccgtccacacgttgcggacgtactcaagttctgccggcgagccgatg8388
<210>41
<211>8930
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>pcambia1300-m
<400>41
gttggcaagctgctctagccaatacgcaaaccgcctctccccgcgcgttggccgattcat60
taatgcagctggcacgacaggtttcccgactggaaagcgggcagtgagcgcaacgcaatt120
aatgtgagttagctcactcattaggcaccccaggctttacactttatgcttccggctcgt180
atgttgtgtggaattgtgagcggataacaatttcacacaggaaacagctatgaccatgat240
tacgaattccggggtaccgctctagatagcttggcactggccgtcgttttacaacgtcgt300
gactgggaaaaccctggcgttacccaacttaatcgccttgcagcacatccccctttcgcc360
agctggcgtaatagcgaagaggcccgcaccgatcgcccttcccaacagttgcgcagcctg420
aatggcgaatgctagagcagcttgagcttggatcagattgtcgtttcccgccttcagttt480
aaactatcagtgtttgacaggatatattggcgggtaaacctaagagaaaagagcgtttat540
tagaataacggatatttaaaagggcgtgaaaaggtttatccgttcgtccatttgtatgtg600
catgccaaccacagggttcccctcgggatcaaagtactttgatccaacccctccgctgct660
atagtgcagtcggcttctgacgttcagtgcagccgtcttctgaaaacgacatgtcgcaca720
agtcctaagttacgcgacaggctgccgccctgcccttttcctggcgttttcttgtcgcgt780
gttttagtcgcataaagtagaatacttgcgactagaaccggagacattacgccatgaaca840
agagcgccgccgctggcctgctgggctatgcccgcgtcagcaccgacgaccaggacttga900
ccaaccaacgggccgaactgcacgcggccggctgcaccaagctgttttccgagaagatca960
ccggcaccaggcgcgaccgcccggagctggccaggatgcttgaccacctacgccctggcg1020
acgttgtgacagtgaccaggctagaccgcctggcccgcagcacccgcgacctactggaca1080
ttgccgagcgcatccaggaggccggcgcgggcctgcgtagcctggcagagccgtgggccg1140
acaccaccacgccggccggccgcatggtgttgaccgtgttcgccggcattgccgagttcg1200
agcgttccctaatcatcgaccgcacccggagcgggcgcgaggccgccaaggcccgaggcg1260
tgaagtttggcccccgccctaccctcaccccggcacagatcgcgcacgcccgcgagctga1320
tcgaccaggaaggccgcaccgtgaaagaggcggctgcactgcttggcgtgcatcgctcga1380
ccctgtaccgcgcacttgagcgcagcgaggaagtgacgcccaccgaggccaggcggcgcg1440
gtgccttccgtgaggacgcattgaccgaggccgacgccctggcggccgccgagaatgaac1500
gccaagaggaacaagcatgaaaccgcaccaggacggccaggacgaaccgtttttcattac1560
cgaagagatcgaggcggagatgatcgcggccgggtacgtgttcgagccgcccgcgcacgt1620
ctcaaccgtgcggctgcatgaaatcctggccggtttgtctgatgccaagctggcggcctg1680
gccggccagcttggccgctgaagaaaccgagcgccgccgtctaaaaaggtgatgtgtatt1740
tgagtaaaacagcttgcgtcatgcggtcgctgcgtatatgatgcgatgagtaaataaaca1800
aatacgcaaggggaacgcatgaaggttatcgctgtacttaaccagaaaggcgggtcaggc1860
aagacgaccatcgcaacccatctagcccgcgccctgcaactcgccggggccgatgttctg1920
ttagtcgattccgatccccagggcagtgcccgcgattgggcggccgtgcgggaagatcaa1980
ccgctaaccgttgtcggcatcgaccgcccgacgattgaccgcgacgtgaaggccatcggc2040
cggcgcgacttcgtagtgatcgacggagcgccccaggcggcggacttggctgtgtccgcg2100
atcaaggcagccgacttcgtgctgattccggtgcagccaagcccttacgacatatgggcc2160
accgccgacctggtggagctggttaagcagcgcattgaggtcacggatggaaggctacaa2220
gcggcctttgtcgtgtcgcgggcgatcaaaggcacgcgcatcggcggtgaggttgccgag2280
gcgctggccgggtacgagctgcccattcttgagtcccgtatcacgcagcgcgtgagctac2340
ccaggcactgccgccgccggcacaaccgttcttgaatcagaacccgagggcgacgctgcc2400
cgcgaggtccaggcgctggccgctgaaattaaatcaaaactcatttgagttaatgaggta2460
aagagaaaatgagcaaaagcacaaacacgctaagtgccggccgtccgagcgcacgcagca2520
gcaaggctgcaacgttggccagcctggcagacacgccagccatgaagcgggtcaactttc2580
agttgccggcggaggatcacaccaagctgaagatgtacgcggtacgccaaggcaagacca2640
ttaccgagctgctatctgaatacatcgcgcagctaccagagtaaatgagcaaatgaataa2700
atgagtagatgaattttagcggctaaaggaggcggcatggaaaatcaagaacaaccaggc2760
accgacgccgtggaatgccccatgtgtggaggaacgggcggttggccaggcgtaagcggc2820
tgggttgtctgccggccctgcaatggcactggaacccccaagcccgaggaatcggcgtga2880
cggtcgcaaaccatccggcccggtacaaatcggcgcggcgctgggtgatgacctggtgga2940
gaagttgaaggccgcgcaggccgcccagcggcaacgcatcgaggcagaagcacgccccgg3000
tgaatcgtggcaagcggccgctgatcgaatccgcaaagaatcccggcaaccgccggcagc3060
cggtgcgccgtcgattaggaagccgcccaagggcgacgagcaaccagattttttcgttcc3120
gatgctctatgacgtgggcacccgcgatagtcgcagcatcatggacgtggccgttttccg3180
tctgtcgaagcgtgaccgacgagctggcgaggtgatccgctacgagcttccagacgggca3240
cgtagaggtttccgcagggccggccggcatggccagtgtgtgggattacgacctggtact3300
gatggcggtttcccatctaaccgaatccatgaaccgataccgggaagggaagggagacaa3360
gcccggccgcgtgttccgtccacacgttgcggacgtactcaagttctgccggcgagccga3420
tggcggaaagcagaaagacgacctggtagaaacctgcattcggttaaacaccacgcacgt3480
tgccatgcagcgtacgaagaaggccaagaacggccgcctggtgacggtatccgagggtga3540
agccttgattagccgctacaagatcgtaaagagcgaaaccgggcggccggagtacatcga3600
gatcgagctagctgattggatgtaccgcgagatcacagaaggcaagaacccggacgtgct3660
gacggttcaccccgattactttttgatcgatcccggcatcggccgttttctctaccgcct3720
ggcacgccgcgccgcaggcaaggcagaagccagatggttgttcaagacgatctacgaacg3780
cagtggcagcgccggagagttcaagaagttctgtttcaccgtgcgcaagctgatcgggtc3840
aaatgacctgccggagtacgatttgaaggaggaggcggggcaggctggcccgatcctagt3900
catgcgctaccgcaacctgatcgagggcgaagcatccgccggttcctaatgtacggagca3960
gatgctagggcaaattgccctagcaggggaaaaaggtcgaaaaggtctctttcctgtgga4020
tagcacgtacattgggaacccaaagccgtacattgggaaccggaacccgtacattgggaa4080
cccaaagccgtacattgggaaccggtcacacatgtaagtgactgatataaaagagaaaaa4140
aggcgatttttccgcctaaaactctttaaaacttattaaaactcttaaaacccgcctggc4200
ctgtgcataactgtctggccagcgcacagccgaagagctgcaaaaagcgcctacccttcg4260
gtcgctgcgctccctacgccccgccgcttcgcgtcggcctatcgcggccgctggccgctc4320
aaaaatggctggcctacggccaggcaatctaccagggcgcggacaagccgcgccgtcgcc4380
actcgaccgccggcgcccacatcaaggcaccctgcctcgcgcgtttcggtgatgacggtg4440
aaaacctctgacacatgcagctcccggagacggtcacagcttgtctgtaagcggatgccg4500
ggagcagacaagcccgtcagggcgcgtcagcgggtgttggcgggtgtcggggcgcagcca4560
tgacccagtcacgtagcgatagcggagtgtatactggcttaactatgcggcatcagagca4620
gattgtactgagagtgcaccatatgcggtgtgaaataccgcacagatgcgtaaggagaaa4680
ataccgcatcaggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcg4740
gctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcagg4800
ggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaa4860
ggccgcgttgctggcgtttttccataggctccgcccccctgacgagcatcacaaaaatcg4920
acgctcaagtcagaggtggcgaaacccgacaggactataaagataccaggcgtttccccc4980
tggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtccgc5040
ctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctcagttc5100
ggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccg5160
ctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgcc5220
actggcagcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacaga5280
gttcttgaagtggtggcctaactacggctacactagaaggacagtatttggtatctgcgc5340
tctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccggcaaacaaac5400
caccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaagg5460
atctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaactc5520
acgttaagggattttggtcatgcattctaggtactaaaacaattcatccagtaaaatata5580
atattttattttctcccaatcaggcttgatccccagtaagtcaaaaaatagctcgacata5640
ctgttcttccccgatatcctccctgatcgaccggacgcagaaggcaatgtcataccactt5700
gtccgccctgccgcttctcccaagatcaataaagccacttactttgccatctttcacaaa5760
gatgttgctgtctcccaggtcgccgtgggaaaagacaagttcctcttcgggcttttccgt5820
ctttaaaaaatcatacagctcgcgcggatctttaaatggagtgtcttcttcccagttttc5880
gcaatccacatcggccagatcgttattcagtaagtaatccaattcggctaagcggctgtc5940
taagctattcgtatagggacaatccgatatgtcgatggagtgaaagagcctgatgcactc6000
cgcatacagctcgataatcttttcagggctttgttcatcttcatactcttccgagcaaag6060
gacgccatcggcctcactcatgagcagattgctccagccatcatgccgttcaaagtgcag6120
gacctttggaacaggcagctttccttccagccatagcatcatgtccttttcccgttccac6180
atcataggtggtccctttataccggctgtccgtcatttttaaatataggttttcattttc6240
tcccaccagcttatataccttagcaggagacattccttccgtatcttttacgcagcggta6300
tttttcgatcagttttttcaattccggtgatattctcattttagccatttattatttcct6360
tcctcttttctacagtatttaaagataccccaagaagctaattataacaagacgaactcc6420
aattcactgttccttgcattctaaaaccttaaataccagaaaacagctttttcaaagttg6480
ttttcaaagttggcgtataacatagtatcgacggagccgattttgaaaccgcggtgatca6540
caggcagcaacgctctgtcatcgttacaatcaacatgctaccctccgcgagatcatccgt6600
gtttcaaacccggcagcttagttgccgttcttccgaatagcatcggtaacatgagcaaag6660
tctgccgccttacaacggctctcccgctgacgccgtcccggactgatgggctgcctgtat6720
cgagtggtgattttgtgccgagctgccggtcggggagctgttggctggctggtggcagga6780
tatattgtggtgtaaacaaattgacgcttagacaacttaataacacattgcggacgtttt6840
taatgtactgaattaacgccgaattaattcgggggatctggattttagtactggattttg6900
gttttaggaattagaaattttattgatagaagtattttacaaatacaaatacatactaag6960
ggtttcttatatgctcaacacatgagcgaaaccctataggaaccctaattcccttatctg7020
ggaactactcacacattattatggagaaactcgagcttgtcgatcgacagatccggtcgg7080
catctactctatttctttgccctcggacgagtgctggggcgtcggtttccactatcggcg7140
agtacttctacacagccatcggtccagacggccgcgcttctgcgggcgatttgtgtacgc7200
ccgacagtcccggctccggatcggacgattgcgtcgcatcgaccctgcgcccaagctgca7260
tcatcgaaattgccgtcaaccaagctctgatagagttggtcaagaccaatgcggagcata7320
tacgcccggagtcgtggcgatcctgcaagctccggatgcctccgctcgaagtagcgcgtc7380
tgctgctccatacaagccaaccacggcctccagaagaagatgttggcgacctcgtattgg7440
gaatccccgaacatcgcctcgctccagtcaatgaccgctgttatgcggccattgtccgtc7500
aggacattgttggagccgaaatccgcgtgcacgaggtgccggacttcggggcagtcctcg7560
gcccaaagcatcagctcatcgagagcctgcgcgacggacgcactgacggtgtcgtccatc7620
acagtttgccagtgatacacatggggatcagcaatcgcgcatatgaaatcacgccatgta7680
gtgtattgaccgattccttgcggtccgaatgggccgaacccgctcgtctggctaagatcg7740
gccgcagcgatcgcatccatagcctccgcgaccggttgtagaacagcgggcagttcggtt7800
tcaggcaggtcttgcaacgtgacaccctgtgcacggcgggagatgcaataggtcaggctc7860
tcgctaaactccccaatgtcaagcacttccggaatcgggagcgcggccgatgcaaagtgc7920
cgataaacataacgatctttgtagaaaccatcggcgcagctatttacccgcaggacatat7980
ccacgccctcctacatcgaagctgaaagcacgagattcttcgccctccgagagctgcatc8040
aggtcggagacgctgtcgaacttttcgatcagaaacttctcgacagacgtcgcggtgagt8100
tcaggctttttcatatctcattgccccccgggatctgcgaaagctcgagagagatagatt8160
tgtagagagagactggtgatttcagcgtgtcctctccaaatgaaatgaacttccttatat8220
agaggaaggtcttgcgaaggatagtgggattgtgcgtcatcccttacgtcagtggagata8280
tcacatcaatccacttgctttgaagacgtggttggaacgtcttctttttccacgatgctc8340
ctcgtgggtgggggtccatctttgggaccactgtcggcagaggcatcttgaacgatagcc8400
tttcctttatcgcaatgatggcatttgtaggtgccaccttccttttctactgtccttttg8460
atgaagtgacagatagctgggcaatggaatccgaggaggtttcccgatattaccctttgt8520
tgaaaagtctcaatagccctttggtcttctgagactgtatctttgatattcttggagtag8580
acgagagtgtcgtgctccaccatgttatcacatcaatccacttgctttgaagacgtggtt8640
ggaacgtcttctttttccacgatgctcctcgtgggtgggggtccatctttgggaccactg8700
tcggcagaggcatcttgaacgatagcctttcctttatcgcaatgatggcatttgtaggtg8760
ccaccttccttttctactgtccttttgatgaagtgacagatagctgggcaatggaatccg8820
aggaggtttcccgatattaccctttgttgaaaagtctcaatagccctttggtcttctgag8880
actgtatctttgatattcttggagtagacgagagtgtcgtgctccaccat8930
- 还没有人留言评论。精彩留言会获得点赞!