一种人DIP-InDel基因座荧光标记试剂盒及检测方法与流程
本发明属于生物检测技术领域,涉及一种不平衡混合样本的鉴定,特别是涉及一种用于双人不平衡混合斑次要成分确定和胎儿亲子鉴定的dip-indel基因座试剂盒及其检测方法。
背景技术:
混合斑是最常见的法医检材之一,可由不同个体的同种或不同种体液、分泌液混合而成。随着dna提取技术和检测技术的不断提高,混合dna样本的检出率大大提高,且根据案件不同,近年来对含量极低dna、降解dna等复杂情况的分析要求逐渐提升。
插入缺失多态性(deletion/insertionpolymorphisms,dip)多指人类基因组中长度小于300bp的2个插入缺失标记(insertion-deletion,indel)的组合。因此dip-indel是指在一个插入缺失多态性位点后连锁另一个插入缺失多态性位点。目前已有利用常染色体上的dip-str,dip-snp等多种dip-连锁下游多态性对混合斑进行研究,但dip-indel相关研究很少。
对于可明确为双人混合斑的案件,已知主要组分分型(一般为受害人)的情况下,对其中微量组分(一般为加害人)进行分析时,常因其含量极低引起检测结果波动。在亲子鉴定中,常用的str基因座具有较高突变率,鉴定时会出现被检亲代与被检子代之间出现不符合孟德尔遗传规律的现象。尤其是针对胎儿的亲子鉴定,因鉴定大多采用胎儿羊水为样本,传统的羊膜腔穿刺术和绒毛吸取术操作有创伤性,严重可导致宫内感染、流产、胎儿死亡等不良并发症,因此开发一种针对双人不平衡混合斑的快速鉴定试剂盒对提高目前亲子鉴定的安全性和准确锁定嫌疑人方面都有帮助。
技术实现要素:
本发明的目的在于提供一种人dip-indel基因座荧光标记试剂盒及检测方法,提高分析双人不平衡dna混合样本的准确度和降低胎儿亲子鉴定的难度。
为实现上述目的,本发明提供了一种人dip-indel基因座荧光标记试剂盒,所述试剂盒包含了45条特异性多重pcr引物、pcrmastermix、4对带荧光标记的通用引物、ddh2o、用于检测扩增产物的去离子甲酰胺和size-500plus内标。
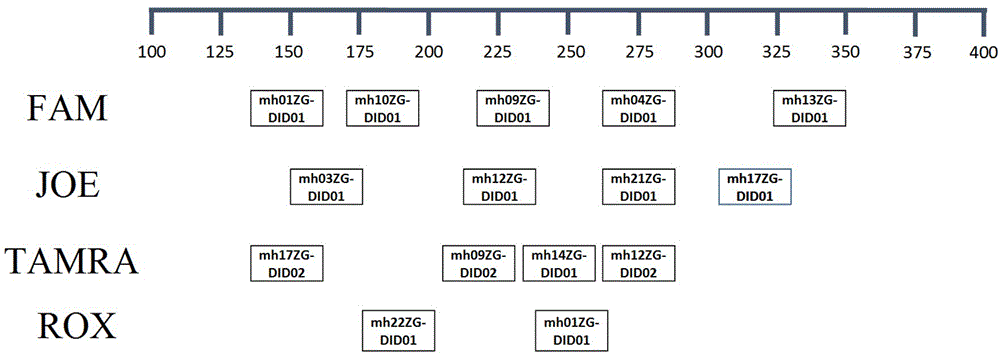
本发明所述人dip-indel基因座荧光标记试剂盒中包括的45条特异性多重pcr引物的核苷酸序列如seqno.1~45所示,用于检测本发明筛选出的以下15个人dip-indel基因座:mh01zg-did01、mh10zg-did01、mh09zg-did01、mh04zg-did01、mh13zg-did01、mh03zg-did01、mh12zg-did01、mh21zg-did01、mh17zg-did01、mh17zg-did02、mh09zg-did02、mh14zg-did01、mh12zg-did02、mh22zg-did01、mh01zg-did01。
上述15个人dip-indel基因座是基于以下特点筛选得到的。
1、所有indel的杂合度het>0.2。
2、一个dip-indel标记中所选indel之间的物理距离小于200bp。
3、每个indel中插入或缺失的长度为3~15bp。
4、扩增片段的长度小于300bp。
5、每个dip-indel位点均位于非编码区且与基因表达无关。
进一步地,所述15个人dip-indel基因座的相关信息见表1所示。
其中,具体地,所述15个人dip-indel基因座与45条特异性多重pcr引物按照如下关系相对应。
mh01zg-did01的pcr引物为seqidno.1的fl上游引物及seqidno.2的fs上游引物和seqidno.3的下游引物。
mh10zg-did01的pcr引物为seqidno.4的fl上游引物及seqidno.5的fs上游引物和seqidno.6的下游引物。
mh09zg-did01的pcr引物为seqidno.7的fl上游引物及seqidno.8的fs上游引物和seqidno.9的下游引物。
mh04zg-did01的pcr引物为seqidno.10的fl上游引物及seqidno.11的fs上游引物和seqidno.12的下游引物。
mh13zg-did01的pcr引物为seqidno.13的fl上游引物及seqidno.14的fs上游引物和seqidno.15的下游引物。
mh03zg-did01的pcr引物为seqidno.16的fl上游引物及seqidno.17的fs上游引物和seqidno.18的下游引物。
mh12zg-did01的pcr引物为seqidno.19的fl上游引物及seqidno.20的fs上游引物和seqidno.21的下游引物。
mh21zg-did01的pcr引物为seqidno.22的fl上游引物及seqidno.23的fs上游引物和seqidno.24的下游引物。
mh17zg-did01的pcr引物为seqidno.25的fl上游引物及seqidno.26的fs上游引物和seqidno.27的下游引物。
mh17zg-did02的pcr引物为seqidno.28的fl上游引物及seqidno.29的fs上游引物和seqidno.30的下游引物。
mh09zg-did02的pcr引物为seqidno.31的fl上游引物及seqidno.32的fs上游引物和seqidno.33的下游引物。
mh14zg-did01的pcr引物为seqidno.34的fl上游引物及seqidno.35的fs上游引物和seqidno.36的下游引物。
mh12zg-did02的pcr引物为seqidno.37的fl上游引物及seqidno.38的fs上游引物和seqidno.39的下游引物。
mh22zg-did01的pcr引物为seqidno.40的fl上游引物及seqidno.41的fs上游引物和seqidno.42的下游引物。
mh01zg-did01的pcr引物为seqidno.43的fl上游引物及seqidno.44的fs上游引物和seqidno.45的下游引物。
本发明试剂盒中的4对带荧光标记的通用引物的核苷酸序列如seqno.46~53所示。
本发明中使用的荧光标记染料分别为:蓝色荧光标记染料羧基荧光素(fam)、绿色荧光标记染料2,7-二甲基-4,5-二氯-6-羧基荧光素(joe)、黄色荧光标记染料四甲基-6羧基罗丹明(trama)、红色荧光标记染料罗丹明(rox)。内标选用strtyper-21g的橙色内标size-500plus,但本发明不限于此,只要由橙色标记,长度范围在75~500之间的内标均可使用。
进一步地,根据对应通用引物标记荧光染料的不同,15个人dip-indel基因座被分为4组,其中第1组基因座的通用引物为seqidno.46所示的上游引物和seqidno.47所示的下游引物,上游引物5’端带有fam荧光标记;第2组基因座的通用引物为seqidno.48所示的上游引物和seqidno.49所示的下游引物,上游引物5’端带有joe荧光标记;第3组基因座的通用引物为seqidno.50所示的上游引物和seqidno.51所示的下游引物,上游引物5’端带有tamra荧光标记;第4组基因座的通用引物为seqidno.52所示的上游引物和seqidno.53所示的下游引物,上游引物5’端带有rox荧光标记。
本发明所述人dip-indel基因座的分组以及上下游引物浓度具体见表2。
本发明还提供了一种使用所述人dip-indel基因座荧光标记试剂盒检测双人混合样本中微量成分的方法,包括以下步骤。
1)、提取待测样本dna为模板,利用所述45条人dip-indel基因座特异性引物和4对通用引物构建多重pcr扩增体系,进行多重pcr扩增得到扩增产物。
2)、利用试剂盒中的去离子甲酰胺和size-500plus内标处理扩增产物,热变性后使用基因分析仪(3130geneticanalyzer)和分析软件(genemapperidv3.2)检测并对结果进行分析。
与现有技术相比,本发明具有以下有益效果。
本发明选取的15个人dip-indel基因座突变率低、多态性良好、且无stutter峰影响分型图谱。
在明确为双人混合斑的案件中,可检测出含量极低的次要成分,在混合dna已降解的情况下也可直接锁定嫌疑人。
对胎儿的亲子鉴定中,因微量胎儿dna会过胎盘进入孕妇血液,只需对孕妇外周血中次要dna成分进行分析,可以在仅提取孕妇血液的情况下完成亲子鉴定,保护了胎儿和孕妇安全。
本发明的人dip-indel基因座荧光标记试剂盒可以满足针对两样本的不平衡混合dna中的次要成分的检测,完成法医鉴定和医学检测多方面的需要。与现有的不平衡混合斑试剂盒、亲子鉴定方法结合使用,可以进一步提高鉴定结论的准确性。
附图说明
图1是基因座排布示意图。
图2是人群频率调查中随机个体的分型结果图。
图3是不同dna含量时的灵敏度分型结果,(a)dna含量1ng,(b)dna含量2ng,(c)dna含量4ng,(d)dna含量6ng。
图4是使用strtyper-21gstrkit试剂盒和本发明所述试剂盒对不同单位降解检材的分型结果,(a)strtyper-21gstrkit检测0.003单位检材,(b)strtyper-21gstrkit检测0.006单位检材,(c)本发明所述试剂盒检测0.003单位检材,(d)本发明所述试剂盒检测0.006单位检材。
图5是模拟混合斑中主、次要成分及两者不同比例混合的分型结果,(a)主要成分,(b)次要成分,(c)两者比例为10:1、(d)两者比例为20:1、(e)两者比例为100:1。
图6是针对mh04zg-did01基因座的模拟混合斑主、次要成分及两者不同比例混合的分型结果。其中,(a)主要成分,(b)次要成分,(c)两者比例为10:1、(d)两者比例为50:1、(e)两者比例为100:1、(f)两者比例为1000:1。
具体实施方式
下述实施例仅为本发明的优选技术方案,并不用于对本发明进行任何限制。对于本领域技术人员而言,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
实施例1:人群频率调查。
1、收集个体检材。
本实验中共采集200份无关个体样本均属于山西地区汉族人群。基因组dna的提取参照《ga/t383-2014法庭科学dna实验室检验规范》进行。
2、扩增及检测。
以提取的dna为模板,进行pcr扩增。pcr反应体系为(10μl):5μl2×pcrmastermix,根据表2计算加入不同体积的引物,10ng模板dna,ddh2o补至10μl。
扩增程序为:95℃10min;95℃30s,56℃30s,72℃30s,5个循环;94℃30s,52℃30s,68℃30s,25个循环;68℃30min。
检测扩增产物:扩增产物1.5μl、去离子甲酰胺10μl、size-5000plus0.2μl。变性方法:95℃处理5min后,-20℃处理3min。
使用3130geneticanalyzer(appliedbiosystem,usa)和genemapperidv3.2(appliedbiosystems,usa)软件进行分析。图2为随机无关个体分型结果。经过计算得到各人群在每个dip-indel位点的等位基因频率,结果见表3。
实施例2:种属特异性研究。
1、收集动物检材。
本实验中动物和细菌样本dna分别来自于牛、鱼、猪、兔、鼠、羊和链球菌。使用凯杰(dneasyblood&tissuekit)试剂盒进行基因组dna的提取。
2、扩增及检测。
按实施例1中的方法进行pcr扩增、检测和结果分析,结果证实本试剂盒具有种属特异性。
实施例3:灵敏度检测。
1、制备dna样本。
利用凯杰(dneasyblood&tissuekit)试剂盒提取dna。使用含不同dna模板量(6ng,4ng,2ng和1ng)的单源dna确定本体系的灵敏度。
2、扩增及检测。
按实施例1中的方法进行pcr扩增、检测和结果分析。图3证实本试剂盒灵敏度良好,最低可检测含量低至2ng的dna。
实施例4:已降解检材的检测。
1、制备降解检材。
使用dnasei(无rnase)(dneasyblood&tissuekit,transgen)制备人工降解检材。用0.003和0.006单位的dnasei(无rnase)处理50μl新鲜血液,以获得人工降解dna。
2、扩增及检测。
按实施例1中的方法利用本发明所述试剂盒和strtyper-21gstr试剂盒(healthgenetechnologies)分别对降解检材进行pcr扩增、检测和结果分析。
图4表明,本试剂盒针对降解样本的分型效果优于strtyper-21gstrkit试剂盒效果。
实施例5:模拟混合斑检材的检测。
1、制备模拟混合斑。
采用两人dna进行不同比例的混合,混合比例分别为:1:10;1:20;1:100;1:1000。
2、扩增及检测。
按实施例1中的方法进行扩增、检测和结果分析。图5是模拟混合斑中主要、次要成分及两者不同比例混合样品的分型结果,证明本试剂盒针对一方含量低至1%~2%的混合斑检测效果良好。针对单一基因座mh04zg-did01,可检测到低至0.1%的混合斑,检测结果见图6。
序列表
<110>山西医科大学
<120>一种人dip-indel基因座荧光标记试剂盒及检测方法
<160>53
<170>siposequencelisting1.0
<210>1
<211>25
<212>dna
<213>人工序列()
<400>1
aactgctctccttgattctcacttc25
<210>2
<211>24
<212>dna
<213>人工序列()
<400>2
aactgctctccttgattcttctca24
<210>3
<211>24
<212>dna
<213>人工序列()
<400>3
aagaatctcagcagaggtgtgaac24
<210>4
<211>23
<212>dna
<213>人工序列()
<400>4
ccatccatttctaaccccaacaa23
<210>5
<211>23
<212>dna
<213>人工序列()
<400>5
ccatccatttctaaccccaaagg23
<210>6
<211>24
<212>dna
<213>人工序列()
<400>6
cctgattcactaagatttggctcc24
<210>7
<211>26
<212>dna
<213>人工序列()
<400>7
cccattttctgtatcctcttgttcat26
<210>8
<211>26
<212>dna
<213>人工序列()
<400>8
cccattttctgtatcctcatttgttg26
<210>9
<211>25
<212>dna
<213>人工序列()
<400>9
gatgcttttccttctattcccttct25
<210>10
<211>23
<212>dna
<213>人工序列()
<400>10
aagcaagactgtggagggcctaa23
<210>11
<211>23
<212>dna
<213>人工序列()
<400>11
aagcaagactgtggactcatctg23
<210>12
<211>23
<212>dna
<213>人工序列()
<400>12
tgagccttgtattcacagaccca23
<210>13
<211>21
<212>dna
<213>人工序列()
<400>13
taagataagcatcaggggtaa21
<210>14
<211>21
<212>dna
<213>人工序列()
<400>14
taagataagcatcaggggata21
<210>15
<211>23
<212>dna
<213>人工序列一种()
<400>15
agttccttaacagggtttcaagc23
<210>16
<211>23
<212>dna
<213>人工序列()
<400>16
ccagactcttttaaggttcacca23
<210>17
<211>23
<212>dna
<213>人工序列()
<400>17
ccagactcttttaagggcttttc23
<210>18
<211>23
<212>dna
<213>人工序列()
<400>18
ccaggactatgaataccatggat23
<210>19
<211>23
<212>dna
<213>人工序列()
<400>19
gtttggaaacatagtacaagtat23
<210>20
<211>23
<212>dna
<213>人工序列()
<400>20
gtttggaaacatagtacaaggaa23
<210>21
<211>24
<212>dna
<213>人工序列()
<400>21
atatgtaatgctatggcactgctc24
<210>22
<211>23
<212>dna
<213>人工序列()
<400>22
acaaactggcaccttcactccct23
<210>23
<211>23
<212>dna
<213>人工序列()
<400>23
tcaaactggcaccctcactcttg23
<210>24
<211>24
<212>dna
<213>人工序列()
<400>24
acactgtgagaattcagagcaagg24
<210>25
<211>24
<212>dna
<213>人工序列()
<400>25
gggcaagttcaacttgttcaactt24
<210>26
<211>24
<212>dna
<213>人工序列()
<400>26
gggcaagttcaacttaaccctcaa24
<210>27
<211>21
<212>dna
<213>人工序列()
<400>27
agcagaaccttcccagtagcc21
<210>28
<211>25
<212>dna
<213>人工序列()
<400>28
gagcacatgtgtatgtatgactgtg25
<210>29
<211>25
<212>dna
<213>人工序列()
<400>29
gagcacatgtgtatgtatgtgacta25
<210>30
<211>23
<212>dna
<213>人工序列()
<400>30
gttcttctcctgaccctgcatga23
<210>31
<211>23
<212>dna
<213>人工序列()
<400>31
tagaggcctatggactgtggact23
<210>32
<211>23
<212>dna
<213>人工序列()
<400>32
tggaggcctatggactgttttgg23
<210>33
<211>23
<212>dna
<213>人工序列()
<400>33
agcctccttagcatttggttggt23
<210>34
<211>23
<212>dna
<213>人工序列()
<400>34
aaggttctaagggaatagcaggg23
<210>35
<211>23
<212>dna
<213>人工序列()
<400>35
aaggttctaagggaatagctgcc23
<210>36
<211>24
<212>dna
<213>人工序列()
<400>36
gcagttccatagaggtgatactca24
<210>37
<211>23
<212>dna
<213>人工序列()
<400>37
ctccctcacataccaagtgcaga23
<210>38
<211>23
<212>dna
<213>人工序列()
<400>38
ctccctcacataccaagtgctac23
<210>39
<211>23
<212>dna
<213>人工序列()
<400>39
gcatcactagcaccctagtccaa23
<210>40
<211>24
<212>dna
<213>人工序列()
<400>40
atgtagagggcaaagtgcaaagtg24
<210>41
<211>23
<212>dna
<213>人工序列()
<400>41
atgtagagggcaaagtgcaccca23
<210>42
<211>22
<212>dna
<213>人工序列()
<400>42
tctgccttctctgaattgctgg22
<210>43
<211>26
<212>dna
<213>人工序列()
<400>43
gcgcttccactctgccctcctaacag26
<210>44
<211>26
<212>dna
<213>人工序列()
<400>44
gcgcttccaactctgaaatatttttc26
<210>45
<211>21
<212>dna
<213>人工序列()
<400>45
acaaccacctcactgctttcc21
<210>46
<211>24
<212>dna
<213>人工序列()
<400>46
tgtcattgtcggcgcaactatcgg24
<210>47
<211>24
<212>dna
<213>人工序列()
<400>47
tgagctacagcaccagatccagca24
<210>48
<211>24
<212>dna
<213>人工序列()
<400>48
ccgttgtactttgtttcgcgcttg24
<210>49
<211>24
<212>dna
<213>人工序列()
<400>49
cctgacctgttggagtttgcttcc24
<210>50
<211>24
<212>dna
<213>人工序列()
<400>50
gcccttttgtctttggcgctggta24
<210>51
<211>24
<212>dna
<213>人工序列()
<400>51
tctattctcactccgctgaaactg24
<210>52
<211>24
<212>dna
<213>人工序列()
<400>52
gtaacatggagcaggtcgcggatt24
<210>53
<211>24
<212>dna
<213>人工序列()
<400>53
agcctctgtagccgttgctaccct24
- 还没有人留言评论。精彩留言会获得点赞!