人类Y染色体35个STR基因座的荧光标记复合扩增试剂盒及其应用的制作方法
本发明属于法医遗传学领域,涉及一种荧光标记复合扩增检验系统,具体为人类y染色体35个str基因座的荧光标记复合扩增试剂盒及其应用。
背景技术:
上世纪90年代,荧光标记的短串联重复序列基因座(shorttandemrepeat,str)被首次提出,并作为遗传标记进行法医物证学相关方面的研究及鉴定。str标记与其他遗传标记物相比,str基因座片段小、容易扩增,更适用于微量和降解检材,多个str基因座可以同时进行复合扩增,混合检材、dna分子降解的检材和微量生物检材等疑难生物检材能够成功进行dna检验及分型,因而具有快速、高效、准确、灵敏、信息量大等优点。近年来,str分型技术被广泛的应用于法医物证中的个体识别和亲子鉴定,尤其是在dna数据库的建设方面,与以往rflp等技术相比,str复合扩增技术具有极大的优越性。
y染色体str遗传标记是指存在于人类y染色体非重组区域的短串联重复序列。y染色体str遗传标记与常染色体str遗传标记相比,具有男性特有、父系遗传及单倍型遗传等三大特征,将其独特性与str位点分型检测的优越性相结合,可进行法医学个体识别、亲子鉴定及dna家谱构建等。
y-str独特的优势在于同一父系所有男性样本分型一致,可以作为家系排查的工具,从而有效缩小侦查范围,低突变基因座更加适用于家系排查,但低突变基因座往往多态性较低,不利于家系之间的区分。法医上将突变率高于1%的基因座称为快速突变基因座(rapidlymutatingy-str)。快速突变基因座具有较高的相关男性区分能力,而且一般具有较高的多态性。因此,y-str试剂盒需要基因座的数量要多,同时基因座的多态性要高,以保证试剂盒具有足够高的整体多态性。
技术实现要素:
为了克服现有技术的缺陷,获得一种具有高分辨率且能够满足y-str数据库建设及男性个体家系排查的试剂盒,本发明提供了人类y染色体35个str基因座的荧光标记复合扩增试剂盒及其应用。
其技术方案是这样的,人类y染色体35个str基因座的荧光标记复合扩增试剂盒,35个str基因座包括28个低突变y-str基因座dys392、dys389i、dys447、dys389ii、dys438、dys527a、dys527b、dys596、dys391、dys456、dys19、dys448、dys385a、dys385b、dys549、dys437、dys481、dys533、dys390、dys458、dys460、dys393、y_gata_h4、dys439、dys635、dys444、dys643、dys557,以及7个快速突变y-str基因座dys627、dys576、dys570、dyf387s1a、dyf387s1b、dys449、dys518;
所述试剂盒共包括31对特异性引物,用于扩增所述35个str基因座,所述特异性引物的序列为:dys392、seqidno:1~2;dys389i和dys389ii、seqidno:3~4;dys447、seqidno:5~6;dys438、seqidno:7~8;dys527a和dys527b、seqidno:9~10;dys596、seqidno:11~12;dys391、seqidno:13~14;dys456、seqidno:15~16;dys19、seqidno:17~18;dys448、seqidno:19~20;dys385a和dys385b、seqidno:21~22;dys549、seqidno:23~24;dys437、seqidno:25~26;dys481、seqidno:27~28;dys533、seqidno:29~30;dys390、seqidno:31~32;dys627、seqidno:33~34;dys458、seqidno:35~36;dys460、seqidno:37~38;dys393、seqidno:39~40;y_gata_h4、seqidno:41~42;dys439、seqidno:43~44;dys635、seqidno:45~46;dys444、seqidno:47~48;dys643、seqidno:49~50;dys557、seqidno:51~52;dys576、seqidno:53~54;dys570、seqidno:55~56;dyf387s1a和dyf387s1b、seqidno:57~58;dys449、seqidno:59~60;dys518、seqidno:61~62;
所述特异性扩增引物分为五组:dys392、dys389i、dys447、dys389ii、dys438、dys527a、dys527b和dys596为第一组,dys391、dys456、dys19、dys448、dys385a、dys385b和dys549为第二组,dys437、dys481、dys533、dys390、dys627、dys458和dys460为第三组,dys393、y_gata_h4、dys439、dys635、dys444、dys643和dys557为第四组,dys576、dys570、dyf387s1a、dyf387s1b、dys449和dys518为第五组;每对引物中至少一条采用荧光染料标记其5’端。
优选的,所述特异性引物在扩增体系中的终浓度为:dys392、0.072μm;dys389i和dys389ii、0.078μm;dys447、0.096μm;dys438、0.06μm;dys527a和dys527b、0.06μm;dys596、0.12μm;dys391、0.06μm;dys456、0.054μm;dys19、0.144μm;dys448、0.054μm;dys385a和dys385b、0.084μm;dys549、0.36μm;dys437、0.072μm;dys481、0.084μm;dys533、0.12μm;dys390、0.12μm;dys627、0.264μm;dys458、0.36μm;dys460、0.42μm;dys393、0.09μm;y_gata_h4、0.144μm;dys439、0.084μm;dys635、0.12μm;dys444、0.18μm;dys643、0.42μm;dys557、0.48μm;dys576、0.108μm;dys570、0.156μm;dyf387s1a和dyf387s1b、0.192μm;dys449、0.3μm;dys518、0.528μm。
该试剂盒包括的35个基因座对应的引物及其浓度具体如表1所示:
表1.35个ystr基因座引物信息
优选的,所述荧光染料为:6-fam、hex、tamra、rox、atto中任意一种,且每组采用的荧光标记不同,内标选用橙色荧光siz。
优选的,所述试剂盒包括反应混合液(reactionmix)、特异性复合扩增引物、热启动taq酶、dna标准品、sdh2o、35个基因座的等位基因和荧光分子量内标;其中反应混合液的组成为:mgcl27.5mm,tris-hcl125mm,kcl125mm,dntps7.5mm,bsa2g/l。各组分及其含量如下:reactionmix10.0μl,基因组dnaxμl含量为0.125-5ng,引物混合物5.0μl,热启动taq酶(5u/μl)0.5μl,sdh2o补足至25.0μl。
以上所述的人类y染色体35个str基因座的荧光标记复合扩增试剂盒在法医鉴定、亲权鉴定或dna家谱构建中的应用。步骤如下:收集基因组dna,进行pcr扩增,分析扩增产物;其中pcr扩增程序为:变性95℃2min,循环94℃30s,60℃1min,65℃1min,30个循环,终止延伸72℃20min,4℃维持。
优选的,法医鉴定、亲权鉴定或dna家谱构建中采用的样品来源包括利用chelex法、磁珠提取法或有机抽提法提取的人基因组dna;或采用免提取的滤纸、fta卡、棉签、纱布中任意一种载体收集的人类血液或口腔细胞。检材的来源包括人的血液、血痕、精液、唾液、体液、毛发、肌肉或组织器官。
上述人类y染色体35个str基因座的荧光标记复合扩增试剂盒的使用方法,其特征在于:包括以下步骤:
a、配制扩增体系,扩增体系为:reactionmix10.0μl,基因组dnaxμl含量为0.125-5ng,引物混合物5.0μl,热启动taq酶(5u/μl)0.5μl,sdh2o补足至25.0μl;
b、扩增热循环,扩增程序为,初始变性,95℃2分钟;热循环,94℃30秒,60℃1min,68℃1min,30个循环;终延伸,72℃20分钟;保温,4℃维持;其中,扩增后的样品应避光保存;
c、扩增产物在遗传分析仪上荧光检测,将12.5μl上样混合物与1μl扩增产物或系统中35个y基因座等位基因分型标准物allelicladder(无锡中德美联生物技术有限公司)混合,95℃变性3分钟,冰浴3分钟,采用遗传分析仪进行电泳检测,其中,电泳采用多道或单道毛细管电泳;
所述上样混合物的配制如下:0.5μl分子量内标agcumarkersiz-500×(进样数)+(12μl去离子甲酰胺)×(进样数);
d、分型分析,用片段分析软件genemapperid-x分析步骤c中遗传分析仪检测收集的数据。
本发明的人类y染色体35个str基因座的荧光标记复合扩增试剂盒,具有如下有益效果:(1)本发明所述试剂盒包括28个低突变率和7个快速突变、多态性较高的y-str基因座,兼顾了男性家系排查和同一父系不同男性个体之间的区分,为市面上基因座数量最多的y-str检测产品;(2)本发明所述试剂盒中引物具有特异性强,灵敏度高,分型结果准确的优点,完全能够满足实际案件检验、dna数据库建设以及亲子鉴定的需要;(3)本发明所述试剂盒具有很强的检材适应性。
附图说明
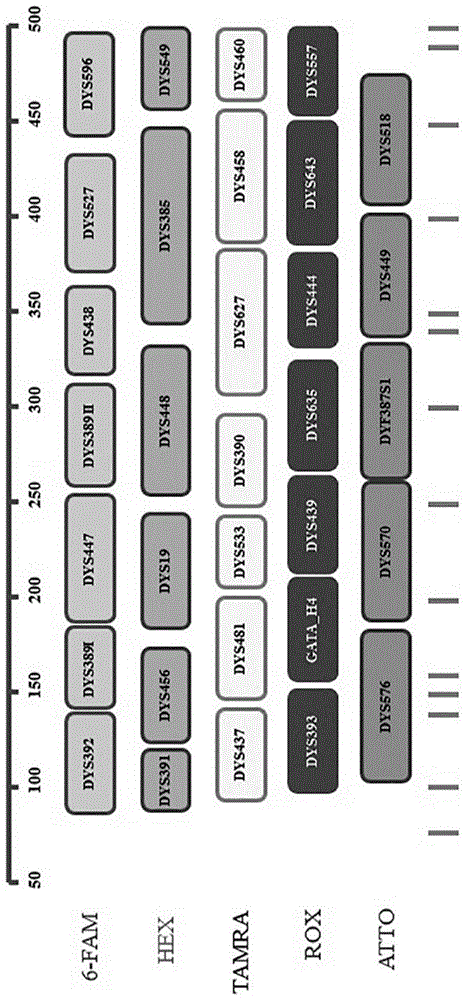
图1为本试剂盒的35个y基因座的排布示意图。
图2为本试剂盒标准dna9948分型图。
图3为35个y基因座等位基因分型标准物:allelicladder图。
图4为家系样本1的y-str分型图。
图5为家系样本2的y-str分型图。
具体实施方式
以下实施例进一步说明本发明的内容,但不应理解为对本发明的限制。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改和替换,均属于本发明的范围。
若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
实施例1
一、基因座筛选
通过对dys19、dys385a/b、dys389i、dys389ii、dys390、dys391、dys392、dys393、dys437、dys438、dys439、dys448、dys456、dys458、dys635、y_gata_h4、dys481、dys533、dys576、dys643、dys460、dys549、dyf387s1a、dyf387s1b、dys449、dys518、dys627、dys570、dys527a/b、dys447、dys444、dys557、dys596、dys446、dys510、dys622、dys443、dys587、dys522、y_gata_a10、dys520、dys552、dys593、dys531、dys459a/b、dys508、dys388、dys617、dys645、dys713、dys630、dyf404s1、dys626、dys526a/b共59个y染色体短串联重复序列基因座及其等位基因遗传特征进行了深入的研究,筛选出35个y染色体短串联重复序列基因座:dys392、dys389i、dys447、dys389ii、dys438、dys527a、dys527b、dys596、dys391、dys456、dys19、dys448、dys385a、dys385b、dys549、dys437、dys481、dys533、dys390、dys627、dys458、dys460、dys393、y_gata_h4、dys439、dys635、dys444、dys643、dys557、dys576、dys570、dyf387s1a、dyf387s1b、dys449、dys518。这35个y染色体短串联重复序列基因座在人群中具有较低的遗传多态性和良好的等位基因频率分布,复合本发明对于位点的要求。每个基因座相应信息见表2:
表2.35个ystr基因座信息
二、基因座排布
根据以上35个基因座,设计了独特的基因座排布方式及化学荧光染料标记的方法:dys392、dys389i、dys447、dys389ii、dys438、dys527a、dys527b和dys596为第一组,荧光染料标记物为6-fam;dys391、dys456、dys19、dys448、dys385a/b、dys549为第二组,荧光染料标记物为hex;dys437、dys481、dys533、dys390、dys627、dys458、dys460为第三组,荧光染料标记物为tamra;dys393、y_gata_h4、dys439、dys635、dys444、dys643、dys557为第四组,荧光染料标记物为rox;dys576、dys570、dyf387s1a、dyf387s1b、dys449、dys518为第五组,荧光染料标记物为atto,基因座排布如图1。
三、特异性引物设计及复合扩增条件的建立
通过基因座名称或者y染色体位置,利用ucsc或ncbi网站进行基因座序列下载;其次,根据每个基因座重复单元两侧的序列进行引物设计。
(1)特异性引物设计
在设计引物时,使用的设计软件最佳搭配是premier和oligo合并使用,以premier进行自动搜索,oligo进行分析评价,引物碱基分布要随机、tm值相近、gc含量在40%~60%之间、引物自身及引物之间不应存在互补序列;同时也要保证引物扩增的特异性,在ncbi数据库中使用primer-blast软件对设计的引物进行比对分析,要充分考虑引物3’端的特异性,因为引物3’端序列同源性较高的话容易导致错误引发。
随着复合扩增系统中引物数量的增加,不同基因座引物之间的相互干扰也越来越严重,反应体系的动力学变得越来越复杂,因此需要设计大量的引物序列进行复杂的测试,最终保证试剂盒的扩增特异性及效率。
(2)复合扩增条件的建立
先对35个基因座的单一扩增条件进行优化,在成功地建立了单个基因座扩增条件的基础上,研究35个基因座复合扩增pcr反应条件,通过大量的反复实验确定了复合扩增中的各个参数,如,循环参数、退火温度、缓冲液离子强度、酶量、复合扩增反应体积的变化以及模板dna量等,使扩增产物达到平衡、特异的要求,建立起复合扩增体系,同时扩增出35个基因座,最终引物序列及浓度见表1。
四、调整pcr反应混合液
pcr体系中,mg2+浓度分别测试1.0mm、1.5mm、2.0mm、2.5mm、3.0mm5个梯度,dntps浓度分别测试0.15mm、0.2mm、0.25mm、0.3mm、0.35mm5个梯度,热启动taq酶含量分别测试1.0u、1.5u、2.0u、2.5u、3.0u共5梯度,tris-hcl浓度定为10mm,kcl浓度定为40mm。通过设计正交实验,最终将mg2+浓度定为2.0mm,dntps浓度定为0.25mm,热启动taq酶含量2.0u,tris-hcl浓度定为10mm,kcl浓度定为40mm,利用以上材料制备成反应混合液reactionmix,加入到pcr体系中。最终pcr体系各组成如下:reactionmix10.0μl,基因组dnaxμl含量为0.125-5ng,引物混合物5.0μl,热启动taq酶(5u/μl)0.5μl,sdh2o补足至25.0μl。
实施例2
人类y染色体35个str基因座的荧光标记复合扩增试剂盒的使用方法,包括以下步骤,
a、配制扩增体系,扩增体系为:reactionmix10.0μl,基因组dnaxμl含量为0.125-5ng,引物混合物5.0μl,热启动taq酶(5u/μl)0.5μl,sdh2o补足至25.0μl;
b、扩增热循环,扩增程序为,初始变性,95℃2分钟;热循环,94℃30秒,60℃1min,68℃1min,30个循环;终延伸,72℃20分钟;保温,4℃维持;其中,扩增后的样品应避光保存;
c、扩增产物在遗传分析仪上荧光检测,将12.5μl上样混合物与1μl扩增产物或系统中35个y基因座等位基因分型标准物allelicladder(无锡中德美联生物技术有限公司)混合,95℃变性3分钟,冰浴3分钟,采用遗传分析仪进行电泳检测,其中,电泳采用多道或单道毛细管电泳;
所述上样混合物的配制如下:0.5μl分子量内标agcumarkersiz-500×(进样数)+(12μl去离子甲酰胺)×(进样数);
d、分型分析,用片段分析软件genemapperid-x分析步骤c中遗传分析仪检测收集的数据,其中,dna标准对照品9948和等位基因分型标准物的分型结果如图2、图3所示。
实施例3同一家系中个体排查的应用
(1)收集家系排查中的家系样本:共2份,样本由某公安局提供。
(2)样本dna提取:采用法庭科学dna实验室检验规范【ga-t383-2002】中硅珠法提取方法。
(3)利用本发明试剂盒、agcudatabasey30试剂盒分别对以上两个家系样本进行分型检测,然后将两种试剂盒的分型结果进行对比。其中,本发明试剂盒检测的两个家系样本的y-str分型图如图4、图5所示。
分型结果比对如下表:
结果显示,使用agcudatabasey30检测这两份家系样本,所得分型完全一致,无法进行个体的区分识别。而本试剂盒中加入了7个高突变基因座,大大提升了试剂盒的个体识别能力,扩增相同的2份家系样本,dys627、dyf381s1a/b、dys518共3个基因座发现分型不一致情况,可有效区分个体,提高了试剂盒个体识别能力。
序列表
<110>安徽省公安厅物证鉴定中心
安徽安科生物工程(集团)股份有限公司
无锡中德美联生物技术有限公司
<120>人类y染色体35个str基因座的荧光标记复合扩增试剂盒及其应用
<141>2019-10-31
<160>62
<170>siposequencelisting1.0
<210>1
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>1
aaacagagggatcattaaacct22
<210>2
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>2
atttttttcttgtatcaccattta24
<210>3
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>3
tccaactctcatctgtattatct23
<210>4
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>4
acacaattatccctgagtagc21
<210>5
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>5
atacattttagggagacataagg23
<210>6
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>6
gggaagtacatggaagcgg19
<210>7
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>7
aagataaataataatagtattag23
<210>8
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>8
cgtggtggcagacgcctataatcc24
<210>9
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>9
gaaggaaggaaggaaatgaaac22
<210>10
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>10
cataaaaagtgtgatatttcaat23
<210>11
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>11
ctttcagcaaccttatttcagataac26
<210>12
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>12
cattttattattgttctttcactttat27
<210>13
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>13
tcattcaatcatacacccata21
<210>14
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>14
gagggataggtaggcaggcagat23
<210>15
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>15
caacagaccatctgcaggct20
<210>16
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>16
cagtttaagaagttttggg19
<210>17
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>17
agtcaatttgatctcatacctagac25
<210>18
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>18
actctccttaacccagatgga21
<210>19
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>19
tctctatctccctttctgtctc22
<210>20
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>20
agaggctagaaaaaagggaacccagct27
<210>21
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>21
gggtgtcatgaactgaaatga21
<210>22
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>22
ggtaagggctgctgaccag19
<210>23
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>23
aactatgctggcctaatagaatga24
<210>24
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>24
gaaataataaggtagacatagca23
<210>25
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>25
tagaccggatgggcatgcact21
<210>26
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>26
tctatcatctgtgaatgatgtctat25
<210>27
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>27
gctaaaaggaatgtggctaac21
<210>28
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>28
cagagccccacaacccaagaag22
<210>29
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>29
gatatagataaatattaggtagatgac27
<210>30
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>30
ctgatcatgaataaatacatttc23
<210>31
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>31
aatatcacagaacatcgtaatcc23
<210>32
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>32
cattgcaatgtgtatactcaga22
<210>33
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>33
agtgcagtggcattatctgtgc22
<210>34
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>34
taaaaacttgccatttgctcattatc26
<210>35
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>35
agagacaatgtttcacca18
<210>36
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>36
ggcgtggtggcaggaac17
<210>37
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>37
gatagacagatatatctaataggtaga27
<210>38
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>38
gcaggaagattaaaggttgtcta23
<210>39
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>39
tgtcattcctaatgtggtcttctac25
<210>40
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>40
tgtctcatagaaaagacatacat23
<210>41
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>41
cacttgtatcctaggaatcatcatta26
<210>42
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>42
cacctatctatgtatctatctatctat27
<210>43
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>43
gttagacctaaaaccataacaact24
<210>44
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>44
gtggattatgcctgtaatcccaac24
<210>45
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>45
gtgagacactcttgatatctatggat26
<210>46
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>46
aacaatgtatgtgcaagtgtctt23
<210>47
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>47
gttgatgtgattataagtgatttatg26
<210>48
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>48
tgcctttggatcccttagacagcc24
<210>49
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>49
gtgcagccccactttctggttcac24
<210>50
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>50
gccacccttcccttgagttc20
<210>51
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>51
ggaagtcttgactctcagac20
<210>52
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>52
aatgatatgtcacactgccccgtt24
<210>53
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>53
agagatgaggtcttgctatgttgctt26
<210>54
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>54
aaaagccaagacaaatacgctta23
<210>55
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>55
tcattcattttacgtttcataac23
<210>56
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>56
gtttcttgactatgctgaataattttca28
<210>57
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>57
ctgtcacccaggctggagtgc21
<210>58
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>58
cacacttctcacactgtggcacc23
<210>59
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>59
aatcacacaagttaaggga19
<210>60
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>60
cctcagcctcctgagtagctggga24
<210>61
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>61
cctgcctcagcctcatgagtagttg25
<210>62
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>62
actaagtccactattcttggagc23
- 还没有人留言评论。精彩留言会获得点赞!