一种激素性股骨头坏死易感基因panel的制作方法
本发明属于基因检测领域,具体涉及一种用于激素性股骨头坏死易感性检测的基因panel,还涉及该基因panel在基因检测装置中的应用。
背景技术:
糖皮质激素因其抗炎、抗过敏、抗休克、非特异性抑制免疫等多种作用,被广泛应用于自身免疫性疾病、过敏反应性疾病、严重感染和器官移植后状态等,但激素性股骨头坏死(glucocorticoid-associatedosteonecrosisoffemoralhead,ga-onfh)是其严重并发症。ga-onfh好发于30-40岁的青壮年,往往起病隐匿,常在发现时即已发生股骨头塌陷变形,致残率高,对个人及社会带来巨负担,是骨科的难治性疾病。
然而,临床实践证明并非每一个接受糖皮质激素治疗的患者都会发生ga-onfh,这一比例在接受激素治疗的系统性红斑狼疮病人中为0.8%-33%,在肾移植病人中约为6%,在急性淋巴细胞白血病或非霍奇金淋巴瘤病人中约为15.5%。虽然激素剂量被报道可能与ga-onfh的发生存在相关性,但在实际临床工作中,接受相似激素治疗方案的患者,也并非均会发生股骨头坏死,这说明激素性股骨头坏死是用药因素和个体因素共同作用而导致的复杂性疾病。遗传因素作为最重要的个体因素之一,被报道与ga-onfh的发生相关。随着分子生物学与遗传学的深入研究,越来越多的结果证实ga-onfh具有极大的遗传易感性,但现有的研究基于候选单核苷酸多态性位点(snp)分型方法,一次检测一个到数个位点,无法全面地反映ga-onfh的遗传易感性,因此无法建立准确地、适用于临床实践的预测模型。
用于高通量的筛查基因组多态性位点的方法包括芯片(microarrays)技术和二代测序(nextgenerationsequencing)技术。芯片技术只能筛查数据库已知的多态性位点,无法检测新发现或数据库未录入的多态性位点,开发拓展性能有限;二代测序技术,包括全基因组测序、靶向捕获测序、多重pcr靶向测序。其中,全基因组测序可以在全基因组水平上检测多态性位点,但测序数据量大,成本高昂;目标区域靶向捕获测序,例如全外显子捕获测序,适合大数据的靶向捕获,操作复杂且开发成本高;多重pcr靶向多态性位点检测是一种多重pcr和高通量测序相结合的高效检测技术,通过对多个待检位点设计特异性引物,利用多重pcr技术进行扩增,在保证扩增均一性的前提下,对snp、indel等几百甚至上千个多态性位点进行快速靶向线性扩增后,结合高通量测序技术实现对大样本多位点的检测,操作简单灵活,通过两轮pcr就能完成文库构建,适合中小通量的数据量。
目前,尚无用于预测ga-onfh发生的基因集panel,也无二代测序方法在预测ga-onfh中的应用。既往研究只基于对已有ga-onfh发病机制的研究,对少数与激素代谢、血管功能、骨代谢及脂代谢等关键基因位点进行检测。但ga-onfh是复杂的全身代谢性疾病和多基因相关疾病,目前对ga-onfh发病机制的研究仍不清楚,既往研究涵盖的基因位点有限,需要二代测序方法进行更高通量的检测,以确定用于ga-onfh风险检测的更优化的snp组合。
技术实现要素:
为了弥补现有激素性股骨头坏死易感性预测和检测技术的上述不足,发明人对已知的骨坏死及糖皮质激素代谢相关基因的进行归纳和分析,发现了许多基因与ga-onfh高度相关,从而开发出一种新的激素性股骨头坏死易感性基因panel,该基因panel覆盖度广,能够用于从ga-onfh风险评估到ga-onfh的诊断、预后等ga-onfh发生发展的各个阶段,并且可移植性强,适用于目前国内市场的测序仪器或新开发的检测平台。具体而言,本发明包括如下技术方案。
一种激素性股骨头坏死(ga-onfh)易感基因panel,其为包括下述587个人类基因组位点的基因集:
rs710322、rs2294486、rs2294487、rs150880809、rs7407、rs144487103、rs12738235、rs10909811、rs78132938、rs11121552、rs9435659、rs761421、rs2076603、rs4920608、rs67232976、rs6666034、rs2064374、rs2295942、rs3748705、rs600674、rs12568483、rs74843031、rs10493845、rs78167983、rs4658219、rs147504208、rs79090643、rs6424379、rs11579366、rs2004659、rs9728345、rs2231375、rs2280474、rs4971066、rs17472444、rs7550383、rs10919082、rs4656656、rs4656657、rs7526721、rs10800420、rs3748618、rs2274227、rs3795503、rs10797850、rs1256930、rs10900525、rs11120575、rs1059704、rs2297545、rs2297543、rs2076149、rs9435867、rs7539016、rs7549137、rs7551511、rs2049182、rs10489691、rs3795375、rs3795374、rs963982、rs963981、rs4659948、rs562038978、rs111896385、rs1056104、rs7560262、rs58882124、rs200809231、rs187904341、rs937725、rs12233086、rs11691981、rs777432、rs12052654、rs13025791、rs13025959、rs2255161、rs11686946、rs35720878、rs2124971、rs2272499、rs16860497、rs2293649、rs56084828、rs2305414、rs1046356、rs2287600、rs13021295、rs59458664、rs3214826、rs2305541、rs2290464、rs2924811、rs2971863、rs1470864、rs2167885、rs78122518、rs72982319、rs143258246、rs838543、rs838549、rs78451449、rs151152774、rs79471829、rs117751285、rs200139615、rs3790993、rs200039497、rs456168、rs6796483、rs6777976、rs61743511、rs61743461、rs2070987、rs1546737、rs10933973、rs7614116、rs75144949、rs3732755、rs12492699、rs150242411、rs583550、rs35070271、rs55683908、rs843358、rs843357、rs11924016、rs371821065、rs6762208、rs1823238、rs6804448、rs138704337、rs28434055、rs322117、rs11355796、rs16844401、rs75501914、rs730420、rs2269877、rs62289296、rs62289297、rs4320134、rs3755863、rs8192678、rs11940551、rs6837020、rs77939156、rs4031826、rs2219471、rs3214870、rs3816584、rs2305946、rs7655964、rs1870377、rs7349683、rs3836713、rs861340、rs13128286、rs3966087、rs2175766、rs61758879、rs2241894、rs13147012、rs6847454、rs1039808、rs3756122、rs17024037、rs12650966、rs2289318、rs12648678、rs29675、rs1126417、rs162850、rs162848、rs332811、rs37368、rs4371784、rs3797209、rs1423099、rs10070440、rs423906、rs401302、rs3749669、rs59653731、rs10477486、rs6888031、rs11283943、rs2227950、rs2303076、rs2306960、rs3812036、rs70997617、rs3817064、rs3817063、rs75304543、rs368030749、rs372469036、rs778456436、rs1749158、rs2842949、rs6239、rs11754288、rs13204445、rs3749877、rs3749878、rs76902576、rs80125253、rs2297740、rs7762830、rs12435、rs1132742、rs1131123、rs1131500、rs1140546、rs1051488、rs3808343、rs117545108、rs4720537、rs4222、rs67361882、rs2286213、rs4721888、rs73705179、rs78155900、rs114097201、rs367830344、rs375743758、rs4646453、rs71540916、rs71540918、rs1424426、rs12671813、rs2278130、rs2069459、rs2069456、rs72494453、rs61013791、rs7829535、rs658948、rs2280335、rs35858677、rs145012061、rs34462909、rs17092126、rs1078363、rs6996616、rs2278467、rs1042381、rs10956412、rs17056759、rs6435、rs6397、rs12549574、rs11793555、rs7853758、rs2027433、rs3737309、rs2015408、rs2275137、rs10796042、rs2282383、rs6602051、rs7100510、rs1954181、rs12256835、rs2297329、rs2297328、rs11008968、rs148671234、rs138210257、rs10827116、rs2230396、rs2230395、rs2230394、rs2660169、rs735877、rs3736583、rs3736582、rs7912404、rs4986789、rs7894、rs4575219、rs3739968、rs6578504、rs11603496、rs3740620、rs11605894、rs11605072、rs398075678、rs96489、rs331510、rs2657167、rs10836954、rs11037444、rs60832895、rs11037445、rs16930982、rs16930998、rs55802517、rs12272856、rs1043388、rs1043390、rs1128396、rs2072651、rs10458926、rs10833050、rs10766524、rs2280331、rs369353409、rs574381、rs626670、rs2286163、rs200951888、rs57503021、rs7932320、rs7130258、rs66861805、rs7950735、rs291241、rs7931870、rs2186627、rs12790613、rs503223、rs1893764、rs79715120、rs79669172、rs77206991、rs79417294、rs114979979、rs75994039、rs75240316、rs59358830、rs57751948、rs775990209、rs2075626、rs58283839、rs2241281、rs2970827、rs2241280、rs2241279、rs12426675、rs1800692、rs11542844、rs2273986、rs7138535、rs4763232、rs7135018、rs12313469、rs10772423、rs12370363、rs78152338、rs2708381、rs2600357、rs2600356、rs2599404、rs2600355、rs11557132、rs61928643、rs9943714、rs9943809、rs2280446、rs6581565、rs825074、rs10861953、rs4072888、rs12821855、rs73166888、rs1451772、rs1817104、rs1669412、rs9315906、rs7982426、rs520342、rs3092904、rs12429818、rs7331894、rs1805097、rs2275841、rs2228036、rs1713417、rs2297612、rs4981349、rs74036552、rs150516189、rs1808975、rs2235962、rs3742885、rs181572、rs75391113、rs60593979、rs2180770、rs3837659、rs3829955、rs7153601、rs11626446、rs17127245、rs4401027、rs60585083、rs11366248、rs2260160、rs2467426、rs12441861、rs2277531、rs3759791、rs12702、rs2228368、rs3736180、rs8023508、rs12592155、rs28706938、rs28673176、rs2277598、rs57373048、rs17875502、rs397954069、rs28504011、rs11259927、rs2304796、rs3743262、rs10664668、rs4646626、rs3832984、rs12447306、rs2667661、rs2667660、rs2240691、rs2302607、rs115465506、rs1050113、rs14122、rs11865800、rs11076125、rs11076176、rs12691052、rs9928398、rs2241619、rs2241620、rs58353328、rs3803640、rs11557187、rs2077412、rs2663345、rs2286672、rs10999、rs3744400、rs16956647、rs11078698、rs76317718、rs148830167、rs61274670、rs2074274、rs2074273、rs2074272、rs111232194、rs2074146、rs4796030、rs2074519、rs3744372、rs2304967、rs143471015、rs1719152、rs16532、rs2227322、rs25645、rs4072639、rs2070833、rs756100328、rs79526563、rs2301682、rs143818567、rs2285524、rs4793639、rs3216895、rs2301627、rs2301625、rs3785925、rs2075555、rs2075554、rs2256835、rs2665799、rs2302133、rs2302134、rs10605889、rs7208382、rs11077414、rs9909216、rs12941264、rs690371、rs2467577、rs2070919、rs690514、rs2070918、rs1688149、rs492095、rs35286723、rs2306690、rs552432、rs2305027、rs2240900、rs7243349、rs17851892、rs7234849、rs3763951、rs713041、rs35870594、rs10421267、rs11671881、rs8120、rs2292151、rs7255265、rs2240745、rs2277969、rs35647251、rs137943075、rs7245563、rs2056820、rs56156262、rs36008189、rs12459147、rs34433978、rs3833221、rs305968、rs12975986、rs34650210、rs305975、rs73039860、rs1799782、rs17725124、rs2547238、rs45468391、rs8100114、rs4802905、rs12609379、rs12977654、rs56960413、rs862708、rs78628129、rs398059497、rs41275620、rs6133723、rs2295576、rs1078761、rs6030462、rs3746504、rs3737063、rs3918254、rs13969、rs140429049、rs8133052、rs1056892、rs11701124、rs8131313、rs8132521、rs35796750、rs881712、rs2839077、rs59769541、rs11464071、rs11089978、rs11089977、rs165657、rs361666、rs361762、rs362003、rs362124、rs362132、rs361611、rs361580、rs362011、rs361721、rs1476576、rs11089442、rs2074731、rs3218318、rs2072862、rs5758651、rs6006950、rs200564038、rs140313、rs372438307、rs376698610、rs6654760、rs2071776、rs2854412、rs6616890、rs143921252。
上述激素性股骨头坏死(ga-onfh)易感基因panel可以用于构建基于多态性位点的ga-onfh风险预测模型。
上述ga-onfh风险预测模型选自下组:以or值作为权重的遗传风险评分(anoddsratioweightedgeneticriskscore,or_grs)模型、可释方差遗传风险评分(plainedvarianceweightedgeneticriskscore,ev_grs)模型。
其中,在以or值作为权重的遗传风险评分(or_grs)模型中,计算所有纳入模型snp的or值权重,该评分方法的计算公式如下:
ωi=ln(ori),
上述or_grs模型又可以是基于or_grs风险评分的单因素逻辑回归分析模型和基于or_grs风险评分的多因素逻辑回归分析模型,其中,
基于or_grs风险评分的单因素逻辑回归分析模型的训练公式为:
logitp(d=1|g)=α+β(or_grs)。
基于or_grs风险评分的多因素逻辑回归分析模型是同时考虑or_grs、性别(sex)、年龄(age)、原发病(primarydisease)进行的多因素逻辑回归分析模型训练,公式如下:logitp(d=1|g)
=α+β1(or_grs)+β2(sex_0)+β3(sex_1)+β4(age_0)+β5(age_1)
+β6(primarydisease_1)+β7(primarydisease_2)。
在可释方差遗传风险评分(ev_grs)模型中,同时考虑snp效应和风险等位基因频率(即最小等位基因频率maf),该评分方法的计算公式如下:
上述ev_grs模型又可以是ev_grs单因素逻辑回归分析模型或者ev_grs多因素逻辑回归分析模型,其中,
ev_grs单因素逻辑回归分析模型训练的回归公式为:
logitp(d=1|g)=α+β(ev_grs)。
ev_grs多因素逻辑回归分析模型是同时考虑grs、性别(sex)、年龄(age)、原发病(primarydisease)四个因素进行的多因素逻辑回归分析模型训练,公式为:
logitp(d=1|g)=
α+β1(ev_grs)+β2(sex_0)+β3(sex_1)+β4(age_0)+β5(age_1)+
β6(primarydisease_1)+β7(primarydisease_2)。
上述ga-onfh风险预测模型可以通过编程、通过数学软件包形式被输入计算机或者基因检测装置比如illumina测序平台,从而预测受试者是否属于激素性股骨头坏死易感人群,方便医生制定给药方案。
本发明的另一个方面在于提供一种激素性股骨头坏死易感性检测装置和配套的试剂盒,其包括用于检测上述基因panel中基因的引物或者dna/rna探针。
例如,用于检测基因panel中人类基因组位点的引物序列如表2所示。
上述的基因panel和引物适用于多重pcr靶向测序、ga-onfh预测,通过检测生物样本中的基因组,可以进行ga-onfh的风险预测。
本发明构建的基因panel在用于预测ga-onfh风险时,具有如下优点:
1.属于首个预测ga-onfh的基因panel。通过对目前公认的常用数据库中骨坏死及糖皮质激素代谢相关基因进行筛选,最终选择的基因覆盖度广,与ga-onfh相关性高。该技术的开发并不局限于是否接受糖皮质激素治疗或是否患有骨坏死,是首个可靠的用于预测ga-onfh发生的基因检测panel。
2.检测成本低,适用范围更广。本发明开发的panel适用于主流illumina测序平台,相对于目前其他二代测序技术检测方法而言,本发明的panel检测平台对基因的检测和分析成本更低,极具推广价值。
3.分析便捷,可移植性强,可操作性强。该技术中的信息分析形成的软件包能够安装于多个平台,从下机数据到各种变异结果一步即可完成,分析时间大大缩短,为患者争取宝贵的检测分析时间;结果呈现更加直观,可直接用于指导实际临床工作。
附图说明
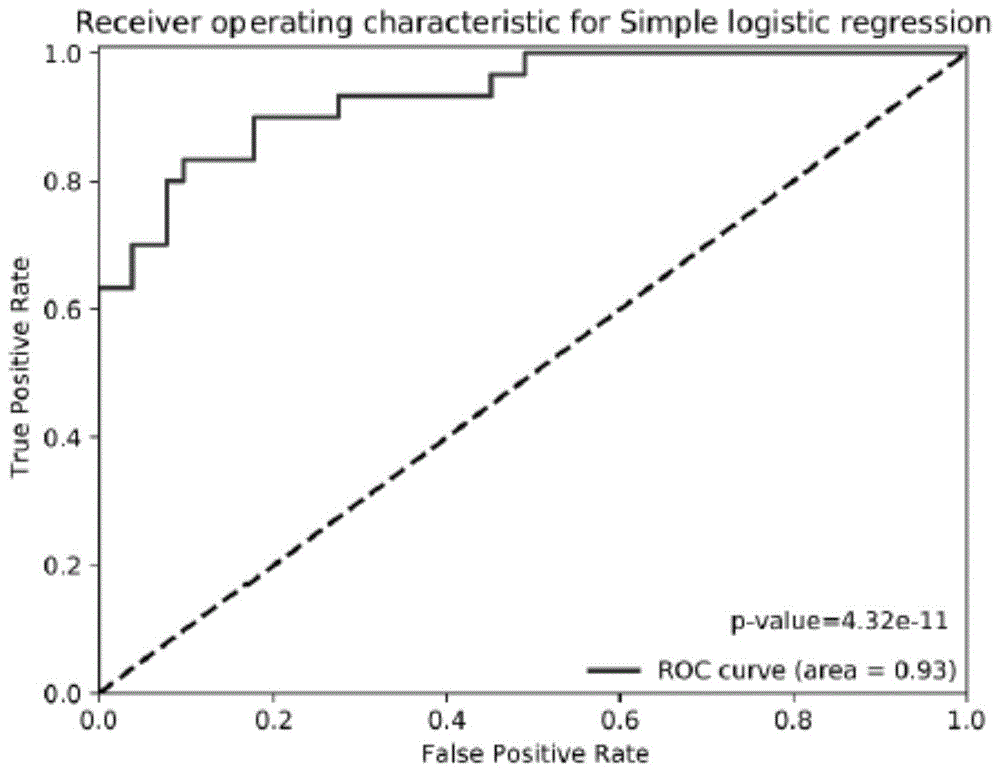
图1显示了验证本发明基因panel的or_grs单因素逻辑回归分析模型的训练集验证结果的受试者工作特征曲线(receiveroperatingcharacteristic,roc)。纵坐标为真阳性率(truepositiverate,tpr)或者灵敏度(sensitivity),横坐标为假阳性率(falsepositiverate,fpr)或者特异性(specificity)。
图2显示了验证本发明基因panel的or_gr单因素逻辑回归分析模型的训练集分层5折交叉验证结果的roc曲线。
图3显示了验证本发明基因panel的ev_grs单因素逻辑回归分析模型的训练集验证结果的roc曲线。
图4显示了验证本发明基因panel的ev_grs单因素逻辑回归分析模型的训练集分层5折交叉验证结果的roc曲线。
图5显示了验证本发明基因panel的or_grs多因素逻辑回归分析模型的训练集验证结果的roc曲线。
图6显示了验证本发明基因panel的or_grs多因素逻辑回归分析模型的训练集分层5折交叉验证结果的roc曲线。
图7显示了验证本发明基因panel的ev_grs多因素逻辑回归分析模型的训练集验证结果的roc曲线。
图8显示了验证本发明基因panel的ev_grs多因素逻辑回归分析模型的训练集分层5折交叉验证结果的roc曲线。
图9显示了验证本发明基因panel的or_grs多因素逻辑回归分析模型对测试集预测结果的roc曲线。
图10显示了验证本发明基因panel的ev_grs单因素逻辑回归分析模型对测试集预测结果的roc曲线。
图11显示了验证本发明基因panel的or_grs多因素逻辑回归分析模型对测试集预测结果的roc曲线。
图12显示了验证本发明基因panel的ev_grs多因素逻辑回归分析模型对测试集预测结果的roc曲线。
具体实施方式
以下结合具体实施例对本发明做进一步详细说明。应理解,以下实施例仅用于说明本发明而非用于限定本发明的范围。
本文中的基因panel又可称为基因集(geneset),是所有与激素性股骨头坏死(ga-onfh)易感性相关的基因集合或基因组合。
基因(检测)panel是高通量基因检测和基因测序发展起来后用的一个词语,它是指在检测中将若干基因对应的探针设计到同一张捕获芯片上以捕获目标dna并用于后续的基因测序。在检测中不只是检测一个位点、一个基因,而是同时检测多个位点、多个基因、多个位点,这些位点和基因需要按照一个标准进行选择和组合,需要一个检测panel。
本发明应用基因panel进行ga-onfh易感性相关基因检测的手段主要是多重pcr靶向测序装置和配套的试剂盒。
在优选的实施方式中,该试剂盒除了各种引物外,还可分别包括下述物品中的至少之一:携带工具,其空间划分为可以收容一种或多种容器、96孔板或板条的限定空间,该容器例如是试剂盒、药瓶、试管、和类似物,每样容器都含有一个单独的用于本发明方法的组分;说明书,其可以写在瓶子、试管和类似物上,或者写在一张单独的纸上,或者在容器的外部或内部,例如是带有操作演示视频app下载窗口比如二维码的纸件,说明书也可以是多媒体的形式,比如cd、u盘、网盘等。
筛选构建ga-onfh预测基因panel和多重pcr引物设计的方法包括如下步骤:
1、从已公开文献资源中选择已公开骨坏死及糖皮质激素代谢相关基因,所述已公开骨坏死及糖皮质激素代谢相关基因包括表1中所列人类基因位点。
2、将上述骨坏死及糖皮质激素代谢相关基因合并,去冗余,并通过ncbiofficename、hgncapprovedofficialsymbol系统确定标准基因名,获得ga-onfh预测靶向测序基因集,如下述表1所示。
表1、多态性位点基因集列表
3、多重pcr引物设计:根据步骤2中获得的ga-onfh预测靶向测序基因集,从人类基因组中寻找可以设计多重pcr引物的设计区域,设计并生成多重pcr引物;所述引物经过合理的设计,能覆盖基因集多态性位点,用于单管多重pcr扩增时特异性良好,引物彼此之间不引发引物二聚体,且均一性较好。结果如表2所示。
表2、用于检测基因panel中人类基因组位点的引物序列
4、靶向测序结果的生物信息学分析:将下机得到的.bcl文件,使用数据拆分软件bcl2fastq(v2.20.0.422),依据各个样本的sampleindex拆分成各样本的原始的.fastq文件,即rawdata;使用trimmomatic(v0.36)去除rawdata中的接头序列、低质量碱基等,生成cleandata.;使用bwa(v0.7.17-r1188),将cleandata比对到hg19参考基因组,产生.sam文件;使用samtools(v1.7),将.sam文件进行排序并转换成.sorted.bam文件;使用varscan(v2.4.3),以.sorted.bam为输入,进行snp和indel变异检测,并产生.vcf文件;从.vcf文件中提取相关信息,最终生成各位点的基因型信息。
5、建立基于多态性位点的ga-onfh风险预测模型:
1)以or值作为权重的遗传风险评分(anoddsratioweightedgeneticriskscore,or_grs),计算所有纳入模型snp的or值权重,该评分方法的计算公式如下:
ωi=ln(ori),
a、or_grs单因素逻辑回归分析模型训练,公式为:
logitp(d=1|g)=α+β(or_grs),参数值α=0.64[-0.22,1.49],β=0.35[0.20,0.51]。该模型内部验证训练集得到的auc为0.93(p-value=4.32e-11),测试集预测得到的auc值为0.95(p-value=1.15e-11)。
b、同时考虑or_grs、性别(sex)、年龄(age)、原发病(primarydisease)进行多因素逻辑回归分析模型训练,公式如下:
logitp(d=1|g)
=α+β1(or_grs)+β2(sex_0)+β3(sex_1)+β4(age_0)+β5(age_1)
+β6(primarydisease_1)+β7(primarydisease_2),
参数值分别为:α=0.11[-0.63,0.86],β1=0.54[0.22,0.85],β2=0.98[-0.272.23],β3=-0.86[-2.40,0.67],β4=1.15[-0.07,2.37],β5=-1.04[-2.37,0.30],β6=-1.61[-4.25,1.03],β7=-2.63[-0.04,5.30]。该模型内部验证训练集得到的auc为0.97(p-value=1.72e-12),测试集预测得到的auc值为0.97(p-value=3.19e-13)。
2)可释方差遗传风险评分(plainedvarianceweightedgeneticriskscore,ev_grs),同时考虑snp效应和风险等位基因频率(即最小等位基因频率maf),该评分方法的计算公式如下:
a、ev_grs单因素逻辑回归分析模型训练,回归公式为:
logitp(d=1|g)=α+β(ev_grs),
参数值为:α=0.90[-0.02,1.82],β=0.57[0.31,0.82]。该模型进行内部验证所得到的auc为0.93(p-value=4.92e-11),测试集预测得到的auc值为0.95(p-value=1.60e-11)。
b、同时考虑grs、性别(sex)、年龄(age)、原发病(primarydisease)四个因素进行多因素逻辑回归分析模型训练,公式为:
logitp(d=1|g)=
α+β1(ev_grs)+β2(sex_0)+β3(sex_1)+β4(age_0)+β5(age_1)+
β6(primarydisease_1)+β7(primarydisease_2),
参数值分别为:α=0.35[-0.40,1.10],β1=0.81[0.36,1.26],β2=1.02[-0.22,2.26],β3=-0.67[-2.12,0.79],β4=1.19[-0.03,2.41],β5=-0.84[-2.09,0.41],β6=-1.85[-4.55,0.85],β7=2.44[-0.07,4.94]。该模型进行内部验证所得auc为0.96(p-value=1.84e-12),测试集预测得到的auc值为0.97(p-value=3.19e-13)。
上述的auc值(曲线下面积)高达0.93-0.97,证明本发明筛选构建的基因panel用于预测ga-onfh具有高度的可靠性和准确性。
本领域技术人员容易理解,上述ga-onfh风险预测模型很容易通过编程、或者通过数学软件包形式被输入到计算机、基因检测装置比如illumina测序仪、云服务器的信息处理模块。根据受试者的基因检测结果,能够方便地预测出受试者的激素性股骨头坏死易感性,为医生给药方案选择提供参考。
如下以ga-onfh易感性基因panel的筛选和验证实施例说明本发明的技术效果。实施例中涉及到的百分含量,除特别说明外(比如明示为体积百分比率或比例),皆指质量百分含量。
实施例1多态性位点检测
仪器:pcr仪、qubit荧光定量仪、安捷伦4200片段分析仪、illumina高通量测序仪。
按下述步骤检测多态性位点:
1、样本dna提取:全血样本,采用qiaampdnabloodminikit核酸提取试剂盒,参考试剂盒说明书进行操作,提取样本中的dna,并用quibt定量,-20℃冻存,使用前室温解冻。
2、dna文库构建:
1)第一轮多重pcr扩增
a)按照表制备多重pcr反应液,dna的起始量为40ng;混匀并短暂离心,将反应混合液收集至pcr管底部。
b)将pcr管放入带有加热盖的热循环器中,并使用下列程序执行多重pcr扩增反应:
2)第一轮磁珠纯化
a)向30μlpcr产物加入27μl室温平衡后的ampurexp磁珠,用移液器轻缓吸打混匀20次;
b)室温孵育5min后,将pcr管置于磁力架上静置5min;
c)彻底移除上清,将pcr管从磁力架取下,向管内加入50μlyfbufferb用移液器轻缓吸打混匀20次;
d)室温孵育5min后,将pcr管置于磁力架上静置5min;
e)移除上清,pcr管继续放置在磁力架上,向管内加入200μl80%乙醇溶液,静置30s;
f)移除上清,pcr管继续放置在磁力架上,向管内加入200μl80%乙醇溶液,静置30s后彻底移除上清(使用10μl移液器移除底部残留乙醇溶液);
g)室温静置3-5min,使残留乙醇彻底挥发;
h)将pcr管从磁力架取下,加入24μlnuclease-freewater,用移液器轻缓吸打混匀20次,重悬磁珠,避免产生气泡,室温静置3min;
i)将pcr管重新置于磁力架上,静置3min;
j)用移液器吸取20μl上清液,转移到新的200μlpcr管内,管内上清液为多重pcr产物。
3)第二轮接头序列pcr反应
a)按照表制备pcr反应液,混匀并短暂离心,将反应混合液收集至pcr管底部。
b)将pcr管放入带有加热盖的热循环器中,并使用下列程序执行多重pcr扩增反应
4)第二轮磁珠纯化
a)向30μlpcr产物加入27μl室温平衡后的ampurexp磁珠,用移液器轻缓吸打混匀20次;
b)室温孵育5min后,将pcr管置于磁力架上静置5min;
c)彻底移除上清,将pcr管从磁力架取下,向管内加入50μlyfbufferb用移液器轻缓吸打混匀20次;
d)室温孵育5min后,将pcr管置于磁力架上静置5min;
e)移除上清,pcr管继续放置在磁力架上,向管内加入200μl80%乙醇溶液,静置30s;
f)移除上清,pcr管继续放置在磁力架上,向管内加入200μl80%乙醇溶液,静置30s后彻底移除上清(使用10μl移液器移除底部残留乙醇溶液);
g)室温静置3-5min,使残留乙醇彻底挥发;
h)将pcr管从磁力架取下,加入24μlnuclease-freewater,用移液器轻缓吸打混匀20次,重悬磁珠,避免产生气泡,室温静置3min;
i)将pcr管重新置于磁力架上,静置3min;
j)用移液器吸取20μl上清液,转移到新的200μlpcr管内,管内上清液为多重pcr测序文库。
5)文库定量及片段分析
a)取1μl文库使用
b)取1μl文库样本使用agilent4200bioanalyzersystem(highsensitivitydnakit)进行文库片段长度和纯度测量,正常文库的靶片段分布区间在300bp–450bp之间,主峰在409bp左右。
3、上机测序:将建好的文库,准确定量并稀释到合适浓度,参考illumina相应操作说明书,将每个样本的dna扩增文库按照500m数据量进行上机测序。
4、生物信息学分析
1)数据拆分:将下机得到的.bcl文件,使用数据拆分软件bcl2fastq(v2.20.0.422),依据各个样本的sampleindex拆分成各样本的原始的.fastq文件,即rawdata。
2)数据清洗:使用trimmomatic(v0.36)去除rawdata中的接头序列、低质量碱基等,生成cleandata。
3)序列比对:使用bwa(v0.7.17-r1188),将cleandata比对到hg19参考基因组,产生.sam文件。
4)比对结果排序:使用samtools(v1.7),将.sam文件进行排序并转换成.sorted.bam文件。
5)多态性位点检测:使用varscan(v2.4.3),以.sorted.bam为输入,进行snp和indel变异检测,并产生.vcf文件。从.vcf文件中提取相关位点信息,最终生成各位点的基因型信息。
实施例2遗传风险评分的计算及模型训练数据准备
1、按照实施例1的方法,对162例回顾性样本进行panel检测,获得样本的多态性位点基因型信息。
2、or(oddratio)值计算:一个snp位点有两个等位基因,记为allele1、allele2。假设allele1为风险等位基因,那么按照下面的公式计算风险等位基因的or值:
等位基因1(allele1)的or值:oddsratio=(a/b)/(c/d)=ad/bc
3、风险等位基因人群频率收集:考虑到各数据库中的中国人群量较小,从ensembl数据库(http://grch37.ensembl.org/index.html)上收集风险等位基因在亚种人群中的人群频率。以千人基因组计划为主,在千人基因组数据库中搜索不到的位点,从gnomadad数据库中收集。
4、snp位点间的连锁不平衡分析:使用r包ldlinkr对所有位点在亚洲人群中进行连锁不平衡分析,互相存在连锁不平衡的位点只保留其中的一个。
5、计算每个样本的遗传风险分值grs(geneticriskscore):我们以g表示所有snp位点风险等位基因的集合向量(gi表示第i个snp位点的风险等位基因的数量)。对于野生基因型,因不含有风险等位基因,故gi=0;对于杂合突变基因型,含有1个风险等位基因,故gi=1;对于纯合突变基因型,含有2个风险等位基因,故gi=2。我们采用了两种风险评分方法计算grs,以评估不同评分方法对坏死风险预测模型的影响。
1)以or值作为权重的遗传风险评分(anoddsratioweightedgeneticriskscore,or_grs):考虑snp对坏死的不同效应,以snp效应作为权重,计算所有纳入模型snp的or值权重,该评分方法的计算公式如下:
ωi=ln(ori)
2)可释方差遗传风险评分(plainedvarianceweightedgeneticriskscore,ev_grs),同时考虑snp效应和风险等位基因频率(即最小等位基因频率maf),该评分方法的计算公式如下:
6、分层抽样:考虑到随机抽样可能会造成测试集和预测集样本种类上的偏倚,进而影响最终的训练及预测效果,因此采用分层抽样。
1)样本标记:对样本按照疾病风险、性别、年龄区间、是否为坏死组进行4个字符的标记。其中疾病风险3种,分别为1、2、3;性别2种,女性标记为0,男性标记为1;年龄区间2种,<40岁标记为0,>=40岁标记为1;是否坏死涉及2种情形,对照标记为0,坏死标记为1。162个样本最终产生22种标记,其中12种为对照组的,另外10种为坏死组的。
2)样本分层:按照22种标记对162个患者的样本进行分层,主要考虑疾病、兼顾性别和年龄因素,并尽可能保证测试集和预测集的三种因素涉及患者的比例大体一致。
3)抽取训练集:按照各层在训练集和预测集比例,在各层内部随机抽取训练集,其余样本做测试集。
7、产生单因素模型和多因素模型测试、训练数据矩阵:对于单因素模型,仅考虑grs一个因素,分别在or_grs风险评分和ev_grs风险评分下的构建训练集、测试集矩阵。对于多因素模型,则同时考虑grs因素、性别因素、年龄因素、原发病因素,在or_grs风险评分和ev_grs风险评分下的构建训练集、测试集矩阵时加入年龄因素、性别因素、原发病因素的数据。此时,由于性别、年龄、原发病属于分类变量,因此这些变量需要先转化成哑变量。性别涉及2个哑变量,即sex_0、sex_1;年龄涉及2个哑变量,即age_0、age_1;原发病涉及3个哑变量,分别代表已知低坏死风险原发病(primarydisease_1)、未知坏死风险原发病(primarydisease_2)和高坏死风险原发病(primarydisease_3),原发病的风险划分以临床经验为依据,低风险疾病主要包括原发性血管炎、腹膜后纤维化、白塞氏病、干燥综合征和急性肾衰竭;高风险疾病主要包括系统性红斑狼疮和成人斯蒂尔病,其他疾病列入未知坏死风险原发病。为避免过拟合,只使用其中2个哑变量进行分析,即primarydisease_1、primarydisease_2。
实施例3基于or_grs风险评分的单因素逻辑回归分析模型的训练及内部验证
1、基于or_grs风险评分的单因素逻辑回归分析模型训练:仅考虑grs一个因素,在or_grs风险评分下对所有训练集样本进行单因素逻辑回归分析模型训练。回归公式如下:
logitp(d=1|g)=α+β(or_grs)。
2、or_grs风险评分训练得到的单因素回归模型的参数值分别为α=0.64[-0.22,1.49],β=0.35[0.20,0.51]。
3、采用训练集对该模型进行内部验证,auc为0.93(p-value=4.32e-11),结果如图1所示。
4、采用分层5折交叉验证该模型,auc为0.94±0.03,结果如图2所示。
5、采用自助法对基于or_grs风险评分的单因素逻辑回归分析模型进行内部验证:对训练集进行1000次的有放回的随机抽样,产生1000个测试集,auc为0.93±0.03。
实施例4基于ev_grs风险评分的单因素逻辑回归分析模型的训练及内部验证
1、基于ev_grs风险评分的单因素逻辑回归分析模型训练:仅考虑grs一个因素,在ev_grs风险评分下对所有训练集样本进行单因素逻辑回归分析模型训练。回归公式如下:
logitp(d=1|g)=α+β(ev_grs)。
2、ev_grs风险评分训练得到的单因素回归模型的参数值分别为α=0.90[-0.02,1.82],β=0.57[0.31,0.82]。
3、采用训练集对该模型进行内部验证,auc为0.93(p-value=4.92e-11),结果如图3所示。
4、采用分层5折交叉验证该模型,auc为0.95±0.05,结果如图4所示。
5、采用自助法对基于ev_grs风险评分的单因素逻辑回归分析模型进行内部验证:对训练集进行1000次的有放回的随机抽样,产生1000个测试集,auc为0.93±0.03。
实施例5基于or_grs风险评分的多因素逻辑回归分析模型的训练及内部验证
1、多因素逻辑回归分析模型训练及模型预测:同时考虑grs、性别、年龄、原发病四个因素,在or_grs风险评分下对所有训练集样本进行多因素逻辑回归分析模型训练。回归公式如下:
logitp(d=1|g)
=α+β1(or_grs)+β2(sex_0)+β3(sex_1)+β4(age_0)+β5(age_1)
+β6(primarydisease_1)+β7(primarydisease_2)
2、or_grs风险评分训练得到的多因素回归模型的参数值分别为α=0.11[-0.63,0.86],β1=0.54[0.22,0.85],β2=0.98[-0.272.23],β3=-0.86[-2.40,0.67],β4=1.15[-0.07,2.37],β5=-1.04[-2.37,0.30],β6=-1.61[-4.25,1.03],β7=-2.63[-0.04,5.30]。
3、采用训练集对该模型进行内部验证,auc为0.97(p-value=1.72e-12),结果如图5所示。
4、采用分层5折交叉验证该模型,auc为0.96±0.02,结果如图6所示。
5、采用自助法对基于or_grs风险评分的多因素逻辑回归分析模型进行内部验证:对训练集进行1000次的有放回的随机抽样,产生1000个测试集,auc为0.96±0.02。
实施例6基于ev_grs风险评分的多因素逻辑回归分析模型的训练及内部验证
1、多因素逻辑回归分析模型训练及模型预测:同时考虑grs、性别、年龄、原发病四个因素,在ev_grs风险评分下对所有训练集样本进行多因素逻辑回归分析模型训练。回归公式如下:
logitp(d=1|g)
=α+β1(ev_grs)+β2(sex_0)+β3(sex_1)+β4(age_0)+β5(age_1)
+β6(primarydisease_1)+β7(primarydisease_2)
2、ev_grs风险评分训练得到的多因素回归模型的参数值分别为α=0.35[-0.40,1.10],β1=0.81[0.36,1.26],β2=1.02[-0.22,2.26],β3=-0.67[-2.12,0.79],β4=1.19[-0.03,2.41],β5=-0.84[-2.09,0.41],β6=-1.85[-4.55,0.85],β7=2.44[-0.07,4.94]。
3、采用训练集对该模型进行内部验证,auc为0.96(p-value=1.84e-12),结果如图7所示。
4、采用分层5折交叉验证该模型,auc为0.97±0.03,结果如图8所示。
5、采用自助法对基于ev_grs风险评分的多因素逻辑回归分析模型进行内部验证:对测试集进行1000次的有放回的随机抽样,产生1000个测试集,auc为0.96±0.02。
实施例7基于or_grs风险评分的单因素逻辑回归分析模型的预测
使用测试集对基于or_grs风险评分的单因素逻辑回归分析模型进行测试,该模型对测试集预测得到的auc值为0.95(p-value=1.15e-11),结果如图9所示。
实施例8基于ev_grs风险评分的单因素逻辑回归分析模型的预测
使用测试集对基于ev_grs风险评分的单因素逻辑回归分析模型进行测试,该模型对测试集预测得到的auc值为0.95(p-value=1.60e-11),结果如图10所示。
实施例9基于or_grs风险评分的多因素逻辑回归分析模型的预测
使用测试集对基于or_grs风险评分的多因素逻辑回归分析模型进行测试,该模型对测试集预测得到的auc值为0.97(p-value=3.19e-13),结果如图11所示。
实施例10基于ev_grs风险评分的多因素逻辑回归分析模型的预测
使用测试集对基于ev_grs风险评分的多因素逻辑回归分析模型进行测试,该模型对测试集预测得到的auc值为0.97(p-value=3.19e-13),结果如图12所示。
上述各实施例的auc值为0.93-0.97,表明了本发明的基因panel在预测ga-onfh易感性中具有的高度可靠性和准确率。
以上所述仅为本发明的较佳实施例,并非对本发明任何形式上和实质上的限制,应当指出,对于本技术领域的普通技术人员,在不脱离本发明方法的前提下,还将可以做出若干改进和补充,这些改进和补充也应该视为本发明的保护范围。凡熟悉本专业的技术人员,在不脱离本发明的精神和范围的情况下,当可利用以上所揭示的技术内容而做出的些许更动、修改与演变的等同变化,均为本发明的等效实施例;同时,凡依据本发明的实质技术对上述实施例所作的任何等同变化的更动、修饰与演变,均仍属于本发明的技术方案的范围内。
序列表
<110>复旦大学附属中山医院
骏实生物科技(上海)有限公司
<120>一种激素性股骨头坏死易感基因panel
<130>shpi2010270
<160>38
<170>siposequencelisting1.0
<210>1
<211>27
<212>dna
<213>人工序列()
<400>1
gaattattgctctgtttctgaacgatt27
<210>2
<211>19
<212>dna
<213>人工序列()
<400>2
gggcttgtcagaggcagca19
<210>3
<211>28
<212>dna
<213>人工序列()
<400>3
tttttgaaccactgctaacaatcactaa28
<210>4
<211>17
<212>dna
<213>人工序列()
<400>4
gccttgtccatggcgct17
<210>5
<211>28
<212>dna
<213>人工序列()
<400>5
tttttgaaccactgctaacaatcactaa28
<210>6
<211>17
<212>dna
<213>人工序列()
<400>6
gccttgtccatggcgct17
<210>7
<211>18
<212>dna
<213>人工序列()
<400>7
cttggagggcagcggaag18
<210>8
<211>18
<212>dna
<213>人工序列()
<400>8
catctgtgtgcgggaccc18
<210>9
<211>18
<212>dna
<213>人工序列()
<400>9
ggtcccgcacacagatgg18
<210>10
<211>25
<212>dna
<213>人工序列()
<400>10
gtcaaactccggatcctctaattca25
<210>11
<211>21
<212>dna
<213>人工序列()
<400>11
cggatgtccctgactctctct21
<210>12
<211>16
<212>dna
<213>人工序列()
<400>12
cgggtcctcggctcca16
<210>13
<211>16
<212>dna
<213>人工序列()
<400>13
ggcagggcaagggctg16
<210>14
<211>16
<212>dna
<213>人工序列()
<400>14
ccaggtcctcgccgga16
<210>15
<211>21
<212>dna
<213>人工序列()
<400>15
agactgcaccaaacctcacag21
<210>16
<211>21
<212>dna
<213>人工序列()
<400>16
tgggaatatggcaaggcagtc21
<210>17
<211>22
<212>dna
<213>人工序列()
<400>17
ccgtattgtgccagctagaagt22
<210>18
<211>17
<212>dna
<213>人工序列()
<400>18
cccgcagtggcaccatt17
<210>19
<211>18
<212>dna
<213>人工序列()
<400>19
ccccacgttcgttccctg18
<210>20
<211>20
<212>dna
<213>人工序列()
<400>20
aggaagtagccccctagctg20
<210>21
<211>19
<212>dna
<213>人工序列()
<400>21
gttcctggccatcgacctg19
<210>22
<211>17
<212>dna
<213>人工序列()
<400>22
ggacgctgactggcacc17
<210>23
<211>18
<212>dna
<213>人工序列()
<400>23
gagccaaagcccccagac18
<210>24
<211>21
<212>dna
<213>人工序列()
<400>24
catgggagactgaatggtggg21
<210>25
<211>18
<212>dna
<213>人工序列()
<400>25
ccccaagccctagcaagc18
<210>26
<211>30
<212>dna
<213>人工序列()
<400>26
tctagaagaaaagcggcatgataaaataaa30
<210>27
<211>20
<212>dna
<213>人工序列()
<400>27
gctggggtaggagctagaca20
<210>28
<211>23
<212>dna
<213>人工序列()
<400>28
tgtggagaaaaatgaagctggga23
<210>29
<211>18
<212>dna
<213>人工序列()
<400>29
cctagggagggacggtgg18
<210>30
<211>30
<212>dna
<213>人工序列()
<400>30
aaatacttacaataaggccattctctgttc30
<210>31
<211>33
<212>dna
<213>人工序列()
<400>31
tgtgctgtgtaagttacattcattaactaatat33
<210>32
<211>33
<212>dna
<213>人工序列()
<400>32
tataggaaaatgaaagttaggattttgagactc33
<210>33
<211>30
<212>dna
<213>人工序列()
<400>33
gttaagattagaagcctccattgaaagaat30
<210>34
<211>23
<212>dna
<213>人工序列()
<400>34
cttagacagcaaggggtcatacc23
<210>35
<211>33
<212>dna
<213>人工序列()
<400>35
tttttgagctgtttctaaaatatctttccaaaa33
<210>36
<211>21
<212>dna
<213>人工序列()
<400>36
cctatgtaacaaggcaccgct21
<210>37
<211>21
<212>dna
<213>人工序列()
<400>37
gccataacactgcccatctga21
<210>38
<211>35
<212>dna
<213>人工序列()
<400>38
gctgttcaaaatttcttaatataacagtcactaat35
- 还没有人留言评论。精彩留言会获得点赞!