一种PCR随机引物和使用其构建靶向测序文库的方法与流程
本发明属于生物技术领域,具体涉及一种pcr随机引物和使用其构建靶向测序文库的方法。
背景技术:
下一代测序(nextgenerationsequencing,ngs)也称为深度测序或大规模平行测序,可同时对数百万小片段进行测序。ngs已广泛应用于众多领域,其中最常用的是基因组dna变异分析和rna表达分析。这些分析应用可扩展到整个基因组和整个外显子组,还可专门测序特定区域和基因组合。
ngs测序前,有几种方法可以富集目标区域,最常用的两种方法是:1)基于探针捕获,通过设计与靶dna序列互补的核酸探针来捕获靶dna;这类方法因为需要合成探针,所以成本高,实验步骤冗长。2)基于多重pcr,通过设计靶dna特异性的dna引物进行pcr扩增来富集靶dna;相比于基于探针捕获的方法,这类方法成本较低,步骤简短。
ngs和靶向序列捕获技术的进步使其可用于测序异质混合物中的低频突变。然而,pcr和测序方法的系统误差使ngs的进一步发展受到了限制。文库制备、靶向序列捕获和测序均采用dna聚合酶以及扩增步骤,这些过程会引入偏差,包括重复、相应的不均匀扩增以及因聚合酶误差导致的假象,这种误差会引入原始样品中不存在的序列变化。为了对扩增和测序过程中出现的随机错误进行纠错,使用上述方法建库的过程中可以对每个模板分子加上独特的分子标记(umi)。测序后具有同样分子标记且对比到基因组同一位置的读序(reads)可归为一组,并视为来源于同一个模板分子。由于扩增和测序错误是随机发生的,同组中如果有少于一半的读序中存在序列与其它读序不同的序列,该序列可视为扩增或测序错误,忽略不计,从而达到纠错的效果。
基于多重pcr的二代测序靶向技术的建库方法可以分成2种:1)使用成对的特异性引物进行常规pcr扩增对靶dna区域进行捕获和富集;其中umi可以包含在引物中,在前2个pcr循环中被整合至扩增子。2)首先对dna片段用链接酶加上通用接头,其中umi可包含在接头内,然后使用特异性引物和通用引物进行靶dna富集。在上述基于多重pcr的建库方法中。方法1)需要至少一对引物来富集一个靶区域。引物设计是有条件限制的,比如说在很难在序列复杂度低的区域进行引物设计。这样很多时候成对引物跨度的区域会比较大。如果起始dna片段小于引物跨越的长度,靶区域将得不到扩增。这在分子诊断中可能会造成灵敏度降低和假阴性。方法2)使用单端特异性引物,避免了使用双端引物时,短模板不能被富集的情形。但是要使用链接酶加上包含通用序列的接头。连接酶链接的效率一般在30%左右,意味着只有30%的起始dna能被加上接头,转化为测序文库,损失剩下的70%。同样地,这在分子诊断中也会造成灵敏度降低和假阴性。
技术实现要素:
本发明旨在至少解决现有技术中存在的技术问题之一,为此,本发明提供了一种pcr随机引物和使用其构建靶向测序文库的方法。
本发明所采用的技术方案如下文所述。
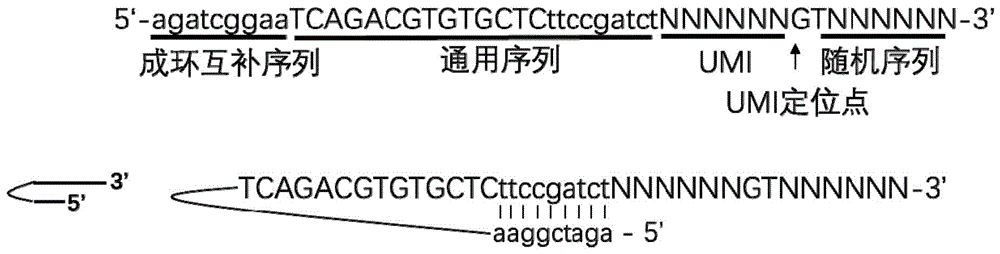
本发明的第一方面涉及一种pcr随机引物,所述pcr随机引物从5’端到3’端依次包括成环互补序列、通用序列、umi序列、umi定位点和随机序列,其中,所述成环互补序列与所述通用序列中的部分序列反向互补。
根据本发明的一些实施方式,所述pcr随机引物从5’端到3’端依次由成环互补序列、通用序列、umi序列、umi定位点和随机序列组成。
根据本发明的一些实施方式,所述成环互补序列与所述通用序列中连接umi序列端的部分序列反向互补。
根据本发明的一些实施方式,所述成环互补序列的长度为4~20bp。例如,可以是4bp、5bp、6bp、7bp、8bp、9bp或更长。
根据本发明的一些实施方式,所述成环互补序列选自seqidno:1~5。
根据本发明的一些实施方式,所述通用序列的长度为18~30bp。例如,可以是20bp、23bp、25bp、28bp或30bp。
根据本发明的一些实施方式,所述通用序列选自seqidno:6~9。
根据本发明的一些实施方式,所述umi序列的长度为4~20bp。例如,可以是4bp、5bp、8bp、11bp或更长。
根据本发明的一些实施方式,所述umi定位点的长度为1~6bp。例如,可以是2bp、3bp、4bp或5bp。
根据本发明的一些实施方式,所述umi定位点选自seqidno:10~13。
根据本发明的一些实施方式,所述随机序列的长度为5~15bp。例如,可以是5bp、6bp、7bp、8bp、9bp、10bp或12bp。
根据本发明的一些实施方式,所述pcr随机引物选自seqidno:14~28。
本发明第二方面提供了一种试剂盒,包括本发明第一方面任一项所述的pcr随机引物。
本发明第三方面提供了一种pcr扩增方法,包括用本发明第一方面任一项所述的pcr随机引物或本发明第二方面任一项所述的试剂盒进行pcr扩增。
本发明第四方面提供了一种构建靶向测序文库的方法,包括多轮链置换随机扩增和靶向富集。
根据本发明的一些实施方式,所述构建靶向测序文库的方法,包括如下步骤:
1)将片段化的待测基因组dna与如上所述的pcr随机引物混合,变性后,迅速冷却处理;
2)将核苷酸单体混合物、核酸聚合酶、缓冲液和步骤1)的产物混合,进行第一轮pcr扩增;
3)将第一轮pcr扩增的产物作为模板,利用特异性引物在第二温度循环程序中进行第二轮的pcr扩增。
根据本发明的一些实施方式,步骤1)中,片段化的待测基因组dna与如上所述的pcr随机引物混合后在高温下变性,然后迅速冰浴冷却;
根据本发明的一些实施方式,步骤2)中,核酸聚合酶为具有链置换活性的dna聚合酶,例如可以选自bst3dna聚合酶,omni-ampdna聚合酶和sddna聚合酶。
根据本发明的一些实施方式,步骤2)中,将核苷酸单体混合物、核酸聚合酶、缓冲液和步骤1)的产物混合,并在第一温度循环程序中进行第一轮pcr扩增;
根据本发明的一些实施方式,步骤2)中,第一温度循环程序可以为:
1个循环:25℃5min,30℃5min,55℃15min;
2-4个循环:75℃1min,25℃5min,30℃5min,55℃15min;
1个循环:55℃10min。
根据本发明的一些实施方式,步骤3)中,将第一轮pcr扩增的产物作为模板,利用特异性引物、5’接头引物和3’接头引物在第二温度循环程序中进行第二轮的pcr扩增。
根据本发明的一些实施方式,所述特异性引物的3’端为靶区域特异性序列,5’端与所述5’接头引物的3’端相同。
根据本发明的一些实施方式,步骤3)中,将第一轮pcr扩增的产物作为模板,利用特异性引物在第二温度循环程序中进行第二轮的pcr扩增。
根据本发明的一些实施方式,步骤3)中,将第一轮pcr扩增的产物作为模板,加入无核酸酶去离子水、pcr预混液、特异性引物、5’接头引物和3’接头引物在第二温度循环程序中进行第二轮的pcr扩增。
根据本发明的一些实施方式,步骤3)中,第二温度循环程序可以为:
1个循环:98℃,30s;
25个循环:98℃,5s;65℃,5min;72℃,30s;
1个循环:72℃2min。
根据本发明的一些实施方式,步骤3)之后,还包括将第二轮pcr扩增的产物纯化的步骤。
根据本发明的一些实施方式,采用磁珠进行纯化,也可以采用柱纯化,或进行凝胶电泳后进行dna提取。
本发明的有益效果:
本发明设计了一种pcr随机引物和使用其构建靶向测序文库的方法,构建的文库具有在靶率高、均一性好和、平均转化效率高等优势。
多轮随机链置换反应中的pcr随机引物中引入了umi,用以区分使用同一模板时在不同位点扩增的扩增子。5’端引入成环互补序列,从而避免引物中随机序列的核苷酸与通用序列形成二聚体,充分暴露随机序列来提高与模板退火的效率。
整个多轮随机链置换反应中的温控循环是独特的。不同于传统的mda,可以很大程度减少反应时间;也不同于malbac,每轮都需要添加dna聚合酶来保证反应的进行。
附图说明
图1为本发明pcr随机引物设计示意图;
图2为多轮链置换随机扩增过程示意图;
图3为靶向富集过程示意图;
图4为本发明实施例构建的测序文库的性能参数。
具体实施方式
本发明的第一方面涉及一种pcr随机引物,所述pcr随机引物从5’端到3’端依次包括成环互补序列、通用序列、umi序列、umi定位点和随机序列,其中,所述成环互补序列与所述通用序列中的部分序列反向互补。
如图1所示,为本发明以illuminatrueseq接头序列为例设计的pcr随机引物;该pcr随机引物,5’端到3’端依次由成环互补序列、通用序列、umi序列、umi定位点和随机序列组成,并且所述成环互补序列与所述通用序列的3’端反向互补。
本发明人通过研究发现,设计成环互补序列与通用序列中的部分序列反向互补成环,可以防止3’端随机序列与测序接头自我形成二聚体,充分暴露随机序列,提高了与核酸模板随机配对的效率,提高了与模板退火的效率;通用序列用来作为下一步扩增的引物配对,可以是不同测序平台的测序接头或其它固定序列;umi序列用来区分线性扩增中的不同的扩增子;umi定位点可以用来区分随机序列和umi序列。现有技术中,大都采用连接酶连接测序接头和umi,而本发明则是利用pcr随机引物进行链置换随机扩增的方式引入测序接头和umi,提高了起始核酸分子转化为文库的效率。
本发明中,所述pcr随机引物中,成环互补序列、通用序列、umi序列、umi定位点和随机序列的长度和具体序列可以根据实际情况进行调整。
根据本发明的一些实施方式,所述成环互补序列的长度为4~20bp,或更长。例如,可以是4bp、5bp、6bp、7bp、8bp、9bp或更长。
根据本发明的一些实施方式,所述成环互补序列选自seqidno:1~5。
根据本发明的一些实施方式,所述通用序列的长度为18~30bp。例如,可以是20bp、23bp、25bp、28bp或30bp。
通用序列可以是不同测序平台的测序接头如illumina平台、bgi平台、iontorrent平台、oxfordnanopore平台或pacbio平台的测序接头序列,还可以是其它固定序列。
根据本发明的一些实施方式,所述通用序列选自seqidno:6~9。
根据本发明的一些实施方式,所述umi序列的长度为4~20bp。例如,可以是4bp、5bp、8bp、11bp或更长。
根据本发明的一些实施方式,所述umi定位点的长度为1~6bp。例如,可以是2bp、3bp、4bp或5bp。
根据本发明的一些实施方式,所述umi定位点选自seqidno:10~13。
根据本发明的一些实施方式,所述随机序列的长度为5~15bp。例如,可以是5bp、6bp、7bp、8bp、9bp、10bp或12bp。
根据本发明的一些实施方式,pcr随机引物选自seqidno:14~28。
本发明第二方面提供了一种试剂盒,包括本发明第一方面任一项所述的pcr随机引物。相对于常规引物,本发明试剂盒采用pcr随机引物进行多轮链置换随机扩增,提高起始核酸分子转化为文库的效率。
本发明第三方面提供了一种pcr扩增方法,包括用本发明第一方面任一项所述的pcr随机引物或本发明第二方面任一项所述的试剂盒进行pcr扩增。利用该方法可以有效实现模板的pcr扩增,并且该方法能够增加pcr扩增的特异性,有效降低非特异性产物的产生,并提高扩增效率。
本发明第四方面提供了一种构建靶向测序文库的方法,包括如下步骤:
1)片段化的待测基因组dna与如上所述的pcr随机引物混合后变性,然后迅速冷却处理;
2)将核苷酸单体混合物、核酸聚合酶、限制性内切酶缓冲液和步骤1)的产物混合,并在第一温度循环程序中进行第一轮pcr扩增;
3)将第一轮pcr扩增的产物作为模板,利用特异性引物在第二温度循环程序中进行第二轮的pcr扩增。
本发明的建库过程主要分为多轮链置换随机扩增(步骤1)和步骤2))和靶向富集(步骤3))过程。首先,本发明使用单端特异性引物,避免了使用双端特异性引物的不足。其次,现有技术中大都是对dna片段用连接酶加上通用序列(umi紧邻通用序列),而本发明不使用连接酶加通用序列和umi,而是设计独特的pcr随机引物,通过链置换随机扩增的方式加上通用序列和umi,在此过程中,同一分子会在不同位置被随机扩增多次并标上umi,这种方式不同于只在引物特异性位点扩增的传统pcr,可以鉴别不同pcr随机引物扩增的扩增子,从而降低了扩增中引起的序列错配,便于后续反应中使用通用序列进行的pcr和测序接头的整合;提高了起始核酸分子转化为文库的效率。再者,靶向富集反应即第二轮pcr扩增反应中,使用多轮链置换随机扩增的产物作为模板,靶区域特异性的dna引物进行pcr扩增,对特异性产物进行富集,同时测序接头也在此过程中加到产物两端,形成测序文库。
根据本发明的一些实施方式,步骤1)中,片段化的待测基因组dna与如上所述的pcr随机引物混合后在高温下变性,然后迅速冰浴冷却;
本发明中将片段化的待测基因组dna与如上所述的pcr随机引物混合后在高温下变性,然后迅速冰浴冷却;其中冷却处理是为了防止变性后的单链dna重新变成双链dna。
根据本发明的一些实施方式,步骤2)中,核酸聚合酶为具有链置换活性的dna聚合酶,例如可以选自bst3dna聚合酶,omni-ampdna聚合酶和sddna聚合酶。
本发明在第二个循环以及后面的多轮链置换循环中,不需要加入额外的dna聚合酶,使得整个反应中大大减少了人工操作的时间。
根据本发明的一些实施方式,步骤2)中,第一温度循环程序可以为:
1个循环:25℃5min,30℃5min,55℃15min;
2-4个循环:75℃1min,25℃5min,30℃5min,55℃15min;
1个循环:55℃10min。
本发明的步骤1)和步骤2)属于多轮随机链置换扩增过程,在这一过程中,核酸模板与上述pcr随机引物混合后在高温下变性(95℃),变成单链的dna分子;随后上述pcr随机引物与核酸模板在25℃下退火5分钟,30℃下继续退火5分钟;退火后在链置换dna聚合酶的作用下进行延伸15分钟。由于链置换扩增,在一个延伸反应中从同一个模板会产生多个长度不一的拷贝。每个拷贝都带有一个独特的分子标记(umi);这样经过大于或等于2轮这样的反应,产生的新拷贝分子中有些会在2端都带有固定的序列(5’端是通用序列,3’端是通用序列互补序列),有些只有5’端带有通用序列(图2)。这种扩增方式可以使同一个dna分子在不同位点得到扩增。这不同于传统的pcr,它只在引物特异性位点得到扩增。umi和通用序列整合到扩增子中,便于纠正扩增中的错配,以及后续反应中的使用通用序列进行的pcr和测序接头的整合。不同于mda的长时间恒温扩增,也不同于malbac的每次反应中的酶添加操作,上述操作更为省时,更方便操作。
根据本发明的一些实施方式,步骤3)中,将第一轮pcr扩增的产物作为模板,加入无核酸酶去离子水、pcr预混液、特异性引物、5’接头引物和3’接头引物在第二温度循环程序中进行第二轮的pcr扩增。
本发明利用特异性引物,以第一轮pcr扩增的产物为模板进行pcr,对特异性产物进行富集,同时,测序接头也在此过程中加到产物两端,形成测序文库。5’接头引物和3’接头引物是测序平台特异性的接头引物,可以是illumina平台、bgi平台、iontorrent平台或其它测序平台的接头序列;特异性引物是以预扩增区域为模板设计的引物。特异性引物由2部分组成,3’端是靶区域特性序列,5’端序列与5’接头引物3’端相同(图3)。
根据本发明的一些实施方式,步骤3)中,第二温度循环程序可以为:
1个循环:98℃,30s;
25个循环:98℃,5s;65℃,5min;72℃,30s;
1个循环:72℃2min。
根据本发明的一些实施方式,步骤3)之后,还包括将第二轮pcr扩增的产物纯化的步骤。
根据本发明的一些实施方式,采用磁珠进行纯化。
术语解释:
umi:独特的分子标签。为一段短的dna序列,与待测序dna分子通过生化方法绑定后成为该分子的标签,可用来鉴别该分子。组成umi的核苷酸一般是随机的,通过a,t,g,c的随机组合,每个位置可以有4个选择。一个长度为n的随机dna片段可包含4^n个umi组合。
测序文库:为一个由不同dna分子组成的混和物。在ngs中,通常由于测序平台的不同,文库dna分子的两端有添加的通用接头序列。根据样本来源的不同,文库中的dna可能包含人dna,其它动物的dna,植物dna,或微生物dna。
dna引物:一段由核苷酸组成的单链dna。通常在18-30bp.用来作为dna聚合酶延伸dna链时的起始点。
dna聚合酶:可以单链dna,dna引物,脱氧核糖核苷三磷酸作为反应底物,其它无机盐作为辅助因子来合成dna互补链的酶。
pcr:聚合酶链反应。该反应由dna聚合酶催化完成,需要起始的dna模板,dna引物,脱氧核糖核苷三磷酸作为反应底物,其它无机盐作为辅助因子。产物为与dna模板互补,从引物处延长的单链dna。根据所使用的dna聚合酶的不同,该反应在特定的温度下进行。
链置换扩增:使用具有链置换能力的dna聚合酶进行的dna扩增。在dna聚合酶以dna作为模板延伸互补链的过程中,若下游遇到处于双链状态的dna时,具有链置换能力的dna聚合酶可以解开双链,继续延伸,合成新的互补链,同时旧的互补链被替换下来。
多重pcr:在一个pcr体系中使用大于一条或一对引物进行的pcr反应。
扩增子:由pcr反应产生的dna产物
探针:一定长度范围内的单链核酸,可以是rna或dna。在序列上与靶dna或rna互补。
mda:多重链置换扩增。在该体系中,多个dna引物用来扩增同一个dna靶区域。由于反应体系中用的具有链置换能力的dna聚合酶,比如说phi29dna聚合酶,靶区域可被不同引物同时扩增而富集。
malbac:多轮退火与成环扩增。在该体系中,带通用序列的随机引物被用来作链置换扩增。在多轮链置换扩增中涉及到温度的变化,包括dna模板变性,退火,延伸以及退火使产物成环。产物为5’,3’端互补的dna以及5’端带通用序列的dna。在第一轮后的扩增中,由于高温变性使dna聚合酶失活,要加入新的链置换dna聚合酶。
文库条形码:为短的dna片段,通常为6-12bp;整合到dna文库中用以区分不同的dna文库
reads:读序,指测序过程中单次测序所得到的碱基序列,由一连串的碱基符号(a,t,g,c)组成。根据测序原理的不同,可读出被测序dna模板中序列中碱基或其互补碱基。
以下结合具体的实施例对本发明的技术方案做进一步说明,但本发明并不限于这些具体实施方式。实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
本发明实施例中的待测基因组dna是来源于hek293t细胞系的基因组dna,使用前基因组dna被dna片段化酶(neb)消化成100-300bp的片段。
实施例1
1、引物设计:
第一轮pcr扩增用pcr随机引物(seqidno:14):
5’-agatcggaatcagacgtgtgctcttccgatctnnnnnngtnnnnnn-3’
第二轮pcr扩增用引物:
5’接头序列(seqidno:29):
5’-aatgatacggcgaccaccgagatctacactgttctctacactctttccctacacgacgctcttccga-3’
3’接头序列(seqidno:30):
5’-caagcagaagacggcatacgagataggttggggtgactggagttcagacgtgtgctcttccgatct-3’
特异性引物序列:序列如seqidno:31~161所示。
cctacacgacgctcttccgatctcactcaccgggcgagggcg(seqidno:31)
cctacacgacgctcttccgatctctctgggagatcttcacgctggggg(seqidno:32)
cctacacgacgctcttccgatcttgaaggtgcttggatctggcgctt(seqidno:33)
cctacacgacgctcttccgatctttgatgaagggcgggctcccatc(seqidno:34)
cctacacgacgctcttccgatcttgcccatcaagtggatggcgctg(seqidno:35)
cctacacgacgctcttccgatctaggaagtagcgtggccgccag(seqidno:36)
cctacacgacgctcttccgatcttaccgtgcgggcttcgtgctg(seqidno:37)
cctacacgacgctcttccgatctacggcccccactgcgtcaag(seqidno:38)
cctacacgacgctcttccgatctgagcagcagcgaaagcgccttg(seqidno:39)
cctacacgacgctcttccgatctcctggcctacctggtcgccatg(seqidno:40)
cctacacgacgctcttccgatctcggcagtccggcttggagga(seqidno:41)
cctacacgacgctcttccgatctcgttcatcgggacttggcagccag(seqidno:42)
cctacacgacgctcttccgatctcacaacgtcttggagcgccagagg(seqidno:43)
cctacacgacgctcttccgatctctcttgagggccacaaagtggccac(seqidno:44)
cctacacgacgctcttccgatctggcgctgtgtcctttcaggatggtg(seqidno:45)
cctacacgacgctcttccgatctagagaatctccattttagcacttacctgtgac(seqidno:46)
cctacacgacgctcttccgatcttctggatcagctggatggtcagcg(seqidno:47)
cctacacgacgctcttccgatcttgcctcacctccaccgtgcag(seqidno:48)
cctacacgacgctcttccgatctgcagccgaggaggagctggtg(seqidno:49)
cctacacgacgctcttccgatcttactggtcccgcatggcgct(seqidno:50)
cctacacgacgctcttccgatctggcagtagcggcttctaagggcaag(seqidno:51)
cctacacgacgctcttccgatctggtgtttgtagaggctgctgccctc(seqidno:52)
cctacacgacgctcttccgatctcccaggtcctcggacaccga(seqidno:53)
cctacacgacgctcttccgatctcccgggacgtgcacaacctc(seqidno:54)
cctacacgacgctcttccgatctacccccacccacagatccactg(seqidno:55)
cctacacgacgctcttccgatcttatggcacctgcaactgcttccctg(seqidno:56)
cctacacgacgctcttccgatctttggctggctggcccagttc(seqidno:57)
cctacacgacgctcttccgatctccatgtttggaactggcatcgccatga(seqidno:58)
cctacacgacgctcttccgatctgatggatgtgaaccccgagggcaaa(seqidno:59)
cctacacgacgctcttccgatctgtcaagccctccaacatcctagtcaa(seqidno:60)
cctacacgacgctcttccgatcttggggctgaaccaaggatgatgtgg(seqidno:61)
cctacacgacgctcttccgatctctgagcgtcatctgcccccac(seqidno:62)
cctacacgacgctcttccgatctaaccggggcagggattgcag(seqidno:63)
cctacacgacgctcttccgatctcaaccccaaccagtgctgcca(seqidno:64)
cctacacgacgctcttccgatctgcccactgtgttactgccatcgact(seqidno:65)
cctacacgacgctcttccgatctgatgaatgggccacactcactgtcgatg(seqidno:66)
cctacacgacgctcttccgatctggggagaatgtgaaaattccagtggccatc(seqidno:67)
cctacacgacgctcttccgatctttggctttgtgctcattaccttcagctgc(seqidno:68)
cctacacgacgctcttccgatctcaggggatgagctacctggaggatg(seqidno:69)
cctacacgacgctcttccgatctcacaacaaaacaggtgcaaagctgccag(seqidno:70)
cctacacgacgctcttccgatcttgacagaacgggaagccctcatgtc(seqidno:71)
cctacacgacgctcttccgatctcggctgttagtcactggcagcaac(seqidno:72)
cctacacgacgctcttccgatctctgggccccattcttctcccac(seqidno:73)
cctacacgacgctcttccgatctcctcatgtactggtccctcattgcactg(seqidno:74)
cctacacgacgctcttccgatctgctctgtgctgcatttcagagaacgc(seqidno:75)
cctacacgacgctcttccgatctctggattagctggattgtcagtgcgctt(seqidno:76)
cctacacgacgctcttccgatcttggtgctgaccatgtggacattaggtg(seqidno:77)
cctacacgacgctcttccgatctgctcccaaccaagctctcttgaggat(seqidno:78)
cctacacgacgctcttccgatctccaccgctgtgttgcagctacct(seqidno:79)
cctacacgacgctcttccgatcttccgacccaccatcatggcctg(seqidno:80)
cctacacgacgctcttccgatctcattctaggaagctcaccattcccccaag(seqidno:81)
cctacacgacgctcttccgatcttacccttgtccccaggaagcatacg(seqidno:82)
cctacacgacgctcttccgatctaggggtgaggcagtctttactcacc(seqidno:3383)
cctacacgacgctcttccgatctcgacagccccggtaaatcttggtg(seqidno:84)
cctacacgacgctcttccgatcttcctactcaccatcctgtgtgcagg(seqidno:85)
cctacacgacgctcttccgatctcacactgacgtgcctctccctc(seqidno:86)
cctacacgacgctcttccgatctaccacccccactgaacctctcttac(seqidno:87)
cctacacgacgctcttccgatctccgggctgtcgtggtagacttagaa(seqidno:88)
cctacacgacgctcttccgatctggataggcttgtaagtgcccgaagtgtaag(seqidno:89)
cctacacgacgctcttccgatcttttgggacctccggtcagaaaaccaa(seqidno:90)
cctacacgacgctcttccgatcttggtgtcctcctcctcttccctagataac(seqidno:9)
cctacacgacgctcttccgatctgattcgagaagtgacaggctatgtcctcg(seqidno:92)
cctacacgacgctcttccgatctggttgttgcctcgtgacaaggagatg(seqidno:93)
cctacacgacgctcttccgatctgtcaaggttgctgattttggtcttgccag(seqidno:94)
cctacacgacgctcttccgatctcaccacccctcggaatccct(seqidno:95)
cctacacgacgctcttccgatcttaaggcttacaacgatgtgggcaagacttc(seqidno:96)
cctacacgacgctcttccgatctcctgatgacctgaaggagtttctgcaga(seqidno:97)
cctacacgacgctcttccgatctcacttgatgatactcactgtccatcagcctc(seqidno:98)
cctacacgacgctcttccgatctgggctcccggaagacagtccc(seqidno:99)
cctacacgacgctcttccgatcttgtcctcatgtattggtctctcatggcac(seqidno:100)
cctacacgacgctcttccgatctggctttgaatctttggccagtacctcatg(seqidno:101)
cctacacgacgctcttccgatctagataatgactcacctggggccacatt(seqidno:102)
cctacacgacgctcttccgatctgcctgttttgtgtctactgttctagaaggcaa(seqidno:103)
cctacacgacgctcttccgatctacactgtacaagctctacgagaggtgtg(seqidno:104)
cctacacgacgctcttccgatctagttctttcttttgcacagggcattttggttg(seqidno:105)
cctacacgacgctcttccgatctctggtgaaaacaccgcagcatgtc(seqidno:106)
cctacacgacgctcttccgatctggaattcctgtgcatgaaagcactgc(seqidno:107)
cctacacgacgctcttccgatctgacaggaagagcacagtcactttgactc(seqidno:108)
cctacacgacgctcttccgatctcccaccctaaagacagcaccaatcttt(seqidno:109)
cctacacgacgctcttccgatcttcgagtttgcccccatgtacaagatc(seqidno:110)
cctacacgacgctcttccgatcttggaacttgacgagcagcagaagaag(seqidno:111)
cctacacgacgctcttccgatctcggaagatgaagatttcggatttcggct(seqidno:112)
cctacacgacgctcttccgatctaaaaattgtgaagatctgtgactttggcctgg(seqidno:113)
cctacacgacgctcttccgatcttgatgcccccaagaatcctagtagaatgttt(seqidno:114)
cctacacgacgctcttccgatctgactgttaccaccatacaggcgattaagaag(seqidno:115)
cctacacgacgctcttccgatctaaatgctgaaagctgtaccatacctgtctg(seqidno:116)
cctacacgacgctcttccgatctgcatctgtacagcatgaagtgcaagaac(seqidno:11)
cctacacgacgctcttccgatcttgatctccttagacaactacctttctacggac(seqidno:118)
cctacacgacgctcttccgatcttgttccttcatacacttctccaaaggctc(seqidno:119)
cctacacgacgctcttccgatctgggcttgttttgtatcaactgtccttgttg(seqidno:120)
cctacacgacgctcttccgatctattgccaacatgacttacttgatccccataag(seqidno:121)
cctacacgacgctcttccgatctacattacatacttaccatgccactttcccttg(seqidno:122)
cctacacgacgctcttccgatctttctgaattagctgtatcgtcaaggcactc(seqidno:123)
cctacacgacgctcttccgatcttttgaaagagaacacacttactctccacgtc(seqidno:124)
cctacacgacgctcttccgatctcacagtggagcgaattcctttggaaa(seqidno:125)
cctacacgacgctcttccgatctaacctcaatctctttgtccgtggtgtta(seqidno:126)
cctacacgacgctcttccgatctaaggacttttaccctcttcagctcagt(seqidno:127)
cctacacgacgctcttccgatctgggaccattatggacatcaggacattc(seqidno:128)
cctacacgacgctcttccgatctgtagatatgatgcagccattgacctgtttac(seqidno:129)
cctacacgacgctcttccgatctcctcttattgttccctacagattgcgag(seqidno:130)
cctacacgacgctcttccgatctctggtgccctataacaatgaatgaccaaa(seqidno:131)
cctacacgacgctcttccgatctcctccaacctaatagtgtattcacagagact(seqidno:132)
cctacacgacgctcttccgatcttggaatgccagaactacaatcttttgatgac(seqidno:133)
cctacacgacgctcttccgatctttcagggtttctgaatacctgaggtttttc(seqidno:134)
cctacacgacgctcttccgatctagcctggtagccaaaagctaagaat(seqidno:135)
cctacacgacgctcttccgatctctgttttcctgtagcaaaaccagaaatcc(seqidno:136)
cctacacgacgctcttccgatctctatgttcgaacaggtatctaccatggag(seqidno:137)
cctacacgacgctcttccgatcttcggatcacaaagatttgtgattttggtct(seqidno:138)
cctacacgacgctcttccgatctgtgcctaaattgtttgtagggttggtta(seqidno:139)
cctacacgacgctcttccgatctgcaagctttctcacaagcatttggtt(seqidno:140)
cctacacgacgctcttccgatctggtggtctttgggatcctcatc(seqidno:141)
cctacacgacgctcttccgatcttccgggctttacgcaaataagtaaga(seqidno:142)
cctacacgacgctcttccgatctctggatcccagaaggtgagaaagtta(seqidno:143)
cctacacgacgctcttccgatctgcttggttctgatgtttgtagtgtagc(seqidno:144)
cctacacgacgctcttccgatctacattttcgtaagtgttactcaagaagcagaa(seqidno:145)
cctacacgacgctcttccgatcttacagagtagacacaatgagtgaagctc(seqidno:146)
cctacacgacgctcttccgatctcctggtcatttatagaaaccgaggtatgaa(seqidno:147)
cctacacgacgctcttccgatctctaattcatctggagatcaaacccgcaa(seqidno:148)
cctacacgacgctcttccgatctaacagttatgattttgcagaaaacagatctgtatt(seqidno:149)
cctacacgacgctcttccgatctgttttgaaatgtgttttataatttagactagtgaa(seqidno:150)
cctacacgacgctcttccgatctcccatactttggatgatagaaacttcatctt(seqidno:151)
cctacacgacgctcttccgatctataggtttcatggactcagttactacctg(seqidno:152)
cctacacgacgctcttccgatcttggaaaaatagcctcaattcttaccatc(seqidno:153)
cctacacgacgctcttccgatctgacattttgtacttcttcaacgcgaag(seqidno:154)
cctacacgacgctcttccgatcttggcacattattctggggaatgtatat(seqidno:155)
cctacacgacgctcttccgatcttgagtattgttaaccttgcagaatgg(seqidno:156)
cctacacgacgctcttccgatcttgatctatttttccctttctcccca(seqidno:157)
cctacacgacgctcttccgatcttcctagagagttagagtaacttcaatgtcttt(seqidno:158)
cctacacgacgctcttccgatctctttgggttataaatagtgcactcagaat(seqidno:159)
cctacacgacgctcttccgatctctcctttaccagaatatgagagtaaagg(seqidno:160)
cctacacgacgctcttccgatctcttttagatctgagatgcacaataaaacag(seqidno:161)
2、基于多重pcr的靶向测序文库的构建
包括多轮链置换随机扩增(步骤1)和步骤2))和靶向富集(步骤3))过程。
1)10ng的片段化的待测基因组dna与1μl10μm的pcr随机引物混合后加无核酸酶去离子水补足至11μl,在95℃下变性2min,然后迅速移到冰浴中冷却3分钟;
2)准备4μl以下酶混合物,加到步骤1)的产物中。
酶混合物:1.5μl10xcutsmart缓冲液(neb),1μl10mmdntps(neb),12单位的bst3dna聚合酶(neb)。
按以下条件在pcr仪中进行反应:
1个循环:25℃5min,30℃5min,55℃15min;
3个循环:75℃1min,25℃5min,30℃5min,55℃15min;
1个循环:55℃10min。
得到第一轮pcr扩增的产物,在8℃保存。
3)第二轮的pcr扩增中各组分的用量如表1所示。
表1第二轮pcr扩增的各组分用量
在pcr仪中,反应条件如下:
1个循环:98℃,30s;
25个循环:98℃,5s;65℃,5min;72℃,30s;
1个循环:72℃2min。
反应完成后,产物用1倍体积的spriselect磁珠(beckmancoulter)进行纯化,纯化产物在bgi2000测序仪上进行双端测序。测序长度为100bp/端。
3、结果分析:
测序完成后,与人基因组序列进行序列匹配后,计算了评价靶向建库方法的3个参数:
1)在靶率:反映靶向建库方法富集靶区域的特异性。在靶率=匹配到预扩增靶区域的reads数/总reads数。
2)均一性:反映靶向建库方法富集众多靶区域是否存在偏好。如果偏好过大,表明有些靶区域没有得到很好的富集或有些靶区域被过于富集。均一性=匹配到靶区域的reads数超过平均在靶reads数的20%的靶区域数目/总的靶区域数目。
3)平均转化效率:反映出靶向建库方法对起始投入的dna分子捕获成库的效率。在测序达到饱和的状态下,平均转化效率=捕获的在靶平均分子数/投入的靶分子数
在此实施例中投入了约10nghek293t细胞系基因组dna,约合3300个投入的靶分子。
在此实施例中,样本测序总读序为2507765对,其中2256611对读序可以匹配到基因组中,匹配率为90%。1157871对读序可以正确匹配到预扩增区域,根据umi对在靶读序进行去重后,共有98150个dna分子。
根据上述公式进行计算,结果如图4所示,为本发明构建的测序文库的性能参数;其中,在靶率为46.2%。去重后均一性为97.7%,在测序总读序数为2507765的情况下平均转化效率为22.9%。随着测序深度的增加,会检测到更到的起始模板分子,预估计算所得平均转化效率会进一步加大。
本领域技术人员应该理解的是,本发明的使用不受限于上述特定应用。就本文描述或描绘的特定元素和/或特征而言,本发明也不局限于其优选实施方案。应当理解的是,本发明不限于所公开的实施方案例或各个实施方案,且在不脱离由以下权利要求所阐述和限定的本发明的范围的情况下能够进行许多重新布置、修改和替换。
sequencelisting
<110>广州迈景基因医学科技有限公司
<120>一种pcr随机引物和使用其构建靶向测序文库的方法
<130>111
<160>161
<170>patentinversion3.3
<210>1
<211>9
<212>dna
<213>人工序列(artificialsequence)
<400>1
agatcggaa9
<210>2
<211>7
<212>dna
<213>人工序列(artificialsequence)
<400>2
aagtgga7
<210>3
<211>8
<212>dna
<213>人工序列(artificialsequence)
<400>3
ctgagtcg8
<210>4
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>4
agatcg6
<210>5
<211>4
<212>dna
<213>人工序列(artificialsequence)
<400>5
agat4
<210>6
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>6
tcagacgtgtgctcttccgatct23
<210>7
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>7
gaacgacatggctacgatccgactt25
<210>8
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>8
ccatctcatccctgcgtgtctccgactcag30
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
gacgtgtgctcttccgatct20
<210>10
<211>2
<212>dna
<213>人工序列(artificialsequence)
<400>10
gt2
<210>11
<211>3
<212>dna
<213>人工序列(artificialsequence)
<400>11
gtg3
<210>12
<211>4
<212>dna
<213>人工序列(artificialsequence)
<400>12
gtga4
<210>13
<211>5
<212>dna
<213>人工序列(artificialsequence)
<400>13
gtgac5
<210>14
<211>45
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(37)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(40)..(45)
<223>nisa,c,g,ort
<400>14
agatcggaatcagacgtgtgctcttccgatctnnnnngtnnnnnn45
<210>15
<211>46
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(38)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(41)..(46)
<223>nisa,c,g,ort
<400>15
aagtggagaacgacatggctacgatccgacttnnnnnngtnnnnnn46
<210>16
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(39)..(44)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(47)..(52)
<223>nisa,c,g,ort
<400>16
ctgagtcgccatctcatccctgcgtgtctccgactcagnnnnnngtnnnnnn52
<210>17
<211>42
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(30)..(34)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(37)..(42)
<223>nisa,c,g,ort
<400>17
agatcggaagacgtgtgctcttccgatctnnnnngtnnnnnn42
<210>18
<211>39
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(27)..(31)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(34)..(39)
<223>nisa,c,g,ort
<400>18
agatcggacgtgtgctcttccgatctnnnnngtnnnnnn39
<210>19
<211>37
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(25)..(29)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(32)..(37)
<223>nisa,c,g,ort
<400>19
agatgacgtgtgctcttccgatctnnnnngtnnnnnn37
<210>20
<211>46
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(37)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(41)..(46)
<223>nisa,c,g,ort
<400>20
agatcggaatcagacgtgtgctcttccgatctnnnnngtgnnnnnn46
<210>21
<211>47
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(37)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(42)..(47)
<223>nisa,c,g,ort
<400>21
agatcggaatcagacgtgtgctcttccgatctnnnnngtgannnnnn47
<210>22
<211>48
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(37)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(43)..(48)
<223>nisa,c,g,ort
<400>22
agatcggaatcagacgtgtgctcttccgatctnnnnngtgacnnnnnn48
<210>23
<211>45
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(37)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(40)..(45)
<223>nisa,c,g,ort
<400>23
agatcggaatcagacgtgtgctcttccgatctnnnnngtnnnnnn45
<210>24
<211>48
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(40)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(43)..(48)
<223>nisa,c,g,ort
<400>24
agatcggaatcagacgtgtgctcttccgatctnnnnnnnngtnnnnnn48
<210>25
<211>51
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(43)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(46)..(51)
<223>nisa,c,g,ort
<400>25
agatcggaatcagacgtgtgctcttccgatctnnnnnnnnnnngtnnnnnn51
<210>26
<211>45
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(38)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(41)..(45)
<223>nisa,c,g,ort
<400>26
agatcggaatcagacgtgtgctcttccgatctnnnnnngtnnnnn45
<210>27
<211>48
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(38)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(41)..(48)
<223>nisa,c,g,ort
<400>27
agatcggaatcagacgtgtgctcttccgatctnnnnnngtnnnnnnnn48
<210>28
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(33)..(38)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(41)..(52)
<223>nisa,c,g,ort
<400>28
agatcggaatcagacgtgtgctcttccgatctnnnnnngtnnnnnnnnnnnn52
<210>29
<211>67
<212>dna
<213>人工序列(artificialsequence)
<400>29
aatgatacggcgaccaccgagatctacactgttctctacactctttccctacacgacgct60
cttccga67
<210>30
<211>66
<212>dna
<213>人工序列(artificialsequence)
<400>30
caagcagaagacggcatacgagataggttggggtgactggagttcagacgtgtgctcttc60
cgatct66
<210>31
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>31
cctacacgacgctcttccgatctcactcaccgggcgagggcg42
<210>32
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>32
cctacacgacgctcttccgatctctctgggagatcttcacgctggggg48
<210>33
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>33
cctacacgacgctcttccgatcttgaaggtgcttggatctggcgctt47
<210>34
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>34
cctacacgacgctcttccgatctttgatgaagggcgggctcccatc46
<210>35
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>35
cctacacgacgctcttccgatcttgcccatcaagtggatggcgctg46
<210>36
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>36
cctacacgacgctcttccgatctaggaagtagcgtggccgccag44
<210>37
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>37
cctacacgacgctcttccgatcttaccgtgcgggcttcgtgctg44
<210>38
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>38
cctacacgacgctcttccgatctacggcccccactgcgtcaag43
<210>39
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>39
cctacacgacgctcttccgatctgagcagcagcgaaagcgccttg45
<210>40
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>40
cctacacgacgctcttccgatctcctggcctacctggtcgccatg45
<210>41
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>41
cctacacgacgctcttccgatctcggcagtccggcttggagga43
<210>42
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>42
cctacacgacgctcttccgatctcgttcatcgggacttggcagccag47
<210>43
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>43
cctacacgacgctcttccgatctcacaacgtcttggagcgccagagg47
<210>44
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>44
cctacacgacgctcttccgatctctcttgagggccacaaagtggccac48
<210>45
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>45
cctacacgacgctcttccgatctggcgctgtgtcctttcaggatggtg48
<210>46
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>46
cctacacgacgctcttccgatctagagaatctccattttagcacttacctgtgac55
<210>47
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>47
cctacacgacgctcttccgatcttctggatcagctggatggtcagcg47
<210>48
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>48
cctacacgacgctcttccgatcttgcctcacctccaccgtgcag44
<210>49
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>49
cctacacgacgctcttccgatctgcagccgaggaggagctggtg44
<210>50
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>50
cctacacgacgctcttccgatcttactggtcccgcatggcgct43
- 还没有人留言评论。精彩留言会获得点赞!