一种RBS优化提高γ-谷氨酰胺转肽酶表达量的方法与流程
本发明涉及一种rbs优化提高γ-谷氨酰胺转肽酶表达量的方法,属于基因工程领域。
背景技术:
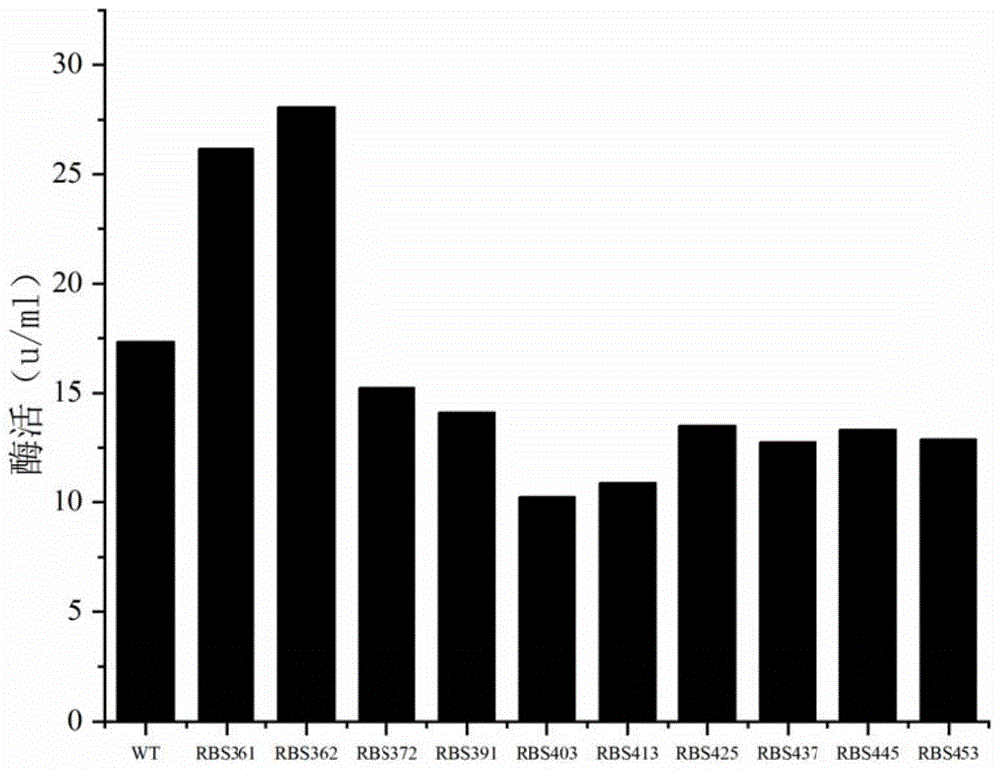
:γ-谷氨酰胺转肽酶(γ-glutamyltranspeptidase,γ-gt,ec2.3.2.2)负责催化γ-谷氨酰类化合物的γ-谷氨酰分子转移到其他氨基酸、短肽等受体分子上(转肽反应)或水分子上(水解反应),广泛存在于哺乳动物和细菌中。γ-谷氨酰胺转肽酶可以用于临床诊断、催化合成氨基酸衍生物方面、催化合成药物或药物前体、催化合成茶氨酸、谷胱甘肽等,还可以用于改善某些苦味氨基酸的风味。这对于医学、化工行业以及食品的发展意义重大。同时,γ-谷氨酰胺转肽酶在工业上也用来生产l-茶氨酸,因此对于γ-谷氨酰胺转肽酶酶活的提高对于工业生产也具有很大的意义。发明人课题组前期利用pxmj19质粒,实现了b.subtilis168来源的γ-谷氨酰胺转肽酶基因在c.crenatumsdnn403的表达。同时发现γ-谷氨酰胺转肽酶蛋白的信号肽片段可以在钝齿棒杆菌中被识别,从而引导该目的蛋白分泌到胞外,但是,胞外γ-谷氨酰胺转肽酶活力仅为10.31u/ml,并申请了相关专利(申请号201610454875.8),而一种提高γ-谷氨酰胺转肽酶表达量的方法(申请号:201610576989.x)中提到以钝齿棒状菌为宿主,并在γ-谷氨酰胺转肽酶基因后加poly(a/t)尾巴,酶活也仅仅才有20.62u/ml;但是,相比于工业化生产要求,该酶的表达量偏低,会增加上游生产成本。因此,目前急需得到一种提高γ-谷氨酰胺转肽酶表达量的方法,以满足工业化的生产需求以及降低生产成本。技术实现要素:本发明提供了一种载体,所述载体是将载体pma5上核苷酸序列如seqidno.2所示的原始rbs序列替换为核苷酸序列如seqidno.3所示的序列得到的,命名为pma5-rbs361;或者所述载体是将载体pma5上核苷酸序列如seqidno.2所示的原始rbs序列替换为核苷酸序列如seqidno.4所示的序列得到的,命名为pma5-rbs362。在本发明的一种实施方式中,所述表达载体还包括目的基因。本发明还提供了含有上述载体的微生物细胞。在本发明的一种实施方式中,所述微生物细胞为细菌或真菌。在本发明的一种实施方式中,所述微生物细胞为枯草芽孢杆菌。在本发明的一种实施方式中,所述微生物细胞为枯草芽孢杆菌168。本发明还提供了一种提高目的基因表达量的方法,所述方法是利用上述表达载体将目的基因在宿主细胞中进行重组表达。在本发明的一种实施方式中,所述目的基因为编码γ-谷氨酰胺转肽酶的基因。在本发明的一种实施方式中,所述γ-谷氨酰胺转肽酶的氨基酸序列如seqidno.1所示。本发明还提供了上述载体,或上述微生物细胞,或上述方法在制备l-茶氨酸中的应用。本发明还提供了一种制备l-茶氨酸的方法,所述方法为将携带编码γ-谷氨酰胺转肽酶基因上述表达载体转入宿主细胞中构建得到重组菌,利用所述重组菌转化底物l-谷氨酰胺和乙胺生产得到l-茶氨酸。有益效果:本发明对表达载体pma5的rbs序列进行改造,提高了酶的翻译效率从而提高酶活,采用本发明提供的载体pma5-rbs361和pma5-rbs362构建的重组枯草芽孢杆菌中,使目的基因γ-谷氨酰胺转肽酶的酶活得到了提高;其表达量较原始pma5-ggt/b.subtilis168分别提高了1.51倍和1.62倍。附图说明图1:含有不同rbs序列的重组菌发酵得到的胞外粗酶液中γ-谷氨酰胺转肽酶的酶活。图2:rbs361、rbs362序列、原始rbs序列的sd结构。具体实施方式下述实施例所涉及的pma5质粒购自生物风公司,b.subtilis168感受态细胞、大肠杆菌jm109感受态细胞均购自invitrogen公司。下述实施例中所涉及的培养基如下:发酵培养基:葡萄糖10g/l,胰蛋白胨15g/l,酵母提取物20g/l,mgso41g/l和k2hpo4·3h2o2g/l,添加卡那霉素(50mg/ml)至终浓度为50μg/ml,调节ph为7.2。lb固体培养基:酵母提取物5g/l,胰蛋白胨10g/l、氯化钠10g/l、琼脂20g/l。lb液体培养基:酵母提取物5g/l,胰蛋白胨10g/l、氯化钠10g/l。下述实施例中所涉及的检测方法如下:γ-谷氨酰胺转肽酶酶活测定方法:采用比色法测定,具体方法如下:取培养48h的菌液50ml,于4℃条件下10000r/min离心30min收集上清,上清液中即为目的蛋白。酶活测定方法如下:1ml的标准反应体系中包含100mmtris-hcl(ph10),2.5mmγ-l-谷氨酰基-对硝基苯胺一水合物,60mm双甘二肽和5μl适当稀释的酶液。37℃反应15min后添加0.2ml3.5mol的醋酸终止反应,不添加双甘二肽作为对照,410nm处测定吸光度值。通过实验组和对照组的吸光度差值即可计算转肽酶活。转肽酶活定义:在37℃反应条件下,每分钟内由γ-l-谷氨酰基-对硝基苯胺一水合物经转肽反应生成1μmol对硝基苯胺所需要的酶量定义为一个单位标准酶活。实施例1:γ-谷氨酰胺转肽酶的重组载体的构建γ-谷氨酰胺转肽酶ggt基因的获得:化学合成核苷酸序列如seqidno.5所示的γ-谷氨酰胺转肽酶ggt基因。分别用bamhi、mlui双酶切pma5质粒与上述获得的ggt基因,得到酶切后的产物,将酶切后的产物经纯化后用同源重组连接酶37℃连接1h,得到连接产物;将得到的连接产物通过化学法转化大肠杆菌jm109感受态细胞,得到转化产物;将转化产物涂布含氨苄霉素(100mg/l)的lb固体培养基,挑取正确克隆子,提取质粒测序,双酶切验证构建的重组质粒,得到重组原始质粒,命名为pma5-wt-ggt。测序工作由上海生工完成。实施例2:含有不同rbs序列的基因的获得以pma5-wt-ggt质粒作为模板,以表1中的引物序列,构建含有不同rbs序列(如表2)的基因片段。分别得到经改造后的基因:rbs361-ggt,rbs362-ggt,rbs372-ggt,rbs391-ggt,rbs403-ggt,rbs413-ggt,rbs425-ggt,rbs437-ggt,rbs445-ggt,rbs453-ggt。表1引物序列表2不同rbs的序列名称序列wtgccacctaaaaaggagcgattta(seqidno.2)rbs361actgtattcactaaaaaaggtaggtcgtatttc(seqidno.3)rbs362ccgaacctaggcccacaaggaggtcgtaag(seqidno.4)rbs372aaaggttagctctggc(seqidno.25)rbs391gtttgtctttaatctagaaaaaggaggtattttc(seqidno.26)rbs403cccgtccgcggccaggagcccttacc(seqidno.27)rbs413aacagatacttctattctaggtcgctaa(seqidno.28)rbs425cttttattctgactaaaggaagcc(seqidno.29)rbs437cgcctaaacctaaattgtcttcgtt(seqidno.30)rbs445ttaggttagctctggc(seqidno.31)rbs453aacggttagctctggc(seqidno.32)实施例3:含有不同rbs序列的γ-谷氨酰胺转肽酶的重组载体的构建(1)以pma5质粒为模板,分别用下列引物序列pma5-f、pma5-r反向pcr,再用dpni37℃酶切1小时,得到酶切产物。pma5-f:acgcgttctagaggtcgaaattcacctcgaaagca(seqidno.34)pma5-r:acaaatgtgaggcattttcgctctttccggcaacc(seqidno.35)(2)将步骤(1)得到的酶切产物以及实施例2得到的含有不同rbs序列的ggt基因:rbs361-ggt,rbs362-ggt,rbs372-ggt,rbs391-ggt,rbs403-ggt,rbs413-ggt,rbs425-ggt,rbs437-ggt,rbs445-ggt,rbs453-ggt,经纯化后用同源重组连接酶37℃连接1h,得到连接产物。(3)将步骤(2)得到的连接产物通过化学法转化大肠杆菌jm109感受态细胞,得到转化产物。将转化产物涂布含氨苄霉素(100mg/l)的lb固体培养基,挑取正确克隆子,提取质粒测序,用bamhi、mlui双酶切验证构建的重组质粒,得到重组突变体质粒,测序工作由上海生工完成。将重组突变体质粒分别命名为:pma5-rbs361-ggt,pma5-rbs362-ggt,pma5-rbs372-ggt,pma5-rbs391-ggt,pma5-rbs403-ggt,pma5-rbs413-ggt,pma5-rbs425-ggt,pma5-rbs437-ggt,pma5-rbs445-ggt,pma5-rbs453-ggt。实施例4:产γ-谷氨酰胺转肽酶枯草芽孢杆菌工程菌构建分别将实施例1和实施例3得到的重组原始质粒及重组突变体质粒化学法转化入b.subtilis168感受态细胞,得到转化子,取转化子涂布卡那霉素(50mg/ml)lb固体培养基中。37℃培养12h,挑取阳性转化子验证。验证成功即得到以下重组菌,分别命名为:b.subtilis168/pma5-wt-gg,b.subtilis168/pma5-rbs361-ggt,b.subtilis168/pma5-rbs362-ggt,b.subtilis168/pma5-rbs372-ggt,b.subtilis168/pma5-rbs391-ggt,b.subtilis168/pma5-rbs403-ggt,b.subtilis168/pma5-rbs413-ggt,b.subtilis168/pma5-rbs425-ggt,b.subtilis168/pma5-rbs437-ggt,b.subtilis168/pma5-rbs445-ggt,b.subtilis168/pma5-rbs453-ggt。实施例5:γ-谷氨酰胺转肽酶高效表达及酶活测定将实施例4获得的重组菌的单菌落分别接种于10ml含卡那霉素(50mg/ml)的lb液体培养基中,37℃,200rmp,培养10h后,得到种子液;将种子液按6%(v/v)的接种量转接于发酵培养基中,37℃培养48h,得到发酵液;将发酵液于4℃、10000r/min离心10min,上清为胞外粗酶液,用于酶活力的测定,酶活如表3所示:表3:含有不同rbs序列的重组菌发酵得到的胞外粗酶液中γ-谷氨酰胺转肽酶的酶活由表3和图1可知,酶的表达量提高最多的是采用rbs362序列,其酶活是原始序列的1.62倍,其次是是采用rbs361序列,其酶活是原始序列的1.51倍。对于两株表达量提高的rbs序列的sd结构进行分析(结果图2所示),从图2中可知采用人工rbs361、rbs362序列较原始rbs序列的sd的二级结构有明显的改善。虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。sequencelisting<110>江南大学<120>一种rbs优化提高γ-谷氨酰胺转肽酶表达量的方法<130>baa200900a<160>35<170>patentinversion3.3<210>1<211>584<212>prt<213>人工序列<400>1metlysargileserleuthrvalleuserilecysleuphevalphe151015serphepheleuprovalserglnvalthralaasngluthrhisgly202530asnlysvalalavalglylysaspglymetvalalathralahispro354045leualasergluileglyalaaspvalleulyslysglyglyasnala505560valaspalaalavalalaileglntyralaleuasnvalthrglupro65707580metmetserglyileglyglyglyglyphemetmetvaltyraspgly859095lysthrlysgluthrserileileasnserarggluargalaprogln100105110glyalathrproaspmetpheleuthraspaspglylysvalilepro115120125phealagluargserthrhisglyasnalavalglyvalproglythr130135140vallysglyleuglualaalaleuasplystrpglythrargsermet145150155160lysgluleuilegluproserileglnleualagluaspglypheglu165170175ileaspservalleualalysalaileaspasphisglnalalysleu180185190lyslysthralaalaalaproilepheleuproasnaspglnproleu195200205glugluglyaspleuleuvalglnproglyleualalysthrphelys210215220leuilealalyslysglyserlysalaphetyrgluglylysvalala225230235240lysalaleualaasnthrvalglnasppheglyglythrmetthrser245250255lysaspilelysargtyrgluvallysthrasplysproiletrpgly260265270asptyrlysglytyrglnleualasermetproproprosersergly275280285glyvalphemetleuglnileleulysileleuasphispheasnleu290295300serglntyraspprolysserpheglulystyrglnleuleualaglu305310315320thrmethisleusertyralaaspargalaalatyralaglyasppro325330335gluphevalaspvalproleulysglyleuleuaspaspasptyrile340345350sergluargalaserleuileglnleuaspglnmetasnargserpro355360365lysgluglyaspprotrpalatyrgluaspglulysasnproserpro370375380ilevalproglnprogluasplysthrileglygluthrthrhisphe385390395400thrvalalaaspglntrpglyasnvalvalserphethrthrthrile405410415gluglnleupheglythrglyileleuvalproglutyrglyphephe420425430leuasnasngluleuthrasppheaspalaargproglyglyalaasn435440445gluvalglnproasnlysargproleusersermetthrprothrile450455460ilephelysaspglygluprovalmetthrvalglyserproglygly465470475480thrthrileilealaservalserglnthrileleuasnleuleuglu485490495tyraspmetgluleuglnaspalavalglugluproargiletyrthr500505510asnserleuthrsertyrargtyrgluvalglyvalproleuaspval515520525argthrlysleuasnaspmetglyhisglnpheglyserserproile530535540aspileglyasnvalglnalaleuleuileasparglysalaglythr545550555560phethrglyvalalaaspserthrargasnglythralavalglyval565570575asnleulysvalalaalaaspgln580<210>2<211>23<212>dna<213>人工序列<400>2gccacctaaaaaggagcgattta23<210>3<211>33<212>dna<213>人工序列<400>3actgtattcactaaaaaaggtaggtcgtatttc33<210>4<211>30<212>dna<213>人工序列<400>4ccgaacctaggcccacaaggaggtcgtaag30<210>5<211>1755<212>dna<213>人工序列<400>5atgaaacgcatttctctaactgttttgtccatatgtctgtttgttttctcgttttttctg60ccggtcagccaagtcactgcaaacgaaactcatgggaataaagtagctgttggcaaagat120gggatggtggctactgcacatccgcttgcatcggagattggtgctgacgtattgaaaaaa180ggtgggaacgcagtggatgctgctgttgccattcagtacgcacttaacgtaacagagcca240atgatgtctggaattggcggcggcggatttatgatggtttatgatgggaagacaaaggaa300acatccatcatcaatagcagagagcgagcaccacagggcgcaacacctgacatgttttta360acagatgatggaaaagtgattccgtttgctgagcgatctacacatggaaatgcggtaggt420gttccagggactgtaaaaggtcttgaagctgcattagataagtggggtactcgttctatg480aaggaattgattgagccctccattcaacttgcagaagatggttttgaaattgattctgtc540ttggcaaaagcgattgatgatcatcaagcaaaattgaaaaaaacggccgcagcgccaatt600tttcttccaaatgatcagccgctcgaagaaggagatctgcttgtccagccaggtcttgca660aaaacatttaaacttattgcgaaaaaaggaagcaaagcgttttatgaaggaaaagtagca720aaggcacttgcaaatacagttcaagattttggcgggacgatgacttcaaaagatatcaaa780cgttatgaagtcaagactgacaagccaatctggggagactataaaggatatcagcttgca840agcatgccaccaccaagctcaggcggggtgtttatgctacaaattctcaaaatacttgac900cattttaacctgtctcagtatgaccccaaatcattcgaaaaatatcagcttcttgctgaa960acgatgcacctctcctatgctgacagagccgcatatgccggcgaccccgaattcgtagat1020gttccactaaaaggactattagacgatgattacatttcagaaagagcctctctcattcaa1080ttagatcaaatgaatcgcagcccgaaagaaggagatccttgggcatatgaggatgaaaaa1140aatccatccccaattgttcctcagccagaagataaaaccattggtgagacgacccatttt1200actgttgcagatcagtggggaaatgttgtgtcattcaccactaccattgaacaattattt1260ggtacagggattcttgttccagagtatggatttttcttaaataatgaactaactgatttt1320gatgcacgacctggcggtgcaaatgaagtccagccaaataaacgtccattatcaagtatg1380accccgacgatcatttttaaagatggagagcctgtcatgactgttggatctcctggtgga1440acgacgatcattgcttctgtttcacagacgattctcaatttacttgaatatgacatggag1500cttcaggatgcggtagaagagccaagaatctacacaaacagcttaacttcgtatcgttat1560gaagtaggtgtccctctagatgtaagaacaaagttaaatgatatgggtcaccagtttggc1620agttcacctattgatatagggaatgtgcaagctttactgattgatcgaaaagcagggaca1680tttactggagtagctgattcaacgagaaatggaactgctgttggtgtcaatctaaaggta1740gctgcagatcaatag1755<210>6<211>50<212>dna<213>人工序列<400>6gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>7<211>65<212>dna<213>人工序列<400>7gaaaatgcctcacatttgtccgaacctaggcccacaaggaggtcgtaagcatatgagtta60tgcag65<210>8<211>50<212>dna<213>人工序列<400>8gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>9<211>48<212>dna<213>人工序列<400>9cgaaaatgcctcacatttgtaaaggttagctctggccatatgagttat48<210>10<211>50<212>dna<213>人工序列<400>10gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>11<211>66<212>dna<213>人工序列<400>11cgaaaatgcctcacatttgtgtttgtctttaatctagaaaaaggaggtattttccatatg60agttat66<210>12<211>50<212>dna<213>人工序列<400>12gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>13<211>58<212>dna<213>人工序列<400>13cgaaaatgcctcacatttgtcccgtccgcggccaggagcccttacccatatgagttat58<210>14<211>50<212>dna<213>人工序列<400>14gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>15<211>60<212>dna<213>人工序列<400>15cgaaaatgcctcacatttgtaacagatacttctattctaggtcgctaacatatgagttat60<210>16<211>50<212>dna<213>人工序列<400>16gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>17<211>56<212>dna<213>人工序列<400>17cgaaaatgcctcacatttgtcttttattctgactaaaggaagcccatatgagttat56<210>18<211>50<212>dna<213>人工序列<400>18gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>19<211>57<212>dna<213>人工序列<400>19cgaaaatgcctcacatttgtcgcctaaacctaaattgtcttcgttcatatgagttat57<210>20<211>50<212>dna<213>人工序列<400>20gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>21<211>48<212>dna<213>人工序列<400>21cgaaaatgcctcacatttgtttaggttagctctggccatatgagttat48<210>22<211>50<212>dna<213>人工序列<400>22gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>23<211>48<212>dna<213>人工序列<400>23cgaaaatgcctcacatttgtaacggttagctctggccatatgagttat48<210>24<211>50<212>dna<213>人工序列<400>24gtgaatttcgacctctagaacgcgtctattgatctgcagctacctttaga50<210>25<211>16<212>dna<213>人工序列<400>25aaaggttagctctggc16<210>26<211>34<212>dna<213>人工序列<400>26gtttgtctttaatctagaaaaaggaggtattttc34<210>27<211>26<212>dna<213>人工序列<400>27cccgtccgcggccaggagcccttacc26<210>28<211>28<212>dna<213>人工序列<400>28aacagatacttctattctaggtcgctaa28<210>29<211>24<212>dna<213>人工序列<400>29cttttattctgactaaaggaagcc24<210>30<211>25<212>dna<213>人工序列<400>30cgcctaaacctaaattgtcttcgtt25<210>31<211>16<212>dna<213>人工序列<400>31ttaggttagctctggc16<210>32<211>16<212>dna<213>人工序列<400>32aacggttagctctggc16<210>33<211>65<212>dna<213>人工序列<400>33cgaaaatgcctcacatttgtactgtattcactaaaaaaggtaggtcgtatttccatatga60gttat65<210>34<211>35<212>dna<213>人工序列<400>34acgcgttctagaggtcgaaattcacctcgaaagca35<210>35<211>35<212>dna<213>人工序列<400>35acaaatgtgaggcattttcgctctttccggcaacc35当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1