高丝氨酸乙酰转移酶突变体及其在生产O-乙酰高丝氨酸中的应用的制作方法
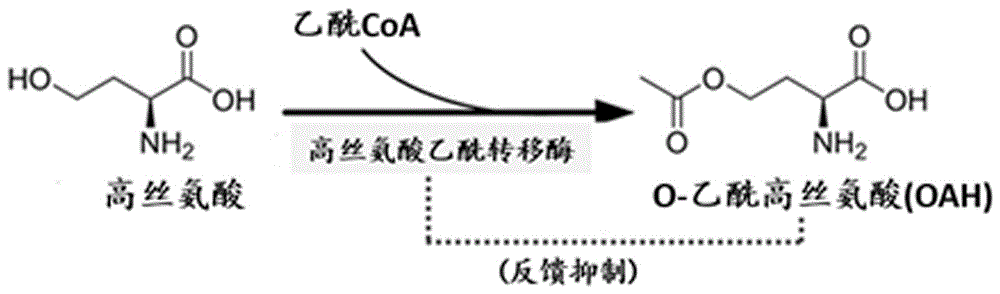
本发明涉及一种酶活性和抗反馈抑制提升的高丝氨酸乙酰转移酶突变体及其在制备o-乙酰高丝氨酸中应用,属于生物工程领域。
背景技术:
l-甲硫氨酸是一种重要的含硫氨基酸,广泛应用于饲料,食品,医药以及化妆品等领域,具有广阔的应用前景(huangjf,etal.metabolicengineeringofescherichiacoliformicrobialproductionofl-methionine.biotechnolbioeng,2017,114:843-51)。目前,甲硫氨酸主要通过化学合成的方法进行生产。然而由于其能耗高,工艺路线复杂,生产成本高,环境污染大,从而极大地限制了大规模甲硫氨酸的工业化生产。同时化学合成法的产物为dl-甲硫氨酸的混合物,分离难度大,仅能应用于饲料添加。随着能源和环境危机的日益严峻,利用成本低廉、环境友好、产物单一的微生物发酵法合成l-甲硫氨酸的研究越来越受到关注。然而由于细胞中l-甲硫氨酸合成途径复杂且受到严谨的调控作用,目前l-甲硫氨酸细胞工厂的产量和得率都比较低,尚无法满足工业化生产的需求。
o-乙酰高丝氨酸(oah)是l-甲硫氨酸合成的重要前体,能够在乙酰高丝氨酸巯解酶(ec2.5.1.49)的作用下直接与甲硫醇反应生成l-甲硫氨酸(willket.methionineproduction-acriticalreview.applmicrobiolbiot,2014,98:9893-914)。基于上述工艺路线,一种偶联发酵-酶转化的工艺路线被开发并用于l-甲硫氨酸的生产。该工艺首先通过微生物发酵合成oah,然后再通过酶催化缩合甲硫醇生成l-甲硫氨酸,可以获得较高的产率。在该工艺中,通过发酵法合成oah是制约l-甲硫氨酸合成的关键问题之一。高得率的oah发酵生产能够有效降低生产成本,提高l-甲硫氨酸的生产效率。在微生物细胞中,o-乙酰高丝氨酸是由高丝氨酸和乙酰辅酶a在高丝氨酸乙酰转移酶(homoserineo-acetyltransferase,hat,ec:2.3.1.31)的催化作用下形成的。然而研究表明,高丝氨酸乙酰转移酶不仅具有较低的催化活性,还受到产物oah的强烈反馈抑制,从而限制了oah的高效生产(bourhyp,etal.homoserineo-acetyltransferase,involvedintheleptospirameyerimethioninebiosyntheticpathway,isnotfeedbackinhibited.jbacteriol,1997,179:4396-98)。因此,构建酶活性和抗反馈抑制能力提高的高丝氨酸乙酰转移酶突变体,对oah的高效生产具有非常重要的意义。
技术实现要素:
本发明的首要目的在于提供一种高丝氨酸乙酰转移酶突变体,以使得其酶活性和抗反馈抑制能力得到提高。
本发明提供一种来源于leptospirameyeri的高丝氨酸乙酰转移酶突变体,其特征在于,相对于seqidno.2所示氨基酸序列而言,仅存在第118位由甘氨酸突变为缬氨酸,第147位由苯丙氨酸突变为异亮氨酸,第182位由甲硫氨酸突变为苏氨酸或者第240位甲硫氨酸突变为甘氨酸中的一个或多个位点突变。优选实施方式之一是,仅存在第118位由甘氨酸突变为缬氨酸,第147位由苯丙氨酸突变为异亮氨酸,第182位由甲硫氨酸突变为苏氨酸或者第240位甲硫氨酸突变为甘氨酸中的一个位点突变;仅存在第118位由甘氨酸突变为缬氨酸和第147位由苯丙氨酸突变为异亮氨酸的组合突变;仅存在第118位由甘氨酸突变为缬氨酸和第182位由甲硫氨酸突变为苏氨酸的组合突变;仅存在第118位由甘氨酸突变为缬氨酸和第240位甲硫氨酸突变为甘氨酸的组合突变;仅存在第147位由苯丙氨酸突变为异亮氨酸和第182位由甲硫氨酸突变为苏氨酸的组合突变;仅存在第147位由苯丙氨酸突变为异亮氨酸和第240位甲硫氨酸突变为甘氨酸的组合突变;仅存在第182位由甲硫氨酸突变为苏氨酸和第240位甲硫氨酸突变为甘氨酸的组合突变;仅存在第118位由甘氨酸突变为缬氨酸,第147位由苯丙氨酸突变为异亮氨酸和第182位由甲硫氨酸突变为苏氨酸的组合突变;仅存在第147位由苯丙氨酸突变为异亮氨酸,第182位由甲硫氨酸突变为苏氨酸和第240位甲硫氨酸突变为甘氨酸的组合突变;仅存在第118位由甘氨酸突变为缬氨酸,第182位由甲硫氨酸突变为苏氨酸以及第240位甲硫氨酸突变为甘氨酸中的组合突变;或者仅存在第118位由甘氨酸突变为缬氨酸,第147位由苯丙氨酸突变为异亮氨酸,第182位由甲硫氨酸突变为苏氨酸和第240位甲硫氨酸突变为甘氨酸的组合突变。
其中最优选地,仅存在第147位由苯丙氨酸突变为异亮氨酸,第182位由甲硫氨酸突变为苏氨酸以及第240位甲硫氨酸突变为甘氨酸中的组合突变。
本发明进一步提供所述高丝氨酸乙酰转移酶突变体的编码基因。优先地实施方式中,所述高丝氨酸乙酰转移酶突变体的编码基因的核苷酸序列是在seqidno.1所示核苷酸酸序列的基础进行突变获得的。在更具体地实施方式中,所述的高丝氨酸乙酰转移酶突变体的编码基因核苷酸序列优选为seqidno.3(编码g118v-f147i-m182t)、seqidno.4(编码g118v-f147i-m240g)、seqidno.5(编码f147i-m182t-m240g)以及seqidno.6(编码g118v-f147i-m182t-m240g)所示。
本发明还提供含有如所述这高丝氨酸乙酰转移酶突变体的编码基因的表达载体和宿主细胞。
本发明还提供所述的高丝氨酸乙酰转移酶突变体或其编码基因在制备o-乙酰高丝氨酸或甲硫氨酸中的应用。在具体实施方式中,其是通过构建微生物细胞工厂用于发酵生产o-乙酰高丝氨酸或甲硫氨酸。在更具体的实施方式中,所述微生物细胞是大肠杆菌或谷氨酸棒杆菌。
本发明是通过下述的技术思路实现的:将来源于leptospirameyeri的高丝氨酸乙酰转移酶(hat)基因构建到表达载体上,获得带有目的酶编码基因的重组质粒。将上述重组质粒转入大肠杆菌感受态中,进行目的酶诱导表达,并对酶活性和抗反馈抑制能力进行测定。本发明采用易错pcr随机突变方法所述的高丝氨酸乙酰转移酶基因进行定向进化。通过对目的基因片段进行pcr扩增,获得目的基因突变序列文库,并将其构建到表达载体上,获得高丝氨酸乙酰转移酶突变体质粒文库。
本发明采用高通量筛选的方法对所述的高丝氨酸乙酰转移酶突变体质粒文库进行高通量筛选,获得了三个酶活性提高的突变体,其突变位点分别为g118v、f147i和m182t(即原始基因序列的第118位由甘氨酸突变为缬氨酸,第147位由苯丙氨酸突变为异亮氨酸,第182位由甲硫氨酸突变为苏氨酸)。
本发明还对所述的高丝氨酸乙酰转移酶进行同源建模,并对底物通道3å范围内的氨基酸残基(l236,m240,y267,e270,p348,a349,l356和p357)进行饱和定点突变,经过高通量筛选获得了酶活性和抗反馈抑制能力提高的突变体m240g(即原始基因序列的第240位甲硫氨酸突变为甘氨酸)。最后还所获得的酶活性提高的突变体g118v、f147i、m182t和m240g进行组合突变,获得了最优的突变组合f147i-m182t-m240g,该突变体具有最高的酶活性和抗反馈抑制能力。
其中,在进行研究时,先对所述高丝氨酸乙酰转移酶(hat)基因进行密码子优化(优化后的核苷酸序列如seqidno.1所示),以更有利于表达和达到最终效果。
本发明针对高丝氨酸乙酰转移酶催化活性低和/或反馈抑制强烈等问题,分别利用易错pcr随机突变和基于结构信息的定点饱和突变方法获得了多种酶活性和/或抗反馈抑制能力提高的高丝氨酸乙酰转移酶突变体。在最最优的突变组合体即f147i-m182t-m240g,其酶活性比野生型提高了15.2倍,并具有较强的反馈抑制抗性,当10mmo-乙酰高丝氨酸存在时仍能保持42.7%的剩余酶活性,因此本发明的高丝氨酸乙酰转移酶突变体能够为高效生产o-乙酰高丝氨酸及下游代谢产物奠定了良好基础。
附图说明
图1高丝氨酸乙酰转移酶催化反应示意图。
图2高丝氨酸乙酰转移酶有益突变位点的组合突变体的酶活。
具体实施方式
下面通过实施例来进一步说明本发明的内容,但不应理解为对本发明的限制。实施例中所使用的实验方法如无特殊说明,均为常规方法。实施例中所使用的材料、试剂等如无特殊说明,均可从商业途径得到。
实施例1:高丝氨酸乙酰转移酶的诱导表达和酶活测定
1.1重组质粒的构建
本发明将来源于leptospirameyeri的高丝氨酸乙酰转移酶基因进行按照大肠杆菌的密码子使用频率进行密码子优化,核苷酸序列如seqidno.1所示,并进行基因合成(其编码的氨基酸序列如seqidno.2)。利用限制性内切酶ndei和xhoi连接至pet21b表达载体上,构建重组质粒。
1.2将重组质粒的转化至大肠杆菌bl21(de3)中
具体转化方法为:(1)取10μl重组质粒于50μl大肠杆菌bl21(de3)感受态细胞中,冰浴30min;(2)42℃水浴热激90s,快速置于冰上1~2min;(3)加入新鲜lb液体培养基(蛋白胨10g/l,酵母粉5g/l,nacl10g/l)600μl,于37ºc振荡培养60min;(4)取200μl菌液涂布于含有100μg/ml氨苄青霉素的lb固体培养基表面,37ºc培养12~16h。
1.3高丝氨酸乙酰转移酶的诱导表达
具体方法为:挑取单菌落至含有氨苄青霉素的lb液体培养基中,37ºc、200r/min培养过夜。将过夜培养的种子液以1%的接种量转接到新鲜的含有氨苄青霉素的lb液体培养基中,37ºc、200r/min培养至od600达到0.6~0.8。随后在培养基中添加终浓度为0.4mm的异丙基-β-d-硫代半乳糖苷(iptg),30ºc继续培养6~10h。
1.4高丝氨酸乙酰转移酶的酶活测定
收集诱导表达的菌体,离心去除培养基,加入5ml50mmtris-hcl缓冲液重悬,使用超声波细胞破碎仪破碎细胞,设定200w功率,超声2s间歇1s,超声10min;随后在高速冷冻离心机上8000×g离心10min,收集上清用于酶活测定。酶活测定体系为:50mmtris-hcl(ph7.0),2mm高丝氨酸(homoserine),0.12mmacetyl-coa,1mm5,5'-二硫代双(2-硝基苯甲酸)dtnb,适当稀释的粗酶液。以不加homoserine样品为空白对照,室温下使用酶标仪测定412nm的吸光值。由于dtnb不稳定,见光易分解,需现配现用,并在测定时需重复制作coa标准曲线。标准曲线的测定体系为:50mmtris-hcl(ph7.0),2mmhomoserine,0~0.1mmcoa,1mmdtnb。metx的酶活单位定义为:单位时间内(min)生成1μmolcoa所需的酶量。
经酶标仪检测,来源于leptospirameyeri的高丝氨酸乙酰转移酶活性为0.85±0.21u/mgprotein,并受到oah高丝氨酸强烈的反馈抑制,当10mmoah存在时,仅能保存23%的剩余酶活(0.19u/mg蛋白质)。
实施例2:采用易错pcr方法定向进化高丝氨酸乙酰转移酶
2.1高丝氨酸乙酰转移酶突变体文库的构建
本发明首先采用易错pcr的方法构建高丝氨酸乙酰转移酶突变体文库。本发明采用的易错pcr体系为:10xtaq缓冲液,5μl;25mmol/lmg2+,14μl;10mmol/lmn2+,0-5μl;10pmol引物1,2μl;10pmol引物2,2μl;10mmol/ldntp,4μl;50ngdna模板;takarartaqdna聚合酶,5u;加ddh2o补充至50μl。易错pcr程序为:95ºc,3min;95ºc,30s;58ºc,30s;72ºc,2min;循环35times,72ºc延伸5min。
将上述获得的pcr产物经1%的琼脂糖凝胶电泳检测后,采用胶回收试剂盒进行切胶回收。然后通过限制性内切酶ndei和xhoi酶切,酶切体系为:10μl10x缓冲液;5μlxhoi;5μlndei;500ng目的片段;加ddh2o补充至100μl。随后连接至用同样内切酶处理的pet21b表达载体上,构建高丝氨酸乙酰转移酶突变体质粒文库。然后将上述获得的质粒文库按照实施例1中的第1.2点所示方法转化至大肠杆菌bl21(de3)菌株中。
2.2高丝氨酸乙酰转移酶突变体文库的高通量筛选
将实施例2中的第2.1点获得的含有高丝氨酸乙酰转移酶突变体文库的大肠杆菌,挑取单克隆接种至已含有800μllb液体培养基的96深孔板中,37ºc、800r/min震荡培养。当菌体浓度od600达到0.6~0.8时,加入终浓度0.4mmiptg诱导剂,30ºc、800r/min继续震荡培养12h。4ºc5000´g离心10min,去除培养基上清。随后加入1ml已含有2mg/ml溶菌酶和0.05%tritonx-100的裂解缓冲液(20mmna2hpo4,200mmnacl,ph7.0),37ºc孵育2h,4ºc5000´g离心10min,收集上清用于酶活测定。
按照实施例1中的第1.4点所述的酶活测定方法,对上述获得的突变体文库进行酶活测定,从约1000个突变体中筛选获得三个酶活明显提升的候选突变体,即g118v、f147i和m182t。然后将三个含有候选突变体的菌株按照实施例1.3所示的方法,重新进行诱导表达和酶活验证。
结果表明,高丝氨酸乙酰转移酶突变体g118v、f147i和m182t的酶活性可达8.37±0.42、7.68±0.51和6.49±0.24u/mg蛋白质,分别是野生型高丝氨酸乙酰转移酶活性的9.7倍、8.9倍和7.5倍。然而,上述三个突变体仍然受到目标产物oah的较明显反馈抑制作用,在10mmoah存在时,三个突变体仅能保留20%~25%的剩余酶活。
实施例3:3å范围内底物通道氨基酸残基的定点饱和突变
本发明首先利用软件rosettacm,以来源于leptospirainterrogans的高丝氨酸乙酰转移酶为模板(pdbid:2pl5),通过同源建模的方法构建了leptospirameyeri的高丝氨酸乙酰转移酶的三维模型。随后利用rosetta分子对接方法,将coa对接至上述构建的高丝氨酸乙酰转移酶三维模型中。根据同源建模和分子对接结果分析,对底物通道3å范围内8个可变的氨基酸残基进行定点饱和突变,分别为:l236、m240、y267、e270、p348、a349、l356和p357。
本发明所采用定点饱和突变具体方法为:在待突变的氨基酸位点设计随机的兼并引物nnk(其中n代表a、c、g或t,k代表g或t),如下所示;
以含有密码子优化后高丝氨酸乙酰转移酶基因的pet21b质粒为模板,利用上述兼并引物,通过pcr扩增获得基因突变文库;随后采用基于goldengate克隆的片段装配方法连接成完整质粒文库。goldengate组装体系为:10xt4连接酶缓冲液,1.5μl;100xbsa,1.5μl;bsai内切酶,1μl;t4ligase,1μl;dna片段,100ng;补充ddh2o至15μl。goldengate组装程序为:37°c,3min;22°c,4min;循环30次;22°c,20min;50°c,2min;80°c,2min;16°c,5min。将goldengate反应液直接转化e.colibl21(de3)感受态,构建待突变氨基酸位点的饱和突变文库。为了保证文库的覆盖率,每个点的饱和突变均随机选择95个单克隆进行验证,将获得的单克隆接种至含有800μllb液体培养基的96深孔板中,37ºc、800r/min震荡培养。当菌体浓度od600达到0.6~0.8时,加入终浓度0.4mmiptg诱导剂,30ºc、800r/min继续震荡培养12h。4ºc5000xg离心10min,去除培养基上清。随后加入1ml已含有2mg/ml溶菌酶和0.05%tritonx-100的裂解缓冲液(20mmna2hpo4,200mmnacl,ph7.0),37ºc孵育2h,4ºc5000xg离心10min,收集上清用于酶活测定。
按照实施例1中的第1.4点所述的酶活测定方法,将上述获得的突变体文库进行酶活测定。从8个位点的饱和突变中筛选获得1个酶活性提高的候选突变体,即m240g。然后将该突变体菌株按照实施例1中的第1.3点所示的方法重新进行诱导表达和酶活验证。
结果表明,突变体m240g酶活性达到2.73±0.35u/mg蛋白质,是野生型的3.21倍。同时该突变体酶有较强的反馈抑制能力,在10mmoah存在时,仍能保留54.2%的剩余酶活(1.48u/mg蛋白质)。
实施例4:有益突变位点的组合突变进一步提高酶活性
4.1有益突变位点的组合突变
为了进一步提高酶的催化能力,将上述获得突变位点g118v、f147i、m182t和m240g进行组合突变。本发明所涉及的定点突变采用quikchange定点突变试剂盒(quikchangesite-directedmutagenesiskit)完成,所涉及的引物如下:
quikchange定点突变体系为:10´phusion缓冲液,5μl;10μmol引物1,2μl;10μmol引物2,2μl;10mmol/ldntp,4μl;50ngdna模板;phusionhigh-fidelitydna聚合酶,5u;补充ddh2o至50μl。pcr程序为:98ºc,30s;98ºc,10s;58ºc,30s;72ºc,5min;循环30次,72ºc延伸5min。
将上述获得的反应液转化至大肠杆菌bl21(de3)感受态中,具体转化方法如实施例1中的第1.2点所示。挑取单菌落进行测序验证,最终构建了6个双突变体组合(g118v-f147i、g118v-m182t、g118v-m240g、f147i-m182t、f147i-m240g和m182t-m240g),3个三突变体组合(g118v-f147i-m182t、f147i-m182t-m240g和g118v-m182t-m240g)以及1个四突变体组合(g118v-f147i-m182t-m240g)。
4.2高丝氨酸乙酰转移酶突变体诱导表达及酶活测定
将上述获得的含有高丝氨酸乙酰转移酶组合突变体的菌株接种于含有氨苄青霉素的lb液体培养基中,37ºc、200r/min培养过夜。将过夜培养的种子液以1%的接种量转接到新鲜的含有氨苄青霉素的lb液体培养基中,37ºc、200r/min培养至od600达到0.6~0.8。随后在培养基中添加终浓度为0.4mmiptg,30ºc继续培养6~8h。收集诱导表达的菌体,离心去除培养基,加入5ml50mmtris-hcl缓冲液重悬,使用超声波细胞破碎仪破碎细胞,设定200w功率,超声2s间歇1s,超声10min;随后在高速冷冻离心机上8000xg离心10min,收集上清用于酶活测定。
按照实施例1中的第1.4点所述的方法,进行酶活分析,测定结果如表1和图2所示。表中所列出的突变体均表现相对于贤野生型来说有提高的酶活性(至少是约9.4倍),其中高丝氨酸乙酰转移酶突变体f147i-m182t-m240g表现出最高的酶催化能力,其酶活性为12.96±0.19u/mg蛋白质,是野生型的15.25倍。所列出的突变体均表现增加的抗反馈抑制能力,其中仍以高丝氨酸乙酰转移酶突变体f147i-m182t-m240g表现最优,在10mmoah存在时,突变体能保留42.7%的剩余酶活,酶性活仍达5.54±0.23u/mg蛋白质。其中本实施例中,g118v-f147i-m182t突变的编码核苷酸序列如seqidno.3所示,g118v-f147i-m240g如seqidno.4所示,f147i-m182t-m240g如seqidno.5所示,g118v-f147i-m182t-m240g如seqidno.6所示。
表1高丝氨酸乙酰转移酶有益突变位点的组合突变体的酶活
因此,所述的高丝氨酸乙酰转移酶突变体具有明显优于野生型的催化活性,具有良好的应用前景。
实施例5:构建工程菌株发酵生产o-乙酰高丝氨酸
基于实施例4获得的高丝氨酸乙酰转移酶组合突变体,构建微生物细胞工厂用于发酵生产o-乙酰高丝氨酸或甲硫氨酸。以野生型大肠杆菌w3110或谷氨酸棒杆菌atcc13032为出发背景菌株,采用强启动子对最优高丝氨酸乙酰转移酶组合突变体f147i-m182t-m240g进行优化表达。利用引物对(metx-f:5’-ctagaagcttatgccaacctccgaacagaac-3’和metx-r:5’-ctagggtaccttacaggaacacgccttcatc-3’),扩增获得f147i-m182t-m240g突变体编码基因,经过hindiii和kpni双酶切后,亚克隆至表达载体pxmj19(大肠杆菌-谷氨酸棒杆菌穿梭表达载体),获得重组表达质粒pxmj19-f147i-m182t-m240g。随后,将对照表达质粒pxmj19和重组表达质粒pxmj19-f147i-m182t-m240g,转入野生型大肠杆菌w3110中,分别获得菌株ec-pxmj19和ec-pxmjmut;将对照表达质粒pxmj19和重组表达质粒pxmj19-f147i-m182t-m240g,转入野生型谷氨酸棒杆菌atcc13032中,分别获得菌株cg-pxmj19和cg-pxmjmut.
将上述获得的菌株接种于lb培养基中,过夜培养后,接种于含有50ml新鲜的种子培养基(5g/l酵母粉,5g/l蛋白胨,15g/l葡萄糖,尿素0.1g/l,玉米浆15g/l,硫酸铵15g/l,ph为6.5)的500ml三角瓶中,培养16h至对数中期。按照10%接种量接种至新鲜发酵培养基中(葡萄糖50g/l,玉米浆20g/l,(nh4)2so420g/l,kh2po41g/l,k2hpo41g/l,mgso4·7h2o0.01g/l,mnso4·h2o0.01g/l,feso4·7h2o0.01g/l,柠檬酸钠0.5g/l,维生素b12mg/l,生物素0.1mg/l),进行摇瓶发酵。
菌株发酵培养60h后,取样使用高效液相色谱测定发酵液中o-乙酰高丝氨酸含量。结果如表2所示。
表2菌株生产o-乙酰高丝氨酸的发酵分析
从结果可知,在大肠杆菌或谷氨酸棒杆菌中过表达高丝氨酸乙酰转移酶突变体f147i-m182t-m240g后,相较于原始出发菌而言,菌株oah发酵生产能力分别提升13.2倍和10.8倍。因此,构建酶活性和抗反馈抑制能力提高的高丝氨酸乙酰转移酶突变体,对于大肠杆菌或谷氨酸棒杆菌等底盘菌株高效生产o-乙酰高丝氨酸具有重要的应用价值。
序列表
<110>中国科学院天津工业生物技术研究所
<120>高丝氨酸乙酰转移酶突变体及其在生产o-乙酰高丝氨酸中的应用
<160>32
<170>siposequencelisting1.0
<210>1
<211>1137
<212>dna
<213>leptospirameyeri
<400>1
atgccaacctccgaacagaacgaattctcccacggctctgtcggcgtcgtgtacacccaa60
tccatccgcttcgaatccctcaccctggaaggtggcgagaccatcacccctctggaaatt120
gcctacgagacctacggcaccctgaacgagaagaaagacaacgccatcctggtctgccat180
gccctgtccggtgacgcccacgcagcaggcttccacgaaggtgacaagcgccctggctgg240
tgggattactacatcggcccaggcaagtcttttgacaccaaccgctacttcatcatctcc300
tccaacgtgatcggcggttgcaaaggttcctccggcccactcaccatcaacggcaagaac360
ggtaagccatttcagtccaccttcccattcgtgtccatcggtgatatggtcaacgcccag420
gagaagctgatctcccatttcggcatccacaagctcttcgcagtggccggtggttctatg480
ggcggcatgcaggccctgcaatggtccgtcgcctaccctgaccgcctcaagaactgcatt540
gtgatggcctcctcctctgagcactccgcccagcagatcgcatttaacgaggtgggccgt600
caggccatcctgtccgaccctaactggaaccaaggcctgtatacccaggagaaccgccct660
tccaagggtctggcactggcccgtatgatgggccacatcacctacctgtccgacgagatg720
atgcgcgaaaaattcggccgcaagccaccaaagggcaacatccagtccaccgacttcgca780
gtgggctcctacctgatttaccagggcgagtccttcgtcgaccgcttcgatgcaaactcc840
tacatctacgtcaccaaggcactggaccacttctccctgggcaccggcaaggaactgacc900
aaggtgctcgccaaagtccgctgccgcttcctcgtggtcgcctatacctccgattggctg960
tacccaccataccagtccgaggagatcgtgaagtccctcgaagtgaacgcagtgcctgtc1020
tccttcgtggaactcaataacccagcaggccacgattctttcctgctgccatccgaacag1080
caggattccatcctgcgcgacttcctgtcctctactgatgaaggcgtgttcctgtaa1137
<210>2
<211>378
<212>dna
<213>leptospirameyeri
<400>2
mptseqnefshgsvgvvytqsirfesltleggetitpleiayetygtlnekkdnailvch60
alsgdahaagfhegdkrpgwwdyyigpgksfdtnryfiissnviggckgssgpltingkn120
gkpfqstfpfvsigdmvnaqeklishfgihklfavaggsmggmqalqwsvaypdrlknci180
vmasssehsaqqiafnevgrqailsdpnwnqglytqenrpskglalarmmghitylsdem240
mrekfgrkppkgniqstdfavgsyliyqgesfvdrfdansyiyvtkaldhfslgtgkelt300
kvlakvrcrflvvaytsdwlyppyqseeivkslevnavpvsfvelnnpaghdsfllpseq360
qdsilrdflsstdegvfl378
<210>3
<211>1137
<212>dna
<213>人工序列
<400>3
atgccaacctccgaacagaacgaattctcccacggctctgtcggcgtcgtgtacacccaa60
tccatccgcttcgaatccctcaccctggaaggtggcgagaccatcacccctctggaaatt120
gcctacgagacctacggcaccctgaacgagaagaaagacaacgccatcctggtctgccat180
gccctgtccggtgacgcccacgcagcaggcttccacgaaggtgacaagcgccctggctgg240
tgggattactacatcggcccaggcaagtcttttgacaccaaccgctacttcatcatctcc300
tccaacgtgatcggcggttgcaaaggttcctccggcccactcaccatcaacgtcaagaac360
ggtaagccatttcagtccaccttcccattcgtgtccatcggtgatatggtcaacgcccag420
gagaagctgatctcccatatcggcatccacaagctcttcgcagtggccggtggttctatg480
ggcggcatgcaggccctgcaatggtccgtcgcctaccctgaccgcctcaagaactgcatt540
gtgacggcctcctcctctgagcactccgcccagcagatcgcatttaacgaggtgggccgt600
caggccatcctgtccgaccctaactggaaccaaggcctgtatacccaggagaaccgccct660
tccaagggtctggcactggcccgtatgatgggccacatcacctacctgtccgacgagatg720
atgcgcgaaaaattcggccgcaagccaccaaagggcaacatccagtccaccgacttcgca780
gtgggctcctacctgatttaccagggcgagtccttcgtcgaccgcttcgatgcaaactcc840
tacatctacgtcaccaaggcactggaccacttctccctgggcaccggcaaggaactgacc900
aaggtgctcgccaaagtccgctgccgcttcctcgtggtcgcctatacctccgattggctg960
tacccaccataccagtccgaggagatcgtgaagtccctcgaagtgaacgcagtgcctgtc1020
tccttcgtggaactcaataacccagcaggccacgattctttcctgctgccatccgaacag1080
caggattccatcctgcgcgacttcctgtcctctactgatgaaggcgtgttcctgtaa1137
<210>4
<211>1137
<212>dna
<213>人工序列
<400>4
atgccaacctccgaacagaacgaattctcccacggctctgtcggcgtcgtgtacacccaa60
tccatccgcttcgaatccctcaccctggaaggtggcgagaccatcacccctctggaaatt120
gcctacgagacctacggcaccctgaacgagaagaaagacaacgccatcctggtctgccat180
gccctgtccggtgacgcccacgcagcaggcttccacgaaggtgacaagcgccctggctgg240
tgggattactacatcggcccaggcaagtcttttgacaccaaccgctacttcatcatctcc300
tccaacgtgatcggcggttgcaaaggttcctccggcccactcaccatcaacgtcaagaac360
ggtaagccatttcagtccaccttcccattcgtgtccatcggtgatatggtcaacgcccag420
gagaagctgatctcccatatcggcatccacaagctcttcgcagtggccggtggttctatg480
ggcggcatgcaggccctgcaatggtccgtcgcctaccctgaccgcctcaagaactgcatt540
gtgatggcctcctcctctgagcactccgcccagcagatcgcatttaacgaggtgggccgt600
caggccatcctgtccgaccctaactggaaccaaggcctgtatacccaggagaaccgccct660
tccaagggtctggcactggcccgtatgatgggccacatcacctacctgtccgacgaggga720
atgcgcgaaaaattcggccgcaagccaccaaagggcaacatccagtccaccgacttcgca780
gtgggctcctacctgatttaccagggcgagtccttcgtcgaccgcttcgatgcaaactcc840
tacatctacgtcaccaaggcactggaccacttctccctgggcaccggcaaggaactgacc900
aaggtgctcgccaaagtccgctgccgcttcctcgtggtcgcctatacctccgattggctg960
tacccaccataccagtccgaggagatcgtgaagtccctcgaagtgaacgcagtgcctgtc1020
tccttcgtggaactcaataacccagcaggccacgattctttcctgctgccatccgaacag1080
caggattccatcctgcgcgacttcctgtcctctactgatgaaggcgtgttcctgtaa1137
<210>5
<211>1137
<212>dna
<213>人工序列
<400>5
atgccaacctccgaacagaacgaattctcccacggctctgtcggcgtcgtgtacacccaa60
tccatccgcttcgaatccctcaccctggaaggtggcgagaccatcacccctctggaaatt120
gcctacgagacctacggcaccctgaacgagaagaaagacaacgccatcctggtctgccat180
gccctgtccggtgacgcccacgcagcaggcttccacgaaggtgacaagcgccctggctgg240
tgggattactacatcggcccaggcaagtcttttgacaccaaccgctacttcatcatctcc300
tccaacgtgatcggcggttgcaaaggttcctccggcccactcaccatcaacggcaagaac360
ggtaagccatttcagtccaccttcccattcgtgtccatcggtgatatggtcaacgcccag420
gagaagctgatctcccatatcggcatccacaagctcttcgcagtggccggtggttctatg480
ggcggcatgcaggccctgcaatggtccgtcgcctaccctgaccgcctcaagaactgcatt540
gtgacggcctcctcctctgagcactccgcccagcagatcgcatttaacgaggtgggccgt600
caggccatcctgtccgaccctaactggaaccaaggcctgtatacccaggagaaccgccct660
tccaagggtctggcactggcccgtatgatgggccacatcacctacctgtccgacgaggga720
atgcgcgaaaaattcggccgcaagccaccaaagggcaacatccagtccaccgacttcgca780
gtgggctcctacctgatttaccagggcgagtccttcgtcgaccgcttcgatgcaaactcc840
tacatctacgtcaccaaggcactggaccacttctccctgggcaccggcaaggaactgacc900
aaggtgctcgccaaagtccgctgccgcttcctcgtggtcgcctatacctccgattggctg960
tacccaccataccagtccgaggagatcgtgaagtccctcgaagtgaacgcagtgcctgtc1020
tccttcgtggaactcaataacccagcaggccacgattctttcctgctgccatccgaacag1080
caggattccatcctgcgcgacttcctgtcctctactgatgaaggcgtgttcctgtaa1137
<210>6
<211>1137
<212>dna
<213>人工序列
<400>6
atgccaacctccgaacagaacgaattctcccacggctctgtcggcgtcgtgtacacccaa60
tccatccgcttcgaatccctcaccctggaaggtggcgagaccatcacccctctggaaatt120
gcctacgagacctacggcaccctgaacgagaagaaagacaacgccatcctggtctgccat180
gccctgtccggtgacgcccacgcagcaggcttccacgaaggtgacaagcgccctggctgg240
tgggattactacatcggcccaggcaagtcttttgacaccaaccgctacttcatcatctcc300
tccaacgtgatcggcggttgcaaaggttcctccggcccactcaccatcaacgtcaagaac360
ggtaagccatttcagtccaccttcccattcgtgtccatcggtgatatggtcaacgcccag420
gagaagctgatctcccatatcggcatccacaagctcttcgcagtggccggtggttctatg480
ggcggcatgcaggccctgcaatggtccgtcgcctaccctgaccgcctcaagaactgcatt540
gtgacggcctcctcctctgagcactccgcccagcagatcgcatttaacgaggtgggccgt600
caggccatcctgtccgaccctaactggaaccaaggcctgtatacccaggagaaccgccct660
tccaagggtctggcactggcccgtatgatgggccacatcacctacctgtccgacgaggga720
atgcgcgaaaaattcggccgcaagccaccaaagggcaacatccagtccaccgacttcgca780
gtgggctcctacctgatttaccagggcgagtccttcgtcgaccgcttcgatgcaaactcc840
tacatctacgtcaccaaggcactggaccacttctccctgggcaccggcaaggaactgacc900
aaggtgctcgccaaagtccgctgccgcttcctcgtggtcgcctatacctccgattggctg960
tacccaccataccagtccgaggagatcgtgaagtccctcgaagtgaacgcagtgcctgtc1020
tccttcgtggaactcaataacccagcaggccacgattctttcctgctgccatccgaacag1080
caggattccatcctgcgcgacttcctgtcctctactgatgaaggcgtgttcctgtaa1137
<210>7
<211>41
<212>dna
<213>人工序列
<223>n=a或c或g或t,k=g或t
<400>7
caccaggtctcaatcacctacnnktccgacgagatgatgcg41
<210>8
<211>35
<212>dna
<213>人工序列
<400>8
caccaggtctcatgatgtggcccatcatacgggcc35
<210>9
<211>44
<212>dna
<213>人工序列
<223>n=a或c或g或t,k=g或t
<400>9
caccaggtctcatccgacgagnnkatgcgcgaaaaattcggccg44
<210>10
<211>35
<212>dna
<213>人工序列
<400>10
caccaggtctcacggacaggtaggtgatgtggccc35
<210>11
<211>39
<212>dna
<213>人工序列
<223>n=a或c或g或t,k=g或t
<400>11
caccaggtctcatacctgattnnkcagggcgagtccttc39
<210>12
<211>35
<212>dna
<213>人工序列
<400>12
caccaggtctcaggtaggagcccactgcgaagtcg35
<210>13
<211>39
<212>dna
<213>人工序列
<223>n=a或c或g或t,k=g或t
<400>13
caccaggtctcataccagggcnnktccttcgtcgaccgc39
<210>14
<211>38
<212>dna
<213>人工序列
<400>14
caccaggtctcaggtaaatcaggtaggagcccactgcg38
<210>15
<211>41
<212>dna
<213>人工序列
<223>n=a或c或g或t,k=g或t
<400>15
caccaggtctcacaataacnnkgcaggccacgattctttcc41
<210>16
<211>37
<212>dna
<213>人工序列
<400>16
caccaggtctcaattgagttccacgaaggagacaggc37
<210>17
<211>41
<212>dna
<213>人工序列
<223>n=a或c或g或t,k=g或t
<400>17
caccaggtctcataacccannkggccacgattctttcctgc41
<210>18
<211>42
<212>dna
<213>人工序列
<400>18
caccaggtctcagttattgagttccacgaaggagacaggcac42
<210>19
<211>37
<212>dna
<213>人工序列
<223>n=a或c或g或t,k=g或t
<400>19
caccaggtctcatttcctgnnkccatccgaacagcag37
<210>20
<211>33
<212>dna
<213>人工序列
<400>20
caccaggtctcagaaagaatcgtggcctgctgg33
<210>21
<211>33
<212>dna
<213>人工序列
<223>n=a或c或g或t,k=g或t
<400>21
caccaggtctcatgctgnnktccgaacagcagg33
<210>22
<211>35
<212>dna
<213>人工序列
<400>22
caccaggtctcaagcaggaaagaatcgtggcctgc35
<210>23
<211>41
<212>dna
<213>人工序列
<400>23
tcaccatcaacgtcaagaacggtaagccatttcagtccacc41
<210>24
<211>37
<212>dna
<213>人工序列
<400>24
tggcttaccgttcttgacgttgatggtgagtgggccg37
<210>25
<211>37
<212>dna
<213>人工序列
<400>25
ctcccatatcggcatccacaagctcttcgcagtggcc37
<210>26
<211>40
<212>dna
<213>人工序列
<400>26
gaagagcttgtggatgccgatatgggagatcagcttctcc40
<210>27
<211>38
<212>dna
<213>人工序列
<400>27
cctcaagaactgcattgtgacggcctcctcctctgagc38
<210>28
<211>37
<212>dna
<213>人工序列
<400>28
ggaggccgtcacaatgcagttcttgaggcggtcaggg37
<210>29
<211>28
<212>dna
<213>人工序列
<400>29
acgagggaatgcgcgaaaaattcggccg28
<210>30
<211>28
<212>dna
<213>人工序列
<400>30
cggccgaatttttcgcgcattccctcgt28
<210>31
<211>31
<212>dna
<213>人工序列
<400>31
ctagaagcttatgccaacctccgaacagaac31
<210>32
<211>31
<212>dna
<213>人工序列
<400>32
ctagggtaccttacaggaacacgccttcatc31
- 还没有人留言评论。精彩留言会获得点赞!