用于分离生物体内特异性蛋白-DNA复合体的方法、融合蛋白及其制备方法
用于分离生物体内特异性蛋白
‑
dna复合体的方法、融合蛋白及其制备方法
技术领域
1.本发明涉及生物工程技术领域,特别涉及一种用于分离生物体内特异性蛋白
‑
dna复合体的方法、融合蛋白及其制备方法。
背景技术:
2.染色质免疫共沉淀技术(chromatin immunoprecipitation assay,chip)一直是体内研究蛋白质
‑
dna相互作用的主要方法,其过程包括细胞与甲醛交联固定、染色质片段化、引入抗体富集、收集与目标蛋白结合的染色质进行分析,最终获得蛋白质在基因组上的结合位点。chip 技术也存在一定的局限性,例如需要高度特异性抗体,交联过程中或者无效抗体造成的假阴性信号,甲醛固定过程中的假阳性信号等。
3.近些年来,基因组技术的创新与进步为人们更好地了解蛋白质在染色质上的分布提供了新的工具。比如核酸酶靶向切割和释放(cut&run)技术,其主要通过靶标特异性一抗和pag
‑
微球菌核酸酶(pag
‑
mnase)分离体内特异性蛋白
‑
dna复合体,利用ca
2+
控制酶的活性,切割下来的dna片段建库测序,从而获得蛋白特异结合的dna片段,但此方法需要高特异性的目标蛋白一抗。
技术实现要素:
4.本发明的目的之一是提供一种能够用于分离生物体内特异性蛋白
‑
dna复合体的融合蛋白的制备方法,采用该方法制备的融合蛋白分离蛋白
‑
dna复合体,可以简化操作步骤且不需要用到抗原特异性抗体。
5.为了实现上述目的,本发明采用如下技术方案:用于分离生物体内特异性蛋白
‑
dna复合体的融合蛋白的制备方法,包括以下步骤:一、纳米抗体核酸酶融合蛋白表达载体的构建:将与目的蛋白具有相同标签的纳米抗体构建于合适的载体上,获取微球菌核酸酶的dna序列,将所述微球菌核酸酶的dna序列插入载体中纳米抗体序列的前面或后面,构建纳米抗体微球核酸酶融合蛋白表达载体;二、纳米抗体核酸酶融合蛋白的纯化:将步骤一中构建的纳米抗体微球核酸酶融合蛋白表达载体于合适的表达系统中表达后,提取纳米抗体核酸酶融合蛋白并进行纯化处理。
6.在本发明的一个实施例中,所述纳米抗体为绿色荧光蛋白纳米抗体。
7.在本发明的一个实施例中,上述步骤二中的表达系统为大肠杆菌表达系统。
8.在本发明的一个实施例中,步骤一中,先将绿色荧光蛋白纳米抗体构建在padl
‑
10b载体上, 得到padl
‑
10b
‑
nanobody质粒,之后通过无缝克隆将微球菌核酸酶的dna序列插入padl
‑
10b
‑
nanobody载体中纳米抗体序列的后面,并通过pcr扩增绿色荧光蛋白纳米抗体
‑
微球核酸酶融合基因序列,之后再次采用无缝克隆将绿色荧光蛋白纳米抗体
‑
微球核酸
酶融合基因序列构建到pet28a质粒上,从而得到绿色荧光蛋白纳米抗体
‑
微球核酸酶融合蛋白表达载体。
9.其中,在步骤一中,是从密码子优化后的微球菌核酸酶dna片段中克隆获取微球核酸酶的dna序列,用以进行pcr扩增的引物为:padl
‑
10
‑
yj
‑
homo
‑
fp:ccgggaggccaaggcggtggttctgagggtggtggctccggtggaggggcaacttcaac ;padl
‑
10
‑
yj
‑
homo
‑
rp:atttaacaaaaatttaacgcgaattttaacaaaatattattagtgatgatgatgatgatg) 。
10.进一步地,在前述实施例中,采用pcr扩增绿色荧光蛋白纳米抗体
‑
微球核酸酶融合基因序列时,用以进行pcr扩增的引物为:pet28a
‑
yj
‑
homo
‑
fp:catatggctagcatgactggtggacagcaaatgggtcgcatggatcaagtccaactggt;pet28a
‑
yj
‑
homo
‑
rp:gttagcagccggatctcagtggtggtggtggtggtgttattgacctgaatcagcgttgt 。
11.另外,本发明还涉及一种用于分离生物体内特异性蛋白
‑
dna复合体的融合蛋白,其是采用上面所述的方法制备得到。
12.基于同样的发明构思,本发明还涉及一种用于分离生物体内特异性蛋白
‑
dna复合体的方法,该方法是利用标签的纳米抗体与目的蛋白中带有的相同标签结合,将微球菌核酸酶带到目的蛋白与染色质结合位点处,通过核酸酶的激活使染色质裂解,释放出蛋白
‑
dna复合体。
13.进一步地,是通过将与目的蛋白具有相同标签的纳米抗体与微球菌核酸酶融合形成融合蛋白并将融合蛋白与目的蛋白结合,以此利用标签的纳米抗体与目的蛋白中带有的相同标签结合,将微球菌核酸酶带到目的蛋白与染色质结合位点处,通过核酸酶的激活使染色质裂解,释放出蛋白
‑
dna复合体。
14.其中,可以通过前面所述的制备方法将与目的蛋白具有相同标签的纳米抗体与微球菌核酸酶融合形成融合蛋白。
15.本发明首次提出将纳米抗体和微球菌核酸酶形成融合蛋白,融合蛋白与目的蛋白结合时,利用标签的纳米抗体与目的蛋白中所带有的相同标签结合,将微球菌核酸酶带到目的蛋白与染色质结合位点处,通过核酸酶的激活促使染色质裂解,就可释放出蛋白
‑
dna复合体。上述纳米抗体和微菌球核酸酶形成的融合蛋白可应用于蛋白质
‑
dna相互作用的研究中,在研究蛋白质特异结合dna序列时,不需要目标蛋白特异性一抗且操作步骤简单,为含有相同标签的不同蛋白质寻找生物体内染色质结合序列提供新的技术手段。
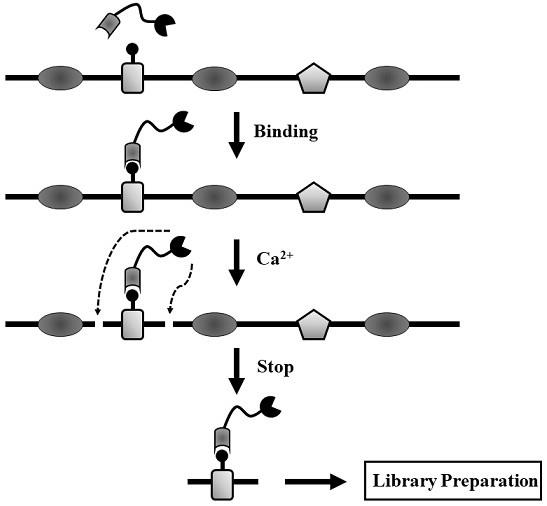
16.附图说明:图1为纳米抗体和微菌球核酸酶形成的融合蛋白分离生物体内特异性蛋白
‑
dna复合体的原理图。
17.图2为pet28a
‑
nanogm载体示意图。
18.图3中,a为除盐后提纯nanogm聚丙烯酰胺凝胶电泳图,b
‑
d为nanogm应用可行性体外验证结果。
具体实施方式
19.本发明的关键之处在于利用标签的纳米抗体与目的蛋白中带有的相同标签结合,将微球菌核酸酶带到目的蛋白与染色质结合位点处,通过核酸酶的激活促使染色质裂解,释放出蛋白
‑
dna复合体,不需要额外提供抗原特异性的抗体,之后消化复合体中蛋白即可对dna进行测序,确定结合位点,操作步骤更为简单。
20.为了便于本领域技术人员的理解,下面以绿色荧光蛋白纳米抗体
‑
微球菌核酸酶融合蛋白(nanogm)为例,结合附图对本发明作进一步的说明,实施例提及的内容并非对本发明的限定。
21.需要提前说明的是,本实施例中所用到的大都为常规实验材料,都是通用型的产品,可以通过市场途径外购获得,为简化表述,不对其生产厂商作详细说明,应当指出的是,以下实施例中所用到的微球菌核酸酶dna片段是先将微球菌核酸酶的氨基酸序列转化为优化密码子的核苷酸序列,再发给外部合作公司合成。
22.一、nanogm蛋白表达载体的构建:1.1从基因库中获得微球菌核酸酶的氨基酸序列,利用在线工具“密码子优化工具”,将其氨基酸序列转化为优化密码子的核苷酸序列,在本实施例中,密码子优化后的微球菌核酸酶dna片段由ige生物技术有限公司合成。
23.1.2将绿色荧光蛋白纳米抗体构建在padl
‑
10b载体上, 得到padl
‑
10b
‑
nanobody质粒。
24.1.3绿色荧光蛋白纳米抗体
‑
微球核酸酶融合蛋白表达质粒pet28a
‑
nanogm克隆过程如下:设计引物(padl
‑
10
‑
yj
‑
homo
‑
fp/ padl
‑
10
‑
yj
‑
homo
‑
rp),利用pcr扩增出微球核酸酶的dna序列;采用无缝克隆的方法插入将微球核酸酶的dna序列插入质粒padl
‑
10b
‑
nanobody中绿色荧光蛋白纳米抗体序列的后面,构建padl
‑
10b
‑
nanogm质粒。随后,设计引物(pet28a
‑
yj
‑
homo
‑
fp/ pet28a
‑
yj
‑
homo
‑
rp),利用pcr技术扩增nanogm序列,再次利用无缝克隆技术,将nanogm构建在pet28a质粒上,得到pet28a
‑
nanogm绿色荧光蛋白纳米抗体
‑
微球核酸酶融合蛋白表达载体。pcr引物如下:padl
‑
10
‑
yj
‑
homo
‑
fp:ccgggaggccaaggcggtggttctgagggtggtggctccggtggaggggcaacttcaac ;padl
‑
10
‑
yj
‑
homo
‑
rp:atttaacaaaaatttaacgcgaattttaacaaaatattattagtgatgatgatgatgatg) 。
25.pet28a
‑
yj
‑
homo
‑
fp:catatggctagcatgactggtggacagcaaatgggtcgcatggatcaagtccaactggt;pet28a
‑
yj
‑
homo
‑
rp:gttagcagccggatctcagtggtggtggtggtggtgttattgacctgaatcagcgttgt 。
26.二、nanogm蛋白的表达和纯化:2.1 pet28a
‑
nanogm质粒转化大肠杆菌bl21(de3)菌株。
26.2.2经过重组蛋白小规模诱导条件筛选,iptg的最佳浓度为0.5 mm,诱导温度为37℃。
27.2.3根据小规模诱导条件扩大诱导规模,从250 ml lb培养基(tryptone 10 g,
yeast extract 5 g,nacl 10 g, 1 l ddh2o)中提取融合蛋白。
28.2.4 8000 g离心10 min,弃上清,收集细菌。
29.2.5 用50 ml裂解缓冲液(ph8.0, 50 mm na2hpo4, 0.3 m nacl)重悬沉淀。
30.2.6超声波将得到的混合物在冰上破碎,直到溶液澄清。
31.2.7超声破碎后,4 ℃,13000 rmp离心15 min,收集上清置冰上。
32.2.8将1 ml ni
‑
nta琼脂糖珠(qiagen,1018244)吸至50ml混合物中,室温孵育1小时。
33.2.9用裂解缓冲液清洗纯化柱,调整流速。
34.2.10将孵育后的蛋白混合物转移到纯化柱中,依次用3个柱体积的洗涤缓冲液i (ph8.0, 50 mm na2hpo4, 0.5 m nacl)和ii (ph8.0, 50 mm na2hpo4, 0.5 m nacl, 10 mm咪唑)洗涤琼脂糖珠。
35.2.11用3 ml洗脱缓冲液(ph7.5, 20 mm tris, 100 mm nacl, 250 mm咪唑)洗脱nanogm融合蛋白。
36.2.12利用除盐柱将提纯的nanogm融合蛋白储存在10 mm tris
‑
hcl缓冲液中,nanogm融合蛋白基因序列如seq id no.1所示。
37.三、 nanogm应用可行性体外验证:3.1上述纯化的nanogm融合蛋白聚丙烯酰胺凝胶电泳结果如图3中a所示:第1
‑
5泳道为his纯化的不同洗脱顺序的蛋白质样品。
38.3.2 nanogm蛋白与绿色荧光蛋白体外结合实验结果如图3中b所示,证明nanogm可以很好的结合gfp。
39.3.3图3中c显示nanogm可以有效地切割dna,证明了nanogm的核酸酶活性;3.4图3中d表明nanogm切割dna的活性可以被ca
2+
有效控制。
40.四、nanogm在蛋白质
‑
dna相互作用研究中的应用:4.1果蝇胚胎收集:(1)本实验以含有gfp(绿色荧光蛋白)的果蝇株为材料,将果蝇于25 ℃培养箱中培养。
41.(2)收集隔夜(8
‑
12 h)的胚胎,用胚胎洗脱液(0.7% nacl, 0.04% triton
‑
x 100)将胚胎从琼脂板上洗下来。
42.(3)利用50%新鲜漂白水去除胚胎外壳约1 min,随后胚胎洗脱液洗2次。
43.(4)准备好的胚胎保存在
‑
80℃,备用。
44.4.2胚胎细胞核的提取:(1)将30 ul上述处理好的胚胎重悬于500 ul缓冲液b中(ph7.5, 15 mm tris
‑
hcl, 15 mm nacl, 60 mm kcl, 0.34 m蔗糖,0.5 mm亚精胺,0.1% β
‑
巯基乙醇,0.25 mm pmsf, 2 mm edta, 0.5 mm egta),用研磨杵研磨胚胎。
45.(2)将研磨后的混合物转移至1.5 ml ep管中, 4 ℃,5000 g离心5 min ,弃上清。
46.(3)用500 ul缓冲液a (ph7.5, 15 mm tris
‑
hcl, 15 mm nacl, 60 mm kcl, 0.34 m蔗糖,0.5 mm亚精胺,0.1% β
‑
巯基乙醇,0.25 mm pmsf)重悬,4 ℃ ,5000 g离心5 min。
47.(4)利用缓冲液a洗细胞核两次。
48.(5)细胞核重悬于600 ul的wbsed溶液中(0.5 mm spermidine,0.05% digitonin,1片small roche complete edta
‑
free protease inhibitor;wbe至12 ml),wbe(20 mm hepes ph7.5, 150 mm nacl, 0.1% bsa, 2 mm edta)。
49.4.3细胞核与磁珠孵育:(1)吸取20 ul(每个样品)的bio
‑
mag plus concanavalin a beads(polysciences, inc. #86057),转移到800 ul的结合缓冲液(20 mm hepes ph7.9,10 mm kcl,1 mm cacl2,1 mm mncl2)中。
50.(2)将ep管放置在磁力架上,用1 ml结合缓冲液清洗两次。
51.(3)加入300 ul结合缓冲液重悬磁珠。
52.(4)轻轻涡旋上述处理好的细胞核,缓缓加入含有磁珠的ep管中。
53.(5)室温下,旋转孵育5
‑
10 min。
54.4.4细胞核的封闭:(1)将ep管放置在磁力架上,静置60s后移走液体部分。
55.(2)500 ul wbsed溶液清洗细胞核一次。
56.(3)加入1 ml wbsed溶液,用移液枪轻轻混匀,在室温孵育5 min。
57.4.5. nanogm的孵育结合:(1)将ep管置于磁力架上,静置60 s后移走液体部分。
58.(2)将细胞核样品重悬于含0.6 ul nanogm的wbsed中。
59.(3)置于4 ℃中旋转孵育2 h。
60.(4)离心(不超过100 g),1 min,用1 ml wbsed洗涤两次。
61.4.6 nanogm消化dna:(1)细胞核重悬于150 ul wbc缓冲液(20 mm hepes ph 7.5,150 mm nacl,0.1% bsa,12 mm cacl2)中,4 ℃孵育2min。
62.(2)加入200 ul 2
ꢀ×ꢀ
stop溶液(340 mm nacl,20 mm edta,4 mm egta,50
ꢀµ
g/ml rnase)停止消化。
63.(3)4 ℃,16000 g离心5 min,将上清液转移到新ep管中。
64.4.7 dna提取:(1)将样品置于37 ℃水浴锅中孵育30 min。
65.(2)每个样品中加入2 ul的10% sds和2.5 ul的20 mg/ml蛋白酶k,颠倒混匀。
66.(3)70 ℃金属浴中孵育10 min。
67.(4)每个样品中加300 ul 的dna提取液(酚:氯仿:异戊醇=25:24:1),涡旋混合。
68.(5)将每个样品转移到一个准备好的phase lock管中,以13000 rpm 离心10 min。
69.(6)每个样品加300 ul氯仿,颠倒混匀。
70.(7)将上清转移到新的ep管中,在每个管中加入50 ul的ampure xp beads。
71.(8)将样品放置在磁力架上,静置2 min,随后将上清液转移到新的ep管中。
72.(9)每管加入1 ml100%乙醇,轻轻混合,
‑
20 ℃过夜。
73.(10)4 ℃,13000 rpm 离心10 min,用20 ul ddh2o溶解dna。
74.(11)最后,将dna样品送往测序公司建库测序即可。
75.上述实施例中采用了不同于核酸酶靶向切割和释放(cut&run)技术的手段,通过
将纳米抗体和微球菌核酸酶形成融合蛋白,融合蛋白与目的蛋白结合时,利用标签的纳米抗体与目的蛋白中所带有的相同标签结合,将微球菌核酸酶带到目的蛋白与染色质结合位点处,通过核酸酶的激活促使染色质裂解,就可释放出蛋白
‑
dna复合体。同时,在上述实施例中,以nanogm为例,给出了其在蛋白质
‑
dna相互作用研究中的应用方式,在研究蛋白质特异结合dna序列时,不需要额外提供抗原特异性的抗体且步骤简单,为含有相同标签的不同蛋白质寻找生物体内染色质结合序列提供了新的技术手段。
76.上述实施例为本发明较佳的实现方案,除此之外,本发明还可以其它方式实现,在不脱离本技术方案构思的前提下任何显而易见的替换均在本发明的保护范围之内。最后,应该强调的是,为了让本领域普通技术人员更方便地理解本发明相对于现有技术的改进之处,本发明的一些描述已经被简化,并且为了清楚起见,本申请文件还省略了一些其它元素,本领域普通技术人员应该意识到这些省略的元素也可构成本发明的内容。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1