玉米穗腐病抗性显著相关SNP分子标记组合、及其筛选方法和应用与流程
本发明涉及数量遗传学及玉米育种,特别涉及玉米穗腐病性状,具体是指一种玉米穗腐病抗性显著相关snp分子标记组合、及其筛选方法和应用。
背景技术:
1、玉米穗腐病是由多种病原菌引起的、世界玉米种植区普遍发生的一种真菌性病害。fusarium graminearum是引起我国玉米穗腐病发生的主要致病菌之一,病原菌侵染果穗后,不仅可以造成玉米产量的损失和商业品质的下降,而且还会产生脱氧雪腐镰孢菌烯醇和玉米赤霉烯酮等多种毒素,影响食品与饲料安全,直接威胁到人畜健康。
2、由于致病菌遗传多样性丰富,可以适应多种环境和不同杀菌剂,导致通过栽培管理和化学手段防控效果不理想,使用抗病品种是降低玉米穗腐病发生的有效手段。通过鉴定和克隆抗病基因有助于抗病品种的选育,但大量研究表明,玉米穗腐病抗性属复杂数量性状,由微效多基因控制,易受环境影响。利用传统育种方法和分子标记辅助选择对该性状进行遗传改良选择效率较低、效果较差。
3、全基因组选择是随着现代测序技术发展而来的、一种利用覆盖全基因组高密度分子标记进行预测的育种方法,该方法主要是通过已知表型和基因型的训练群体,构建统计模型,预测仅已知基因型的育种群体的育种值。由于是利用全基因组的分子标记进行选择,可以估算出所有的遗传效应,解释全部的遗传变异,即便是分子标记的效应微效,也能估算出来,因此全基因组选择比较适合用于由微效多基因控制的复杂数量性状的遗传改良。
4、因此,希望提供一种玉米穗腐病抗性显著相关snp分子标记组合,其能够用于构建玉米穗腐病抗性全基因组选择数据模型,从而显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本。
技术实现思路
1、为了克服上述现有技术中的缺点,本发明的一个目的在于提供一种玉米穗腐病抗性显著相关snp分子标记组合,其能够用于构建玉米穗腐病抗性全基因组选择数据模型,从而显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
2、本发明的另一目的在于提供一种玉米穗腐病抗性显著相关snp分子标记组合的筛选方法,其设计巧妙,操作简便,适于大规模推广应用。
3、本发明的另一目的在于提供一种玉米穗腐病抗性显著相关snp分子标记组合在构建玉米穗腐病抗性全基因组选择数据模型中的应用,由此构建的构建玉米穗腐病抗性全基因组选择数据模型能够显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
4、本发明的另一目的在于提供一种玉米穗腐病抗性全基因组选择数据模型构建方法,其设计巧妙,操作简便,由此构建的构建玉米穗腐病抗性全基因组选择数据模型能够显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
5、本发明的另一目的在于提供一种玉米穗腐病抗性全基因组选择数据模型,其能够显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
6、本发明的另一目的在于提供一种玉米穗腐病抗性全基因组选择数据模型在预测玉米穗腐病抗性中的应用,从而能够显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
7、为达到以上目的,在本发明的第一方面,提供一种玉米穗腐病抗性显著相关snp分子标记组合,其特点是,所述玉米穗腐病抗性显著相关snp分子标记组合包括snp1~snp69,其中:
8、所述snp1、所述snp2、所述snp3、所述snp4、所述snp5、所述snp6、所述snp7、所述snp8、所述snp9、所述snp10、所述snp11、所述snp12和所述snp13分别位于玉米的第1染色体的第43645851位碱基、第49802535位碱基、第59419876位碱基、第69429811位碱基、第81312482位碱基、第163156463位碱基、第183882633位碱基、第190231452位碱基、第197499264位碱基、第199720627位碱基、第199916785位碱基、第213511580位碱基和第278099674位碱基,所述第43645851位碱基为a或g,所述第49802535位碱基为a或g,所述第59419876位碱基为t或c,所述第69429811位碱基为t或g,所述第81312482位碱基为a或c,所述第163156463位碱基为a或g,所述第183882633位碱基为t或c,所述第190231452位碱基为t或c,所述第197499264位碱基为a或g,所述第199720627位碱基为a或g,所述第199916785位碱基为a或g,所述第213511580位碱基为t或c,所述第278099674位碱基为a或c;
9、所述snp14、所述snp15、所述snp16、所述snp17、所述snp18和所述snp19分别位于所述玉米的第2染色体的第6422884位碱基、第10149988位碱基、第40362374位碱基、第200079778位碱基、第206597745位碱基和第239867263位碱基,所述第6422884位碱基为t或g,所述第10149988位碱基为a或c,所述第40362374位碱基为c或g,所述第200079778位碱基为t或c,所述第206597745位碱基为t或c,所述第239867263位碱基为a或g;
10、所述snp20、所述snp21、所述snp22、所述snp23、所述snp24和所述snp25分别位于所述玉米的第3染色体的第629072位碱基、第16413990位碱基、第160126277位碱基、第185399398位碱基、第212721112位碱基和第225639028位碱基,所述第629072位碱基为t或c,所述第16413990位碱基为t或c,所述第160126277位碱基为t或c,所述第185399398位碱基为t或g,所述第212721112位碱基为a或g,所述第225639028位碱基为a或c;
11、所述snp26、所述snp27、所述snp28、所述snp29、所述snp30和所述snp31分别位于所述玉米的第4染色体的第80308919位碱基、第165573983位碱基、第166840292位碱基、第214745547位碱基、第216218588位碱基和第227982406位碱基,所述第80308919位碱基为a或c,所述第165573983位碱基为a或g,所述第166840292位碱基为t或c,所述第214745547位碱基为a或g,所述第216218588位碱基为a或g,所述第227982406位碱基为a或c;
12、所述snp32、所述snp33、所述snp34、所述snp35、所述snp36、所述snp37、所述snp38和所述snp39分别位于所述玉米的第5染色体的第2444523位碱基、第67326175位碱基、第118284375位碱基、第174142666位碱基、第184890420位碱基、第199518834位碱基、第221353359位碱基和第223581183位碱基,所述第2444523位碱基为t或c,所述第67326175位碱基为t或c,所述第118284375位碱基为t或g,所述第174142666位碱基为a或c,所述第184890420位碱基为a或g,所述第199518834位碱基为a或g,所述第221353359位碱基为t或c,所述第223581183位碱基为t或c;
13、所述snp40、所述snp41和所述snp42分别位于所述玉米的第6染色体的第94243988位碱基、第159543194位碱基和第163565455位碱基,所述第94243988位碱基为t或c,所述第159543194位碱基为a或g,所述第163565455位碱基为t或g;
14、所述snp43、所述snp44、所述snp45、所述snp46、所述snp47、所述snp48、所述snp49、所述snp50、所述snp51、所述snp52、所述snp53、所述snp54和所述snp55分别位于所述玉米的第7染色体的第9654032位碱基、第22231435位碱基、第23766644位碱基、第30366330位碱基、第41809741位碱基、第84902618位碱基、第103037600位碱基、第121214812位碱基、第位124925724碱基、第159951197位碱基、第162528814位碱基、第164524282位碱基和第180741651位碱基,所述第9654032位碱基为t或c,所述第22231435位碱基为t或c,所述第23766644位碱基为a或g,所述第30366330位碱基为t或c,所述第41809741位碱基为t或c,所述第84902618位碱基为t或g,所述第103037600位碱基为a或g,所述第121214812位碱基为t或c,所述第位124925724碱基为a或g,所述第159951197位碱基为t或g,所述第162528814位碱基为a或c,所述第164524282位碱基为t或g,所述第180741651位碱基为a或g;
15、所述snp56、所述snp57、所述snp58、所述snp59和所述snp60分别位于所述玉米的第8染色体的第13775624位碱基、第80844720位碱基、第104001857位碱基、第139410361位碱基和第176473798位碱基,所述第13775624位碱基为a或g,所述第80844720位碱基为a或g,所述第104001857位碱基为t或c,所述第139410361位碱基为a或g,所述第176473798位碱基为a或g;
16、所述snp61、所述snp62、所述snp63和所述snp64分别位于所述玉米的第9染色体的第13707254位碱基、第22155950位碱基、第23991241位碱基和第112271967位碱基,所述第13707254位碱基为t或c,所述第22155950位碱基为t或c,所述第23991241位碱基为a或c,所述第112271967位碱基为a或g;
17、所述snp65、所述snp66、所述snp67、所述snp68和所述snp69分别位于所述玉米的第10染色体的第10705997位碱基、第15723458位碱基、第122855713位碱基、第135749532位碱基和第137106570位碱基,所述第10705997位碱基为t或c,所述第15723458位碱基为t或c,所述第122855713位碱基为a或t,所述第135749532位碱基为t或c,所述第137106570位碱基为t或g;
18、所述snp1~所述snp69所在的染色体和位置均基于玉米自交系b73基因组v4版本确定。
19、在本发明的第二方面,提供一种上述的玉米穗腐病抗性显著相关snp分子标记组合的筛选方法,其特点是,包括以下步骤:
20、(1)田间试验
21、将多份玉米自交系材料在至少一个种植环境下种植,采用完全随机区组设计,每小区选取生长正常、吐丝期基本一致的玉米植株若干株,在吐丝10d后,接种fusariumgraminearum菌液,待玉米成熟后,调查果穗的发病情况;
22、(2)表型数据分析
23、表型数据分析采用r 4.0.2软件进行,描述性统计采用所述r 4.0.2软件的psych包中的describe语言,方差分析采用所述r 4.0.2软件的lme4包中的lmer语言,最佳线性无偏预测值blup的估算采用所述lme4包中的所述lmer语言;
24、(3)基因型分析
25、采用snp芯片分析所述玉米自交系材料的基因型,过滤掉最小等位基因频率小于5%和缺失率大于20%的低质量snp分子标记,获得高质量snp分子标记;
26、(4)全基因组关联分析
27、采用所述r 4.0.2软件的3vmrmlm包的压缩方差组分混合模型的“single_env”方法进行全基因组关联分析,获得玉米穗腐病抗性显著相关snp分子标记组合。
28、较佳地,在所述步骤(1)中,多份所述玉米自交系材料分为p群、tangsipingtou群、lancaster群、bsss群和waxy群,所述p群包括99份所述玉米自交系材料,所述tangsipingtou群包括51份所述玉米自交系材料,所述lancaster群包括36份所述玉米自交系材料,所述bsss群包括76份所述玉米自交系材料,所述waxy群包括72份所述玉米自交系材料;所述种植环境的数目为5个;所述fusarium graminearum菌液的浓度为1×106个孢子/ml;所述的调查果穗的发病情况具体是将所述玉米收获后自然晒干,根据发病面积占果穗面积的比例分为1级~7级,其中:1级:没有发病;2级:发病面积占果穗面积的1%~3%;3级:发病面积占果穗面积的4%~10%;4级:发病面积占果穗面积的11%~25%;5级:发病面积占果穗面积的26%~50%;6级:发病面积占果穗面积的51%~75%;7级:发病面积占果穗面积的76%~100%。
29、较佳地,在所述步骤(3)中,所述snp芯片采用 maize56k snp array,所述过滤采用tassel 5.0软件进行。
30、较佳地,在所述步骤(4)中,所述“single_env”方法考虑q+k,设置lod≥3,其余参数为所述4.0.2软件的默认值。
31、在本发明的第三方面,提供一种上述的玉米穗腐病抗性显著相关snp分子标记组合在构建玉米穗腐病抗性全基因组选择数据模型中的应用。
32、在本发明的第四方面,提供一种玉米穗腐病抗性全基因组选择数据模型构建方法,其特点是,将上述的玉米穗腐病抗性显著相关snp分子标记组合作为固定效应加入全基因组选择数据模型中从而构建玉米穗腐病抗性全基因组选择数据模型。
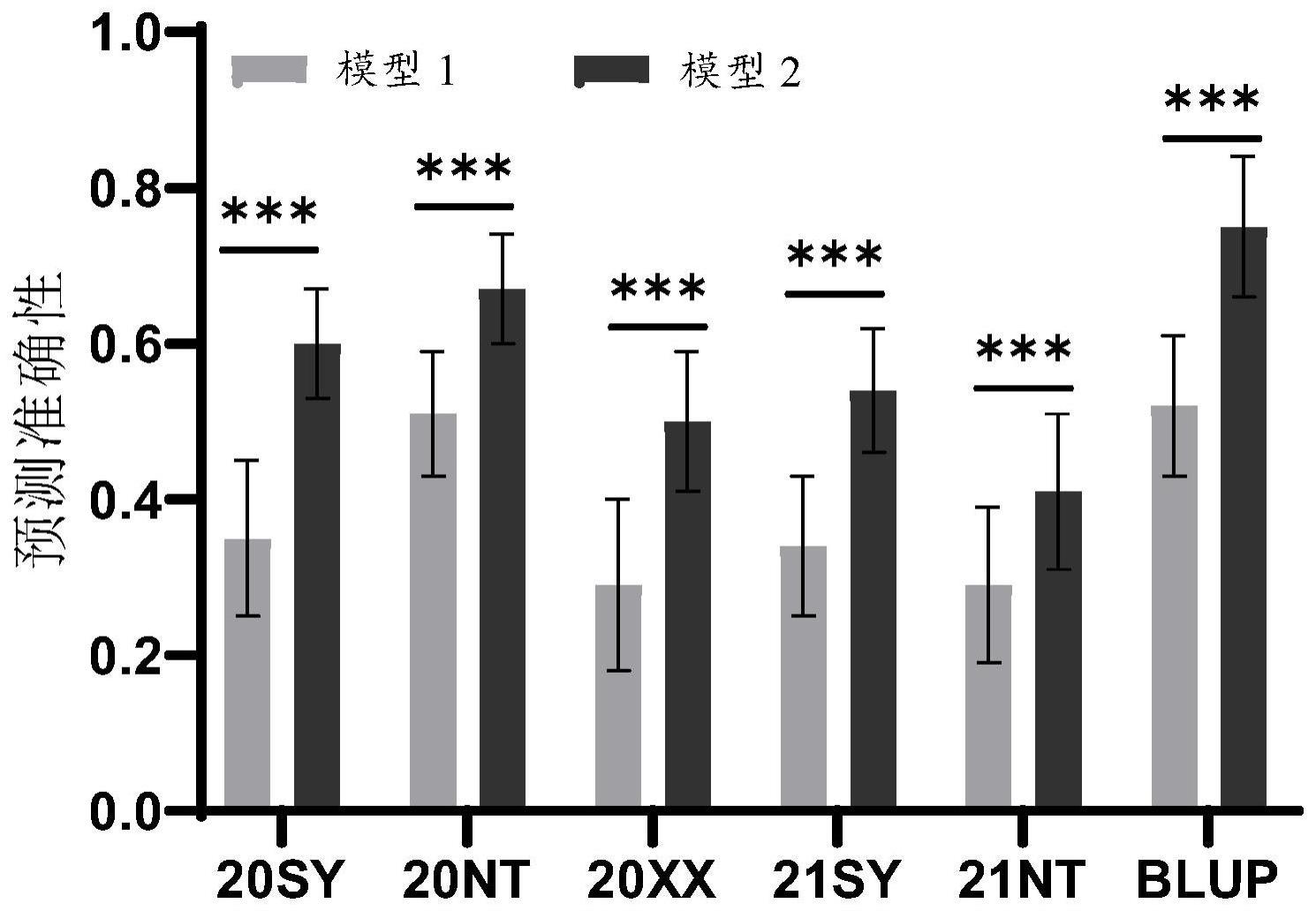
33、较佳地,所述全基因组选择数据模型是采用r 4.0.2软件的gapit包构建的gblup随机效应模型,所述gblup随机效应模型为:y=1nμ+zu+ε,其中y表示表型值,μ表示总体均值,1n表示元素为1的1×n维向量,u表示随机效应,z表示与随机效应相关的n×m的设计矩阵,n表示个体数,m表示分子标记数,ε表示剩余效应;
34、所述玉米穗腐病抗性全基因组选择数据模型是采用所述gapit包构建的gblup固定效应模型,所述gblup固定效应模型为:y=1nμ+zu+xβ+ε,其中β表示所述固定效应,x表示与所述固定效应相关的设计矩阵,其余同所述gblup随机效应模型。
35、在本发明的第五方面,提供一种玉米穗腐病抗性全基因组选择数据模型,其特点是,采用上述的玉米穗腐病抗性全基因组选择数据模型构建方法构建而成。
36、在本发明的第六方面,提供一种上述的玉米穗腐病抗性全基因组选择数据模型在预测玉米穗腐病抗性中的应用。
37、本发明的有益效果主要在于:
38、1、本发明的玉米穗腐病抗性显著相关snp分子标记组合包括snp1~snp69,其中:snp1~snp13位于玉米的第1染色体,snp14~snp19位于玉米的第2染色体,snp20~snp25位于玉米的第3染色体,snp26~snp31位于玉米的第4染色体,snp32~snp39位于玉米的第5染色体,snp40~snp42位于玉米的第6染色体,snp43~snp55位于玉米的第7染色体,snp56~snp60位于玉米的第8染色体,snp61~snp64位于玉米的第9染色体,snp65~snp69位于玉米的第10染色体,其能够用于构建玉米穗腐病抗性全基因组选择数据模型,从而显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
39、2、本发明的玉米穗腐病抗性显著相关snp分子标记组合的筛选方法,包括以下步骤:(1)田间试验,将多份玉米自交系材料在至少一个种植环境下种植,采用完全随机区组设计,接种fusarium graminearum菌液,调查果穗的发病情况;(2)表型数据分析,采用r4.0.2软件进行;(3)基因型分析,采用snp芯片分析,过滤掉最小等位基因频率小于5%和缺失率大于20%的低质量snp分子标记;(4)全基因组关联分析,采用r 4.0.2软件的3vmrmlm包的压缩方差组分混合模型的“single_env”方法进行,因此,其设计巧妙,操作简便,适于大规模推广应用。
40、3、本发明的玉米穗腐病抗性显著相关snp分子标记组合在构建玉米穗腐病抗性全基因组选择数据模型中的应用,由此构建的构建玉米穗腐病抗性全基因组选择数据模型能够显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
41、4、本发明的玉米穗腐病抗性全基因组选择数据模型构建方法是将玉米穗腐病抗性显著相关snp分子标记组合作为固定效应加入全基因组选择数据模型中从而构建玉米穗腐病抗性全基因组选择数据模型,设计巧妙,操作简便,由此构建的构建玉米穗腐病抗性全基因组选择数据模型能够显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
42、5、本发明的玉米穗腐病抗性全基因组选择数据模型采用玉米穗腐病抗性全基因组选择数据模型构建方法构建而成,能够显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
43、6、本发明的玉米穗腐病抗性全基因组选择数据模型在预测玉米穗腐病抗性中的应用,从而能够显著提高玉米穗腐病抗性预测准确性,进而提高育种效率,降低育种成本,适于大规模推广应用。
- 还没有人留言评论。精彩留言会获得点赞!