幽门螺旋杆菌四价黏附因子GEM颗粒疫苗及制备方法与应用与流程
本发明涉及生物医学及生物工程领域,具体而言,涉及幽门螺旋杆菌四价黏附因子gem颗粒疫苗及制备方法与应用。
背景技术:
幽门螺旋杆菌(hp)是胃炎、胃溃疡和胃癌的重要致病因子,世界感染率超过50%,我国hp感染率高达58.07%,且呈现明显的家庭聚集性,形式更加严峻。目前,临床治疗hp感染性胃病的方法主要是多联抗生素疗法,但是面临着抗生物导致hp耐药性、治疗后再感染、抗生素破坏肠道微生态平衡、药物毒副作用和病人依从性差等问题,难以在hp感染人群中大规模推广使用,研发有效的hp疫苗具有很好的应用前景。hp对人类胃部的感染,关键的第一步是通过黏附因子黏附胃黏膜细胞。
口服类疫苗的免疫原性一般较弱,需使用大量抗原多次接种,且免疫应答持续时间较短,容易产生免疫耐受。微皱褶细胞(m细胞)是胃肠道黏膜免疫系统中一种特化的抗原转运细胞,散布于黏膜上皮细胞间,能将抗原从胃肠腔内转运到上皮下的淋巴组织,诱导黏膜免疫反应。m细胞是胃肠道黏膜免疫系统的“入口”,其对抗原的摄取是启动胃肠黏膜免疫应答关键的第一步。因此,“抗原能否被m细胞大量摄取”是口服类疫苗免疫成败的关键所在。利用“与m细胞表面受体相结合的m细胞靶向肽”,将抗原靶向递呈给m细胞,增加其对抗原的摄取和转运效率,激发高水平黏膜免疫应答,是一种提高口服类疫苗免疫效果的有效策略。
口服类疫苗在激发胃肠道黏膜免疫应答和特异性细胞免疫应答具有一定优势,但是直接口服给药,疫苗效果往往甚微,其中主要原因是:胃肠道中存在大量的酸和蛋白水解酶,会破坏疫苗抗原。因此,研究安全有效的黏膜免疫投递系统是口服类疫苗研究领域的一个热点。乳酸菌是一群在人体肠道内的常见益生细菌,作为口服类疫苗的黏膜免疫投递系统,具安全性高等独特优势。另外,将抗原展示在乳酸菌表面更符合黏膜疫苗的要求。锚定蛋白是细菌表面展示技术中的核心组件。n-乙酰胞壁质酶(acma)是在乳酸菌表面展示系统中常用的一种锚定蛋白。然而,在胞内表达的acma往往跨膜转运活性不强。基于acma设计更为有效的乳酸菌细胞表面展示系统,仍是一项亟待研究的工作。
技术实现要素:
本发明的第一方面,公开了幽门螺旋杆菌四价黏附因子gem颗粒疫苗,其特征在于,在gem颗粒表面展示有重组抗原same-fade;重组抗原same-fade含有核心组件same和幽门螺旋杆菌黏附因子多表位肽fade。
幽门螺旋杆菌四价黏附因子gem颗粒疫苗gem-same-fade特点如下:(1)在gem颗粒表面展示有重组抗原same-fade;重组抗原same-fade含有核心组件same和幽门螺旋杆菌黏附因子多表位肽fade。(2)gem颗粒疫苗gem-same-fade表面展示的重组抗原same-fade具有m细胞靶向的性质,可增加胃肠道m细胞对重组抗原same-fade的摄取量和转运效率,增强胃肠道黏膜特异性免疫应答。(3)gem颗粒疫苗gem-same-fade可激发机体产生针对幽门螺旋杆菌多种关键黏附因子的特异性黏膜免疫应答。
gem颗粒表面展示技术是一种基于革兰氏阳性增强基质颗粒(即gem颗粒)的非活性、非遗传修饰的新型乳酸菌表面展示技术,称之为gem颗粒表面展示技术,又叫细菌样颗粒表面展示技术。gem颗粒表面展示技术主要利用热酸处理乳酸菌,去除蛋白质和核酸等大分子物质,制备仅含有细胞壁肽聚糖骨架的空心颗粒,叫gem颗粒;将高纯度疫苗抗原与gem颗粒混合,疫苗抗原通过与其偶联的锚定蛋白展示在gem颗粒表面,制备的疫苗叫gem颗粒疫苗,又叫细菌样颗粒疫苗。
在前述的第一方面的一些实施例中,黏附因子多表位肽fade的氨基酸序列如seqidno.1所示,编码黏附因子多表位肽fade的核苷酸序列如seqidno.2所示。
幽门螺旋杆菌的关键黏附因子主要有尿素酶、黏附素hpaa、外膜蛋白lpp20、cagl等。目前国内外尚无上市可用的hp疫苗,全菌疫苗或亚单位疫苗,存在毒副作用、免疫特异性不高、保护性较低等问题。表位疫苗是一种新型疫苗,较之传统疫苗具有高安全性、高特异性和设计灵活等优势,是发展hp疫苗尤其治疗性hp疫苗的新策略。hp黏附因子多表位疫苗cfade含来自hp四种关键黏附因子(尿素酶、lpp20、hpaa和cagl)的多表位肽fade和霍乱毒素b亚基(ctb),研究已证实cfade具有很好的防治hp感染的免疫效果。幽门螺旋杆菌黏附因子多表位肽fade是来自hp黏附因子多表位疫苗cfade中除去ctb的多表位肽部分,主要包含来自hp四种关键黏附因子尿素酶、lpp20、hpaa和cagl的优势表位或表位区段,可刺激机体产生针对幽门螺旋杆菌关键黏附因子的t细胞免疫应答和特异性体液免疫应答。
在前述的第一方面的一些实施例中,核心组件same的氨基酸序列如seqidno.3所示,编码核心组件same的核苷酸序列如seqidno.4所示。
在前述的第一方面的一些实施例中,重组抗原same-fade的氨基酸序列如seqidno.5所示;编码重组抗原same-fade的核苷酸序列如seqidno.6所示。
本申请的第二方面,公开了幽门螺旋杆菌四价黏附因子gem颗粒疫苗在制备防治幽门螺旋杆菌引起的疾病的药物中的应用。
本申请的第三方面,幽门螺旋杆菌四价黏附因子gem颗粒疫苗的制备方法,包括以下步骤:
合成如序列seqidno.4所示的same基因片段,将same基因片段通过酶切和连接,克隆到pczn1载体上,得到pesame重组载体;
通过扩增得到如序列seqidno.2所示的黏附因子多表位肽fade基因片段,双酶切所述fade片段和pesame重组载体;将pesame重组载体线性化片段与fade片段连接,得到pesame-fade重组载体;
将pesame-fade重组载体转化进表达宿主细胞,通过培养和诱导剂诱导表达,纯化得到重组抗原same-fade;
通过三氯乙酸制备乳酸菌的gem颗粒,将制备的gem颗粒和重组抗原same-fade混合重悬,得到gem颗粒疫苗gem-same-fade。
在前述第三方面的一些实施例中,诱导剂为iptg,诱导剂的工作浓度为0.5mmol/l。
在实施例中,通过iptg的诱导,重组表达菌株能快速大量的表达重组抗原same-fade。
在前述第三方面的一些实施例中,表达宿主细胞为大肠杆菌;
优选地,大肠杆菌选自arcticexpress(de3),dh5α,top10,bl21(de3),bl21(de3)plyss或rosetta;
进一步优选地,大肠杆菌为arcticexpress(de3)。
在实施例中,蛋白表达的宿主细胞有多种,本实施例中优选大肠杆菌中的arcticexpress(de3)菌株进行表达。
通过大肠杆菌的表达系统,能快速高效的表达出目的蛋白,提高重组蛋白的收率和质量。
在前述第三方面的一些实施例中,纯化重组蛋白包括层析和透析;柱层析选用ni-ida-sepharosecl-6b亲和层析柱;透析的透析液为20mmtris-hcl,0.10mnacl,ph8.0的透析液。
在实施例中,通过亲和层析和透析的处理,不仅能提高重组蛋白的浓度和质量,还能通过透析等方法,除去蛋白中的一些毒素。提高了蛋白的浓度,能提高蛋白的免疫效果,透析除去毒素,能提高的蛋白的安全性。
在前述第三方面的一些实施例中,层析缓冲液的流速为0.5-1ml/min,透析的透析液为20mmol/ltris-hcl,0.10mol/lnacl,ph8.0的溶液。
在实施例中,通过合适流速的缓冲液的冲洗,就能较好的纯化出蛋白质。通过合适浓度的透析液的透析,可以较好的将毒素等除去,又不至于破坏蛋白质的结构和功能。
与现有技术相比,本申请的有益效果为:本申请提供的幽门螺旋杆菌四价黏附因子gem颗粒疫苗gem-same-fade,主要是将含有核心组件same和幽门螺旋杆菌黏附因子多表位肽fade的重组抗原same-fade展示在gem颗粒表面制备而成,具有良好的抗原表面展示性质。幽门螺旋杆菌四价黏附因子gem颗粒疫苗gem-same-fade同时具有良好的胃肠道m细胞靶向性,可高效地将重组抗原same-fade靶向递送给胃肠道m细胞,激发胃肠道产生针对幽门螺旋杆菌多种黏附因子的特异性黏膜免疫应答。幽门螺旋杆菌四价黏附因子gem颗粒疫苗gem-same-fade可应用于防治幽门螺旋杆菌相关性胃病。
附图说明
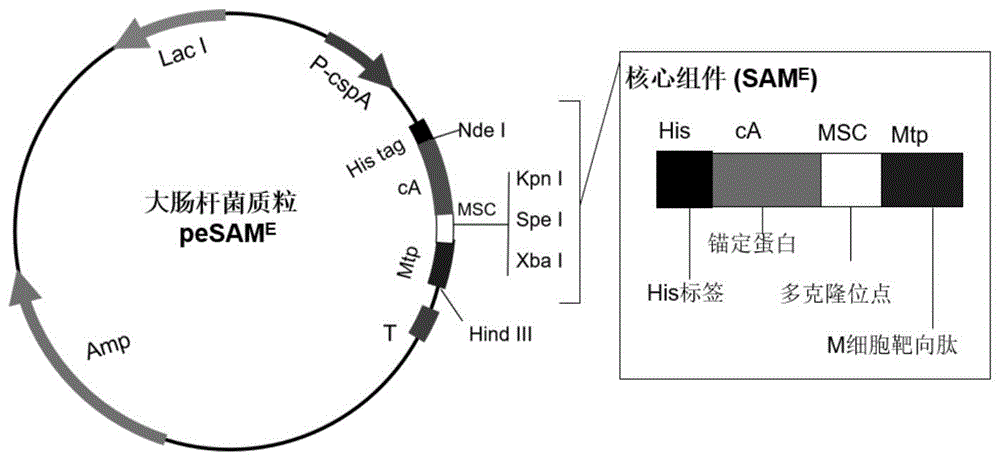
图1是实施例1中大肠杆菌质粒pesame的示意图。
图2是实施例1中大肠杆菌质粒pesame的双酶切电泳图。
图3是实验例2中大肠杆菌质粒pesame-fade的双酶切电泳图。
图4是实验例3中大肠杆菌e.coli-pesame-fade的诱导表达结果图。
图5是实验例4中gem颗粒疫苗gem-same-fade的抗原表面展示性鉴定图。
图6是实验例5中gem颗粒疫苗gem-same-fade的m细胞靶向性鉴定图。
图7是实验例6中gem颗粒疫苗gem-same-fade免疫治疗幽门螺旋杆菌感染效果图。
图8是实验例7中gem颗粒疫苗gem-same-fade免疫治疗后诱导小鼠产生抗体的示意图。
具体实施方式
下面将结合实施例对本申请的实施方案进行详细描述,但是本领域技术人员将会理解,下列实施例仅用于说明本申请,而不应视为限制本申请的范围。实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。
材料
1.iptg溶液:称取1.2giptg置于50ml离心管中,加入40ml无菌水,充分混匀溶解后,定容至50ml。用0.22μm滤器过滤除菌,小份分装,-20℃保存。
2.氨苄青霉素(amp)贮液(100mg/ml):称取100mg氨苄青霉素(amp)溶于1ml无菌水,制得浓度为100mg/ml的贮存液,通过0.22µm细菌滤器过滤除菌,溶液分装保存于-20℃冰箱中。
3.lb培养基:(1)lb液体培养基:称取10g胰蛋白胨、5g酵母提取物和5gnacl,加蒸馏水至1000ml,调整ph至7.4,高压蒸气灭菌。(2)lb固体培养基:1.5g琼脂粉/100mllb培养液,高压灭菌后,倾倒平板。
4.dna电泳缓冲液(50×tae):称取242gtris、37.2gna2edta·2h2o和57.1ml冰乙酸,加水至1000ml,使用时稀释50倍。
5.sds-page电泳缓冲液(5×):称取tris粉末15.1g、甘氨酸94g、sds5.0g;加入约800ml的去离子水,搅拌溶解;加去离子水定容至1l,室温保存;注意:加水时应让水延着壁缓缓流下,以避免由于sds的原因产生很多泡沫。
6.考马斯亮蓝蛋白染色试剂:(1)考马斯亮蓝r-250染液:0.29g考马斯亮蓝r-250溶解在250ml脱色液中。(2)固定液:500ml乙醇、100ml冰醋酸用蒸馏水稀释至1000ml。(3)脱色液:250ml乙醇、80ml冰醋酸用蒸馏水稀释至1000ml。(4)保存液:25ml87%甘油溶于225ml脱色液中。
7.实验动物:balb/c小鼠为spf级,雄性,8~10周龄,购自宁夏医科大学实验动物中心。
8.elisa试剂:(1)包被液:1.6gna2co3,2.9gnahco3,0.2gnan3,加双蒸水至1l,调ph值至9.6。(2)洗涤液:分别称取0.2gkh2po4,2.9gna2hpo4∙12h2o,8.0gnacl,0.2gkcl,0.5mltween-20,加入ddh2o定容至1000ml(pbst)。(3)封闭液:称取3.0gbsa溶解于100ml洗涤缓冲液中,过滤除菌后4℃保存。(4)底物液:可溶性单组份tmb底物溶液。(5)终止液:量取蒸馏水178.3ml,逐滴加入浓硫酸21.7ml(1mh2so4)。
9.bhi血平板:称取3.5gbhi干粉,加蒸馏水93ml,1.5g琼脂粉,121℃灭菌13min,待冷却至60℃以下,加入7ml脱纤维羊血,多粘菌素b(终浓度5μg/ml),万古霉素(终浓度10μg/ml)和甲氧苄氨嘧啶(终浓度5μg/ml),分装至培养皿,冷却后备用。
以下结合实施例对本申请的特征和性能作进一步的详细描述。
实施例1:大肠杆菌质粒pesame的构建
根据核心组件same的氨基酸序列seqidno.3,设计优化其编码基因并合成核心组件same的基因序列如seqidno.4所示。
提取pczn1载体质粒,并用ndei/hindiii双酶切pczn1载体质粒和核心组件same;37℃,2h;利用dna回收试剂盒回收same基因和pczn1载体双酶切产物。将回收的pczn1线性化载体和same基因片段通过互补的粘性末端,在t4dna连接酶的作用下连接成闭合环状的dna分子,即形成大肠杆菌质粒pesame。
质粒结构如图1所示,将连接完成的的质粒检测,通过双酶切并进行电泳检测,结果如图2所示,图中泳道1为大肠杆菌质粒pesame,泳道2为双酶切的结果;双酶切结果与实际一致。送样进行测序,测序结果准确,且无移码突变。
实施例2:大肠杆菌重组质粒pesame-fade的构建
将得到的大肠杆菌重组质粒pesame和扩增得到的fade基因基因片段(带酶切位点)分别用kpnⅰ和xbaⅰ双酶切;37℃,2h;利用dna回收试剂盒回收fade基因和pesame双酶切线性化片段。将回收的pesame线性化载体和fade基因通过互补的粘性末端,在t4dna连接酶的作用下连接成闭合环状的dna分子,即形成大肠杆菌重组质粒pesame-fade。
将大肠杆菌重组质粒pesame-fade通过双酶切后进行电泳,结果如图3所示,图中泳道1为大肠杆菌重组质粒pesame-fade;泳道2为ndeⅰ和hindⅲ酶切后的大肠杆菌重组质粒pesame-fade;与实际一致。将获得的重组大肠杆菌重组质粒pesame-fade进行测序验证,结果显示构建的大肠杆菌重组质粒pesame-fade序列准确且无移码突变。
实施例3:重组抗原same-fade的表达、纯化和检测
将大肠杆菌重组质粒pesame-fade转化宿主细胞进行蛋白表达和纯化。
(1)e.coli-pesame-fade的诱导表达:将验证正确的重组表达质粒pesame-fade转化进大肠杆菌arcticexpress(de3)中。将阳性克隆子接种于含50μg/mlamp的3mllb培养液的试管中,37℃,220rpm,振摇过夜;次日,按1:100接种于50μg/mlamp的30mllb培养液中,37℃,220rpm,振摇培养约2h,至菌体od600为0.6-0.8;然后,加入iptg至终浓度为0.5mm,11℃,220rpm,振摇过夜,诱导重组蛋白表达。
(2)sds-page检测e.coli-pesame-fade的表达情况:e.coli-pesame-fade经诱导表达后,取出1ml培养物,10000g室温离心2min,弃上清,用100μl上样缓冲液重悬菌体沉淀;剩余培养物4000g,离心10min,弃上清,用pbs重悬菌体沉淀;重悬液进行超声波破碎后,分别取上清液与沉淀液加入上样缓冲液重悬,进行sds-page检测分析。
(3)重组抗原same-fade的纯化:利用低压层析系统,包涵体溶液以0.5ml/min流速上样至ni-idabinding-buffer预平衡的ni-ida-sepharosecl-6b亲和层析柱;用ni-idabinding-buffer以0.5ml/min流速冲洗,至流出液od280值到达基线;用ni-idawashing-buffer以1ml/min流速冲洗,至流出液od280值到达基线;用ni-idaelution-buffer以1ml/min流速洗脱目的蛋白,收集流出液;上述收集的蛋白溶液加入透析袋中,使用20mmtris-hcl,0.10mnacl,ph8.0进行透析过夜,进行12%sds-page和westernblot鉴定分析。
sds-page检测结果如图4a所示,诱导表达后,经sds-page检测,表明:基因工程菌株e.coli-pesame-fade在约97kd处出现目的蛋白条带,与重组蛋白same-fade的理论大小相符合(图4a);经ni-ida蛋白纯化后,可获得高纯度重组蛋白same-fade,纯度约为97.85%,纯化的效果较好(图4b);mouseanti-fade可识别same-fade,但是鼠正常血清不是识别same-fade(图4c),表达的蛋白可以被相应的抗体识别。
实施例4:gem颗粒疫苗gem-same-fade的制备及抗原表面展示性质的鉴定
通过三氯乙酸制备乳酸菌的gem颗粒,将制备的gem颗粒与纯化的重组抗原same-fade混合重悬,制备gem颗粒疫苗gem-same-fade。
将gem-same-fade、gem-same、same-fade和gem颗粒分别包被至elsia板,4℃过夜。洗4次后,加入300μl/孔封闭液,37℃,2h。洗4次后,加mouseanti-fade(1:3000)到elisa板,100μl/孔,37℃,60min。洗4次后,加入hrp标记的羊抗鼠igg(1:10000),100μl/孔,37℃,60min。洗4次后,加入tmb显色液,室温,10min,加100μl终止液,测定od450值。
结果如图5所示,gem颗粒疫苗gem-same-fade和重组抗原same-fade能够与mouseanti-fade发生特异性反应,但gem-same和gem不能与mouseanti-fade发生特异性反应,说明gem颗粒疫苗gem-same-fade表面展示有same-fade抗原,gem-same表面无same-fade抗原。
实施例5:gem颗粒疫苗gem-same-fade的m细胞靶向性的鉴定
balb/c小鼠禁食一晚(约12h),麻醉,打开腹部,将回肠中区约2cm长度结扎形成一个封闭的回肠袢,将100μlgem颗粒疫苗gem-same-fade和gem颗粒注入回肠袢,约1h后,切除回肠袢,pbs反复冲洗三次后,用4%多聚甲醛固定。组织切片经3%山羊血清封闭后,接着分别与rabbitanti-fade多克隆抗体和goatanti-rabbitiggh&l(alexafluor®647)抗体孵育。派尔淋巴结上的m细胞由mouseanti-gp2mab(alexafluor®488)进行标记;细胞核由dapi进行染色。
结果如图6所示,与对照gem颗粒相比,gem颗粒疫苗gem-same-fade具有良好的m细胞靶向性质,可明显与胃肠道m细胞相结合;而对照中均没有靶向结果。
实施例6:gem颗粒疫苗gem-same-fade治疗hp感染效果的鉴定
(1)小鼠实验流程
实验分组:gem-same-fade免疫组、gem-same免疫组和gem颗粒免疫组;每组10只小鼠,共30只。
hp感染小鼠模型的制备:各组小鼠分别于1、3、5、7天灌胃幽门螺旋杆菌,共计4次,每次300μl/只,浓度为1×1010cfu/ml。
小鼠免疫:分别于15,22,29,36天免疫gem颗粒疫苗gem-same-fade、gem-same和gem,共计4次,每次300μl/只,浓度为1×1010cfu/ml(即3×109cfu)。
小鼠处死:在第50天将小鼠处死,取样进行检验。
(2)小鼠胃部hp的定量培养检测
称取一定重量的小鼠胃组织,用生理盐水冲去内容物,在0.5ml生理盐水中制备组织均浆液,吸取20μl均浆液,按照1:10、1:100和1:1000倍稀释。每个稀释度各取100μl均匀涂布在bhi血平板上,37℃,微需氧培养3~5天。用细菌革兰氏染色和过氧化氢触酶等方法鉴定hp菌落。对鉴定为阳性的hp菌落进行计数,菌落计数换算成每克胃组织菌落形成单位数(cfu/g),计算公式为:hp定植密度=hp菌落数×稀释度/胃重量。
(3)小鼠胃部组织病理学检测:
he染色:用石蜡包埋经10%甲醛固定的小鼠胃组织,切成约6μm厚的组织切片,he染色观察胃组织炎症情况,并进行组织病理学评分。
评分标准如下:
无明显可见白细胞(淋巴细胞和中性粒细胞)浸润为0分;
在胃粘膜固有层深部有少许散在白细胞浸润为1分;
在胃粘膜深部至中部固有层有中等程度白细胞浸润为2分;
在胃粘膜深部至中部固有层有大量白细胞浸润,少量微脓肿为3分;
在胃粘膜固有层全层至黏膜下层有严重弥散性白细胞浸润,微脓肿较多为4分。
免疫组织化学染色:石蜡切片脱蜡后,经正常山羊血清封闭,室温,10min;滴加适当比例稀释的rabbitanti-hp,37℃,1h;经洗涤后,滴加羊抗兔igg-hrp,37℃,10min;dab显色,冲洗,复染,封片。
结果如图7所示,经gem-same-fade免疫治疗hp感染的小鼠后,hp定量培养和尿素酶活性检测,结果表明:gem-same-fade免疫组小鼠胃部的hp定植数显著减少,尿素酶活性降低(图7a和图7b);胃组织病理学实验结果表明:gem-same和gem免疫组小鼠的胃黏膜和胃黏膜下层有大量白细胞浸润,胃部炎症比较明显,有明显hp定植;而gem-same-fade免疫组小鼠的胃部炎症显著减轻,无明显hp定植(图7c、图7d和图7e)。
实施例7:gem颗粒疫苗gem-same-fade免疫治疗后小鼠特异性抗体的检测
hp感染的小鼠经gem颗粒疫苗gem-same-fade免疫治疗后,处死,取血清,1:1000稀释,取胃黏液和肠液1:5倍稀释。按100μl/孔加入已包被hp裂解液的elisa板,37℃,60min;洗4次后,加hrp标记羊抗鼠igg或iga(1:10000),100μl/孔,37℃,60min;洗4次后,加tmb显色液,室温避光10min,加100μl终止液,测定od450值。
结果如图8所示,其中图8a为血清igg抗体检测,图8b为血清iga抗体检测,图8c为黏膜siga抗体检测。elisa实验结果表明gem-same-fade免疫组小鼠能产生hp特异性igg、iga和siga;然而,gem-same和gem免疫组小鼠不能够产生hp特异性igg、iga和siga(图8a、图8b和图8c)。
综上可以看出,本发明提供的gem颗粒疫苗gem-same-fade具有胃肠道m细胞靶向投递的性质,可诱导胃肠道产生针对幽门螺旋杆菌的特异性免疫应答,具有防治幽门螺旋杆菌感染相关性胃病的潜力。
以上所描述的实施例是本申请一部分实施例,而不是全部的实施例。本申请的实施例的详细描述并非旨在限制要求保护的本申请的范围,而是仅仅表示本申请的选定实施例。基于本申请中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本申请保护的范围。
sequencelisting
<110>宁夏医科大学
<120>幽门螺旋杆菌四价黏附因子gem颗粒疫苗及制备方法与应用
<160>6
<170>patentinversion3.5
<210>1
<211>628
<212>prt
<213>人工序列
<400>1
glyilelysleuasntyrvalglualavalalaleuileseralahis
151015
ilemetgluglualaargalaglylyslysthralaalagluleumet
202530
glngluglyargthrleuleulysproaspaspvalmetaspglyval
354045
alasermetilehisgluvalglyileglualametpheproaspgly
505560
servalgluleuileaspileglyglyasnargargilepheglyphe
65707580
asnalaleuvalaspargglnalaaspasngluserlyslysileala
859095
leuhisargalalysgluargglyphehisglylyslysservalglu
100105110
leuileaspileglyglyasnargargilepheglypheasnalaleu
115120125
valaspargglnalaaspasngluserlyslysilealaleuhisarg
130135140
alalysgluargglyphehisglysercyshishisleuasplysser
145150155160
ilelysgluaspvalglnphealaaspserargileargproglnthr
165170175
ilealaalagluaspthrleuhisaspmetglyilepheserilethr
180185190
serseraspserglnalametglyargvalglygluvalilethrarg
195200205
thrtrpglnthralaasplysasnlyslyscyshishisleuasplys
210215220
serilelysgluaspvalglnphealaaspserargileargprogln
225230235240
thrilealaalagluaspthrleuhisaspmetglyilepheserile
245250255
thrserseraspserglnalametglyargvalglygluvalilethr
260265270
argthrtrpglnthralaasplysasnlyslystyrglulystyrser
275280285
glyvalpheleuglyargalagluaspleuilethrasnasnaspval
290295300
asptyrserthrasnglnalathralalysalaargalaasnleuala
305310315320
alaasnleuglysersertrpproleutyrseraspalaserglyleu
325330335
glysersertrpproleutyrseraspalaserglyleuglyserser
340345350
trpproleutyrseraspalaserglyleuglupheglnalaleuasp
355360365
glulysileleuleuleulysproalapheglntyrseraspasnile
370375380
alalysglutyrgluasnlysphelysasnglnthrthrleulysval
385390395400
glugluileleuglnasnglnglytyrlysvalileasnvalaspser
405410415
serasplysaspasppheserphealaglnlyslysgluglytyrleu
420425430
alavalalametileglygluilevalleuargproaspprolysarg
435440445
thrileglnlyslyssergluproglyleuleupheserthrglyleu
450455460
asplysmetgluglyvalleuileproalaglyphevallysvalthr
465470475480
ileleugluprometserglygluserleuaspserphethrmetasp
485490495
leusergluleuaspileglnglulyspheleulysleugluaspile
500505510
thrserglyleulysglnleuaspserthrtyrglngluthrasngln
515520525
glnvalleulysasnleuaspgluilepheserthrthrserproser
530535540
alaasnasngluileglyglngluaspalaleuasnilelyslysala
545550555560
alailealaleuargglyaspleualaleuleulysalaasnpheglu
565570575
alaasngluleuphepheilesergluaspvalilephelysthrtyr
580585590
metserserprogluleuleuleuthrtyrmetlysileasnproleu
595600605
aspglnasnthralagluglnglncysglyileserasplysvalleu
610615620
valleutyrcys
625
<210>2
<211>1884
<212>dna
<213>人工序列
<400>2
ggcatcaaactcaactatgtagaggcagtggccttaatttccgcccatatcatggaagaa60
gcgcgtgcagggaagaaaaccgcggcggaactgatgcaggaaggccgcaccctgctgaag120
cctgacgacgttatggatggtgttgcgtccatgatccacgaagtaggcattgaagcgatg180
ttcccggatggtagcgtggagctgattgatatcggtggtaaccgtcgcatcttcggtttc240
aacgccttagtggaccgtcaggcggacaacgagagcaagaaaattgctctacaccgtgca300
aaagagcgcggctttcacggcaagaaaagcgtcgaactgatcgatattgggggtaaccgc360
cgcattttcggcttcaacgcgctggtagatcggcaggctgataacgaaagcaaaaagatt420
gctctgcaccgtgctaaagaacgtggtttccacggttcttgtcaccacttagacaaatcg480
attaaagaagatgtccaattcgctgatagccgtatccgcccgcagaccatcgcggcggag540
gacaccctgcacgacatgggcatcttcagcatcacctccagcgacagccaagcaatgggt600
cgtgtaggggaagtgatcacccgtacctggcaaactgcggataaaaacaaaaaatgccac660
cacctggacaaaagtattaaggaggacgtgcagtttgcagattcccgtatccgtccgcaa720
accatcgctgcagaagatactctgcatgacatgggcatcttcagtattacctcaagcgac780
tcccaggcgatgggccgtgttggcgaagtgatcactcgtacatggcagactgcggacaaa840
aataaaaagtacgagaaatattcaggtgtttttctgggtcgcgcagaggatctgattacc900
aacaacgacgtggactactcaacgaaccaggctacggcgaaagcgcgcgcaaatctcgca960
gctaacctcggttctagttggccgctgtactctgacgcaagcggtctgggctcctcttgg1020
ccgctgtattccgatgcctctggcctgggttcctcctggccactgtactccgacgccagc1080
ggtctggaattccaggcgctggacgaaaaaatccttctgctgaaaccagccttccagtac1140
tccgataacattgcaaaagaatatgaaaacaaattcaaaaaccagacgactctgaaggtt1200
gaggaaattctgcaaaaccagggttacaaggttatcaacgtcgactcttcagacaaagat1260
gacttctcgtttgctcagaaaaaagaaggctatttggcagtcgcaatgatcggtgagatc1320
gttctgcgtccggacccgaaacgtaccatccagaaaaaatctgaaccgggcctgctgttc1380
agtacaggcctggataaaatggaaggtgtgctcatcccggccggcttcgttaaagtgacc1440
atcctggaaccgatgtctggtgaatctctggacagcttcaccatggatcttagtgaactg1500
gatatccaggaaaagttcttaaagcttgaagacattacgagcggcctgaaacaactggat1560
agcacctatcaggaaacgaaccagcaagttctgaaaaacctggacgaaatttttagcacc1620
acgtcaccgtcggctaacaatgaaatcggccaggaagatgcgctgaacatcaaaaaagca1680
gcaatcgcactgcgtggtgacctggcactgctgaaagctaactttgaagcgaatgaactg1740
tttttcatctcagaagatgtcatcttcaaaacctatatgagctctccggaactgctgctg1800
acgtacatgaaaattaacccgctggatcagaataccgcggaacagcaatgcggcatctcc1860
gacaaagtgctggttctgtattgt1884
<210>3
<211>264
<212>prt
<213>人工序列
<400>3
metasnhislysvalhishishishishishismetthrthrtyrthr
151015
vallysserglyaspthrleutrpglyileserglnargtyrglyile
202530
servalalaglnileglnseralaasnasnleulysserthrileile
354045
tyrileglyglnlysleuvalleuthrglyseralaserserthrasn
505560
serglyglyserasnasnseralaserthrthrprothrthrserval
65707580
thrproalalysprothrserglnthrthrvallysvallyssergly
859095
aspthrleutrpalaleuservallystyrlysthrserilealagln
100105110
leulyssertrpasnhisleuserseraspthriletyrileglygln
115120125
asnleuilevalserglnseralaalaalaserasnproserthrgly
130135140
serglyserthralathrasnasnserasnserthrserserasnser
145150155160
asnalaserilehislysvalvallysglyaspthrleutrpglyleu
165170175
serglnlysserglyserproilealaserilelysalatrpasnhis
180185190
leuserseraspthrileleuileglyglntyrleuargilelysgly
195200205
thrthrserserargcyslysserthrhisproleusercysserphe
210215220
hisglnleuproalaargserproleuproserleuaspalaglygln
225230235240
tyrvalleuvalmetlysalaasnsersertyrserglyasntyrpro
245250255
tyrserileleupheglnlysphe
260
<210>4
<211>795
<212>dna
<213>人工序列
<400>4
atgaatcacaaagtgcatcatcatcatcatcatatgactacttataccgtcaaatctggt60
gatactctttggggaatctcacaaagatatggaattagtgtcgctcaaattcaaagtgcg120
aataatcttaaaagtaccattatctacattggtcaaaaacttgtactgacaggttcagct180
tcttctacaaattcaggtggttcaaacaattccgcaagcactactccaaccacttctgtg240
acacctgcaaaaccaacttcacaaacaactgttaaggttaaatccggagataccctttgg300
gcgctatcagtaaaatataaaactagtattgctcaattgaaaagttggaatcatttaagt360
tcagataccatttatattggtcaaaatcttattgtttcacaatctgctgctgcttcaaat420
ccttcgacaggttcaggctcaactgctaccaataactcaaactcgacttcttctaactca480
aatgcctcaattcataaggtcgttaaaggagatactctctggggactttcgcaaaaatct540
ggcagcccaattgcttcaatcaaggcttggaatcatttatctagcgatactattttaatt600
ggtcagtatctacgaataaaaggtaccactagttctagatgtaaatcaacacatccatta660
tcatgttcatttcatcaattaccagctcgttcaccattaccatcattagatgctggtcaa720
tatgttttagttatgaaagctaattcatcatattcaggtaattatccatattcaatttta780
tttcaaaaattttga795
<210>5
<211>890
<212>prt
<213>人工序列
<400>5
metasnhislysvalhishishishishishismetthrthrtyrthr
151015
vallysserglyaspthrleutrpglyileserglnargtyrglyile
202530
servalalaglnileglnseralaasnasnleulysserthrileile
354045
tyrileglyglnlysleuvalleuthrglyseralaserserthrasn
505560
serglyglyserasnasnseralaserthrthrprothrthrserval
65707580
thrproalalysprothrserglnthrthrvallysvallyssergly
859095
aspthrleutrpalaleuservallystyrlysthrserilealagln
100105110
leulyssertrpasnhisleuserseraspthriletyrileglygln
115120125
asnleuilevalserglnseralaalaalaserasnproserthrgly
130135140
serglyserthralathrasnasnserasnserthrserserasnser
145150155160
asnalaserilehislysvalvallysglyaspthrleutrpglyleu
165170175
serglnlysserglyserproilealaserilelysalatrpasnhis
180185190
leuserseraspthrileleuileglyglntyrleuargilelysgly
195200205
thrglyilelysleuasntyrvalglualavalalaleuileserala
210215220
hisilemetgluglualaargalaglylyslysthralaalagluleu
225230235240
metglngluglyargthrleuleulysproaspaspvalmetaspgly
245250255
valalasermetilehisgluvalglyileglualametpheproasp
260265270
glyservalgluleuileaspileglyglyasnargargilephegly
275280285
pheasnalaleuvalaspargglnalaaspasngluserlyslysile
290295300
alaleuhisargalalysgluargglyphehisglylyslysserval
305310315320
gluleuileaspileglyglyasnargargilepheglypheasnala
325330335
leuvalaspargglnalaaspasngluserlyslysilealaleuhis
340345350
argalalysgluargglyphehisglysercyshishisleuasplys
355360365
serilelysgluaspvalglnphealaaspserargileargprogln
370375380
thrilealaalagluaspthrleuhisaspmetglyilepheserile
385390395400
thrserseraspserglnalametglyargvalglygluvalilethr
405410415
argthrtrpglnthralaasplysasnlyslyscyshishisleuasp
420425430
lysserilelysgluaspvalglnphealaaspserargileargpro
435440445
glnthrilealaalagluaspthrleuhisaspmetglyilepheser
450455460
ilethrserseraspserglnalametglyargvalglygluvalile
465470475480
thrargthrtrpglnthralaasplysasnlyslystyrglulystyr
485490495
serglyvalpheleuglyargalagluaspleuilethrasnasnasp
500505510
valasptyrserthrasnglnalathralalysalaargalaasnleu
515520525
alaalaasnleuglysersertrpproleutyrseraspalasergly
530535540
leuglysersertrpproleutyrseraspalaserglyleuglyser
545550555560
sertrpproleutyrseraspalaserglyleuglupheglnalaleu
565570575
aspglulysileleuleuleulysproalapheglntyrseraspasn
580585590
ilealalysglutyrgluasnlysphelysasnglnthrthrleulys
595600605
valglugluileleuglnasnglnglytyrlysvalileasnvalasp
610615620
serserasplysaspasppheserphealaglnlyslysgluglytyr
625630635640
leualavalalametileglygluilevalleuargproaspprolys
645650655
argthrileglnlyslyssergluproglyleuleupheserthrgly
660665670
leuasplysmetgluglyvalleuileproalaglyphevallysval
675680685
thrileleugluprometserglygluserleuaspserphethrmet
690695700
aspleusergluleuaspileglnglulyspheleulysleugluasp
705710715720
ilethrserglyleulysglnleuaspserthrtyrglngluthrasn
725730735
glnglnvalleulysasnleuaspgluilepheserthrthrserpro
740745750
seralaasnasngluileglyglngluaspalaleuasnilelyslys
755760765
alaalailealaleuargglyaspleualaleuleulysalaasnphe
770775780
glualaasngluleuphepheilesergluaspvalilephelysthr
785790795800
tyrmetserserprogluleuleuleuthrtyrmetlysileasnpro
805810815
leuaspglnasnthralagluglnglncysglyileserasplysval
820825830
leuvalleutyrcysserargcyslysserthrhisproleusercys
835840845
serphehisglnleuproalaargserproleuproserleuaspala
850855860
glyglntyrvalleuvalmetlysalaasnsersertyrserglyasn
865870875880
tyrprotyrserileleupheglnlysphe
885890
<210>6
<211>2673
<212>dna
<213>人工序列
<400>6
atgaatcacaaagtgcatcatcatcatcatcatatgactacttataccgtcaaatctggt60
gatactctttggggaatctcacaaagatatggaattagtgtcgctcaaattcaaagtgcg120
aataatcttaaaagtaccattatctacattggtcaaaaacttgtactgacaggttcagct180
tcttctacaaattcaggtggttcaaacaattccgcaagcactactccaaccacttctgtg240
acacctgcaaaaccaacttcacaaacaactgttaaggttaaatccggagataccctttgg300
gcgctatcagtaaaatataaaactagtattgctcaattgaaaagttggaatcatttaagt360
tcagataccatttatattggtcaaaatcttattgtttcacaatctgctgctgcttcaaat420
ccttcgacaggttcaggctcaactgctaccaataactcaaactcgacttcttctaactca480
aatgcctcaattcataaggtcgttaaaggagatactctctggggactttcgcaaaaatct540
ggcagcccaattgcttcaatcaaggcttggaatcatttatctagcgatactattttaatt600
ggtcagtatctacgaataaaaggtaccggcatcaaactcaactatgtagaggcagtggcc660
ttaatttccgcccatatcatggaagaagcgcgtgcagggaagaaaaccgcggcggaactg720
atgcaggaaggccgcaccctgctgaagcctgacgacgttatggatggtgttgcgtccatg780
atccacgaagtaggcattgaagcgatgttcccggatggtagcgtggagctgattgatatc840
ggtggtaaccgtcgcatcttcggtttcaacgccttagtggaccgtcaggcggacaacgag900
agcaagaaaattgctctacaccgtgcaaaagagcgcggctttcacggcaagaaaagcgtc960
gaactgatcgatattgggggtaaccgccgcattttcggcttcaacgcgctggtagatcgg1020
caggctgataacgaaagcaaaaagattgctctgcaccgtgctaaagaacgtggtttccac1080
ggttcttgtcaccacttagacaaatcgattaaagaagatgtccaattcgctgatagccgt1140
atccgcccgcagaccatcgcggcggaggacaccctgcacgacatgggcatcttcagcatc1200
acctccagcgacagccaagcaatgggtcgtgtaggggaagtgatcacccgtacctggcaa1260
actgcggataaaaacaaaaaatgccaccacctggacaaaagtattaaggaggacgtgcag1320
tttgcagattcccgtatccgtccgcaaaccatcgctgcagaagatactctgcatgacatg1380
ggcatcttcagtattacctcaagcgactcccaggcgatgggccgtgttggcgaagtgatc1440
actcgtacatggcagactgcggacaaaaataaaaagtacgagaaatattcaggtgttttt1500
ctgggtcgcgcagaggatctgattaccaacaacgacgtggactactcaacgaaccaggct1560
acggcgaaagcgcgcgcaaatctcgcagctaacctcggttctagttggccgctgtactct1620
gacgcaagcggtctgggctcctcttggccgctgtattccgatgcctctggcctgggttcc1680
tcctggccactgtactccgacgccagcggtctggaattccaggcgctggacgaaaaaatc1740
cttctgctgaaaccagccttccagtactccgataacattgcaaaagaatatgaaaacaaa1800
ttcaaaaaccagacgactctgaaggttgaggaaattctgcaaaaccagggttacaaggtt1860
atcaacgtcgactcttcagacaaagatgacttctcgtttgctcagaaaaaagaaggctat1920
ttggcagtcgcaatgatcggtgagatcgttctgcgtccggacccgaaacgtaccatccag1980
aaaaaatctgaaccgggcctgctgttcagtacaggcctggataaaatggaaggtgtgctc2040
atcccggccggcttcgttaaagtgaccatcctggaaccgatgtctggtgaatctctggac2100
agcttcaccatggatcttagtgaactggatatccaggaaaagttcttaaagcttgaagac2160
attacgagcggcctgaaacaactggatagcacctatcaggaaacgaaccagcaagttctg2220
aaaaacctggacgaaatttttagcaccacgtcaccgtcggctaacaatgaaatcggccag2280
gaagatgcgctgaacatcaaaaaagcagcaatcgcactgcgtggtgacctggcactgctg2340
aaagctaactttgaagcgaatgaactgtttttcatctcagaagatgtcatcttcaaaacc2400
tatatgagctctccggaactgctgctgacgtacatgaaaattaacccgctggatcagaat2460
accgcggaacagcaatgcggcatctccgacaaagtgctggttctgtattgttctagatgt2520
aaatcaacacatccattatcatgttcatttcatcaattaccagctcgttcaccattacca2580
tcattagatgctggtcaatatgttttagttatgaaagctaattcatcatattcaggtaat2640
tatccatattcaattttatttcaaaaattttga2673
- 还没有人留言评论。精彩留言会获得点赞!