具有极低水平的大麦醇溶蛋白的大麦的制作方法
本发明涉及生产适用于患有乳糜泻的受试者食用的食物或基于麦芽的饮料的方法。具体来说,本发明涉及生产具有极低水平的大麦醇溶蛋白的食物或基于麦芽的饮料的方法。也提供生产可用于本发明的方法的谷粒的大麦植物。
发明背景
乳糜泻(CD)是T-细胞介导的因食用来由谷粒诸如小麦、大麦和黑麦的谷醇溶蛋白触发的肠病。CD的临床症状包括疲劳、腹泻、腹胀、体重减轻、贫血和神经障碍(Green等人,2006)。CD与肠恶性肿瘤的增加率相关,诸如肠癌风险增加10倍、非霍奇金氏淋巴瘤(non-Hodgkin lymphoma)风险增加3至6倍以及肠T-细胞淋巴瘤风险增加28倍(Green等人,2009)以及与贫血、骨质疏松症、神经性缺损和其它自身免疫疾病诸如糖尿病的增加率相关(Skovbjerg等人,2005)。在大部分研究的谷醇溶蛋白,α-麦醇溶蛋白情况中,毒性主要由单肽中单谷氨酰胺介导(Anderson等人,2000;Shan等人,2002),产生破坏性级联反应而最终损伤小肠绒毛,减少营养吸收和影响健康。在利用小麦、大麦和黑麦短期饮食刺激后,现在利用从HLA-DQ2+腹腔外周血液中分离的无偏T-细胞群体广泛地定位来自小麦、大麦和黑麦的所有已知谷醇溶蛋白的腹腔毒性表位(Tye-Din等人,2010)。出人意料的是,仅三种高免疫原性肽(来源自α-麦醇溶蛋白(ELQPFPQPELPYPQPQ,SEQ ID NO:1)、ω-麦醇溶蛋白/C-大麦醇溶蛋白(EQPFPQPEQPFPWQP,SEQ ID NO:2),和B-大麦醇溶蛋白(EPEQPIPEQPQPYPQQ,SEQ ID NO:3))可能占据90%的由小麦、大麦和黑麦蛋白的完全补充引发的腹腔特异性响应。
目前对CD的唯一治疗是终生避免食用谷蛋白,谷蛋白由小麦(麦醇溶蛋白、麦谷蛋白)、黑麦(黑麦精)、大麦(大麦醇溶蛋白)和燕麦(燕麦蛋白)中所见的类似蛋白的家族组成。然而,此类膳食昂贵(Lee等人,2007)且与低纤维和高糖摄取相关(Kupper等人,2005;Wild等人,2010;Ohlund等人,2010),其本身就是健康风险。避免食用谷蛋白导致大部分(但非全部乳糜泻患者)健康统计的标准化(Lanzini等人,2009;Rubio-Tapia等人,2010)。全球范围内大多数群体中约1%罹患乳糜泻,然而,高达50%的成人仍然未确诊或未表现显性症状(Catassi等人,1994;Fowell等人,2006)。
小麦族(Triciceae)谷物中种子蛋白的主要家族是白蛋白、球蛋白和谷蛋白样蛋白,总称为谷醇溶蛋白(Shewry和Tatham,1990)。白蛋白和球蛋白广泛分布于开花植物中,但谷醇溶蛋白限于草,特别是亚科早熟禾亚科(Pooideae)(Hausch等人,2002)。谷醇溶蛋白如此命名是因为它们含有高比例的氨基酸脯氨酸和谷氨酰胺,在消化期间抗蛋白水解(Hausch等人,2002)。α-麦醇溶蛋白基因是待克隆的第一个谷醇溶蛋白基因且目前已知很多关于该蛋白在乳糜泻中的作用(Kasarda等人,1984)。该谷醇溶蛋白的毒性集中在以谷氨酰胺残基Q65作为关键性氨基酸的单肽57-QLQPFPQPQLPYPQPQS-73(SEQ ID NO:4)。该单谷氨酰胺例如突变为赖氨酸消除了该谷醇溶蛋白的腹腔毒性(Anderson等人,2000)。部分水解肽通过未知机制穿过上皮并进入固有层;此处Q65通过组织转谷氨酰胺酶(tTG)去酰胺化为E65(谷氨酸),从而增加了肽的免疫刺激潜力(Skovbjerg等人,2004)。负电荷促进肽与DQ2(或较不常见的DQ8)受体在抗原呈递细胞表面的结合,从而使所述肽呈递至靶向肠道的具体谷蛋白特异性、DQ2限制性、CD4+T-细胞。因此激活的CD4-T细胞经历克隆扩增且继而通过乳糜泻特征的抗-TG2和抗-谷蛋白抗体特征的最终产生有助于谷蛋白-特异性-和TG2-特异性-B-细胞的扩增。通过分泌促炎细胞因子也出现细胞介导的Th1响应(Tjon等人,2010)。因此,通过引入单个负电荷残基促进的简单蛋白相互作用引发一系列特异性和靶向的级联,最终导致肠道绒毛的破坏。
大麦(Hordeum vulgare L.)是一种用于生产用于酿造和食物工业的麦芽的广泛种植的谷物。发芽大麦是啤酒的主要成分,为发酵提供碳水化合物来源。不幸的是,对于腹腔疾病患者,大麦啤酒也含有低但腹腔毒性水平的大麦醇溶蛋白(谷蛋白)(Dostalek等人,2006)。该大麦醇溶蛋白占一半的大麦谷粒蛋白(Moravcova等人,2009)且由四种多基因家族构成:B-大麦醇溶蛋白(30-45kDa;70%的大麦醇溶蛋白含量)和C-大麦醇溶蛋白(45-75kDa;20%的大麦醇溶蛋白含量)在谷粒蛋白中占据优势,而D-(105kDa)和γ-大麦醇溶蛋白(35-40kDa)为少量组分(Shewry等人,1999)。发芽的高粱、小米和荞麦用于啤酒生产的无谷蛋白的替代品(Wijngaard等人,2007),然而,用这些谷粒难以重现大麦啤酒的质量和低成本生产。
食品法典委员会在2008采用的用于无谷蛋白食物的WHO标准定义要求由谷物诸如小麦和大麦制备的食物必须含有小于20mg/kg(20ppm)谷蛋白才能标注“无谷蛋白”。使用质谱法评估食物和饮料中谷蛋白含量的精确定量方法由Colgrave等人(2012)报道。
WO2009/021285描述将大麦醇溶蛋白(特别是大麦醇溶蛋白B和C)含量降至小于野生型水平10%的大麦谷粒的生产。然而,仍然需要低得多的水平的大麦醇溶蛋白的谷粒用于生产可由患有CD的人食用的食物和饮料。
因此,需要具有实质上更低水平的诱导CD的大麦醇溶蛋白的大麦,其能够用于生产CD易感受试者的食品和饮品中。
发明概要
本发明人已生产具有极低水平大麦醇溶蛋白的大麦谷粒。该谷粒可用于生产罹患乳糜泻的受试者可食用的众多食物和基于麦芽的饮料。
在第一方面,本发明提供一种生产食物或基于麦芽的饮料成分,或食物或基于麦芽的饮料的方法,所述方法包括(i)加工大麦谷粒以生产麦芽、麦芽汁、面粉或全麦,和/或(ii)将大麦谷粒或从所述谷粒生产的麦芽、麦芽汁、面粉或全麦与至少一种其它食物或饮料成分混合,其中所述大麦谷粒、麦芽、麦芽汁、面粉或全麦包含约50ppm或更少的大麦醇溶蛋白,从而生产所述食物或基于麦芽的饮料成分、食物或基于麦芽的饮料。
在另一方面,本发明提供一种生产食物或基于麦芽的饮料成分或食物或基于麦芽的饮料的方法,所述方法包括(i)加工包含约50ppm或更少大麦醇溶蛋白的大麦谷粒以产生麦芽、麦芽汁、面粉或全麦,和/或(ii)将大麦谷粒或从所述谷粒中产生的麦芽、麦芽汁、面粉或全麦与至少一种其它食物或饮料成分混合,其中所述大麦谷粒、麦芽、麦芽汁、面粉或全麦包含约50ppm或更少的大麦醇溶蛋白,从而生产所述食物或基于麦芽的饮料成分、食物或基于麦芽的饮料。
在一个实施方案中,所述谷粒、麦芽、麦芽汁、面粉或全麦包含约20ppm或更少、约10ppm或更少、约5ppm或更少、约0.05ppm至约50ppm、或约0.05ppm至约20ppm、约0.05ppm至约10ppm、约0.05ppm至约5ppm、约0.1ppm至约5ppm、约3.9ppm或约1.5ppm的大麦醇溶蛋白。
在另一个实施方案中,所述谷粒的平均重量为至少约35mg、至少约39mg、至少约41mg、至少约47mg、约35mg至约60mg、约40mg至约60mg、约45mg至约60mg、约39.1mg、约41.8mg或约47.2mg。
在另一个实施方案中,所述谷粒、麦芽、麦芽汁、面粉或全麦包含20ppm或更少的大麦醇溶蛋白,且所述谷粒的平均重量为约40mg至约60mg。在另一个实施方案中,所述谷粒、麦芽、麦芽汁、面粉或全麦包含20ppm或更少的大麦醇溶蛋白,且谷粒的平均重量为约45mg至约60mg。特别提到的这些组合并不排除这些特征的其它组合。
在另一个实施方案中,至少约80%、至少约90%、至少约95%、约80%至约98%,或约80%至约93%的谷粒不能通过2.8mm筛。
在另一个实施方案中,所述谷粒、麦芽、麦芽汁、面粉或全麦包含20ppm或更少的大麦醇溶蛋白,且所述谷粒的平均重量为约40mg至约60mg,且至少约80%的谷粒不能通过2.8mm筛。在另一个实施方案中,谷粒、麦芽、麦芽汁、面粉或全麦包含20ppm或更少的大麦醇溶蛋白,且谷粒的平均重量为约45mg至约60mg,且至少约80%的谷粒不能通过2.8mm筛。特别提到的这些组合并不排除这些特征的其它组合。
在又一个实施方案中,所述谷粒来自具有至少40%、约40%至约60%、约40%至55%,或约40%至约50%的收获指数的植物。在一个优选的实施方案中,所述谷粒来自具有约40%至约50%的收获指数的植物。
在另一个实施方案中,所述谷粒具有小于约5、小于约4、小于约3.8、约2至约5,或约2.5至约3.8的长度与厚度之比。
在另一个实施方案中,由所述谷粒生产的面粉或全麦包含约10ppm或更少、约5ppm或更少、约0.05ppm至约10ppm、或约0.05ppm至约5ppm、约3.9ppm或约1.5ppm的大麦醇溶蛋白。
在另一个实施方案中,由所述谷粒生产的麦芽或麦芽汁包含小于约50ppm或小于约20ppm的大麦醇溶蛋白。
在另一个实施方案中,所述谷粒,或由所述谷粒生产的麦芽、麦芽汁、面粉或全麦具有野生型水平的小于10%、小于5%或小于2%的水平,或缺乏以下物质中一种或多种或所有:
i)包含如SEQ ID NO:53提供的氨基酸序列的B-大麦醇溶蛋白,
ii)包含如SEQ ID NO:54提供的氨基酸序列的B-大麦醇溶蛋白,
iii)包含如SEQ ID NO:55提供的氨基酸序列的C-大麦醇溶蛋白,以及
iv)包含如SEQ ID NO:56提供的氨基酸序列的D-大麦醇溶蛋白,
其中小于10%、小于5%或小于2%的每一个水平相对于大麦品种Bomi、Sloop、Baudin、Yagan、Hindmarsh,或Commander的野生型大麦谷粒,或由所述谷粒制得的麦芽、麦芽汁、面粉或全麦。
在一个实施方案中,B-大麦醇溶蛋白为至少B1-大麦醇溶蛋白(例如包含如SEQ ID NO:78提供的氨基酸序列)和B3-大麦醇溶蛋白(例如包含如SEQ ID NO:79提供的氨基酸序列)。在另一个实施例中,C-大麦醇溶蛋白包含如SEQ ID NO:80提供的氨基酸序列。在又一个实施例中,D-大麦醇溶蛋白包含如SEQ ID NO:76提供的氨基酸序列。
在一个实施方案中,所述谷粒,或由所述谷粒生产的麦芽、麦芽汁、面粉或全麦具有野生型水平的小于10%、小于5%或小于2%的水平,或还缺乏;
i)包含如SEQ ID NO:57提供的氨基酸序列的γ-大麦醇溶蛋白,和/或
ii)包含如SEQ ID NO:52提供的氨基酸序列的燕麦蛋白样A蛋白。
其中小于10%、小于5%或小于2%的每一个水平相对于大麦品种Bomi、Sloop、Baudin、Yagan、Hindmarsh,或Commander的野生型大麦谷粒,或由所述谷粒生产的麦芽、麦芽汁、面粉或全麦。在一个实施例中,γ-大麦醇溶蛋白包含如SEQ ID NO:81提供的氨基酸序列。在又一个实施例中,燕麦蛋白样A蛋白包含如SEQ ID NO:84提供的氨基酸序列。
优选地,在上述实施方案中,γ-大麦醇溶蛋白是γ1-大麦醇溶蛋白和γ2-大麦醇溶蛋白。
在一个实施方案中,所述谷粒对于其中缺失大部分或全部B-大麦醇溶蛋白编码基因的Hor2基因座的等位基因是纯合的,或其中由所述谷粒生产的麦芽、麦芽汁、面粉或全麦包含含有其中缺失大部分或全部B-大麦醇溶蛋白编码基因的Hor2基因座的等位基因的DNA。
在另一个实施方案中,所述谷粒对于Hor3基因座处的编码D-大麦醇溶蛋白的基因的无效等位基因是纯合的,或其中由所述谷粒生产的麦芽、麦芽汁、面粉或全麦包含含有编码D-大麦醇溶蛋白的基因的无效等位基因的DNA,所述无效等位基因优选地包含终止密码子、剪接位点突变、移码突变、插入、缺失或编码截短的D-大麦醇溶蛋白,或其中缺失大部分或全部D-大麦醇溶蛋白编码基因。
在另一个实施方案中,截短的D-大麦醇溶蛋白在编码氨基酸编号150的三联体具有终止密码子。
在另一个实施方案中,所述谷粒对于导致所述谷粒缺乏C-大麦醇溶蛋白的大麦Lys3基因座的等位基因是纯合的,或其中由所述谷粒生产的麦芽、麦芽汁、面粉或全麦包含含有在Lys3基因座的等位基因的DNA。
在另一个实施方案中,相比于来自相应的野生型大麦植物的谷粒或由来自相应的野生型大麦植物的谷粒以相同方式生产的麦芽、麦芽汁、面粉或全麦,谷粒、麦芽、麦芽汁、面粉或全麦包含约1%或更少、约0.01%或更少、约0.007%或更少、约0.0027%或更少、约0.001%至约1%、约0.001%至约0.01%、约0.007%、或约0.0027%的水平的大麦醇溶蛋白。
在又一个实施方案中,所述谷粒来自具有野生型大麦植物的谷粒产量的至少60%、至少80%、至少90%、约60%至100%、约70%至100%、约80%至100%、约60%、约70%、约80%,或约90%的植物。
在另一个实施方案中,所述谷粒的平均重量为野生型大麦植物的谷粒的至少约70%、至少约80%、至少约90%、至少约95%,或约100%。
在又一个实施方案中,相比于相应的野生型大麦植物,所述谷粒、或由所述谷粒生产的麦芽、麦芽汁、面粉或全麦包含约10%或更少、约5%或更少、约2%或更少、约0.1%至约10%、或约0.1%至约10%的以下物质中一种或多种或所有:
i)包含如SEQ ID NO:53提供的氨基酸序列的B-大麦醇溶蛋白,
ii)包含如SEQ ID NO:54提供的氨基酸序列的B-大麦醇溶蛋白,
iii)包含如SEQ ID NO:55提供的氨基酸序列的C-大麦醇溶蛋白,以及
iv)包含如SEQ ID NO:56提供的氨基酸序列的D-大麦醇溶蛋白。
在另一个实施方案中,相比于相应的野生型大麦植物,所述谷粒,或由所述谷粒生产的麦芽、麦芽汁、面粉或全麦还包含约10%或更少、约5%或更少、约1%或更少、约0.1%至约10%,或约0.1%至约10%以下物质;
i)包含如SEQ ID NO:57提供的氨基酸序列的γ-大麦醇溶蛋白,和/或
ii)包含如SEQ ID NO:52提供的氨基酸序列的燕麦蛋白样A蛋白。
在另一个实施方案中,相比于相应的野生型大麦植物的量,所述谷粒,或由所述谷粒生产的麦芽、麦芽汁、面粉或全麦还包含约60%或更少的γ3-大麦醇溶蛋白,所述γ3-大麦醇溶蛋白包含其序列如SEQ ID NO:58提供的氨基酸,诸如包含其序列如SEQ ID NO:83提供的氨基酸的γ3-大麦醇溶蛋白。
野生型大麦植物的实例包括但不限于Bomi、Sloop、Baudin、Yagan、Hindmarsh,或Commander。
在另一个实施方案中,所述谷粒的淀粉含量为至少约50%(w/w)。更优选地,所述谷粒的淀粉含量为约50%至约70%(w/w)。
在另一个实施方案中,由所述谷粒生产的面粉的腹腔毒性为由相应的野生型大麦植物的谷粒生产的面粉小于约5%,或小于约1%。
在另一个实施方案中,平均谷粒重量为以下谷粒的至少1.05倍、至少1.1倍或1.05至1.3倍
i)所述谷粒对于缺失大部分或全部B-大麦醇溶蛋白编码基因的Hor2基因座的等位基因是纯合的,
ii)所述谷粒对于导致谷粒缺乏C大麦醇溶蛋白的大麦的Lys3基因座处的等位基因是纯合的,和
iii)所述谷粒对于编码全长蛋白的D大麦醇溶蛋白的野生型等位基因是纯合的。
在又一个实施方案中,所述谷粒来自具有比以下植物的谷粒产量高至少1.20倍,或至少1.35倍,或1.2至1.5倍,或1.2至2.0倍的谷粒产量的植物
i)所述植物对于缺失大部分或全部B-大麦醇溶蛋白编码基因的Hor2基因座的等位基因是纯合的,
ii)所述植物对于导致谷粒缺乏C大麦醇溶蛋白的大麦的Lys3基因座的等位基因是纯合的,和
iii)所述植物对于编码全长蛋白的D大麦醇溶蛋白的野生型等位基因是纯合的。
例如,关于上述两个实施方案,具有i)至iii)中界定的特征的谷粒可为WO 2009/021285中描述的G1*谷粒。
在另一个实施方案中,至少约50%、至少约60%、至少约70%、至少约80%、至少约90%,或至少约95%的大麦谷粒基因组与大麦野生型栽培品种诸如但不限于Sloop、Hindmarsh、Oxford或Marati me的基因组具有同一性。
在一个实施方案中,所述谷粒来自非转基因植物。
在一个替代实施方案中,所述谷粒来自转基因植物。
在另一个实施方案中,所述植物包含编码下调谷粒中至少一种大麦醇溶蛋白生产的多核苷酸的转基因。优选地,该实施方案的多核苷酸是反义多核苷酸、有义多核苷酸、催化多核苷酸、人造微小RNA或下调一种或优选地多种编码大麦醇溶蛋白的基因的表达的双链RNA分子。
在另一个实施方案中,所述方法包括由谷粒生产的面粉或全麦。
在另一个实施方案中,所述方法包括由谷粒生产的麦芽。
在一个实施方案中,基于麦芽的饮料为啤酒且所述方法包括使谷粒或由此来源的破裂谷粒发芽。在一个实施方案中,所述方法还包括将干燥的发芽谷粒分离成两种或多种胚乳部分、内皮细胞层部分、外壳部分、初生叶(acrospire)部分和麦芽细根部分;以及组合和混合预确定量的两种或多种所述部分。
在一个实施方案中,至少约50%的所述谷粒在吸涨后3天内发芽。
在另一个实施方案中,食物成分或基于麦芽的饮料成分为面粉、淀粉、麦芽或麦芽汁或其中所述食物为发酵或未发酵的面包、意大利面、面条、早餐谷物、零食食物、蛋糕、糕点或含有基于面粉沙司的食物。
在一个实施方案中,所述基于麦芽的饮料为啤酒或威士忌。
在一个实施方案中,所述食物或基于麦芽的饮料用于人食用。
在另一个实施方案中,在食用所述食物或饮料后,乳糜泻的至少一种症状不被患有所述疾病的受试者产生。
在另一方面,本发明提供生产包含约50ppm或更少的大麦醇溶蛋白的谷粒的大麦植物。
也提供本发明大麦植物的谷粒。
本发明的谷粒和/或本发明的大麦植物的谷粒可以包含一种或多种上述界定的特征。
在一个实施方案中,所述大麦谷粒能够产生本发明的大麦植物。
在一个实施方案中,已对所述谷粒进行加工以便其不能发芽。
在一个实施方案中,所述谷粒是无壳的。
在另一方面,本发明提供一种生产大麦谷粒的方法,所述方法包括;
a)种植本发明的大麦植物,
b)收获所述谷粒,和
c)任选地加工所述谷粒。
在一个实施方案中,该方法包括在至少一公顷面积的田间栽培至少10,000株植物。
在另一个实施方案中,本发明提供一种生产面粉、全麦、淀粉、麦芽、麦芽汁或从谷粒中获得的其它产品的方法,所述方法包括;
a)获得本发明的谷粒,以及
b)加工所述谷粒以生产面粉、全麦、淀粉、麦芽、麦芽汁或其它产品。
也提供由本发明的大麦植物或本发明的谷粒生产的产品。
在一个实施方案中,所述产品为食物成分、基于麦芽的饮料成分、食品或基于麦芽的饮品。
在一个实施方案中,所述基于麦芽的饮品为啤酒或威士忌。
在另一个实施方案中,该产品是非食品。实例包括但不限于薄膜、涂料、粘合剂、建筑材料和包装材料。
也提供使用本发明的方法生产的食物或基于麦芽的饮料。
在另一方面,提供包含一种或多种大麦谷粒蛋白和小于0.9ppm大麦醇溶蛋白的啤酒。
在一个实施方案中,所述啤酒包含至少约2%、至少约3%、至少约4%或至少约5%的乙醇。
在另一方面,本发明提供面粉或全麦,其包含一种或多种大麦谷粒蛋白和小于约50ppm或小于约20ppm的大麦醇溶蛋白。
在另一方面,本发明提供麦芽或麦芽汁,其包含一种或多种大麦谷粒蛋白和小于约50ppm或小于约20ppm的大麦醇溶蛋白。
在另一方面,本发明提供一种鉴定编码截短的D大麦醇溶蛋白的大麦D大麦醇溶蛋白基因的等位基因的方法,所述方法包括
i)自大麦植物获得包含核酸的样品,和
ii)分析样品以确定编码D大麦醇溶蛋白的开放阅读框的位置450存在或不存在鸟嘌呤残基,
其中存在鸟嘌呤表示D大麦醇溶蛋白基因编码截短的D大麦醇溶蛋白。
在一个实施方案中,步骤b)包括使用引物GGCAATACGAGCA GCAAAC(SEQ ID NO:66)和CCTCTGTCCTGGTTGTTGTC(S EQ ID NO:67)或其一种或两种变体扩增基因组DNA,以及使扩增产物与限制性内切酶KpnI接触,其中不存在裂解表示D大麦醇溶蛋白基因编码截短的D大麦醇溶蛋白。
在另一方面,本发明提供一种避免或降低受试者乳糜泻发病率或严重性的方法,所述方法包括使所述受试者经口施用本发明的食物或基于麦芽的饮料或本发明的谷粒,其中所述乳糜泻的发病率和严重性的降低与所述受试者经口施用相同量的来自野生型大麦谷粒的相应的食物或基于麦芽的饮料有关。
也提供本发明的食物或基于麦芽的饮料的用途,其用于制造向受试者经口施用以避免或降低乳糜泻的发病率或严重性的食物或饮料。
在另一方面,本发明提供一种用于鉴定可用于生产由患有乳糜泻的受试者食用的食物和/或基于麦芽的饮料的大麦谷粒的方法,其包括
a)获得一种或多种以下材料;
i)来自能够生产所述谷粒的植物的样品,
ii)谷粒,
iii)由所述谷粒生产的麦芽,和/或
iv)所述谷粒的提取物,
b)分析来自步骤a)的材料的B、C和D大麦醇溶蛋白水平、来源自B、C和D大麦醇溶蛋白的肽,和/或B、C和D大麦醇溶蛋白基因的等位基因,
c)筛选包含约50ppm或更少的大麦醇溶蛋白的用于生产由患有乳糜泻的受试者食用的食物和/或基于麦芽的饮料的谷粒。
在一个实施方案中,来自步骤a)的该材料包含基因组DNA且步骤b)包括鉴定编码以下一种或多种或所有的基因的等位基因的存在或不存在;
i)包含如SEQ ID NO:53提供的氨基酸序列的B-大麦醇溶蛋白,
ii)包含如SEQ ID NO:54提供的氨基酸序列的B-大麦醇溶蛋白,
iii)包含如SEQ ID NO:55提供的氨基酸序列的C-大麦醇溶蛋白,和
iv)包含如SEQ ID NO:56提供的氨基酸序列的D-大麦醇溶蛋白,
其中等位基因的不存在鉴定包含约50ppm或更少的大麦醇溶蛋白的谷粒。
本文的任意实施方案应加以必要的变通以应用于任意其它实施方案,除非另外特定说明。
本发明范围不受限于本文描述的特定实施方案(其仅用于示例的目的)。功能等效的产品、组合物和方法明确属于本文描述的本发明范围内。
在本说明书中,除非另外特定说明或上下文另外要求,对单步骤、物质组合物、步骤组或物质组合物组的引述应理解为涵盖那些步骤、物质组合物、步骤组或物质组合物组中一种及多种(即一种或多种)。
在下文中通过以下非限制实施例并参照附图来描述本发明。
附图简述
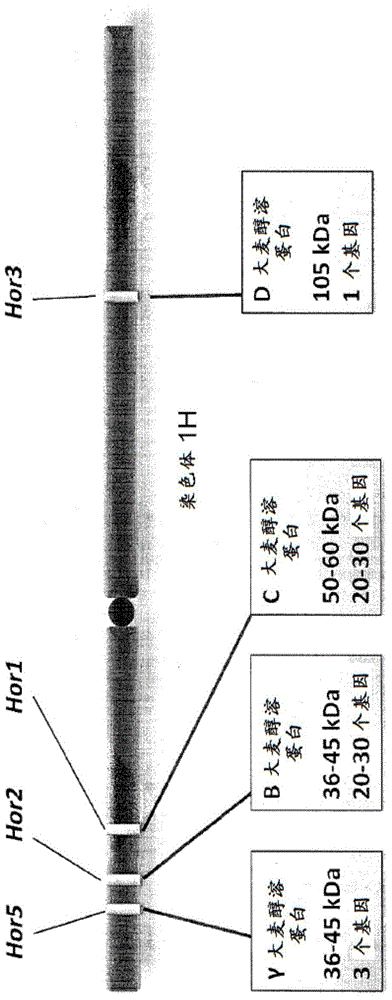
图1.大麦染色体1的示意图。
图2.啤酒中的相对大麦醇溶蛋白量。在啤酒中检测的表示最大量的大麦醇溶蛋白的所选肽或相关多肽的峰面积(A)QQCCQPLAQISEQAR(SEQ ID NO:5)表示燕麦蛋白样A蛋白;(B)VFLQQQCSPVR(SEQ ID NO:53)表示B1-大麦醇溶蛋白;(C)VFLQQQCSPVPMPQR(SEQ ID NO:54)表示B3-大麦醇溶蛋白;(D)ELQESSLEACR(SEQ ID NO:56)表示D-大麦醇溶蛋白;以及(E)QQCCQQLANINEQSR(SEQ ID NO:58)表示γ-大麦醇溶蛋白-3。这些代表性肽被用于说明野生型(Sloop)和分别由56和1508制造的两种大麦醇溶蛋白缺失啤酒以及在60种市售啤酒中主要大麦醇溶蛋白的相对量。看见不含谷蛋白的啤酒17、47、49-52、54的小峰面积是由于信号中的低水平噪声而不是由于大麦醇溶蛋白的检测。
图3.来自Sloop(野生型)和Ethiopia R118(无效)的D大麦醇溶蛋白氨基酸序列的对比。
图4.来自Sloop(野生型)和Ethiopia R118(无效)的D大麦醇溶蛋白氨基酸和编码核苷酸序列的对比。显示用于每个等位基因检测的引物位置连同只存在于野生型序列中的Kpn1裂解位点。
图5.通过MRM MS利用峰面积确定ULG3.0系的大麦醇溶蛋白含量且指示相对于Sloop(100%)的百分比。
图6.通过MRM MS利用峰面积确定ULG3.0系的大麦醇溶蛋白含量且指示相对于Sloop(100%)的百分比。
图7.通过MRM MS如界定的肽的峰面积确定ULG3.0啤酒的大麦醇溶蛋白含量。显示峰面积,以及相对于Sloop(100%)的百分比。
图8.相比于野生型品种Sloop、Baudin、Commander和Hindmarsh,通过MRM MS检测面粉中ULG3.0(T2-4-8)和ULG3.2候选系的大麦醇溶蛋白和大麦醇溶蛋白样蛋白。每个图显示来自每个大麦醇溶蛋白家族的代表性肽的总峰面积(3MRM转移)的平均值+/-SD。在每种情况中,所述肽(序列列于图中)映射燕麦蛋白样A蛋白B1-B3-、C-、D-、γ1-或γ3-大麦醇溶蛋白。Uniprot登录号在符号表中给出(例如F2EGD5用于第一实施例)。
序列表说明
SEQ ID NO:1和4-小麦α-麦醇溶蛋白肽。
SEQ ID NO 2、3、6至11、16至59和85-大麦的大麦醇溶蛋白肽。
SEQ ID NO:5–小麦燕麦蛋白样A肽。
SEQ ID NO:12至15-黑麦大麦醇溶蛋白肽。
SEQ ID NO:60至71-寡核苷酸引物。
SEQ ID NO:72–编码大麦cv.Sloop D-大麦醇溶蛋白的基因组区。
SEQ ID NO:73–编码大麦cv.Ethiopia R118D-大麦醇溶蛋白(空)的基因组区。
SEQ ID NO:74–大麦cv.Sloop D-大麦醇溶蛋白。
SEQ ID NO:75–大麦cv.Ethiopia R1118D-大麦醇溶蛋白。
SEQ ID NO:74–大麦cv.Sloop D-大麦醇溶蛋白。
SEQ ID NO:75–大麦cv.Ethiopia R118D-大麦醇溶蛋白。
SEQ ID NO:76–编码大麦cv.Sloop D大麦醇溶蛋白的开放阅读框。
SEQ ID NO:77–编码大麦cv.Ethiopia R118D大麦醇溶蛋白的开放阅读框。
SEQ ID NO:78–野生型大麦B1-大麦醇溶蛋白的实例(登录:Q40020)。
SEQ ID NO:79–野生型大麦B3-大麦醇溶蛋白的实例(登录:Q4G3S1)。
SEQ ID NO:80–野生型大麦C-大麦醇溶蛋白的实例(登录:Q40055)。
SEQ ID NO:81–野生型大麦γ1-大麦醇溶蛋白的实例(登录:P17990)。
SEQ ID NO:82–野生型大麦γ2-大麦醇溶蛋白的实例(登录:Q70IB4)。
SEQ ID NO:83–野生型大麦γ3-大麦醇溶蛋白的实例(登录:P80198)。
SEQ ID NO:84–野生型大麦燕麦蛋白样A蛋白的实例(登录:F2EGD5)。
具体实施方式
一般技术和定义
除非另有其它限定,如本文使用的所有技术和科学术语具有与本领域(例如,在植物培育、食物技术、细胞培养、分子遗传学、免疫学、蛋白化学和生物化学中)技术人员通常理解的相同含义。
除非另有说明,本发明利用的重组蛋白、细胞培养和免疫技术是本领域技术人员熟知的标准步骤。此类技术在以下来源的文献中描述和解释,诸如J.Perbal,A Practical Guide to Molecular Cloning,John Wiley和Sons(1984),J.Sambrook等人,Molecular Cloning:A Laboratory Manual,Cold Spring Harbour Laboratory Press(1989),T.A.Brown(编者),Essential Molecular Biology:A Practical Approach,第一1和2卷,IRL Press(1991),D.M.Glover和B.D.Hames(编者),DNA Cloning:A Practical Approach,第1-4卷,IRL Press(1995和1996),和F.M.Ausubel等人(编者),Current Protocols in Molecular Biology,Greene Pub.Associates and Wiley-Interscience(1988,包括所有至今的更新),Ed Harlow和David Lane(编者)Antibodies:A Laboratory Manual,Cold Spring Harbour Laboratory,(1988),及J.E.Coligan等人(编者)Current Protocols in Immunology,John Wiley&Sons(包括所有至今的更新)。
如本文使用的术语“大麦”是指大麦属(Genus Hordeum)的任意种类,包括其祖细胞以及由与其它种类杂交而生产的其后代。大麦的优选形式是大麦(Hordeum vulgare)种类。目前在世界范围内商业种植的大多数大麦栽培品种的谷粒已覆盖(去壳)颖果,在颖果中所谓的壳(其为外部鞘膜和内部托苞)在成熟期连接果皮表皮。其它栽培品种(称为叫做去壳或裸大麦)为免脱粒且在脱粒过程中很容易除去壳。尽管精磨后可使用去壳谷粒,但是优选裸大麦谷粒用于人食用,然而,优选去壳大麦谷粒用于酿造工业和动物饲料。去壳大麦的特点由位于染色体7H长臂的命名为nud的单隐性基因控制(Kikuchi等人,2003)。
乳糜泻或脂泻病是小肠自身免疫疾病,其一般发生在早期婴儿期后的所有年龄组的易患个体中。它影响大约1%印-欧群体,但是其明显确诊不足。乳糜泻由与麦醇溶蛋白的反应引起,麦醇溶蛋白为一种在小麦(和包括其它栽培品种诸如大麦和黑麦的小麦族相似蛋白)中发现的谷蛋白。暴露于麦醇溶蛋白时,酶组织转谷氨酰胺酶修饰所述蛋白,且免疫系统与肠组织交叉反应,引起炎性反应。这导致小肠内壁平整,其干扰营养吸收。唯一有效的治疗是终生无谷蛋白饮食。这种症状有几个其它称谓,包括:脂泻病(带结扎线)、腹腔口炎性腹泻、非热带性口炎性腹泻、特有口炎性腹泻、麸质肠病或谷胶肠病,和谷蛋白不耐受症。乳糜泻的症状因人而异。乳糜泻的症状可以包括以下中的一种或多种:排气、反复腹胀和疼痛、慢性腹泻、便秘、苍白、恶臭,或脂肪粪便、体重减轻/体重增加、疲劳、未知因素的贫血(引起贫血的低量红血细胞)、骨或关节疼痛、骨质疏松、骨质减少、行为改变、腿麻木(由于神经损伤)、肌肉痉挛、癫痫发作、停经(通常因过度体重减轻)、不育、反复性流产、发育迟缓、婴儿不能茁壮成长、流产、口腔内苍白溃疡、所谓的口疮性溃疡、牙齿变色或牙釉质流失和所谓的疱疹样皮炎的瘙痒皮疹。一些更常见的症状包括:疲倦、间歇性腹泻、腹痛或腹部绞痛、消化不良、胃肠胀气、腹胀和体重减轻。乳糜泻可例如如WO 01/025793中描述进行诊断。
如本文使用的术语“在Hor3基因座处的编码D-大麦醇溶蛋白的基因的无效等位基因”是指不编码D-大麦醇溶蛋白的蛋白的此位的任意等位基因,或如果蛋白被编码,则它对患有乳糜泻的受试者无免疫原性(诸如图3中提供的截短的D-大麦醇溶蛋白)。
如本文使用,本文在引述物质的上下文中使用的术语“缺乏”意指该物质不存在本发明的大麦谷粒或其衍生产品,或当使用本领域已知的方法对该物质实施测定时,该物质在本发明的谷粒或产品中探测不到。也就是说,该物质可以不足以被检测到的水平或在测定那个物质的标准差内存在。例如,在引述大麦醇溶蛋白的上下文中,术语“缺乏”意指在测定诸如例如MRM MS测定、ELISA测定或2D-凝胶电泳测定中探测不到特定大麦醇溶蛋白,诸如本文示例。缺乏的物质不能在一种类型的测定或多种类型的测定中探测到。应理解,被认为在本发明的谷粒或产品中缺乏的物质在相应的野生型或产品中是存在的,如本领域已知测定法所容易确定的。
如本文使用的术语“大麦醇溶蛋白”例如当用于短语“约50ppm或更少的大麦醇溶蛋白”和相似短语时是指包括B-、C-、D-和γ-大麦醇溶蛋白的总大麦醇溶蛋白。
术语“种子”和“谷粒”在本文相互交换使用。“谷粒”一般是指成熟、收获的谷粒,但也可指根据上下文诸如例如研磨或抛光的加工后(其中大多数谷粒保持完整)或吸涨或发芽后的谷粒。成熟谷粒通常具有小于约18-20%的含水量。野生型大麦谷粒(全谷粒)一般含有9-12%的蛋白且该蛋白的约30-50%是谷醇溶蛋白,通常为35%,所以野生型大麦谷粒具有约3-4重量%的谷醇溶蛋白。谷醇溶蛋白几乎完全在胚乳中,胚乳为约70重量%的全谷粒。
如本文使用的术语“收获指数”是指收获的谷粒重量以占该植物总重量的百分比计。
如本文使用的术语“相应的野生型”大麦植物是指包含至少50%,更优选至少75%,更优选至少95%,更优选至少97%,更优选至少99%,以及甚至更优选99.5%的本发明的植物的基因型的植物,但是生产具有非修饰的大麦醇溶蛋白水平的谷粒。在一个实施方案中,所述“相应的野生型”大麦植物是用于植物育种实验以引入在谷粒中降低大麦醇溶蛋白产生的基因变体的栽培品种。在另一个实施方案中,所述“相应的野生型”大麦植物是引入了在谷粒中降低大麦醇溶蛋白产生的转基因的亲本栽培品种。在另一个实施方案中,所述“相应的野生型”大麦植物是在申请日用于以下大麦谷粒的商业生产的栽培品种,诸如但不限于Bomi、Sloop、Carlsberg II、K8、L1、Vlamingh、Stirling,Hamelin、Schooner、Baudin、Commander、Gairdner、Buloke、WI3586-1747、WI3416、Flagship、Cowabbie、Franklin、SloopSA、SloopVic、Quasar、VB9104、Grimmett、Cameo*Arupo 31-04、Prior、Schooner、Unicorn、Harrington、Torrens、Galleon、Morex、Dhow、Capstan、Fleet、Keel、Maritime、Yarra、Dash、Doolup、Fitzgerald、Molloy、Mundah、Oxford、Onslow、Skiff、Unicorn、Yagan、Chebec、Hindmarsh、Chariot、Diamant、Korál、Rubín、Bonus、Zenit、Akcent、Forum、Amulet、Tolar、Heris、Maresi、Landora、Caruso、Miralix、Wikingett Brise、Caruso、Potter、Pasadena、Annabell、Maud、Extract、Saloon、Prestige、Astoria、Elo,、Cork、Extract、Laura。在一个实施方案中,所述“相应的野生型”大麦植物因为其包含编码功能性大麦醇溶蛋白的蛋白(包括由Hor2、Hor1、Hor3和Hor5位编码的B、C、D和γ-大麦醇溶蛋白)的功能性大麦醇溶蛋白基因全补体,所以产生具有未修饰的大麦醇溶蛋白水平的谷粒。
如本文使用的术语“一种或多种大麦谷粒蛋白”是指由大麦谷粒而非大麦醇溶蛋白产生的天然存在蛋白。此类蛋白的实例是本领域的技术人员已知的。特定的实例包括但不限于大麦白蛋白诸如9kDa脂质转运蛋白1(LTP1)(参见Douliez等人(2000)综述且以Swiss-prot登录号P07597为例)、α-淀粉酶胰蛋白酶抑制剂(CMd、CMb、CMa)、蛋白Z(参见Brandt等人(1990)和Genbank登录号P06293),和Colgrave等人(2012)鉴定的蛋白,包括其加工(成熟)形式,以及变性形式和/或因生产本发明的麦芽、面粉、全麦、食物或基于麦芽的饮料而产生的其片段。
如本文使用的“谷粒的平均重量”优选地通过从植物(或在相同条件下种植的遗传上相同的植物)中获得至少25、至少50或至少100,更优选约100单独谷粒和确定所述谷粒的平均重量来确定。
如本文使用的术语“麦芽”用于指大麦麦芽,“面粉”指大麦面粉,“全麦”指大麦全麦,以及“啤酒”指使用大麦作为提供可发酵碳水化合物的其主要成分而生产的啤酒,除了明确阐明麦芽、面粉、全麦或啤酒来自其它大麦来源的情况。如本文使用的“麦芽汁”是指在啤酒或威士忌酿造期间从淀粉糖化过程中提取的液体。麦芽汁含有通过酿造酵母发酵以产生酒精的糖。更具体地是,本发明的麦芽、麦芽汁、面粉、啤酒、全麦、食品等的来源为来自大麦谷粒的加工(例如研磨和/或发酵)。本发明的谷粒、麦芽、麦芽汁、面粉、全麦或啤酒可与不是从大麦来源而来的谷粒、麦芽、麦芽汁、面粉、全麦或啤酒混合或掺合。这些术语包括从包括大麦的谷粒混合物中生产的麦芽、麦芽汁、面粉、啤酒、全麦、食品等。在一个优选的实施方案中,至少10%或至少50%的用于生产麦芽、麦芽汁、面粉、啤酒、全麦、食品等的谷粒是大麦谷粒。
如本文使用的术语“植物”作为名词是指整体植物诸如例如种植在田间中用于商业大麦生产的植物。“植物部分”是指植物的植物性结构(例如叶、茎)、根、花器官/结构、种子(包括胚芽、胚乳和种皮)、植物组织(例如维管组织、基本组织等)、细胞、淀粉颗粒或其后代。
“转基因植物”、“基因修饰的植物”或其变异是指含有在相同种类的野生型植物、变体或栽培品种中未发现的基因构造(“转基因”)的植物。本文所指的“转基因”具有生物技术领域常用的含义,且包括通过重组DNA或RNA技术生产的或改变的且被引入植物细胞的基因序列。所述转基因可包括来源自植物细胞的遗传序列。通常,所述转基因通过人为操作诸如例如通过转化但是可使用本领域技术人员认识到的任意方法引入植物中。
“核酸分子”指多核苷酸诸如例如DNA、RNA或寡核苷酸。其可为基因组或合成来源、双链或单链,以及与碳水化合物、脂类、蛋白或其它材料组合以实施本文界定的具体活性的DNA或RNA。
如本文使用的“可操作地连接”是指两种或多种核酸(例如DNA)片段之间的功能性关系。通常,其指转录调控元件(启动子)到转录序列的功能性关系。例如,如果启动子在适当细胞中刺激或调节编码序列的转录,则其可操作地连接编码序列,诸如本文界定的多核苷酸。一般地,可操作地连接转录序列的启动子转录调节元件与转录序列物理上毗邻,即它们是顺式作用。然而,一些转录调节元件诸如增强子不需要物理上毗邻或紧邻它们增强其转录的编码序列。
如本文使用的术语“基因”是取它的广义含义且包括脱氧核糖核苷酸序列,所述序列包含结构基因的蛋白编码区且包括邻近编码区5’和3’端的序列(其距任一端至少约2kb),且其涉及基因表达。位于编码区5’且在mRNA上存在的序列称为5’非转录序列。位于编码区3’或下游且在mRNA上存在的序列称为3’非转录序列。术语“基因”涵盖cDNA和基因的基因组形式。基因的基因组形式或克隆含有可被称为“内含子”或“干预区”或“干预序列”的非编码序列中断的编码区。内含子是被转录到核RNA((hnRNA)的基因片段;内含子可以含有调节元件诸如增强子。因为将内含子从核或初级转录物中除去或“切除”;所以在信使RNA(mRNA)转录物中不存在内含子。转录期间mRNA功能为在新生多肽中规定氨基酸的序列或顺序。术语“基因”包括编码所有或部分本文描述的本发明的蛋白的合成或融合分子和与上述任一互补的核苷酸序列。
如本文使用的术语“其它食物或饮料成分”是指当以食物或饮料的一部分提供时,适合于动物食用的任意物质,优选的是适合人食用的任意物质。实例包括但不限于来自其它植物种类的谷粒、糖等,但排除水。
本发明的植物通常具有一种或多种基因变异,每一变异导致至少一种大麦醇溶蛋白,更优选地至少B、C和D-大麦醇溶蛋白水平的降低。如本文使用的术语“其中每一变异导致至少一种大麦醇溶蛋白水平降低的基因变异”是指降低大麦醇溶蛋白产生的大麦植物的任意多机组合形式。基因变异可以包括例如大麦醇溶蛋白基因或其部分的缺失,或降低大麦基因转录的突变。此类基因变异的实例呈现在56、527和1508和Ethiopia R118中。因此,此类植物可用作亲本植物以产生本发明的植物。本发明的植物可从这些大麦突变体任意之间杂交的后代中产出。在一个优选的实施方案中,本发明的植物不是来自包含存在于这些系中的hor2和lys3突变的56和1508之间杂交的后代且其是用于编码D-大麦醇溶蛋白的基因的野生型。在一个实施方案中,所述植物编码包含其序列如SEQ ID NO:58提供的氨基酸的γ3-大麦醇溶蛋白,诸如包含其序列如SEQ ID NO:83提供的氨基酸的γ3-大麦醇溶蛋白。比如,所述植物可以具有功能野生型γ3-大麦醇溶蛋白基因诸如大麦栽培品种Bomi、Sloop、Baudin、Yagan、Hindmarsh,或Commander的γ3-大麦醇溶蛋白基因。在一个实施方案中,所述植物不是来自527和1508之间杂交的后代。
除非有相反的说明,否则本文使用的短语“约”是指据此讨论的值的合理范围。在优选的实施方案中,术语“约”是指规定值的+/-10%,更优选+/-5%,更优选+/-1%。
除非有特定说明,否则关于重量和百分比,单位为w/w。
术语“和/或”,例如“X和/或Y”应理解为“X和Y”或“X或Y”的意思且应被理解为明确支持这两种意思或任一意思。
在该说明书中,词“包含”或其变形将理解为包括阐述的元素、整数或步骤、或元素、整数或步骤的组,但是不排除任何其它元素、整数或步骤、或元素或整数或步骤的组。
谷醇溶蛋白和大麦醇溶蛋白
谷物谷醇溶蛋白(小麦中称为麦醇溶蛋白、大麦中称为大麦醇溶蛋白、黑麦中称为裸麦醇溶蛋白、燕麦中称为燕麦蛋白,和玉米中称为玉米醇溶蛋白)在所有谷物谷粒中是主要的胚乳贮存蛋白,除了燕麦和大米(Shewry和Halford,2002)。大麦醇溶蛋白占大麦种子中总蛋白的35-50%。(Jaradat,1991)。按迁移率降序,将它们分为四组:A(也称为γ大麦醇溶蛋白)、B、C和D(Field等人,1982)。B、C、D和γ-大麦醇溶蛋白分别由染色体1H上Hor2、Hor1、Hor3和Hor5位编码,染色体1H在图1中给出。
B大麦醇溶蛋白是主要的蛋白部分,与C大麦醇溶蛋白的硫含量不同(Kreis和Shewry,1989)。B大麦醇溶蛋白占全部的70-80%且C大麦醇溶蛋白占10-20%(Davies等人,1993)。一般认为A大麦醇溶蛋白不是贮存部分,而D大麦醇溶蛋白与高分子量麦谷蛋白同源。大麦醇溶蛋白,连同其余的相关谷物谷醇溶蛋白不在合子胚芽自身中表达,不像其它贮存蛋白诸如油菜籽蛋白(napin);它们被认为在种子发育的中至后期只在淀粉质胚乳中表达。
大麦的大麦醇溶蛋白氨基酸序列和编码它们的基因的实例提供于WO 2009/021285中。
制麦芽
本发明提供的基于麦芽的饮料涉及酒精饮料(包括蒸馏饮料)和将麦芽用作它们的起始材料的一部分或全部而生产的非酒精饮料。实例包括啤酒、发泡酒(happoshu,低麦芽啤酒饮料)、威士忌、基于麦芽的低酒精饮料(例如含有低于1%酒精的基于麦芽的饮料)和非酒精饮料。
制麦芽是受控的浸泡和发芽,随后干燥大麦谷粒的过程。事件的顺序对于引起谷粒修饰的众多酶的合成很重要,其为原则上解聚合沉寂胚乳细胞壁和调动谷粒营养的过程。在后续的干燥过程中,由于化学褐变反应产生香味和颜色。尽管麦芽的首要用途是生产饮料,但它还可以用于其它工业过程,例如在烘培工业中作为酶源,或在食物工业中作为调味剂和着色剂,例如作为麦芽或作为麦芽面粉,或间接地作为麦芽糖浆等。
在一个实施方案中,本发明涉及产生麦芽组合物的方法。所述方法优选地包含以下步骤:
(i)提供本发明的大麦植物的谷粒,
(ii)浸泡所述谷粒,
(iii)在预确定的条件下使浸泡的谷粒发芽,和
(iv)干燥所述发芽谷粒。
例如,所述麦芽可由Hoseney(Principles of Cereal Science and Technology,第二版,1994:American Association of Cereal Chemists,St.Paul,Minn.)中描述的任意方法产生。然而,用于产生麦芽的任意其它合适方法也可用于本发明,诸如用于产生特殊麦芽的方法,包括但不限于烘烤麦芽的方法。一种非限制性实例描述于实施例6中。
可只使用由本发明的大麦植物生产的谷粒或包括其它谷粒的混合物制备麦芽。
麦芽主要用于酿造啤酒,但也用于生产蒸馏的烈性酒。酿造包括麦芽汁产生、主要和二次发酵和后处理。首先研磨麦芽,水中搅拌且加热。在此“淀粉糖化”期间,制麦芽时激活的酶将籽粒的淀粉降解为发酵糖。澄清产生的麦芽汁,加入酵母,发酵该混合物且进行后处理。
在另一个实施方案中,麦芽汁组合物可由麦芽制备。所述麦芽汁可为第一和/或第二和/或进一步的麦芽汁。一般来讲,麦芽汁组合物将具有高含量的氨基氮和可发酵碳水化合物,主要是麦芽糖。通常,通过用水孵育麦芽即通过淀粉糖化制备麦芽汁。在淀粉糖化期间,麦芽/水组合物可补充有额外的富含碳水化合物的组合物(例如大麦、玉米或大米辅助剂)。未发芽的谷物辅助剂常不含有活性酶,且因此依靠麦芽或外来酶提供糖转化所必要的酶。
一般来讲,麦芽汁产生过程的第一步是研磨麦芽以使水在淀粉糖化阶段水可进入谷粒颗粒,其基本上是用酶促解聚底物而制麦芽过程的延伸。淀粉糖化期间,研磨的麦芽用液体部分诸如水孵育。保持温度恒定(等温淀粉糖化)或逐渐升温。在任一情况下,将制麦芽和淀粉糖化中产生的可溶底物提取到所述液体部分中,然后通过过滤将其分离为麦芽汁和残留固体颗粒(称为废谷粒)。该麦芽汁也称为第一麦芽汁。过滤后,获得第二麦芽汁。通过重复该步骤可制备进一步的麦芽汁。用于制备麦芽汁的合适步骤的非限制性实例描述于Hoseney(同上)中。
该麦芽汁组合物可通过用一种或多种合适酶(诸如酶组合物或酶混合物组合物,例如Ultraflo或Cereflo(Novozymes))孵育本发明的大麦植物或其部分而制备。该麦芽汁组合物也可通过使用麦芽和未发芽的大麦植物或其部分的混合物制备,在所述制备期间任选地加入一种或多种合适的酶。另外,在麦芽汁发酵期间可加入特异性破坏涉及乳糜泻的毒性氨基键的脯氨酰基-肽链内切酶来降低残留大麦醇溶蛋白的毒性(De Angelis等人,2007;Marti等人,2005;Stepniak等人,2006)。
谷粒加工
使用本领域已知的任意技术可加工本发明的大麦谷粒以生产食物成分、饮料成分、食物或饮料,或非食品。
在一个实施方案中,该产品是全谷粒面粉(超细研磨的全谷粒面粉,诸如由约100%的谷粒制造的超细研磨的全谷粒面粉、全谷粒面粉或面粉)。该全谷粒面粉包括精制面粉组分(精制面粉或精制面粉)和粗糙部分(超细研磨的粗糙部分)。
所述精制面粉可为例如通过粉碎和筛选干净的大麦制备的面粉。为了包括在精制大麦面粉目录中,食物药品管理局(FDA)要求面粉要符合一定的粒径标准。精制面粉的粒径被描述成其不小于98%通过具有开口不大于指定的“212微米(U.S.线70)”金属编织网布的那些开口的布的面粉。
所述粗糙部分包括麸皮和胚芽中的至少一种。比如,胚芽是大麦籽粒中发现的胚胎植物。胚芽包括脂类、纤维、维他命、蛋白、矿物质和植物营养物,诸如类黄酮。麸皮包括多种细胞层且具有显著量的脂类、纤维、维他命、蛋白、矿物质和植物营养物,诸如类黄酮。进一步地,所述粗糙部分可包括糊粉层,所述糊粉层也包括脂类、纤维、维他命、蛋白、矿物质和植物营养物,诸如类黄酮。所述糊粉层(虽然技术上视为胚乳的一部分)展示许多与麸皮相同的特征且因此通常在研磨过程期间与麸皮和胚芽一起除去。所述糊粉层含有蛋白、维他命和植物营养物,诸如阿魏酸。
进一步地,所述粗糙部分可以与所述精制面粉组分掺合。优选地,所述粗糙部分与所述精制面粉组分均匀掺和。均匀掺合所述粗糙部分和所述精制面粉组分可以有助于降低在运输期间颗粒的尺寸层化。所述粗糙部分可以与所述精制面粉组分混合以形成全谷粒面粉,因此相比于精制面粉,提供具有增加的养分价值、纤维含量和抗氧化剂能力的全谷粒面粉。例如,所述粗糙部分或全谷粒面粉可在烘烤食物、零食产品和食品中以各种量使用以取代精制或全谷粒面粉。本发明的全谷粒面粉(即,超细研磨的全谷粒面粉)也可直接市售给消费者以食用其家制烘烤产品。在一个示例性实施方案中,所述全谷粒面粉的造粒情况为以便于全谷粒面粉的98重量%的颗粒小于212微米。
在另外的实施方案中,为了稳定全谷粒面粉和/或粗糙部分,使在全谷粒面粉和/或粗糙部分的麸皮和胚芽中所见的酶失活。本发明预期失活也意指抑制、变性等。稳定是一种使用蒸汽、加热、辐射或其它处理以使麸皮和胚芽层中所见的酶失活的过程。麸皮和胚芽中天然存在的酶将催化面粉中化合物的变化,不利地影响面粉的烹饪特性和保质期。失活酶不催化面粉中所见的化合物变化,从而稳定的面粉保持它的烹饪特性且具有较长的保质期。例如,本发明可施用双-蒸汽研磨技术以粉碎粗糙部分。一旦分离和稳定粗糙部分,则接下来通过粉碎机(优选凹口研磨机)磨碎所述粗糙部分以形成具有粒径分布小于或等于约500微米的粗糙部分。在一个示例性实施方案中,正常操作所述凹口研磨机的尖端速度一般在115m/s与144m/s之间,高的尖端速度生成热量。该过程中生成的热量和气流导致所述粗糙部分的微生物负载降低。在另一个实施方案中,在凹口研磨机中粉碎前,所述粗糙部分可具有95,000菌落形成单位/克(cfu/g)的平均需氧平板计数和1,200cfu/g平均大肠杆菌计数。通过凹口研磨机后,所述粗糙部分可具有10,000cfu/g平均需氧平板计数和900cfu/g平均大肠杆菌计数。因此,在本发明的粗糙部分中微生物负载可明显降低。筛查后,可将具有粒径大于500微米的任意磨碎的粗糙部分返回到该过程中进一步研磨。
在另外的实施方案中,所述全谷粒面粉或所述粗糙部分可以是食品的组分。例如,食品可以是百吉饼、饼干、面包、馒头、羊角面包、团子(dumpling)、英国松饼、松饼、皮塔面包、速制面包、冷藏/冷冻面团产品、面团、烘培豆、墨西哥玉米煎饼、辣椒食物(chili)、玉米卷、玉米粉蒸肉、玉米粉圆饼、菜肉馅饼、即食谷物、即食膳食、馅料、微波膳食、巧克力蛋糕、蛋糕、奶酪蛋糕、咖啡蛋糕、曲奇饼干、甜点、油酥点心、甜卷饼、块状糖、派皮、派馅料、婴儿食物、奶油面包粉、糊状物、面包制品、肉汁粉、肉增量剂、素肉、调味粉、汤粉、肉汁、奶油炒面糊、色拉酱调料、汤、酸奶油、面条、披萨、日本拉面、炒面、拌面、冰淇淋包容物、雪糕、冰淇淋蛋卷、冰淇淋三明治、薄脆饼干、油煎面包块、油炸圈饼、蛋卷、挤压点心、水果和谷粒条、微波零食产品、营养棒、薄烤饼、半烘烤面包产品、脆饼干、布丁、基于格兰诺拉麦片的产品、碎零食、零食食物、零食粉、华夫饼干、比萨饼皮、动物食物或宠物食物。
在替代的实施方案中,所述全谷粒或粗糙部分可以是营养补充剂的组分。比如,该营养补充剂可以是加入饮食中的含有一种或多种成分的产品,通常包括:维他命、矿物质、草药、氨基酸、酶、抗氧化剂、草药、香料、益生菌、提取物、益生元和纤维。本发明的全谷粒面粉或粗糙部分包括维他命、矿物质、氨基酸、酶和纤维。比如,所述粗糙部分含有浓缩量的膳食纤维以及其它对饮食健康很重要的基础营养,诸如B-维他命、硒、铬、锰、镁和抗氧化剂。例如,22克本发明的粗糙部分递送个体每日纤维推荐量的33%。进一步地,14克是需要递送个体每日纤维推荐量的20%的全部量。因此,对于个体纤维食用需求,所述粗糙部分是优良的补充源。因此,在本发明的实施方案中,所述全谷粒面粉或粗糙部分可以是营养补充剂的组分。营养补充剂可以包括将有助于个体整体健康的任何已知营养成分,实例包括但不限于维他命、矿物质、其它纤维组分、脂肪酸、抗氧化剂、氨基酸、肽、蛋白、黄体素、核糖、Ω-3脂肪酸,和/或其它营养成分。
在另外的实施方案中,所述全谷粒面粉或粗糙部分可以是纤维补充剂或其组分。目前许多纤维补充剂诸如欧车前果壳、纤维素衍生物和水解的瓜尔豆胶具有超出其纤维含量的有限营养值。另外,许多纤维补充剂具有不期望的质地且口味不佳。由所述全谷粒面粉或粗糙部分制造的纤维补充剂可有助于递送纤维以及蛋白和抗氧化剂。纤维补充剂可以但不限于以下方式递送:即时混合饮料、即饮饮料、营养条、威化饼、曲奇饼干、薄饼干、凝胶丸粒(gel shot)、胶囊、咀嚼物、咀嚼片和丸剂。一个实施方案以风味奶昔(flavored shake)或麦芽类型饮料的形式递送纤维补充剂,该实施方案作为用于儿童的纤维补充剂特别具有吸引力。
在另一个实施方案中,研磨过程可用于制造多谷粒面粉、多大麦面粉或多谷粒粗糙部分。例如,可磨碎来自一种大麦类型的麸皮和胚芽且与另一种大麦类型的磨碎胚乳或全谷粒大麦面粉掺合。可替代地,可磨碎一种谷粒类型的麸皮和胚芽且与另一种谷粒类型的磨碎胚乳或全谷粒面粉掺合。在另一个实施方案中,来自第一类型的大麦或谷粒的麸皮和胚芽可与来自第二类型的大麦或谷粒的麸皮和胚芽掺合以生产多谷粒粗糙部分。本发明预期涵盖混合一种或多种谷粒的麸皮、胚芽、胚乳和全谷粒面粉中一种或多种的任意组合。此多谷粒、多大麦途径可用于制造定制化面粉且利用多种类型的谷粒或大麦的品质和营养含量制造一种面粉。
本发明的全谷粒面粉可通过多种研磨过程生产。一个示例性实施方案涉及在无需将谷粒胚乳、麸皮和胚芽分离成单独流的情况,将谷粒粉碎成单流。将干净和回火的谷粒运输至第一通道粉碎器中,诸如锤磨机、辗压机、钉式研磨机、冲击锤磨机、圆盘粉碎机、空气磨碎机、凹口研磨机等。在一个实施方案中,粉碎机可以为凹口研磨机。粉碎后,排放谷粒并将其运输至筛粉机。可使用本领域已知的任意筛粉机筛选磨碎的颗粒。通过筛选机筛网的材料是本发明的全谷粒面粉且不需要进一步加工。留在筛网上的材料称为第二部分。第二部分需要额外降低颗粒。因此,可将此第二部分运输至第二通道粉碎机。粉碎后,可将第二部分运输至第二筛粉机。通过第二筛选机筛网的材料是本发明的全谷粒面粉。留在筛网上的材料称为第四部分且需要进一步加工以降低粒径。将在第二筛选机筛网上的第四部分运回第一通道粉碎机或第二通道粉碎机以通过反馈环路进一步加工。在本发明的一个替代实施方案中,该过程可以包括多个第一通过粉碎机以提供更高的系统能力。
预期本发明的全谷粒面粉、粗糙部分和/或谷粒产品可以通过本领域已知的任意研磨过程生产。进一步地,预期本发明的全谷粒面粉、粗糙部分和/或谷粒产品可以通过众多其它过程修饰或加强,诸如:发酵、速溶化、挤压、密封、烘烤等。
下调大麦醇溶蛋白生产的多核苷酸
在一个实施方案中,本发明和/或用于本发明的方法的谷粒来自转基因大麦植物,所述大麦植物包含编码下调谷粒中至少一种大麦醇溶蛋白生产的多核苷酸的转基因。此类多核苷酸的实例包括但不限于反义多核苷酸、有义多核苷酸、催化多核苷酸、人造微小RNA或双链RNA分子。当存在于谷粒中时,这些多核苷酸中每一个导致用于翻译的大麦醇溶蛋白mRNA下降。
反义多核苷酸
术语“反义多核苷酸”应被理解为意指与编码大麦醇溶蛋白且能够干扰转录后事件诸如mRNA翻译的特异性mRNA分子的至少一部分互补的DNA或RNA分子或其组合。反义法的用途是本领域熟知的(参见例如G.Hartmann和S.Endres,Manual of Antisense Methodology,Kluwer(1999))。植物中的反义技术的使用由Bourque(1995)和Senior(1998)综述。Senior(1998)阐述,反义法是目前用于操控基因表达的非常完善的技术。
本发明的大麦植物中的反义多核苷酸在生理条件下将与靶多核苷酸杂交。如本文使用的术语“在生理条件下杂交的反义多核苷酸”意指多核苷酸(其完全或部分单链)至少能够与编码蛋白(诸如,在大麦细胞中正常条件下的大麦的大麦醇溶蛋白)的mRNA形成双链多核苷酸。
反义分子可以包括对应于结构基因的序列或有效控制基因表达或剪接事件的序列。例如,反义序列可对应于本发明的基因的靶向的编码区,或5’-未翻译区(UTR)或3’-UTR或这些组合。其可以与内含子序列部分互补(转录期间或之后可切除内含子),优选地仅与靶基因的外显子序列互补。考虑到UTR的通常较大的趋异性,靶向这些区为基因抑制提供较大特异性。
反义序列的长度是至少19个连续核苷,优选至少50个核苷,更优选地是至少100、200、500或1000个核苷。可使用与整个基因转录物互补的全长序列。最优选的长度是100-2000个核苷。反义序列与靶向的转录物的同一性程度应为至少90%且更优选95-100%。反义RNA分子理所当然地可包含用以稳定该分子的不相关序列。
催化多核苷酸
术语催化多核苷酸/核酸是指特异性识别特定底物并催化该底物化学修饰的DNA分子、含DNA的分子(本领域中也称为“脱氧核酶”)、或RNA或含RNA的分子(也称为“核酶”)。催化核酸中的核酸碱基可以是碱基A、C、G、T(和U,对于RNA而言)。
通常,催化核酸含有特异性识别靶核酸的反义序列和核酸切割酶促活性(在本文也称为“催化结构域”)。在本发明中特别有用的核酶类型是锤头状核酶(Haseloff and Gerlach 1988,Perriman等人,1992)和发夹状核酶(hairpin ribozyme)(Shippy等人,1999)。
可使用本领域熟知的方法化学合成本发明的大麦植物的核酶和编码核酶的DNA。所述核酶也可由可操作地与RNA聚合酶启动子连接的DNA分子(一旦转录,则产生RNA分子)制备,例如,用于T7RNA聚合酶或SP6RNA聚合酶的启动子。当载体也含有可操作地与DNA分子连接的RNA聚合酶启动子时,可通过用RNA聚合酶和核苷酸孵育体外产生核酶。在一个单独的实施方案中,可将DNA插入表达盒或转录盒中。合成后,可通过连接到能够稳定核酶并耐受RNA酶的DNA分子来修饰RNA分子。
正如本文描述的反义多核苷酸,催化多核苷酸也应能够在“生理条件下”(即大麦细胞中的那些条件)杂交靶核酸分子(例如编码大麦的大麦醇溶蛋白的mRNA)。
RNA干扰
RNA干扰(RNAi)特别用于特异性抑制具体蛋白产生。虽然不希望被理论所限制,但是Waterhouse等人(1998)已提供了dsRNA(双链体RNA)可用于减少蛋白产生的机制的模型。这种技术依赖于dsRNA分子的存在,该分子含有与目标基因的mRNA或其部分基本上一致的序列,在此情况中为根据本发明编码多肽的mRNA。常规地,该dsRNA可在重组载体或宿主细胞中由单启动子产生,其中有义和反义序列通过无关序列相接,这使得有义和反义序列杂交与形成环结构的无关序列形成dsRNA分子。用于本发明的合适的dsRNA分子的设计和产生是在本领域技术人员熟知的,尤其考虑Waterhouse等人(1998)、Smith等人(2000)、WO 99/32619、WO 99/53050、WO 99/49029和WO 01/34815。
在一个实例中,引入的DNA至少部分地指引双向(双链)RNA产物的合成,与将待失活的靶基因有同源性。因此,该DNA包含正义和反义序列两者,当被转录成RNA时,所述正义和反义序列可以杂交以形成双链RNA区。在优选的实施方案中,所述正义序列和反义序列被包含内含子的间隔区隔开,在转录成RNA时,所述内含子被切除。这种安排已显示引起更高效率的基因沉默。所述双链区可以包含一种或两种RNA分子,该RNA分子由一种或两种DNA区转录而来。双链分子的存在被认为引起了内生植物系统的响应,该响应破坏双链RNA,且也破坏了靶植物基因的同源RNA转录物,有效地降低或消除靶基因的活性。
杂交的所述正义和反义序列的长度各为至少19个连续的核苷酸,优选至少30个或50个,更优选至少100、200、500或1000个核苷酸。可使用对应于整个基因转录物的全长序列。所述长度最优选100-2000个核苷酸。所述正义和反义序列与靶向的转录物的同一性程度应为至少85%,优选至少90%且更优选95-100%。RNA分子理所当然地可以包含用以稳定该分子的不相关序列。RNA分子可以在RNA聚合酶II或RNA聚合酶III启动子的控制下表达。后者的实例包括tRNA或snRNA启动子。
优选的小干扰RNA(“siRNA”)分子包含与靶mRNA的约19-21个毗邻核苷酸相同的核苷酸序列。优选地,以二核苷酸AA开始的靶mRNA序列包含约30-70%的GC-含量(优选30-60%、更优选40-60%和更优选约45%-55%),且不具有与待引入的大麦植物的基因组中非靶的任意核苷酸序列的高百分比同一性,例如,如由标准BLAST检索确定。
微小RNA
微小RNA调节是向基因调节演进的RNA沉默路径的明显特定分支,其从常规RNAi/PTGS分离。微小RNA是在以特征性反向重复组构的基因样元素中编码的小RNA的特定种类。转录时,微小RNA基因产生随后加工成微小RNA的茎环前体RNA。微小RNA的长度通常为约21个核苷酸。将释放的miRNA并入含有产生序列-特异性基因阻遏的Argonaute蛋白的特定子集的RISC样复合物中(参见,例如Millar和Waterhouse,2005;Pasquinelli等人,2005;Almeida和Allshire,2005)。
共抑制
另一种可用的分子生物学方式是共抑制。共抑制的机制尚未完全清楚,但其被认为涉及转录后的基因沉默(PTGS),且从这一点来看,与很多反义抑制的实例相似。共抑制涉及以相对于启动子的有义方向将基因或其片断的额外拷贝引入植物中以进行表达。有义片段的大小、其与靶基因区的对应和其与靶基因的序列同一性程度见上述的反义序列。在一些情况中,基因序列的其它拷贝干扰靶植物基因的表达。关于实施共抑制方式的方法参见WO 97/20936和EP 0465572。
核酸构建体
用于产生转基因植物的核酸构建体可使用标准技术轻易产生。
当插入编码mRNA的区时,构建体可包含内含子序列。这些内含子序列可有助于转基因在植物中的表达。术语“内含子”以其常见含义使用,指转录但不编码蛋白且在翻译前被剪接掉RNA的基因片段。如果转基因编码翻译产物,则可将内含子并入5’-UTR或编码区中,反之,可并入转录区中任何位置。然而,在优选的实施方案中,任意多肽编码区以单开放阅读框提供。如本领域技术人员所知,此类开放阅读框可通过反转录编码多肽的mRNA而获得。
为了确保编码目标mRNA的基因的合适的表达,所述核酸构建体通常包含一种或多种调节元件诸如启动子、增强子,以及转录终止或聚腺苷酸化序列。此类元件在本领域是熟知的。
包含调节元件的转录起始区可在植物中提供调节或构建表达。优选地,表达至少发生在种子的细胞中。
已描述植物细胞中为活性的大量构建启动子。植物中构建表达的合适的启动子包括但不限于花椰菜花叶病毒(CaMV)35S启动子、玄参花叶病毒(FMV)35S、甘蔗杆菌状病毒启动子、鸭跖草属黄斑驳病病毒启动子、来自核酮糖-1,5-二磷酸羧化酶的小亚基的光诱导型启动子、水稻胞质丙糖磷酸异构酶启动子、拟南芥属(Arabidopsis)的腺嘌呤磷酸核糖基转移酶启动子、水稻肌动蛋白1基因启动子、甘露氨酸合成酶合成酶及章鱼氨酸合成酶启动子、Adh启动子、蔗糖合成酶启动子、R基因复合体启动子,和叶绿素α/β结合蛋白基因启动子。这些启动子已用于创建在植物中表达的DNA载体;参见,例如WO84/02913。所有这些启动子已被用于创建各种类型的植物可表达的重组DNA载体。
这些启动子可通过诸如温度、光或压力的因素调节。通常,调节元件将为待表达的基因序列的5’。构建体也可以含有其它增强转录的元件诸如nos 3’或ocs 3’聚腺苷酸化区或转录终止子。
该5’非翻译的前导序列可以来源自选择用于表达异源基因序列的启动子,且如需要时可特异性修饰以增加mRNA的翻译。关于转基因最佳表达的综述,参见Koziel等人(1996)。该5’非翻译区也可从植物病毒RNA(尤其是烟草花叶病毒、烟草蚀刻病毒、玉米矮花叶病毒、苜蓿花叶病毒)、从合适的真核基因、植物基因(小麦和玉米叶绿素a/b结合蛋白基因前导)或从合成的基因序列中获得。本发明不限于其中非翻译区来源自伴随启动子序列的5’非翻译序列的构建体的用途。前导序列也可来源自非相关的启动子或编码序列。用于本发明的上下文中的前导序列包含玉米Hsp70前导(US 5,362,865和US 5,859,347)和TMVΩ元件。
转录终止通过在嵌合载体中可操作地连接至目标多核苷酸的3’非翻译DNA序列完成。重组DNA分子的3’非翻译区含有在植物中用以使腺苷酸核苷酸添加到RNA的3’端的聚腺苷酸化信号。该3’非翻译区可从在植物细胞中表达的各种基因获得。胭脂碱合成酶3’未翻译区、来自豌豆小亚单位核酮糖二磷酸羧化酶-加氧酶基因的3’未翻译区、来自大豆7S种子贮存蛋白基因的3’未翻译区通常用于此方面。含有农杆菌属(Agrobacterium)肿瘤诱导(Ti)质粒基因的聚腺苷酸信号的3’转译、非翻译区也合适。
通常,所述核酸构建体包含选择性标记物。选择性标记物有助于鉴定和筛选利用外来核酸分子转化的植物或细胞。所述选择性标记基因可向大麦细胞提供抗生素或除草剂抗性,或允许利用底物诸如甘露糖。选择性标记物优选地向大麦细胞赋予潮霉素抗性。
优选地,将所述核酸构建体稳定地并入植物基因组。由此,核酸包含允许分子并入基因组的适当元件,或将构建体置于可并入植物细胞的染色体中的适当载体中。
本发明的一个实施方案包括重组载体的用途,该重组载体包括至少本文所列的转基因,将其插入能够将核酸分子递送至宿主细胞中的任意载体中。此类载体含有异源性核酸序列,其是在天然状态下不与本发明的核酸分子邻近的核酸序列,且该核酸序列优选地来源自非衍生核酸分子来源的那些种类的种类。该载体可以是RNA或DNA,原核或真核,且通常为病毒或质粒。
适用于植物细胞稳定转染或用于确立转基因植物的大量载体已描述于例如Pouwels等人,Cloning Vectors:A Laboratory Manual,1985,增刊,1987;Weissbach和Weissbach,Methods for Plant Molecular Biology,Academic Press,1989;以及Gelvin等人,Plant Molecular Biology Manual,Kluwer Academic Publishers,1990中。通常,植物表达载体包括例如一种或多种在5’和3’调节序列和主要的选择性标记物的转录控制下的克隆植物基因。此类植物表达载体也可以含有启动子调节区(例如控制可诱导或构建的表达、环境或发育调节的表达或细胞或组织特异性表达的调节区)、转录起始位点、核糖体结合位点、RNA加工信号、转录终止位点,和/或聚腺苷酸化信号。
转基因植物
本发明的上下文中所界定的转基因大麦植物包括植物(以及所述植物的部分和细胞)和它们的后代,该后代已使用重组技术进行基因修饰以在所需的植物或植物器官中引起至少一种多核苷酸和/或多肽的产生。可使用本领域已知的技术产生转基因植物,所述技术诸如A.Slater等人,Plant Biotechnology-The Genetic Manipulation of Plants,Oxford University Press(2003),以及P.Christou和H.Klee,Handbook of Plant Biotechnology,John Wiley和Sons(2004)中通常描述的那些。
在一个优选的实施方案中,转基因植物对于引入的各基因是纯合的,(转基因)后代不分离所需的表型。转基因植物也可对于诸如例如在由杂交种子培育的F1后代中引入的转基因是杂合的。此类植物可提供诸如杂交优势的优点,这在本领域是熟知的。
描述了四种用于将基因直接递送到细胞中的一般方法:(1)化学法(Graham等人,1973);(2)物理法诸如显微注射(Capecchi,1980);电穿孔(参见,例如WO 87/06614、US 5,472,869、5,384,253、WO 92/09696和WO 93/21335);以及基因枪(参见,例如US 4,945,050和US 5,141,131);(3)病毒载体(Clapp,1993;Lu等人,1993;Eglitis等人,1988);以及(4)受体-介导的机制(Curiel等人,1992;Wagner等人,1992)。
可使用的加速方法包括例如微粒轰击等。一个用于将转化核酸分子递送到植物细胞中的方法实例是微粒轰击。该方法由Yang等人,Particle Bombardment Technology for Gene Transfer,Oxford Press,Oxford,England(1994)综述。非生物颗粒(微粒)可以用核酸包覆且通过推动力递送到细胞中。示例性颗粒包括由钨、金、铂等组成的那些。微粒轰击除了是可重复地转化单子叶植物的有效手段外,其一个特别优点是其既不需要原生质体的分离,也不需要对土壤杆菌(Agrobacterium)感染的敏感性。通过加速将DNA递送到玉米(Zea)细胞中的方法的说明性实施方案是生物弹α-颗粒递送系统,其可用于通过筛网诸如不锈钢或Nytex筛网将包覆有DNA的颗粒推向覆盖有悬浮培养的玉米细胞的过滤器表面。适用于本发明的颗粒递送系统是来自Bio-Rad实验室的氦加速PDS-1000/He枪。
为了轰击,悬浮液中的细胞可在过滤器中浓缩。将含有待轰击的细胞的过滤器以适当的距离置于微粒挡板下。如果需要,可将一个或多个筛网置于枪和待轰击的细胞之间。
可替代地,未成熟的胚胎或其它靶细胞可以布置在固体培养基上。待轰击的细胞以适当的距离置于微粒挡板下。如果需要,也将一个或多个筛网置于加速设备和待轰击的细胞之间。通过使用本文提出的技术,可获得多达1000或更多个瞬时表达标记基因的细胞的焦点。轰击后48小时后,焦点中表达外来基因产物的细胞数通常为一至十且平均为一至三。
在轰击转化中,可以优化轰击前的培养条件和轰击参数以产生最大数量的稳定转化体。在这种技术中,轰击的物理和生物参数都是重要的。物理因素是涉及操控DNA/微粒沉淀物的那些或影响大或微小微粒的射程和速度的那些。生物因素包括涉及在轰击之前和紧接轰击之后的细胞操控、靶细胞的渗透压调节以有助于减少与轰击相关联的损伤的所有步骤,以及转化DNA的性质,诸如线性化DNA或者完整的超螺旋质粒。相信预轰击操控对于未成熟胚胎的成功转化是非常重要的。
在另一个可替代的实施方案中,可稳定转化质粒。公开的关于在高等植物中转化质粒的方法包括含有选择性标记物的DNA的颗粒枪递送以及通过同源重组将DNA靶向质粒基因组(U.S.5,451,513、U.S.5,545,818、U.S.5,877,402、U.S.5,932479和WO 99/05265)。
由此,预期可能希望在小规模研究中调整轰击参数的各个方面以完全优化所述条件。可能特别希望调整物理参数诸如间隙距离、飞行距离、组织距离和氦气压力。还可以通过修改影响受体细胞的生理状态从而可能影响转化和整合效率的条件使损伤减少因素最小化。例如,可以调整渗透状态、组织水合和次培养期或受体细胞的细胞周期以优化转化。进行其它常规调整将是根据本公开的本领域技术人员所熟知的。
农杆菌属介导的转移是将基因引入植物细胞的普遍适用系统,因为DNA可被引入整个植物组织中,因此没有必要由原生质体再生完整的植物。使用农杆菌属介导的植物整合载体将DNA引入植物细胞中是本领域熟知的(参见,例如US 5,177,010、US 5,104,310、US 5,004,863、US 5,159,135)。进一步地,T-DNA的整合是相对精确的过程,导致很少的重组。待转移的DNA区被边界序列界定,且通常将间插DNA插入植物基因组中。
现代农杆菌属转化载体能够在大肠杆菌(E.coli)中以及农杆菌属中复制,使得操控方便,如(Klee等人,In:Plant DNA Infectious Agents,Hohn和Schell,编,Springer-Verlag,New York,第179-203页(1985)中所描述。此外,用于农杆菌属介导的基因转移的载体的技术进步已经改善了载体中基因和限制性位点的排列以利于构建能表达各种多肽编码基因的载体。所描述的载体具有由启动子和聚腺苷酸位点侧面连接的便捷的多连接子区以直接表达插入的多肽编码基因,且这些载体适用于本发明目的。另外,可将含有有臂和无臂Ti基因两者的农杆菌用于转化。在其中农杆菌属介导的转化有效的那些植物品种中,因为基因转移的简便性和确定性,所以其为一种选择的方法。
使用农杆菌属转化法形成的转基因植物通常在一种染色体上含有单个基因位点。此类转基因植物可被认为对于所加入的基因是半合子的。更优选的是对于加入的结构基因是纯合的转基因植物,即含有两种添加的基因的转基因植物,一种基因在染色体对的各染色体的相同位点上。纯合的转基因植物可通过使含有单个加入的基因的独立分离子转基因植物有性交配(自交),使所产生的一些种子发芽并分析所得植物的目标基因。
还应理解的是,两种不同的转基因植物也可配对以产生包含两种独立分离的外源基因的后代。合适的后代的自交可产生对于两种外源基因是纯合的植物。还预期与亲本植物回交和与非转基因植物异型杂交,如无性繁殖。关于通常用于不同特性和农作物的其它育种方法的描述可参见Fehr,In:Breeding Methods for Cultivar Development,Wilcox J.编,American Society of Agronomy,Madison Wis.(1987)。
植物原生质体的转化可以使用基于磷酸钙沉淀、聚乙二醇处理、电穿孔以及这些处理的组合而实现。这些体系应用到不同植物品种取决于由原生质体再生特定植物株的能力。描述了由原生质体再生谷物的说明性方法(Fujimura等人,1985;Toriyama等人,1986;Abdullah等人,1986)。
也可使用的其它细胞转化的方法且所述方法包括但不限于通过将DNA直接转移到花粉中,通过将DNA直接注射到植物生殖器官中,或通过将DNA直接注射到未成熟胚胎细胞中,然后使干燥胚胎再水合,将DNA引入植物中。
从单个植物原生质体转化体或各种转化的外植体再生、发育和培养植物是本领域熟知的(Weissbach等人,In:Methods for Plant Molecular Biology,Academic Press,San Diego,Calif.,(1988))。这种再生和生长过程通常包括以下步骤:转化细胞的选择、培养那些个体化细胞通过胚胎发育的常见阶段和通过生根小植株阶段。相似地,再生转基因胚胎和种子。然后,将所得到的转基因的生根幼苗种植到诸如土壤的合适的生长培养基中。
含有外来、外源基因的植物的发育或再生是本领域熟知的。优选地,再生植物自花授粉以提供纯合的转基因植物。否则,从再生植物获得的花粉与农艺重要株系的种子生长植物杂交。相反,这些重要株系的植物的花粉用于对再生植物授粉。使用本领域技术人员熟知的方法培养含有所需的外源核酸的本发明转基因植物。
主要通过使用根癌农杆菌属(Agrobacterium)转化双子叶植物,和获得转基因植物的方法已经公开,其用于棉花(U.S.5,004,863、U.S.5,159,135、U.S.5,518,908);大豆(U.S.5,569,834、U.S.5,416,011);芸苔属(U.S.5,463,174);花生(Cheng等人,1996);以及豌豆(Grant等人,1995)。
用于谷物植物诸如大麦转化以通过引入外源核酸将基因变异引入植物中且用于自原生质体或未成熟植物胚胎再生植物的方法是本领域熟知的,参见例如CA 2,092,588、AU 61781/94、AU 667939、US 6,100,447、PCT/US97/10621、US 5,589,617、US 6,541,257和WO 99/14314。优选地,通过根癌农杆菌介导的转化步骤产生转基因大麦植物。可将携带所需的核酸构建体的载体引入组织培养的植物或外植体的可再生大麦细胞,或合适的植物体系诸如原生质体。
可再生大麦细胞优选地来自未成熟胚胎、成熟胚胎的盾片、由这些衍生的愈伤组织,或分生组织。
为确认所述转基因在转基因细胞和植物中的存在,可以使用本领域技术人员已知的方法进行聚合酶链反应(PCR)扩增或Southern印迹分析。根据所述产物的性质,可以多种方式检测转基因的表达产物,且所述方式包括蛋白质印迹(Western blot)和酶测定。一种特别用于在不同植物组织中定量蛋白表达和检测复制的方式是使用报告基因,诸如GUS。一旦获得转基因植物后,就可以培养所述植物,以生产具有所需的表型的植物组织或部分。可以收获所述植物组织或植物部分和/或收集种子。所述种子可以用作培养其它植物的来源,所述植物包含具有所需的特征的组织或部分。
突变植物的产生
许多本领域已知的技术可用于通过突变内源性大麦醇溶蛋白基因产生具有降低水平的大麦醇溶蛋白的大麦,所述技术包括但不限TILLING、锌指核酸酶、TAL效应子核酸酶(TALEN),和簇生规则间隔短回文重复(Clustered Regularly Interspaced Short Palindromic Repeat,CRSPR)。
TILLING
使用称为TILLING(基因组中靶向诱导的局部损害)的过程可产生本发明的植物。在第一步骤中,在植物群中通过用化学诱变剂处理种子(或花粉)诱导引入的突变(诸如新单碱基对变化),且然后促进植物传代,其中突变将会稳定遗传。提取DNA且保存来自所有群成员的种子以创建可以在一段时间反复使用的资源。
对于TILLING测定,设计PCR引物以特异性扩增目标单个基因靶。如果靶是基因家族的成员或多倍体基因组的一部分,则特异性尤其重要。接着,可使用染色标记的引物来扩增从多个个体收集的DNA的PCR产物。将这些PCR产物变性且重新退火以允许错配碱基对的形成。错配或异源双链体表示天然存在的单核苷酸多态性(SNP)(即,所述群中几种植物可能携带相同多态性)和诱导的SNP(即,仅很少个体植物可能展示突变)。异源双链体形成后,使用识别和切割错配DNA的核酸内切酶(诸如Cel I)是探寻TILLING群中新SNP的关键。
使用该方式,可筛选数千植物以用单碱基变化以及在任意基因或基因组的特定区中的小插入或缺失(1-30bp)来鉴定任意个体。测定的基因组片段的大小无论在何处可以是0.3至1.6kb。在8倍的收集时,每次测定的1.4kb片段(不考虑片段的末端,其中由于噪音SNP探测有问题)和96个泳道的这个组合允许每次测定筛选高达百万的基因组DNA的碱基对,使TILLING成为高产量技术。
TILLING进一步描述于Slade和Knauf(2005)以及Henikoff等人,(2004)中。
除了有效突变检测,高产量TILLING技术对于检测自然多态性是理想的。因此,通过异源化为已知序列询问未知同源DNA揭示多态性位点的数量和位置。鉴定核苷酸变化以及小插入和缺失,包括至少一些重复数多态性。将此称为Ecotilling(Comai等人,2004)。
每个SNP通过在一些核苷酸内的大概位置记录。因此,每个单元型可基于它的迁移率归档。序列数据可使用用于错配切割测定的相同扩增的DNA的等份以相对小增量的努力获得。通过它与多态性邻近选择用于单反应的左或右测序引物。Sequencher软件进行多个比对且发现碱基变化,这在每种情况下证实凝胶带。
进行Ecotilling可比全测序更便宜,该方法目前常用于大多数SNP发现。可筛选含有阵列生态型DNA的平板,而不是来自突变植物的DNA收集。因为在凝胶上以几乎碱基对分辨率检测且背景图案在整个泳道上是均匀的,所以可匹配具有相同大小的带,从而在单步骤中发现和基因分型SNP。以这种方式,SNP的最终测序简单且有效,通过以下事实更是如此:用于筛选的等份相同PCR产物可经历DNA测序。
使用位点特异性核酸酶的基因组编辑
基因组编辑使用工程化的核酸酶,该核酸酶由融合至非特异性DNA切割模块的序列特异性DNA结合结构域组成。这些嵌合核酸酶通过诱导刺激细胞的内源性细胞DNA修复机制以修复诱导的断裂的靶向的DNA双链断裂能实现有效且精确的基因修饰。此类机制包括,例如容易出错的非同源末端连接(NHEJ)和同源介导的修复(HDR)。
在具有扩展的同源臂的供体质粒的存在下,HDR可导致单个或多个转基因的引入以纠正或取代现有基因。在供体质粒不存在时,NHEJ介导的修复产出引起基因破坏的靶的小插入或缺失突变。
用于本发明的方法的工程化核酸酶包括锌指核酸酶(ZFN)和转录激活因子样(TAL)效应子酶(TALEN)。
通常通过质粒DNA、病毒载体或体外转录的mRNA将核酸酶编码基因递送到细胞中。荧光代用品受体载体的使用也允许富集ZFN-和TALEN-修饰的细胞。作为ZFN基因递送系统的替代,细胞可与纯化的ZFN蛋白接触,该蛋白能够穿过细胞膜且诱导内源性基因破坏。
复杂基因组常含有多个与预期的DNA靶相同或高度同源的序列拷贝,潜在地导致脱靶活性和细胞毒性。为解决这个,可使用基于结构(Miller等人,2007;Szczepek等人,2007)及选择(Doyon等人,2011;Guo等人,2010)的方式生成具有最佳切割特异性且降低的毒性的改进ZFN和TALEN异二聚体。
锌指核酸酶(ZFN)包含DNA结合结构域和DNA切割结构域,其中该DNA结合结构域由至少一个锌指组成且可操作地连接至DNA切割结构域。该锌指DNA结合结构域在蛋白的N-端且该DNA切割结构域位于所述蛋白的C-端。
ZFN必须具有至少一个锌指。在一个优选的实施方案中,ZFN具有至少三个锌指以具有足以在宿主细胞或生物体中用于靶向的基因重组的特异性。通常,具有三个以上锌指的ZFN将与每个其它锌指具有逐渐增大的特异性。
锌指结构域可来源自任意类别或类型的锌指。在具体的实施方案中,该锌指域包含Cis2His2类型的锌指,该锌指一般由例如锌指转录因子TFIIIA或Sp1表示。在一个优选的实施方案中,该锌指结构域包含三种Cis2His2类型的锌指。可以改变ZFN的DNA识别和/或结合特异性以在细胞DNA的任意选择位点实现靶向的基因重组。此类改变可使用已知的分子生物学和/或化学合成技术(参见,例如,Bibikova等人,2002)实现。
ZFN DNA切割结构域来源自非特异性DNA切割结构域的类别,例如II型限制性内切酶例如FokI的DNA切割结构域(Kim等,1996)。其它有用的内切酶可以包括例如HhaI、HindIII、Nod、BbvCI、EcoRI、BglI,和AlwI。
在ZFN的切割和识别结构域之间的连接子(如果存在),包含选择的氨基酸残基序列以使得所得的连接子是可变的。或者,为了最大化靶位点特异性,构建无连接子的构建体。无连接子的构建体具有结合6bp间隔的识别位点并随后对其进行切割的强烈倾向。然而,具有0-18个氨基酸的连接子长度,ZFN介导的切割发生在间隔5-35bp的识别位点之间。对于给定的连接子长度,将限制与结合和二聚作用一致的识别位点之间的距离。(Bibikova等人,2001)。在一个优选的实施方案中,切割结构域和识别结构域之间没有连接子,且靶基因座包含通过六个核苷酸间隔子分开的彼此方向相反的两个9核苷酸识别位点。
为了根据本发明的优选实施方案靶向基因重组或突变,两个9bp锌指DNA识别序列必须在宿主DNA中鉴定。这些识别位点彼此方向相反且被约6bp的DNA分隔。然后,通过设计生成ZEN,且产生在靶基因座特异性结合DNA的锌指组合,然后将锌指连接至DNA切割结构域。
ZFN活性可通过使用瞬时低温培养条件而改善以增加核酸酶表达水平(Doyon等人,2010)和位点特异性核酸酶与DNA端加工酶的共递送(Certo等人,2012)。通过使用刺激HDR而不激活易出错NHE-J修复路径的锌指切口酶(ZFNickases)可改善ZFN介导的基因组编辑特异性(Kim等人,2012;Wang等人,2012;Ramirez等人,2012;McConnell Smith等人,2009)。
转录激活因子样(TAL)效应子核酸酶(TALEN)包含TAL效应子DNA结合结构域和内切酶结构域。
TAL效应子是通过病原注射至植物细胞中的植物病原细菌的蛋白,其中它们行进至细胞核且作为转录因子以启动特异性植物基因。TAL效应子的一级氨基酸序列指示它结合的核苷酸序列。因此,可预测TAL效应子的靶位点,且可工程化及生成TAL效应子以结合特定核苷酸序列。
与TAL效应子编码核酸序列融合的是编码核酸酶或核酸酶一部分的序列,通常是来自型II型限制性内切酶诸如FokI的非特异性切割结构域(Kim等人,1996)。其它有用的内切酶可以包括例如HhaI、HindIII、Nod、BbvCI、EcoRI、BglI和AlwI。事实是一些仅作为二聚物的内切酶(例如FokI)可被利用以强化TAL效应子的靶向特异性。例如,在一些情况下,可将每个FokI单体与识别不同DNA靶序列的TAL效应子序列融合,且仅当两个识别位点紧密接近时,与失活的单体一起创建功能性酶。通过要求DNA结合以激活核酸酶,可创建高位点特异性限制性酶。
序列特异性TALEN可识别细胞中存在的预选定靶核苷酸序列中的特定序列。因此,在一些实施方案中,可扫描靶核苷酸序列的核酸酶识别位点,且基于靶序列可选择特定核酸酶。在其它情况下,可工程化TALEN以靶向特定细胞序列。
使用可编程的RNA导向的DNA内切酶的基因组编辑
与上述的位点特异性核酸酶不同,簇生规则间隔短回文重复(CRISPR)/Cas体系提供ZFN和TALEN的替代用于诱导靶向的基因改变。在细菌中,该CRISPR体系经由RNA导向的DNA切割提供对侵入性外来DNA的后天免疫。
CRISPR体系依靠CRISPR RNA(crRNA)及反式激活嵌合RNA(tracrRNA)用于侵入性外来DNA的序列特异性沉默。CRISPR/Cas体系存在三种类型:在II型体系中,Cas9用作RNA导向的DNA内切酶,其在crRNA–tracrRNA靶识别时切割DNA。CRISPR RNA碱基对与tracrRNA形成双RNA结构用于将Cas9内切酶导向互补的DNA位点以切割。
通过表达Cas内切酶的质粒和必需crRNA组分的共递送,针对植物细胞,CRISPR体系可为便携式。可将Cas内切酶转化成切口酶以提供对DNA修复机制的其它控制(Cong等人,2013)。
CRISPR位点是散置的短序列重复(SSR)的不同类别,所述SSR在大肠杆菌中第一次识别(Ishino等人,1987;Nakata等人,1989)。相似的散置SSR已在地中海富盐菌(Haloferax mediterranei)、酿脓链球菌(Streptococcus pyogenes)、鱼腥藻属(Anabaena)和结核分枝杆菌(Mycobacterium tuberculosis)(Groenen等人,1993;Hoe等人,1999;Masepohl等人,1996;Mojica等人,1995)中鉴定。
CRISPR位点在重复结构上与其它SSR不同,这些重复结构被称作短规则隔开重复(SRSR)(Janssen等人,2002;Mojica等人,2000)。该重复是以簇发生的短元素,其总是被唯一的具有恒定长度的间插序列规则地间隔(Mojica等人,2000)。尽管该重复序列在菌株之间高度保守,但间插的重复数和间隔区序列在菌株与菌株之间不同(van Embden等人,2000)。
CRISPR位点的常见结构特征在Jansen等人,(2002)中被描述成(i)多个短直接重复的存在,其表明在给定位点无序列变异或有非常小的序列变异;(ii)非重复的间隔序列在相似大小的重复之间的存在;(iii)在携有多个CRISPR位点的大多数种类中数百个碱基对的常见前导序列的存在;(iv)位点中长开放阅读框的不存在;以及(v)一种或多种cas基因的存在。
CRISPR通常是24-40bp的部分短回文序列,其含有高达11bp的内部和终端反向重复。尽管已检测分离的元素,但它们一般以由唯一的间插20-58bp序列分隔的重复单元的簇(每个基因组高达约20或更高)安排。CRISPR一般在给定的基因组中是均质的,它们大多数是相同的。然而,在例如古细菌(Archaea)中有异质性的实例(Mojica等人,2000)。
如本文使用的术语“cas基因”是指一种或多种一般耦合或接近或在侧面CRISPR位点附近的cas基因。Cas蛋白家族的全面综述在Haft等人(2005)中给出。在给定的CRISPR位点,大量cas基因可随种类变化。
实施例
实施例1.材料和方法
植物材料
大麦系cv Sloop(野生型)从澳大利亚冬性谷物保藏中心(Tamworth,澳大利亚)获得。56(不表达B-大麦醇溶蛋白)和1508(不表达C-大麦醇溶蛋白及表达降低的D-和B-大麦醇溶蛋白)(Doll,1973;Doll,1983)从北欧种质库(Alnarp,瑞典)获得。1508是乙撑亚胺诱导的突变体,其携带降低C-大麦醇溶蛋白积累的染色体5H上的lys3a基因突变。这些系的每一个都可公开获得。在白天25℃且晚上20℃的温室条件下种植植物且收获经检查排除污染的种子。对于制麦芽和酿造实验,栽培品种植物Sloop、56和1508在堪培拉的CSIRO Ginninderra实验站的田间并排种植且各收获10kg谷粒。使用标准技术,将谷粒制麦芽且发酵20L批次的啤酒。
从面粉中提取谷醇溶蛋白
为了提取谷醇溶蛋白(醇溶性蛋白),使用标准技术将谷粒研磨成全麦面粉。将水洗全麦面粉中的谷醇溶蛋白(10g)溶于55%(v/v)丙-2-醇(HPLC级)、2%(w/v)二硫苏糖醇(DTT)中,在65℃下孵育45分钟,且用两体积丙-2-醇在-20℃过夜沉淀。将沉淀的谷醇溶蛋白溶于8M尿素、1%DTT、25mM三乙醇胺-HCl(pH 6)中,且通过快速蛋白液相色谱法(FPLC)在用在1%(v/v)三氟乙酸(TFA)中3%至60%乙腈的30mL线性梯度(以2mL/min)洗脱的4mL Resource RPC柱(GE Healthcare,悉尼,NSW,澳大利亚)上进行纯化。
过滤器辅助的样品制备(FASP)方法
将50μL的大麦醇溶蛋白提取物转移至PALL Nanosep 10MWCO过滤器中。加入200μL的8M于0.1M Tris/HCl pH 8.5(UA)中的尿素(Sigma)且在约20℃下以14000rpm离心混合物15分钟。丢弃管流出液。将另外的200μl UA加入过滤器单元中且以14,000rpm再次离心15分钟。加入100μl IAA溶液(0.05M于UA中的碘乙酰胺)且将样品在设定为20℃的热混合器中以600rpm混合1分钟且然后在不混合下孵育20分钟。将100μl UA加入过滤器单元中且以14,000×g离心15分钟。此步骤重复两次。将100μl 0.05M于中的NH4HCO3(碳酸氢铵,ABC)的水溶液加入过滤器单元中且以14,000×g离心10分钟。此步骤重复两次。加入具有15μL胰蛋白酶原液(1.5μg/μl,Sigma)的40μl ABC且每个样品在热混合器中以600rpm混合1分钟。将该过滤器单元在湿室中于37℃下孵育4-18h用于酶消化。将该过滤器单元转移至新的收集管中且以14,000rpm离心10分钟。加入40μl ABC且将该过滤器单元以14,000rpm再次离心10分钟。此步骤重复一次。通过加入15μL 5%的甲酸酸化流出部分且将其冻干。将提取的干燥肽再悬浮于30μL 0.5%的甲酸中用于MRM分析。
麦芽汁和啤酒的制备
在乔·怀特微生物制麦系统(Joe White Micromalting System)中在多个800g罐中将大麦制麦芽。浸泡方案涉及:浸泡8h,静置9h,在17℃下浸泡5h(Sloop);浸泡8h,静置10h,在17℃下浸泡5h(56);以及浸泡7h,静置8h,在17℃下浸泡3h(1508)。Sloop在16℃下发芽超过94h,且在15℃下两种大麦醇溶蛋白缺失突变体发芽超过94h。干燥炉程序在50-80℃下进行超过21h。将干燥的麦芽进行淀粉糖化,如在Colgrave等人(2012)中所描述。在指示的淀粉酶静置时间后,使醪液煮沸且沸腾1h以制得麦芽汁。在沸腾期间,煮沸的麦芽汁因Tettnang啤酒花变苦以达到21-22IBU。过夜冷却麦芽汁至20℃且然后用富酶泰斯公司(Fermentis)US-05酵母在18-20℃下发酵以在约2周后完成。将未过滤的啤酒装桶,且使其在装瓶前强制碳化。
啤酒的分析
获得选择的60种市售啤酒,如Colgrave等人(2012)的附加表1中所列举。从每个的两种不同瓶中取一式三份样品(1mL)且在减压下脱气以除去CO2。取等份(100μL)脱气啤酒且通过加入20μL 50mM DTT在N2中60℃下还原30分钟。将20μL 100mM碘乙酰胺(IAM)加入这些溶液中且在室温下孵育样品15分钟。将5μL 1mg/mL胰蛋白酶(Sigma)或胰凝乳蛋白酶(Sigma)加入各溶液中且在37℃下孵育样品过夜。通过加入10μL 5%甲酸酸化消化的肽溶液且使其通过10kDa MW过滤器(Pall,澳大利亚)。将滤液冻干且在1%甲酸中复溶且保存在4℃下直到分析。
未消化的麦芽汁和啤酒的分析
通过14,000rpm离心30分钟使来源自野生型(Sloop)大麦和大麦醇溶蛋白缺失突变体的麦芽汁和啤酒(0.1mL)通过10kDa分子量截止过滤器(Pall)以制得符合LC-MS/MS的肽级分。在QStar Elite质谱仪上分析肽级分(10μL)。
Q-TOF MS
在使用Vydac MS C18 具有5μm粒径的柱(150mm×0.3mm)(Grace Davison,Deerfield,美国)的Shimadzu nano HPLC(Shimadzu Scientific,Rydalmere,澳大利亚)系统上使用2-42%溶剂B的线性梯度以3μL/min的流速色谱法分离样品20分钟。流动相由溶液A(0.1%甲酸)和溶液B(0.1%甲酸/90%乙腈/10%水)组成。以具有纳米级电子喷射离子化源的标准MS/MS数据依赖型采集模式使用QStar Elite QqTOF质谱仪(Applied Biosystems)。使用生厂商的‘智能出口(Smart Exit)’收集调查MS光谱(m/z 400-1800)1s,接着收集最强母离子的三个MS/MS测量值(对于MS/MS,10个计数/第二阀值、2+至5+电荷状态和m/z 100-1600质量范围)。从重复MS/MS采集中排除先前靶向的母离子30秒(100mDa的质量公差)。
线性离子阱(三重四极)MS
在装备有以正离子模式操作的TurboV离子源的Applied Biosystems 4000QTRAP质谱仪(Applied Biosystems,Framingham,MA,美国)上分析还原的和烷基化的胰蛋白酶肽。在使用Phenomenex Kinetex C18(2.1mm x 10cm)柱的Shimadzu Nexera UHPLC(Shimadzu)上使用5-45%乙腈(ACN)的线性梯度以400μL/min的流速色谱法分离样品15分钟。来自HPLC的洗脱液直接耦合到质谱仪上。获得数据且使用Analyst 1.5softwareTM对其进行处理。信息依赖型采集(IDA)分析使用作为调查扫描的质量范围350-1500的加强型MS(EMS)扫描进行且引发串联质谱采集。选择超过界定阈值(100,000个计数)的电荷状态2-5的头两种离子且首先使其经历增强型分辨率(ER)扫描,然后获得质量范围125-1600的增强型产品扫描(EPI)。
质谱和数据库搜索的分析
将利用Paragon算法的ProteinPilotTM 4.0软件(Applied Biosystems)用于鉴定蛋白。针对经由电脑模拟的Uniprot(版本2011/05)和NCBI(版本2011/05)数据库的小麦族蛋白的胰蛋白酶或胰凝乳蛋白酶消化物搜索串联质谱数据。将所有搜索参数界定为经半胱氨酸烷基化、经作为消化酶的胰蛋白酶或胰凝乳蛋白酶修饰的碘乙酰胺。设定修饰为这个软件包提供的“通用后处理”及“生物”修饰设置,所述修饰由126种可能修饰组成,例如乙酰化、甲基化和磷酸化。通用后处理修饰设置含有51种可能因样品处置产生的潜在修饰,例如氧化、脱水和脱酰胺化。该分析中包括具有一种漏切的肽。
定制谷物数据库的构建和应用
通过包括所有报告的来自NCBI、TIGR基因指数的核苷酸记录,或属于小麦属(Triticum)、大麦属(Hordeum)、燕麦属(Avena)、黑麦属(Secale)和小黑麦属(Triticosecale)种类的TIGR植物转录物组件的蛋白序列构建非冗余定制谷物种子贮藏蛋白数据库。对于上述种类,核苷酸序列以六框翻译,剪切以仅保持最长阅读框。然后将所得蛋白序列设置成非冗余。仅从开始至结束100%匹配的序列一起折叠以维持所有变异。最终,筛选这些文件以仅保持含有谷蛋白、麦醇溶蛋白、麦谷蛋白、大麦醇溶蛋白、燕麦蛋白或裸麦醇溶蛋白文字的记录。针对定制谷物数据库搜索串联质谱数据。
原型肽的蛋白比对和鉴定
比对Uniprot数据库中的所有已知大麦醇溶蛋白和TIGR数据库中预测的大麦醇溶蛋白。在每个家族中(B、C、D或γ),选择常见的肽作为该家族的代表。确定每个肽的MRM转换,在所述肽中前体离子(Q1)m/z基于大小和预期的电荷,且使用已知片段化模式和/或在特征工作流程中收集的数据预测片段离子(Q3)m/z值。在初步分析中使用了多达六个转换且精炼MRM转换且每个肽选择前两个MRM转换用于最终的方法,其中最强MRM转换被用作定量因素且第二最强转换被用作定性因素。
MRM质谱
MRM实验被用于大麦醇溶蛋白来源的胰蛋白酶肽的定量。对于IDA-和MRM-引发的MS/MS实验,扫描速度设置为1000Da/s且在具有氮气的碰撞室中使用取决于前体离子大小和电荷的滚动碰撞能将肽片段化。使用预定的MRM扫描实验(其针对每个MRM转换使用120s检测窗口和1s循环时间)实现大麦醇溶蛋白肽的定量。第一个四极子用于选择分析物(所谓的前体离子)的荷质比(m/z)。然后将前体离子传送到碰撞室中(第二个四极子)。发生碰撞诱导解离(CID),导致传送到第三个四极子的片段离子产生。质量选择的第二阶段发生,特异性靶向已知片段离子的m/z值。质量选择的两个阶段被称为Q1和Q3,其指它们在其中发生的四极子。因此,Q1至Q3的转换被称为MRM转换且对于选择的分析物具有高特异性和选择性。
大麦醇溶蛋白的相对定量
每个大麦醇溶蛋白的相对定量通过对每个肽的最强MRM转换的峰面积积分而进行。通过取瓶A和B(表示生物重复)中的两种重复注射液(在不同天)的平均值而确定平均峰面积。结果以相对于所有含全谷蛋白啤酒的平均大麦醇溶蛋白含量的每个大麦醇溶蛋白的蛋白的百分比表示。
实施例2.大麦面粉中大麦醇溶蛋白的表征
通过溶于乙醇溶液中从大麦面粉(野生型cv Sloop)中提取的大麦醇溶蛋白通过如实施例1中描述的FPLC纯化。使纯化的大麦醇溶蛋白级分还原、烷基化并经历胰蛋白酶或胰凝乳蛋白酶消化。酶促消化后,通过LC-MS/MS分析低于-10kDa级分以鉴定存在于来自面粉的纯化的谷醇溶蛋白级分中的大麦醇溶蛋白以提供一整套预期在由该面粉酿造的啤酒中发现的大麦醇溶蛋白蛋白。使用1%假发现率(FDR),在胰蛋白酶消化后鉴定共144种蛋白且在胰凝乳蛋白酶消化后鉴定共55种蛋白。
表1列出了从胰蛋白酶消化后的面粉中检测到的大麦醇溶蛋白的蛋白产物。从最丰富的蛋白检测到的是先前已报告的B3-大麦醇溶蛋白(登录号P06471,Kristoffersen等人,2000)、γ-3-大麦醇溶蛋白(P80198,Fasoli等人,2010)和预测的γ-1-大麦醇溶蛋白(P17990,Cameron-Mills等人,1988)。同样地,检测到大量D-大麦醇溶蛋白(Uniprot:Q84LE9,Gu等人,2003)。检测到具有较少丰度的其它蛋白,包括两种γ-大麦醇溶蛋白和一种B1-大麦醇溶蛋白,它们不存在于NCBI或Uniprot数据库(表1)中。通过在NCBI、TIGR基因指数或TIGR植物转录物组件中的核苷酸条目搜索包含翻译的谷物蛋白的定制数据库而鉴定这些蛋白。检测到几种与预测γ-麦醇溶蛋白和谷醇溶蛋白(来自小麦)和燕麦蛋白样蛋白-A(来自小麦和山羊麦)匹配的肽。基于由通过SDS-PAGE分离的16-17kDa蛋白带的胰蛋白酶消化所得的15种氨基酸肽(QQCCQPLAQISEQAR,SEQ ID NO:5)的检测,报告燕麦蛋白样A蛋白存在于啤酒中(Picariello等人,2011)。如通过BLASTp搜索所确定,肽序列匹配大麦的单一蛋白中一条序列,另外的13个氨基酸(aa)肽与同样的预测蛋白序列(Uniprot:F2EGD5)匹配。还检测到定位至来自圆柱山羊草(Aegilops cylindrica)(铰链的山羊麦)的燕麦蛋白样A蛋白(Uniprot:Q2A782)的11aa(MVLQTLPSMCR,SEQ ID NO:6)肽。翻译的EST数据库(TIGR)的随后搜索揭示解释该11aa肽的大麦(H.vulgare)蛋白。基于预测的燕麦蛋白样蛋白-A和γ-大麦醇溶蛋白蛋白质之间的同源性,这些包括在后续的分析中。
单肽鉴定示意大麦醇溶蛋白级分内的C-大麦醇溶蛋白的存在,然而,已知C-大麦醇溶蛋白蛋白质的蛋白序列比对揭示在这些富含谷氨酰胺的蛋白中不存在胰蛋白酶切割位点。结果,大麦醇溶蛋白级分的胰凝乳蛋白酶消化产生对具有高达80%序列覆盖率的C-大麦醇溶蛋白的鉴定(表2),突出对替代性消化策略的需求以表征此类大麦醇溶蛋白。
表1.胰蛋白酶消化后在大麦面粉的FPLC纯化级分中鉴定的谷醇溶蛋白的蛋白。
列出每个蛋白的登录号:*表明支持有注释的转录物证据的蛋白鉴定(Uniprot);**表明支持基因组来源的预测蛋白的蛋白鉴定(Uniprot);***表明在针对未注释的TIGR EST的数据库搜索中鉴定的新型蛋白。
表2.胰凝乳蛋白酶消化后在大麦面粉的FPLC纯化级分中鉴定的谷醇溶蛋白蛋白质。
列出每个蛋白的登录号:*表明支持有注释的转录物证据的蛋白鉴定(Uniprot);**表明支持基因组来源的预测的蛋白的蛋白鉴定(Uniprot);***表明在针对未注释的TIGR EST的数据库搜索中鉴定的新型蛋白。
实施例3.麦芽汁和啤酒中大麦醇溶蛋白的特征
然后进行麦芽汁(在酿造期间从淀粉糖化过程中提取的液体)及啤酒分析,且鉴定相似的一套谷醇溶蛋白(表3)。在麦芽汁中鉴定共27种蛋白且在啤酒中鉴定79种,最丰富的蛋白为非特异性脂质转运蛋白1(LTP1)和α-淀粉酶胰蛋白酶抑制剂(CMd、CMb、CMa)。在麦芽汁中鉴定的谷蛋白蛋白包括燕麦样A蛋白(18种肽)、γ-大麦醇溶蛋白-3(10种肽)和D-大麦醇溶蛋白(4种肽),它们以前在富含大麦醇溶蛋白级分中有观察到。然而值得注意的是,>50%肽是半-胰蛋白酶(在不是Lys/Arg的位点一端裂解),表明在酿造过程中发生显著的蛋白降解。以>60%序列覆盖率(12种肽)检测从EST数据库搜索中鉴定的燕麦样A蛋白(GenBank登录号BE195337),从而增加了该蛋白鉴定的可信度。
C-大麦醇溶蛋白明显不存在于啤酒中,但为了确保这个不是归咎于低量胰蛋白酶位点的假阳性,进行胰凝乳蛋白酶消化,其确认在啤酒中不存在C-大麦醇溶蛋白(表3)。C-大麦醇溶蛋白的不存在可能与它们在水中的不溶性有关。C-大麦醇溶蛋白由多种具有共有序列PQQPFPQQ(SEQ ID NO:7)的八肽重复组成,致使它们高度不溶。在麦芽汁中鉴定出了许多C-大麦醇溶蛋白降解产物,但其并不幸免于导致啤酒产生的酿造和过滤步骤中。
为了表征肽片段,使麦芽汁和啤酒通过10kDa分子量截留过滤器且在无酶促消化下对其分析。1D-PAGE分析揭示过滤步骤有效地将蛋白从啤酒中除去。MS分析揭示存在几种截短的或降解的大麦醇溶蛋白产物。表4列出鉴定的肽片段。除了大麦醇溶蛋白肽,鉴定了麦芽汁和啤酒中来源自包括丝氨酸蛋白酶抑制蛋白-Z4、非特异性脂质转运蛋白1、α-淀粉酶、β-淀粉酶、hordoindoles(B1,B2)和GAPDH的大量大麦蛋白的肽(列于表4和Colgrave等人,2012的补充表2中)。有趣的是,在麦芽汁中观察到大量C-大麦醇溶蛋白片段,在啤酒中只检测到微量水平的C-大麦醇溶蛋白肽。
啤酒在酶促消化不存在下的表征清楚地表明除了完整的大麦醇溶蛋白,存在大量部分降解的大麦醇溶蛋白片段且这些也可导致腹腔毒性。许多这些肽片段含有可以在乳糜泻患者中引起免疫反应的运行Gln和Pro。检测到的潜在免疫原性肽的实例是FVQPQQQPFPLQPHQP(燕麦蛋白样A;GenBank:TA31086,SEQ ID NO:8)、YPEQPQQPFPWQQPT(γ-1-大麦醇溶蛋白;P17990,SEQ ID NO:9)、LERPQQLFPQWQPLPQQPP(γ-3-大麦醇溶蛋白;P80198,SEQ ID NO:10)和LIIPQQPQQPFPLQPHQP(C-大麦醇溶蛋白;P17991,SEQ ID NO:11),其中加下划线的序列与先前报道的免疫原性肽具有高同源性(Tye-Din等人,2010;Kahlenberg等人,2006)。
表3.在麦芽汁和啤酒中鉴定的谷醇溶蛋白蛋白质。
列出每个蛋白的登录号:*表明支持有注释的转录物证据的蛋白鉴定(Uniprot);**表明支持基因组来源的预测蛋白的蛋白鉴定(Uniprot);***表明在针对未注释的TIGR EST的数据库搜索中鉴定的新型蛋白。
表4.在啤酒的低于-10kDa级分检测的来源自大麦醇溶蛋白蛋白的肽。
粗体的表位显示与谷蛋白表位的高序列同源性,所述谷蛋白表位由针对黑麦谷醇溶蛋白产生的Mendez R4MAb识别(QQPFP(SEQ ID NO:12)、QQQFP(SEQ ID NO:13)、LQPFP(SEQ ID NO:14)、QLPFP(SEQ ID NO:15))a。这些肽可以在CD患者中引起免疫反应。
a.Kahlenberg等人,2006。
b.列出每个蛋白的登录号:*表明支持有注释的转录物证据的蛋白鉴定(Uniprot);**表明支持基因组来源的预测蛋白的蛋白鉴定(Uniprot);***表明在针对未注释的TIGR EST的数据库搜索中鉴定的新型蛋白。
实施例4.通过MRM质谱对啤酒中的大麦醇溶蛋白相对定量
实施例2和3中纯化的大麦醇溶蛋白和啤酒的蛋白组表征可阐明存在于大麦中的主要大麦醇溶蛋白蛋白。多种反应监测(MRM)被视为肽和蛋白定量的有用工具。在这种方法中,蛋白经胰蛋白酶消化后,蛋白水解片段通过HPLC以色谱法分离且通过MRM质谱进行分析。第一个四极子(Q1)选择第一个肽m/z值(前体质量)且将该离子传送至碰撞室(Q2)。碰撞诱导的解离导致与蛋白水解片段的氨基酸序列有关的片段离子系列产生。然后在第三个四极子(Q3)中选择诊断片段离子且将其传送至允许目标肽定量的检测器中。通常,每种肽使用三种MRM转换且每种蛋白使用至少两种肽。
从每个蛋白家族中,选择一种单一同种型以监测由选择性育种大麦系酿造的啤酒的谷蛋白含量(见下文)。选择多种胰蛋白酶肽(≥2种肽/蛋白)用于开发定量测定。当这些肽先前已在探索实验中检测到,使用肽m/z和片段离子信息确定待使用的MRM转换。MRM测定中包括了大量最初未鉴定的肽,以至于每种蛋白使用最少两种肽。在这些情况下,滞留时间是未知的且在第一通过实验中不能调度MRM转换。确定肽的滞留时间且在后续实验中使用预定的MRM转换。
来源自单一优良澳大利亚制麦芽大麦(“Sloop”)的啤酒被用于开发和改进MRM方法,因为该啤酒含有大麦醇溶蛋白蛋白的完全互补,而大麦变体(56和1508)被预期为低或不含B-和C-大麦醇溶蛋白。当使用MRM分析来分析由野生型和两种大麦醇溶蛋白突变体大麦谷粒(56和1508)的样品酿造的啤酒时,在野生型大麦啤酒中清晰地观察到所有八种选择的肽(每种肽三种MRM转换)。56啤酒显示D-大麦醇溶蛋白肽的量大约降低三倍且明显不存在B-大麦醇溶蛋白肽。1508啤酒显示测量的每种肽的量进一步降低,但观察到微量燕麦蛋白样A蛋白。
通过检验单一栽培品种大麦啤酒(“Sloop”)评估分析方法的再现性。首先,使啤酒进行多次冻融循环(1、10、20或50次循环)且甚至在50次循环后未观察到每个监测的MRM转换的峰面积有明显变化(变异系数,CV<15%)。其次,通过六次重复消化检测消化效率,得出CV<15%。通过以CV<15%重复四次注射每个消化物来评估分析的重现性。最后,评估两种不同啤酒瓶之间的大麦醇溶蛋白含量的变化且发现为<10%。
市售啤酒的分析
通过分析60种选择的市售啤酒的谷蛋白含量进一步验证量化啤酒的大麦醇溶蛋白的MRM方法,所述市售啤酒包括标注的低谷蛋白和无谷蛋白啤酒。通过还原、烷基化和消化(实施例1)处理来自各个瓶中的重复样品且通过MRM质谱进行分析。图2显示相对于Sloop,市售啤酒中燕麦蛋白样A蛋白(A)、B-大麦醇溶蛋白(B,C)、D-大麦醇溶蛋白(D)和γ-大麦醇溶蛋白(E)的相对定量。相对于非无谷蛋白啤酒的平均值,观察到市售啤酒类型(存在的大麦醇溶蛋白家族)及量(对于任意给定的大麦醇溶蛋白蛋白,平均在1-380%cf.之间)不同。八种啤酒(编号:17、47、49、50、51、52、58和60)标记为无谷蛋白,因为它们由高粱麦芽、画眉草、大米、小米或玉米酿造。这些谷物缺乏存在于大麦和小麦中的谷蛋白蛋白。MRM测定证实它们不含靶向的大麦醇溶蛋白蛋白和大麦醇溶蛋白相关的蛋白(燕麦蛋白样A)。啤酒17和50-52基于高粱,啤酒47未指定由什么酿造,啤酒49基于小米,啤酒58是由高粱麦芽、画眉草和大米酿造以及啤酒60是非谷物来源的啤酒。在已被分类为低谷蛋白(<10ppm)的两种啤酒(57和59)的检验中,相对大麦醇溶蛋白的含量类似于测试的啤酒的整个范围内的平均大麦醇溶蛋白含量。啤酒57显示低燕麦蛋白样A蛋白水平(平均~50%cf.),但出人意料地显示显著水平的来源自B1-(平均>300%cf.)、D-(~105%)和γ3-大麦醇溶蛋白(~62%)的肽。啤酒59显示低但显著水平的B1-、D-和γ-大麦醇溶蛋白(分别为55%、42%和92%)且与在含谷蛋白的啤酒中观察到的那些相同水平的燕麦蛋白样A蛋白。
结论
由大麦制造的啤酒含有来源自用于酿造过程的谷粒的谷蛋白。可使用ELISA测量啤酒中的谷蛋白水平,然而,使用当前ELISA技术时有许多与大麦醇溶蛋白的精确测量相关的限制。在上述实施例中,开发质谱测定以表征纯化的大麦醇溶蛋白制品、麦芽汁和啤酒中完整一套大麦醇溶蛋白,且以实施最丰富大麦醇溶蛋白的相对定量。使用质谱的测定对于面粉、麦芽汁和啤酒中的大麦醇溶蛋白(谷蛋白)的测量是稳定而敏感的且可容易地应用于麦芽。
实施例5.大麦Hor3基因中无效突变的鉴定
由染色体1H的长臂上Hor3基因编码105kDa的大麦D-大麦醇溶蛋白(Gu等人,2003)。称为Ethiopian R118的大麦野生种族被认为不积累D-大麦醇溶蛋白(Brennan等人,1998)且从The John Innes Centre Public Collections的公开获得的种质收集获得(登录号3771)。该系是大麦的野生种族,非常不适合用于谷粒的商业产生。
Ethiopia R118谷粒在温室中播种。观察到,所得植物分类为2-行和6-行表型以及黑色、着色种子和绿色种子。选择产生绿色种子的2-行系且与cv Sloop杂交。选择对于产生D-大麦醇溶蛋白呈阴性的F2半-种子。将该系的植物与野生型Sloop回交且再次选择对于D-大麦醇溶蛋白无效的F2植物。将该植物与Sloop回交且后代植物再次在具有约87.5%Sloop基因组的遗传背景下回交以产生对于D-大麦醇溶蛋白产生呈阴性的BC2系。
从分离自每个Sloop BC2、来源自Ethiopia R118的D-大麦醇溶蛋白阴性植物和野生型Sloop。这通过具有标准条件的PCR实现且生成一系列3重叠片段。用于扩增的引物对为:
片段1
5’Dhor1 GACACATATTCTGCCAAAACCCC(SEQ ID NO:60)和
3’Dhor3 ACGAGGGCGACGATTACCGC(SEQ ID NO:61)
片段2
5’Dhor1b GAGATCAATTCATTGACAGTCCACC(SEQ ID NO:62),和
3’Dhor1 CTTGTCCTGACTGCTGCGGAGAAA(SEQ ID NO:63)
片段3
5’Dhor2 GCAACAAGGACACTACCCAAGTATG(SEQ ID NO:64),和
3’Dhor2 GCTGACAATGAGCTGAGACATGTAG(SEQ ID NO:65)
纯化扩增产物且确定每个的核苷酸序列。来自Sloop的装配序列为2838bp长(SEQ ID NO:72)且来源自Ethiopia R118的序列为2724bp长(SEQ ID NO:73)。两种蛋白的预测的ATG翻译起始密码子在各自序列的398-400位置处。对于Sloop,终止密码子在核苷酸位置2641-2643处,其编码747个氨基酸残基蛋白。来自Ethiopia R118的序列分析揭示:相比于Sloop,在位置848(相对于起始密码子的位置450)处C变化为G,这将框内TAG终止密码子引入Hor3基因,这导致蛋白位置150处核苷酸的截短(图3)。这截短了含有鉴定为对乳糜泻患者具有免疫原性的表位的脯氨酸和谷氨酰胺富集结构域前面的D-大麦醇溶蛋白多肽。
该核苷酸取代消除了存在于栽培品种Sloop的野生型核苷酸序列中的KpnI位点且允许开发共显性CAPS标记物。为此,使用引物5’Dhor-标记物(GGCAATACGAGCAGCAAAC,SEQ ID NO:66)和3’Dhor-标记物(CCTCTGTCCTGGTTGTTGTC,SEQ ID NO:67)扩增272bp的DNA片段(图4)。然后将扩增产物用限制性酶KpnI孵育且在琼脂糖凝胶上电泳。通过KpnI消化来自野生型大麦Sloop的片段以产生164bp和108bp的两种片段。相反,不消化来自D-无效Ethiopia R118的272bp片段。因此,这个方法明确区分用于编码D-大麦醇溶蛋白的基因的野生型和无效等位基因。
实施例6.大麦Hor2基因座处缺失突变的分子标记物的鉴定
大麦染色体1H短臂上的Hor2基因座包含约20-30个基因的家族,每个编码约36-45kDa的大小范围的B-大麦醇溶蛋白(Anderson等人,2013)。56是γ-射线诱导的突变,该突变具有包括所有或几乎所有B-大麦醇溶蛋白基因的约86kb的缺失(Kreis等人,(1983))。该突变用于生成如由Tanner等人(2010)先前描述的Hor2-lys3a双重突变,表示总大麦醇溶蛋白的显著降低的水平。
如下使用标准PCR条件,设计且测试分子标记物以检测野生型Hor2基因座并将它与Hor2-缺失基因座进行区分。不存在800bp的B1-大麦醇溶蛋白PCR带诊断出不存在(缺失)Hor2基因座(即,Hor2处存在突变体等位基因)。用于扩增的引物对为:
3’B1 Hor TCGCAGGATCCTGTACAACG(SEQ ID NO:68)
5’B1 Hor CAACAATGAAGACCTTCCTC(SEQ ID NO:69)
使用以下引物对,对每个DNA样品也进行在450bp处产生特有PCR带的对照PCR反应,表明存在γ-大麦醇溶蛋白基因座且确保样品DNA的质量足以用于Hor2反应:
5’γ Hor 3 CGAGAAGGTACCATTACTCCAG(SEQ ID NO:70)
3’γ 3全 AGTAACAATGAAGGTCCATCG(SEQ ID NO:71)
实施例7.三重无效大麦突变体的生成
在温室中种植在WO2009/021285中鉴定为G1*的Hor2-lys3a双重突变体大麦系的F5植物以产生F6后代。然后将F6植物种植在田间以产生F7后代。为了将Hor2-lys3a突变和Hor3无效突变合并,F7代植物与来源自Ethiopia R118(实施例5)的D-大麦醇溶蛋白阴性BC2植物杂交且F1后代自交以产生F2种子。将F2种子切半且使半-胚芽发芽,且如实施例5和6通过B-和D-大麦醇溶蛋白PCR筛选幼苗的γ-大麦醇溶蛋白。每个包含胚乳的种子的剩余一半在含有8M尿素、1%DTT和由SDS-PAGE分离的提取蛋白的溶液中磨碎。在大约50kDa处不存在特有的蛋白带,表明不存在C-大麦醇溶蛋白蛋白。从约300粒F2种子中鉴定称为T1、T2和T3的三种三重-无效大麦醇溶蛋白。通过孟德尔式遗传学(Mendelian genetics)的组合三种隐性突变(每种为纯合状态)的预期频率是1/64,推测Hor2(B-大麦醇溶蛋白)和Hor3(D-大麦醇溶蛋白)基因座在染色体1H上相隔足够远以易于重组。
维持对称为T1、T2和T3的三种突变(Hor2-lys3a-Hor2)中每一种为纯合的三种植物且使其繁殖高达三代单种子后代,每代中选择12粒最重的种子。来自这些系的F3种子的平均种子重量为:T1,38.2mg;T2,37.0mg;T3,39mg。测量每种系的种子产量(每20穗的种子克数)和植物高度。丢弃产生不良充穗的植物。选择两种F4系:T2-4-8和T2-6-A5且进一步进行田间研究。其中,因为稍佳的谷粒产量选择T2-4-8且命名为大麦ULG3.0。
与粒度和形状以及谷粒产量指标有关的大麦谷粒重要表型是不能通过具有2.8、2.5、2.2和2.0mm筛目尺寸的筛(特别是2.8mm筛)的谷粒的百分比。相对于野生型大麦,较小的谷粒使得加工和制麦芽效率更低。该表型称为“2.8mm筛选”且以不能通过特殊筛的谷粒的百分比表示。对于野生型栽培品种诸如Sloop,2.8mm筛选参数通常为95-98%。对于大麦醇溶蛋白缺陷系诸如Hor2-lys3a双重突变体(ULG2.0),2.8mm筛选参数一般为约53%。有时,取决于生长条件例如干旱,其会低于10%且大部分谷粒可通过2.5mm筛。对于ULG3.0,2.8mm筛选参数为约54%。田间种植谷粒的平均重量(mg)为:Sloop,53.6+/-0.9,ULG2.0,33.5+/-0.4;ULG3.0,39.1+/-0.3。因此,相对于野生型Sloop(100%),ULG3.0提供69%的谷粒产量,相比而言,ULG2.0为50%。因此,相比于ULG2.0系,ULG3.0表示谷粒产量的实质性改善。然而,对于大麦ULG3.0,2.8mm筛选参数仍然是个问题。
实施例8.其它具有增加的产量的三重无效大麦突变体的生成
尽管相比于ULG2.0,ULG3.0大麦系产生增加的谷粒产量,但其仍然需要更多的增加。因此,ULG3.0植物与野生型栽培品种Sloop、Baudin和Yagan植物和鉴定为含50%每种亲本种质的大麦醇溶蛋白三重无效系的植物杂交。这些大麦醇溶蛋白三重-无效系互交,且也与野生型栽培品种Hindmarsh和Commander杂交。包含所有这些无效突变的后代与Sloop、Baudin、Hindmarsh和Commander植物和通过单粒传代产生的更纯合系回交两次。选择产生的众多所得系中的一种且将其命名为大麦ULG3.1。
与Sloop、Yagan和Baudin植物互交,进行第二轮的互交以组合所有三种以每个包含三重无效突变的植物起始的亲本栽培品种的遗传背景。从互交的F1植物,预期所有后代包含所有三种突变。在田间以行栽种每个谱系约1000粒F2种子,且选择相对较矮的F2植物和产生较大的优良充穗的F3种子。选择早熟和晚熟植物。在田间种植的下代中,选择另外展示相对高的谷粒淀粉酶、相对高收获指数和穗长、最佳高度(半矮化)、缺乏倒伏和对白粉病有疾病抗性的系。1500个家族中,选择最优的约20个。最优的20个系的数据提供于表5中。改良单个种子重量(籽粒重),使其超出ULG 3.0的41.8mg/种子的单个种子重量,观察到系P12072-2的最高种子重量为48.4mg/种子。通过使收获指数(种子形成效率的量度)增加到高于40%实现种子大小的这种改良,其中系P12124-1中测量的最高收获指数为46.5%。最重要的是,2.8mm筛选百分比也从ULG3.0的53.5%改良为高于80%,系P12140-1高达97.3%。
通过单粒传代将选择的系固定以获得对于在Hor2-lys3a-Hor3处的三种无效等位基因是纯合的植物且将其命名为ULG3.2。与相比于Sloop的约97%、Hindmarsh的约85%、野生型栽培品种Oxford的96%和Maratime的98%,田间种植的几种重复份中ULG3.2的2.8mm筛选参数范围为80-93%。
平均种子重量和厚度为:ULG2.0,33.4mg,2.4mm;ULG3.0,41.8mg,2.5mm;ULG3.2,47.2mg,2.8mm。
表5.ULG 3.2系的第二次筛选(2012年6月)。
实施例9.三重无效大麦突变体中大麦醇溶蛋白水平的测量
通过多个反应监测质谱(MRM MS)确定ULG3.0面粉中大麦醇溶蛋白含量
为了精确地测量大麦醇溶蛋白含量,如下使用MRM MS测定。研磨谷粒和半谷粒以产生面粉,所述作为全麦粉具有与整个谷粒一样的成分。使用200μL含有55%(v/v)异丙醇和2%(w/v)二硫苏糖醇(DTT)的溶液,提取20mg面粉样品中的谷醇溶蛋白多肽。通过使用10kDa MW截留过滤器单元离心三次,使等效于5mg面粉的提取物的等份试样经历缓冲液交换到于0.1M Tris-HCl(pH 8.5)中的8M尿素中。通过加入100μL 50mM碘乙酰胺并在室温下孵育1小时使多肽中的半胱氨酸烷基化。将缓冲液交换为100μL 50mM碳酸氢铵(pH8.5),且用10μL(20μg)胰蛋白酶将多肽在37℃下消化18小时。通过10kDa过滤器过滤收集肽、干燥且复溶在30μL 1%(v/v)甲酸中。经10分钟,通过液相色谱在具有Phenomemenex柱(Kinetex,1.7μm,C18,100×2.1mm)的Shimadzu Nexera HPLC上利用5%B至40%B梯度以0.4mL/min的流速将肽分离。溶剂A为0.1%(v/v)甲酸水溶液,溶剂B为含有0.1%(v/v)甲酸的90%(v/v)乙腈。HPLC洗脱液直接耦合到质谱仪上且在靶向大麦醇溶蛋白来源的胰蛋白酶肽的4000QTRAP质谱仪上进行MRM分析。使用Analyst v1.5软件和使用了(峰面积积分)的MultiQuant v2.0.2软件分析数据。
图5显示从选择的B-大麦醇溶蛋白、C-大麦醇溶蛋白、D-大麦醇溶蛋白、γ-3-大麦醇溶蛋白(G3)和γ-1-大麦醇溶蛋白(G1)获得的数据。图5示出了以下的每种标准化为Sloop(100%)水平的肽MRM转换的平均峰面积:四种来自每个对照大麦(野生型,cv Sloop)的半谷粒的重复注射液、大麦醇溶蛋白单一-无效系:56、1508和来源自Ethiopia R118的D-无效系、大麦醇溶蛋白双重-无效系ULG2.0及三重-无效系T2-4-8和T2-6-A5(圆圈标示)。选择一种原型肽代表每个大麦醇溶蛋白家族,即:对于B-大麦醇溶蛋白,TLPTMCSVNVPLYR(SEQ ID NO:48);对于D-大麦醇溶蛋白,DVSPECRPVALSQVVR(SEQ ID NO:49);对于C-大麦醇溶蛋白,LPQKPFPVQQPF(SEQ ID NO:50);对于G3-大麦醇溶蛋白,QQCCQQLANINEQSR(SEQ ID NO:50)和对于G1-大麦醇溶蛋白,CTAIDSIVHAIFMQQGR(SEQ ID NO:51)。这些肽频繁且以相对高的丰度出现在野生型大麦中,且在那个基础上进行选择。
可看出,来自系T2-4-8的三重-无效ULG3.0谷粒和第二种三重-无效系T2-6-A5没有可检测到水平的B-、C-、D-或最出人意料的γ-1-大麦醇溶蛋白。也就是,相对于野生型,观察到低于1%的水平。以同样的方式,在ULG3.0谷粒中检测不到γ-2-大麦醇溶蛋白。存在相对低水平的γ-3-大麦醇溶蛋白(圆圈标示),相比于Sloop中的G3水平,其以约20%的水平存在。γ-3-大麦醇溶蛋白是次要大麦醇溶蛋白;Sloop中γ-3大麦醇溶蛋白含量远低于总大麦醇溶蛋白含量的1%。单一-无效和双重-无效谷粒不积累适当的大麦醇溶蛋白,例如56和ULG2.0如预期那样不积累B-大麦醇溶蛋白,且1508和ULG2.0如预期那样不积累C-大麦醇溶蛋白。D-无效谷粒展示野生型水平的B-和C-大麦醇溶蛋白,但不积累D-大麦醇溶蛋白。
当使用几种不同的肽序列、尤其是存在于野生型D-大麦醇溶蛋白朝向C端(恰好在B-大麦醇溶蛋白突变体中终止密码子的位置后)的D-大麦醇溶蛋白肽AQQLAAQLPAMCR(SEQ ID NO:85)重复分析时,可得到相似的结果(图6)。面粉中也不存在燕麦蛋白样A蛋白。
显然,从大麦醇溶蛋白三重无效T2-4-8和T2-6-A5中获得的谷粒不含有可检测水平的B-、C-、D-大麦醇溶蛋白和选择的γ-1-(P17990)和γ-2-大麦醇溶蛋白。对γ-1和γ-2大麦醇溶蛋白的观察结果是发明者最出人意料,因为三重无效突变体系被认为不含有任何可完全使相应基因沉默的突变。
如由MRM确定的ULG3.0谷粒的低大麦醇溶蛋白含量如下通过二维凝胶电泳方法证实。根据Tanner等人(2013),用0.006%(w/v)胶体考马斯G250(Colloidal Coomassie G250)染色50μg来自每个大麦醇溶蛋白无效系T2-4-8和T2-6-A5以及对照大麦cv 56(每个加标有1μg标志多肽标准物BSA、大豆胰蛋白酶抑制剂和马肌红蛋白)的面粉提取物的乙醇可溶性蛋白,并与20、30、40、50、60、80和kDa的标准蛋白对比(M;Benchmark Protein Ladder,Invitrogen)。切去2D凝胶的斑点且通过胰蛋白酶肽的质谱鉴定来自对照56的以下蛋白:C-大麦醇溶蛋白、γ-2-大麦醇溶蛋白(γ2)、γ-3-大麦醇溶蛋白(γ3)和γ-1-大麦醇溶蛋白(γ1)。通过与56凝胶对比鉴定凝胶中来自ULG3.0谷粒的γ-1-、γ-2和γ-3-大麦醇溶蛋白的预测位置。在ULG3.0面粉中仅观察到γ-3-大麦醇溶蛋白,未检测到其它三种多肽。通过三种方法将每个斑点的γ-3-大麦醇溶蛋白浓度测量为:1)50μg蛋白的所有斑点体积的百分比:ULG3.0平均γ3含量为13.5±1.6ppm;2)相对于1μg BSA的斑点强度:ULG3.0平均γ3含量为10.9±1.3ppm;3)相对于1μg BSA的斑点体积(强度×面积):ULG3.0平均γ3含量为3.4±0.41ppm。
如由MRM确定的ULG3.0谷粒的低大麦醇溶蛋白含量通过ELISA方法如下再次被证实。将20mg全麦粉样品或半谷粒胚乳压碎且用0.5ml MilliQ水通过在96孔振动磨碎机(Retsch Gmbh,Rheinische)中以30/s震荡3×30秒洗涤三次且以14,000rpm离心5分钟。对于对照系Sloop、56、1508和对于ULG2.0、大麦醇溶蛋白三重-无效系T1、T2和T3以及来自T2-4-8和T2-6-A5的单粒后代传代物,将面粉中的谷醇溶蛋白提取到由0.5ml 50%(v/v)异丙醇/1%(w/v)DTT组成的乙醇溶液中。根据Bradford(1976)确定蛋白浓度且40ng(1900ng,对于三重-无效谷粒)醇溶性蛋白利用含有ELISA体系稀释液的溶液稀释,加入恒定过量的0.2mM H2O2以猝灭从初始提取物中剩余的任意DTT猝灭。将稀释的蛋白溶液加入到ELISA平板孔(ELISASystems,Windsor,Queensland,澳大利亚)中,根据生厂商的指导说明洗涤且在37℃下显色15分钟。对照0-50ng Sloop总大麦醇溶蛋白标准校准对照提取物中大麦醇溶蛋白的量。对照0-5ng ULG2.0总大麦醇溶蛋白标准校准三重-无效大麦醇溶蛋白含量。如由Tanner等人(2010)所描述制备Sloop和ULG2.0大麦醇溶蛋白。
通过ELISA方法,相对于野生型cv Sloop,双重-无效面粉样品的总大麦醇溶蛋白含量为2.9%,而两种选定的大麦醇溶蛋白三重-无效系:T2-4-8和T2-6-A5的剩余大麦醇溶蛋白含量为3.9和1.5ug/g(百万分率,ppm;表6),均显著低于无谷蛋白食物中谷蛋白20ppm的FSANZ法定水平且比野生型cv Sloop谷粒低大约15,000倍。
通过多个反应监测质谱(MRM MS)确定ULG3.0啤酒中大麦醇溶蛋白含量
通过检测特异性肽序列的MRM MS,使用实施例3和4中描述的稍修饰的方法,测量由大麦ULG3.0谷粒酿造的啤酒中的大麦醇溶蛋白含量。数据示于表7中。该测定显示,缺乏燕麦蛋白样A蛋白、B1-和B3-大麦醇溶蛋白、D-大麦醇溶蛋白以及降低水平的γ-1-大麦醇溶蛋白和γ-3-大麦醇溶蛋白,因为这些未检测到高出质谱中的背景噪音(图7)。该测定也显示不存在C-大麦醇溶蛋白。
表6.大麦醇溶蛋白单-、双重-和三重-无效系中大麦醇溶蛋白含量的总结。
通过MRM MS确定ULG3.1和ULG3.2面粉中大麦醇溶蛋白含量
按照对ULG3.0谷粒的描述,通过MRM MS确定从来自ULG3.1系和ULG3.2的10种候选系的谷粒研磨面粉中大麦醇溶蛋白含量。将半谷粒研磨成面粉且将20mg面粉(n=4个重复份)的谷醇溶蛋白提取、烷基化、胰蛋白酶消化以及如上所述通过MRM MS对其进行分析。将数据在图8中作图,该数据显示ULG3.1和由双重亲本互交系(命名为043-2–148-2)产生的系的每个半谷粒的每种肽(三种MRM转换的总和)的平均峰面积。这些在比较对照大麦(野生型栽培品种Sloop、Baudin、Commander和Hindmarsh)与三重-无效系T2-4-8时进行作图。显示选定的原型肽的峰面积,每个大麦醇溶蛋白家族代表、Uniprot登录和氨基酸序列如下:
A-F2EGD5_QQCCQPLAQISEQAR(SEQ ID NO:52;来自F2EGD5,在燕麦蛋白样A蛋白中央)
B1-Q40020_VFLQQQCSPVR(SEQ ID NO:53;靠近B1-大麦醇溶蛋白的N端)
B3-Q4G3S1_VFLQQQCSPVPMPQR(SEQ ID NO:54)
C-Q40055_QLNPSHQELQSPQQPFLK(SEQ ID NO:55;靠近C-大麦醇溶蛋白的N端)
D-Q84LE9_ELQESSLEACR(SEQ ID NO:56;来自Q84LE9,在Y150终止密码子之前)
G1-P17990_APFVGVVTGVGGQ(SEQ ID NO:57;来自P17990,C端肽),和
G3-P80198_QQCCQQLANINEQSR(SEQ ID NO:58;来自P80198,在γ3-大麦醇溶蛋白中央)。
图8中显示的双-亲本互交谷粒中的D-大麦醇溶蛋白水平与来自ULG2.0和大麦醇溶蛋白三重无效系T2-4-8和T2-6-A5的谷粒中的D-大麦醇溶蛋白水平相似,接近零,从而通过2D PAGE证实该观察:在来自T2-4-8和T2-6-A5系的谷粒中未检测到D-大麦醇溶蛋白。这些双-亲本互交谷粒中的γ-1-大麦醇溶蛋白水平也与ULG2.0和大麦醇溶蛋白三重无效系T2-4-8和T2-6-A5中的γ-1-大麦醇溶蛋白水平相似。几种ULG3.2系具有接近零水平的肽APFVGVVTGVGGQ(SEQ ID NO:59)。通过T2-4-8和T2-6-A5系的2D PAGE未检测到γ-1-大麦醇溶蛋白。该双-亲本互交谷粒中的γ-3-大麦醇溶蛋白水平也与ULG2.0和大麦醇溶蛋白三重无效系中的那些相似,除了γ-3-大麦醇溶蛋白水平非常低的系124.1(ULG3.2)。通过T2-4-8和T2-6-A5系的2D PAGE也检测到降低水平的γ-3-大麦醇溶蛋白。
有趣的是,观察到Hor2和lys3a突变在ULG2.0中降低D-大麦醇溶蛋白含量方面存在协同作用,即使不存在Hor3突变(D-大麦醇溶蛋白)。以类似的方式,所有三种突变(Hor2-lys3a-Hor3)的存在对降低如由上述肽界定的γ-1-和γ-2-大麦醇溶蛋白积累有协同作用。
实施例10.三重无效去壳大麦突变体的生成
用于选择体ULG3.0、ULG3.1和ULG3.2的大麦谷粒全部去壳,这对于发酵工业是有利的,因为在麦芽汁过滤的最终阶段(过滤(lautering))废壳形成过滤床。然而,大麦谷粒壳具有大量微型二氧化硅穗且因此在人食用前需要通过脱壳除去壳。替代方式是通过遗传手段产出去壳谷粒。因此,ULG3.0植物与命名为Barleymax II(WO2011/011833)的去壳大麦品种以及选定的去壳、大麦醇溶蛋白B-、C-、D-三重无效突变体(Hor2-lys3a-Hor3)植物(对于SSIIa基因,它是野生型)杂交。
一些F6大麦醇溶蛋白三重-无效选择体(例如A7_1)在三轮单粒后代后在面粉中含有低于0.1ppm的总大麦醇溶蛋白。
实施例11.大麦的诱变
为了分离大麦中选择基因的突变体,如由Caldwell(2004)所描述,进行乙基甲磺酸酯(EMS)诱变。如下对大约45,000(1.5kg)粒ULG3.0谷粒诱变:在室温通风下,将谷粒在2.5L蒸馏水中吸收4小时。吸涨期间,每小时更换水。然后将种子在2.5L现制备的于0.1M磷酸盐缓冲液(pH7)中的30mM EMS室温通风孵育16h。然后室温下,用2.5L 100mM硫代硫酸钠将种子洗涤10分钟。用硫代硫酸盐重复洗涤,且然后室温下,用2×2L蒸馏水将谷粒通风彻底冲洗30分钟。在次日田间播种之前,将种子在吸附性滤纸上,在流动气流下过夜风干。将大量M2种子收获、汇集且分析突变体。此类处理后,突变频率是每1000粒种子目标基因中大约一个突变体。使用γ-3-大麦醇溶蛋白特异性单克隆抗体,通过半粒的斑点印迹,筛选种子的γ-3-大麦醇溶蛋白表达损失,且从数万个M2谷粒中鉴定40粒产生阴性信号的谷粒。这些谷粒中γ-3-大麦醇溶蛋白的缺乏通过蛋白质印迹使用单克隆抗体且通过特异性肽(SEQ ID NO:58)的质谱证实。将包含来自每个阴性谷粒的胚胎的半谷粒涂铺在培养基上以使其发芽。在大多数半谷粒中胚胎不发芽,但少量发芽。
实施例12.由低大麦醇溶蛋白大麦产生啤酒
使用由ULG2.0谷粒产生的麦芽,通过标准方法进行酿造试验,且与对照野生型大麦cv.Gairdner对比。Gairdner为高产量的中到晚熟的半矮化2-行大麦品种,其广泛种植于整个澳大利亚谷物种植区。在有利的条件下,其产生优良的粒度,从而产生适度的提取水平、发酵和淀粉酶力,且因此代表工业“标准”,所以它用作酿造评价试验中的对照是理想的。相比于Gairdner麦芽(5.0%),从ULG2.0产生的麦芽具有5.7%的稍高含水量且外观与Gairdner麦芽显著不同,因为该谷粒外观凹皱。ULG2.0麦芽的淀粉酶力(DP)是54WK,比Gairdner的DP(299WK)低很多。制麦芽的大麦一般具有至少250KW的DP。然而,20分钟的淀粉糖化时间后ULG2.0麦芽对于淀粉是阴性的且达到1.7°Plato的LG结果。
两次通过2-辊压机将麦芽研磨且达到满意的谷粒破裂。由于它的谷粒形态,该麦芽将极适于在6-辊压机中的更复杂研磨。可替代地,可使用与醪液过滤器相连的锤式粉碎机,而不是用于麦芽汁分离的过滤槽。通过标准方法在初始温度65℃下将研磨的产物淀粉糖化20分钟,然后在74℃下浆煮5分钟,加入额外的喷洒液体。总体来说,相比于规则的麦芽,ULG2.0麦芽的皱形态使得它难被满意地研磨和淀粉糖化。然后将淀粉糖化的产物过滤,其中ULG2.0的皱形态再次引起麻烦。过滤床因沟流而解体,所以需要停止径流且将过滤床再次倾斜。由于无效研磨,在过滤过程中损失了相当多的潜在提取物,当目标为实现14°Plato时,实现总计10.96°和11.12°Plato的釜值。pH、EBC颜色和β-葡聚糖水平是可接受的。研磨低效也意味着无有效形成壳床以作为过滤器介质,从而造成更低的预期提取物。尽管麦芽汁澄清度因30L径流改善为可接受的,但是由于床形成不良,所以径流的澄清度最初非常浑浊。
将所得的麦芽汁用酵母菌株葡萄汁酵母(Saccharomyces uvarum)A在18.5℃下发酵120小时。尽管仍然有显著水平的联乙酰,甚至在达到发酵重力(fermentation gravity)的结束后给定延长的联乙酰静置阶段后,但发酵曲线正常,发酵开始时无延长的停滞阶段。联乙酰静置是将啤酒致冷至0℃前将啤酒置于较高发酵温度下以使酵母再吸收和代谢联乙酰。一般来讲,在发酵结束时,需要24小时以使得酵母有时间分解这个产物。两次ULG2.0酿造试验并不如此。加入30mg/L异构酒花提取物并加入二氧化硅水凝胶澄清液体。最终嫩啤酒具有比对照酿造品更低的物理稳定性。初始的冷却浑浊被认为高且强制冷却浑浊结果超出常规标准。然而,冷却浑浊不形成颗粒。
使最终啤酒经受一组受训过的酿造师进行感官分析,且不管研磨和过滤过程中的困难,该结果出人意料的好(考虑到与麦芽的酿造性能有关问题)。DMS(甲硫醚)是主要风味;然而这不会被认为是异议。在澳大利亚啤酒中,DMS的存在常被认为是错误的风味,但是在欧洲尤其是德国的啤酒中却通常被普遍接受。酿造师的评述是:ULG2.0啤酒与对照啤酒没有太多的不同且它们作为啤酒是非常合格的,且使人联想到德国啤酒。ULG2.0啤酒中无明显的“颗粒状”或“谷物”类型调味剂且不烈或不涩,ULG2.0啤酒可为称为“无谷蛋白”的市售啤酒的典型。总体来说,风味性质是可接受的和合理的。
通过用于ULG2.0的相同方法由ULG3.0的大麦谷粒制备啤酒,且由ULG3.2大麦谷粒制备啤酒。相对于ULG3.0谷粒,ULG3.0谷粒的制麦芽有所改善,主要因为谷粒皱更少而改善了研磨步骤。由ULG3.0制备的啤酒具有良好质量以及可接受且合理的风味。ULG3.2谷粒的改善谷粒形态(大小和形状)使得其酿造过程中更容易研磨和过滤,提供具有低于1ppm的总大麦醇溶蛋白的啤酒。
实施例13.使用ULG大麦的食物的生产
使用ULG3.0和ULG3.2大麦系烘培两种小型(10g)面包。为了测试的目的烘培小型面包,但该方法可易于扩大成商业量。一种面包用如上述研磨的100%ULG大麦面粉作为面粉成分制作,而第二种面包用30%面粉和70%市售非谷蛋白面粉诸如小米面粉作为面粉成分制作。面粉(13.02g)和其它成分混合成面团以在35-g混粉图中使面团形成时间达到峰值。在每种情况下基于13.02g面粉所使用的配方为:面粉100%、盐2%、干酵母1.5%、植物油2%和改进剂1.5%。加水水平基于对整个配方调整的微量Z-臂水吸收值。在40℃和85%室湿度下,以两阶段检验步骤进行模制和筛选。在Rotel炉中190℃下烘培14分钟。
面包包含小于20ppm的谷蛋白含量且适于患有CD的受试者食用。
本领域技术人员将认识到,在不偏离如宽泛描述的本发明的精神或范围的情况下,可以对本发明进行多种如具体实施方案所示的变形和/或修改。因此本发明的实施方案在各方面都被认为是说明性的而不是限制性的。
本申请要求2013年6月13日提交的AU 2013902140和2013年7月11日提交的AU 2013902565优先权,它们两者的全部内容通过引用并入本文。
本文讨论和/或参考的所有出版物都整体并入本文。
对包括在本说明书中的文献、行为、材料、装置、物品等的任何讨论仅仅是为了给本发明提供一个背景的目的。不应认为承认任何或所有这些内容形成部分现有技术基础或是与本发明相关的领域的常见的一般常识,因为在它在本申请的每项权利要求的优先权日之前已经存在。
参考文献
Abdullah et al.(1986)Biotechnology 4:1087.
Almeida and Allshire(2005)TRENDS Cell Biol 15:251-258.
Anderson et al.(2000)Nature Medicine 6:337-342.
Anderson et al.(2013)Genome 56:179-185.
Bibikova et al.(2001)Mol.Cell.Biol.21:289-287.
Bibikova et al.(2002)Genetics 161:1169-1175.
Bourque(1995)Plant Sci.105:125-149.
Brandt et al.(1990)Eur J Biochem 194:499-505.
Brennan et al.(1998)J Cereal Sci.28:291-299.
Caldwell(2004)The Plant Journal 40:143-150.
Cameron-Mills et al.,(1998).Plant Mol Biol 11:449-461.
Capecchi(1980)Cell 22:479-488.
Catassi et al.(1994)Lancet 343:200-203.
Certo et al.(2012)Nat Methods 8:941-943.
Cheng et al.(1996)Plant Cell Rep.15:653-657.
Clapp(1993)Clin.Perinatol.20:155-168.
Colgrave et al.,(2012)J.Proteome Res.11:386-396.
Comai et al.(2004)Plant J 37:778-786.
Curiel et al.(1992)Ham.Gen.Ther.3:147-154.
Davies et al.(1993)Cell Biology International Reports 17:195-202.
De Anglis et al.(2007)J Food Protection 70:135-144.
Doll(1983)Barley seed proteins and possibilities for their improvement.In″Seed Proteins:Biochemistry,Genetics,Nutritional Value",Gottschalk W,Muller HP(eds).Martinus Nijhoff,The Hague:207-223.
Doll et al(1973)Barley Genetics Newsletter 3:12-13.
Dostalek et al.(2006).Food Additives and Contaminants 23:1074-1078.
Doyon et al.(2010)Nat.Methods 7:459-460.
Doyon et al.(2011)Nat.Methods 8:74-79.
Eglitis et al.(1988)Biotechniques 6:608-614.
Fasoli et al.,(2010).J.Proteome Res.9:5262-6269.
Field et al.(1982)Theoretical and Applied Genetics 62:329-336.
Fowell et al.(2006)Qjm-an InternationalJournal of Medicine 99:453-460.
Fujimura et al.(1985)Plant Tissue Culture Letters 2:74.
Graham et al.(1973)Virology 54:536-539.
Grant et al.(1995)Plant CellRep.15:254-258.
Green(2009)Journal of the American Medical Association 302:1225-1226.
Green and Jabri(2006)Annual Review of Medicine 57:207-221.
Groenen et al.(1993)Mol.Microbiol.10:1057-1065.
Gu et al.(2003)Genome 46:1084-1097.
Guo et al.(2010)J.Mol.Biol.400:96-107
Haft et al.(2005)Computational Biology 1(6):e60
Haseloff and Gerlach(1988)Nature 334:585-591.
Hausch et al.(2002)Gastroenterology 122:A 180.
Henikoff et al.(2004)Plant Pbysiol135:630-636.
Hoe et al.(1999)Emerg.Infect.Dis.5:254-263.
Ishino et al.(1987)J.Bacteriol.169:5429-5433.
Janssen et al.(2002)OMICS J.Integ.Biol.6:23-33.
Jaradat(1991)Theor Appl Genet 83:164-168.
Kahlenberg et al.(2006)European Food Research Technology.222:78-82.
Kasarda et al.(1984)PNAS 81:4712-4716.
Kikuchi et al.,(2003).Theor Appl Genetics 108:73-78.
Kim et al.(1996)Proc.Natl.Acad.Sci.USA 93:1156-1160
Kim et al.(2012)Genome Res.22:1327-1333.
Koziel et al.(1996)Plant Mol.Biol.32:393-405.
Kreis and Shewry(1989)BioEssays 10:201-207.
Kreis et al.(1983)Cell:34:161-167.
Kristofiersen et al.,(2000)Electrophoresis 21:3693-3700.
Kupper(2005)Gastroenterology 128:S 121-127.
Lanzini et al.(2009)Alimentary Pharmacology&Therapeutics 29:1299-1308.
Lee et al.(2007).Journal of Human Nutrition and Dietetics 20:423-430.
Lu et al.(1993)J.Exp.Med.178:2089-2096.
Marti et al.(2005)J pharmacol Exp Therapeut 312:19-26.
Masepohl et al.(1996)Biochim.Biophys.Acta 1307:26-30.
McConnell Smith et al.(2009)PNAS 106:5099-5104.
Millar and Waterhouse(2005)Funct Integr Genomics 5:129-135.
Miller et al.(2007)Nat.Biotechnol.25:778-785.
Mojica et al.(1995)Mol.Microbiol.17:85-93.
Mojica et al.(2000)Mol.Microbiol.36:244-246.
Moravcova et al.(2009).Journal of Chromatography a 1216:3629-3636.
Nakata et al.(1989)J.Bacteriol.171:3553-3556.
Ohlund et al.(2010).Journal of Human Nutrition and Dietetics 23:294-300.
Pasquinelli et al.(2005)Curr Opin Genet Develop 15:200-205.
Perriman et al.(1992)Gene 113:157-163.
Picariello et al.(2011).Food Chemistry 124:1718-1726.
Ramirez et al.(2012)Nucleic Acids Res.40:5560-5568.
Rubio-Tapia et al.(2010).American Journal of Gastroenterology 105:1412-1420.
Senior(1998)Biotech.Genet.Engin.Revs.15:79-119.
Shan et al.(2002).Science 297:2275-2279.
Shewry(1995)Biological Reviews of the Cambridge Philosophical Society 70:375-426.
Shewry and Halford(2002)Journal of Experimental Botany 53:947-958.
Shewry and Tatham(1990)BiochemicalJournal 267:1-12.
Shewry,et al.,(1999)The prolamins of the Triticeae.In Seed Proteins,Klewer:London,1999;pp 35-78.
Shippy et al.(1999)Mol.Biotech.12:117-129.
Skovbjerg et al.(2005)Diabetologia 48:1416-1417.
Skovbjerg et al.,(2004).Biochim.et Biophys.Aeta-Mol.Bas.of Dis.1690:220-230.
Slade and Knauf(2005)Transgenic Res 14:109-115.
Smith et al.(2000)Nature 407:319-320.
Stepniak et al.(2006)Am J Physiol-Gastrointest Liver Physiol 291:621-629.
Szczepek et al.(2007)Nat.Biotechnol.25:786-793.
Tanner et al.(2013)Plos One:8:e56456.
Tanner et al.,(2010).Aliment Pharmacol Ther.32:1184-1191.
Tjon et al.(2010)Immunogenetics 62:641-651.
Toriyama et al.(1986)Theor.Appl.Genet.205:34.
Tye-Din et al.(2010)Science Transl ational Medicine 2:41-51.
van Embden et al.(2000)J.Bacteriol.182:2393-2401.
Wagner et al.(1992)Proc.Natl.Acad.Sci.USA 89:6099-6103.
Wang et al.(2012)Genome Res.22:1316-1326.
Waterhouse et al.(1998)Proc.Natl.Acad.Sci.USA 95:13959-13964.
Wijngaard and Arendt(2007)Brewer&Distiller International 3:31-32.
Wild et al.(2010).Alimentary Pharmacology&Therapeutics 32:573-581.
- 还没有人留言评论。精彩留言会获得点赞!