水稻杂种花粉育性基因座Sc的等位基因Sc‑j和Sc‑i的分离克隆和在育种中的应用的制作方法
本发明涉及植物基因工程技术领域,具体地,涉及水稻杂种花粉育性基因座sc的等位基因sc-j和sc-i的分离克隆和在育种中的应用。
背景技术:
植物通过不同品种间的杂交产生杂种。杂种在生长势、适应性、产量等方面具有优势,称为杂种优势。利用杂种优势可以提高农作物的产量、品质和抗性。亚洲栽培稻分为籼稻亚种(oryzasativassp.indica)和粳稻亚种(o.sativassp.japonica),它们间的杂种有强大的杂种优势,增产潜力很大。另一方面,通过栽培稻与野生稻杂交和回交等育种方法,可以将存在于野生稻的一些有用性状如抗病、抗虫、高产等导入栽培稻,培育出优良新品种。但是,籼粳亚种间杂交以及栽培稻与野生稻杂交产生的杂种存在育性低或不育的问题,称为杂种不育(hybridsterility)。所述的杂种不育是稻属物种分化过程中产生的生殖隔离的机制之一,属于合子后生殖隔离(postzygoticreproductiveisolation)。杂种不育现象严重阻碍了将水稻杂种优势转化为更高的稻谷产量和野生稻有用基因的利用。
在水稻中有多个基因座(loci)与水稻杂种育性有关。所述的基因座是细胞遗传学水平的概念,是指染色体的一个特定区域,可能包含1个或1个以上的相关基因控制同一性状。这些杂种育性基因座中,有些是导致雌配子不育,如s5,s7,s8,s9,s15,s17,有些是导致雄配子(花粉)不育,如sa,sb,sc,sd,se,sf(kinoshita1995,ricegenet.newsl.,12:9-15;ikehashi等1988,japanj.breed.38:283-291;ikehashi等1996,ricegenetiii,pp404-408;wang等1996,theorapplgenet92:183-190;wang等1998,ricegenetnewsl15:151-154;张桂权等1989,中国水稻科学3:97-101;张桂权等1993,遗传学报20:222-228;张桂权等1993,遗传学报20:541-551;张桂权等1994,遗传学报21:34-41)。这些基因座在不同品种或品系或野生稻材料中可以有不同程度的变异分化,产生多种复等位基因。根据携带这些复等位基因的水稻亚种类型以及对杂种育性的效应,它们被划分为籼型(s-i)、粳型(s-j)、中性型(s-n)、或其它过渡类型。所述的基因符号s代表杂种育性基因座,如sa,sc等指特定的杂种不育基因座,i,j,n分别代表籼型(indica)、粳型(japonica)、和中性型(neutral)的复等位基因座,如sc-i、sc-j、或sc-n。中性型也称为亲和型(compatibility)或广亲和型(widecompatibility)。
在水稻杂种中,分化的杂种不育基因座在杂合状态下往往发生不亲和互作,导致携带某一等位基因的雄配子(或雌配子)发育不正常,称为杂种的等位基因特异杀配子(allele-specificgameteelimination)效应。但是,这些基因座在纯合状态下(如纯合的亲本)不影响配子的正常发育。大部分杂种不育基因座的遗传行为符合“单座位孢子体-配子体互作”的模式(oka,1974,genetics77,521-534)。例如,用分别携带sc-j/sc-j基因型的水稻粳稻和sc-i/sc-i基因型的近等基因系杂交,产生的杂种一代(f1)的基因型为sc-j/sc-i。其花药减数分裂产生的雄配子(花粉)中,携带sc-i等位基因座的正常可育,而携带sc-j等位基因的雄配子发育异常,因此表现为约50%的花粉不育即半不育(图1)。但杂种雌配子的育性不受该杂合基因座的影响。结果导致杂种二代(f2)代群体中携带sc-j/sc-j基因型的植株的比例严重偏少,即偏离sc-i/sc-i:sc-i/sc-j:sc-j/sc-j=1:2:1的孟德尔分离规律(图1)。
目前已报道了3个单座位的籼粳杂种不育基因的克隆,包括s5,sa,hsa1(chen等2008,proc.natl.acad.sci.usa105,11436-11441;yang等2012,science337,1336-1340;long等2008,proc.natl.acad.sci.usa105,18871-18876;kubo等2016,mol.plant9,221-232).sc是控制水稻杂种花粉育性的主要基因座之一(yang等2004,chinesesciencebulletin49,1718-1721)。
技术实现要素:
本发明为了在杂交水稻育种中有效地克服杂种不育性问题,通过图位克隆技术在sc座位克隆了杂种育性相关基因,包括籼型和粳型等位基因;发现其中籼型等位基因座sc-i由2个或3个串联重复拷贝构成。在此基础上,本发明提出一种利用基因编辑技术定点突变sc-i的部分拷贝,创建亲和型等位基因sc-n,以恢复杂种中sc-j花粉育性,克服杂种籼粳杂种雄性不育的杂交育种方法。
本发明的第一个目的是分离克隆水稻杂种花粉育性基因座sc的dna序列及其编码的蛋白质序列。
本发明的第二个目的阐明sc基因的功能和产生杂种不育的分子遗传机制。
本发明的第三个目的是以其分子遗传机制为依据,提供创建人工的亲和型sc-n基因,培育杂种育性亲和系的方法和在籼粳杂交育种的应用。
为了实现上述目的,本发明是通过以下技术方案予以实现的:
水稻杂种花粉育性基因座sc的控制花粉育性的等位基因,包括粳稻中的单拷贝等位基因sc-j,其核苷酸序列如seqidno:1所示;和籼稻中的串联重复拷贝等位基因sc-ia和sc-ib,sc-ia和sc-ib的核苷酸序列如seqidno:2和seqidno:3所示。seqidno:1所示序列中,1~1000bp为启动子序列,2285~3678bp为内含子序列,1001~2284bp和3679~3735bp为外显子序列。seqidno:2所示序列中,1~1000bp为启动子序列,2276~3673bp为内含子序列,1001~2275bp和3674~3730bp为外显子序列。seqidno:3所示序列中,1~1000bp为启动子序列,2276~3669bp为内含子序列,1001~2275bp和3670~3726bp为外显子序列。
等位基因sc-j编码的蛋白序列如seqidno:4所示,等位基因sc-ia编码的蛋白序列如seqidno:5,等位基因sc-ib编码的蛋白序列如seqidno:6所示。
本发明发现粳稻的sc-j和籼稻的sc-i编码含有duf1618结构域的蛋白,为水稻花粉正常发育所必需。sc-j只有一个基因拷贝,sc-i在不同的品种具有2个或3个串联基因拷贝。
本发明还发现在籼稻和粳稻杂种中,sc-i多拷贝基因的高水平表达产物能抑制sc-j的表达,导致杂种花粉不育。因此,本发明建立了一种培育水稻杂种育性亲和系的方法,具体步骤为,利用基因组编辑技术将含有sc-i等位基因的水稻品种中的sc-ia或/和sc-ib部分基因拷贝定点突变,降低sc-i基因座的活性基因拷贝数,产生杂种亲和系。作为最优的实施方式,一种培育水稻杂种育性亲和系的方法,具体步骤为,利用基因组编辑技术将含有sc-i等位基因的水稻品种中的sc-ia或/和sc-ib部分基因拷贝定点突变,最后只保留一个sc-ia基因拷贝,或只保留一个sc-ib,从而降低sc-i基因座的活性基因拷贝数,产生杂种亲和系。
因为sc-i在不同的品种具有2个或3个串联基因拷贝,所以对于含有sc-ia、sc-ib1和sc-ib2的水稻品种,利用基因组编辑技术将sc-ia、sc-ib1或sc-ib2中的任意一个或任意两个基因拷贝进行定点突变,降低sc-i基因座的活性基因拷贝数,产生杂种亲和系,其中sc-ib1和sc-ib2的核苷酸序列同sc-ib,sc-ib1和sc-ib2是sc-ib的两个基因拷贝的命名。作为优选的实施方式,具体步骤为利用植物crispr/cas9基因编辑系统(ma等2015,mol.plant8,1274-1284),对籼稻sc-i区域针对sc-ib1和sc-ib2的编码区进行特异性突变,但保留sc-ia的正常功能,这些部分基因的突变可降低sc-i活性基因拷贝数,而剩余一个基因拷贝(sc-ia)的表达水平可以维持其对花粉育性的功能。
对于含有sc-ib1和sc-ib2的水稻品种,利用基因组编辑技术将sc-ib1或sc-ib2中的任意一个基因拷贝进行定点突变,降低降低sc-i基因座的活性基因拷贝数,产生杂种亲和系;sc-ib1和sc-ib2的核苷酸序列相同,是sc-ib的两个基因拷贝。优选地,本发明所述的基因组编辑技术为crispr/cas9基因编辑系统,但并不仅限于这种基因编辑技术。
作为一种优选的实施方案,利用crispr/cas9基因编辑技术将籼稻sc-i座位的sc-ib1或sc-ib2中的任意一个基因拷贝进行定点突变的具体方法为,
s1.利用sc-ib基因编码区第235位的g碱基变异,设计一个对sc-ib基因特异的crispr/cas9基因编辑靶点,构建表达此特异靶点sgrna的crispr/cas9载体;
s2.以此基因修饰载体转化含有sc-i等位基因的水稻品种,获得sc-ib等位基因特异性突变,而sc-ia等位基因为野生型的突变体。
本发明所述的培育水稻杂种育性亲和系的方法在水稻杂交育种中的应用,具体应用方法为将所述方法培育得到的水稻杂种育性亲和系和粳稻品种杂交,培育sc座位不产生花粉败育的杂种,用于水稻杂交育种。
应当理解,基于可编程核酸酶如tallens和crispr/cas9/cpf1等基因编辑技术在植物尤其水稻的应用已经相当成熟(ma等2016,molecularplant,10.1016/j.molp.2016.04.009)。在本发明提供的基因序列、基因功能、和等位基因表达水平与杂种不育的关系等信息的基础上,本领域技术人员可对这些基因选择1个或若干个不同于本发明实施的靶点进行基因删除或碱基突变,容易达到本发明的相同或相似的杂种亲和性效果。因此,本发明包含对这些基因及其启动子调控区的任意片段删除和编码区的任意位点的编辑突变。
与现有技术相比,本发明具有如下有益效果:
本发明提供的杂种育性基因及其亲和系创建方法具有重要的应用价值。本发明方法可以快速培育杂种育性亲和系,这些亲和系也可以与其它水稻品种或品系杂交,培育出新的杂种育性亲和系。这些杂种育性亲和系用于籼粳稻杂交育种,可克服sc杂种不育,提高杂种的育性,达到增产的目的。
附图说明
图1显示水稻sc基因的遗传模式及其基因型对花粉育性的效应。粳稻t65和近等基因系e5产生的正常可育花粉,其杂种f1产生约一半的花粉不育(箭头指败育花粉),以及f2群体产生的基因型分离严重偏离1:2:1,其中sc-j纯合体的数目很少,表明含有sc-j的花粉败育。
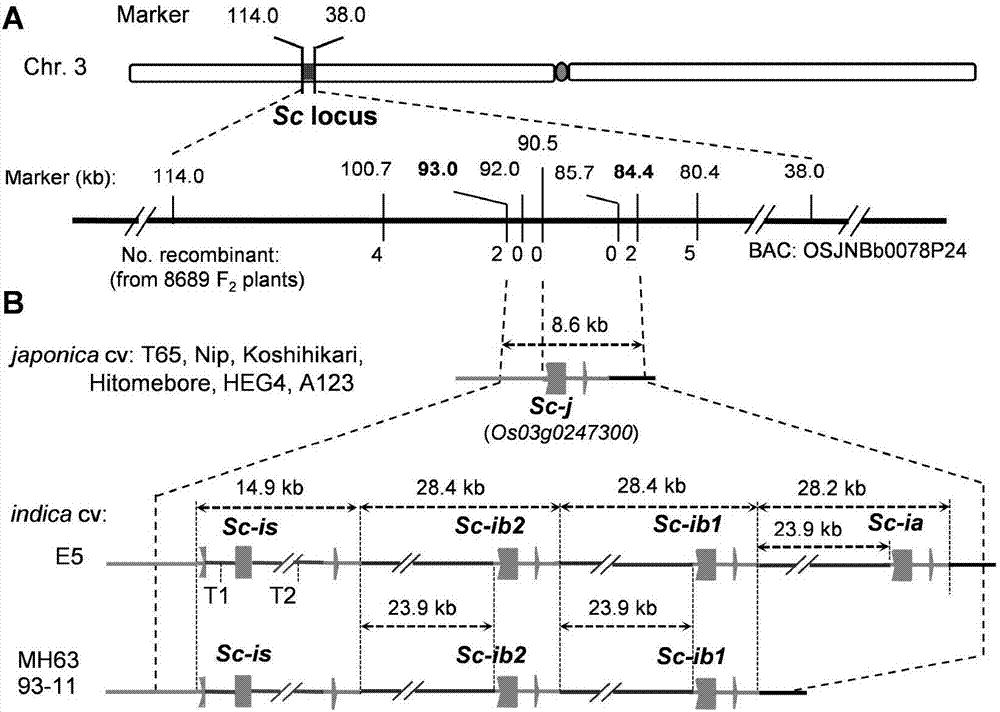
图2为水稻sc基因座在第3染色体的精细定位和基因序列结构;图(a)各标记的数字同时表示在一个公开的细菌人工染色体(bac)上的位置(kb);图(b)显示的基因组测序分析表明,sc基因座在粳稻只有1个基因os03g0247300,命名为sc-j;不同的籼稻品种的sc基因座含有2-3个os03g0247300同源串联重复基因,命名为sc-ia,sc-ib1,sc-ib2;sc-is为转座子插入破坏的假基因;sc-ia,sc-ib1和sc-ib2的启动子和上游序列与sc-j的启动子和上游序列完全不同;sc-j,sc-ia,和sc-ib(sc-ib1和sc-ib2)序列如seqidno:1~3所示。
图3为sc-j的功能研究;图(a)为将sc-j反义表达载体转化t65,获得的转基因半合植株(t1代),sc-j在其表达高峰的二核早期(early-bicellularstage)的表达水平下降;图(b)为反义转基因t1代的转基因半合植株表现花粉半不育(箭头指败育花粉);相对表达值为对内参基因osactin1的比值。
图4为定量rt-pcr检测粳稻t65,近等基因系e5,及其杂种f1的sc基因表达;图(a)以sc等位基因通用引物(p1,p2)检测不同发育时期花药的表达水平,结果显示t65的sc-j主要在二核早期特异表达;图(b)为根据sc-j的特异5’非翻译区设计的sc-j特异引物(p3,p4)检测比较杂种f1和t65在二核早期花药的每拷贝sc-j的表达水平,结果显示f1的sc-j表达水平大大低于t65的sc-j表达水平;相对表达值为对osactin1的比值。
图5为以crispr/cas9基因编辑系统修饰sc-i的部分重复拷贝;根据sc-ib1和sc-ib1的编码区第235位的g碱基变异形成tgg即pam元件(是cas9-sgrna复合体识别结合靶点序列的必需元件),设计一个对sc-ib/sc-ib1基因特异的crispr/cas9基因编辑靶点(下画线);e5-ed1和e5-ed2为特异突变了sc-ib1和sc-ib2的基因编辑亲和系。
图6为以基因编辑亲和系克服杂种不育性;图(a)为以e5-ed1和e5-ed2与粳稻t65杂交,获得花粉育性提高的基因编辑杂种f1(箭头指败育花粉);图(b)为sc-ib基因编辑杂种f2群体中sc-j纯合个体数增加,其基因型分离符合1:2:1(ns表示差异不显著,jj,ij,ii分别表示sc-j纯合体,杂合体,和sc-i纯合体,包括野生型和sc-ib编辑突变型);图(c)显示基因编辑杂种的sc-j表达水平与正常杂种相比有大幅度提高。
具体实施方式
下面结合说明书附图和具体实施例对本发明作出进一步地详细阐述,所述实施例只用于解释本发明,并非用于限定本发明的范围。下述实施例中所使用的试验方法如无特殊说明,均为本领域技术人员所熟知的常规手段和使用标准的遗传学和分子生物学操作条件。
实施例1:sc的精细定位克隆和序列结构分析
本发明人的前期研究已将sc定位在水稻第3染色体上的大致区域(yang等2004.chinesesciencebulletin49,1718-1721)。本发明用如图2所示的图位克隆方法进一步克隆sc基因。根据公开的水稻基因组序列数据库的资料(http://www.ncbi.nlm.nih.gov),在该区域发展了多个分子标记(表1)。用t65(sc-j/sc-j基因型)和它的近等基因系e5(sc-i/sc-i)杂交,产生f1和f2群体,经过连锁分析8689株f2个体,把sc精细定位在粳稻一个8.6kb的区间(图2a)。根据水稻基因组序列数据库genbank(http://www.ncbi.nlm.nih.gov),在定位的粳稻8.6kb区间内有1个注释编号为os03g0247300的基因,本发明将它作为sc-j基因,其核苷酸序列如seqidno:1,编码具有seqidno:4所示的氨基酸多肽序列,含有一个duf1618保守结构域。
根据水稻基因组序列数据库genbank(http://www.ncbi.nlm.nih.gov),在籼稻品种93-11的os03g0247300等位区间有较大的结构和序列变异。但是由于93-11的该区间的序列不完全,本发明用聚合酶链式反应(pcr)技术(使用toyobokodfxdna聚合酶,按产品说明书设定条件操作)从93-11和籼稻品种明恢63以及近等基因系e5扩增出sc-i区间的基因组片段并测定其序列。结果表明,e5的os03g0247300对应基因(含有与sc-j同源的启动子序列)的编码区存在2个转座子插入,因此是一个失活的假基因(sc-is)。在sc-is下游存在3个分别为28.2和28.5kb的顺向重复序列,每个重复序列含有一个os03g0247300同源基因,但它们的启动子和上游序列(共23.9kb)与sc-j的启动子和上游序列完全不同。本发明将此重复序列的重组基因命名为sc-ia,sc-ib1和sc-ib2(图2b)。sc-ia的核苷酸序列如seqidno:2,编码具有seqidno:5所示的氨基酸多肽序列;sc-ib1和sc-ib2具有相同的核苷酸序列如seqidno:3,编码具有seqidno:6所示的氨基酸多肽序列。93-11和明恢63含有sc-is,sc-ib1和sc-ib2拷贝,但没有sc-ia拷贝。这些发现表明,籼稻的sc-i座位发生了大的结构变异和基因拷贝数变异,是1个复合基因座,而粳稻的sc-j是1个简单基因座。序列比较表明,sc-j,sc-ia,sc-ib1(sc-ib2)的编码区之间存在19个单碱基变异和3个3碱基插入缺失变异。
表1本发明所用的引物序列
实施例2:sc基因的功能验证
用rt-pcr技术从粳稻扩增sc-jcdna片段(位于编码区第514-1284bp),以反向连接到玉米ubiquitin基因启动子连接,构建成反义基因表达双元载体。以此载体转化t65,获得转化体。转基因半合体(1条染色体有转基因另1条同源染色体没有转基因)的sc-j表达水平降低(图3a),有大致一半的花粉不育(图3b)。证明sc-j是花粉配子体育性基因。
实施例3:sc杂种不育机理分析
以引物p3和p8(扩增所有sc等位基因)进行定量rt-pcr,对t65,e5,杂种f1的表达分析表明,sc-j主要在二核早期花粉特异表达,表达水平较低。sc-i重复基因在花药小孢子母细胞到二核早期花粉的不同发育阶段表达,且表达水平较高(图4a)。以sc-j特异引物(p3,p4)进行定量rt-pcr表达分析表明,f1的sc-j每个拷贝的表达水平只相当于其亲本t65的约1/5(图4),表明在杂种中sc-j的表达受到抑制而产生花粉败育。
实施例4:杂种亲和系的创建方法
为了阐明杂种中sc-j的表达抑制是否与sc-i多个拷贝的高水平表达有关,以及开发创建杂种亲和系的新方法,本发明根据sc-ib1和sc-ib1的编码区第235位的g碱基变异形成tgg即crispr/cas9基因编辑的必需元件pam,设计一个对sc-ib1/sc-ib2基因特异的crispr/cas9基因编辑靶点(5’-gcggaaggcgatcagggtag-3’),利用植物crispr/cas9基因编辑系统及其载体构建方法(ma等2015,mol.plant8,1274-1284),构建了1个crispr/cas9基因修饰载体。以此载体转化e5获得转化体,经pcr扩增含有靶点序列的片段进行测序,鉴定到sc-ib1和sc-ib2发生突变(sc-ia为野生型)的特异突变体e5-ed1和e5-ed2(图5)。
实施例5:杂种亲和系的应用
将上述实施例4获得的杂种亲和系与粳稻杂交获得f1杂种。观察表明,这些f1杂种都能不同程度恢复花粉育性(图6a)。f2基因型分离分析表明,各群体的sc-j纯合体的比例大幅度上升,其群体的分离比符合1:2:1(图6b)。以sc-j特异引物(p1,p2)进行定量rt-pcr表达分析表明,这些基因编辑亲和系组合f1杂种的sc-j表达水平比野生型f1的显著提高(图6c)。因此,本发明创建的基因编辑杂种亲和系具有亲和基因型sc-n,能够用于水稻杂交育种中克服sc杂种不育问题。
sequencelisting
<110>华南农业大学
<120>水稻杂种花粉育性基因座sc的等位基因sc-j和sc-i的分离克隆和在育种中的
应用
<130>
<160>6
<170>patentinversion3.3
<210>1
<211>3735
<212>dna
<213>sc-j基因
<400>1
aaatgggctaagtattttattcacaagtgaaacgcccgagactaatgtaaattagtgtta60
gtatagtaactttaggcttgtcgattagtgcgtgaaaagcaatagttagataaatttggg120
tttactagactaatctttgcgccacacaatcatttgcatgtcaaaactacctataaaacc180
actgtaggggttatttgaaccgattttcatacttcaaaggtagaaatctagtttcgtact240
ttataagtggttaaatagaccaaccccaacacttcagggtattccttatatctactatag300
ttttaactttgagttaaatcttaaggtggtccatgccctacggcagtagcggagccagaa360
atttaaatctgtggggtcaattactagtggtttgtatttaagtgggatcatttctcttta420
ttgttttggatttatacataaaaattgaggtttataggacgttttttaaaaagctggggt480
cggccgaccccacagaaatggcctaaatccaccactgccccaggtgccacctagttaact540
ctgcatatgggcctatgatcccttcgatcacttttcattactacaatggtgtttgaaact600
gatggatattaagctggtgcatgtagaatgaagaaaataattaacacattattgattaaa660
atttaattattaaaaacttgacaaattaatttatttgataattttaatgtaacttttata720
tagaaagtttgaaaagtgtaataacggaaactgagttaaaatctgtacgtgttaaatata780
ttattcccctttttaaatttatagaagaagttaaatatgaacgttggaaaacgctgaaca840
agtgaacatatcccgcgatccgcatttgttgcgactcgtttcgatcaactgattctcctt900
cccggccggacatccacatcatcctgcgatcgttcgtttccctagtcgttccaggctttt960
tcagctctactcccctccacccaggcgcaaatcatccgccatggctcccgcctgggtctt1020
gctcgatcgcgtcgtcaagcccgccgtcttcgacgaagaagagagcaaaggtaagggaga1080
atcgaccggcgccccagtgaagtacctgccagccagattaaggcaggaagtccccgccgg1140
catgcgggacgtgaagccctacccagaggttgcggatcctcccattatatctcgcttctc1200
catgctgatttcgcggaaggcgatcagggtagtgaaatccgtccgtgtccggtgcgccga1260
caagagccttgtcttgttctacgccggcaccggcttccccggcttctcgtcccacggctg1320
ccacctgatctacgacgccatcgacggctctctcactgcggtgcacacattcccctttcc1380
tgtgtccggagtcgtctgggtcggcagagccgctgtcctgcgccatgccggcggcggagg1440
aggaggaggagacggtactaccgcctcctatgtcatcgccgagctgctcaggccgtttca1500
tggctcccttcccgatgccacgctcgtgatgtggttgtcgaattcccccgcgtcgacctc1560
cggcagcaacggccaatgggtgaaggaggacgttcgccttcccggcgaggtgtgcacggg1620
caccgaccccttcaccaccgacttggtgttctcgttcggtgaatcgtgcctgtgctgggc1680
cgatctgttcatgggcatcctgttctgcgaccttgcgacgctgcgtgcacctcggttccg1740
tttcatcccattgcccaaggcttgctccttcgaccccgtcggcaagtatggccggcctca1800
catgcccgagttccgttccatgggccgcgtcaacggtgtcatcaggttgatcgacatgga1860
gggttttaccaacgagtacctggccgtggatgaggtgaagctgaccatctggaccctgtc1920
agccgacctcagcgagtgggagaaaggcccggtgtgtactgtaggagacatttgggccag1980
tgaggaattcgtcgccatggggttgccgcagcttagaccgatgtgccctgtcctgagcat2040
ggttgacgaagatgtcgtctgtgtagtcatgactgaagtcgagattgaggagagcgatgt2100
cacagatttcgacgatgagggcaacaagctcaagttcaaggcccagtacgttcttgacat2160
cgacgtgcggcgtaagcgggtgttatccatcacccagcaccacattgaaagtatgggtga2220
tcttataccagatctcatcgcctgtgagttcactgcatactcggagctctcaaaaggcat2280
gcaggtattactactacgacacccaacatacttagttatttactcacttgaagtatattg2340
ttctgttaacacaataacactttccacagttctaatgcttggttatcatgtccagtcagt2400
tacatcttagtatctcacgagtaacggagtaagtatattaggctagttatcctactgaac2460
tgaactctagatagcatatgtgtgtgctaatcagccatcatgattgtctaatgccaaatg2520
acttagctcttttgccatgctacagccctcctcgtccaaaccaggaacgccattttttca2580
ttctgttggtacttctaggaactggagaacgctgtcatagttactttcacttcactacaa2640
agttgtgatggcttgtccagtagatgtatccaattgaattcttgattcagaattgatata2700
gacctattttatttaaagatgttacatgtggtaaattcaaactgagcaccaagtcaccag2760
caaggttctgtaatgaaatttgtgaaaagtgcaatctatccataacgtgtgggcttggtt2820
ggttttagctattttacttaaatgcctcttagaattgaccttatcagcatgcccttgtgt2880
gagtattgctcattattattttagagagtacagacaaatggggccaatgataggtatgtt2940
gcggcgaggcaactacatcatctgaccaaacctgaccataattcttgtcgcagttgtacg3000
aaaatctcttttcttttttacttctgaagtttttttagcgttgtgtgttggaaaagagat3060
ttgtctaactaaaattctctatgtaataagtatagaatcgtatcctctataccatagtat3120
atggaaatatcatgtgtaccagtgtaaggccctaactgagttatgttactttccctagct3180
ggtactccctccatttcacattataagactttctagtattgcacacattcatatatatgt3240
taatgaatctaaacatatactccctccgttttgaaatgtttgacaccgttgactttttag3300
cacatgtttgaccattcgtcttattcaaaaacttttgtgaaatatgtaaaattatatgcc3360
tacataaaaatatatttaacaatgaatcaaatgataggaaaataattaataattacttaa3420
attttttgaataagacgaacggtcaaacatgtgctaaaaagtcaacggcgtcaaatatgt3480
gtagtagatgtcctaagactatctaggtttctcaatgatgaattttcatttatatatatt3540
tggccacatattaatttcctgtctagctctgttaattgtaactttggtatgcactgaggg3600
aattattgaagtggattaagaaccattaggccattttaaatttgtctagtttgacataca3660
ttaatttgtttctgctaggcaatggtggaaggaaatgagggtgaggaaagcacaaagcgg3720
atgaaggttaaatga3735
<210>2
<211>3730
<212>dna
<213>sc-ia基因
<400>2
gttgactagtgttgtagatccagtatagcaaaagaccaagaattaaaacaacggcttaaa60
aacagcacaaacagaatgctaaaaggttgcaaaccagtaagcaagtataatggacgacac120
attttgttcggatcatcacctctttctccttatccatcagaaaacaccgagacagcgaaa180
ggtcgacgagcccatccttggaagtggacgccaactccgtagccgccactgcagcctcgt240
gcgaggcagccgctgatgtaggcgccggcggcacttgatcgtccacgatcagcttctgcc300
tcagatctttcttctccagcgttgctgccggttccaacgacgatggaaccgccatgggag360
ctagtaaggacaccaccatcggacgcgaatcagaacagcggcagccggcgcttgacgtgc420
cgcgcccgtgagcaccgagcccgtcggcgtcgcctcggtccctgccgcggcagcggctgc480
ggctgcggcgtcgacggatcattagggtgcggaaggagtgagagagcggacagcggagga540
acaaggtgctgtctctcacccttccttccactgcacctatagccttttcatctcctgcac600
ctatgcctagtccacaagacagaggcgtcctcctctcctctctctcctctctcctctcac660
cggtgcataatgaacctagtctacaaactaggctccctttgtgtttgttgcgggtggcat720
atccaccgtccattagcctctctatctccgccgccatatccaccgtccattagtctcccg780
atccccgccactctacctttgatggggcccgtttccgatctggactcgtcttctccctct840
cgacggcacagtgggatctgggaagagggaagggtttgctcgatccctttgttcttgccg900
tctcttgcactgatctagctaattctttctgctggggcactcgaaaaaaaactcttcgac960
tgctccccgtactccaaacccacccacgcgcaaatccgcgatggctcccacctgggtctt1020
gctcgatcgcttcgtcaagcccactatcttcgacgaagaagagagcaaaggtaagggaga1080
atcgaccggcgccccagtgaagtaccttccagccagattaaggcaggaagtccccgccgg1140
catgcgggacgtgaagccctacccagaggttgcggatcctcccattgtatctcgcttctc1200
catgctgatttcgcggaaggcgatcagggtagtgaaatccgtccgtgtcgagtgcgccga1260
caagagccttgtcttgttctacgccggcaccggcttccccggcttctcccacggctgcca1320
cctgatctacgacgccatcgacggttctctcactgcagtgcacacgttcccctttcctgt1380
gtccggagtcgtctgggtcggcacagccgctgtcctgcgccatgccggcggcggcggagg1440
aggagacggtactaccgcctatgtcatcgccgagctgctcaggccgttccatggctccct1500
tcccgatgccacgctcgtgatgtggttgtcgaattcccccgcgtcgacctccggcagcaa1560
tggccaatgggtgaaggaggacgttcgccttcccggcgaggtgtgcacgggcaccgaccc1620
cttcaccaccgacttggtgttctcgttcggtgaatcgtgcctgtgctgggccgatctgtt1680
catgggcatcctgttctgcgaccttgcgacgctgcgtgcacctcggttccgtttcatccc1740
attgcccaaggcttgctccttcgaccccgtcggcaagtatggccggcctcacatgcccga1800
gttccgttccatgggccgcgtcaacggtgtcatcaggttgatcgacatggagggttttac1860
caacgagtacctggccgtggatgaggtgaagctgaccatctggaccctgtcagccgacct1920
cagcgagtgggagaaaggcccggtgtgtactgtaggagacatttgggccagtgaggaatt1980
cgtcgccatggggttgccgcagcttagaccgatgtgccctatcctgagcatggttgacga2040
agatgtcgtctgtgtagtcatgactgaagtcgagattgaggagagcgatgtcacagattt2100
cgacgatgagggcaacaagctcaagttcaaggcccagtacgttcttgacatcgacgtgcg2160
gcgtaagcgggtgttatccatcacccagcaccacattgaaagtatgggtgatcttatacc2220
agatctcatcgcctgtgagttcactgcatactcggagctctcaaaaggcatgcaggtatt2280
actactacgacacccaacatacttagttatttactcacttgaagtatatatattgttctg2340
ttaacacaataacactttccacagttctaatgcttggttatcatgtccagtcagttacat2400
cttagtatctcacgagtaacggagtaagtatattaggctagttatcctactgaactgaac2460
tctagatagcatatgtgtgtgctaatcagccatcatgattgtctaatgccaaatgactta2520
gctcttttgccatgctacagccctcctcgtccaaaccaggaacgccatttttgcattctg2580
ttggtacttctaggaactggagaacgctgtcatagttactttcacttcactacaaagttg2640
tgatggcttgtccagtagatgtatccaattgaattcttgattcagaattgatatagacct2700
attttatttaaagatgttacatgtggtaaattcaaactgagcaccaagtcaccagcaagg2760
ttctgtaatgaaatttgtgaaaagtgcaatctatccataacgtgtgggcttggttgattt2820
tagctattttacttaaatgcctcttagaattgaccttatcagcatgcccttgtgtgagta2880
ttgctcattattattttagagagtacagacaaatggggccaatgataggtatgttgcggc2940
gaggcaactacatcatctgaccaaacctgaccataattcttgtcgcagttgtacgaaaat3000
ctcttttcttttttacttctgaagtttttttagcgttgtgtgttggaaaagagatttgtc3060
taactaaaattctctatgtaataagtatagaatcgtatcctctataccatagtatatgga3120
aatatcatgtgtaacagtgtaaggccctaactgagttatgttactttccctagctggtac3180
tccctccatttcatattataagactttctattattgcacacattcatatatatgttaatg3240
aatctaaacatatactccctccgttttgaaatgtttgacaccgttgactttttagcacat3300
gtttgatcattcgtcttattcaaaaacttttgtgaaatatgtaaaattatatgcctacat3360
aaaaatatatttaacaatgaatcaaatgataggaaaataattaataattacttaaatttt3420
ttgaataagacgaacggtcaaacatgtgctaaaaagtcaacggcgtcaaatatgtgtagt3480
agatgtcctaagactatctaggtttctcaatgatgaattttcatttatatatatttggcc3540
acatattaatttcctgtctagctctgttaattgtaactttggtatgcactgagggaatta3600
ttgaagtggattaagaaccattaggccattttaaatttgtctagtttgacatacattaat3660
ttgtttctgctaggcaatggtggaaggaaatgagggtgaggaaagcacaaagcggatgaa3720
ggttaaatga3730
<210>3
<211>3726
<212>dna
<213>sc-ib1或sc-ib2基因
<400>3
gttgactagtgttgtagatccagtatagcaaaagaccaagaattaaaacaacggcttaaa60
aacagcacaaacagaatgctaaaaggttgcaaaccagtaagcaagtataatggacgacac120
attttgttcggatcatcacctctttctccttatccatcagaaaacaccgagacagcgaaa180
ggtcgacgagcccatccttggaagtggacgccaactccgtagccgccactgcagcctcgt240
gcgaggcagccgctgatgtaggcgccggcggcacttgatcgtccacgatcagcttctgcc300
tcagatctttcttctccagcgttgctgccggttccaacgacgatggaaccgccatgggag360
ctagtaaggacaccaccatcggacgcgaatcagaacagcggcagccggcgcttgacgtgc420
cgcgcccgtgagcaccgagcccgtcggcgtcgcctcggtccctgccgcggcagcggctgc480
ggctgcggcgtcgacggatcattagggtgcggaaggagtgagagagcggacagcggagga540
acaaggtgctgtctctcacccttccttccactgcacctatagccttttcatctcctgcac600
ctatgcctagtccacaagacagaggcgtcctcctctcctctctctcctctctcctctcac660
cggtgcataatgaacctagtctacaaactaggctccctttgtgtttgttgcgggtggcat720
atccaccgtccattagcctctctatctccgccgccatatccaccgtccattagtctcccg780
atccccgccactctacctttgatggggcccgtttccgatctggactcgtcttctccctct840
cgacggcacagtgggatctgggaagagggaagggtttgctcgatccctttgttcttgccg900
tctcttgcactgatctagctaattctttctgctggggcactcgaaaaaaaactcttcgac960
tgctccccgtactccaaacccacccacgcgcaaatccgcgatggctcccacctgggtctt1020
gctcgatcgcttcgtcaagcccactatcttcgacgaagaagagagcaaaggtaagggaga1080
atcgaccggcgccccagtgaagtaccttccagccagattaaggcaggaagtccccgccgg1140
catgcgggacgtgaagccctacccagaggttgcggatcctcccattgtatctcgcttctc1200
catgctgatttcgcggaaggcgatcagggtagtggaatccatccatgtcgagtgcgccga1260
caagagccttgtcttgttctacgccggcaccggcttccccggcttctcccacggctgcca1320
cctgatctacgacgccatcgacggttctctcactgcagtgcacacgttcccctttcctgt1380
gtccggagtcgtctgggtcggcacagccgctgtcctgcgccatgccggcggcggcggagg1440
aggagacggtactaccgcctatgtcatcgccgagctgctcaggccgttccatggctccct1500
tcccgatgccacgctcgtgatgtggttgtcgaattcccccgcgtcgacctccggcagcaa1560
tggccaatgggtgaaggaggacgttcgccttcccggcgaggtgtgcacgggcaccgaccc1620
cttcaccaccgacttggtgttctcgttcggtgaatcgtgcctgtgctgggccgatctgtt1680
catgggcatcctgttctgcgaccttgcgacgctgcgtgcacctcggttccgtttcatccc1740
attgcccaaggcttgctccttcgaccccgtcggcaagtatggccggcctcacatgcccga1800
gttccgttccatgggccgcgtcaacggtgtcatcaggttgatcgacatggagggttttac1860
caacgagtacctggccgtggatgaggtgaagctgaccatctggaccctgtcagccgacct1920
cagcgagtgggagaaaggcccggtgtgtactgtaggagacatttgggccagtgaggaatt1980
cgtcgccatggggttgccgcagcttagaccgatgtgccctgtcctgagcatggttgacga2040
agatgtcgtctgtgtagtcatgactgaagtcgagattgaggagagcgatgtcacagattt2100
cgacgatgagggcaacaagctcaagttcaaggcccagtatgttcttgacatcgacgtgcg2160
gcgtaagcgggtgttatccatcacccagcaccacattgaaagtatgggtgatcttatacc2220
agatctcatcgcctgtgagttcactgcatacttggagctctcaaaaggcatgcaggtatt2280
actactacgacacccaacatacttagttatttactcacttgaagtatattgttctgttaa2340
cacaataacactttccacagttctaatgcttggttatcatgtccagtcagttacatctta2400
gtatctcacgagtaacggagtaagtatattaggctagttatcctactgaactgaactcta2460
gatagcatatgtgtgtgctaatcagccatcatgattgtctaatgccaaatgacttagctc2520
ttttgccatgctacagccctcctcgtccaaaccaggaacgccatttttgcattctgttgg2580
tacttctaggaactggagaacgctgtcatagttactttcacttcactacaaagttgtgat2640
ggcttgtccagtagatgtatccaattgaattcttgattcagaattgatatagacctattt2700
tatttaaagatgttacatgtggtaaattcaaactgagcaccaagtcaccagcaaggttct2760
gtaatgaaatttgtgaaaagtgcaatctatccataacgtgtgggcttggttgattttagc2820
tattttacttaaatgcctcttagaattgaccttatcagcatgcccttgtgtgagtattgc2880
tcattattattttagagagtacagacaaatggggccaatgataggtatgttgcggcgagg2940
caactacatcatctgaccaaacctgaccataattcttgtcacagttgtacgaaaatctct3000
tttcttttttacttctgaagtttttttagcgttgtgtgttggaaaagagatttgtctaac3060
taaaattctctatgtaataagtatagaatcgtatcctctataccatagtatatggaaata3120
tcatgtgtaacggtgtaaggccctaactgagttatgttacttttcctagctggtactccc3180
tccatttcacattataagactttctagtattgcacacattcatatatatgttaatgaatc3240
taaacatatactccctccgttttgaaatgtttgacaccgttgactttttagcacatgttt3300
gatcattcgtcttattcaaaaacttttgtgaaatatgtaaaattatatgcctacataaaa3360
atatatttaacaatgaatcaaatgataggaaaataattaataattacttaaattttttga3420
ataagacgaacggtcaaacatgtgctaaaaagtcaacggcgtcaaatatgtgtagtagat3480
gtcctaagactatctaggtttctcaatgatgaattttcatttatatatatttggccacat3540
attaatttcctgtctagctctgttaattgtaactttggtatgcactgagggaattattga3600
agtggattaagaaccattaggccattttaaatttgtctagtttgacatacattaatttgt3660
ttctgctaggcaatggtggaaggaaatgagggtgaggaaagcacaaagcggatgaaggtt3720
aaatga3726
<210>4
<211>446
<212>prt
<213>sc-j蛋白
<400>4
metalaproalatrpvalleuleuaspargvalvallysproalaval
151015
pheaspgluglugluserlysglylysglygluserthrglyalapro
202530
vallystyrleuproalaargleuargglngluvalproalaglymet
354045
argaspvallysprotyrprogluvalalaaspproproileileser
505560
argphesermetleuileserarglysalaileargvalvallysser
65707580
valargvalargcysalaasplysserleuvalleuphetyralagly
859095
thrglypheproglypheserserhisglycyshisleuiletyrasp
100105110
alaileaspglyserleuthralavalhisthrphepropheproval
115120125
serglyvalvaltrpvalglyargalaalavalleuarghisalagly
130135140
glyglyglyglyglyglyaspglythrthralasertyrvalileala
145150155160
gluleuleuargprophehisglyserleuproaspalathrleuval
165170175
mettrpleuserasnserproalaserthrserglyserasnglygln
180185190
trpvallysgluaspvalargleuproglygluvalcysthrglythr
195200205
aspprophethrthraspleuvalpheserpheglyglusercysleu
210215220
cystrpalaaspleuphemetglyileleuphecysaspleualathr
225230235240
leuargalaproargpheargpheileproleuprolysalacysser
245250255
pheaspprovalglylystyrglyargprohismetprogluphearg
260265270
sermetglyargvalasnglyvalileargleuileaspmetglugly
275280285
phethrasnglutyrleualavalaspgluvallysleuthriletrp
290295300
thrleuseralaaspleuserglutrpglulysglyprovalcysthr
305310315320
valglyaspiletrpalaserglugluphevalalametglyleupro
325330335
glnleuargprometcysprovalleusermetvalaspgluaspval
340345350
valcysvalvalmetthrgluvalgluileglugluseraspvalthr
355360365
asppheaspaspgluglyasnlysleulysphelysalaglntyrval
370375380
leuaspileaspvalargarglysargvalleuserilethrglnhis
385390395400
hisileglusermetglyaspleuileproaspleuilealacysglu
405410415
phethralatyrsergluleuserlysglymetglnalametvalglu
420425430
glyasngluglyglugluserthrlysargmetlysvallys
435440445
<210>5
<211>443
<212>prt
<213>sc-ia蛋白
<400>5
metalaprothrtrpvalleuleuaspargphevallysprothrile
151015
pheaspgluglugluserlysglylysglygluserthrglyalapro
202530
vallystyrleuproalaargleuargglngluvalproalaglymet
354045
argaspvallysprotyrprogluvalalaaspproproilevalser
505560
argphesermetleuileserarglysalaileargvalvallysser
65707580
valargvalglucysalaasplysserleuvalleuphetyralagly
859095
thrglypheproglypheserhisglycyshisleuiletyraspala
100105110
ileaspglyserleuthralavalhisthrphepropheprovalser
115120125
glyvalvaltrpvalglythralaalavalleuarghisalaglygly
130135140
glyglyglyglyaspglythrthralatyrvalilealagluleuleu
145150155160
argprophehisglyserleuproaspalathrleuvalmettrpleu
165170175
serasnserproalaserthrserglyserasnglyglntrpvallys
180185190
gluaspvalargleuproglygluvalcysthrglythraspprophe
195200205
thrthraspleuvalpheserpheglyglusercysleucystrpala
210215220
aspleuphemetglyileleuphecysaspleualathrleuargala
225230235240
proargpheargpheileproleuprolysalacysserpheasppro
245250255
valglylystyrglyargprohismetproglupheargsermetgly
260265270
argvalasnglyvalileargleuileaspmetgluglyphethrasn
275280285
glutyrleualavalaspgluvallysleuthriletrpthrleuser
290295300
alaaspleuserglutrpglulysglyprovalcysthrvalglyasp
305310315320
iletrpalaserglugluphevalalametglyleuproglnleuarg
325330335
prometcysproileleusermetvalaspgluaspvalvalcysval
340345350
valmetthrgluvalgluileglugluseraspvalthrasppheasp
355360365
aspgluglyasnlysleulysphelysalaglntyrvalleuaspile
370375380
aspvalargarglysargvalleuserilethrglnhishisileglu
385390395400
sermetglyaspleuileproaspleuilealacysgluphethrala
405410415
tyrsergluleuserlysglymetglnalametvalgluglyasnglu
420425430
glyglugluserthrlysargmetlysvallys
435440
<210>6
<211>443
<212>prt
<213>sc-ib1或sc-ib2蛋白
<400>6
metalaprothrtrpvalleuleuaspargphevallysprothrile
151015
pheaspgluglugluserlysglylysglygluserthrglyalapro
202530
vallystyrleuproalaargleuargglngluvalproalaglymet
354045
argaspvallysprotyrprogluvalalaaspproproilevalser
505560
argphesermetleuileserarglysalaileargvalvalgluser
65707580
ilehisvalglucysalaasplysserleuvalleuphetyralagly
859095
thrglypheproglypheserhisglycyshisleuiletyraspala
100105110
ileaspglyserleuthralavalhisthrphepropheprovalser
115120125
glyvalvaltrpvalglythralaalavalleuarghisalaglygly
130135140
glyglyglyglyaspglythrthralatyrvalilealagluleuleu
145150155160
argprophehisglyserleuproaspalathrleuvalmettrpleu
165170175
serasnserproalaserthrserglyserasnglyglntrpvallys
180185190
gluaspvalargleuproglygluvalcysthrglythraspprophe
195200205
thrthraspleuvalpheserpheglyglusercysleucystrpala
210215220
aspleuphemetglyileleuphecysaspleualathrleuargala
225230235240
proargpheargpheileproleuprolysalacysserpheasppro
245250255
valglylystyrglyargprohismetproglupheargsermetgly
260265270
argvalasnglyvalileargleuileaspmetgluglyphethrasn
275280285
glutyrleualavalaspgluvallysleuthriletrpthrleuser
290295300
alaaspleuserglutrpglulysglyprovalcysthrvalglyasp
305310315320
iletrpalaserglugluphevalalametglyleuproglnleuarg
325330335
prometcysprovalleusermetvalaspgluaspvalvalcysval
340345350
valmetthrgluvalgluileglugluseraspvalthrasppheasp
355360365
aspgluglyasnlysleulysphelysalaglntyrvalleuaspile
370375380
aspvalargarglysargvalleuserilethrglnhishisileglu
385390395400
sermetglyaspleuileproaspleuilealacysgluphethrala
405410415
tyrleugluleuserlysglymetglnalametvalgluglyasnglu
420425430
glyglugluserthrlysargmetlysvallys
435440
- 还没有人留言评论。精彩留言会获得点赞!